Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Reactivación de modelos celulares desmembranados en Chlamydomonas reinhardtii
En este artículo
Resumen
La reactivación in vitro de las células móviles es un experimento crucial para comprender los mecanismos de la motilidad celular. El protocolo describe la reactivación de los modelos de células desmembranadas de Chlamydomonas reinhardtii, un organismo modelo para estudiar los cilios/flagelos.
Resumen
Desde el experimento histórico sobre la contracción del músculo glicerinado mediante la adición de ATP, que Szent-Györgyi demostró a mediados del siglo 20, la reactivación in vitro de las células desmembranadas ha sido una forma tradicional y potente de examinar la motilidad celular. La ventaja fundamental de este método experimental es que la composición de la solución de reactivación puede cambiarse fácilmente. Por ejemplo, un entorno de alta concentraciónde Ca 2+ que ocurre solo temporalmente debido a la excitación de la membrana in vivo se puede replicar en el laboratorio. Los cilios eucariotas (también conocidos como flagelos) son elaboradas máquinas de motilidad cuyos mecanismos reguladores aún no se han aclarado. El alga verde unicelular Chlamydomonas reinhardtii es un excelente organismo modelo en el campo de investigación de los cilios. Los experimentos de reactivación utilizando modelos celulares demembranados de C. reinhardtii y sus derivados, como los axonemas demembranados de cilios aislados, han contribuido significativamente a comprender los mecanismos moleculares de la motilidad ciliar. Esos experimentos aclararon que el ATP energiza la motilidad ciliar y que varias señales celulares, incluyendo Ca2+, cAMP y especies reactivas de oxígeno, modulan los movimientos ciliares. El método preciso para la demembranación de las células de C. reinhardtii y la reactivación de los modelos celulares se describe aquí.
Introducción
La reactivación in vitro de células móviles desmembranadas es una herramienta valiosa para estudiar las bases moleculares del mecanismo regulador de la motilidad celular. Szent-Györgyi demostró por primera vez la contracción in vitro de las fibras musculares esqueléticas de conejo extraídas con 50% de glicerol mediante la adición de trifosfato de adenosina (ATP)1. Este experimento fue el primero en demostrar que el ATP energiza la contracción muscular. La metodología pronto se aplicó al estudio de la motilidad ciliar/flagelar energizada por ATP, como los flagelos 2 de los espermatozoides, los ciliosde Paramecium 3 y los cilios de Chlamydomonas reinhardtii (también llamados flagelos)4 utilizando detergentes no iónicos para la demembranación.
El alga verde unicelular C. reinhardtii es un organismo modelo para el estudio de los cilios: nada con dos cilios golpeándolos como la braza de un humano5. El movimiento ciliar es impulsado por la dineína, una proteína motora basada en microtúbulos dirigidos de extremo negativo 6,7. Las dineínas ciliares se pueden clasificar en dineínas del brazo externo y dineínas del brazo interno. Los mutantes que carecen de cada tipo de dineína se han aislado como mutantes de natación lenta con diferentes anomalías de motilidad. El análisis detallado de la motilidad in vitro de estos mutantes ha avanzado significativamente en la investigación de la dineína8.
Se han logrado muchos hallazgos importantes utilizando este método y sus derivados desde que se estableció el experimento de reactivación in vitro de células C. reinhardtii desmembranadas (modelos celulares). La reactivación de modelos celulares en una serie de tampones de Ca2+, por ejemplo, mostró9 que dos cilios están regulados de manera diferente por Ca2+ submicromolar, y este control asimétrico de los cilios permite la orientación fototáctica de C. reinhardtii10. Además, ambos cilios muestran la conversión de la forma de onda del modo de natación hacia adelante (llamado forma de onda asimétrica) al modo de natación hacia atrás (llamado forma de onda simétrica que aparece durante un corto período cuando las células están foto-o-mecano-chocadas)11,12. Esta conversión de forma de onda está regulada por Ca2+ submilimolar, que se demostró mediante la reactivación del llamado aparato nucleoflagelar (un complejo que contiene dos cilios, los cuerpos basales, las estructuras que unen los cuerpos basales con el núcleo y el remanente del núcleo)11 o axonemas demembranados de cilios aislados13. Aparte de Ca2+, el aplomo redox (reducción-oxidación) es una señal que regula la frecuencia de latido ciliar, que se demostró mediante la reactivación de modelos celulares en tampones redox que contienen diferentes proporciones de glutatión reducido vs. glutatión oxidado14. Además, el monofosfato de adenosina cíclico (cAMP) regula asimétricamente dos cilios, lo que se demostró mediante la reactivación de axonemas con cAMP15 enjaulado fotocleavable. Estos hallazgos in vitro, combinados con hallazgos genéticos, han llevado a una comprensión más profunda de los mecanismos moleculares de la regulación de los cilios en C. reinhardtii.
Aquí se describe un protocolo para reactivar los modelos celulares. El método es simple, permite varias modificaciones y se puede aplicar a múltiples organismos que se mueven con cilios. Sin embargo, debido a que las células desmembranadas son frágiles, se requieren algunos consejos para reactivar la motilidad de los modelos celulares con buena eficiencia al tiempo que se evita la deciliación.
Protocolo
Para el presente estudio se utilizó una cepa de tipo salvaje de Chlamydomonas reinhardtii, CC-125. CC-125 se obtuvo del Centro de Recursos de Chlamydomonas (ver Tabla de Materiales) y se mantuvo en un medio de Tris-acetato-fosfato (TAP)16, 1.5% agarosa a 20-25 °C.
1. Cultivo celular
- Cultivo de Chlamydomonas reinhardtii (CC-125) en el medio TAP16 en un período de luz-oscuridad de 12 h/12 h (condiciones de luz para el período de luz: ~50 fotones μmol m−2 s−1 de luz blanca) a 20-25 °C durante 2 días (Figura 1).
NOTA: Las células deben estar en una fase de crecimiento logarítmico medio (Película 1). El cultivo largo (>4 días, en el crecimiento logarítmico tardío o en la fase estacionaria) disminuye la eficiencia de reactivación de los modelos de células desmembranadas.

Figura 1: Cultivo líquido después de 2 días de cultivo. A partir de una placa de agar TAP-1.5%, un trozo de células de tipo salvaje para llenar el bucle de platino se inoculó a ~ 150 ml de medio líquido TAP en un matraz. La densidad celular después del cultivo de 2 días fue de 2,3 × 106 células/ml. Haga clic aquí para ver una versión más grande de esta figura.
Película 1: Natación de células vivas. Las células se observaron bajo un microscopio con una lente de objetivo 10x y un condensador de campo oscuro de inmersión en aceite. Barra de escala = 100 μm. Haga clic aquí para descargar esta película.
2. Preparación de modelos de células desmembranadas
NOTA: Antes de comenzar el experimento, mantenga el tampón de lavado a temperatura ambiente y los tampones de demembranación, dilución, reactivación y la solución de ATP en hielo. La composición de estos colchones figura en el cuadro complementario 1.
- Centrifugación ~ 10 ml de cultivo líquido a 1000 × g a 20 °C durante 3 min.
NOTA: En todo el protocolo de este paso, no utilice artículos de plástico en autoclave (tubos, puntas de pipeta, etc.), disminuyendo la eficiencia de reactivación. Para los tubos cónicos, los reutilizados son preferibles. La densidad celular después de un cultivo de 2 días puede ser típicamente de 1.0-5.0 × 106 células/ ml. Cuando la densidad celular es menor que esto, tome suficiente volumen de cultivo para contener ~ 5 × 107 células. - Deseche el sobrenadante, primero por decantación y luego por una pipeta Pasteur, y vuelva a suspender los precipitados en ~ 5 ml de tampón de lavado.
- Células centrífugas en el tampón de lavado a 1000 × g durante 3 min a 20 °C.
- Deseche el sobrenadante cuidadosamente con una pipeta (Figura 2).
NOTA: Las pipetas Pasteur son preferibles. Cuando use micropipetas, evite usar puntas de pipeta que hayan sido esterilizadas en autoclave. - Superponga ~ 0.5 ml de tampón de demembranación en un gránulo celular, agite el tubo suavemente con la mano para suspender aproximadamente las células en el tampón y coloque el tubo en hielo (Figura 3A).
NOTA: No es necesario suspender completamente el pellet en este momento. Elevar la concentración de MgSO4 a 15 mM en los tampones de demembranación y reactivación cuando se reactivan los modelos celulares, con una concentración final de ATP >1 mM para una reactivación estable17. - Suspenda suavemente la gránula de celda restante con una pipeta y vuelva a colocar el tubo sobre hielo (Figura 3B).
- Tome 5-10 μL de los modelos celulares, diluya 10 veces con el tampón de dilución y observe bajo el microscopio (Película 2) para confirmar que todos los modelos celulares están desmembranados y no nadan (Figura 4).
NOTA: Si algunas células todavía están nadando (vivas), agregue el detergente no iónico utilizado en el tampón de demembranación directamente a la solución del modelo celular a la concentración final de ~ 0.15%. Alternativamente, repita los pasos 2.1-2.5 con la solución de demembranación que contiene 0.15% de detergente.
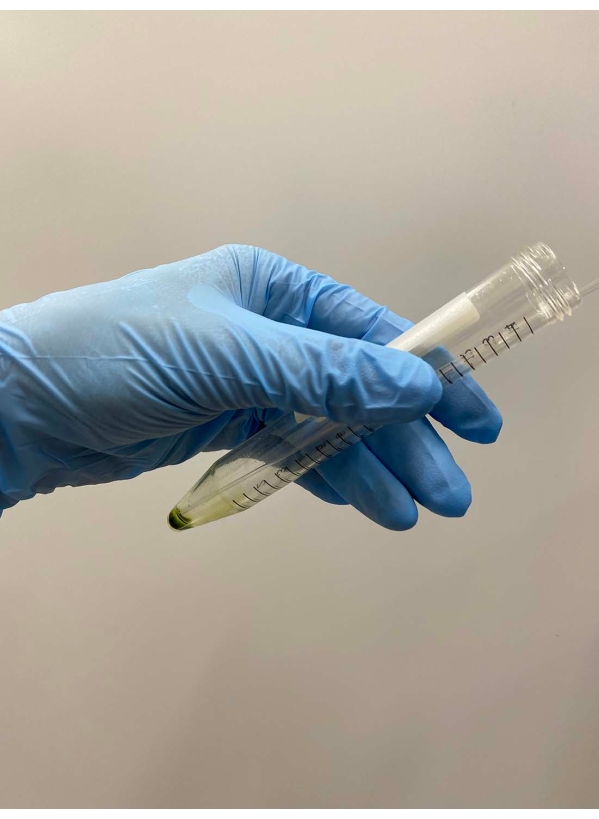
Figura 2: Descartando el sobrenadante. El resto del sobrenadante se retiró cuidadosamente con una pipeta Pasteur después de que el sobrenadante se eliminó por decantación del tubo. Haga clic aquí para ver una versión más grande de esta figura.
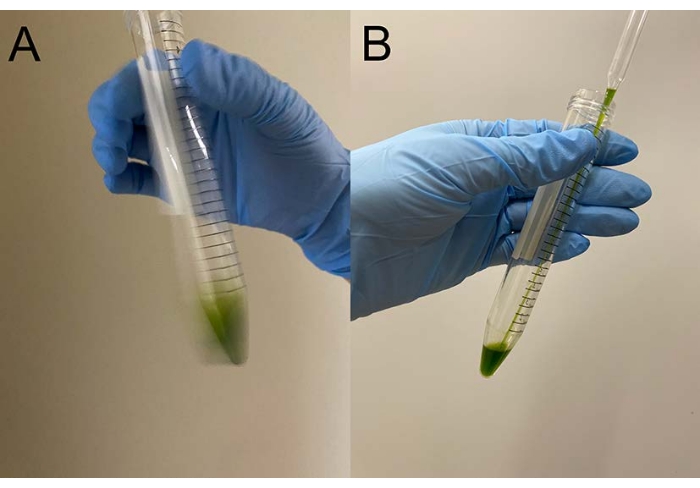
Figura 3: Demembranación. (A) Después de superponer 0,5 ml de solución de demembranación sobre el pellet celular, la solución se mezcló con una mano para suspender las células aproximadamente. (B) Después de la mezcla, el resto del pellet celular quedó completamente suspendido en la solución por una pipeta Pasteur. Haga clic aquí para ver una versión más grande de esta figura.
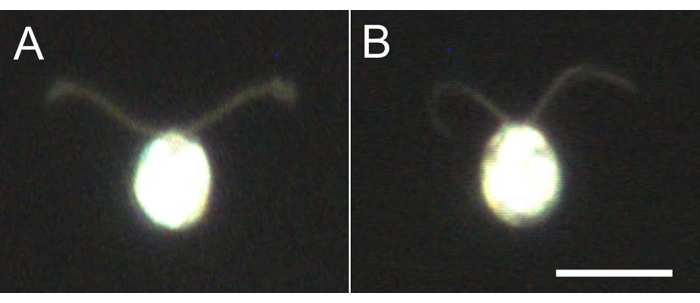
Figura 4: Efectos de la demembranación. (A) Una célula viva pegada en un portaobjetos de vidrio. (B) Un modelo de celda pegado en un portaobjetos de vidrio. Tenga en cuenta que en el modelo celular, los cilios se volvieron ligeramente más delgados. Las imágenes se observaron bajo un microscopio con una lente de objetivo 20x y un condensador de campo oscuro de inmersión en aceite. Barra de escala = 10 μm. Haga clic aquí para ver una versión más grande de esta figura.
Película 2: Confirmación de la demembranación. Se observó la suspensión del modelo celular bajo un microscopio con una lente de objetivo 10x y un condensador de campo oscuro de inmersión en aceite. Ninguna célula estaba nadando. Barra de escala = 100 μm. Haga clic aquí para descargar esta película.
3. Reactivación de modelos de células desmembranadas
- Mezcle 80 μL de solución de reactivación, 10 μL de solución de ATP y 10 μL de modelos celulares en un tubo de 0,5 ml tocando el tubo (Figura 5).
NOTA: La concentración final de ATP debe ser de <3 mM porque la frecuencia de latido ciliar, un parámetro para el movimiento ciliar, aumenta con la concentración de ATP y se satura a 2-3 mM de ATP17.
PRECAUCIÓN: La mezcla por pipeteo o vórtice causa deciliación y disminuye la eficiencia de reactivación.- Para reactivar con <0,2 mM de ATP, agregue un sistema de regeneración de ATP, como 70 U/mL de creatina quinasa y 5 mM de fosfato de creatina (ver Tabla de Materiales).
NOTA: La solución de reactivación se prepara a una concentración más alta (1.125x, Tabla suplementaria 1) que la solución de dilución para que después de mezclar el ATP disuelto en agua, el contenido alcance las siguientes concentraciones finales: 30 mM de Hepes (pH 7.4), 5 mM de MgSO4, 1 mM de ditiotreitol (TDT), 1 mM de EGTA y 50 mM de acetato de potasio (ver Tabla de Materiales).
- Para reactivar con <0,2 mM de ATP, agregue un sistema de regeneración de ATP, como 70 U/mL de creatina quinasa y 5 mM de fosfato de creatina (ver Tabla de Materiales).
- Coloque ~ 30 μL de la solución mezclada en un portaobjetos de vidrio y coloque suavemente una cubierta sobre él con espaciadores para evitar choques mecánicos en los modelos de celdas (Figura 6).
NOTA: Se pueden usar cintas adhesivas de petróleo blanco o de doble cara para hacer los espaciadores. - Observe los modelos de células reactivadas bajo un microscopio (Película 3).

Figura 5: Solución de mezcla tocando el tubo. (A) A 80 μL de la solución de reactivación, se agregaron 10 μL de solución de ATP y 10 μL de modelos celulares en orden secuencial. (B) La solución se mezcló golpeando el tubo con un dedo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Hacer espaciadores en los bordes de las cubiertas. (A) Se aplicó una fina capa de petróleo blanco al dorso de una mano. (B) Se raspó una pequeña cantidad de petróleo blanco con un borde de una cubierta. (C) Se hizo un espaciador en el borde de un cubrehojas. (D) Se hizo otro espaciador en el borde opuesto. Haga clic aquí para ver una versión más grande de esta figura.
Película 3: Natación de modelos celulares reactivados. La motilidad de los modelos celulares se reactivó mediante la adición de ATP a una concentración final de 1 mM, y se observó bajo un microscopio con una lente de objetivo 10x y un condensador de campo oscuro de inmersión en aceite. Barra de escala = 100 μm. Haga clic aquí para descargar esta película.
Resultados
El proceso de demembranación y reactivación en la cepa de tipo salvaje C. reinhardtii (CC-125) se muestra aquí. El cultivo 2 días después de la inoculación se convirtió en un color verde claro (paso 1.1) (Figura 1). Las células se recogieron (paso 2.1), se lavaron (paso 2.2) y se desmembranaron (paso 2.5). Después de la demembranación, todos los modelos celulares se volvieron inmóviles (paso 2.7). Los cilios desmembranados (llamados axonemas) permanecen adheridos al cuer...
Discusión
Hay dos pasos críticos en este protocolo. El primero es un proceso conocido como demembranación, que debe llevarse a cabo de manera suave pero exhaustiva. La deciliación (es decir, la separación de los cilios del cuerpo celular) es inducida por pipeteos vigorosos o vórtices, lo que hace que los modelos celulares queden inmóviles incluso después de la adición de ATP. Típicamente, 5 × 107 células están suspendidas en ~0.5 mL de tampón de demembranación (densidad celular final: 1 × 108 c...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este estudio fue apoyado por subvenciones de la Sociedad Japonesa para la Promoción de la Ciencia KAKENHI (https://www.jsps.go.jp/english/index.html) a N.U. (19K23758, 21K06295) y K.W. (19H03242, 20K21420, 21H00420), de la Ohsumi Frontier Science Foundation (https://www.ofsf.or.jp/en/) a K.W., y de la Alianza Dinámica para la Innovación Abierta Uniendo Humanos, Medio Ambiente y Materiales (http://alliance.tagen.tohoku.ac.jp/english/) a N.U. y K.W. Agradecemos a la Sra. Miyuki Shinohara (Hosei Univ.) por su ayuda en la preparación de las cifras.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.5 mL plastic tube | QSP | 502-PLN-Q | |
| 15 mL conical tube | SARSTEDT | 62.554.502 | |
| Adenosine 5'-triphosphate disodium salt hydrate (ATP) | Sima-Aldrich | A2383 | |
| Centrifuge | KUBOTA | 2800 | |
| Chlamydomonas strain CC-125 | Chlamydomonas Resource Center | https://www.chlamycollection.org/ | |
| Creatine kinase | Merck | CK-RO | |
| Creatine phosphate | Merck | CRPHO-RO | |
| Dithiothreitol (DTT) | Nakalai tesque | 14128-46 | |
| GEDTA(EGTA) | Dojindo | G002 | |
| Hepes | Dojindo | GB70 | |
| Igepal CA-630 | Sigma-Aldrich | I8896 | IUPAC name is octylphenoxypolyethoxyethanol: IGEPAL CA-630 is a substitute for Nonidet P-40 (NP-40); NP-40 is no longer available in Sigma-Aldrich. |
| MgSO4-7H2O | Nakalai tesque | 21002-85 | |
| Microscope | Olympus | BX-53 | |
| Pasteur pipette | fisher scientific | 13-678-20C | |
| Polyethylene glycol, Mr 20,000 | Merck | 8.18897.1000 | |
| Pottasium acetate | Nakalai tesque | 28434-25 | |
| Sodium Hydroxide | Nacalai | 31511-05 | |
| Sucrose | FUJIFILM Wako Pure Chemical Corporation | 196-00015 |
Referencias
- Szent-Gyorgyi, A. Free-energy relations and contraction of actomyosin. Biological Bulletin. 96 (2), 140-161 (1949).
- Hoffman-Berling, H. Adenosintriphosphat als betriebsstoff von zellbewegungen. Biochimica et Biophysica Acta. 14, 182-194 (1954).
- Naitoh, Y., Kaneko, H. Reactivated Triton-extracted models of Paramecium: modification of ciliary movement by calcium ions. Science. 176 (4034), 523-524 (1972).
- Witman, G. B., Plummer, J., Sander, G. Chlamydomonas flagellar mutants lacking radial spokes and central tubules. Structure, composition, and function of specific axonemal components. Journal of Cell Biology. 76 (3), 729-747 (1978).
- Rüffer, U., Nultsch, W. Flagellar coordination in Chlamydomonas cells held on micropipettes. Cell Motility and the Cytoskeleton. 41 (4), 297-307 (1998).
- Sale, W. S., Satir, P. Direction of active sliding of microtubules in Tetrahymena cilia. Proceedings of the National Academy of Sciences of the United States of America. 74 (5), 2045-2049 (1977).
- Fox, L. A., Sale, W. S. Direction of force generated by the inner row of dynein arms on flagellar microtubules. Journal of Cell Biology. 105 (4), 1781-1787 (1987).
- Kamiya, R., Yagi, T. Functional diversity of axonemal dyneins as assessed by in vitro and in vivo motility assays of Chlamydomonas mutants. Zoolog Science. 31 (10), 633-644 (2014).
- Kamiya, R., Witman, G. B. Submicromolar levels of calcium control the balance of beating between the two flagella in demembranated models of Chlamydomonas. Journal of Cell Biology. 98 (1), 97-107 (1984).
- Okita, N., Isogai, N., Hirono, M., Kamiya, R., Yoshimura, K. Phototactic activity in Chlamydomonas 'non-phototactic' mutants deficient in Ca2+-dependent control of flagellar dominance or in inner-arm dynein. Journal of Cell Science. 118, 529-537 (2005).
- Hyams, J. S., Borisy, G. G. Isolated flagellar apparatus of Chlamydomonas: characterization of forward swimming and alteration of waveform and reversal of motion by calcium ions in vitro. Journal of Cell Science. 33, 235-253 (1978).
- Fujiu, K., Nakayama, Y., Yanagisawa, A., Sokabe, M., Yoshimura, K. Chlamydomonas CAV2 encodes a voltage- dependent calcium channel required for the flagellar waveform conversion. Current Biology. 19 (2), 133-139 (2009).
- Bessen, M., Fay, R. B., Witman, G. B. Calcium control of waveform in isolated flagellar axonemes of Chlamydomonas. Journal of Cell Biology. 86 (2), 446-455 (1980).
- Wakabayashi, K., King, S. M. Modulation of Chlamydomonas reinhardtii flagellar motility by redox poise. Journal of Cell Biology. 173 (5), 743-754 (2006).
- Saegusa, Y., Yoshimura, K. cAMP controls the balance of the propulsive forces generated by the two flagella of Chlamydomonas. Cytoskeleton. 72 (8), 412-421 (2015).
- Gorman, D. S., Levine, R. P. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. Proceedings of the National Academy of Sciences of the United States of America. 54 (6), 1665-1669 (1965).
- Takano, W., Hisabori, T., Wakabayashi, K. Rapid estimation of cytosolic ATP concentration from the ciliary beating frequency in the green alga Chlamydomonas reinhardtii. Journal of Biological Chemistry. 296, 100156 (2021).
- Wakabayashi, K., Yagi, T., Kamiya, R. Ca2+-dependent waveform conversion in the flagellar axoneme of Chlamydomonas mutants lacking the central-pair/radial spoke system. Cell Motility and the Cytoskeleton. 38 (1), 22-28 (1997).
- Yueh, Y. G., Crain, R. C. Deflagellation of Chlamydomonas reinhardtii follows a rapid transitory accumulation of inositol 1,4,5-trisphosphate and requires Ca2+ entry. Journal of Cell Biology. 123 (4), 869-875 (1993).
- Wakabayashi, K., Ide, T., Kamiya, R. Calcium-dependent flagellar motility activation in Chlamydomonas reinhardtii in response to mechanical agitation. Cell Motility and the Cytoskeleton. 66 (9), 736-742 (2009).
- Ueki, N., Wakabayashi, K. Detergent-extracted Volvox model exhibits an anterior-posterior gradient in flagellar Ca2. Proceedings of the National Academy of Sciences of the United States of America. 115 (5), 1061-1068 (2018).
- Tanno, A., et al. The four-celled Volvocales green alga Tetrabaena socialis exhibits weak photobehavior and high-photoprotection ability. PLoS One. 16 (10), 0259138 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados