Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Desarrollo de líneas celulares musculares knock-out utilizando la edición de genes CRISPR/Cas9 mediada por lentivirus
En este artículo
Resumen
El protocolo describe cómo generar mioblastos knock-out utilizando CRISPR/Cas9, desde el diseño de arneses guía hasta la clonación celular y caracterización de los clones knock-out.
Resumen
Una aplicación importante de las repeticiones palindrómicas cortas interespaciadas reguladoras agrupadas (CRISPR)/Cas 9 es el desarrollo de líneas celulares knock-out, específicamente para estudiar la función de nuevos genes/proteínas asociados con una enfermedad, identificados durante el diagnóstico genético. Para el desarrollo de tales líneas celulares, dos cuestiones principales deben desenredarse: la inserción de las herramientas CRISPR (el Cas9 y el ARN guía) con alta eficiencia en las células elegidas, y la restricción de la actividad cas9 a la deleción específica del gen elegido. El protocolo aquí descrito está dedicado a la inserción de las herramientas CRISPR en células difíciles de transfectar, como las células musculares. Este protocolo se basa en el uso de lentivirus, producidos con plásmidos disponibles públicamente, para los cuales se describen todos los pasos de clonación para dirigirse a un gen de interés. El control de la actividad de Cas9 se ha realizado mediante una adaptación de un sistema previamente descrito llamado KamiCas9, en el que la transducción de las células con un lentivirus que codifica un ARN guía dirigido al Cas9 permite la abolición progresiva de la expresión de Cas9. Este protocolo se ha aplicado al desarrollo de una línea celular muscular humana RYR1-knock out, que se ha caracterizado aún más a nivel proteico y funcional, para confirmar el knockout de este importante canal de calcio implicado en la liberación de calcio intracelular muscular y en el acoplamiento excitación-contracción. El procedimiento descrito aquí se puede aplicar fácilmente a otros genes en las células musculares o en otras células difíciles de transfectar y producir herramientas valiosas para estudiar estos genes en las células humanas.
Introducción
Con el progreso de la secuenciación de genes y la identificación de mutaciones en genes de funciones desconocidas en un tejido específico, el desarrollo de modelos celulares relevantes para comprender la función de un nuevo gen objetivo y confirmar su participación en los mecanismos fisiopatológicos relacionados constituye una herramienta esencial. Además, estos modelos son de gran importancia para futuros desarrollos terapéuticos 1,2, y constituyen una alternativa interesante al desarrollo de modelos animales knock-out en línea recta con las recomendaciones internacionales para la reducción del uso de animales en experimentación. La edición de genes utilizando CRISPR / Cas9 se encuentra entre las herramientas más poderosas actualmente disponibles, lo que ha permitido el desarrollo de muchos modelos knock-out / knock-in, y la validación de genes dirigidos utilizando CRISPR / Cas9 se encuentra entre las aplicaciones más utilizadas de CRISPR / Cas93. El éxito de la edición de genes se basa en la capacidad de introducir las herramientas CRISPR (la guía de ARN y la nucleasa Cas9) en el modelo de célula objetivo, lo que puede ser un desafío en muchas células difíciles de transfectar, como las células musculares4. Este desafío se puede superar con el uso de virus, generalmente lentivirus, que tiene la gran ventaja de transducir eficientemente muchos tipos de células y entregar su transgén. Pero su mayor inconveniente es la integración del transgén en el genoma de la célula huésped, lo que lleva a una posible alteración de los genes localizados en el sitio de integración y a la expresión permanente del transgén, lo que en el caso de la nucleasa Cas9 tendría consecuencias perjudiciales5. Merienne y sus colegas6 han propuesto una solución inteligente, que consiste en la introducción en las células de un ARN guía dirigido al propio gen Cas9, lo que lleva a la inactivación de Cas9. Una adaptación de esta estrategia se presenta aquí como un protocolo fácil de usar y versátil que permite eliminar prácticamente cualquier gen en células difíciles de transfectar.
El objetivo del protocolo aquí presentado es inducir la inactivación de un gen de interés en células musculares inmortalizadas. Se puede utilizar para eliminar cualquier gen de interés, en diferentes tipos de células inmortalizadas. El protocolo aquí descrito contiene pasos para diseñar los ARN guía y su clonación en plásmidos lentivirales, para producir las herramientas CRISPR en vectores lentivirales, para transducir las células con los diferentes lentivirus y para clonar las células para producir una línea celular editada homogénea.
Usando este protocolo, se han desarrollado células musculares esqueléticas humanas inmortalizadas con la deleción del receptor de rianodina tipo 1 (RyR1), un canal de calcio esencial involucrado en la liberación intracelular de calcio y la contracción muscular7. El knock-out (KO) del gen se ha confirmado a nivel de proteína usando Western blot, y a nivel funcional usando imágenes de calcio.
Protocolo
Las biopsias musculares se obtuvieron del Banco de Tejidos para la Investigación (Myobank, socio de la red de la UE EuroBioBank, París, Francia) de acuerdo con las recomendaciones europeas y la legislación francesa. Se obtuvo el consentimiento informado por escrito de todas las personas. Los mioblastos inmortalizados fueron producidos amablemente por el Dr. V. Mouly (Instituto de Miología, París, Francia), y los protocolos fueron aprobados por el comité de ética del Instituto de Miología (MESRI, n AC-2019-3502).
1. Diseño de la guía CRISPR
- Identifique la región del gen que se va a eliminar. Busque su secuencia genómica utilizando herramientas de navegador del genoma como ensembl.org o genome.ucsc.edu y determine las coordenadas cromosómicas de las dos regiones para buscar el ARN guía (ARNg), a ambos lados de la región que se eliminará.
- Para el gen utilizado aquí, obtenga la secuencia FASTA del gen RYR1 y la secuencia del exón 101 de la siguiente manera. En ensembl, busque RYR1 en la versión más reciente del genoma humano, seleccione la primera entrada y haga clic en la transcripción de la secuencia codificante de proteínas. Luego, haga clic en Exones para redirigir a la lista de exones del gen.
- Haga clic en Descargar secuencia y seleccione Sólo secuencia genómica para descargar la secuencia de consenso completa de todo el gen. Desplácese hacia abajo en la lista de los exones e intrones del gen y seleccione el (los) objetivo (s).
- Encuentra la secuencia de nucleótidos correspondiente en el gen. Seleccione las secuencias de nucleótidos de los intrones inmediatamente aguas arriba y aguas abajo del exón que se eliminará, que se utilizarán para buscar los gRNAs.
- Diseñe los dos gRNA, llamados guía 1 y guía 2 aquí, en las regiones identificadas en el paso 1.1 (intrones aguas arriba y aguas abajo de la región que se eliminará) utilizando herramientas en línea como Crispor.tefor.net8. Elija los dos gRNAs separados por unos pocos cientos de pares de bases (bp), la secuencia de cada gRNA es exactamente de 20 nucleótidos de largo sin el motivo adyacente del protoespaciador (PAM). Elija las mejores guías disponibles para limitar el fuera del objetivo. Consulte la Figura 1 para ver un ejemplo de diseño de guía.
- Se espera que la secuencia entre las dos guías se elimine, y para dar lugar a la eliminación del gen de interés, elija así la posición de las dos guías de modo que elimine una secuencia esencial o un exón esencial en el gen de interés. Asegúrese de que la secuencia / exón eliminado no esté presente exclusivamente en una transcripción alternativa del gen y / o que codifique una parte importante de la proteína, por lo que su eliminación dará como resultado un knock-out funcional.
NOTA: Aunque se predice que el sitio de escisión cas9 ocurrirá 3 pb aguas arriba del motivo adyacente del protoespaciador (PAM), también puede ocurrir la escisión a una distancia mayor, por lo que una buena solución no es depender de la localización precisa del sitio de escisión, como la escisión en intrón.
- Se espera que la secuencia entre las dos guías se elimine, y para dar lugar a la eliminación del gen de interés, elija así la posición de las dos guías de modo que elimine una secuencia esencial o un exón esencial en el gen de interés. Asegúrese de que la secuencia / exón eliminado no esté presente exclusivamente en una transcripción alternativa del gen y / o que codifique una parte importante de la proteína, por lo que su eliminación dará como resultado un knock-out funcional.
- Determinar la secuencia del complemento inverso (RC) para cada ARNg, sin el PAM, con el fin de tener las siguientes secuencias: Guía 1 y Guía 1-RC, Guía 2 y Guía 2-RC.
- Ordenar los cebadores presentados en la Tabla 1 para realizar la clonación de los plásmidos. A lo largo del protocolo, utilice cebadores a una concentración de 10 nM en H2O estéril.
NOTA: Las secuencias añadidas a los ARNs en estos cebadores (en negrita y subrayados) corresponden a la secuencia del plásmido antes y después del ARN guía, promotor y transactivante-Crispr (tracrRNA), respectivamente y no deben modificarse para asegurar una buena superposición entre los cebadores y el plásmido.
2. Clonación de plásmidos
NOTA: En este paso, los gRNAs se insertarán en la columna vertebral del plásmido para la producción de lentivirus. Un casete que codifica los dos gRNAs se produce primero por sucesivas reacciones en cadena de la polimerasa (PCR), utilizando los cebadores superpuestos. El nuevo cassette se inserta en el plásmido de la columna vertebral lentiviral #87919.
- Obtener los siguientes plásmidos: plásmido #87919, codificando para los ARN guía CRISPR en un vector lentiviral y plásmido #87904 codificando para la secuencia SpCas9 en un vector lentiviral.
- Construcción de casetes
NOTA: El protocolo de clonación se resume en Figura 2.- Ejecutar una reacción de PCR (A), con 2 μL de plásmido #87919, 2 μL de primer_XmaIF, 2 μL de primer_Guide1R, 25 μL de mezcla de polimerasa y 19 μL de H2O. Ejecute el siguiente programa de PCR (programa 1): desnaturalización inicial 5 min a 98 °C, seguido de 30 ciclos de: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 s a 72 °C, y un alargamiento final de 7 min a 72 °C. La Tm de los cebadores descritos en el paso 1.4 es de 60 °C.
NOTA: Aunque el tiempo de elongación en el programa 1 parece bastante largo, este tiempo de elongación se ha elegido para garantizar la producción de suficiente material del tamaño correcto. De hecho, debido a las secuencias repetidas en el plásmido (la secuencia de tracrRNA después de las guías de ARN se repite tres veces en el plásmido # 87919) la amplificación por PCR del ADN esperado es difícil, y se producen bandas más pequeñas adicionales en las PCR sucesivas. Así, debido a la competencia entre los diferentes productos de PCR, o bien se ha aumentado el tiempo de elongación (para favorecer el más largo y tener suficiente material purificado al final), o bien se ha utilizado una PCR de tacto (programa 2) para el fragmento largo (como se describe para la PCR (final) en el paso 2.2.6). - Separe los productos de PCR en un gel de agarosa al 1% en tampón Tris-Borate-EDTA (TBE), extirpe y purifique el fragmento de 300 pb. Realice la purificación utilizando un kit dedicado siguiendo las instrucciones del fabricante, con elución en un volumen final de 20 μL. Utilice el fragmento purificado directamente o guárdelo a -20 °C para el paso 2.2.5.
- Ejecutar una reacción de PCR (B), con 2 μL de plásmido #87919, 2 μL de primer_Guide1F, 2 μL de primer_Guide2R, 25 μL de mezcla de polimerasa y 19 μL de H2O utilizando el programa de PCR 1. Separe los productos de PCR en un gel de agarosa al 1% en TBE, excise y purifique el fragmento de 400 pb en 20 μL de tampón de elución. Utilice el fragmento purificado directamente o guárdelo a -20 °C para el paso 2.2.5.
- Ejecutar una reacción de PCR (C), con 2 μL de plásmido #87919, 2 μL de primer_Guide2F, 2 μL de primer_BlpIR, 25 μL de mezcla de polimerasa y 19 μL de H2O utilizando el programa de PCR 1. Separe los productos de PCR en un gel de agarosa al 1% en tampón TBE. Extirpar y purificar el fragmento de 600 pb en 20 μL de tampón de elución. Utilice el fragmento purificado directamente o guárdelo a -20 °C para el paso 2.2.6.
NOTA: Otro fragmento a 900 pb puede ser visible, debido a la hibridación del cebador en las regiones repetidas en el plásmido, como se describe en la NOTA del paso 2.2.1. Si está presente, esta banda debe descartarse. - Ejecutar una reacción de PCR (D), con 2 μL de elución PCR A (del paso 2.2.2), 2 μL de elución PCR B (del paso 2.2.3), 2 μL de primer_XmaIF, 2 μL de primer_Guide2R, 25 μL de mezcla de polimerasa y 19 μL de H2O, con el programa de PCR 1. Separe los productos de PCR en un gel de agarosa al 1% en tampón TBE; extirpar y purificar el fragmento de 700 pb en 20 μL de tampón de elución. Utilice el fragmento purificado directamente o guárdelo a -20 °C para el paso 2.2.6.
NOTA: Otras bandas pueden ser visibles a >1,000 pb, 400 pb y 300 pb, debido a la hibridación de los cebadores en las regiones repetidas y deben descartarse. - Ejecutar una reacción de PCR (final), con 4 μL de PCR C de elución (del paso 2.2.4), 4 μL de PCR D de elución (del paso 2.2.5), 4 μL de primer_XmaIF, 4 μL de primer_BlpIR, 50 μL de mezcla de polimerasa y 34 μL de H2O. El programa utilizado es el siguiente (programa PCR 2): desnaturalización inicial durante 5 min a 98 °C; seis ciclos de: 30 s a 98 °C, 30 s a 66 °C (reducción de la temperatura de hibridación de 1 °C por ciclo), 1 min 45 a 72 °C; 35 ciclos de: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 a 72 °C, y un alargamiento final de 5 min a 72 °C.
NOTA: El programa de PCR para esta amplificación final es una PCR de toque, diferente de la anterior, debido al gran tamaño de la amplificación final que contiene dos secuencias repetidas de tracrRNA justo después de cada guía. - Separe los productos de PCR en un gel de agarosa al 1% en tampón TBE; extirpar y purificar el casete final, que migra a unos 1.300 pb en 20 μL de tampón de elución. Cuantificar el producto eluido. Utilice el fragmento purificado directamente o guárdelo a -20 °C para el paso 2.3.2.1. Este es el producto final que se insertará en el plásmido lentiviral.
NOTA: Otros fragmentos pueden ser visibles a >1000 pb, 400 pb y 300 pb, correspondientes a fragmentos de PCR incompletos que deben descartarse.
- Ejecutar una reacción de PCR (A), con 2 μL de plásmido #87919, 2 μL de primer_XmaIF, 2 μL de primer_Guide1R, 25 μL de mezcla de polimerasa y 19 μL de H2O. Ejecute el siguiente programa de PCR (programa 1): desnaturalización inicial 5 min a 98 °C, seguido de 30 ciclos de: 30 s a 98 °C, 30 s a 60 °C, 1 min 45 s a 72 °C, y un alargamiento final de 7 min a 72 °C. La Tm de los cebadores descritos en el paso 1.4 es de 60 °C.
- Inserción del cassette gRNAs en la columna vertebral del plásmido lentiviral.
- Linealizar el plásmido por doble digestión del plásmido #87919 con las enzimas de restricción XmaI y BlpI.
- Preparar la reacción con 15 μL de tampón recomendado, 15 μL de plásmido (1μg/μL), 7,5 μL de enzima BlpI (a 10 U/μL), 7,5 μL de enzima XmaI (a 10 U/μL) y 112,5 μL de H2O. Incubar durante 1 h a 37 °C, y luego durante 20 min a 65 °C. Cargue la cantidad total en un gel de agarosa al 1%, corte y purifique el plásmido de ~ 10 kb con un kit apropiado. La elución se realiza en 20 μL de tampón, y el producto eluido se cuantifica utilizando la medición de densidad óptica.
NOTA: Utilice un protocolo de purificación apropiado para fragmentos de ADN grandes, como el protocolo descrito por Sun y coll9.
- Preparar la reacción con 15 μL de tampón recomendado, 15 μL de plásmido (1μg/μL), 7,5 μL de enzima BlpI (a 10 U/μL), 7,5 μL de enzima XmaI (a 10 U/μL) y 112,5 μL de H2O. Incubar durante 1 h a 37 °C, y luego durante 20 min a 65 °C. Cargue la cantidad total en un gel de agarosa al 1%, corte y purifique el plásmido de ~ 10 kb con un kit apropiado. La elución se realiza en 20 μL de tampón, y el producto eluido se cuantifica utilizando la medición de densidad óptica.
- Ligar el casete de ARNg y el plásmido. Prepare la mezcla de reacción con casete de ARNg del paso 2.2.7 y el plásmido linealizado del paso 2.3.1.1, agregue 2 μL de enzima y H2O para tener un volumen final de 10 μL. Incube durante 15 min a 50 ° C para producir el plásmido final llamado p_guides.
NOTA: La cantidad de casete de ADN debe estar entre 50-100 ng y la cantidad de plásmido entre 100-200 ng, con una relación molar de 2:1.
- Linealizar el plásmido por doble digestión del plásmido #87919 con las enzimas de restricción XmaI y BlpI.
- Utilice 2 μL del plásmido recién preparado para transformar E.Coli químicamente competente como Stbl3 (50 μL) o XL10-Gold y extiéndalo en una placa de agar LB con 100 μg / ml de ampicilina después de 1 h de crecimiento a 37 ° C sin antibiótico. Incubar a 37 °C durante la noche. Elija algunas colonias y realice una mini preparación utilizando un kit comercial siguiendo las instrucciones del fabricante.
- Realizar una amplificación por PCR en el ADN miniprep con Primer_XmaI y Primer_BlpR utilizando el programa de PCR 1 (ver Figura 2). Separe los productos de PCR en un gel de agarosa al 1% en tampón TBE. Seleccione algunas colonias (~ 5) con una banda en el tamaño esperado de aproximadamente 1300 pb.
- Realizar la secuenciación del ADN de las colonias seleccionadas, utilizando el Primer_XmaI o Primer_BlpR, para confirmar la correcta inserción del casete de ARNg.
NOTA: Una de las colonias verificadas por secuencia se utiliza además en el estudio, y el plásmido se llama p_guides. - Repita el paso 2.2 (construcción de casete) con el Primers-Killer F y R, y el Primers-mCherry F y R. Utilice una colonia verificada por secuencia para un análisis posterior. El plásmido se llama p_Killer.
3. Producción de lentivirus
- Produzca y purifique una gran cantidad de todos los plásmidos requeridos utilizando un kit de preparación maxi sin endotoxinas siguiendo las instrucciones del fabricante. Preparar alícuotas a 2 μg/μL. Conservar a -20 °C
- Preparación de células (Día 1)
- Preparar 18 placas de 145 cm sembradas con 1 x 106 células HEK293 por placa en 16 mL de medio compuesto por piruvato de alta glucosa media de águila modificada (DMEM) de Dulbecco, complementado con 10% de suero fetal bovino (FBS) y 1% de penicilina/estreptomicina. Amplificar las células a 37 °C, en una incubadora de CO2 al 5% durante 3 días.
NOTA: La producción de lentivirus debe realizarse con precaución en un laboratorio de Nivel 2 de Bioseguridad, utilizando equipos de protección adaptados, incluido el traje de protección desechable, la gorra protectora y los guantes. Todos los experimentos deben realizarse bajo una campana de flujo laminar (gabinete de seguridad BSLII) con puntas de filtro. Toda la solución que contenga lentivirus y todos los residuos de plástico/vidrio usados deben estar inactivados con etanol al 70% u otro inactivador de virus.
- Preparar 18 placas de 145 cm sembradas con 1 x 106 células HEK293 por placa en 16 mL de medio compuesto por piruvato de alta glucosa media de águila modificada (DMEM) de Dulbecco, complementado con 10% de suero fetal bovino (FBS) y 1% de penicilina/estreptomicina. Amplificar las células a 37 °C, en una incubadora de CO2 al 5% durante 3 días.
- Transfección de células (Día 4)
- Compruebe la confluencia de las células, para asegurarse de que han alcanzado el 60%-65% de confluencia.
- Preparar la solución de transfección que contenga para cada placa: 20,8 μg del plásmido de interés (p-guías, o p-Killer, o pCas9 #87904), 4,8 μg del plásmido que codifica la envoltura (VSV-G, #8454), 20,8 μg del plásmido psPAX2 (#12260) para el envasado lentiviral, 136 μL de fosfato de calcio y ajustar con H2O a un volumen final de 1.000 μL. Añadir esta solución gota a gota y agitación a 1 mL de solución salina tamponada con HEPES (HBS).
NOTA: No prepare una mezcla para todas las placas al mismo tiempo para garantizar una preparación óptima de los reactivos; preparar una mezcla para seis platos al mismo tiempo, por ejemplo, para 18 platos, preparar tres veces una mezcla para seis platos. - Incubar a temperatura ambiente (RT) durante al menos 10 min y añadir 2 ml de solución gota a gota a gota a las células. Homogeneizar el reactivo de transfección con agitación suave de la placa hacia atrás, hacia adelante, hacia arriba y hacia abajo, incubar a 37 °C, 5% DE CO2 durante al menos 5 h.
NOTA: A partir de este punto y hasta el final de la producción de lentivirus, use equipo de protección adicional, incluido un segundo par de guantes, mangas de protección y un plastrón desechable. - Cinco horas después de la transfección, retire el medio de las placas y enjuague con PBS para deshacerse de los reactivos de transfección. Añadir 12 ml de medio fresco e incubar 48 h a 37 °C, 5% CO2.
- Recolección de las partículas virales (Día 6)
- Recoge y agrupa el medio de todos los platos. Centrifugar a 800 x g durante 5 min a 4 °C, para granular los residuos celulares. Filtre el sobrenadante utilizando un filtro de 0,45 μm (se requieren varios filtros).
- Centrifugadora a 68.300 x g durante 2 h a 4 °C en un rotor de cucharón oscilante. Retire el sobrenadante y deje los tubos boca abajo debajo del gabinete de seguridad en un papel durante 5-10 minutos para eliminar la mayor cantidad de líquido posible, y luego agregue 100 μL de medio de proliferación de HEK por pellet. Después de al menos 2 h a 4 °C, vuelva a suspender los gránulos mediante pipeteo hacia arriba y hacia abajo. Agrupa todos los pellets resuspendidos.
NOTA: Los pellets se pueden dejar en el medio durante la noche a 4 ° C antes de la alícuota. - Lentivirus alícuota en tamaño de muestra de 10 μL o 25 μL (dependiendo del uso) y congelación con nitrógeno líquido. Conservar a -80 °C. No congele una alícuota que haya sido descongelada.
- Repita los pasos 3.2 a 3.4 con otros plásmidos de interés para producir guías LV, LV-Killer y LV-Cas9.
NOTA: Como alternativa, los lentivirus se pueden comprar en una empresa o en una instalación de virus.
4. Titulación de Lentivirus
NOTA: La titulación del virus se realiza en células HEK293. La titulación es importante para incorporar en los pasos posteriores un número preciso de lentivirus por célula (cualquiera que sea el lote de lentivirus), para las células de interés. El número de partículas virales que transducen eficientemente una célula se denomina multiplicidad de infección (MOI): MOI 10 corresponde así a la introducción de 10 partículas virales por célula. Como el ciclo de congelación/descongelación afecta a la viabilidad del lentivirus, la titulación se realiza con una alícuota de lentivirus congelada, y cada experimento posterior se realizará con una nueva alícuota de la misma piscina. Aquí se describe un método de valoración, pero se pueden usar otros métodos.
- El día 1, sembrar 1 x 105 celdas por placa en cinco placas de 35 mm con fundas de vidrio en la parte inferior y dos placas de 35 mm sin fundas.
- En el día 2, a partir de las dos placas sin cubiertas, recoja y cuente el número de células después de la tripsinización, y determine la cantidad promedio de células por placa (N).
- Preparar 100 μL de lentivirus diluido a 1/10 en medio de proliferación. Transducir las cinco placas cultivadas con diferentes volúmenes de virus diluido, de 1 a 50 μL de virus diluido. Para este protocolo, utilice los siguientes volúmenes para transducir las cinco placas: 1 μL, 5 μL, 10 μL, 20 μL, 50 μL. Incubar durante 48 h a 37 °C, en un 5% de CO2.
- El día 4, fije las células mediante la incubación de los coverslips en RT durante 20 a 30 min en paraformaldehído al 4%. Para el LV-Cas9, permeabilizar las células con 0.1% Triton X100 en Phosphate Buffer Saline (PBS) durante 10 min a RT, saturar en PBS-0.1% Triton X100-2% Suero de Cabra- 0.5% Albúmina Sérica Bovina (BSA) durante 20 min en RT, y etiquetar durante 45 min en RT con anticuerpo primario anti-V5 (dilución 1/400) seguido de 30 min de incubación en RT con anticuerpo secundario fluorescente. Etiquete los núcleos con Hoescht (10 μg/mL en PBS) durante 10 min a RT.
- Monte los ladichos en un portaobjetos y observe utilizando un microscopio fluorescente equipado con un objetivo 20x. Para las guías LV y LV-Killer, después de la fijación, proceda directamente al etiquetado de los núcleos y monte las cubiertas. Para cada coverlip, cuente el número total de núcleos (número total de células) en el campo de visión y el número de células marcadas (ya sea con V5 o mCherry), y determine la proporción de células marcadas para cada coverslip.
- Seleccione un cubrehojas en el que la proporción de células transducidas sea de al menos el 10% y no exceda del 50%. Determine la eficiencia de transducción (X) para este deslizamiento de cubierta y observe el volumen de virus diluido (V, en μL) utilizado para obtener esta eficiencia de transducción.
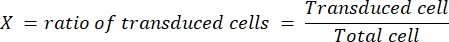
- Determine el título (en partículas infecciosas, representadas como ip/mL) del virus de acuerdo con la siguiente fórmula:

El factor de dilución es la dilución del lentivirus realizada en el paso 4.3. Obtuvimos rutinariamente un título final de 1 x 109 ip/mL para LV-Cas9 y 1 x 1010 ip/mL para LV-guía, determinado en células HEK.
5. Transducción de mioblastos
NOTA: Los mioblastos inmortalizados se transducen sucesivamente con los tres lentivirus producidos previamente. Se mantienen a una densidad inferior al 50% en un medio de proliferación compuesto por F10 de Ham suplementado con 20% de FBS, 2% de penicilina/estreptomicina, 2% de Ultroser G, y cultivados a 37 °C, 5% de CO2.
- Determinar el volumen de lentivirus necesario para tratar el número elegido de células con MOI 10 para LV-Cas9 y LV-guías y MOI 20 para LV-killer, de acuerdo con la siguiente fórmula:
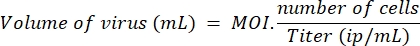
NOTA: En un experimento paralelo, se ha comparado la eficiencia de transducción en mioblastos y HEK, utilizando un lentivirus de control (lenti-GFP), y hemos determinado que se necesitan cinco veces más lentivirus para transducir un mioblasto de manera eficiente en comparación con una célula HEK. Por lo tanto, MOI 10 medido en HEK corresponde a dos partículas virales por mioblasto. Los MOI utilizados aquí se calculan en células HEK. - El día 1, sembrar placas de 96 pocillos con 10.000 células en 100 μL de medio de proliferación por pozo. El día 2, transduzca las celdas debajo del gabinete de seguridad agregando el volumen apropiado de guías LV y LV-Cas9 calculado en el paso 5.1. Devuelva las células a la incubadora hasta el día 7.
NOTA: El uso de MOI superior a 25, especialmente para LV-Cas9, puede no mejorar el número de células editadas debido a una mayor muerte celular a una alta concentración de lentivirus. - El día 7, realice la tripsinización y cuente las células. Sembra las células a una confluencia del 40% al 50% en una nueva placa y devuelve las células a la incubadora. Cinco horas más tarde, transducir las células con LV-Killer a un MOI de 20 (volumen calculado en el paso 5.1). Amplificar las células durante 5 a 10 días después de la transfección, durante al menos dos pasajes, y mantenerlas siempre en una confluencia baja de <50%. Las células están listas para el siguiente paso, la clonación celular, cuando su crecimiento vuelve a la normalidad (estimado por el tiempo de división).
NOTA: La proliferación puede ser un poco más lenta después de la transducción. El crecimiento celular normal se puede determinar antes de este procedimiento experimental mediante la estimación del tiempo de división de las células.
6. Clonación celular
NOTA: Como la transducción de mioblastos es difícil y nunca alcanza el 100% de eficiencia, incluso cuando se usa lentivirus, se requiere clonación celular para obtener una línea celular completamente corregida. Esto solo es posible con células inmortalizadas, o células que se pueden cultivar y amplificar durante unas pocas semanas / meses.
- Tripsinizar y contar las células. Diluir las células en medio de proliferación a 10 células/ml y sembrar las células en 1 célula/pozo en placas de 96 pocillos que contengan 100 μL/pocillo de medio.
NOTA: El número de placas a sembrar depende de la probabilidad de la edición genética esperada, se utilizan rutinariamente de 2 a 10 placas. - Monitoree las células para el crecimiento y amplifique progresivamente cada pozo en una placa más grande hasta alcanzar al menos una placa de 35 mm, manteniendo la confluencia de los mioblastos por debajo del 50%. Este paso puede durar de 2 a 6 semanas, dependiendo de las células utilizadas y su capacidad para crecer una vez aisladas en un pozo.
7. Selección de clones
NOTA: Este paso se realiza para identificar cuáles de los clones en crecimiento se han modificado adecuadamente.
- Diseñe un conjunto de cebadores compuesto por un cebador ubicado antes de la primera guía (Primer_BeforeGuide1F) y otro cebador ubicado después de la segunda guía (Primer_AfterGuide2R) para amplificar la región que encierra la secuencia supuestamente modificada. Consulte la Tabla 1 para los cebadores utilizados aquí.
- Recolecte células de cada clon, ahorre al menos 300,000 células para futuras amplificaciones y extraiga ADN genómico utilizando cualquier protocolo estándar en las células restantes.
- Si está creciendo un gran número de clones, para descartar los no editados, realice una prueba rápida agrupando células de cinco clones en el mismo tubo para extraer el ADN de este grupo y realizar una prueba con PCR. Repita con tantos clones como sea necesario. Luego, separe aún más los grupos que contenían celdas editadas para realizar análisis individuales.
- Controle la edición por PCR de la siguiente manera. Preparar la reacción de PCR con 1 μL de Primer_BeforeGuide1F, 1 μL de Primer_AfterGuide2R, 12,5 μL de mezcla de polimerasa, 3 μL de ADN genómico y 7,5 μL de H2O. Amplificar en un termociclador de acuerdo con las instrucciones del fabricante y los parámetros de los cebadores. Ejecute en un gel de agarosa al 1% para identificar los clones editados.
- Controle la edición mediante la secuenciación de la siguiente manera. Realice la secuenciación de Sanger de los clones seleccionados para confirmar la eliminación e identificar cómo se ha realizado la edición en cada clon. Mantenga más de un clon editado para asegurarse de que solo el gen objetivo ha sido modificado y es responsable del efecto fisiológico observado y mantenga un clon no editado que se utilizará como clon de control (CTRL) en los experimentos posteriores.
- Expanda los clones seleccionados. Una vez que la confluencia de cada clon ha alcanzado alrededor del 50%, tripsinizar las células y colocar las células en un plato más grande, hasta que se hayan producido suficientes células para realizar las caracterizaciones bioquímicas y funcionales (generalmente más de 1 x 106 por clon), y almacenar alícuotas congeladas de cada clon para su uso futuro.
8. Caracterización de clones editados
NOTA: Una vez que unos pocos clones han sido seleccionados y confirmados por secuenciación de ADN, la deleción del gen objetivo se puede confirmar a nivel de proteína utilizando Western blot, y a nivel funcional si hay un ensayo celular funcional disponible para este gen. En el caso de RYR1-KO, como RyR1 es un canal de calcio, la caracterización funcional se ha realizado mediante imágenes de calcio en células cultivadas.
- Expresión de proteínas en clones editados
NOTA: RyR1 se expresa solo en miotubos diferenciados10. Su expresión ha sido evaluada en miotubos utilizando Western blot, para confirmar la deleción a nivel proteico de RyR1, así como la deleción de la proteína Cas9.- Placa 200.000 células en medio de proliferación (descrito anteriormente, paso 5) sobre una superficie de aproximadamente 1,76 cm2 en una placa de 35 mm recubierta con laminina (superficie correspondiente a una gota de 200 μL de laminina a 10 mg/mL en PBS con calcio). Una vez que las células se adhieren a la placa después de ser incubadas durante 2-3 h a 37 ° C, 5% CO2, cambie el medio de cultivo a un medio de diferenciación compuesto de DMEM bajo glucosa + 10% suero de caballo + 1% de penicilina / estreptomicina, y devuelva las células a la incubadora durante 6 días.
- Después de 6 días de diferenciación, recolectar y lisar las células con 200 μL de RIPA suplementado con inhibidores de la proteasa. Determinar la concentración de proteínas utilizando el método folin Lowry11.
- Cargue 15 μg de proteína, después de la desnaturalización durante 30 min a RT en el tampón de desnaturalización laemmli, en un gel de acrilamida de gradiente del 5%-15%. Después de la separación electroforética, transfiera las proteínas en Immobilon P a 0.8 V durante 4 h11.
- Después de la saturación de la membrana durante 30 min a RT en PBS que contiene 0.1% Tween 20 y 5% leche seca sin grasa, incubar la membrana con los anticuerpos primarios diluidos en el mismo tampón durante 2 h a RT o durante la noche a 4 °C, lavar la membrana 5x durante 5 min con PBS-0.1% Tween 20 e incubar la membrana con los anticuerpos secundarios durante 1 h a RT. Los principales anticuerpos utilizados son: anticuerpos contra V5-tag (dilución: 1/5000) para detectar Cas9, anti-GAPDH (dilución: 1/1000) como control de carga, anticuerpo anti-RyR112,13 (dilución: 1/10.000), anticuerpo contra la subunidad alfa 1 de DHPR (dilución: 1/1000) y anticuerpo contra la cadena pesada de miosina MF20 (dilución: 1/1000).
- Lavar la membrana 5x durante 5 min con PBS-0.1% Tween 20, secar el exceso de líquido y añadir el sustrato quimioluminiscente. Proceda según lo recomendado por el proveedor de sustrato para detectar la señal quimioluminiscente.
- Caracterización funcional de clones editados
NOTA: La función de RyR1 se evaluó mediante imágenes de calcio en miotubos diferenciados, producidos a partir de clones CTRL o KO14.- Placa 50.000 células sobre una superficie de 0,2 cm2 en el centro de platos de 35 mm recubiertos con laminina (superficie cubierta por una gota de laminina de 50 μL, a 10 mg/ml en PBS con calcio) e inducir diferenciación durante 6 días como se describe en el paso 8.1.1. Preparar tres placas para cada estimulación, para tener un triplicado biológico.
- Cargue los miotubos con 50 μL de fluo 4-directo, diluido 1:1 en medio de diferenciación e incubado durante 30 min a 37 °C. Enjuague las células dos veces con el tampón KREBS suplementado con glucosa a 1 mg/ml.
- Mida las variaciones de fluorescencia con un microscopio fluorescente invertido o un microscopio confocal utilizando un objetivo 10x. Instale la placa en el escenario del microscopio e inicie la adquisición a 1 fotograma por segundo durante 90 s.
- Eliminar los KREBS restantes y estimular las células en el marco 25 mediante la adición de 2 ml de KCl para la despolarización de la membrana (concentración final de 140 mM) o 2 ml de 4 CmC (concentración final de 500 μM) para la estimulación directa de RyR1. Asegúrese de que al menos 10 miotubos estén presentes en el campo grabado.
- Cuantificar la variación de fluorescencia en cada miotubo, utilizando un software dedicado. Seleccione para el análisis al menos 10 miotubos por plato (idealmente 20-30 miotubos por plato), dibuje una línea (o una Región de Interés (ROI)) en el eje largo de cada miotubo y recoja la fluorescencia F a lo largo de esta línea para todos los fotogramas.
- Determine el valor fluorescente inicial, F0, correspondiente a los fotogramas 1 a 24. Traza la variación fluorescente (F-F0)/F0 en función del tiempo de 0 a 90 s. Repita el experimento tres veces para obtener la variación de fluorescencia de al menos 90 miotubos de tres cultivos diferentes. Agrupa todos los resultados de los 90 miotubos y calcula la media ± SEM de (F-F0)/F0 en cada marco de tiempo. Cuantificar la amplitud máxima de la liberación de calcio para cada estimulación y cada clon.
Resultados
Este protocolo se aplicó a mioblastos inmortalizados de un sujeto sano15 (las llamadas células HM, para mioblastos humanos), en el que previamente se ha caracterizado el RyR116, con el fin de eliminar el gen RYR1 que codifica la proteína RyR1. El diseño de las guías de ARN se realizó para eliminar la secuencia que abarca parte del exón 101 y el intrón 101 del gen. Se prevé que la eliminación de parte del exón 101 provoque la interrupción del marco de le...
Discusión
Un paso importante en el camino hacia la caracterización de genes de función desconocida implicados en patologías es el desarrollo de modelos celulares relevantes para estudiar la función de estos genes. El uso de la edición de genes utilizando CRISPR / Cas9 es un campo de investigación en crecimiento exponencial, y el desarrollo de modelos knock-out como se presenta aquí se encuentra entre sus aplicaciones más utilizadas. En este contexto, proponemos aquí un protocolo versátil para desarrollar un knock-out de ...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este trabajo fue financiado por subvenciones de la Association Française contre les myopathies (AFM-Téléthon) y de Auvergne-Rhône Alpes Région (AURA).
Materiales
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
Referencias
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados