Method Article
Generación, detección de alto rendimiento y biobancos de esferoides cardíacos derivados de células madre pluripotentes inducidos por humanos
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí se presenta un conjunto de protocolos para la generación y criopreservación de esferoides cardíacos (CS) a partir de cardiomiocitos derivados de células madre pluripotentes inducidos por humanos cultivados en un formato multidimensional de alto rendimiento. Este modelo tridimensional funciona como una plataforma robusta para el modelado de enfermedades, exámenes de detección de alto rendimiento y mantiene su funcionalidad después de la criopreservación.
Resumen
Los cardiomiocitos derivados de células madre pluripotentes inducidos por humanos (hiPSC-CM) son de suma importancia para el modelado y la terapéutica de enfermedades cardíacas humanas. Recientemente publicamos una estrategia rentable para la expansión masiva de hiPSC-CM en dos dimensiones (2D). Dos limitaciones principales son la inmadurez celular y la falta de disposición tridimensional (3D) y escalabilidad en plataformas de detección de alto rendimiento (HTS). Para superar estas limitaciones, los cardiomiocitos expandidos forman una fuente celular ideal para la generación de cultivos celulares cardíacos 3D y técnicas de ingeniería de tejidos. Este último tiene un gran potencial en el campo cardiovascular, proporcionando HTS más avanzado y fisiológicamente relevante. Aquí, describimos un flujo de trabajo compatible con HTS con fácil escalabilidad para la generación, mantenimiento y análisis óptico de esferoides cardíacos (CS) en un formato de 96 pocillos. Estos pequeños CS son esenciales para llenar el vacío presente en los modelos actuales de enfermedades in vitro y / o generación para plataformas de ingeniería de tejidos 3D. Los SC presentan una morfología, tamaño y composición celular altamente estructurados. Además, los hiPSC-CM cultivados como CS muestran una mayor maduración y varias características funcionales del corazón humano, como el manejo espontáneo del calcio y la actividad contráctil. Mediante la automatización del flujo de trabajo completo, desde la generación de CS hasta el análisis funcional, aumentamos la reproducibilidad intra e interlote, como lo demuestran las imágenes de alto rendimiento (HT) y el análisis de manejo de calcio. El protocolo descrito permite modelar enfermedades cardíacas y evaluar los efectos farmacológicos / terapéuticos a nivel de una sola célula dentro de un entorno complejo de células 3D en un flujo de trabajo HTS totalmente automatizado. Además, el estudio describe un procedimiento sencillo para la preservación a largo plazo y el biobanco de esferoides enteros, lo que brinda a los investigadores la oportunidad de crear un almacenamiento de tejido funcional de próxima generación. HTS combinado con almacenamiento a largo plazo contribuirá sustancialmente a la investigación traslacional en una amplia gama de áreas, incluyendo el descubrimiento y prueba de fármacos, la medicina regenerativa y el desarrollo de terapias personalizadas.
Introducción
El descubrimiento de células madre pluripotentes inducidas por humanos (hiPSC) ofreció oportunidades sin precedentes para estudiar el desarrollo humano y la enfermedad a nivel celular. Durante la última década, utilizando lecciones de desarrollo, se han establecido varios protocolos para asegurar la diferenciación eficiente de las hiPSCs en cardiomiocitos (CMs)1,2,3,4. Los cardiomiocitos derivados de hiPSC (hiPSC-CM) pueden servir como un recurso para modelar enfermedades cardiovasculares (ECV) heredables genéticamente, probar la seguridad cardíaca para nuevos medicamentos y estrategias regenerativas cardíacas 5,6,7,8. A pesar de la diferenciación cardíaca dirigida de las hiPSC, los números indefinidos de CM siguen siendo un desafío en el campo cardíaco, ya que las hiPSC-CM maduras generalmente no son proliferativas y las células humanas primarias no están disponibles en grandes cantidades.
Recientemente, describimos que la activación concomitante de la señalización Wnt con cultivo de baja densidad celular resultó en una respuesta proliferativa masiva (hasta 250 veces) de hiPSC-CMs 9,10. Esta estrategia rentable para la expansión masiva de hiPSC-CM a través del paso en serie en formato de matraz de cultivo facilita la estandarización y el control de calidad de un gran número de hiPSC-CM funcionales. Además, para mantenerse al día con la demanda de grandes lotes de hiPSC-CMs de varios donantes, se ha descrito el biobanco de hiPSC-CMs10. Sin embargo, las monocapas de cardiomiocitos sembradas en estas placas de cultivo estándar no son representativas de la compleja estructura 3D presente en el corazón. Además, la inmadurez de las hiPSC-CM ha seguido siendo un obstáculo, por lo que no ha logrado imitar el fenotipo biológico y fisiológico del entorno cardiovascular in vivo.
Se han desarrollado nuevos modelos 3D in vitro donde los hiPSC-CM muestran un comportamiento fisiológico más cercano, como la autoorganización 11,12, la remodelación de la matriz extracelular (ECM) 13, la maduración mejorada 14,15,16 y la contracción sincronizada17,18,19 . Los modelos 3D se han utilizado para el descubrimiento de fármacos, pruebas de cardiotoxicidad de fármacos, modelado de enfermedades, terapias regenerativas e incluso los primeros ensayos clínicos 20,21,22,23,24. Uno de los modelos más utilizados es el tejido cardíaco diseñado a base de fibrina (EHT), que exhibe una disposición similar a un tejido y contractilidad cardíaca13,17,25. Anteriormente, mostramos que los EHTs generados a partir de hiPSC-CMs expandidos mostraban una contractilidad comparable a los de hiPSC-CMs no expandidos, demostrando una funcionalidad celular no comprometida después de la expansión9. Sin embargo, a pesar de que la generación de EHTs a partir de hiPSC-CMs ha sido bien establecida, se anticipan nuevos desarrollos con respecto al establecimiento de una plataforma de evaluación de HT. Aquí, la rápida generación de un gran número de esferoides cardíacos (CS) autoagregantes en formato de 96 pocillos permite una mejora en las condiciones 3D para fines de detección de alto rendimiento (HTS).
En general, la ventaja de los CS como cultivo celular 3D es su alta reproducibilidad y escalabilidad. En particular, los CS combinados con el manejo robótico de muestras pueden estandarizar y automatizar el cultivo de CS, el tratamiento farmacológico y el análisis de alto contenido20. Aquí, describimos protocolos optimizados para generar CS de alta pureza y alta calidad, que pueden ser criopreservados y examinados eficientemente para la función cardíaca mediante la realización de mediciones transitorias de Ca2+ utilizando un sistema óptico de adquisición y análisis de calcio. Este modelo proporciona una herramienta simple pero poderosa para realizar pantallas de alto rendimiento en cientos a miles de esferoides17,18.
Protocolo
NOTA: los hiPSC-CM utilizados en este estudio fueron generados de acuerdo con los protocolos de cultivo de hiPSC y diferenciación de CM descritos anteriormente26,27. Opcionalmente, los hiPSC-CM se pueden expandir y crioppreservar como se publicó recientemente antes de iniciar el protocolo CS (sección 4)10.
1. Preparación de medios de cultivo celular, soluciones y alícuotas
- Preparar el medio basal
- Equilibre la penicilina-estreptomicina y el medio (RPMI 1640) a temperatura ambiente (RT) y asegúrese de que se haya descongelado por completo. Mezclar 500 mL del medio y 5 mL de pluma/estreptococo. Conservar a 4 °C durante un máximo de 8 semanas; equilibrar a 37 °C antes de su uso.
- Preparar RPMI + B27
- Equilibre el suplemento de B27 y el medio basal a RT. Asegúrese de descongelar el suplemento por completo. Mezclar 490 ml del medio basal y 10 ml del suplemento 50x B27. Conservar a 4 °C durante un máximo de 2 semanas; equilibrar a 37 °C antes de su uso.
- Preparar medios de rechapado hiPSC-CM
- Agregue un inhibidor de la proteína quinasa (ROCK) que contiene una bobina enrollada asociada a Rho (concentración final de 2 μM) y un reemplazo sérico knockout (KSR) al 10% a los medios RPMI + B27. Agregue el inhibidor de ROCK directamente al medio RM según sea necesario. No almacene los medios de cultivo una vez complementados.
- Preparar medios de descongelación CM
- Agregue una concentración de 1:100 de suplemento de supervivencia celular (por ejemplo, Revitacell) y 20% de KSR a RPMI + B27 y equilibre a 37 ° C antes de usar.
- Preparar suplemento de maduración
- La fórmula28 del medio de maduración descrita anteriormente consiste en: 3 mM de glucosa, 10 mM de L-lactato, 5 mg/mL de vitamina B12, 0,82 mM de biotina, 5 mM de creatina monohidrato, 2 mM de taurina, 2 mM de L-carnitina, 0,5 mM de ácido ascórbico, 1x NEAA, 0,5% (p/v) albumax, 1x B27 y 1% de KOSR. Para preparar una botella llena (500 ml) de suplemento de maduración, retire 65 ml de una botella de DMEM sin glucosa y complemente con 2.7 g de glucosa, 5.6 g de L-lactato, 0.025 mg de vitamina B12, 1 mg de biotina, 3.73 g de monohidrato de creatina, 1.25 g de taurina, 1.975 g de L-carnitina, 0.7125 g de ácido ascórbico, 50 ml de NEAA, 12,5 g de albumax y 5 ml de penicilina-estreptomicina.
- Filtrar a través de una unidad de filtro desechable estéril con una membrana de polietersulfona (PES) de poros de 0,22 μm.
- Alícuota en 45 mL (para preparar 500 mL de medio de maduración) o 4.5 mL (para preparar 50 mL de medio de maduración). Conservar a 20 °C durante un máximo de 6 meses.
- Preparar los medios de maduración
- Equilibre el suplemento B27, knockout SR, penicilina-estreptomicina, el suplemento de maduración28 y el medio DMEM sin glucosa en RT. Asegúrese de descongelar el suplemento por completo. Mezcle 435 ml del medio DMEM sin glucosa con 10 ml del suplemento 50x B27, 5 ml de penicilina-estreptomicina, 5 ml de knockout SR y 45 ml de suplemento de maduración. Conservar a 4 °C durante un máximo de 2 semanas; equilibrar a 37 °C antes de su uso.
- Preparar flúor medio brillante
- Equilibrar la penicilina-estreptomicina y el medio fluorobrito DMEM en RT. Asegúrese de que el suplemento esté completamente descongelado. Mezcle 500 ml del medio de fluorbrita DMEM con 5 ml de penicilina-estreptomicina. Conservar a 4 °C durante un máximo de 1 mes; equilibrar a 37 °C antes de su uso.
- Preparar la solución detergente no iónica
- Mezcle 20% p/v detergente no iónico en polvo (por ejemplo, F-127) con PBS. Filtrar con un filtro de 0,22 μm y conservar a 4 °C durante un máximo de 6 meses; equilibrar en RT antes de su uso.
- Preparar medio de colorante de calcio
- Mezclar la solución detergente no iónica (concentración final de 0,04% v/v) y 0,1x del colorante de calcio (por ejemplo, Cal520 AM) en un medio brillante de flúor. En un tubo cónico de 50 ml, añadir 10 μL de Cal520 y 20 μL de la solución detergente no iónica. Mezclar hasta que se disuelva completamente. Mantenga la solución en la oscuridad antes de agregarla a las células.
2. Preparación de tampones
- Preparar el tampón de permeabilización y bloqueo: Este tampón contiene 10 ml de PBS, 5% peso / v BSA y 0.3% v / v Triton-X-100.
- Prepare el tampón de citometría de flujo: Este tampón contiene 50 ml de PBS, 1% de BSA en peso / v y 0.3% v / v Triton-X-100.
- Tampón de lavado por citometría de flujo: Este tampón contiene 50 ml de PBS y 1% de BSA en peso/v.
- Tampón de lavado esferoide (SWB): Este tampón contiene 1 ml de Triton-X-100, 2 ml de SDS al 10% (p/v en DPBS) y 2 g de BSA en 1 L de PBS.
NOTA: SWB puede conservarse a 4 °C durante un máximo de 2 semanas. - Prepare la solución de incrustación (ES): Para preparar 100 ml de la solución de incrustación, mezcle 50 ml de glicerol con 9,09 ml de dH2O, 1 ml de tampón Tris (1 M, pH 8,0) y 200 μL de EDTA (0,5 M). Añadir 22,7 g de fructosa y mezclar en RT en la oscuridad hasta que se disuelva. Cuando esté claro, añadir 22,2 g de fructosa y mezclar hasta que se disuelva. A continuación, añadir 15 g de urea y mezclar hasta que se disuelva (conservar a 4 °C en la oscuridad).
- Prepare el búfer PBT (PBS con interpolación 20). Este búfer contiene PBS/Tween-20 (0,1% v/v). Para 1 L de PBS, agregue 1 ml de Tween-20.
3. Preparación de moléculas pequeñas
- Reconstituir el polvo de tiazovivina (inhibidor de ROCK) en alícuotas de 10 mM de 50 μL en DMSO y almacenar a -20 °C durante un máximo de 6 meses. Proteger de la luz.
- Preparar 2,5 mM de alícuotas de 10 μL cada una de Cal-520 AM en DMSO y almacenarlas a -20 °C durante un máximo de 6 meses. Proteger de la luz.
4. Generación de esferoides cardíacos
NOTA: Para cantidades mayores de CS, siembre hasta 1 millón de CM en una placa de fijación ultra baja de 6 pocillos con 2 ml de medios de rechapado hiPSC-CM. Este estudio utilizó un mínimo de 2.500 (2,5k CS) hasta 20.000 (20k CS) hiPSC-CMs por pocillo de una placa de 96 pocillos.
- Para una placa de 96 pocillos, preparar un cultivo celular que contenga un mínimo de 2 x 106 cardiomiocitos derivados de células madre pluripotentes inducidos por humanos (hiPSC-CM)10.
- Cuando los hiPSC-CM cultivados alcancen la confluencia, agregue 0,1 ml/cm2 de solución estéril de desprendimiento cardíaco (p. ej., triplé) a cada pocillo. Incubar la placa a 37 °C durante 15 min.
- Usando una pipeta de 5 ml, disocie mecánicamente las células enjuagando con 2 ml de medio basal caliente para hacer una suspensión de una sola célula. Confirme el desprendimiento con un microscopio de campo brillante (aumento 4x); Las células se verán blancas y tendrán una forma redonda.
- Transfiera la suspensión celular a un tubo cónico de 15 ml y centrífuga durante 3 min a 300 x g.
- Aspirar el sobrenadante y resuspender las células en 1 ml de medios de rechapado hiPSC-CM.
- Usando una punta de pipeta de 1.000 μL, disociar mecánicamente el pellet celular. La solución parece homogénea después de tres o cuatro mezclas. Cuenta las celdas. Transfiera la cantidad apropiada de células en 100 μL del medio de rechapado a cada pocillo de 96 pocillos de fondo redondo de unión ultrabaja.
- Coloque la placa de CS en un agitador orbital a 70 rpm en la incubadora durante 24 h. Ajuste las condiciones de la incubadora a 37 °C, 5% de CO2, 21% deO2 y 90% de humedad.
- Aspirar 50 μL de medio de cada pocillo y añadir 100 μL de RPMI + B27 medio por pocillo durante las primeras 48 h.
NOTA: Mantenga siempre 50 μL del medio en la placa de 96 pocillos para evitar la aspiración accidental y la ruptura del esferoide. - Aspirar 100 μL del medio de cada pocillo y añadir 100 μL del medio de maduración por pocillo. Mantenga las células en el medio de maduración y refresque el medio cada 2-3 días.
5. Criopreservación de CSs
NOTA: Los CS se pueden crioppreservar para su almacenamiento a largo plazo. La criopreservación se puede realizar a partir del día 3 después de la generación de CS. Los CS se pueden criopreservar directamente en los pocillos de una placa de 96 pocillos o como una suspensión de CS en crioviales.
- Pre-enfríe la placa colocando la placa en hielo durante 10 minutos.
- Centrifugar la placa esferoide durante 3 min a 70 x g.
- Retire el sobrenadante hasta que queden 50 μL y agregue 200 μL de medio de congelación hiPSC helado por pocillo.
NOTA: Mantenga la suspensión de CS en hielo durante todo el procedimiento. En el caso de una placa de 6 pocillos con esferoides, congelar un pocillo en un medio de congelación criovial de 500 μL. - Selle la placa con una película de sellado de placas.
NOTA: La placa de 96 pocillos debe almacenarse en una caja de poliestireno o, cuando no esté disponible, se puede hacer un molde de silicona como se describe en el paso 5.5.1. - Para garantizar un intercambio de calor uniforme entre la placa del pozo y el congelador, coloque la placa cuidadosamente en una caja de poliestireno o en un molde de silicona.
- Para preparar el molde de silicio: Mezcle vigorosamente dos componentes del kit de elastómero de silicio en una proporción de 10: 1. Desburbujear la solución con una bomba de vacío durante 15-20 min. Posteriormente, colte la solución dentro de la parte inferior de la placa del pozo y desburbujee con una bomba de vacío durante 10 minutos. Colocar el molde en un horno y curar a 60 ºC durante 8 h para obtener un elastómero semiflexible que se despega de la placa.
- Congelar la placa a -80 °C durante un mínimo de 4 h en la caja de poliestireno o en el molde de silicona preparado.
- Transfiera la placa a un tanque de nitrógeno líquido o a un congelador de -150 °C para su almacenamiento a largo plazo.
6. Descongelación de esferoides cardíacos
NOTA: No descongele más de una placa a la vez para garantizar un proceso de descongelación rápido.
- Preparar 20 ml de medio basal precalentado a 37 °C en un tubo cónico de 50 ml.
- Recoja la placa celular con CS del nitrógeno líquido y colóquela en la incubadora durante 15 minutos. Ajuste las condiciones de la incubadora a 37 °C, 5% de CO2, 21% deO2 y 90% de humedad.
- Retire el sobrenadante y los restos del pellet celular, y resuspenda cada pocillo en un medio basal cálido. Use 200 μL de medio por pocillo.
- Centrifugar durante 3 min a 70 x g.
- Repita los pasos 6.3 y 6.4.
- Retirar el sobrenadante hasta que quede el pellet celular y añadir 200 μL de medio de descongelación CM en cada pocillo.
- Coloque la placa de CS en un agitador orbital a 70 rpm en una incubadora durante 24 h. Ajuste las condiciones de la incubadora a 37 °C, 5% de CO2, 21% deO2 y 90% de humedad.
- Aspirar 50 μL del medio de cada pocillo y añadir 100 μL de RPMI + medio B27 por pocillo durante las primeras 48 h.
- Aspirar 100 μL del medio de cada pocillo y añadir 100 μL de medio de maduración por pocillo. Mantenga las células en el medio de maduración y refresque el medio cada 2-3 días.
7. Evaluación de transitorios intracelulares de Ca2+
NOTA: Los CS están en cultivo por un total de 3 semanas; 2 semanas antes de la congelación y 1 semana después de la descongelación. Los controles "frescos" coinciden con la edad.
- Después de 1 semana de cultivo, los CS descongelados son óptimos para el manejo de imágenes ópticas de calcio. Use un colorante de calcio (por ejemplo, Cal520AM) para evaluar la absorción y liberación de Ca2+ de las células.
- Trátelos con 100 μL de medio de colorante cálcico por pocillo e incubar durante 60 minutos en la incubadora. Ajuste las condiciones de la incubadora a 37 °C, 5% de CO2, 21% deO2 y 90% de humedad.
NOTA: Cal520AM es sensible a la luz. Realice todos los procedimientos de carga y experimentos en la oscuridad. - Preparar el sistema de adquisición y análisis de calcio.
- Alimente el microscopio, asegurando que la opción de control ambiental esté activada.
- Ajuste las dimensiones de apertura de la cámara y el encuadre para minimizar el área de fondo.
NOTA: Aquí se utilizó el microscopio Leica Thunder DMi8; Otros sistemas de microscopio también son aplicables hasta que permitan una frecuencia de muestreo superior a 30 fotogramas / segundo (fps).
- Grabe un video con un flujo consistente de 2-10 picos dentro de 10 s y escanee a través de la placa de 96 pocillos, inicialmente moviéndose hacia la izquierda, luego hacia abajo en forma de zigzag para cubrir toda la placa. Mida la señal de calcio usando un láser de 488 nm; Establezca el contraste en un fondo negro con una señal verde brillante durante la liberación de calcio.
- Después de adquirir los transitorios Ca2+ , analice los datos con el software de análisis de trazas de fluorescencia (por ejemplo, CyteSeer, Vala Sciences) de acuerdo con las instrucciones del fabricante.
8. Análisis de citometría de flujo de esferoides cardíacos disociados
NOTA: En este estudio, se utilizó citometría de flujo para determinar la viabilidad de los CS antes y después del proceso de descongelación.
- Recoger los CS en un tubo cónico de 15 ml utilizando una pipeta de 5 ml para evitar el daño esferoide y centrifugar durante 3 min a 70 x g. Aspirar el sobrenadante y añadir 1 ml de PBS.
- Centrifugar durante 3 min a 200 x g. Aspirar el sobrenadante y disociar los SC agregando 1 ml de solución de desprendimiento cardíaco (por ejemplo, triplo). Incubar el tubo a 37 °C durante 15 min.
- Usando una pipeta de 5 ml, disocie mecánicamente las células enjuagando con 2 ml de medio basal hasta que se puedan ver células individuales cuando se observan bajo el microscopio.
- Centrifugar durante 3 min a 200 x g.
- Aspirar el sobrenadante y fijar los CM con 200 μL de solución de paraformaldehído (PFA) al 4% en 1x PBS. Incubar durante 10 min en RT.
- Centrifugar durante 3 min a 200 x g. Aspirar el sobrenadante y añadir 1 ml de PBS.
NOTA: Punto de pausa: Los hiPSC-CM fijos pueden almacenarse a 4 °C durante un máximo de 4 semanas. - Transfiera la suspensión celular a un tubo FACS y centrífuga durante 3 min a 200 x g. Aspirar el sobrenadante y resuspender 1 x 105 células en 50 μL del tampón de permeabilización.
- Incubar las celdas durante 30 min a 4 °C.
- Para el análisis de citometría de flujo de inmunofluorescencia, realice los pasos 8.9.1-8.9.4.
- Resuspender las células en el tampón de citometría de flujo (50 μL) que contiene el anticuerpo α-actinina a una dilución de 1:300. En otro tubo FACS, resuspender 1 x 105 células en el tampón de citometría de flujo (50 μL) con el control de isotipo respectivo (por ejemplo, IgM de ratón FITCH, isotipo κ [dilución 1:200]). Del mismo modo, resuspender 1 x 105 células en 50 μL de tampón de citometría de flujo para control negativo.
- Incubar las celdas durante 30 min a 4 °C.
- Añadir 2,5 ml de tampón de citometría de flujo y centrifugar las células a 200 x g durante 3 min a 4 °C; Deseche el sobrenadante y repita este paso de lavado dos veces.
- Resuspender las células en 50 μL de tampón de citometría de flujo con el anticuerpo secundario cabra-anti-ratón (dilución 1:300).
NOTA: Coloque el tubo en la oscuridad ya que la solución de anticuerpos secundarios es sensible a la luz.
- Para comprobar la viabilidad con yoduro de propidio (PI), añadir 150 μL de PI por muestra (1:1.000) e incubar durante 15 min.
NOTA: Coloque el tubo en la oscuridad ya que la solución PI es sensible a la luz. - Ajuste las compuertas de acuerdo con la estrategia de compuerta estándar como se muestra en la Figura suplementaria 1 y analice las células con un citómetro de flujo.
9. Tinción por inmunofluorescencia de esferoides 3D enteros
NOTA: Este protocolo se basa en el protocolo para imágenes 3D de alta resolución de organoides completos tras el etiquetado inmunofluorescente, que se publicó previamente29 y se ajustó para esferoides cardíacos. Durante el procedimiento, todas las puntas de pipeta y tubos cónicos se pueden recubrir con BSA-PBS al 1% en peso / v para evitar que los esferoides se adhieran a los plásticos. Para recubrir los materiales, sumérjase en el BSA-PBS al 1%. Tenga cuidado de no dañar los esferoides utilizando la pipeta de 5 ml, evitando interrupciones mecánicas.
- Recoger los CS en un tubo recubierto de 15 ml con una pipeta de 5 ml. Los esferoides son visibles para el ojo. Recolecta ~20-50 esferoides por combinación de anticuerpos. Centrifugar durante 3 min a 70 x g y aspirar el sobrenadante.
- Resuspenda cuidadosamente los esferoides en 1 ml de solución helada de paraformaldehído al 4% (PFA) en 1x PBS usando una punta recubierta de 1 ml.
- Fijar a 4 °C durante 45 min. Después de 20 minutos, resuspenda suavemente los esferoides con una punta recubierta de 1 ml. Esto iguala la fijación entre todos los esferoides.
- Agregue 10 ml de PBS helado al tubo y mezcle suavemente invirtiendo el tubo. Incubar durante 10 min a 4 °C y girar a 70 x g durante 3 min.
NOTA: A partir de este paso, generalmente no es necesario el recubrimiento de puntas y tubos cónicos, ya que los CS no se adhieren a la punta después de la fijación. - Bloquear los CS resuspendiendo el pellet en SWB helado (200 μL de SWB por pocillo) y transferir los esferoides a una placa de cultivo de suspensión de 24 pocillos.
NOTA: Los CS de un pellet grande se pueden dividir en múltiples pocillos para realizar diferentes tinciones. Use ~20-50 CS por combinación de anticuerpos. - Incubar a 4 °C durante al menos 15 min.
- Agregue 200 μL del SWB en un pozo vacío para que sirva como pozo de referencia.
NOTA: Para la tinción inmunofluorescente, también se pueden usar placas de 48 o 96 pocillos para reducir el uso de anticuerpos. Sin embargo, los resultados de tinción y lavado pueden reducirse debido al menor volumen por pozo. - Permita que los esferoides se asienten en la parte inferior de la placa, dejando la placa inclinada en un ángulo de 45° durante 5 min.
- Retire el SWB, dejando los CS en 200 μL del SWB (use el pocillo de referencia para estimar el volumen mínimo de 200 μL).
- Añadir 200 μL del SWB con los anticuerpos primarios 2x concentrados (p. ej., ɑ-actinina [1:200] y troponina T [1:200]) e incubar durante la noche a 4 °C mientras se balancea/agita (40 rpm en una agitadora horizontal).
- Al día siguiente, agregue 1 ml del SWB a cada pocillo.
- Deje que los esferoides se asienten en la parte inferior de la placa dejando la placa en un ángulo de 45° durante 5 minutos.
- Retire el SWB, dejando 200 μL en la placa. Añadir 1 ml de SWB y lavar durante 2 h con balanceo/agitación lenta.
- Repita los pasos 9.12 y 9.13 dos veces más.
- Deje que los CS se asienten en la parte inferior de la placa dejando la placa inclinada a 45° durante 5 minutos. Retire el SWB, dejando 200 μL en cada pocillo
- Añadir 200 μL del SWB con anticuerpos secundarios, anticuerpos conjugados y colorantes 2x concentrados (p. ej., DAPI [1 μg/ml], ratón-AF488 [1:500], conejo-AF568 [1:500]), e incubar durante la noche a 4 °C en la oscuridad, mientras se balancea/agita lentamente.
- Al día siguiente, repita los pasos 9.12 y 9.13 dos veces más.
- Transfiera con cuidado los CS a un tubo de 1,5 ml y gire hacia abajo a 70 x g durante 3 min.
- Elimine la mayor cantidad posible de SWB pipeteando sin interrumpir los CS.
- Añadir la solución de incrustación (ES; al menos 50 μL, a RT) con una punta de 200 μL con el extremo cortado y resuspender suavemente para evitar la formación de burbujas e incubar a RT durante 20 min.
- Mientras tanto, cree un recipiente cuadrado en un portaobjetos de vidrio con esmalte de uñas o sellador de silicona.
- Corte el extremo de una punta de 200 μL y transfiera los CS en ES al centro del recipiente cuadrado.
- Coloque un cubreobjetos cuadrados en la parte superior. Para reducir las burbujas de aire, coloque primero un lado del cubreobjetos, luego baje lentamente el cubreobjetos de un lado a otro hasta que no quede aire atrapado debajo de la superficie, y luego suelte el cubreobjetos.
- Empuje suavemente todos los bordes del cubreobjetos para sellarlo al esmalte de uñas o al sellador de silicona.
- Deje la diapositiva durante la noche en RT. Al día siguiente, la diapositiva está lista para la obtención de imágenes.
NOTA: El aclaramiento óptico por el ES puede causar una contracción menor del tejido. Sin embargo, esto no puede afectar la morfología general de los CS. El procedimiento de tinción se puede pausar aquí almacenando los portaobjetos a 4 °C (durante al menos 1 semana) o a -20 °C (durante al menos 6 meses).
Resultados
El protocolo que se muestra en la Figura 1A describe la generación de CS a partir de hiPSC-CM previamente expandidos. Los CS adquieren una estructura 3D para el día 1 después de la siembra en placas de fondo redondo de fijación ultra baja y se pueden cultivar hasta por 6 semanas (Figura 1B). Según lo evaluado por tinción de inmunofluorescencia, la mayoría de las células en SC de 3 semanas de edad expresaron proteínas sarcoméricas como α-actinina y troponina T y mostraron una organización regular del sarcómero (Figura 1C). Para la cuantificación de células positivas para α-actinina, se realizó un análisis de citometría de flujo. De acuerdo con los resultados de inmunofluorescencia, los datos de citometría de flujo demostraron niveles altos comparables de α-actinina tanto en el día 0 (76,9% ± 16,6%) como en los CS de 3 semanas de edad (71,1% ± 22,7%) (Figura 1D), lo que indica una composición celular constante y altamente pura durante el cultivo. Hubo un aumento de la expresión de los genes cardíacos para uniones (GJA1, JPH2 y PKP2), desmosomas (DES) y mitocondrias (ATP5A) en esferoides derivados de hiPSC-CM (día 42) versus hiPSC-CM cultivados en 2D durante 90 días (Figura 1E). La expresión de estos genes es un sello distintivo de la interacción célula-célula y la maduración30.
Posteriormente, las propiedades funcionales de los CS, a saber, la velocidad de batido y el manejo de Ca2+, se evaluaron en diferentes puntos de tiempo (Figura 2). Los parámetros transitorios de calcio, como el tiempo de subida, la hora pico, el tiempo de decaimiento y la duración transitoria del calcio (CTD90) se evaluaron como se indica en la Figura 2A, B. El porcentaje de superación de CS es similar en las primeras 3 semanas después de la generación, pero disminuyó significativamente en la semana 6 (Wk6) CS (Figura 2C). La tasa de batidos se redujo significativamente en Wk3 en comparación con Wk1 y, similar al porcentaje de CS superados, disminuyó drásticamente en Wk6 (Figura 2D). En Wk6, se observó un deterioro del CS, lo que puede explicar la caída tanto en la tasa de latido como en el número de SC de latido. La medición de los parámetros transitorios de calcio indicó un valor máximo significativamente mayor en Wk2 (Figura 2E), mientras que el tiempo de subida, el tiempo de decaimiento y CTD90 aumentaron significativamente en Wk3 en comparación con Wk1 (Figura 2F-H ). Tomados en conjunto, estos resultados muestran que los esferoides derivados de hiPSC-CM son funcionalmente óptimos alrededor de las semanas 2 y 3 después de la generación.
La Figura 3 muestra el efecto del tamaño del esferoide en la velocidad de batido y el manejo del calcio. Los CS se generaron sembrando 2.5 x 10 4, 5 x 10 4, 10 x 10 4 y 20 x 10 4 hiPSC-CM en un pocillo de una placa de 96 pocillos para un total de 24 CS / pocillos por condición (Figura 3A). Como era de esperar, el tamaño del esferoide aumentó a medida que aumentaba el número de células utilizadas, variando de 178 ± 36 μm a 351 ± 65 μm (Figura 3A, panel derecho). Los transitorios de Ca2+ se midieron en CS de 3 semanas de edad en las cuatro densidades de siembra diferentes (Figura 3B). Las mediciones de los CS de batido indicaron que solo alrededor del 50% de los CS de tamaño más pequeño (2.5K y 5K-CS) estaban latiendo, mientras que el porcentaje de CS de mayor tamaño (10K y 20K-CS) fue significativamente mayor (alrededor del 85%) (Figura 3C). Una tasa de latido similar (aproximadamente 28 lpm) fue mostrada por 5K-, 10K- y 20K-CSs, que fue significativamente mayor en comparación con 2.5K-CSs (Figura 3D). Los valores máximos de las imágenes de calcio fueron similares en todas las condiciones probadas (Figura 3E), sin embargo, el tiempo de aumento (Figura 3F), el tiempo de decaimiento (Figura 3G) y CTD90 (Figura 3H) aumentaron significativamente en CS de mayor tamaño (10K y 20K-CS) en comparación con los más pequeños (2.5K y 5K-CS). Tomados en conjunto, estos resultados muestran que los esferoides derivados de hiPSC-CM son óptimos para la detección del manejo del calcio cuando se utiliza una densidad de siembra entre 10K y 20K hiPSC-CMs/pocillo.
A continuación, evaluamos el impacto de la criopreservación en la viabilidad y función de la CS. Antes del análisis, los CS descongelados se mantuvieron en cultivo durante 1 semana (Figura 4A). Como se muestra en las pruebas de viabilidad celular de citometría de flujo (Figura 4B) y Calcein-AM (Figura 4C), la criopreservación no afectó la viabilidad celular dentro de las CS. Además, los SC descongelados mostraron niveles de expresión similares de proteínas sarcoméricas en comparación con los CS frescos emparejados por edad (Figura 4D). Estos datos indican que los SC pueden ser criopreservados eficientemente para el posterior análisis de la función cardíaca y la detección de alto rendimiento.
Finalmente, la actividad de batido y el manejo de Ca2+ se midieron tanto en CS frescos como criopreservados (Figura 5). El porcentaje de superación de CS se midió en diferentes puntos de tiempo después de la descongelación, respectivamente, a los 2, 5 y 7 días. Si bien la mayoría de los CS frescos mostraron actividad de latido a lo largo del tiempo, claramente los CS criopreservados necesitaron hasta 1 semana de cultivo para recuperar su actividad de batido (Figura 5B). No hubo cambios significativos en la tasa de batido de los CS descongelados versus frescos; sin embargo, no se observó actividad espontánea de batidos en algunos CS congelados (Figura 5C). Aunque los valores máximos se redujeron significativamente en los CS congelados/descongelados en comparación con los frescos (Figura 5D), no se observaron cambios significativos en el tiempo de subida, el tiempo de descomposición y el CTD90 de los CS congelados/descongelados en comparación con los frescos (Figura 5E-G). Estos datos indican que, después de la descongelación, es importante dejar que los CS se recuperen en la incubadora durante al menos 1 semana antes de medir la actividad de latido y el transitorio Ca2+.
Tomados en conjunto, estos resultados muestran que la criopreservación de esferoides derivados de hiPSC-CM preserva la viabilidad de los cardiomiocitos, la estructura sarcomérica y sus características funcionales, como la actividad espontánea de latido y el manejo del calcio. Por lo tanto, los esferoides derivados de hiPSC-CM representan un modelo adecuado para recapitular con precisión la electrofisiología cardíaca in vitro.
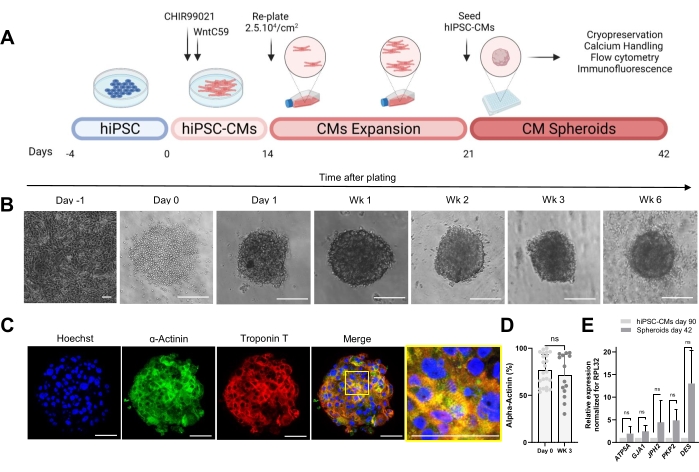
Figura 1: Generación de esferoides cardíacos . (A) Representación esquemática de la diferenciación cardíaca dirigida basada en Wnt, la posterior expansión de hiPSC-CMs y la generación de CSs. Creado con biorender.com. (B) Imágenes de campo claro en diferentes puntos de tiempo de cultivo de CS. Barra de escala, 200 μm. Wk representa semana. (C) Imágenes representativas de inmunofluorescencia para proteínas sarcoméricas cardíacas α-actinina y troponina T en CS de 3 semanas de edad. Inmunofluorescencia: Hoechst (azul), α-actinina (verde) y troponina T (rojo). Barra de escala, 200 μm. La imagen combinada ampliada a la derecha muestra la organización del sarcómero. Barra de escala, 50 μm. (D) Cuantificación por citometría de flujo de células positivas para α-actinina antes (día 0) y 3 semanas después de la formación de CS. (n = 14-23 por condición. (E) RT-qPCR realizada en hiPSC-CMs cultivadas durante 90 días (2D) y muestras esferoides cultivadas durante 42 días para establecer niveles de expresión de diferentes genes cardíacos relacionados con uniones celulares, filamentos intermedios y mitocondrias. (n = 1-3 lotes). Los datos se representan como media ± DE. NS (no significativo) calculado por una prueba t no pareada. Haga clic aquí para ver una versión más grande de esta figura.
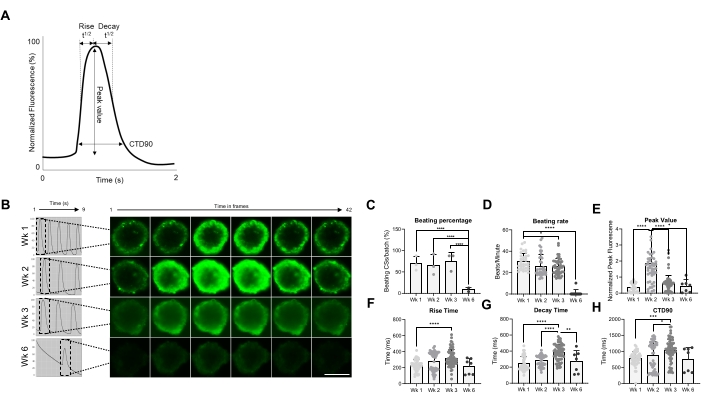
Figura 2: Tasa de batido y manejo de calcio en CSs en diferentes semanas post generación. (A) Ejemplos de parámetros transitorios de calcio calculados por el algoritmo de análisis de ciencias Vala en Cyteseer Software. (B) Trazas transitorias de calcio representativas e imágenes de lapso de tiempo de los CS en diferentes puntos de tiempo (semanas) posteriores a la generación. Barra de escala, 200 μm. (C) La cuantificación del curso temporal de la actividad de batido espontáneo se expresa como el porcentaje de SC de batido. (D) Tasa de batido de CS durante el tiempo de cultivo. (E-H) Cuantificación de los transitorios de calcio que muestran el valor máximo, el tiempo de subida, el tiempo de decaimiento y CTD90. Los datos mostrados son medias ± DE. Réplicas biológicas = tres, réplicas técnicas = 38, 50, 66 y 7, respectivamente. *p < 0,05, ****p < 0,001; ANOVA unidireccional seguido de la prueba de comparaciones múltiples post hoc de Tukey. Abreviaturas; CTD = duración transitoria del calcio, Wk = semana, CS = esferoides cardíacos humanos. Haga clic aquí para ver una versión más grande de esta figura.
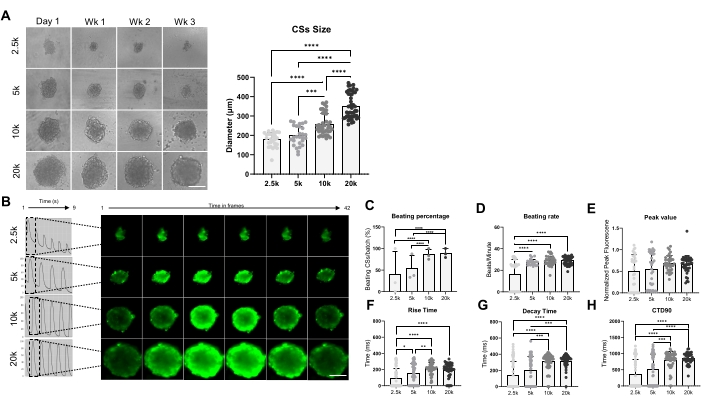
Figura 3: Tasa de batido y manejo de calcio en CSs generados utilizando diferentes densidades de siembra celular. (A) Imágenes de campo claro (izquierda) y mediciones de tamaño (derecha) de CS generadas utilizando diferentes números de hiPSC-CM. Barra de escala, 200 μm. (B) Trazas transitorias representativas de calcio e imágenes de lapso de tiempo de los 2.5K-20K-CS. (C,D) Porcentaje de batir y tasa de batir de 2.5K-20K-CSs. (E-H) Valor máximo, tiempo de subida, tiempo de decaimiento y CTD90 en 2.5K-20K-CSs. Los datos son medias ± DE. Réplicas biológicas = tres, réplicas técnicas = 28-39. *p < 0,05, ****p < 0,001; ANOVA unidireccional seguido de la prueba de comparaciones múltiples post hoc de Tukey. Abreviaturas: CTD = duración transitoria del calcio, Wk = semana, k = x 1.000 células, CS = esferoides cardíacos. Haga clic aquí para ver una versión más grande de esta figura.
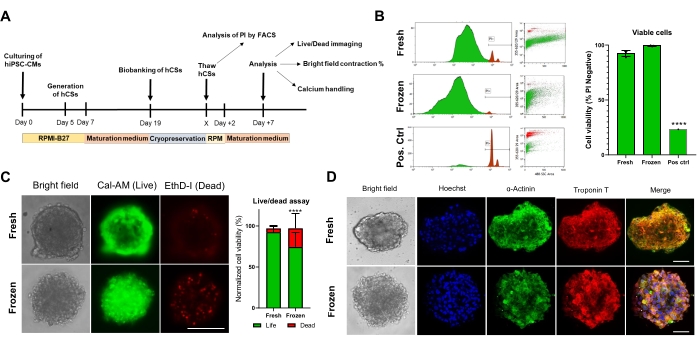
Figura 4. Efecto de la criopreservación sobre la viabilidad y estructura de los esferoides cardíacos. (A) Representación esquemática de la generación de CS, biobancos posteriores y descongelación. (B) Prueba de viabilidad celular de citometría de flujo en CS frescos y criopreservados. Como control positivo, se utilizó un tratamiento con solución Triton-X al 10% durante 5 min. (n = 4 por condición). Los datos se representan como media ± DE. ****p < 0,001; ANOVA unidireccional seguido de la prueba de comparaciones múltiples post hoc de Tukey. (C) Prueba de viabilidad celular Calcein-AM en CS frescos versus descongelados después de 7 días de cultivo (n = 15-17 por condición, ****p < 0.001, por prueba t pareada; barra de escala, 200 μm). (D) Tinción representativa de campo claro (izquierda) e inmunofluorescencia para la expresión de α-actinina y troponina T en CS frescos y descongelados. Inmunofluorescencia: Hoechst (azul), α-actinina (verde) y troponina T (rojo). Las imágenes combinadas de la derecha muestran estrías de sarcómero en los CS. Barra de escala, 50 μm. Abreviaturas: X = día de descongelación de elección, PI = yoduro de propidio, Cal-AM = calceína-AM, EthD-I = Ethidium Homodimer I. Haga clic aquí para ver una versión más grande de esta figura.
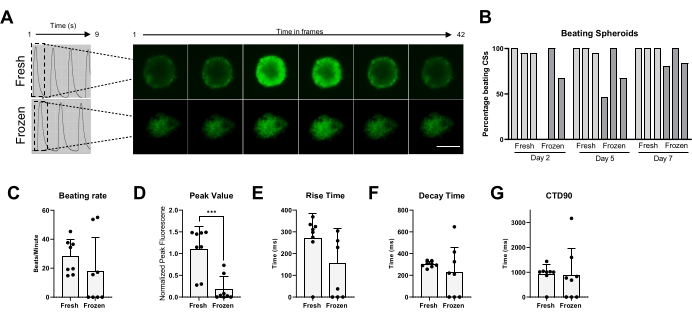
Figura 5: Transitorios de calcio en CS frescos versus descongelados. (A) Trazas transitorias representativas de calcio e imágenes de lapso de tiempo de las CS antes de la criopreservación y 1 semana después de la descongelación. (B) Porcentaje de latidos de esferoides cardíacos frescos y congelados/descongelados. Las barras representan experimentos individuales. (C) Tasa de latido de esferoides cardíacos frescos y congelados/descongelados. (D-G) Cuantificación de parámetros transitorios de calcio: valor pico, tiempo de subida, tiempo de decaimiento y CTD90. Los datos son medias ± DE. *p < 0,05, ****p < 0,001; ANOVA unidireccional seguido de la prueba de comparaciones múltiples post hoc de Tukey. Abreviaturas; CTD = duración transitoria del calcio, CSs = esferoides cardíacos. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Estrategias representativas de compuerta para el análisis de citometría de flujo. (A) Estrategia representativa de gating para hiPSC-CM positivas para α-actinina en una población pura versus control negativo y control de isotipo. El número de células analizadas positivas para α-actinina es 25 x 105. Abreviaturas; SSC = dispersión lateral, PI+ = yoduro de propidio positivo. (B) Estrategia representativa para el análisis de viabilidad tanto en fresco, descongelado, el control positivo (Triton-X) como el control negativo (sin teñir). Haga clic aquí para descargar este archivo.
Discusión
El descubrimiento de fármacos cardíacos se ve obstaculizado por la dependencia de modelos animales y celulares no humanos con un rendimiento inadecuado y fidelidad fisiológica para realizar lecturas con precisión. La biología de hiPSC-CM junto con la instrumentación HT y las sondas fisiológicas tiene el potencial de reintroducir modelos humanos en las primeras etapas del modelado de enfermedades cardíacas y el descubrimiento de fármacos. Desarrollamos un método de generación de tejido cardíaco 3D que produce CS funcionales y de alta calidad para una plataforma óptima de modelado de enfermedades cardíacas y detección de fármacos. Además, la combinación de la tecnología esferoide en sistemas de biorreactores 3D para la producción industrial de EV permite un paso necesario hacia la traducción clínica de la terapia basada en EV. El método descrito aquí se basa en varios factores cruciales y es una variante de los protocolos existentes 9,10,28,29. Estos métodos incluyen: 1) la generación de construcciones de tejido 3D, 2) el número de células y el momento óptimos antes de la detección, 3) mejorar la sensibilidad y la capacidad de alto rendimiento de los instrumentos, y 4) poder congelar los esferoides antes de cualquier análisis funcional. A diferencia de los protocolos descritos anteriormente, el protocolo propuesto describe la generación de hasta 1.500 esferoides por día y la idoneidad para HTS. El análisis convencional de cien compuestos sobre 6 x 0,5 dosis logarítmicas para 10 réplicas utilizando sistemas existentes de imágenes de calcio de 96 pocillos o tejidos cardíacos de ingeniería multiplexada de 24 pocillos requiere aproximadamente 500 millones a 3 mil millones de hiPSC-CM31,32. La aplicación propuesta hace que los exámenes cardíacos sean menos costosos y efectivos en el tiempo en comparación con los sistemas convencionales, ya que las placas de 96 pocillos requerían solo el 10% de la densidad de siembra en comparación con el método descrito. Además, en comparación con protocolos anteriores, como el método de gota colgante, la generación de esferoides por autoagregación en placas de fijación ultra bajas permite obtener imágenes automatizadas de alta calidad de microtejidos individuales33.
Este pequeño modelo 3D imita el fenotipo biológico y fisiológico del entorno cardiovascular in vivo . Como se demostró anteriormente, los transitorios de calcio aumentan dramáticamente en las construcciones de tejido cardíaco 3D en comparación con los cultivos celulares monocapa2D 34.
A continuación, descubrimos que la densidad de siembra y el tiempo de cultivo adecuado también son factores críticos para una detección exitosa de CS. Las densidades de 10K-20K hiPSC-CMs por esferoide y cribado entre las semanas 2-3 después de la generación fueron óptimas, mientras que los esferoides demasiado pequeños o demasiado viejos muestran un manejo alterado del calcio (Figura 2 y Figura 3). Por lo tanto, es importante mantener las densidades de siembra lo más consistentes posible, ya que el tamaño influye en los parámetros funcionales. Además, aunque este método óptico proporciona buenos resultados para cultivos 3D vivos como un tejido completo, la obtención de datos dentro de esferoides más grandes a nivel (sub) celular es un desafío sin depender de métodos histológicos que consumen mucho tiempo. Recientemente, se han publicado varios enfoques que utilizan la "compensación óptica", que permite la adquisición de esferoides 3D completos con la oportunidad de cuantificación de marcadores de una sola célula. Aquí, adaptamos un protocolo de 3 días desde la recolección de CS hasta el análisis de imágenes, que está optimizado para imágenes 3D utilizando microscopía confocal29 (Figura 1C y Figura 4D).
Por último, con el aumento de las aplicaciones de tejido cardíaco 3D y las aplicaciones comerciales, la demanda de almacenamiento a largo plazo y biobancos específicos para pacientes de varios donantes está aumentando. La criopreservación es una estrategia efectiva para generar placas HTS a partir de múltiples lotes a lo largo del tiempo. La congelación de hiPSC-CMs ha sido descrita previamente y no es diferente en comparación con otros tipos de células cultivadas 10,35,36. Recientemente, se han descrito enfoques para congelar placas con células2D 37. Aquí, encontramos que el kit de criopreservación PSC es la condición más óptima en comparación con otros tres (datos no mostrados) y utilizamos este medio para la congelación eficiente de esferoides. Después de la criopreservación, la viabilidad sigue siendo alta (Figura 4B, C), pero las propiedades electrofisiológicas de los CS se ven afectadas y se requiere un período de incubación después de la descongelación. De hecho, 1 semana después de la descongelación, los CS mostraron una actividad espontánea de batido y manejo del calcio. Sin embargo, se ha descrito que los hiPSC-CM frescos y recuperados no siempre muestran propiedades moleculares y fisiológicas idénticas38. Esta limitación debe considerarse cuando se utilizan hiPSC-CM criopreservados para evaluar lecturas cardíacas inducidas por fármacos. Además, aunque modulamos eficazmente el número de células por esferoide y el momento óptimo de la imagen transitoria de calcio, los esferoides cardíacos podrían mejorarse mezclando células cardiomiocitos derivadas de hiPSC con células endoteliales, fibroblastos, uniones célula-célula y matrices extracelulares, como quitosano, colágeno IV, fibronectina, matrigel o laminina, imitando el entorno cardíaco in vivo 39, 40. En general, proponemos un protocolo paso a paso para generar de manera eficiente CS que sean adecuados para aplicaciones posteriores, como el modelado de enfermedades y la detección de medicamentos HT.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Nos gustaría reconocer a VALA sciences por el paquete de software Cyteseer y la optimización del análisis automatizado de calcio 3D. Deseamos agradecer el apoyo de la fundación PLN (RM). P.A.D. y F.S. son compatibles con CUREPLaN Leducq. J.P.G.S. cuenta con el apoyo de H2020-EVICARE (#725229) del Consejo Europeo de Investigación (ERC). J.W.B. cuenta con el apoyo de UMC Utrecht Clinical Fellowship, Netherlands Heart Institute Fellowship y CVON-Dosis young talent grant; Netherlands Heart Foundation (CVON-Dosis 2014-40). N.C. cuenta con el apoyo del Programa de Gravitación "Regeneración Impulsada por Materiales" de la Organización Holandesa para la Investigación Científica (RegmedXB #024.003.013), y las Acciones Marie Skłodowska-Curie (Acuerdo de subvención RESCUE #801540). V.S.-P. cuenta con el apoyo del Fondo de la Alianza (UMCU, UU, TU/e). A.v.M. cuenta con el apoyo del proyecto financiado con fondos europeos BRAVE (H2020, ID:874827)
Materiales
| Name | Company | Catalog Number | Comments |
| 24 wells suspenion plate | Corning | 3738 | |
| 96 wells Ultra-Low Attachment Multiple Well Plate | Corning | CLS3474-24EA | |
| Albumax | Thermo Fisher Scientific | 11020021 | |
| Anti-α-Actinin (Sarcomeric) antibody | Sigma-Aldrich | A7811 | Dilution: 1:200 |
| Anti-Cardiac Troponin T antibody (ab45932) | Abcam | ab45932 | Dilution: 1:200 |
| Ascorbic acid | Sigma-Aldrich | A8960 | |
| B-27 supplement | Thermo Fisher Scientific | 17504-044 | |
| Biotin | Sigma-Aldrich | B4639 | |
| Bovine serum albumin fraction V (BSA) | Roche | 10735086001 | |
| Cal-520, AM | Abcam | ab171868 | |
| Confocal microscope | Leica | DMi8 | |
| Confocal microscope software | Leica | Las X | |
| Conical tubes 15 mL | Greiner Bio-One | 5618-8271 | |
| Creatine monohydrate | Sigma-Aldrich | C3630 | |
| DAPI | Thermo Fisher Scientific | D3571 | Concentration: 1 µg/mL |
| DMEM no glucose | Thermo Fisher Scientific | 11966025 | |
| EDTA | Thermo Fisher Scientific | 15575020 | |
| Fructose | Sigma-Aldrich | 76050771.05 | |
| Glucose | Sigma-Aldrich | G7021 | |
| Glycerol | Boom | 76050771.05 | |
| Goat anti-mouse Alexa Fluor 488 | Invitrogen | A11029 | Dilution: 1:500 |
| Goat anti-rabbit Alexa Fluor 568 | Invitrogen | A11011 | Dilution: 1:500 |
| Horizontal shaker | IKA | 4003000 | |
| Human induced pluripotent stem cell lines | (Stanford Cardiovascular Institute (S-CVI) Biobank) | CVI-273 (control 1) | |
| Human induced pluripotent stem cell lines | Germany | 141 (control 2) 144 (control 3) | |
| Hydrochloric acid (HCl) | Ajax Firechem | 265.2.5L-PL | 10 M stock solution, corrosive |
| Isotype control, FITC mouse IgM κ isotype | BD | 556652 | |
| KnockOut Serum Replacement | Thermo Fisher Scientific | 10828 | Protect from light |
| L-carnitine | Sigma-Aldrich | C0283 | |
| Myocyte calcium and contractility system | Leica | Thunder, DMi8 | |
| Non essential amino acids (NEAA) | Thermo Fisher Scientific | 11140 | |
| Paraformaldehyde solution 4% in 1x PBS, pH 7.0–7.6 | Santa Cruz | SC281692 | Hazardous |
| PBS, pH 7.4 | Thermo Fisher Scientific | 10010023 | |
| Penicillin/streptomycin | Thermo Fisher Scientific | 15140 | |
| PES Membrane Vacuum Filter system | Corning | 431097 | |
| PI/RNase Staining Solution | Invitrogen | F10797 | Dilution: 1:1000 |
| Pluronic F-127 | Sigma-Aldrich | P2443 | |
| PSC Cryopreservation Kit | Thermo Fisher Scientific | A2644601 | |
| RevitaCell | Thermo Fisher Scientific | A2644501 | |
| RPMI 1640 medium | Thermo Fisher Scientific | 11875 | |
| Silicone Elastomer Kit | SYLGARD | 184 | |
| Sodium dodecyl sulfate solution (10%) | Sigma-Aldrich | 71736 | |
| Sodium L-Lactate | Sigma-Aldrich | 71718 | |
| Taurine | Sigma-Aldrich | T0625 | |
| Tris Fisher | Scientific | 11486631 | |
| Triton X-100 | Merck | X100-1L | Hazardous |
| Trypan blue solution, 0.4% | Thermo Fisher Scientific | 15250061 | |
| TrypLE Select Enzyme (10x) | Thermo Fisher Scientific | A1217701 | |
| Tween-20 | Sigma-Aldrich | P1379 | |
| Urea | Sigma-Aldrich | 51456 | |
| Vitamin B12 | Sigma-Aldrich | V6629 | |
| Y-27632 dihydrochloride (Rho-kinase inhibitor) | Tocris | 1254 | Protect from light |
Referencias
- Burridge, P. W., et al. Chemically defined and small molecule-based generation of human cardiomyocytes. Nature Methods. 11 (8), 855-860 (2014).
- Kattman, S. J., et al. Stage-specific optimization of activin/nodal and BMP signaling promotes cardiac differentiation of mouse and human pluripotent stem cell lines. Cell Stem Cell. 8 (2), 228-240 (2011).
- Lian, X., et al. Robust cardiomyocyte differentiation from human pluripotent stem cells via temporal modulation of canonical Wnt signaling. Proceedings of the National Academy of Sciences. 109 (27), 1848-1857 (2012).
- Paige, S. L., et al. Endogenous Wnt/beta-catenin signaling is required for cardiac differentiation in human embryonic stem cells. PLoS One. 5 (6), 11134 (2010).
- Gintant, G., et al. Use of human induced pluripotent stem cell-derived cardiomyocytes in preclinical cancer drug cardiotoxicity testing: A scientific statement from the American Heart Association. Circulation Research. 125 (10), 75-92 (2019).
- Ahmed, R. E., et al. A brief review of current maturation methods for human induced pluripotent stem cells-derived cardiomyocytes. Frontiers in Cell and Developmental Biology. 19 (8), 178 (2020).
- Liu, C., et al. Generating 3D human cardiac constructs from pluripotent stem cells. EBioMedicine. 76, 103813 (2022).
- Musunuru, K., et al. Induced pluripotent stem cells for cardiovascular disease modeling and precision medicine: A scientific statement from the American Heart Association. Circulation: Genomic and Precision Medicine. 11 (1), 000043 (2018).
- Buikema, J. W., et al. Wnt activation and reduced cell-cell contact synergistically induce massive expansion of functional human iPSC-derived cardiomyocytes. Cell Stem Cell. 27 (1), 50-63 (2020).
- Maas, R. G. C., et al. Massive expansion and cryopreservation of functional human induced pluripotent stem cell-derived cardiomyocytes. Cell STAR Protocols. 2 (1), 100334 (2021).
- Tremblay, C., et al. A new construction technique for tissue-engineered heart valves using the self-assembly method. Tissue Engineering Part C: Methods. 20 (11), 905-915 (2014).
- Lewis-Israeli, Y. R., et al. Self-assembling human heart organoids for the modeling of cardiac development and congenital heart disease. Nature Communications. 12 (1), 5142 (2021).
- Goldfracht, I., et al. Engineered heart tissue models from hiPSC-derived cardiomyocytes and cardiac ECM for disease modeling and drug testing applications. Acta Biomaterialia. 1 (92), 145-159 (2019).
- Fleischer, S., et al. Comprehensive human stem cell differentiation in a 2D and 3D mode to cardiomyocytes for long-term cultivation and multiparametric monitoring on a multimodal microelectrode array setup. Biosensors and Bioelectronics. 126, 624-631 (2019).
- Branco, M. A., et al. Transcriptomic analysis of 3D cardiac differentiation of human induced pluripotent stem cells reveals faster cardiomyocyte maturation compared to 2D culture. Science Reports. 9 (1), 9229 (2019).
- Ergir, E., et al. Generation and maturation of human iPSC-derived cardiac organoids in long term culture. bioRxiv. , (2022).
- Lemoine, M. D., et al. Human iPSC-derived cardiomyocytes cultured in 3D engineered heart tissue show physiological upstroke velocity and sodium current density. Scienctific Reports. 7 (1), 5464 (2017).
- Kofron, C. M., et al. A predictive in vitro risk assessment platform for pro-arrhythmic toxicity using human 3D cardiac microtissues. Science Reports. 11 (1), 10228 (2021).
- Giacomelli, E., et al. Human-iPSC-derived cardiac stromal cells enhance maturation in 3D cardiac microtissues and reveal non-cardiomyocyte contributions to heart disease. Cell Stem Cell. 26 (6), 862-879 (2020).
- Richards, D. J., et al. Human cardiac organoids for the modelling of myocardial infarction and drug cardiotoxicity. Nature Biomedical Engineering. 4 (4), 446-462 (2020).
- Tenreiro, M. F., et al. Next generation of heart regenerative therapies: progress and promise of cardiac tissue engineering. npj Regenerative Medicine. 6 (1), 30 (2021).
- Hansen, A., et al. Development of a drug screening platform based on engineered heart tissue. Circulation Research. 107 (1), 35-44 (2010).
- McDermott-Roe, C., et al. Investigation of a dilated cardiomyopathy-associated variant in BAG3 using genome-edited iPSC-derived cardiomyocytes. Journal of Clinical Investigation Insight. 4 (22), 128799 (2019).
- National Library of Medicine (U.S.). Safety and efficacy of induced pluripotent stem cell-derived engineered human myocardium as biological ventricular assist tissue in terminal heart failure. National Library of Medicine. , (2020).
- Ronaldson-Bouchard, K., et al. Advanced maturation of human cardiac tissue grown from pluripotent stem cells. Nature. 556 (7700), 239-243 (2018).
- Oh, J. G., et al. Generation of ventricular-like HiPSC-derived cardiomyocytes and high-quality cell preparations for calcium handling characterization. Journal of Visualized Experiments. 155, 60135 (2020).
- Lian, X., et al. Directed cardiomyocyte differentiation from human pluripotent stem cells by modulating Wnt/β-catenin signaling under fully defined conditions. Nature Protocols. 8 (1), 162-175 (2013).
- Feyen, D. A. M., et al. Metabolic maturation media improve physiological function of human iPSC-derived cardiomyocytes. Cell Reports. 32 (3), 107925 (2020).
- van Ineveld, R. L., et al. Single-cell resolution three-dimensional imaging of intact organoids. Journal of Visualized Experiments. (160), e60709 (2020).
- Guo, Y., Pu, W. T. Cardiomyocyte maturation: New phase in development. Circulation Research. 126 (8), 1086-1106 (2020).
- Ding, B., et al. Three-dimensional renal organoids from whole kidney cells: Generation, optimization, and potential application in nephrotoxicology in vitro. Cell Transplantation. 29, 963689719897066 (2020).
- Denning, C., et al. Cardiomyocytes from human pluripotent stem cells: From laboratory curiosity to industrial biomedical platform. Biochimica Biophysica Acta. 1863, 1728-1748 (2016).
- Amaral, R. L. F., et al. Comparative analysis of 3D bladder tumor spheroids obtained by forced floating and hanging drop methods for drug screening. Frontiers in Physiology. 8, 605 (2017).
- Daily, N. J., et al. Improving cardiac action potential measurements: 2D and 3D cell culture. Journal of Bioengineering and Biomedical Science. 5 (3), 168 (2015).
- Preininger, M. K., et al. Cryopreservation of human pluripotent stem cell-derived cardiomyocytes: Strategies, challenges, and future directions. Advances in Experimental Medicine and Biology. 951, 123-135 (2016).
- Kim, Y. Y., et al. Cryopreservation of human embryonic stem cells derived-cardiomyocytes induced by BMP2 in serum-free condition. Reproductive Science. 18 (3), 252-360 (2011).
- Daily, M. I., et al. Cryopreservation of primary cultures of mammalian somatic cells in 96-well plates benefits from control of ice nucleation. Cryobiology. 93, 62-69 (2020).
- Zhang, J. Z., et al. Effects of cryopreservation on human induced pluripotent stem cell-derived cardiomyocytes for assessing drug safety response profiles. Stem Cell Reports. 16 (1), 168-181 (2021).
- Yeh, H. -. Y., et al. The calcium-dependent regulation of spheroid formation and cardiomyogenic differentiation for MSCs on chitosan membranes. Biomaterials. 33 (35), 8943-8954 (2012).
- Scalise, M., et al. From spheroids to organoids: The next generation of model systems of human cardiac regeneration in a dish. International Journal of Molecular Sciences. 22 (24), 13180 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados