Method Article
Segmentación basada en aprendizaje profundo de tomogramas crioelectrónicos
En este artículo
Resumen
Este es un método para entrenar una U-Net multicorte para la segmentación multiclase de tomogramas crioelectrónicos utilizando una porción de un tomograma como entrada de entrenamiento. Describimos cómo inferir esta red a otros tomogramas y cómo extraer segmentaciones para análisis adicionales, como el promedio de subtomogramas y el rastreo de filamentos.
Resumen
La criotomografía electrónica (crio-ET) permite a los investigadores obtener imágenes de las células en su estado nativo e hidratado a la resolución más alta posible actualmente. Sin embargo, la técnica tiene varias limitaciones que hacen que el análisis de los datos que genera requiera mucho tiempo y sea difícil. La segmentación manual de una sola tomograma puede tomar de horas a días, pero un microscopio puede generar fácilmente 50 o más tomografías al día. Existen programas actuales de segmentación de aprendizaje profundo para crio-ET, pero se limitan a segmentar una estructura a la vez. Aquí, las redes neuronales convolucionales U-Net de múltiples cortes se entrenan y aplican para segmentar automáticamente múltiples estructuras simultáneamente dentro de los criotomogramas. Con un preprocesamiento adecuado, estas redes se pueden inferir robustamente a muchos tomogramas sin la necesidad de entrenar redes individuales para cada tomograma. Este flujo de trabajo mejora drásticamente la velocidad con la que se pueden analizar los tomogramas crioelectrónicos al reducir el tiempo de segmentación a menos de 30 minutos en la mayoría de los casos. Además, las segmentaciones se pueden utilizar para mejorar la precisión del trazado de filamentos dentro de un contexto celular y para extraer rápidamente las coordenadas para el promedio de subtomograma.
Introducción
Los desarrollos de hardware y software en la última década han resultado en una "revolución de resolución" para la criomicroscopía electrónica (crio-EM)1,2. Con detectores mejores y más rápidos3, software para automatizar la recopilación de datos4,5 y avances de aumento de señal como las placas de fase6, la recopilación de grandes cantidades de datos crio-EM de alta resolución es relativamente sencilla.
Cryo-ET ofrece una visión sin precedentes de la ultraestructura celular en un estado nativo e hidratado 7,8,9,10. La limitación principal es el grosor de la muestra, pero con la adopción de métodos como el fresado de haz de iones enfocado (FIB), donde las muestras gruesas de células y tejidos se adelgazan para la tomografía11, el horizonte de lo que se puede obtener una imagen con crio-ET se expande constantemente. Los microscopios más nuevos son capaces de producir más de 50 tomogramas al día, y se prevé que esta tasa aumente debido al desarrollo de esquemas rápidos de recolección de datos12,13. El análisis de las grandes cantidades de datos producidos por cryo-ET sigue siendo un cuello de botella para esta modalidad de imagen.
El análisis cuantitativo de la información tomográfica requiere que primero se anote. Tradicionalmente, esto requiere la segmentación de la mano por parte de un experto, lo que lleva mucho tiempo; Dependiendo de la complejidad molecular contenida en el crio-tomograma, puede tomar horas o días de atención dedicada. Las redes neuronales artificiales son una solución atractiva para este problema, ya que pueden ser entrenadas para hacer la mayor parte del trabajo de segmentación en una fracción del tiempo. Las redes neuronales convolucionales (CNN) son especialmente adecuadas para tareas de visión artificial14 y recientemente han sido adaptadas para el análisis de tomogramas crioelectrónicos15,16,17.
Las CNN tradicionales requieren muchos miles de muestras de entrenamiento anotadas, lo que a menudo no es posible para las tareas de análisis de imágenes biológicas. Por lo tanto, la arquitectura U-Net se ha destacado en este espacio18 porque se basa en el aumento de datos para entrenar con éxito la red, minimizando la dependencia de grandes conjuntos de entrenamiento. Por ejemplo, una arquitectura U-Net puede ser entrenada con sólo unas pocas rebanadas de una sola tomograma (cuatro o cinco rebanadas) y robustamente inferida a otros tomogramas sin volver a entrenar. Este protocolo proporciona una guía paso a paso para entrenar arquitecturas de redes neuronales U-Net para segmentar criotomogramas de electrones dentro de Dragonfly 2022.119.
Dragonfly es un software desarrollado comercialmente utilizado para la segmentación y análisis de imágenes 3D mediante modelos de aprendizaje profundo, y está disponible gratuitamente para uso académico (se aplican algunas restricciones geográficas). Tiene una interfaz gráfica avanzada que permite a un no experto aprovechar al máximo los poderes del aprendizaje profundo tanto para la segmentación semántica como para la eliminación de ruido de imágenes. Este protocolo demuestra cómo preprocesar y anotar tomogramas crioelectrónicos dentro de Dragonfly para entrenar redes neuronales artificiales, que luego se pueden inferir para segmentar rápidamente grandes conjuntos de datos. Además, discute y demuestra brevemente cómo usar datos segmentados para análisis adicionales, como el rastreo de filamentos y la extracción de coordenadas para el promedio de subtogramas.
Protocolo
NOTA: Dragonfly 2022.1 requiere una estación de trabajo de alto rendimiento. Las recomendaciones del sistema se incluyen en la tabla de materiales junto con el hardware de la estación de trabajo utilizada para este protocolo. Todos los tomogramas utilizados en este protocolo se agrupan 4x de un tamaño de píxel de 3.3 a 13.2 ang / pix. Las muestras utilizadas en los resultados representativos se obtuvieron de una empresa (ver la Tabla de materiales) que sigue las pautas de cuidado de los animales que se alinean con los estándares éticos de esta institución. El tomograma utilizado en este protocolo y el ROI múltiple que se generó como entrada de entrenamiento se han incluido como un conjunto de datos incluido en el Archivo Suplementario 1 (que se puede encontrar en https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct) para que el usuario pueda seguir los mismos datos si lo desea. Dragonfly también alberga una base de datos de acceso abierto llamada Infinite Toolbox donde los usuarios pueden compartir redes entrenadas.
1. Configuración
- Cambio del espacio de trabajo predeterminado:
- Para cambiar el espacio de trabajo para que refleje el utilizado en este protocolo, en el lado izquierdo del panel Principal, desplácese hacia abajo hasta la sección Propiedades de vista de escena y anule la selección de Mostrar leyendas. Desplácese hacia abajo hasta la sección Diseño y seleccione la vista Escena única y Cuatro vistas iguales .
- Para actualizar la unidad predeterminada, vaya a Archivo | Preferencias. En la ventana que se abre, cambie la unidad predeterminada de milímetros a nanómetros.
- Combinaciones de teclas predeterminadas útiles:
- Presione Esc para mostrar el punto de mira en las vistas 2D y permitir la rotación del volumen 3D en la vista 3D. Presione X para ocultar el punto de mira en las vistas 2D y permitir la traducción 2D y la traducción de volumen 3D en la vista 3D.
- Desplácese sobre el punto de mira para ver pequeñas flechas en las que se puede hacer clic y arrastrar para cambiar el ángulo del plano de visión en las otras vistas 2D.
- Presione Z para ingresar el estado de zoom en ambas vistas, lo que permite a los usuarios hacer clic y arrastrar a cualquier lugar para acercar y alejar.
- Haga doble clic en una vista de la escena de cuatro vistas para enfocar solo esa vista; Haga doble clic de nuevo para volver a las cuatro vistas.
- Guarde periódicamente el progreso exportando todo en la pestaña Propiedades como un objeto ORS para facilitar la importación. Seleccione todos los objetos de la lista y haga clic derecho en Exportar | Como objeto ORS. Asigne un nombre al archivo y guárdelo. Alternativamente, vaya a Archivo | Guardar sesión. Para utilizar la función de guardado automático en el software, habilítela a través de Archivo | Preferencias | Guardado automático.
2. Importación de imágenes
- Para importar imágenes, vaya a Archivo | Importar archivos de imagen. Haga clic en Agregar, desplácese hasta el archivo de imagen y haga clic en Abrir | Siguiente | Terminar.
NOTA: El software no reconoce los archivos .rec. Todos los tomogramas deben tener el sufijo .mrc. Si utiliza los datos proporcionados, vaya a Archivo | Importar objeto(s). Desplácese hasta el archivo Training.ORSObject , haga clic en Abrir y, a continuación, haga clic en Aceptar.
3. Preprocesamiento (Figura 1.1)
- Cree una escala de intensidad personalizada (utilizada para calibrar las intensidades de imagen en los conjuntos de datos). Ir a Utilidades | Gestor de unidades de dimensión. En la parte inferior izquierda, haga clic en + para crear una nueva unidad de dimensión.
- Elija una función de alta intensidad (brillante) y baja intensidad (oscura) que esté en todos los tomogramas de interés. Asigne a la unidad un nombre y una abreviatura (por ejemplo, para esta escala, establezca las cuentas fiduciales en 0.0 Intensidad estándar y el fondo en 100.0). Guarde la unidad de dimensión personalizada.
NOTA: Una escala de intensidad personalizada es una escala arbitraria que se crea y se aplica a los datos para garantizar que todos los datos estén en la misma escala de intensidad a pesar de haberse recopilado en diferentes momentos o en diferentes equipos. Elija las características claras y oscuras que mejor representen el rango en el que se encuentra la señal. Si no hay fiduciarios en los datos, simplemente elija la característica más oscura que se segmentará (la región más oscura de la proteína, por ejemplo). - Para calibrar imágenes a la escala de intensidad personalizada, haga clic con el botón derecho en el conjunto de datos en la columna Propiedades en el lado derecho de la pantalla y seleccione Calibrar escala de intensidad. En la pestaña Principal en el lado izquierdo de la pantalla, desplácese hacia abajo hasta la sección Sonda . Usando la herramienta de sonda circular con un diámetro apropiado, haga clic en algunos lugares en la región de fondo del tomograma y registre el número promedio en la columna Intensidad sin procesar ; repita el procedimiento para los marcadores fiduciarios y, a continuación, haga clic en Calibrar. Si es necesario, ajuste el contraste para que las estructuras vuelvan a ser visibles con la herramienta Área de la sección Nivelación de ventana de la ficha Principal.
- Filtrado de imágenes:
NOTA: El filtrado de imágenes puede reducir el ruido y aumentar la señal. Este protocolo utiliza tres filtros que están integrados en el software, ya que funcionan mejor para estos datos, pero hay muchos filtros disponibles. Una vez establecido un protocolo de filtrado de imágenes para los datos de interés, será necesario aplicar exactamente el mismo protocolo a todos los tomogramas antes de la segmentación.- En la pestaña principal del lado izquierdo, desplácese hacia abajo hasta el Panel de procesamiento de imágenes. Haga clic en Avanzado y espere a que se abra una nueva ventana. En el panel Propiedades , seleccione el conjunto de datos que desea filtrar y hágalo visible haciendo clic en el icono del ojo situado a la izquierda del conjunto de datos.
- En el panel Operaciones , utilice el menú desplegable para seleccionar Ecualización de histograma (en la sección Contraste ) para la primera operación. Seleccione Agregar operación | Gaussiano (en la sección Suavizado ). Cambie la dimensión del kernel a 3D.
- Agregar una tercera operación; a continuación, seleccione Quitar enfoque (en la sección Enfoque ). Deje la salida para este. Aplíquelo a todos los sectores y deje que se ejecute el filtrado, luego cierre la ventana Procesamiento de imágenes para volver a la interfaz principal.
4. Crear datos de entrenamiento (Figura 1.2)
- Identifique el área de entrenamiento ocultando primero el conjunto de datos sin filtrar haciendo clic en el icono del ojo situado a la izquierda en el panel Propiedades de datos . A continuación, muestre el conjunto de datos recién filtrado (que se denominará automáticamente DataSet-HistEq-Gauss-Unsharp). Utilizando el conjunto de datos filtrado, identifique una subregión del tomograma que contenga todas las características de interés.
- Para crear un cuadro alrededor de la región de interés, en el lado izquierdo, en la pestaña principal , desplácese hacia abajo hasta la categoría Formas y seleccione Crear un cuadro. Mientras esté en el panel Cuatro Vistas , utilice los diferentes planos 2D para ayudar a guiar/arrastrar los bordes de la caja para encerrar solo la región de interés en todas las dimensiones. En la lista de datos, seleccione la región Cuadro y cambie el color del borde para facilitar la visualización haciendo clic en el cuadrado gris junto al símbolo del ojo.
NOTA: El tamaño de parche más pequeño para un U-Net 2D es de 32 x 32 píxeles; 400 x 400 x 50 píxeles es un tamaño de caja razonable para comenzar. - Para crear un ROI múltiple, en el lado izquierdo, seleccione la pestaña Segmentación | Nuevo y marque Crear como Multi-ROI. Asegúrese de que el número de clases corresponde al número de características de interés + una clase de fondo. Asigne un nombre a los datos de entrenamiento multi-ROI y asegúrese de que la geometría corresponde al conjunto de datos antes de hacer clic en Aceptar.
- Segmentación de los datos de entrenamiento
- Desplácese por los datos hasta dentro de los límites de la región en caja. Seleccione Multi-ROI en el menú de propiedades de la derecha. Haga doble clic en el primer nombre de clase en blanco en el ROI múltiple para asignarle un nombre.
- Pinta con el pincel 2D. En la pestaña de segmentación de la izquierda, desplázate hacia abajo hasta Herramientas 2D y selecciona un pincel circular. Luego, seleccione Adaptive Gaussian o Local OTSU en el menú desplegable. Para pintar, mantenga presionada la tecla izquierda y haga clic. Para borrar, mantenga presionada la tecla Mayús a la izquierda y haga clic.
NOTA: El pincel reflejará el color de la clase seleccionada actualmente. - Repita el paso anterior para cada clase de objeto en el ROI múltiple. Asegúrese de que todas las estructuras dentro de la región en caja estén completamente segmentadas o serán consideradas en segundo plano por la red.
- Cuando se hayan etiquetado todas las estructuras, haga clic con el botón derecho en la clase Background en Multi-ROI y seleccione Agregar todos los vóxeles sin etiquetar a la clase.
- Cree un nuevo ROI de clase única llamado Máscara. Asegúrese de que la geometría esté establecida en el dataset filtrado y, a continuación, haga clic en Aplicar. En la pestaña de propiedades de la derecha, haga clic con el botón derecho en el cuadro y seleccione Agregar al ROI. Agrégalo al ROI de la máscara.
- Para recortar los datos de entrenamiento con la máscara, en la pestaña Propiedades , seleccione tanto el ROI múltiple de datos de entrenamiento como el ROI de la máscara manteniendo presionada la tecla Ctrl y haciendo clic en cada uno. A continuación, haga clic en Intersecar debajo de la lista de propiedades de datos en la sección denominada Operaciones booleanas. Asigne al nuevo conjunto de datos el nombre Entrada de entrenamiento recortada y asegúrese de que la geometría corresponde al conjunto de datos filtrado antes de hacer clic en Aceptar.
5. Uso del asistente de segmentación para el entrenamiento iterativo (Figura 1.3)
- Para importar los datos de entrenamiento al asistente de segmentación, primero haga clic con el botón secundario en el conjunto de datos filtrado en la pestaña Propiedades y, a continuación, seleccione la opción Asistente para segmentación . Cuando se abra una nueva ventana, busque la pestaña de entrada en el lado derecho. Haga clic en Importar fotogramas desde un ROI múltiple y seleccione la entrada de entrenamiento recortada.
- (Opcional) Cree un marco de retroalimentación visual para monitorear el progreso del entrenamiento en tiempo real.
- Seleccione un marco de los datos que no esté segmentado y haga clic en + para agregarlo como un nuevo marco. Haga doble clic en la etiqueta mixta situada a la derecha del marco y cámbiela a Supervisión.
- Para generar un nuevo modelo de red neuronal, en el lado derecho de la pestaña Modelos , haga clic en el botón + para generar un nuevo modelo. Seleccione U-Net en la lista y, a continuación, en Dimensión de entrada, seleccione 2,5D y 5 sectores, y, a continuación, haga clic en Generar.
- Para entrenar la red, haga clic en Entrenar en la parte inferior derecha de la ventana SegWiz .
NOTA: El entrenamiento se puede detener temprano sin perder el progreso. - Para usar la red entrenada para segmentar nuevas tramas, cuando se complete la capacitación de U-Net, cree una nueva trama y haga clic en Predecir (abajo a la derecha). A continuación, haga clic en la flecha hacia arriba en la parte superior derecha del fotograma previsto para transferir la segmentación al fotograma real.
- Para corregir la predicción, pulse Ctrl y haga clic en dos clases para cambiar los píxeles segmentados de una a otra. Seleccione ambas clases y pinte con el pincel para pintar solo los píxeles que pertenecen a cualquiera de las clases. Corrija la segmentación en al menos cinco nuevos fotogramas.
NOTA: Pintar con la herramienta de pincel mientras ambas clases están seleccionadas significa que en lugar de borrar con mayús y clic, como lo hace normalmente, convertirá los píxeles de la primera clase a la segunda. Ctrl y haga clic logrará lo contrario. - Para el entrenamiento iterativo, haga clic en el botón Entrenar nuevamente y permita que la red entrene más durante otras 30-40 épocas más, momento en el que detenga el entrenamiento y repita los pasos 4.5 y 4.6 para otra ronda de entrenamiento.
NOTA: De esta manera, un modelo se puede entrenar y mejorar iterativamente utilizando un único conjunto de datos. - Para publicar la red, cuando esté satisfecho con su rendimiento, salga del Asistente para segmentación. En el cuadro de diálogo que aparece automáticamente preguntando qué modelos publicar (guardar), seleccione la red correcta, asígnele un nombre y, a continuación, publíquela para que la red esté disponible para su uso fuera del asistente de segmentación.
6. Aplicar la red (Figura 1.4)
- Para aplicar primero al tomograma de entrenamiento, seleccione el conjunto de datos filtrado en el panel Propiedades . En el panel Segmentación de la izquierda, desplázate hacia abajo hasta la sección Segmento con IA . Asegúrese de que está seleccionado el conjunto de datos correcto, elija el modelo que se acaba de publicar en el menú desplegable y, a continuación, haga clic en Segmento | Todas las rebanadas. Como alternativa, seleccione Vista previa para ver una vista previa de un solo segmento de la segmentación.
- Para aplicarlo a un conjunto de datos de inferencia, importe el nuevo tomograma. Preproceso según el paso 3 (figura 1.1). En el panel Segmentación, vaya a la sección Segmento con IA. Asegúrese de que el tomograma recién filtrado es el conjunto de datos seleccionado, elija el modelo entrenado previamente y haga clic en Segmento | Todas las rebanadas.
7. Manipulación y limpieza de segmentación
- Limpie rápidamente el ruido eligiendo primero una de las clases que tiene ruido segmentado y la característica de interés. Haga clic con el botón derecho | Islas de proceso | Eliminar por Voxel Count | Seleccione un tamaño de vóxel. Comience poco a poco (~ 200) y aumente gradualmente el conteo para eliminar la mayor parte del ruido.
- Para la corrección de segmentación, pulse Ctrl y haga clic en dos clases para pintar sólo los píxeles que pertenecen a esas clases. Ctrl-clic + arrastrar con las herramientas de segmentación para cambiar los píxeles de la segunda clase a la primera y Mayús y clic + arrastrar para lograr lo contrario. Continúe haciendo esto para corregir rápidamente los píxeles etiquetados incorrectamente.
- Separar los componentes conectados.
- Elige una clase. Haga clic con el botón derecho en una clase en Multi-ROI | Separar componentes conectados para crear una nueva clase para cada componente que no esté conectado a otro componente de la misma clase. Utilice los botones situados debajo de Multi-ROI para combinar fácilmente las clases.
- Exporte el ROI como binario/TIFF.
- Elija una clase en Multi-ROI, luego haga clic con el botón derecho y extraiga la clase como ROI. En el panel de propiedades de arriba, seleccione el nuevo ROI, haga clic con el botón derecho | Exportación | ROI como binario (asegúrese de que la opción para exportar todas las imágenes en un archivo esté seleccionada).
NOTA: Los usuarios pueden convertir fácilmente de tiff a formato mrc utilizando el programa IMOD tif2mrc20. Esto es útil para el trazado de filamentos.
- Elija una clase en Multi-ROI, luego haga clic con el botón derecho y extraiga la clase como ROI. En el panel de propiedades de arriba, seleccione el nuevo ROI, haga clic con el botón derecho | Exportación | ROI como binario (asegúrese de que la opción para exportar todas las imágenes en un archivo esté seleccionada).
8. Generación de coordenadas para el promedio de sub-tomogramas a partir del ROI
- Extraer una clase.
- Haga clic con el botón derecho en Clase que se utilizará para promediar | Extraer clase como ROI. Haga clic con el botón derecho en el ROI de la clase | Componentes conectados | Nuevo Multi-ROI (26 conectados).
- Generar coordenadas.
- Haga clic con el botón derecho en el nuevo Multi-ROI | Generador escalar. Expandir Mediciones básicas con conjunto de datos | marque Centro ponderado de masa X, Y y Z. Seleccione el conjunto de datos y el proceso. Haga clic con el botón derecho en Multi-ROI | Exportar valores escalares. Marque Seleccionar todas las ranuras escalares y, a continuación, Aceptar para generar coordenadas mundiales centroides para cada clase en el ROI múltiple como un archivo CSV.
NOTA: Si las partículas están muy juntas y las segmentaciones se tocan, puede ser necesario realizar una transformación de cuenca para separar los componentes en un ROI múltiple.
- Haga clic con el botón derecho en el nuevo Multi-ROI | Generador escalar. Expandir Mediciones básicas con conjunto de datos | marque Centro ponderado de masa X, Y y Z. Seleccione el conjunto de datos y el proceso. Haga clic con el botón derecho en Multi-ROI | Exportar valores escalares. Marque Seleccionar todas las ranuras escalares y, a continuación, Aceptar para generar coordenadas mundiales centroides para cada clase en el ROI múltiple como un archivo CSV.
9. Transformación de cuencas hidrográficas
- Extraiga la clase haciendo clic con el botón derecho en la clase Multi-ROI que se utilizará para promediar | Extraer clase como ROI. Nombra esta máscara de cuenca de ROI.
- (Opcional) Cerrar agujeros.
- Si las partículas segmentadas tienen agujeros o aberturas, ciérrelos para la cuenca. Haga clic en ROI en Propiedades de datos. En la pestaña Segmentación (a la izquierda), vaya a Operaciones morfológicas y use cualquier combinación de Dilate, Erode y Close necesaria para lograr segmentaciones sólidas sin agujeros.
- Invertir el ROI haciendo clic en el ROI | Copiar objeto seleccionado (debajo de Propiedades de datos). Seleccione el ROI copiado y, en el lado izquierdo, en la pestaña Segmentación , haga clic en Invertir.
- Crear un mapa de distancia haciendo clic con el botón derecho en el ROI invertido | Crear mapeo de | Mapa de distancia. Para su uso posterior, haga una copia del mapa de distancia e invierta (haga clic con el botón derecho | Modificar y transformar | Invertir valores | Aplicar). Asigne a este mapa invertido el nombre Paisaje.
- Crea puntos semilla.
- Oculte el ROI y muestre el mapa de distancia. En la pestaña Segmentación , haga clic en Definir rango y disminuya el rango hasta que solo se resalten unos pocos píxeles en el centro de cada punto y ninguno esté conectado a otro punto. En la parte inferior de la sección Rango , haga clic en Agregar a nuevo. Asigne a este nuevo ROI Seedpoints.
- Realizar la transformación de cuencas hidrográficas.
- Haz clic con el botón derecho en Seedpoints ROI | Componentes conectados | Nuevo Multi-ROI (26 conectados). Haga clic con el botón derecho en el ROI múltiple recién generado | Transformación de cuenca. Seleccione el mapa de distancia denominado Paisaje y haga clic en Aceptar; seleccione el ROI denominado Watershed Mask y haga clic en OK para calcular una transformación de cuenca de cada punto de semilla y separar las partículas individuales en clases separadas en el ROI múltiple. Genere coordenadas como en el paso 8.2.
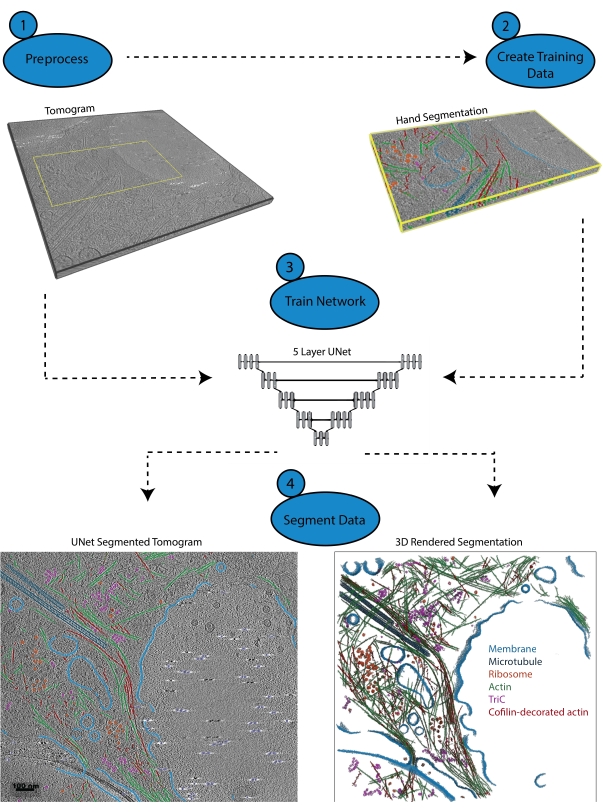
Figura 1: Flujo de trabajo. 1) Preprocesar el tomograma de entrenamiento calibrando la escala de intensidad y filtrando el conjunto de datos. 2) Cree los datos de entrenamiento segmentando a mano una pequeña porción de un tomograma con todas las etiquetas apropiadas que el usuario desea identificar. 3) Usando el tomograma filtrado como entrada y la segmentación manual como salida de entrenamiento, se entrena una U-Net multicorte de cinco capas en el asistente de segmentación. 4) La red entrenada se puede aplicar al tomograma completo para anotarlo y se puede generar una representación 3D de cada clase segmentada. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Siguiendo el protocolo, se entrenó una U-Net de cinco cortes en un solo tomograma (Figura 2A) para identificar cinco clases: membrana, microtúbulos, actina, marcadores fiduciales y antecedentes. La red se entrenó iterativamente un total de tres veces, y luego se aplicó al tomograma para segmentarlo y anotarlo completamente (Figura 2B, C). La limpieza mínima se realizó mediante los pasos 7.1 y 7.2. Los siguientes tres tomogramas de interés (Figura 2D, G, J) se cargaron en el software para su preprocesamiento. Antes de la importación de la imagen, uno de los tomogramas (Figura 2J) requería un ajuste del tamaño de píxel de 17,22 Å/px a 13,3 Å/px, ya que se recogía en un microscopio diferente con un aumento ligeramente diferente. El programa IMOD squeezevol se utilizó para cambiar el tamaño con el siguiente comando:
'squeezevol -f 0.772 inputfile.mrc outputfile.mrc'
En este comando, -f se refiere al factor por el cual alterar el tamaño del píxel (en este caso: 13.3/17.22). Después de la importación, los tres objetivos de inferencia se procesaron previamente de acuerdo con los pasos 3.2 y 3.3, y luego se aplicó la U-Net de cinco sectores. Se volvió a realizar una limpieza mínima. Las segmentaciones finales se muestran en la Figura 2.
Las segmentaciones de microtúbulos de cada tomograma se exportaron como archivos TIF binarios (paso 7.4), se convirtieron a MRC (programa IMOD tif2mrc ) y luego se utilizaron para la correlación de cilindros y el rastreo de filamentos. Las segmentaciones binarias de filamentos dan como resultado un trazado de filamentos mucho más robusto que el rastreo sobre tomogramas. Los mapas de coordenadas del trazado de filamentos (Figura 3) se utilizarán para análisis adicionales, como mediciones de vecinos más cercanos (empaquetamiento de filamentos) y subtomogramas helicoidales promediando filamentos individuales para determinar la orientación de los microtúbulos.
Las redes fallidas o inadecuadamente entrenadas son fáciles de determinar. Una red fallida no podrá segmentar ninguna estructura, mientras que una red inadecuadamente entrenada generalmente segmentará algunas estructuras correctamente y tendrá un número significativo de falsos positivos y falsos negativos. Estas redes pueden corregirse y entrenarse iterativamente para mejorar su rendimiento. El asistente de segmentación calcula automáticamente el coeficiente de similitud de dados de un modelo (llamado puntuación en SegWiz) después de entrenarlo. Esta estadística da una estimación de la similitud entre los datos de entrenamiento y la segmentación de U-Net. Dragonfly 2022.1 también tiene una herramienta incorporada para evaluar el rendimiento de un modelo a la que se puede acceder en la pestaña Inteligencia artificial en la parte superior de la interfaz (consulte la documentación para su uso).
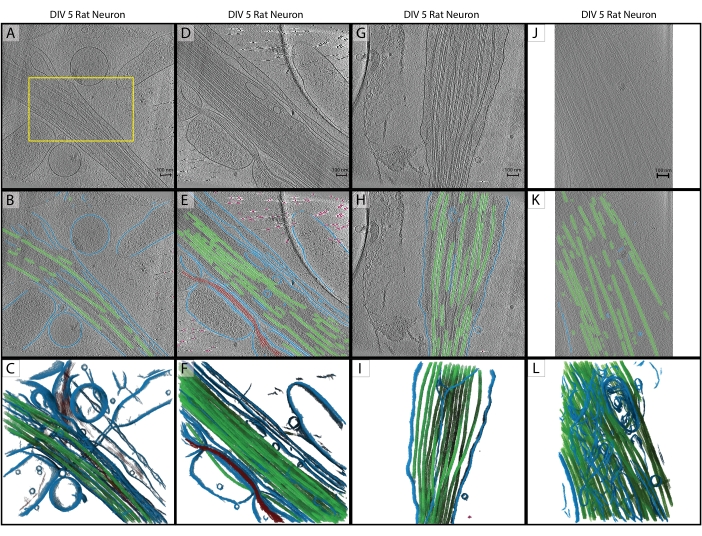
Figura 2: Inferencia. (A-C) Tomografía de entrenamiento original de una neurona de rata del hipocampo DIV 5, recolectada en 2019 en un Titan Krios. Esta es una reconstrucción retroproyectada con corrección CTF en IMOD. (A) El cuadro amarillo representa la región donde se realizó la segmentación de la mano para la entrada de entrenamiento. (B) Segmentación 2D de U-Net después de completar la capacitación. (C) Representación 3D de las regiones segmentadas que muestran membrana (azul), microtúbulos (verde) y actina (rojo). (D-F) DIV 5 neurona de rata del hipocampo de la misma sesión que el tomograma de entrenamiento. (E) Segmentación 2D desde U-Net sin capacitación adicional y limpieza rápida. Membrana (azul), microtúbulos (verde), actina (rojo), fiduciales (rosa). (F) Representación 3D de las regiones segmentadas. (G-I) DIV 5 neurona de rata del hipocampo de la sesión de 2019. (H) Segmentación 2D desde U-Net con limpieza rápida y (I) renderizado 3D. (J-L) Neurona de rata del hipocampo DIV 5, recolectada en 2021 en un Titán Krios diferente con un aumento diferente. El tamaño de píxel se ha cambiado con el programa IMOD squeezevol para que coincida con el tomograma de entrenamiento. (K) segmentación 2D desde U-Net con limpieza rápida, demostrando inferencia robusta en conjuntos de datos con preprocesamiento adecuado y (L) representación 3D de segmentación. Barras de escala = 100 nm. Abreviaturas: DIV = días in vitro; CTF = función de transferencia de contraste. Haga clic aquí para ver una versión más grande de esta figura.
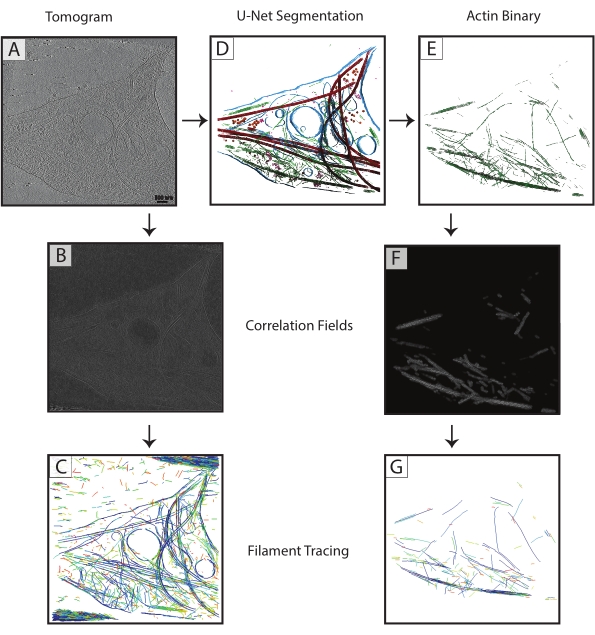
Figura 3: Mejora del trazado de filamentos . (A) Tomograma de una neurona del hipocampo de rata DIV 4, recolectada en un Titán Krios. (B) Mapa de correlación generado a partir de la correlación de cilindros sobre filamentos de actina. (C) Rastreo de filamentos de actina utilizando las intensidades de los filamentos de actina en el mapa de correlación para definir parámetros. El rastreo captura la membrana y los microtúbulos, así como el ruido, mientras intenta rastrear solo la actina. (D) Segmentación U-Net del tomograma. Membrana resaltada en azul, microtúbulos en rojo, ribosomas en naranja, triC en púrpura y actina en verde. (E) Segmentación de actina extraída como una máscara binaria para el rastreo de filamentos. (F) Mapa de correlación generado a partir de la correlación de cilindros con los mismos parámetros de (B). (G) Mejora significativa del trazado de filamentos de solo filamentos de actina del tomograma. Abreviatura: DIV = días in vitro. Haga clic aquí para ver una versión más grande de esta figura.
Archivo complementario 1: El tomograma utilizado en este protocolo y el ROI múltiple que se generó como entrada de entrenamiento se incluyen como un conjunto de datos agrupado (Training.ORSObject). Véase https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct.
Discusión
Este protocolo establece un procedimiento para usar el software Dragonfly 2022.1 para entrenar una U-Net multiclase a partir de un solo tomograma, y cómo inferir esa red a otros tomogramas que no necesitan ser del mismo conjunto de datos. El entrenamiento es relativamente rápido (puede ser tan rápido como 3-5 minutos por época o tan lento como unas pocas horas, dependiendo completamente de la red que se está entrenando y el hardware utilizado), y volver a entrenar una red para mejorar su aprendizaje es intuitivo. Siempre que los pasos de preprocesamiento se lleven a cabo para cada tomografía, la inferencia suele ser robusta.
El preprocesamiento coherente es el paso más crítico para la inferencia de aprendizaje profundo. Hay muchos filtros de imágenes en el software y el usuario puede experimentar para determinar qué filtros funcionan mejor para conjuntos de datos particulares; Tenga en cuenta que cualquier filtrado que se use en el tomograma de entrenamiento debe aplicarse de la misma manera a los tomogramas de inferencia. También se debe tener cuidado de proporcionar a la red información de capacitación precisa y suficiente. Es vital que todas las características segmentadas dentro de las secciones de entrenamiento se segmenten con el mayor cuidado y precisión posible.
La segmentación de imágenes se ve facilitada por una sofisticada interfaz de usuario de nivel comercial. Proporciona todas las herramientas necesarias para la segmentación de manos y permite la simple reasignación de vóxeles de cualquier clase a otra antes del entrenamiento y el reciclaje. Al usuario se le permite segmentar a mano los vóxeles dentro de todo el contexto del tomograma, y se le dan múltiples vistas y la capacidad de girar el volumen libremente. Además, el software proporciona la capacidad de utilizar redes multiclase, que tienden a funcionar mejor16 y son más rápidas que la segmentación con múltiples redes de clase única.
Hay, por supuesto, limitaciones a las capacidades de una red neuronal. Los datos crio-ET son, por naturaleza, muy ruidosos y limitados en el muestreo angular, lo que conduce a distorsiones específicas de orientación en objetos idénticos21. La capacitación depende de un experto para segmentar las estructuras con precisión, y una red exitosa es tan buena (o tan mala) como los datos de capacitación que se le dan. El filtrado de imágenes para aumentar la señal es útil para el entrenador, pero todavía hay muchos casos en los que es difícil identificar con precisión todos los píxeles de una estructura determinada. Por lo tanto, es importante que se tenga mucho cuidado al crear la segmentación de capacitación para que la red tenga la mejor información posible para aprender durante la capacitación.
Este flujo de trabajo se puede modificar fácilmente según las preferencias de cada usuario. Si bien es esencial que todos los tomogramas se procesen exactamente de la misma manera, no es necesario utilizar los filtros exactos utilizados en el protocolo. El software tiene numerosas opciones de filtrado de imágenes, y se recomienda optimizarlas para los datos particulares del usuario antes de emprender un gran proyecto de segmentación que abarque muchos tomogramas. También hay bastantes arquitecturas de red disponibles para usar: se ha encontrado que una U-Net multisegmento funciona mejor para los datos de este laboratorio, pero otro usuario podría encontrar que otra arquitectura (como una U-Net 3D o un Sensor 3D) funciona mejor. El asistente de segmentación proporciona una interfaz cómoda para comparar el rendimiento de varias redes utilizando los mismos datos de entrenamiento.
Herramientas como las que se presentan aquí harán que la segmentación manual de tomogramas completos sea una tarea del pasado. Con redes neuronales bien entrenadas que son robustamente inferibles, es completamente factible crear un flujo de trabajo donde los datos tomográficos se reconstruyan, procesen y segmenten completamente tan rápido como el microscopio pueda recopilarlos.
Divulgaciones
La licencia de acceso abierto para este protocolo fue pagada por Object Research Systems.
Agradecimientos
Este estudio fue apoyado por la Facultad de Medicina de Penn State y el Departamento de Bioquímica y Biología Molecular, así como por la subvención 4100079742-EXT del Fondo de Liquidación del Tabaco (TSF). Los servicios e instrumentos CryoEM y CryoET Core (RRID: SCR_021178) utilizados en este proyecto fueron financiados, en parte, por la Facultad de Medicina de la Universidad Estatal de Pensilvania a través de la Oficina del Vicedecano de Investigación y Estudiantes de Posgrado y el Departamento de Salud de Pensilvania utilizando Fondos de Liquidación de Tabaco (CURE). El contenido es responsabilidad exclusiva de los autores y no representa necesariamente las opiniones oficiales de la Universidad o Facultad de Medicina. El Departamento de Salud de Pensilvania se exime específicamente de responsabilidad por cualquier análisis, interpretación o conclusión.
Materiales
| Name | Company | Catalog Number | Comments |
| Dragonfly 2022.1 | Object Research Systems | https://www.theobjects.com/dragonfly/index.html | |
| E18 Rat Dissociated Hippocampus | Transnetyx Tissue | KTSDEDHP | https://tissue.transnetyx.com/faqs |
| IMOD | University of Colorado | https://bio3d.colorado.edu/imod/ | |
| Intel® Xeon® Gold 6124 CPU 3.2GHz | Intel | https://www.intel.com/content/www/us/en/products/sku/120493/intel-xeon-gold-6134-processor-24-75m-cache-3-20-ghz/specifications.html | |
| NVIDIA Quadro P4000 | NVIDIA | https://www.nvidia.com/content/dam/en-zz/Solutions/design-visualization/productspage/quadro/quadro-desktop/quadro-pascal-p4000-data-sheet-a4-nvidia-704358-r2-web.pdf | |
| Windows 10 Enterprise 2016 | Microsoft | https://www.microsoft.com/en-us/evalcenter/evaluate-windows-10-enterprise | |
| Workstation Minimum Requirements | https://theobjects.com/dragonfly/system-requirements.html |
Referencias
- Bai, X. -C., Mcmullan, G., Scheres, S. H. W. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- de Oliveira, T. M., van Beek, L., Shilliday, F., Debreczeni, J., Phillips, C. Cryo-EM: The resolution revolution and drug discovery. SLAS Discovery. 26 (1), 17-31 (2021).
- Danev, R., Yanagisawa, H., Kikkawa, M. Cryo-EM performance testing of hardware and data acquisition strategies. Microscopy. 70 (6), 487-497 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Tomography 5 and Tomo Live Software User-friendly batch acquisition for and on-the-fly reconstruction for cryo-electron tomography Datasheet. , Available from: https://assets.thermofisher.com/TFS-Assets/MSD/Datasheets/tomography-5-software-ds0362.pdf (2022).
- Danev, R., Baumeister, W. Expanding the boundaries of cryo-EM with phase plates. Current Opinion in Structural Biology. 46, 87-94 (2017).
- Hylton, R. K., Swulius, M. T. Challenges and triumphs in cryo-electron tomography. iScience. 24 (9), (2021).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Oikonomou, C. M., Jensen, G. J. Cellular electron cryotomography: Toward structural biology in situ. Annual Review of Biochemistry. 86, 873-896 (2017).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-the cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Lam, V., Villa, E. Practical approaches for Cryo-FIB milling and applications for cellular cryo-electron tomography. Methods in Molecular Biology. 2215, 49-82 (2021).
- Chreifi, G., Chen, S., Metskas, L. A., Kaplan, M., Jensen, G. J. Rapid tilt-series acquisition for electron cryotomography. Journal of Structural Biology. 205 (2), 163-169 (2019).
- Eisenstein, F., Danev, R., Pilhofer, M. Improved applicability and robustness of fast cryo-electron tomography data acquisition. Journal of Structural Biology. 208 (2), 107-114 (2019).
- Esteva, A., et al. Deep learning-enabled medical computer vision. npj Digital Medicine. 4 (1), (2021).
- Liu, Y. -T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nature Methods. 14 (10), 983-985 (2017).
- Ronneberger, O., Fischer, P., Brox, T. U-net: Convolutional networks for biomedical image segmentation. Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 9351, 234-241 (2015).
- Dragonfly 2021.3 (Computer Software). , Available from: http://www.theobjects.com/dragonfly (2021).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iancu, C. V., et al. A "flip-flop" rotation stage for routine dual-axis electron cryotomography. Journal of Structural Biology. 151 (3), 288-297 (2005).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados