Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Automatización del ensayo de micronúcleos mediante citometría de flujo de imágenes e inteligencia artificial
En este artículo
Resumen
El ensayo de micronúcleos (MN) es una prueba bien establecida para cuantificar el daño del ADN. Sin embargo, calificar el ensayo utilizando técnicas convencionales como la microscopía manual o el análisis de imágenes basado en características es laborioso y desafiante. Este artículo describe la metodología para desarrollar un modelo de inteligencia artificial para calificar el ensayo MN utilizando datos de citometría de flujo de imágenes.
Resumen
El ensayo de micronúcleos (MN) es utilizado en todo el mundo por los organismos reguladores para evaluar los productos químicos en busca de toxicidad genética. El ensayo se puede realizar de dos maneras: marcando MN en células binucleadas bloqueadas por citocinesis una vez divididas o células mononucleadas completamente divididas. Históricamente, la microscopía óptica ha sido el método estándar de oro para calificar el ensayo, pero es laborioso y subjetivo. La citometría de flujo se ha utilizado en los últimos años para calificar el ensayo, pero está limitada por la incapacidad de confirmar visualmente aspectos clave de las imágenes celulares. La citometría de flujo de imágenes (IFC) combina la captura de imágenes de alto rendimiento y el análisis automatizado de imágenes, y se ha aplicado con éxito para adquirir rápidamente imágenes y calificar todos los eventos clave en el ensayo MN. Recientemente, se ha demostrado que los métodos de inteligencia artificial (IA) basados en redes neuronales convolucionales se pueden utilizar para calificar los datos de ensayos MN adquiridos por IFC. En este documento se describen todos los pasos para usar el software de IA para crear un modelo de aprendizaje profundo para calificar todos los eventos clave y aplicar este modelo para calificar automáticamente datos adicionales. Los resultados del modelo de aprendizaje profundo de IA se comparan bien con la microscopía manual, lo que permite una puntuación totalmente automatizada del ensayo MN mediante la combinación de IFC e IA.
Introducción
El ensayo de micronúcleos (MN) es fundamental en toxicología genética para evaluar el daño del ADN en el desarrollo de cosméticos, productos farmacéuticos y productos químicos para uso humano 1,2,3,4. Los micronúcleos se forman a partir de cromosomas enteros o fragmentos de cromosomas que no se incorporan al núcleo después de la división y se condensan en cuerpos pequeños y circulares separados del núcleo. Por lo tanto, MN puede ser utilizado como un punto final para cuantificar el daño del ADN en las pruebas de genotoxicidad1.
El método preferido para cuantificar MN es dentro de células binucleadas una vez divididas (BNC) mediante el bloqueo de la división utilizando citochalasina-B (Cyt-B). En esta versión del ensayo, la citotoxicidad también se evalúa mediante la puntuación de células mononucleadas (MONO) y polinucleadas (POLY). El ensayo también se puede realizar marcando MN en células MONO desbloqueadas, que es más rápido y fácil de puntuar, y la citotoxicidad se evalúa utilizando recuentos celulares previos y posteriores a la exposición para evaluar la proliferación 5,6.
La puntuación física del ensayo se ha realizado históricamente a través de microscopía manual, ya que esto permite la confirmación visual de todos los eventos clave. Sin embargo, la microscopía manual es desafiante y subjetiva1. Por lo tanto, se han desarrollado técnicas automatizadas, incluido el escaneo de portaobjetos de microscopio y la citometría de flujo, cada uno con sus propias ventajas y limitaciones. Mientras que los métodos de escaneo de diapositivas permiten visualizar eventos clave, las diapositivas deben crearse con una densidad celular óptima, lo que puede ser difícil de lograr. Además, esta técnica a menudo carece de visualización citoplasmática, lo que puede comprometer la puntuación de las células MONO y POLY 7,8. Si bien la citometría de flujo ofrece una captura de datos de alto rendimiento, las células deben ser lisadas, lo que no permite el uso de la forma Cyt-B del ensayo. Además, como técnica no imagenológica, la citometría de flujo convencional no proporciona validación visual de eventos clave 9,10.
Por lo tanto, se ha investigado la citometría de flujo por imágenes (IFC) para realizar el ensayo MN. La ImageStreamX Mk II combina la velocidad y la robustez estadística de la citometría de flujo convencional con las capacidades de imagen de alta resolución de la microscopía en un solo sistema11. Se ha demostrado que mediante el uso de IFC, las imágenes de alta resolución de todos los eventos clave se pueden capturar y calificar automáticamente utilizando técnicas basadas en características 12,13 o inteligencia artificial (IA) 14,15. Mediante el uso de IFC para realizar el ensayo MN, se puede lograr la puntuación automática de muchas más células en comparación con la microscopía en un período de tiempo más corto.
Este trabajo se desvía de un flujo de trabajo de análisis de imágenes descrito anteriormente16 y discute todos los pasos necesarios para desarrollar y entrenar un modelo de bosque aleatorio (RF) y / o red neuronal convolucional (CNN) utilizando el software Amnis AI (en adelante, "software de IA"). Se describen todos los pasos necesarios, incluido el llenado de datos reales sobre el terreno utilizando herramientas de etiquetado asistidas por IA, la interpretación de los resultados del entrenamiento del modelo y la aplicación del modelo para clasificar datos adicionales, lo que permite el cálculo de la genotoxicidad y la citotoxicidad15.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Adquisición de datos mediante citometría de flujo de imágenes
NOTA: Consulte Rodrigues et al.16 con las siguientes modificaciones, señalando que las regiones de adquisición que utilizan IFC pueden necesitar ser modificadas para una captura óptima de la imagen:
- Para el método no Cyt-B, realice un recuento de células utilizando un contador de células disponible comercialmente siguiendo las instrucciones del fabricante (consulte la Tabla de materiales) en cada cultivo inmediatamente antes del cultivo e inmediatamente después del período de recuperación.
- Si se ejecutan muestras en un citómetro de flujo de imágenes de una sola cámara, coloque el campo claro (BF) en el canal 4. Reemplace M01 por M04 y M07 por M01.
NOTA: "M" se refiere al canal de la cámara en el IFC. - Utilice el aumento de 40x durante la adquisición.
- En la gráfica de relación de aspecto área BF versus BF durante la adquisición, use las siguientes coordenadas de región:
Coordenadas X: 100 y 900; Coordenadas Y: 0,7 y 1 (método Cyt-B)
Coordenadas X: 100 y 600; Coordenadas Y: 0.7 y 1 - En el gráfico de intensidad de Hoechst, utilice las siguientes coordenadas de región:
Coordenadas X: 55 y 75; Coordenadas Y: 9,5 y 15 (método Cyt-B)
Coordenadas X: 55 y 75; Coordenadas Y: 13 y 21 (método no Cyt-B) - Para eliminar imágenes de objetos apoptóticos y necróticos de los datos, inicie el paquete de software IDEAS 6.3 (en adelante, el "software de análisis de imágenes"; ver Tabla de materiales).
NOTA: El software AI ha sido diseñado para trabajar con archivos .daf que se han procesado utilizando la última versión del software de análisis de imágenes. Asegúrese de que el software de análisis de imágenes esté actualizado. - Guarde este trabajo como un archivo de plantilla (.ast).
2. Creación de archivos .daf para todos los archivos .rif
- El software AI solo permite importar archivos .daf. Cree archivos .daf para todos los archivos .rif del experimento mediante el procesamiento por lotes.
- En el menú Herramientas , haga clic en Archivos de datos por lotes y luego haga clic en Agregar lote.
- En la nueva ventana, seleccione Agregar archivos y seleccione los archivos .rif que se agregarán al lote. En la opción Seleccionar una plantilla o un archivo de análisis de datos (.ast, .daf), seleccione el archivo .ast que se creó anteriormente.
- Asigne un nombre de lote si es necesario y haga clic en Aceptar para crear archivos .daf para todos los archivos .rif cargados.
3. Crear un experimento en el software de IA
- Consulte el diagrama de flujo de la figura 1 que describe el proceso de creación de un modelo de aprendizaje profundo con el software de IA.
- Inicie el software de IA y asegúrese de que la versión más reciente esté instalada haciendo clic en Acerca de en la esquina inferior izquierda de la ventana. Si la versión más reciente no está instalada, póngase en contacto con support@luminexcorp.com para obtenerla.
- La pantalla predeterminada en el software es la pantalla Nuevo experimento . Utilice el icono Carpeta para elegir dónde guardar el experimento y escriba un nombre para el experimento (por ejemplo, "modelo MN").
- En Tipo de experimento, haga clic en el botón de opción situado junto a Entrenar para iniciar un experimento de entrenamiento para comenzar a construir el modelo CNN. Haga clic en Siguiente.
- Opcional: si un modelo ha sido entrenado previamente, se puede utilizar como plantilla para un nuevo modelo de IA y se puede seleccionar como plantilla para la creación de un nuevo modelo desde la pantalla Seleccionar modelo de plantilla . Si no existe ningún modelo de plantilla, simplemente omita este paso haciendo clic en Siguiente.
- La siguiente pantalla es la pantalla Definir nuevo modelo . En Modelo, el nombre que se le dio al modelo en el paso 3.3 se rellenará automáticamente.
- En Descripción, escriba una descripción para el modelo (opcional) y deje el tamaño máximo de imagen en 150 píxeles.
- En Canales, haga clic en Agregar BF para agregar un canal brightfield a la lista. En Nombre, haga doble clic en Brightfield y cambie el nombre de este canal a BF. Haga clic en Agregar FL para agregar un canal fluorescente a la lista. En Nombre, haga doble clic en Fluorescente y cambie el nombre de este canal a ADN.
- En Nombres de clase, haga clic en Agregar. En la ventana emergente, escriba Mononucleado y haga clic en Aceptar. Esto agrega la clase mononucleada a la lista de nombres de clase. Repita este proceso para asegurarse de que las seis clases siguientes están definidas en la lista:
Mononucleado
Mononucleado con MN
Binucleado
Binucleado con MN
Polinucleado
Morfología irregular
Haga clic en Siguiente.
NOTA: Estas seis clases de modelos de verdad básica representarán los eventos clave que se calificarán, así como imágenes con morfología que difiere de los criterios de puntuación aceptados5.- Opcional: Si lo desea, se puede incluir la plantilla de análisis de 1.7 para utilizar las funciones del software de análisis de imágenes. Si desea incluir estas características en el modelo de IA, busque el archivo .ast y, a continuación, en los menús desplegables específicos del canal, elija los subconjuntos de características que desea incluir.
- En Seleccionar archivos, haga clic en Agregar archivos y busque los archivos deseados que se agregarán al software AI para construir los datos de verdad sobre el terreno. Haga clic en Siguiente.
NOTA: Es importante agregar varios archivos de datos (por ejemplo, datos de control positivo y negativo) que contengan un número suficiente de todos los eventos clave.
- A continuación, en la pantalla Seleccionar poblaciones base , busque la población no apoptótica de la jerarquía de población. Haga clic con el botón derecho en la población no apoptótica y seleccione Seleccionar todas las poblaciones coincidentes. Haga clic en Siguiente.
NOTA: Es importante excluir cualquier población que no deba clasificarse (por ejemplo, cuentas, escombros, dobletes, etc.) - Esta pantalla es la pantalla Seleccionar poblaciones de verdad .
- Si las poblaciones de verdad etiquetadas de los eventos clave no se han creado en el software de análisis de imágenes, haga clic en Siguiente.
- Si se han creado poblaciones de verdad etiquetadas en el software de análisis de imágenes, asígnelas a la clase de modelo adecuada.
- Para asignar una población de verdad etiquetada de células MONO con MN, haga clic en la clase Mononucleada con MN en Clases modelo a la izquierda. Luego haga clic en la población de verdad etiquetada apropiada a la derecha que contiene estos eventos.
- Si las poblaciones de verdad etiquetadas se han creado en más de un archivo de datos, haga clic con el botón derecho en una de las poblaciones de verdad y seleccione Seleccionar todas las poblaciones coincidentes para agregar poblaciones etiquetadas de varios archivos a la clase adecuada.
- Una vez que se hayan asignado todas las poblaciones de verdad apropiadas, haga clic en Siguiente.
- En la pantalla Seleccionar canales, elija los canales adecuados para el experimento. Aquí, configure BF en el canal 1 y Hoechst en el canal 7. Haz clic con el botón derecho en un canal y selecciona Aplicar a todo. Haga clic en Siguiente.
- Finalmente, en la pantalla Confirmación , haga clic en Crear experimento.
- El software de IA carga imágenes de los archivos de datos y crea las clases de modelo definidas en el paso 3.5.3 con las imágenes de verdad del terreno que se asignaron en el paso 3.7. Haga clic en Finalizar.
- Una vez creado el experimento, se presentan cinco opciones:
Experimento: proporciona detalles del experimento, incluidos los archivos de datos cargados, los canales elegidos y las clases de modelo de verdad de terreno definidas.
Etiquetado: inicia la herramienta de etiquetado a través de la cual los usuarios pueden completar los datos de verdad del terreno.
Entrenamiento: entrena un modelo basado en los datos de verdad sobre el terreno.
Clasificar: utiliza modelos entrenados para clasificar los datos.
Resultados: proporciona resultados tanto de un experimento de entrenamiento como de un experimento de clasificación.
4. Rellenar los datos de verdad sobre el terreno utilizando herramientas de etiquetado asistidas por IA
- Haga clic en Etiquetado para iniciar la interfaz de la herramienta de etiquetado.
- Haga clic en las herramientas de zoom (iconos de lupa) para recortar las imágenes y facilitar la visualización.
- Haga clic en la barra deslizante para ajustar el tamaño de la imagen y cambiar la cantidad de imágenes que se muestran en la galería.
- Haga clic en la opción Configuración de pantalla y elija Min-Max, que proporciona la mejor imagen de contraste para identificar todos los eventos clave.
- Haga clic en Configurar pantalla de la galería para cambiar el color de la imagen de ADN a amarillo o blanco, lo que mejorará la visualización de objetos pequeños (por ejemplo, MN).
- Haga clic en Clúster para ejecutar el algoritmo para agrupar objetos con morfología similar. Una vez completada la agrupación en clústeres, los clústeres individuales con el número de objetos por clúster se muestran en una lista en Poblaciones desconocidas. Seleccione los clústeres individuales para ver los objetos dentro del clúster y asigne estos objetos a sus clases de modelo adecuadas.
- Después de asignar un mínimo de 25 objetos a cada clase de modelo, el algoritmo Predict estará disponible. Haga clic en Predict.
NOTA: Los objetos que no encajan bien en ninguna población permanecen clasificados como Desconocidos. A medida que se agregan más objetos a las poblaciones de verdad, la precisión de la predicción mejora. - Continúe rellenando las clases del modelo de verdad de base con las imágenes adecuadas hasta que se alcance un número suficiente de objetos en cada clase.
- Una vez que se hayan asignado un mínimo de 100 objetos a cada clase de modelo, haga clic en la pestaña Entrenamiento en la parte superior de la pantalla. Haga clic en el botón Entrenar para crear un modelo utilizando los algoritmos Random Forest y CNN.
NOTA: El software de IA crea modelos utilizando los algoritmos Random Forest y CNN, las casillas de verificación permiten la creación de modelos utilizando solo algoritmos de Random Forest of CNN.
5. Evaluación de la precisión del modelo
- Una vez que se complete el entrenamiento del modelo, haga clic en Ver resultados.
- Utilice la pantalla de resultados para evaluar la precisión del modelo. Usa el menú desplegable para cambiar entre Random Forest y CNN.
NOTA: Las poblaciones de verdad se pueden actualizar y el modelo se puede volver a entrenar o usar tal cual para clasificar datos adicionales.- Para actualizar las poblaciones de verdad, haga clic en Etiquetado en la parte superior y siga la sección 4.
6. Clasificación de datos utilizando el modelo
- Inicie el software de IA. La pantalla predeterminada es la pantalla Nuevo experimento . Utilice el icono Carpeta para elegir dónde guardar el experimento y escriba un nombre para el experimento.
- En Tipo de experimento, haga clic en el botón de opción situado junto a Clasificar para iniciar un experimento de clasificación. Haga clic en Siguiente.
- Haga clic en el modelo que se utilizará para la clasificación, luego haga clic en Siguiente.
- En la pantalla Seleccionar archivos, haga clic en Agregar archivos y busque los archivos que se clasificarán por el modelo CNN. Haga clic en Siguiente.
- A continuación, en la pantalla Seleccionar poblaciones base , haga clic en la casilla de verificación junto a la población no apoptótica en uno de los archivos cargados. Haga clic derecho en la población no apoptótica y haga clic en Seleccionar todas las poblaciones coincidentes para seleccionar esta población de todos los archivos cargados. Haga clic en Siguiente.
- Opcional: si los datos que se van a clasificar contienen poblaciones de verdad, se pueden asignar a las clases de modelo adecuadas en la pantalla Seleccionar poblaciones de verdad. De lo contrario, haga clic en Siguiente para omitir este paso.
- En la pantalla Select Channels (Seleccionar canales ), elija Channel 1 (Canal 1) para el campo claro y Channel 7 (Canal 7 ) para la tinción de ADN. Haga clic derecho en un canal y haga clic en Aplicar a todos. Luego haga clic en Siguiente.
- Finalmente, en la pantalla Confirmación , haga clic en Crear experimento. El software AI carga el modelo seleccionado y todas las imágenes de los archivos de datos elegidos. Haga clic en Finalizar.
- Haga clic en Clasificar para abrir la pantalla de clasificación. Haga clic en el botón Clasificar . Esto inicia el proceso de uso del modelo RF y CNN para clasificar datos adicionales e identificar todos los objetos que pertenecen a las clases de modelo especificadas.
NOTA: Las casillas de verificación se pueden utilizar para seleccionar el modelo RF y/o el modelo CNN. - Una vez completada la clasificación, haga clic en Ver resultados.
- Haga clic en el botón Actualizar DAF para abrir la ventana Actualizar DAF con resultados de clasificación. Haga clic en Aceptar para actualizar los archivos .daf.
7. Generar un informe de los resultados de la clasificación
- En la pantalla Resultados, haga clic en Generar informe. Seleccione la casilla de verificación situada junto a Crear informe para cada DAF de entrada si se requiere un informe individual para cada daf de entrada. Haga clic en Aceptar.
- Una vez completado, abra la carpeta donde se han guardado los archivos de informe. Dentro de la carpeta, hay un experimento .pdf informe y una carpeta Recursos .
- Abra el .pdf para ver el informe. El informe contiene información del modelo y del experimento, la lista de archivos .daf de entrada, los recuentos de clases y porcentajes de clase en formato tabular e histograma, y una matriz de confusión que resume la probabilidad mediana de predicción en todos los archivos .daf de entrada.
- Abra la carpeta Recursos y luego la carpeta CNN . Dentro de esta carpeta se encuentran .png archivos del recuento de clases y gráficos de barras de porcentaje, así como la matriz de confusión. Además, hay .csv archivos que contienen los recuentos de clases y porcentajes para cada archivo de entrada.
8. Determinación de la frecuencia y citotoxicidad de MN
- Cálculo de la frecuencia MN
- Método no Cyt-B: Para determinar la frecuencia MN, abra el archivo class_count.csv del paso 7.4. Para cada archivo de entrada, divida los recuentos en la población "Mononucleada con MN" por los recuentos en la población "Mononucleada" y multiplíquela por 100:

- Método Cyt-B: Para determinar la frecuencia MN, abra el archivo class_count.csv del paso 7.4. Para cada archivo de entrada, divida los recuentos en la población "Binucleada con MN" por los recuentos en la población "Binucleada" y multiplíquela por 100:
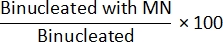
- Método no Cyt-B: Para determinar la frecuencia MN, abra el archivo class_count.csv del paso 7.4. Para cada archivo de entrada, divida los recuentos en la población "Mononucleada con MN" por los recuentos en la población "Mononucleada" y multiplíquela por 100:
- Cálculo de la citotoxicidad
- Método no Cyt-B:
- Utilizando los recuentos celulares iniciales y los recuentos celulares posteriores al tratamiento, calcule primero la duplicación de la población (DP) para cada muestra2:
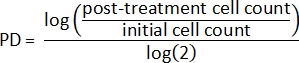
- A continuación,calcule la población relativa duplicando 2:

- Finalmente, calcule la citotoxicidad2 para cada muestra:
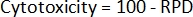
- Utilizando los recuentos celulares iniciales y los recuentos celulares posteriores al tratamiento, calcule primero la duplicación de la población (DP) para cada muestra2:
- Método Cyt-B:
- Para calcular el índice de proliferación de bloques de citocinesis (CBPI)2, use los recuentos en las clases mononucleada, binucleada y polinucleada para cada muestra del archivo de class_count.csv :
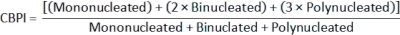
- Para calcular la citotoxicidad2, utilice el CBPI de los cultivos control (C) y cultivos expuestos (T):
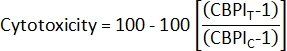
- Para calcular el índice de proliferación de bloques de citocinesis (CBPI)2, use los recuentos en las clases mononucleada, binucleada y polinucleada para cada muestra del archivo de class_count.csv :
- Método no Cyt-B:
Access restricted. Please log in or start a trial to view this content.
Resultados
La figura 1 muestra el flujo de trabajo para usar el software de IA para crear un modelo para el ensayo MN. El usuario carga los archivos .daf deseados en el software de IA, luego asigna objetos a las clases de modelo de verdad del terreno utilizando los algoritmos de etiquetado de clúster asistido por IA (Figura 2) y predicción (Figura 3). Una vez que todas las clases de modelos de verdad básica se han poblado con suficientes ob...
Access restricted. Please log in or start a trial to view this content.
Discusión
El trabajo presentado aquí describe el uso de algoritmos de aprendizaje profundo para automatizar la puntuación del ensayo MN. Varias publicaciones recientes han demostrado que las herramientas intuitivas e interactivas permiten la creación de modelos de aprendizaje profundo para analizar datos de imágenes sin necesidad de un conocimiento computacional profundo18,19. El protocolo descrito en este trabajo utilizando un paquete de software basado en interfaz de...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores son empleados de Luminex Corporation, una compañía de DiaSorin, el fabricante del citómetro de flujo de imágenes ImageStream y el software Amnis AI utilizado en este trabajo.
Agradecimientos
Ninguno.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Falcon | 352096 | |
| Cleanser - Coulter Clenz | Beckman Coulter | 8546931 | Fill container with 200 mL of Cleanser. https://www.beckmancoulter.com/wsrportal/page/itemDetails?itemNumber=8546931#2/10//0/25/ 1/0/asc/2/8546931///0/1//0/ |
| Colchicine | MilliporeSigma | 64-86-8 | |
| Corning bottle-top vacuum filter | MilliporeSigma | CLS430769 | 0.22 µm filter, 500 mL bottle |
| Cytochalasin B | MilliporeSigma | 14930-96-2 | 5 mg bottle |
| Debubbler - 70% Isopropanol | MilliporeSigma | 1.3704 | Fill container with 200 mL of Debubbler. http://www.emdmillipore.com/US/en/product/2-Propanol-70%25-%28V%2FV%29-0.1-%C2%B5m-filtred,MDA_CHEM-137040?ReferrerURL=https%3A%2F%2Fwww.google.com%2F |
| Dimethyl Sulfoxide (DMSO) | MilliporeSigma | 67-68-5 | |
| Dulbecco's Phosphate Buffered Saline 1X | EMD Millipore | BSS-1006-B | PBS Ca++MG++ Free |
| Fetal Bovine Serum | HyClone | SH30071.03 | |
| Formaldehyde, 10%, methanol free, Ultra Pure | Polysciences, Inc. | 04018 | This is what is used for the 4% and 1% Formalin. CAUTION: Formalin/Formaldehyde toxic by inhalation and if swallowed. Irritating to the eyes, respiratory systems and skin. May cause sensitization by inhalation or skin contact. Risk of serious damage to eyes. Potential cancer hazard. http://www.polysciences.com/default/catalog-products/life-sciences/histology-microscopy/fixatives/formaldehydes/formaldehyde-10-methanol-free-pure/ |
| Guava Muse Cell Analyzer | Luminex | 0500-3115 | A standard configuration Guava Muse Cell Analyzer was used. |
| Hoechst 33342 | Thermo Fisher | H3570 | 10 mg/mL solution |
| Mannitol | MilliporeSigma | 69-65-8 | |
| MEM Non-Essential Amino Acids 100X | HyClone | SH30238.01 | |
| MIFC - ImageStreamX Mark II | Luminex, a DiaSorin company | 100220 | A 2 camera ImageStreamX Mark II eqiped with the 405 nm, 488 nm, and 642 nm lasers was used. |
| MIFC analysis software - IDEAS | Luminex, a DiaSorin company | 100220 | "Image analysis sofware" The companion software to the MIFC (ImageStreamX MKII) |
| MIFC software - INSPIRE | Luminex, a DiaSorin company | 100220 | "Image acquisition software" This is the software that runs the MIFC (ImageStreamX MKII) |
| Amnis AI software | Luminex, a DiaSorin company | 100221 | "AI software" This is the software that permits the creation of artificial intelligence models to analyze data |
| Mitomycin C | MilliporeSigma | 50-07-7 | |
| NEAA Mixture 100x | Lonza BioWhittaker | 13-114E | |
| Penicllin/Streptomycin/Glutamine solution 100X | Gibco | 15070063 | |
| Potassium Chloride (KCl) | MilliporeSigma | P9541 | |
| Rinse - Ultrapure water or deionized water | NA | NA | Use any ultrapure water or deionized water. Fill container with 900 mL of Rinse. |
| RNase | MilliporeSigma | 9001-99-4 | |
| RPMI-1640 Medium 1x | HyClone | SH30027.01 | |
| Sheath - PBS | MilliporeSigma | BSS-1006-B | This is the same as Dulbecco's Phosphate Buffered Saline 1x Ca++MG++ free. Fill container with 900 mL of Sheath. |
| Sterile water | HyClone | SH30529.01 | |
| Sterilizer - 0.4%–0.7% Hypochlorite | VWR | JT9416-1 | This is assentually 10% Clorox bleach that can be made by deluting Clorox bleach with water. Fill container with 200 mL of Sterilzer. |
| T25 flask | Falcon | 353109 | |
| T75 flask | Falcon | 353136 | |
| TK6 cells | MilliporeSigma | 95111735 |
Referencias
- Fenech, M., et al. HUMN project initiative and review of validation, quality control and prospects for further development of automated micronucleus assays using image cytometry systems. International Journal of Hygiene and Environmental Health. 216 (5), 541-552 (2013).
- OECD. Test No. 487: In Vitro Mammalian Cell Micronucleus Test. Section 4. OECD Guidelines for the Testing of Chemicals. , OECD Publishing. Paris. (2016).
- Fenech, M. The in vitro micronucleus technique. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 455 (1), 81-95 (2000).
- Bonassi, S., et al. An increased micronucleus frequency in peripheral blood lymphocytes predicts the risk of cancer in humans. Carcinogenesis. 28 (3), 625-631 (2007).
- Fenech, M. Cytokinesis-block micronucleus cytome assay. Nature Protocols. 2 (5), 1084-1104 (2007).
- Fenech, M. Commentary on the SFTG international collaborative study on the in vitro micronucleus test: To Cyt-B or not to Cyt-B. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 607 (1), 9-12 (2006).
- Seager, A. L., et al. Recommendations, evaluation and validation of a semi-automated, fluorescent-based scoring protocol for micronucleus testing in human cells. Mutagenesis. 29 (3), 155-164 (2014).
- Rossnerova, A., Spatova, M., Schunck, C., Sram, R. J. Automated scoring of lymphocyte micronuclei by the MetaSystems Metafer image cytometry system and its application in studies of human mutagen sensitivity and biodosimetry of genotoxin exposure. Mutagenesis. 26 (1), 169-175 (2011).
- Bryce, S. M., Bemis, J. C., Avlasevich, S. L., Dertinger, S. D. In vitro micronucleus assay scored by flow cytometry provides a comprehensive evaluation of cytogenetic damage and cytotoxicity. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 630 (1), 78-91 (2007).
- Avlasevich, S. L., Bryce, S. M., Cairns, S. E., Dertinger, S. D. In vitro micronucleus scoring by flow cytometry: Differential staining of micronuclei versus apoptotic and necrotic chromatin enhances assay reliability. Environmental and Molecular Mutagenesis. 47 (1), 56-66 (2006).
- Basiji, D. A. Principles of Amnis imaging flow cytometry. Methods in Molecular Biology. 1389, 13-21 (2016).
- Rodrigues, M. A. Automation of the in vitro micronucleus assay using the Imagestream® imaging flow cytometer. Cytometry Part A. 93 (7), 706-726 (2018).
- Verma, J. R., et al. Investigating FlowSight® imaging flow cytometry as a platform to assess chemically induced micronuclei using human lymphoblastoid cells in vitro. Mutagenesis. 33 (4), 283-289 (2018).
- Wills, J. W., et al. Inter-laboratory automation of the in vitro micronucleus assay using imaging flow cytometry and deep learning. Archives of Toxicology. 95 (9), 3101-3115 (2021).
- Rodrigues, M. A., et al. The in vitro micronucleus assay using imaging flow cytometry and deep learning. Npj Systems Biology and Applications. 7 (1), 20(2021).
- Rodrigues, M. A. An automated method to perform the in vitro micronucleus assay using multispectral imaging flow cytometry. Journal of Visualized Experiments. (147), e59324(2019).
- Lovell, D. P., et al. Analysis of negative historical control group data from the in vitro micronucleus assay using TK6 cells. Mutation Research/Genetic Toxicology and Environmental Mutagenesis. 825, 40-50 (2018).
- Berg, S., et al. ilastik: interactive machine learning for (bio)image analysis. Nature Methods. 16 (12), 1226-1232 (2019).
- Hennig, H., et al. An open-source solution for advanced imaging flow cytometry data analysis using machine learning. Methods. 112, 201-210 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados