Method Article
Uso de métodos mínimamente invasivos para evaluar la utilización de combustible y los ritmos circadianos en adultos mayores
En este artículo
Resumen
Se presenta un protocolo novedoso y mínimamente invasivo para evaluar el impacto sinérgico de la utilización de combustible y los ritmos circadianos en individuos mayores, utilizando células mononucleares de sangre periférica.
Resumen
El envejecimiento se asocia con múltiples cambios fisiológicos que contribuyen de manera sinérgica e independiente a la discapacidad física y al riesgo de enfermedades crónicas. Aunque la etiología de la discapacidad física relacionada con la edad es compleja y multifactorial, el deterioro de la función mitocondrial parece coincidir con la progresión del deterioro funcional en muchos adultos mayores. La razón por la que hay una disminución de la función mitocondrial con el envejecimiento sigue siendo difícil de alcanzar, pero la ciencia emergente indica que tanto el metabolismo del combustible como los ritmos circadianos pueden influir en la función mitocondrial.
Estudios recientes han establecido que los ritmos circadianos se alteran con el envejecimiento, y que los ritmos circadianos alterados tienen consecuencias patológicas que afectan la función mitocondrial y se superponen con muchas enfermedades crónicas asociadas a la edad. Los métodos cuantitativos actuales para la evaluación directa de la función mitocondrial son invasivos y, por lo general, requieren una biopsia muscular, lo que puede plantear dificultades con el reclutamiento de participantes y la adherencia al estudio, dados los niveles percibidos de dolor y riesgo potenciales. Por lo tanto, se adaptó un protocolo innovador y relativamente no invasivo para evaluar los cambios en la función mitocondrial a nivel celular y los patrones circadianos en adultos mayores. En concreto, se utiliza un analizador de flujo metabólico en tiempo real para evaluar la función bioenergética mitocondrial de los glóbulos blancos bajo disponibilidad diferencial de sustrato.
También se analiza la expresión de los genes del reloj circadiano en los glóbulos blancos para correlacionarse con la bioenergética mitocondrial y los resultados del ritmo circadiano. Se cree que estos enfoques metodológicos innovadores ayudarán a futuros ensayos clínicos al proporcionar métodos mínimamente invasivos para estudiar la preferencia de sustrato mitocondrial y los ritmos circadianos en adultos mayores.
Introducción
Los avances del siglo pasado han provocado un aumento de la esperanza de vida y de la población de adultos mayores. De cara al futuro, se prevé que el porcentaje de adultos de 65 años o más aumente un 5% entre 2020 y 2050 en los Estados Unidos1. Este aumento en la esperanza de vida no implica un aumento en la duración de la salud, el período de la vida asociado con el funcionamiento independiente. La realidad es que el envejecimiento se acompaña de innumerables cambios biológicos que afectan al metabolismo y a la fisiología celular, produciendo disminuciones graduales en el funcionamiento cognitivo y físico 2,3. A medida que la esperanza de vida humana continúa aumentando, existe una mayor necesidad de preservar la capacidad funcional y la independencia a partir de los4 años.
Desde hace mucho tiempo se sabe que la disminución de la función física y de la independencia con la edad es multifactorial, aunque frecuentemente se asocia con la aparición de enfermedades crónicas y eventos desencadenantes agudos5. Por el contrario, se ha demostrado que estas disminuciones en el rendimiento físico y las características musculares se asocian con el desarrollo de discapacidad con la edad, sin una conexión clara con una sola enfermedad6. Con las dificultades para conocer la etiología exacta de la enfermedad crónica y la discapacidad física, se ha pensado que las deficiencias en la función mitocondrial coinciden con el inicio y la progresión de la enfermedad crónica y la pérdida de la función física en adultos mayores 7,8.
Las mitocondrias proporcionan la mayor cantidad de trifosfato de adenosina (ATP), necesario para muchos procesos celulares9. Los tejidos altamente oxidativos dependen de las mitocondrias para una producción adecuada de energía; con el envejecimiento, la capacidad oxidativa y la síntesis de ATP mitocondrial disminuyen. Esta disminución se debe en parte al daño oxidativo del ADN mitocondrial (ADNmt), que resulta en una acumulación incremental de mutaciones y deleciones de ADNmt10. La acumulación de mutaciones y deleciones del ADNmt provoca una disminución en la formación de proteínas funcionales de la cadena de transporte de electrones, lo que provoca una reducción de la capacidad de las células para producir ATP. La disminución de la función mitocondrial asociada a la edad es más notable en los tejidos altamente oxidativos, como el corazón y el músculo esquelético11. Los estudios han demostrado que las mitocondrias del músculo gastrocnemio en muestras de ratas más viejas exhiben una reducción de aproximadamente el 50% en la producción y el contenido de ATP en comparación con las muestras más jóvenes12. Además, se ha demostrado que la capacidad de producción de ATP mitocondrial en el músculo esquelético humano disminuye en aproximadamente un 8% por década de vida13. Estos hallazgos sugieren que las disminuciones relacionadas con la edad en la función mitocondrial pueden contribuir a la disminución de la producción de energía en los organismos.
Se cree que un regulador clave de la actividad mitocondrial es el coactivador del receptor γ activado por proliferadores de peroxisomas (PPARγ)-1 (PGC-1α)14. El deterioro de la actividad de PGC-1α o la disminución de su abundancia conduce a una reducción de la actividad oxidativa mitocondrial y, en consecuencia, a una disminución de la producción de energía. Además, una disminución en la calidad mitocondrial puede afectar la calidad del músculo esquelético y, posteriormente, conducir al desarrollo o exacerbación de sarcopenia, dinapenia y disminución de la capacidad funcional15,16. La evidencia de la disminución concurrente de la función mitocondrial y la calidad del músculo esquelético relacionada con la edad sugiere una conexión entre la discapacidad mitocondrial y la patogénesis del deterioro funcional17. Recientemente, esto ha sido confirmado en adultos mayores funcionales que viven en la comunidad, mostrando que las reducciones en el metabolismo de las mitocondrias del músculo esquelético predicen la disminución de la movilidad en esta población18. Aunque el mecanismo exacto que conduce al deterioro mitocondrial con la edad no está claro, la evidencia reciente ha destacado una interacción recíproca entre el reloj circadiano y la función mitocondrial, con consecuencias para la utilización del combustible mitocondrial y la biogénesis.
Utilización de combustible
La función mitocondrial parece estar influenciada por el metabolismo del combustible y el tipo de combustible utilizado a nivel celular en el tejido muscular esquelético11. Durante los períodos de agotamiento del combustible, específicamente el agotamiento de carbohidratos en los seres humanos, la preferencia por el combustible para la producción de energía (mitocondrial) cambia. A niveles bajos de glucosa, la preferencia por el combustible se aleja de la glucosa y se desplaza hacia los ácidos grasos y los cuerpos cetónicos derivados de ácidos. Este cambio metabólico está marcado por la regulación positiva del metabolismo de los lípidos en los adipocitos, seguida de una mayor liberación de cetonas en la sangre4. El cambio en la utilización de combustible de glucosa a cetonas con una dieta cetogénica parece tener un efecto beneficioso en la producción de especies reactivas de oxígeno mitocondrial, la defensa antioxidante, la síntesis de ATP y la biogénesis20.
El cambio metabólico del metabolismo de los carbohidratos al lipídico se produce en períodos de baja disponibilidad de nutrientes ambientales y cuando las reservas de glucógeno se han agotado. Cuando se inicia este cambio, los triglicéridos almacenados se descomponen en glicerol, un sustrato para la gluconeogénesis, y ácidos grasos libres, que se transportan al hígado para ser oxidados a través de la β-oxidación en acetil coenzima A (acetil CoA). Los cuerpos cetónicos se sintetizan, principalmente en el hígado, mediante una condensación de dos pasos de tres moléculas de acetil-CoA en β-hidroxi-β-metilglutaril-CoA, que luego se procesan en cuerpos cetónicos, incluido el acetoacetato y el hidroxibutirato3-βeta 21. Estos cuerpos cetónicos se distribuyen a los tejidos de todo el cuerpo, y el mayor consumo se produce en el corazón, el cerebro yel músculo esquelético. Con el envejecimiento, la oxidación de los ácidos grasos mitocondriales se ve afectada, lo que afecta el interruptor metabólico22. Se ha propuesto que las deficiencias en la utilización del combustible mitocondrial conducen a una mayor disfunción mitocondrial, lo que a su vez contribuye a las enfermedades relacionadas con la edad y al deterioro funcional23.
Se han estudiado los cambios en el consumo de oxígeno mitocondrial de las células mononucleares de sangre periférica (PBMC) para evaluar los patrones asociados con la disfunción y la vascularización. Hartman et al. llevaron a cabo un estudio que tuvo como objetivo determinar la correlación entre el consumo de oxígeno y la dilatación mediada de diversas maneras, lo que sugiere un vínculo entre la disfunción mitocondrial y la disfunción de las células del músculo liso vascular24. En cuanto a otros órganos, las PBMC se han correlacionado con un mayor funcionamiento cognitivo y cerebral, según lo determinado por la respirometría25. Por lo tanto, la bioenergética y la capacidad respiratoria de PBMC pueden servir como biomarcadores potenciales para evaluar la capacidad funcional de órganos o tejidos en todo el cuerpo.
Ritmo circadiano
Otro factor importante que afecta la salud mitocondrial es el ritmo circadiano. Los ritmos circadianos son oscilaciones de ~24 h en el comportamiento y la fisiología que ocurren en ausencia de señales ambientales26. Estos ritmos funcionan de forma predictiva para apoyar la homeostasis del sistema y los tejidos. El mecanismo que subyace a los ritmos circadianos es un bucle de retroalimentación de transcripción-traducción llamado reloj circadiano27. Se ha demostrado en los últimos 15 años que el mecanismo del reloj circadiano existe en prácticamente todas las células del cuerpo28. Además de mantener el tiempo, el mecanismo del reloj molecular también contribuye a un programa diario de expresión génica, conocido como salida del reloj circadiano29. Los genes de salida del reloj son únicos para cada tipo de tejido y están funcionalmente asociados con vías importantes para el metabolismo celular, la autofagia, la reparación y la homeostasis. La evidencia reciente ha demostrado que la salud mitocondrial depende de la función del reloj circadiano e influye en la función mitocondrial, incluida la biogénesis mitocondrial, la utilización de combustible y la mitofagia30.
La evidencia emergente en estudios preclínicos y clínicos ha demostrado que a lo largo del envejecimiento, hay alteraciones en los ritmos circadianos31. Estos incluyen interrupciones en los ciclos normales de sueño y vigilia, una amplitud disminuida en los ritmos de temperatura corporal central y una capacidad retrasada para adaptarse a los cambios en la fase31. Un estudio, por ejemplo, desafió el sistema circadiano de ratones adultos y viejos (20+ meses) cambiando el horario de luz en 6 h. Se descubrió que los ratones viejos tardaron más en volver a adaptar sus patrones de actividad al nuevo horario de luz32. De acuerdo con los cambios en el comportamiento circadiano, el análisis de los relojes tisulares encontró que tanto los relojes tisulares centrales como los periféricos estaban alterados en la cohorte de envejecimiento.
Más recientemente, varios grupos han realizado análisis transcriptómicos del reloj circadiano y la salida del reloj en diferentes tejidos con la edad de33 años. Los resultados de estos estudios ponen de manifiesto que hay una reprogramación a gran escala de la salida del reloj circadiano con la edad. Esto significa que, a pesar de que el reloj central mantiene una función de temporización, los genes a los que se dirige la expresión diaria son muy diferentes. Por ejemplo, dos estudios han recogido biopsias musculares de sujetos humanos cada 4 h durante 24 h, los resultados determinaron que el pico y el valle de la expresión génica del reloj se invierten entre los roedores nocturnos y los humanos diurnos 34,35,36. Esto indica que cuando se compara la expresión del gen reloj basándose únicamente en la fase activa frente a la fase de reposo (y no en la luz frente a la oscuridad), los patrones de expresión del gen reloj en los músculos son prácticamente los mismos entre las especies. Se propone que este cambio asociado a la edad en la salida del reloj da lugar a deficiencias en la regulación de las vías que incluyen las características conocidas del envejecimiento, como la función mitocondrial, el daño y la reparación del ADN y la autofagia37.
Justificación del estudio
La conexión entre la función mitocondrial y la disminución de la función física está bien establecida. Sin embargo, la causa subyacente de la disfunción mitocondrial sigue siendo un tema de debate. Investigaciones recientes sugieren que la utilización del combustible celular y los ritmos circadianos pueden desempeñar un papel en este proceso. Los métodos tradicionales para evaluar la función mitocondrial, como la medición del consumo de oxígeno mitocondrial en una muestra de biopsia muscular, a menudo se perciben como dolorosos e invasivos, lo que puede desalentar la participación, particularmente en poblaciones con baja masa muscular, como los adultos frágiles y sarcopénicos38.
Dadas estas limitaciones, existe la necesidad de un método menos invasivo para evaluar los cambios en la utilización del combustible celular y el ritmo circadiano en los adultos mayores. Este estudio tiene como objetivo evaluar un protocolo novedoso y mínimamente invasivo que se puede utilizar para evaluar el metabolismo del combustible y el ritmo circadiano en esta población. Los resultados de este estudio contribuirán a una mejor comprensión de los cambios relacionados con la edad y la respuesta a las intervenciones médicas o conductuales, sirviendo de modelo para futuros estudios en este campo.
Protocolo
Los procedimientos que involucran participantes humanos han sido aprobados por el comité de ética de investigación (Política de Ética de Florida 1.0104) y la Junta de Revisión Institucional de la Universidad de Florida.
1. Función mitocondrial
- Aislamiento de células mononucleares de sangre periférica (PBMC)
- Recoja las PBMC utilizando tubos especiales de extracción de sangre de 8 mL (16 mm x 125 mm; que contengan anticoagulante de citrato de sodio 0,1 M o anticoagulante de heparina de sodio) (consulte la Tabla de materiales).
NOTA: Los tubos de extracción de sangre contienen medios de separación de sangre compuestos por un gel de poliéster tixotrópico y un medio de gradiente (consulte la Tabla de materiales) para mejorar la separación de los glóbulos blancos. - Procese los tubos de preparación de células de 8 ml dentro de las 2 horas posteriores a la recolección para obtener resultados viables.
- Mezcle los tubos de extracción de sangre después de llenarlos con sangre (ya sea por inversión suave o en una batidora) y manténgalos a temperatura ambiente (RT) durante un máximo de 2 h.
- Centrifugar los tubos de extracción de sangre en un rotor oscilante adecuado a 2.000 × g durante 15 min (o equivalente a 30.000 G-min; no exceda los 2.000 × g) a RT.
NOTA: Estos tubos son más altos que un tubo de centrífuga normal de 15 ml; Por lo tanto, se debe prestar especial atención al uso del rotor correcto. - Aspire y deseche en un contenedor de desechos biopeligrosos ~ 80% de la capa de plasma, y recoja la capa celular del tubo de recolección de sangre (usando una pipeta de transferencia) en un tubo de centrífuga de 15 mL.
NOTA: Cuando use un tubo de 15 mL, agregue 7 mL de capa celular y llene hasta 14 mL con solución salina estéril tamponada con fosfato (PBS). La relación entre las celdas y el PBS debe ser de 1:1. Use varios tubos si es necesario. Mezclar suavemente invirtiendo. - Centrífuga en un rotor basculante a 900 × g durante 5 min (o equivalente a 4.500 G-min) a RT.
- Sin alterar el pellet de la celda, aspire la solución de PBS y deséchela en un contenedor de residuos biopeligrosos.
- Vuelva a suspender el pellet celular en 1 mL de medio basal (BM; ver Tabla de Materiales) triturando suavemente. Agregue el BM hasta un total de 10 mL y mezcle suavemente invirtiendo. Realice un recuento de células.
- Centrifugar la suspensión de la célula en un rotor basculante a 900 × g durante 5 min en RT.
NOTA: Cuando las células se utilizan el mismo día para la evaluación bioenergética, proceda al paso 1.1.10; Si las celdas están congeladas para su almacenamiento, continúe con el paso 1.1.13. - Calcule el volumen de medio de ensayo (AM; consulte la Tabla de materiales) para la resuspensión de la siguiente célula para lograr la concentración celular deseada.
NOTA: Por ejemplo, para una densidad de siembra de 150.000 células/pocillo en 50 μL, la concentración deseada de la suspensión celular es de 3 millones de células/mL. - Sin alterar la peleta de la célula, aspire el sobrenadante y deséchelo en un contenedor de residuos biopeligrosos.
- Vuelva a suspender el pellet celular suavemente en 1 mL o menos de AM. Agregue AM al volumen total calculado para la concentración celular deseada. Las células ahora están listas para ser sembradas en una placa de cultivo celular recubierta (ver Tabla de Materiales).
NOTA: Los siguientes pasos son para congelar las celdas para su almacenamiento (1.1.13-1.1.16) y no se aplican al procedimiento del mismo día. - Para congelar células, aspire el sobrenadante sin alterar la pelletización de la célula y deséchelo en un contenedor de residuos biopeligrosos.
- Vuelva a suspender suavemente las células en medios de congelación (consulte la Tabla de materiales) a la concentración deseada (5-10 millones de células/mL). Alícuotas de los volúmenes deseados en crioviales marcados.
- Congele lentamente la suspensión de la celda utilizando un recipiente de congelación (consulte la Tabla de materiales) durante un mínimo de 4 h o toda la noche a -80 °C.
- Después de 4 h, o a la mañana siguiente, transfiera los crioviales al almacenamiento de nitrógeno líquido, donde deben residir en la fase de vapor.
- El día antes de la medición, cubra el fondo de los pocillos de una microplaca de cultivo celular (ver Tabla de Materiales) con adhesivo celular (ver Tabla de Materiales). Prepare el adhesivo de acuerdo con las instrucciones del fabricante, con una concentración recomendada de 22,4 μg/μL, diluyendo el volumen adecuado de solución madre adhesiva en bicarbonato sódico 0,1 M (pH 8,0). Aplique 25 μL de adhesivo a cada pocillo de la microplaca de cultivo celular.
- Después de 20 minutos, extrae el líquido restante de cada pocillo y lava los pocillos dos veces con 200 μL de agua estéril purificada. Deje que la placa se seque en la cabina de bioseguridad (durante ~2 h).
- Envuelva el plato en papel de aluminio o colóquelo en un recipiente secundario cerrado y guárdelo en un refrigerador a 4 ° C.
- Recoja las PBMC utilizando tubos especiales de extracción de sangre de 8 mL (16 mm x 125 mm; que contengan anticoagulante de citrato de sodio 0,1 M o anticoagulante de heparina de sodio) (consulte la Tabla de materiales).
- Evaluación bioenergética de PBMCs con analizador de flujo metabólico en tiempo real
NOTA: El uso de un analizador de flujo y el lector multimodo (consulte Tabla de materiales), se determinó una densidad de siembra óptima de 150.000 PBMC por pocillo, una concentración final de cianuro de carbonilo-4 (trifluorometoxi) fenilhidrazona (FCCP) del desacoplador de 2 μM y una concentración final de Hoechst 33342 de 4 μM (con un tiempo de incubación de al menos 20 minutos después de la inyección).- Para hidratar los cartuchos de sensor con hidrobooster, retire el cartucho de sensor con la placa de utilidad de la caja y coloque el cartucho "sensor arriba" en el banco. Pipetee 200 μL de calibrante (consulte la tabla de materiales) en cada pocillo de la placa de servicios y coloque el hidrorefuerzo firmemente en la placa de servicios públicos, seguido del cartucho del sensor. Inspeccione y elimine las burbujas de aire atrapadas y colóquelas en una incubadora sin CO2 a 37 °C durante la noche.
- Encienda el analizador de flujo y, a continuación, abra el software de análisis de ensayos (consulte la tabla de materiales). Ajuste la temperatura a 37 °C. Deje el instrumento encendido durante la noche para permitir la estabilización de la temperatura establecida.
NOTA: Los siguientes pasos de programación (1.2.4-1.2.10) se pueden realizar el día anterior o el día del experimento - Para programar el archivo del software de análisis de ensayos (consulte la tabla de materiales), abra la plantilla para la prueba de tensión de oxidación del sustrato. Vaya a Definiciones de grupo, abra la pestaña Estrategias de inyección , edite la prueba de esfuerzo Inhibidor + Sub Ox a Etomoxir + Prueba de esfuerzo Sub Ox y agregue dos estrategias de inyección más: UK5099 + Prueba de esfuerzo Sub Ox y BTPES + Prueba de esfuerzo Sub Ox. Dentro de cada estrategia de inyección, dentro de la ventana Condición de inyección , haga clic en A para el puerto A e introduzca la concentración final del inhibidor respectivo (Etomoxir: 4 μM; UK5099: 2 μM; BTPES: 3 μM).
- Haga clic en los puertos B, C y D, e introduzca las concentraciones finales respectivas (puerto B: concentración de oligomicina [1,50 μM], puerto C: FCCP [2,0 μM]; puerto D: Rotenona + Antimicina A [0,5 μM]). Añada Hoechst 33342, 4 μM a la ventana de compuestos para el puerto D.
- Abra la pestaña Pretratamientos y elija pretratamientos, si corresponde. Por ejemplo: Control, Experimental o habitual (p. ej., Línea de base y Seguimiento). Agregue descripciones en la ventana de abajo si es necesario.
- Abra la pestaña Medios de ensayo y, en el menú desplegable Medios , elija Medio RPMI, pH 7,4, introduzca el número de lote, el personal que prepara los medios, el tiempo de preparación y los suplementos añadidos a los medios (10 mM de glucosa, 2 mM de glutamina, 1 mM de piruvato; consulte la Tabla de materiales).
- Abra la pestaña Tipo de celda e ingrese PBMC-Subject ID como el Nombre, PBMC como el Tipo de celda, 150000 como la densidad de siembra, Subject ID como la fuente, el personal que preparó las celdas, la fecha de preparación y el día de descongelación de la celda, si corresponde.
- Haga clic en Generar grupos, vaya a Mapa de placas y asigne los pozos de la placa de 96 pocillos a los grupos respectivos. Mantenga los pozos A1, A12, H1 y H12 asignados como pozos de fondo.
- Vaya a Protocolo, marque la casilla Equilibrar (configuración predeterminada) e ingrese la siguiente información: Línea de base: cinco ciclos de medición: 3 min de mezcla, 0 min de espera y 3 min de medición; Medio o inhibidor (puerto A): seis ciclos de medición: 3 min de mezcla, 0 min de espera y 3 min de medición; Oligomicina (puerto B): tres ciclos de medición: 3 min de mezcla, 0 min de espera y 3 min de medición; FCCP (puerto C): tres ciclos de medición: 3 min de mezcla, 0 min de espera y 3 min de medición; Rotenona + Antimicina A + Hoechst 33342 (Puerto D): tres ciclos de medición: 3 min de mezcla, 0 min de espera y 3 min de medición.
- El día de la evaluación bioenergética, saque la microplaca de cultivo celular recubierta del refrigerador y deje que se caliente a RT en el gabinete de bioseguridad.
- Prepare el medio de ensayo (AM) mezclando 97 mL de BM precalentado y suplementos: 1 mL de piruvato (concentración final: 1 mM), 1 mL de glucosa (concentración final 10 mM) y 1 mL de glutamina (concentración final: 2 mM). Coloque la AM a 37 °C en una incubadora sin CO2 hasta su uso.
- Para sembrar las células, agregue 50 μL de suspensión celular (a partir del 1.1.12) que contenga 150,000 PBMC a cada pocillo, excepto A1, H1, A12 y H12 (asignados como pozos de fondo).
CRÍTICO: La densidad óptima de siembra ha sido determinada previamente. - Cubra la placa de células con su tapa y permita 60 minutos de reposo de células en RT en la cabina de bioseguridad para facilitar la distribución uniforme de las células.
CRÍTICO: Observe las células bajo un microscopio para asegurar la distribución celular homogénea deseada a través del pocillo. - Para la obtención de imágenes celulares, encienda el lector multimodo (consulte la Tabla de materiales) unas horas antes de la primera incubación celular; espere a que se complete el protocolo de inicio del lector antes de abrir el software de imágenes (consulte la Tabla de materiales). Abra el software de imagen y ajuste la temperatura de precalentamiento a 37 °C.
- Para preparar los reactivos de ensayo, siga las instrucciones del kit de ensayo para reconstituir los reactivos, con ligeras modificaciones, como se describe en la Tabla 1.
CRÍTICO: Las concentraciones finales óptimas de FCCP y Hoechst33342 se han determinado previamente. - Después del reposo celular de 60 minutos, inspeccione los pocillos con un microscopio de cultivo celular y tome nota de cualquier anomalía visual, como agregaciones celulares.
- Centrifugar la placa de cultivo celular en RT en una centrífuga con un rotor oscilante para microplacas a 60 × g durante 1 min, con una aceleración mínima y una rotura mínima; Gire la placa y vuelva a centrifugar a 40 × g durante 1 min. Ajuste la aceleración y el frenado al mínimo. Incubar las células durante 25-30 min a 37 °C en una incubadora sin CO2 .
NOTA: Cuando se realizan imágenes celulares, la placa celular se incuba en el lector multimodo precalentado (consulte la Tabla de materiales) y se toma una imagen de campo claro de cada pocillo durante este período de incubación (primera incubación celular). - Escanee el código de barras de la placa de cultivo celular con el escáner de código de barras (consulte Tabla de materiales), seleccione Iniciar escaneo de campo claro, coloque la placa de cultivo celular en la bandeja de placas e inicie la retracción de la bandeja. Seleccione los pocillos que contengan celdas para escanear e inicie el escaneo.
- Durante la primera incubación celular, cargue los puertos del cartucho sensor con los reactivos, como se indica en la Tabla 1: puerto (A): 20 μL de AM (controles), etomoxir (inhibidor del transporte de ácidos grasos de cadena larga a las mitocondrias), UK 5099 (ácido 2-ciano-3-(1-fenil-1H-indol-3-il)-2-propenoico; inhibidor del portador de piruvato mitocondrial), o BPTES (Bis-2-(5-fenilacetamido-1, sulfuro de 3,4-tiadiazol-2-il)etilo; inhibidor de la conversión de glutamina en glutamato); puerto (B): 22 μL de oligomicina (inhibidor de la ATP sintasa); puerto (C): 25 μL de FCCP (desacoplador de cianuro de carbonilo-4 (trifluorometoxi)fenilhidrazona); puerto (D): 27 μL de rotenona/antimicina A/Hoechst 33342 (inhibidores de los complejos mitocondriales I y III, respectivamente, y colorante nuclear). Vuelva a colocar el cartucho del sensor en la incubadora sin CO2 a 37 °C durante otros 5 minutos.
NOTA: Siga las instrucciones del fabricante para la carga de puertos. - Saque la placa de cultivo celular de la incubadora o del lector de placas después de la primera incubación celular y después de que se haya completado la obtención de imágenes celulares (si corresponde), y agregue AM tibia a cada pocillo hasta un volumen final de 180 μL por pocillo. Coloque la placa celular a 37 °C en una incubadora sin CO2 durante otros 15-25 minutos (incubación de la segunda célula).
- Inicie el ensayo metabólico iniciando la calibración del sensor durante la incubación de la segunda célula. Haga clic en Ejecutar ensayo cuando el ensayo esté listo para iniciarse con la calibración del cartucho del sensor. Cuando se le solicite, transfiera el cartucho del sensor de la incubadora sin CO2 a 37 °C a la bandeja del analizador de flujo e inicie la calibración. Siga las indicaciones del software de análisis de ensayos y reemplace la placa de utilidad por la placa de celda una vez completada la calibración.
NOTA: La calibración tarda aproximadamente 20 minutos. - Después del ensayo metabólico, siga las instrucciones del software y retire el cartucho del sensor y la placa de celdas del instrumento. Retire el cartucho del sensor de la placa de la celda y cubra la placa de la celda con su tapa.
- Una vez que se haya determinado el tiempo de incubación del tinte de Hoechst en experimentos preliminares, escanee el código de barras de la placa y siga las instrucciones del software para adquirir una imagen fluorescente de cada pocillo que contenga células con el lector multimodo.
NOTA: El software de análisis de imágenes y ensayos está interconectado, lo que permite la importación de los datos de recuento de células al archivo de datos de análisis de ensayos para la normalización de datos al recuento de células.
2. Expresión génica del reloj circadiano
NOTA: La expresión de los genes reloj de los participantes de las PBMC se revisará aislando el ARN utilizando el kit de sangre de ARN (consulte la Tabla de materiales).
- Extraiga 3 mL de la sangre del participante directamente en un tubo de ARN (ver Tabla de Materiales) con 6 mL de reactivos estabilizantes. Vórtice durante 10 s para mezclar completamente.
NOTA: Si los reactivos estabilizadores no se mezclan completamente con la muestra de sangre del participante, existe un mayor riesgo de error en el estudio. - Transfiera 1,0 mL de la muestra de sangre estabilizada a un tubo de 15 mL, con 5 mL de tampón de lisis eritrocitaria. Incubar en hielo durante 10-15 min. Agite el tubo 2 veces durante la incubación. Centrifugar el tubo a 4 °C a 400 × g durante 10 min.
- Identifique la bolita y vierta con cuidado el sobrenadante que contiene glóbulos rojos lisados. Añadir 2 mL de tampón de lisis eritrocitaria (ver Tabla de Materiales), resuspender las células, vórtice brevemente y centrifugar a 400 × g durante 10 min a 4 °C.
- Vierta el sobrenadante y deje el tubo invertido sobre papel absorbente durante 1-2 minutos. Seca cualquier líquido alrededor del borde del tubo con papel limpio antes del siguiente paso.
- Agregue con cuidado 600 μL de tampón de lisis de ARN (consulte la tabla de materiales). Siguiendo el protocolo del fabricante, agregue β-mercaptoetanol y vuelva a suspender el pellet.
- Pipetear el lisado directamente en un homogeneizador de lisado celular desechable (ver Tabla de Materiales), columna de centrifugación colocada en un tubo de recolección de 2 mL y centrifugar durante 2 min a velocidad máxima para homogeneizar. Deseche la columna de centrifugación del homogeneizador de lisado celular desechable y guarde el lisado homogeneizado.
- Añadir 1 volumen (600 μL) de etanol al 70% al lisado homogeneizado y mezclar mediante pipeteo. Pipetear con cuidado la muestra, incluido cualquier precipitado que pueda haberse formado, en una nueva columna de centrifugación en un tubo de recolección de 2 ml, pero no humedezca el borde. Centrífuga durante 15 s a >8.000 × g.
- Transfiera la columna de centrifugado (consulte la Tabla de materiales) a un nuevo tubo de recolección de 2 mL. Aplique 700 μL de tampón de lavado estricto (consulte la tabla de materiales) a la columna de centrifugado y centrifugue durante 15 s a >8.000 × g para lavar. Deseche el flujo.
- Abra con cuidado la columna de centrifugado y agregue 500 μL de tampón de lavado suave (consulte la tabla de materiales). Cierre el tapón y centrifugue a máxima velocidad (20.000 × g) durante 3 min.
- Transfiera la columna de centrifugación a un tubo de microcentrífuga de 1,5 mL y pipetee 30-50 μL de agua libre de RNasa directamente sobre la membrana de sílice. Centrifugar durante 1 min a >8.000 × g para eluir el ARN. Repita una vez más.
NOTA: El ARN se puede almacenar a -80 °C. - Realice el tratamiento con DNasa en una columna utilizando el conjunto de DNasa libre de RNasa (consulte la tabla de materiales), de acuerdo con el protocolo del fabricante.
- Genere ADNc utilizando 500 ng de ARN total y un sistema de reacción en cadena de la polimerasa (PCR) en tiempo real (consulte la tabla de materiales), de acuerdo con el protocolo del fabricante. Diluir todas las muestras de ADNc 1:25 en agua libre de ARNasa y utilizar 4 mL para realizar la PCR cuantitativa con transcripción inversa (qRT-PCR).
- Utilice un método de adición de cebadores (consulte la Tabla de materiales) para completar la qRT-PCR con 10 mM de cada uno de los cebadores que se muestran en la Tabla 2. qRT-PCR completa utilizando un sistema en tiempo real (ver Tabla de Materiales).
- Normalice los niveles de ARNm de los genes elegidos utilizando los niveles de ARNm de Rpl26 . Utilice el método 2-DDCt para calcular la cuantificación relativa.
- Para determinar si la expresión de un ARNm dado exhibe una oscilación circadiana, utilice una prueba basada en la probabilidad deLR_rhythmicity 39 (usando p≤ 0.01) para detectar la ritmicidad circadiana en una condición experimental40.
3. Plan de análisis de datos
NOTA: Se utilizará un inventario médico para categorizar a los participantes en función del uso de medicamentos43.
- Utilice un modelo lineal de efectos mixtos, en el que la edad, el sexo, el peso, la estatura, la presión arterial, la frecuencia cardíaca y otros factores de riesgo deben incluirse como covariables.
- Se debe incluir un término de efecto aleatorio para explicar la correlación de datos dentro del sujeto.
- Para el ajuste del modelo, se debe implementar una selección de variables hacia atrás con el principio jerárquico.
- Desde el modelo ajustado, la medición cambia del punto de tiempo 1 al punto de tiempo 2, dadas todas las covariables en el modelo ajustado.
Resultados
El protocolo propuesto incluye datos preliminares que sirven de validación para la metodología. El protocolo incorpora un analizador de flujo metabólico en tiempo real para examinar la función mitocondrial y la utilización del combustible celular, y la extracción de ARN y qRT-PCR para analizar los genes del ritmo circadiano (por ejemplo, BMAL1, CLOCK, Nfil2, Nr1d1, Dbp, Cry1, Per2).
En la Figura 1 se presenta la tasa de consumo de oxígeno (OCR) de PBMC humanos aislados de cinco participantes de control, 10 días después de un análisis inicial. Los datos se utilizan para comparar los valores previos y posteriores y muestran los valores medios de la respiración basal, la respuesta aguda, la respiración máxima y la capacidad ociosa tras la inyección de un control, etomoxir, UK5099 y BPTES. En particular, la Figura 1C muestra una respuesta aguda negativa significativa después de la inyección de etomoxir, pero no se observaron efectos significativos para la respiración basal, la respiración máxima o la capacidad ociosa.
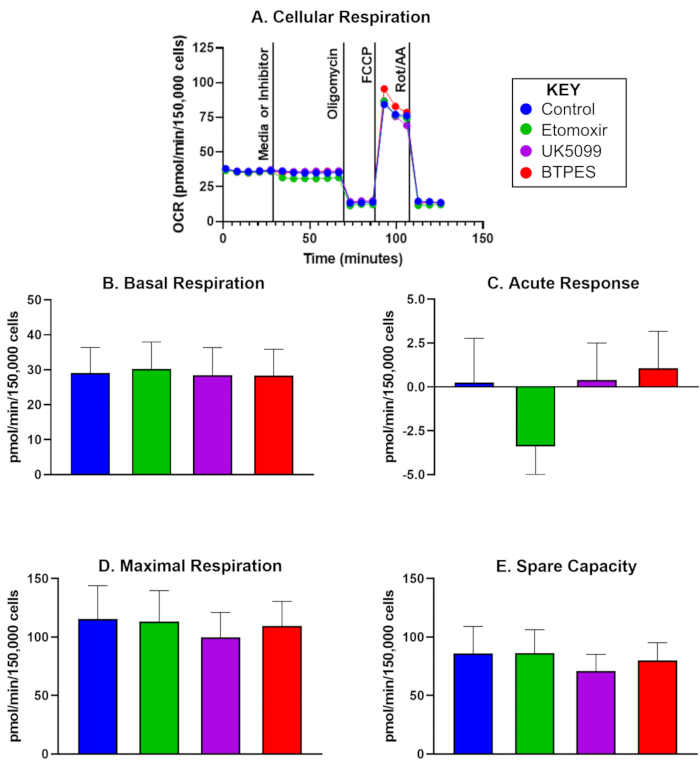
Figura 1: Consumo de oxígeno de células mononucleares aisladas de sangre periférica humana (PBMCs). (A) Tasa de consumo de oxígeno en tiempo real (OCR; pmol/(min∙150.000 células) de PBMCs aisladas de un sujeto control, medida con un analizador de flujo y evaluada con el ensayo de oxidación del sustrato. Las células se sembraron a una densidad de 150.000 células/pocillo. La primera inyección fue media (control) o inhibidora (etomoxir, UK5099 o BPTES; ver texto para más detalles) y ocurrió después de medir la tasa de respiración celular basal. La respuesta aguda a la limitación del sustrato mitocondrial se determinó como la diferencia de OCR basal antes y después de la inyección del inhibidor. La oligomicina, el inhibidor de la ATP sintasa, inhibe la respiración acoplada a la producción de ATP y produce la respiración por fuga de protones. FCCP, el desacoplador, induce la respiración máxima desacoplada; La rotenona y la antimicina A (inhibidores de los complejos I y III, respectivamente) inhiben toda la respiración, excepto la no mitocondrial (consulte el texto para obtener más detalles). (B-E) Cuantificación de la respiración celular (n = 5; los datos se representan como media ± DE). (B) OCR basal antes de la inyección del inhibidor, (C) respuesta aguda al inhibidor (cambio en el OCR en relación con la tasa basal antes de la inyección del inhibidor), (D) OCR máximo y (E) capacidad ociosa (diferencia entre el OCR máximo y el OCR basal después de la primera inyección). La respuesta aguda (C) a la inyección de etomoxir podría sugerir una mayor dependencia del OCR del ácido graso como sustrato energético en condiciones basales en comparación con los otros grupos de sustratos, sin un efecto notable sobre el OCR durante la alta demanda de energía (D). Haga clic aquí para ver una versión más grande de esta figura.
| Compuesto | AM (μL) añadida al compuesto | Culata (μM) | material (μL) para material de trabajo | AM (μL) para material de trabajo | Material de trabajo (μM) | Material de trabajo (μL) [puerto] | Conc. final (μM) |
| Etomoxir | 700 | 160 | 500 | 1500 | 40 | 20 [A] | 4 |
| UK5099 | 700 | 80 | 500 | 1500 | 20 | 20 [A] | 2 |
| BPTES | 700 | 120 | 500 | 1500 | 30 | 20 [A] | 3 |
| oligonucleótido | 420 | 150 | 300 | 2700 | 15 | 22 [B] | 1.5 |
| FCCP | 720 | 100 | 600 | 2400 | 20 | 25 [C] | 2 |
| Podredumbre/AA/H | 540 | 50 | 300 | 2700 | 5 | 27 [D] | 0.5 |
Tabla 1: Preparación de reactivos para el ensayo de oxidación del sustrato y concentraciones de stock, soluciones de trabajo y finales. Todos los reactivos forman parte de la prueba de esfuerzo mitogénico celular o de los kits de prueba de estrés oxidativo del sustrato. Abreviaturas: oligo = oligomicina; FCCP = desacoplador de cianuro de carbonilo-4 (trifluorometoxi) fenilhidrazona; Rot/AA/H = rotenona/antimicina A/Hoechst 33342. Etomoxir, UK5099, BTPES: inhibidores de la oxidación de ácidos grasos, glucosa y glutamina, respectivamente.
| Bmal1 | Adelante – GCACGACGTTCTTTCTTCTGT |
| Reverso – GCAGAAGCTTTTTCGATCTGCTTTT | |
| Reloj | Adelante – CGTCTCAGACCCTTCCTCAAC |
| Reverso – GTAAATGCTGCCTGGGTGGA | |
| Lloro1 | Adelante – ACTGCTATTGCCCTGTTGGT |
| Reverso – GACAGGCAAATAACGCCTGA | |
| Por 1 | Adelante – ATTCGGGTTACGAAGCTCCC |
| Reverso – GGCAGCCCTTTCATCCACAT | |
| Per2 | Adelante – CATGTGCAGTGGAGCAGATTC |
| Reverso – GGGGTGGTAGCGGATTTCAT | |
| Rev-erb α | Adelante – ACAGATGTCAGCAATGTCGC |
| Reverso – CGACCAAACCGAACAGCATC |
Tabla 2: Cebadores genéticos del reloj circadiano.
Discusión
La disminución de la función mitocondrial y la regulación del ritmo circadiano con la edad se consideran cada vez más factores que contribuyen a las enfermedades relacionadas con la edad. La alteración de los ritmos circadianos a través de modificaciones en el estilo de vida, como la dieta y la actividad física, representa una estrategia potencial para promover un envejecimiento saludable y reducir la disminución de la movilidad asociada con el envejecimiento. Sin embargo, los métodos actuales para evaluar directamente la función mitocondrial son invasivos y, a menudo, requieren una biopsia muscular, lo que puede plantear desafíos con el reclutamiento y la retención de participantes debido al dolor y los riesgos percibidos.
La evaluación de los marcadores de la salud circadiana y metabólica a través de métodos menos invasivos, como la extracción de sangre, proporcionaría resultados valiosos para explorar y probar objetivos terapéuticos en estudios futuros. Estos métodos mínimamente invasivos tienen el potencial de hacer avanzar en gran medida el campo al proporcionar nuevos conocimientos sobre la compleja interacción entre el ritmo circadiano y la salud metabólica y su impacto en la función. El objetivo de este estudio es evaluar la relación entre el metabolismo energético celular y el ritmo circadiano. En particular, el análisis de flujo bioenergético se utiliza para evaluar la función mitocondrial en diversas condiciones de disponibilidad de sustrato, junto con el monitoreo de la expresión génica de un grupo de genes circadianos en los glóbulos blancos de los participantes. Mediante el empleo de las dos ramas del análisis, la bioenergética y la expresión génica, se puede lograr una comprensión completa de la relación entre estos dos procesos fundamentales.
El análisis estadístico de estos datos de series temporales desde una perspectiva circadiana ofrece información sobre la fuerza, el alcance y el momento de los ritmos circadianos. En conclusión, la integración del análisis de la expresión génica, la bioenergética celular y las medidas metabólicas a nivel de organismo constituye un enfoque nuevo e innovador que arrojará luz sobre la interacción entre el metabolismo energético y los ritmos circadianos en los seres humanos.
En un estudio piloto, detectamos una respuesta aguda en el OCR de las PBMC a la limitación de la utilización de ácidos grasos (tras la inyección de etomoxir, un inhibidor de la carnitina palmitoil transferasa 1a). Este hallazgo sugiere que en las PBMC de este grupo particular de participantes, podría haber una dependencia de los ácidos grasos como sustrato energético durante la respiración basal. Sin embargo, la respiración máxima no se vio afectada, lo que sugiere que las fuentes de energía alternativas, como la glucosa y la glutamina, pueden compensar la menor utilización de ácidos grasos durante la alta demanda de energía. Los estudios futuros deben investigar si a) la bioenergética de los PBMC refleja la energía de todo el cuerpo y b) si intervenciones como la alimentación restringida en el tiempo podrían afectar las preferencias de sustrato energético.
Hay varios pasos críticos con respecto al análisis de flujo de los PBMC. En primer lugar, antes de evaluar las muestras experimentales, se debe optimizar la densidad de siembra de células (células por pocillo) asegurándose de que haya una distribución uniforme y continua de las células dentro de cada pocillo y en cada placa, la concentración final de FCCP debe optimizarse mediante la ejecución de pruebas de concentraciones utilizando las concentraciones 0, 0,125, 0,25, 0,5, 1,0 y 2,0 μM, y, si corresponde, la tinción Hoechst 33342 debe optimizarse siguiendo las instrucciones del fabricante. En segundo lugar, la normalización de los datos metabólicos a los parámetros celulares es fundamental para la comparabilidad de los datos entre experimentos. En el presente protocolo, se describe el recuento de células después de la conclusión del ensayo del analizador de flujo utilizando células teñidas con Hoechst 33342 y un dispositivo de imagen celular. Si no se dispone de un dispositivo adecuado, se pueden aplicar métodos alternativos de normalización, como la proteína celular total o el contenido de ADN nuclear por pocillo. Hay una modificación notable que se puede utilizar dentro del protocolo, en comparación con las que se han propuesto. Específicamente, el protocolo se puede completar utilizando un kit de ensayo individual para cada uno de los tres inhibidores, en comparación con solo los dos kits propuestos aquí (ver Tabla de Materiales).
El uso de PBMC como sustituto para estudiar la interacción entre el metabolismo energético y los ritmos circadianos en adultos mayores está limitado por la suposición de que su respuesta al tratamiento puede reflejar con precisión la respuesta en otros tejidos y órganos. Aunque este enfoque es novedoso y mínimamente invasivo, es importante reconocer que diferentes tejidos y órganos, como el cerebro, el hígado y el músculo esquelético, pueden reaccionar de manera diferente en diversas condiciones. Un estudio preclínico demostró que la expresión génica reloj estaba alterada en ratones alimentados y en ayunas, lo que llevó a la regulación parcial al alza de los genes objetivo de BMAL1 en el hígado y el tejido muscular, pero a la disminución de otros41. Estos tejidos y órganos periféricos son altamente representativos de los procesos metabólicos y pueden ser influenciados por señales ambientales que impactan en los mecanismos de expresión génicadel reloj 42. Se necesita más investigación para comprender completamente la relación entre los tejidos periféricos, los órganos y el reloj circadiano central.
Otra limitación es que los participantes no están descalificados para tomar cualquier receta, lo que podría plantear limitaciones para el análisis estadístico. Para contrarrestar esta limitación, en futuras investigaciones se utilizará un inventario médico, el cual ha sido validado en poblaciones de ancianos que toman medicamentos43. Los participantes se clasificarán en función de los medicamentos recomendados registrados en la sección de revisión de datos del protocolo. Hay un total de tres categorías, en términos de medicamentos, que han demostrado 1) acelerar el deterioro funcional, 2) ralentizar el deterioro funcional y 3) influir en la función del músculo esquelético.
Por último, la capacidad oxidativa mitocondrial del músculo esquelético humano exhibe un ritmo día-noche, alcanzando su punto máximo entre las 06:00 pm y las 11:00 pm y disminuyendo entre las 08:00 am y las 11:00 am44. Todavía no está claro si esto se mantiene para la capacidad oxidativa mitocondrial de las PBMC. Sin embargo, los datos preliminares sugieren que las PBMC y el metabolismo mitocondrial están relacionados45. Dado que la información sobre las biopsias musculares y las alteraciones en las PBMC no es tan clara, se debe tener precaución al analizar los resultados. Dada esta limitación, es importante tener en cuenta esta información al evaluar y desarrollar un protocolo, ya que puede proporcionar un contexto y una visión valiosos que podrían ayudar a garantizar la validez y eficacia del protocolo.
Hasta donde sabemos, ningún estudio previo ha evaluado los patrones de utilización de combustible o los ritmos circadianos a través de los métodos propuestos en este proyecto. Nuestro objetivo es examinar la capacidad de respuesta de los marcadores de la utilización del combustible mitocondrial y la salud circadiana a los cambios. Este estudio presenta un método mínimamente invasivo para medir un biomarcador altamente sensible, que puede servir como alternativa en futuros estudios intervencionistas donde la biopsia muscular no es factible.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este estudio fue financiado por el Older American's Independence Center (NIH/NIA P30AG028740), con la asistencia del Instituto de Ciencias Clínicas y Traslacionales (NIH/NCRR UL1TR000064).
Materiales
| Name | Company | Catalog Number | Comments |
| Agilent Technologies Cell Imaging | Agilent Technologies | Cell image software | |
| Agilent/Seahorse Wave desktop program | Agilent Technologies | 5994-0039EN | Software used to analyse data from the celluar analyser and stress test assay |
| Agilent/Seahorse XFe96 Flux Analyzer | Agilent Technologies | S7800B | Real-time cellular flux analyzer; flux analyser |
| Bar Code Reader | Agilent Technologies | G2615-90007 | |
| Seahorse Wave Desktop Software | Agilent technologies | Data acquisition software; assay analysis; wave program | |
| Seahorse XF 1.0 M Glucose solution | Agilent Technologies | 103577-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 100 mM Pyruvate solution | Agilent Technologies | 103578-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 200 mM Glutamine solution | Agilent Technologies | 103579-100 | Supplement to basal medium to make assay medium |
| Seahorse XF Cell Mito Stress Test | Agilent Technologies | 103015-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF Mito Fuel Flex Test | Agilent Technologies | 103260-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF RPMI Medium | Agilent Technologies | 103576-100 | Basal medium for PBMCs |
| Seahorse XFe96 FluxPak mini | Agilent Technologies | 102601-100 | Sensor cartridges and cell culture microplates |
| Cytation 1 Cell Imaging Multi-Mode Reader | Agilent/BioTek | Multimode reader to image cells | |
| CPT Sodium Heparin Tube, 16 x 125 mm x 8.0 mL | Becton Dickinson | 362753 | Blood collection tubes for isolation of peripheral blood mononuclear cells |
| CellTak Cell and Tissue Adhesive | Corning | 354240 | Cell adherent to coat cell culture microplate |
| Phosphate Buffered Saline | Corning | 21-040-CV | Buffer to wash blood cells |
| Ficoll Paque Plus | Cytiva | GE17-1440-02 | Gradient medium |
| Lunar Prodigy DXA scanner | General Electric | EN 60601-2-7 5.1 | Whole body lean mass and fat/lean tissue mass ratio |
| Freezing container, Nalgene Mr. Frosty | MilliporeSigma | C1562 | Freezing container used to slow-freeze cell suspension |
| Buffer EL. | Qiagen | 79217 | Erythrocyte lysis buffer |
| Buffer RLT | Qiagen | 79216 | RNA lysis buffer |
| Buffer RPE | Qiagen | 1018013 | Mild washing buffer |
| Buffer RW1 | Qiagen | 1053394 | Stringent washing buffer |
| QIAamp DNA Micro Kit | Qiagen | 56304 | DNA preps: QIAamp MinElute Columns, Proteinase K, Carrier RNA, Buffers, Collection Tubes (2 ml) |
| QIAamp RNA Blood Mini Kit | Qiagen | 52304 | RNA blood kit; Used to isolate RNA |
| QIAshredder | Qiagen | 79656 | disposable cell-lysate homogenizers |
| RNase-Free DNase Set | Qiagen | 79254 | Used to perform DNA digest |
| 2-Mercaptoethanol (Reagent) | Thermo Fisher Scientific | MFCD00004890 | |
| 2-mL collection tubes, 100 count | Thermo Fisher Scientific | AM12480 | |
| Fast SYBR Green Master Mix | Thermo Fisher Scientific | 4385612 | Primers are added to this and used to carry out qRT-PCR |
| Microcentrifuge Tubes, 1.5 mL | Thermo Fisher Scientific | 69715 | Used to hold RNA purification filter during RNA purification |
| Narrow p1000 pipette tips | Thermo Fisher Scientific | 02-707-402 | |
| QuantStudio 3 Real-Time PCR System, MiniAmp Plus Thermal Cycler, and 96-Well Plates Package | Thermo Fisher Scientific | A40393 | |
| Tempus Blood RNA Tube | Thermo Fisher Scientific | 4342792 | RNA Tube |
| Tempus Spin RNA Isolation kit | Thermo Fisher Scientific | 4380204 | RNA extraction and isolation |
Referencias
- UN Department of Economic and Social Affairs. Population Division 2019, World Population Prospects. UN Department of Economic and Social Affairs. , (2019).
- Anton, S., Leeuwenburgh, C. Fasting or caloric restriction for healthy aging. Experimental Gerontology. 48 (10), 1003-1005 (2013).
- Dziechciaż, M., Filip, R. Biological psychological and social determinants of old age: Bio-psycho-social aspects of human aging. Annals of Agricultural and Environmental Medicine. 21 (4), 835-838 (2014).
- Anton, S. D., et al. Flipping the metabolic switch: understanding and applying the health benefits of fasting. Obesity. 26 (2), 254-268 (2018).
- Fried, L. P., Guralnik, J. M. Disability in older adults: evidence regarding significance, etiology, and risk. Journal of the American Geriatrics Society. 45 (1), 92-100 (1997).
- Manini, T. Development of physical disability in older adults. Current Aging Science. 4 (3), 184-191 (2011).
- Chung, H. Y., et al. Molecular inflammation: underpinnings of aging and age-related diseases. Ageing Research Reviews. 8 (1), 18-30 (2009).
- Sun, N., Youle, R. J., Finkel, T. The mitochondrial basis of aging. Molecular Cell. 61 (5), 654-666 (2016).
- Tarasov, A. I., Griffiths, E. J., Rutter, G. A. Regulation of ATP production by mitochondrial Ca2. Cell Calcium. 52 (1), 28-35 (2012).
- Chistiakov, D. A., Sobenin, I. A., Revin, V. V., Orekhov, A. N., Bobryshev, Y. V. Mitochondrial aging and age-related dysfunction of mitochondria. Biomed Research International. 2014, 238463 (2014).
- Boengler, K., Kosiol, M., Mayr, M., Schulz, R., Rohrbach, S. Mitochondria and ageing: role in heart, skeletal muscle and adipose tissue. Journal of Cachexia, Sarcopenia, and Muscle. 8 (3), 349-369 (2017).
- Drew, B., et al. Effects of aging and caloric restriction on mitochondrial energy production in gastrocnemius muscle and heart. American Journal of Physiology Regulatory, Integrative and Comparative Physiology. 284 (2), R474-R480 (2003).
- Short, K. R., et al. Decline in skeletal muscle mitochondrial function with aging in humans. Proceedings of the National Academy of Sciences. 102 (15), 5618-5623 (2005).
- Musci, R. V., Hamilton, K. L., Miller, B. F. Targeting mitochondrial function and proteostasis to mitigate dynapenia. European Journal of Applied Physiology. 118 (1), 1-9 (2018).
- Picca, A., et al. Targeting mitochondrial quality control for treating sarcopenia: lessons from physical exercise. Expert Opinion on Therapeutic Targets. 23 (2), 153-160 (2019).
- Fernandez-Marcos, P. J., Auwerx, J. Regulation of PGC-1α, a nodal regulator of mitochondrial biogenesis. The American Journal of Clinical Nutrition. 93 (4), 884-890 (2011).
- Kim, Y., Triolo, M., Hood, D. A. Impact of aging and exercise on mitochondrial quality control in skeletal muscle. Oxidative Medicine and Cellular Longevity. 2017, 3165396 (2017).
- Wang, H., Hiatt, W. R., Barstow, T. J., Brass, E. P. Relationships between muscle mitochondrial DNA content, mitochondrial enzyme activity and oxidative capacity in man: alterations with disease. European Journal of Applied Physiology and Occupational Physiology. 80 (1), 22-27 (1999).
- Tian, Q., et al. Muscle mitochondrial energetics predicts mobility decline in well-functioning older adults: The baltimore longitudinal study of aging. Aging Cell. 21 (2), e13552 (2022).
- Sardon Puig, L., Valera-Alberni, M., Cantó, C., Pillon, N. J. Circadian rhythms and mitochondria: connecting the dots. Frontiers in Genetics. 9, 452 (2018).
- Gano, L. B., Patel, M., Rho, J. M. Ketogenic diets, mitochondria, and neurological diseases. Journal of Lipid Research. 55 (11), 2211-2228 (2014).
- Liesa, M., Shirihai, O. S. Mitochondrial dynamics in the regulation of nutrient utilization and energy expenditure. Cell Metabolism. 17 (4), 491-506 (2013).
- Lesnefsky, E. J., Chen, Q., Hoppel, C. L. Mitochondrial metabolism in aging heart. Circulation Research. 118 (10), 1593-1611 (2016).
- Hartman, M. L., et al. Relation of mitochondrial oxygen consumption in peripheral blood mononuclear cells to vascular function in type 2 diabetes mellitus. Vascular Medicine. 19 (1), 67-74 (2014).
- Mahapatra, G., et al. Blood-based bioenergetic profiling is related to differences in brain morphology in African Americans with Type 2 diabetes. Clinical Science. 132 (23), 2509-2518 (2018).
- Moore-Ede, M. C. Physiology of the circadian timing system: predictive versus reactive homeostasis. The American Journal of Physiology. 250 (5), R737-R752 (1986).
- Young, M. W. Life's 24-hour clock: molecular control of circadian rhythms in animal cells. Trends in Biochemical Sciences. 25 (12), 601-606 (2000).
- Yoo, S. H., et al. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proceedings of the National Academy of Sciences. 101 (15), 5339-5346 (2004).
- Zhang, R., Lahens, N. F., Ballance, H. I., Hughes, M. E., Hogenesch, J. B. A circadian gene expression atlas in mammals: implications for biology and medicine. Proceedings of the National Academy of Sciences. 111 (45), 16219-16224 (2014).
- de Goede, P., Wefers, J., Brombacher, E. C., Schrauwen, P., Kalsbeek, A. Circadian rhythms in mitochondrial respiration. Journal of Molecular Endocrinology. 60 (3), R115-R130 (2018).
- Hood, S., Amir, S. The aging clock: circadian rhythms and later life. The Journal of Clinical Investigation. 127 (2), 437-446 (2017).
- Sellix, M. T., et al. Aging differentially affects the re-entrainment response of central and peripheral circadian oscillators. The Journal of Neuroscience. 32 (46), 16193-16202 (2012).
- Sato, S., et al. Circadian reprogramming in the liver identifies metabolic pathways of aging. Cell. 170 (4), 664-677 (2017).
- Lundell, L. S., et al. et al. feeding alters lipid and amino acid metabolite rhythmicity without perturbing clock gene expression. Nature Communications. 11 (1), 4643 (2020).
- Perrin, L., et al. Transcriptomic analyses reveal rhythmic and CLOCK-driven pathways in human skeletal muscle. eLife. 7, e34114 (2018).
- Gutierrez-Monreal, M. A., Harmsen, J. -. F., Schrauwen, P., Esser, K. A. Ticking for metabolic health: the skeletal-muscle clocks. Obesity. 28, S46-S54 (2020).
- Wolff, C. A., et al. Defining the age-dependent and tissue-specific circadian transcriptome in male mice. bioRxiv. 42 (1), 111982 (2023).
- Wilson, D., Breen, L., Lord, J. M., Sapey, E. The challenges of muscle biopsy in a community based geriatric population. BMC Research Notes. 11 (1), 830 (2018).
- Ding, H., et al. Likelihood-based tests for detecting circadian rhythmicity and differential circadian patterns in transcriptomic applications. Briefings in Bioinformatics. 22 (6), 224 (2021).
- Ding, Z., Lamb, T. M., Boukhris, A., Porter, R., Bell-Pedersen, D. Circadian clock control of translation initiation factor eIF2α activity requires eIF2γ-dependent recruitment of rhythmic PPP-1 phosphatase in Neurospora crassa. mBio. 12 (3), e00871 (2021).
- Di Francesco, A., Di Germanio, C., Bernier, M., de Cabo, R. A time to fast. Science. 362 (6416), 770-775 (2018).
- Kalfalah, F., et al. Crosstalk of clock gene expression and autophagy in aging. Aging. 8 (9), 1876-1895 (2016).
- Psaty, B. M., et al. Assessing the use of medications in the elderly: methods and initial experience in the cardiovascular health study. Journal of Clinical Epidemiology. 45 (6), 683-692 (1992).
- van Moorsel, D., et al. Demonstration of a day-night rhythm in human skeletal muscle oxidative capacity. Molecular Metabolism. 5 (8), 635-645 (2016).
- Janssen, J. J. E., et al. Extracellular flux analyses reveal differences in mitochondrial PBMC metabolism between high-fit and low-fit females. American Journal of Physiology. Endocrinology and Metabolism. 322 (2), E141-E153 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados