Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Métodos optimizados para la inmovilización superficial de colágenos y ensayos de unión de colágeno
* Estos autores han contribuido por igual
En este artículo
Resumen
Este trabajo presenta un protocolo optimizado para inmovilizar y cuantificar de forma reproducible colágeno tipo I y III en microplacas, seguido de un protocolo mejorado de ensayo de unión in vitro para estudiar las interacciones colágeno-compuesto utilizando un método de fluorescencia resuelto en el tiempo. Se proporciona el posterior análisis de datos paso a paso y la interpretación de los datos.
Resumen
La fibrosis se produce en varios tejidos como respuesta reparadora a una lesión o daño. Sin embargo, si es excesiva, la fibrosis puede provocar cicatrices en los tejidos e insuficiencia orgánica, lo que se asocia con una alta morbilidad y mortalidad. El colágeno es un impulsor clave de la fibrosis, siendo el colágeno tipo I y tipo III los principales tipos involucrados en muchas enfermedades fibróticas. A diferencia de los protocolos convencionales utilizados para inmovilizar otras proteínas (por ejemplo, elastina, albúmina, fibronectina, etc.), no se dispone de protocolos completos para inmovilizar de forma reproducible diferentes tipos de colágeno con el fin de producir recubrimientos estables. La inmovilización del colágeno es sorprendentemente desafiante porque múltiples condiciones experimentales pueden afectar la eficiencia de la inmovilización, incluido el tipo de colágeno, el pH, la temperatura y el tipo de microplaca utilizada. Aquí, se proporciona un protocolo detallado para inmovilizar y cuantificar de manera reproducible los colágenos de tipo I y III, lo que da como resultado geles/películas estables y reproducibles. Además, este trabajo demuestra cómo realizar, analizar e interpretar estudios de unión a fluorescencia resueltos en el tiempo in vitro para investigar las interacciones entre los colágenos y los compuestos candidatos a unirse al colágeno (por ejemplo, un péptido conjugado con un quelato metálico que transporta, por ejemplo, europio [Eu(III)]). Este enfoque se puede aplicar universalmente a diversas aplicaciones biomédicas, incluido el campo de la imagen molecular para desarrollar sondas de imagen dirigidas, el desarrollo de fármacos, los estudios de toxicidad celular, los estudios de proliferación celular y los inmunoensayos.
Introducción
La acumulación de tejido conectivo fibroso como parte del proceso natural de cicatrización de heridas después de una lesión tisular se conoce como fibrosis. Sin embargo, si la deposición de tejido fibroso no termina y continúa más allá de lo necesario para la reparación del tejido, entonces la fibrosis se vuelve excesiva 1,2. La fibrosis excesiva deteriora la fisiología y la función de los órganos y podría provocar daños en los órganos y, potencialmente, insuficiencia orgánica 3,4,5. Dos de los principales impulsores de la fibrosis son las proteínas de la matriz extracelular (MEC), el colágeno tipo I y el tipo III6. El colágeno es una proteína estructural que se encuentra en varios órganos y que constituye aproximadamente un tercio del contenido total de proteínasdel cuerpo humano. Existen 28 tipos diferentes de colágenos identificados por la secuenciación del genoma humano, y los más abundantes son los colágenos fibrilares7. El colágeno fibrilar primario es el colágeno tipo I, que proporciona al ECM resistencia a la tracción y a la deformación8. El colágeno tipo III es un componente estructural que proporciona elasticidad y se colocaliza con el colágeno tipo I. Se expresa durante la embriogénesis y se encuentra naturalmente en pequeñas cantidades en la piel, los músculosy los vasos sanguíneos de los adultos.
La síntesis de colágeno in vivo comienza con un proceso intracelular en el que el ARNm se transcribe en el núcleo y luego pasa al citoplasma, donde se traduce. Después de la traducción, la cadena formada sufre una modificación postraduccional en el retículo endoplásmico, donde se forma el procolágeno (el precursor del colágeno). A continuación, el procolágeno viaja al aparato de Golgi para su modificación final antes de ser excretado al espacio extracelular10. A través de la escisión proteolítica, el procolágeno se transforma en tropocolágeno. A continuación, se reticula a través de una vía de reticulación mediada por enzimas catalizada por la enzima lisil oxidasa (LOX) o a través de una vía de reticulación no enzimática que implica la reacción de Maillard11. Los protocolos in vitro para inmovilizar el colágeno se basan principalmente en la capacidad del colágeno para autoensamblarse. El colágeno se extrae de los tejidos en función de su solubilidad, que depende en gran medida del grado de reticulación de las fibrillas de colágeno individuales7. El colágeno fibrilar se disuelve en ácido acético y las fibrillas pueden reformarse cuando se ajustan el pH y la temperatura12. In vitro, la fibrilogénesis del colágeno puede ser vista como un proceso de dos etapas7. La primera etapa es la fase de nucleación, donde las fibras de colágeno forman dímeros y fibrillas de trímero antes de que se reorganicen para formar una estructura helicoidal triple. La segunda fase es la fase de crecimiento, donde las fibrillas comienzan a crecer lateralmente y dan lugar a la formación característica de la banda D, que generalmente se observa por cambios en la turbidez7. Los estudios de microscopía de fuerza atómica (MFA) también han revelado que el colágeno tipo I y tipo III tiene características diferentes (Tabla 1)13.
Para estudiar las interacciones de unión entre el colágeno y otros compuestos, el colágeno debe inmovilizarse de forma reproducible en los pocillos de las microplacas. Existen varios protocolos para inmovilizar el colágeno soluble 14,15,16. Las microplacas disponibles en el mercado que están prerrecubiertas con colágeno se utilizan normalmente para el cultivo celular. Sin embargo, las microplacas prerrevestidas tienen una capa muy delgada de una cantidad desconocida de colágeno recubierta en los pocillos, lo que las hace inadecuadas para ensayos de unión in vitro. Existen varios retos a la hora de inmovilizar el colágeno en los pocillos de las placas. Uno de los principales desafíos es elegir un tipo adecuado de microplaca, ya que los diferentes tipos de colágeno (por ejemplo, tipo I y III) tienen diferentes propiedades químicas y, por lo tanto, se inmovilizan de manera más estable y efectiva según el material de la microplaca. Otro desafío son las condiciones experimentales del protocolo de inmovilización, ya que el proceso de fibrilogénesis depende de múltiples factores, entre ellos la temperatura, el pH, la concentración de colágeno y la concentración iónica del tampón7.
Para estudiar las interacciones entre el colágeno (el objetivo) y otros compuestos (es decir, un péptido dirigido), también es necesario desarrollar un ensayo de cribado sólido para investigar la especificidad y selectividad del compuesto hacia el objetivo midiendo la constante de disociación, Kd. La posición del equilibrio de formación de un complejo bimolecular entre una proteína (colágeno) y un ligando se expresa en términos de la constante de asociación Ka, cuya magnitud es proporcional a la afinidad de unión. Sin embargo, lo más común es que los bioquímicos expresen relaciones de afinidad en términos de la constante de disociación de equilibrio, Kd, del complejo bimolecular, que se define como Kd = 1/Ka (Kd y es el inverso de Ka). Cuanto menor sea el valorde K d, más fuerte será la fuerza de unión entre la proteína y el ligando. La ventaja de usar Kd para comparar la afinidad de unión de diferentes ligandos para la misma proteína (y al revés) está relacionada con el hecho de que las unidades de Kd para un complejo bimolecular son mol/L (es decir, unidad de concentración). En la mayoría de las condiciones experimentales, el valor de Kd corresponde a la concentración de ligando que conduce a una saturación del 50% de los sitios de unión disponibles en el objetivo en el equilibrio17,18. La constante de disociación se extrae típicamente analizando la ocupación fraccional del receptor (FO), que se define como la relación entre los sitios de unión ocupados y el total de sitios de unión disponibles, en función de la concentración de ligando. Esto se puede hacer siempre que se disponga de un ensayo analítico capaz de distinguir y medir la cantidad de ligando unido.
Los ensayos de unión a ligandos in vitro se pueden realizar utilizando varios métodos bioanalíticos, como la fotometría óptica, los métodos de radioligandos, la espectrometría de masas de plasma acoplado inductivamente (ICP-MS) y la resonancia de plasmones de superficie (SPR). Entre los métodos fotométricos, los basados en la emisión de fluorescencia suelen requerir el marcaje de ligandos o proteínas con fluoróforos para aumentar la sensibilidad y mejorar el límite de detección del ensayo. Los quelatos de ciertos iones lantánidos (III), como el Eu (III), son muy atractivos como fluoróforos, ya que tienen grandes desplazamientos de Stokes, bandas de emisión estrechas (que proporcionan una buena relación señal-ruido), fotoblanqueo limitado y larga vida útil de emisión. Es importante destacar que esta última propiedad permite el uso de fluorescencia resuelta en el tiempo (TRF) de fluoróforos Eu(III) para abolir la autofluorescencia de fondo19. En la versión del inmunoensayo fluorescente de lantánidos mejorado por disociación (DELFIA) del ensayo TRF basado en Eu(III), los ligandos marcados con un quelato Eu(III) no luminiscente se incuban con el receptor inmovilizado en microplacas. El complejo ligando/receptor marcado se separa del ligando no unido, y la fluorescencia de Eu(III) se activa por disociación del complejo Eu(III) a un pH ácido, seguido de una recomplejación con un quelante que mejora la fluorescencia para formar un complejo de Eu(III) altamente fluorescente incrustado en micelas20.
El paso de descomplexión se puede lograr razonablemente con quelantes, como el pentaacetato de dietilentriamina (DTPA), que muestran una cinética de descomplejación rápida. Sin embargo, los complejos Eu(III) con ciertos quelantes macrocíclicos, como el DOTA (ácido 1,4,7,10-tetraazaciclododecano1,4,7,10-tetraacético) y sus derivados monoamida (DO3AAm), muestran una alta estabilidad termodinámica y una inercia cinética muy alta. En este caso, los pasos de descomplejación deben optimizarse con precisión para lograr una activación suficiente y reproducible de TRF21 basada en Eu(III). Cabe destacar que los complejos lantánidos (Ln(III))-DOTA y Ln(III)-DO3AAm son los más comúnmente empleados como agentes de contraste para la obtención de imágenes moleculares in vivo mediante técnicas de resonancia magnética (RM)22. Por lo tanto, el ensayo TRF basado en Ln(III) es la herramienta de elección para estudiar in vitro la afinidad de unión de las sondas moleculares de resonancia magnética con sus objetivos biológicos previstos. En la actualidad, se carece de protocolos completos y reproducibles para inmovilizar el colágeno de tipo I y tipo III, así como de una línea reproducible para realizar experimentos de TRF de unión in vitro con EU(III). Para superar estas limitaciones, se desarrollaron métodos reproducibles para autoensamblar e inmovilizar colágeno tipo I y tipo III y generar geles y películas estables, respectivamente, con la concentración suficiente de colágeno requerida para los ensayos de unión in vitro . Se presenta un protocolo optimizado para Eu(III) TRF de complejos altamente inertes basados en Eu(III)-DO3Aam. Por último, se demuestra un ensayo TRF optimizado en microplacas in vitro de Eu(III) para medir el Kd de los ligandos marcados con Eu(III) hacia el colágeno inmovilizado de tipo I y tipo III (Figura 1).
Protocolo
NOTA: Toda la información del producto utilizada para este trabajo se presenta en la Tabla de Materiales.
1. Inmovilización de colágeno
NOTA: Asegúrese de que cada pocillo de la microplaca utilizada durante el ensayo de unión tenga pocillos adyacentes libres para evitar la fluorescencia cruzada. Realiza esta parte del protocolo con hielo porque el colágeno se autoensambla al aumentar las temperaturas y los niveles de pH. Realice este procedimiento en una campana de cultivo de tejidos y en condiciones estériles porque las microplacas se incuban posteriormente en una incubadora de cultivo de tejidos (TC).
- Inmovilización de colágeno tipo I en las microplacas de 96 pocillos (Figura 2)
Día 1- Prepara una bandeja de silicona con hielo. Coloque el vial que contiene colágeno tipo I, la solución salina fría tamponada con fosfato (PBS) 10x y las microplacas en hielo, y rocíe todo con etanol al 70%. Coloque el material debajo del capó TC.
- Neutraliza el colágeno utilizando volúmenes iguales de colágeno tipo I y 10x PBS (pH 7,4).
- Invierta la solución unas cuantas veces, asegurándose de que no se formen burbujas.
- Añadir 100 μL del colágeno neutralizado a cada dos pocillos y a cada dos filas de la microplaca, e incubar a 37 °C durante 18-20 h para evaporar el colágeno hasta la sequedad.
Día 2 - Lave las microplacas con 100 μL de 1x PBS, pH 7,4, dos veces para eliminar el colágeno no unido.
- Transfiera las microplacas a la incubadora a 37 °C durante otras 2 h para que se sequen antes de usarlas para otros experimentos de unión.
- Inmovilización de colágeno tipo III en las microplacas de 96 pocillos (Figura 3)
Día 1- Prepara una bandeja de silicona con hielo. Coloque el vial que contiene colágeno tipo III, el PBS frío 10x y las microplacas en hielo, y rocíe todo con etanol al 70%. Coloque el material debajo del capó TC.
- Neutralizar el colágeno utilizando volúmenes iguales de colágeno tipo III y 10x PBS (pH 7,4).
- Agregue 70 μL del colágeno neutralizado a cada dos pocillos y cada dos filas de la microplaca, e incube a 37 °C durante 2 h colocando la microplaca debajo de la campana de cultivo de tejidos para evaporar el colágeno hasta la sequedad.
Día 2 - Lave las microplacas con 70 μL de 1x PBS, pH 7,4, dos veces para eliminar el colágeno no unido.
- Transfiera las microplacas a la incubadora durante 1 h a 37 °C y, a continuación, transfiera las microplacas al banco y deje que se sequen durante 1 h antes de utilizarlas en otros experimentos de unión.
2. Evaluación de la estabilidad de los geles/películas de colágeno inmovilizados
- Incubación con PBS durante 1 h
NOTA: Durante el experimento de unión, incubar el colágeno inmovilizado con el compuesto de interés. Es importante investigar la estabilidad del gel o película de colágeno resultante. Para hacer esto, mida la estabilidad de tres condiciones: sin lavado = mide el colágeno inmovilizado directamente después de la incubación; lavado = mide el colágeno inmovilizado después de lavar la placa dos veces con 100 μL de PBS; y 1 h PBS mimic & wash = mide el colágeno inmovilizado después de incubar durante 1 h con PBS seguido de dos lavados con PBS. A continuación, se explica el método de incubación PBS.- Añadir 70 μL de PBS (1x) a cada uno de los pocillos recubiertos de colágeno, e incubar la microplaca a temperatura ambiente durante 1 h.
- Aspirar el exceso de líquido de cada pocillo con una pipeta y lavar con PBS (1x) dos veces antes de realizar el ensayo de cuantificación de proteínas que se describe a continuación.
- Cuantificación de la cantidad de colágeno inmovilizado mediante un ensayo de ácido bicinconínico (BCA)
NOTA: Utilice el kit de ensayo de proteínas BCA de Pierce (tabla de materiales) siguiendo las instrucciones del fabricante. Realice los respectivos estándares de colágeno para este ensayo. El rango de concentración para el colágeno I es de 0 a 3.000 μg/mL y para el colágeno III de 0 a 750 μg/mL. En total, haz 11 estándares por colágeno.- Prepare el volumen total de reactivo de trabajo (WR) necesario siguiendo las instrucciones del fabricante.
- Añadir 25 μL de cada uno de los estándares de colágeno en los pocillos de la microplaca (por duplicado). Estas soluciones se utilizan para dibujar la curva estándar.
- Añadir 200 μL de solución de reactivo de trabajo a cada uno de los pocillos que contengan los patrones y a los pocillos recubiertos con concentraciones desconocidas de colágeno.
- Coloque la microplaca en un agitador de platos durante 30 s. Cubra las microplacas e incube a 37 °C durante 30 min.
- Retire las microplacas y deje enfriar a temperatura ambiente. Mida la absorbancia a 560 nm con un lector de placas.
- Traza una curva de calibración comparando el A560 (AU) con la concentración (μg/mL) de las 11 soluciones estándar, y utiliza la curva de calibración para calcular la cantidad de colágeno.
3. Ensayo de unión al ligando TRF de europio (III) (Figura 1)
NOTA: El compuesto utilizado es un candidato a péptido de unión al colágeno (CBP) marcado con un único complejo Eu(III)-DO3AAm, denominado Eu(III)-DO3AAm-CBP (Figura 4).
- Incubación de las placas recubiertas de colágeno con el compuesto Eu(III)-DO3AAm-CBP
- Prepare soluciones del compuesto Eu(III)-DO3AAm-CBP con concentraciones que oscilan entre 0,1-15 μM (0,1 μM, 0,5 μM, 1 μM, 3 μM, 5 μM, 7 μM, 10 μM y 15 μM) en 1x PBS.
- Agregue 75 μL de cada concentración de compuesto en los pocillos recubiertos de colágeno (Placa A). Realiza el experimento por triplicado para calcular la cantidad de compuesto que se une al colágeno.
- Utilice una segunda placa sin recubrimiento (placa B) y agregue 75 μL de cada compuesto a los pocillos vacíos para calcular la unión no específica del compuesto a la placa. Utilice triplicados para cada concentración.
- Incubar las microplacas durante 1 h a temperatura ambiente.
- Con una pipeta, aspire y deseche el exceso de solución de cada pocillo, y lave los pocillos con 1x PBS dos veces para eliminar el exceso de compuesto suelto. Realice este paso utilizando las microplacas recubiertas de colágeno y sin recubrimiento.
- A una tercera placa sin recubrimiento (placa C), agregue 10 μL del mismo rango de concentraciones de Eu(III)-DO3AAm-CBP (por duplicado). Utilice la lectura de fluorescencia del Eu(III)-DO3AAm-CBP en solución para hacer una curva de calibración.
NOTA: No lave ni aspire la solución de esta placa.
- Extracción ácida de lecturas de europio (III) y fluorescencia resuelta en el tiempo (TRF)
NOTA: Consulte la información complementaria sobre la preparación y calibración de los volúmenes de la solución ácida (AS) y la solución tampón (BS). Los volúmenes de AS y BS requeridos para lograr de manera reproducible un pH óptimo fueron 54 μL y 46 μL, respectivamente, en este trabajo. Realice la siguiente operación en la placa A, la placa B y la placa C.- Añadir 54 μL de solución ácida (AS) a cada pocillo y colocar la placa en la incubadora a 37 °C durante 90 min, cubriendo las microplacas con papel de aluminio para evitar la evaporación. La temperatura y el tiempo de incubación deben controlarse cuidadosamente para lograr una descomplejación reproducible.
- Añada 46 μL de solución tampón (BS) a cada pocillo y agite suavemente la placa durante 30 s.
- Añadir 100 μL de solución de mejora (ES) y agitar la placa durante 30 s.
- Espere 30 minutos antes de leer la placa con un lector de placas TRF. Utilice los parámetros enumerados en la Tabla 2.
4. Análisis de datos
- Cuantificación de la concentración de colágeno inmovilizado en los pocillos
- Obtener la ecuación de la curva de calibración del A560 (AU) frente a la concentración (μg/mL) de las 11 soluciones patrón.
- Utilice las lecturas de absorbancia obtenidas de los pocillos que contienen los estándares de colágeno.
- Tabular los valores promedio de los pocillos duplicados y trazar la absorbancia media frente a las concentraciones conocidas de proteínas (colágeno) (μg/mL) para obtener la ecuación de la curva estándar.
- Utilice los valores de absorbancia para calcular la masa (μg) y la concentración (M) de colágeno inmovilizado.
- Calcule los valores promedio de absorbancia en los tres pocillos que contenían colágeno inmovilizado y registre la desviación estándar.
- Utilice la ecuación de la curva estándar obtenida de la curva estándar de colágeno (paso 2.2.6) para convertir la absorbancia medida de los pocillos recubiertos de colágeno en concertación. A partir de esto, se calculó la concentración de colágeno que se inmovilizó dentro de los pocillos experimentales en μg/mL.
- Convierta la concentración calculada en el paso 4.1.2.2 (μg/mL) primero a gramos/litro y luego, en función del peso molecular del colágeno, a molar (M).
- Por último, se calcula la masa del colágeno inmovilizado en cada pocillo dividiendo la concentración por el volumen de colágeno añadido al pocillo (100 μL para el colágeno tipo I y 70 μL para el colágeno tipo III).
- Obtener la ecuación de la curva de calibración del A560 (AU) frente a la concentración (μg/mL) de las 11 soluciones patrón.
- Cálculo de la constante de disociación (Kd) (Figura 4)
- Extraiga las lecturas de fluorescencia.
- Exporte las lecturas de fluorescencia del lector de placas a una hoja de cálculo.
NOTA: En los ensayos de unión, es importante tener en cuenta la posible unión no específica de un compuesto a la superficie plástica de las placas. - Calcule los valores medios de las mediciones por triplicado de cada concentración de compuesto para las tres placas diferentes: las lecturas de unión específicas de los pocillos recubiertos (Placa A), la unión no específica de los pocillos no recubiertos (Placa B) y el Eu(III)-DO3AAm-CBP total en solución en los pocillos no recubiertos (Placa C).
- Determine los valores de fluorescencia para el compuesto unido restando las lecturas de fluorescencia de los pocillos sin recubrimiento (Placa B) de las de los pocillos recubiertos (Placa A).
Ecuación 1: Determinación de la fluorescencia ligada17:
Fluoresceescnece ligada = Específica (pocillos recubiertos) - Inespecífica (pocillos no recubiertos) - Genere una curva de calibración utilizando las lecturas del compuesto marcado con Eu(III) en solución (Placa C). Trazar las lecturas de fluorescencia obtenidas frente a la concentración del compuesto marcado con Eu(III). Realice un ajuste de regresión lineal.
- Exporte las lecturas de fluorescencia del lector de placas a una hoja de cálculo.
- Convierta las lecturas de fluorescencia en concentraciones.
- Convierta las lecturas de la fluorescencia ligada (paso 4.2.3) en concentración utilizando la curva de fluorescencia estándar a partir de los datos generados utilizando las concentraciones de compuestos en solución (paso 4.2.1.4).
NOTA: Al comparar las propiedades de unión de un compuesto hacia diferentes proteínas objetivo que inmovilizan a diferentes concentraciones, será necesario considerar estas últimas al calcular la cantidad de compuesto unido al objetivo (es decir, compuesto/proteína unido). - Divida la concentración del compuesto unido por la concentración de la proteína inmovilizada en el pocillo.
NOTA: Para este cálculo, utilice la concentración de colágeno inmovilizado que se calculó después de que los pocillos se incubaron con PBS durante 1 h (el llamado experimento de imitación de PBS; sección 2.1 anterior). Esto es para tener en cuenta las posibles pérdidas de colágeno durante la etapa de incubación y la etapa de lavado que no contribuirán a la señal de fluorescencia final. - Represente los datos utilizando un diagrama de dispersión que tenga las concentraciones del compuesto en el eje x (μM) y el compuesto/proteína unido en el eje y.
- Convierta las lecturas de la fluorescencia ligada (paso 4.2.3) en concentración utilizando la curva de fluorescencia estándar a partir de los datos generados utilizando las concentraciones de compuestos en solución (paso 4.2.1.4).
- Obtenga los valores Kd .
- Ajuste los datos adquiridos en el paso 4.2.2.3 utilizando dos posibles modelos cinéticos de unión: unión de un sitio y unión de un sitio con una pendiente de colina. Las ecuaciones para cada modelo se muestran en la Figura 6.
- Elija el modelo que proporcione un ajuste no ambiguo con el valor R cuadrado más alto al ajustar los datos.
- Excluya los valores atípicos (s) para cada conjunto de lecturas de fluorescencia por concentración por placa.
- Calcule el valor final de Kd y presente los datos como la media ± la desviación estándar de experimentos independientes.
NOTA: Para obtener resultados sólidos, realice mediciones triplicadas dentro de cada placa y al menos tres experimentos independientes con diferentes microplacas.
- Calcula la ocupación fraccional (FO).
NOTA: A partir de la Ecuación 2, se desconoce la concentración del objetivo y, por lo tanto, al usar el álgebra y el Kd, de la Ecuación 3, surge una ecuación viable para calcular la ocupación fraccional en forma de Ecuación 4.
Ecuación 2: Definición de ocupación fraccional17:
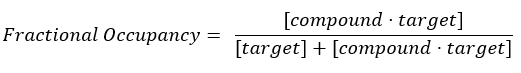
Ecuación 3: La constante de disociación, Kd, que es la concentración a la que el compuesto ocupa el 50% del objetivo en el equilibrio17:
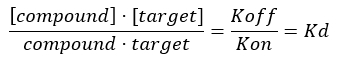
Ecuación 4: Ecuación reordenada para calcular la ecuación FO17:
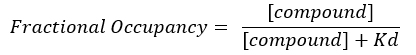
- Calcule el FO utilizando los valores independientes de Kd obtenidos para cada placa individual. Represente los resultados, la media y las desviaciones estándar del FO frente a la concentración del compuesto.
- Reporte el FO con valores que van de 0 a 1 o como un porcentaje con valores que van de 0% a 100%.
- Extraiga las lecturas de fluorescencia.
Resultados
Evaluación de la estabilidad y concentración de colágeno tipo I y tipo III inmovilizado en geles/películas
La cuantificación de la concentración de colágeno inmovilizado por pocillo se realizó utilizando tres condiciones diferentes: a) en pocillos sin lavado con PBS después de inmovilizar las proteínas (sin lavado); b) en pocillos con una etapa de lavado (dos veces con PBS) después de la inmovilización para eliminar cualquier proteína no recubierta; c) en...
Discusión
Este trabajo presenta un método reproducible para inmovilizar colágeno tipo I y tipo III. También se muestra un protocolo para adquirir, analizar e interpretar in vitro los datos de unión a TRF de Eu(III) para caracterizar las propiedades de unión de un ligando candidato hacia el colágeno de tipo I y III. Los protocolos de inmovilización de colágeno tipo I y tipo III que aquí se presentan se desarrollaron y optimizaron teniendo en cuenta trabajos publicados previamente ...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos a los siguientes financiadores por apoyar este trabajo: (1) el Consejo de Investigación Médica del Reino Unido (MR/N013700/1) y el King's College de Londres, miembro de la Asociación de Formación Doctoral en Ciencias Biomédicas del MRC; (2) Subvención del programa BHF RG/20/1/34802; (3) Subvención para proyectos BHF PG/2019/34897; (4) Subvención del Centro King's BHF para la Excelencia en Investigación RE/18/2/34213; (5) el Programa de la Iniciativa Científica del Milenio ANID - ICN2021_004; y (6) FB210024 de subvención basal ANID.
Materiales
| Name | Company | Catalog Number | Comments |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 10x PBS | Gibco | 14200075 | Use this to make 1x PBS by diluting in water (1:10) |
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M HCL | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| 2M Sodium hydroxide +2M Glycine | Made in house and details are in the supporting document | ||
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| Cell-star 96 well microplate | Greiner Bio-One | 655 160 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| DELFIA enhacement solution | Perkin Elmer | 1244-104 | |
| Ice | |||
| Ice | |||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Infinite 200 PRO NanoQuant microplate reader | TECAN | ||
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| Non-binding (NBS) 96 well microplates | Corning | 3641 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH electrode Inlab Routine | Mettler Toledo | 51343050 | |
| pH meter (sevenCompact) | Mettler Toledo | ||
| pH meter (sevenCompact) | Mettler Toledo | ||
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Pierce BCA protein assay kit | Thermofisher | 23227 | |
| Tissue culture incubator (37 °C, 5% CO2) | |||
| Type I bovine collagen, 3 mg/mL | Corning | 354231 | |
| Type III human placenta collagen, 0.99 mg/mL | Advanced Biomatrix | 5021 |
Referencias
- Distler, J. H. W., et al. Review: Frontiers of antifibrotic therapy in systemic sclerosis. Arthritis and Rheumatology. 69 (2), 257-267 (2017).
- Wynn, T. A. Fibrotic disease and the TH1/TH2 paradigm. Nature Reviews Immunology. 4 (8), 583-594 (2004).
- Saha, P., et al. Magnetic resonance T1 relaxation time of venous thrombus is determined by iron processing and predicts susceptibility to lysis. Circulation. 128 (7), 729-736 (2013).
- Mirshahi, M., et al. Defective thrombolysis due to collagen incorporation in fibrin clots. Thrombosis Research. 8, 73-80 (1988).
- Comerota, A. J. The ATTRACT trial: Rationale for early intervention for iliofemoral DVT. Perspectives in Vascular Surgery and Endovascular Therapy. 21 (4), 221-225 (2009).
- Bateman, E. D., Turner-Warwick, M., Adelmann-Grill, B. C. Immunohistochemical study of collagen types in human foetal lung and fibrotic lung disease. Thorax. 36 (9), 645-653 (1981).
- Pawelec, K. M., Best, S. M., Cameron, R. E. Collagen: A network for regenerative medicine. Journal of Materials Chemistry B. 4 (40), 6484-6496 (2016).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Copes, F., Pien, N., Van Vlierberghe, S., Boccafoschi, F., Mantovani, D. Collagen-based tissue engineering strategies for vascular medicine. Frontiers in Bioengineering and Biotechnology. 7, 166 (2019).
- Veis, A. The biochemistry of collagen. Annals of Clinical and Laboratory Science. 5 (2), 123-131 (1975).
- Bielajew, B. K., Hu, J. C., Athanasiou, K. A. Collagen: Quantification, biomechanics, and role of minor subtypes in cartilage. Nature Reviews Materials. 5, 730-747 (2020).
- Zhao, Z., et al. Structural and functional plasticity of collagen fibrils. DNA and Cell Biology. 38 (4), 367-373 (2019).
- Eryilmaz, E., Teizer, W., Hwang, W. In vitro analysis of the co-assembly of type-I and type-III collagen. Cellular and Molecular Bioengineering. 10 (1), 41-53 (2017).
- Jagnow, J., Clegg, S. Klebsiella pneumoniae MrkD-mediated biofilm formation on extracellular matrix- and collagen-coated surfaces. Microbiology. 149 (9), 2397-2405 (2003).
- O'Sullivan, D., O'Neill, L., Bourke, P. Direct plasma deposition of collagen on 96-well polystyrene plates for cell culture. ACS Omega. 5 (39), 25069-25076 (2020).
- Caravan, P., et al. Collagen-targeted MRI contrast agent for molecular imaging of fibrosis. Angewandte Chemie - International Edition. 46 (43), 8171-8173 (2007).
- Copeland, R. A. . Enzymes: A Practical Introduction to Structure, Mechanism, and Data Analysis. , (2000).
- Salahudeen, M. S., Nishtala, P. S. An overview of pharmacodynamic modelling, ligand-binding approach and its application in clinical practice. Saudi Pharmaceutical Journal. 25 (2), 165-175 (2017).
- Bünzli, J. C. G., Piguet, C. Taking advantage of luminescent lanthanide ions. Chemical Society Reviews. 34 (12), 1048-1077 (2005).
- Hemmilii, I. Luminescent lanthanide chelates - A way to more sensitive diagnostic methods. Journal of Alloys and Compounds. 225 (1-2), 480-485 (1995).
- De Silva, C. R., Vagner, J., Lynch, R., Gillies, R. J., Hruby, V. J. Optimization of time-resolved fluorescence assay for detection of europium-tetraazacyclododecyltetraacetic acid-labeled ligand-receptor interactions. Analytical Biochemistry. 398 (1), 15-23 (2010).
- Digilio, G., Lacerda, S., Lavin Plaza, B., Phinikaridou, A. Extracellular matrix targeted MRI probes. Analysis & Sensing. 3 (1), (2022).
- Phinikaridou, A., et al. Tropoelastin: A novel marker for plaque progression and instability. Circulation. Cardiovascular imaging. 11 (8), 007303 (2018).
- Guzaeva, T. V., et al. Protein A used in DELFIA for the determination of specific antibodies. Immunology Letters. 35 (3), 285-289 (1993).
- Nasiri, A. H., Nasiri, H. R. Polymerase assays for lead discovery: An overall review of methodologies and approaches. Analytical Biochemistry. 563, 40-50 (2018).
- Capuana, F., et al. Imaging of dysfunctional elastogenesis in atherosclerosis using an improved gadolinium-based tetrameric MRI probe targeted to tropoelastin. Journal of Medicinal Chemistry. 64 (20), 15250-15261 (2021).
- Drescher, D. G., Drescher, M. J., Ramakrishnan, N. A. Surface plasmon resonance (SPR) analysis of binding interactions of proteins in inner-ear sensory epithelia. Methods in Molecular Biology. 493, 323-343 (2009).
- Murali, S., Rustandi, R. R., Zheng, X., Payne, A., Shang, L. Applications of surface plasmon resonance and biolayer interferometry for virus-ligand binding. Viruses. 14 (4), 717 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados