Uso de imágenes de células vivas de STED para visualizar la ultraestructura de la membrana interna mitocondrial en modelos de células neuronales
En este artículo
Resumen
Este protocolo presenta un flujo de trabajo para la propagación, diferenciación y tinción de células SH-SY5Y cultivadas y neuronas primarias del hipocampo de rata para la visualización y el análisis de la ultraestructura mitocondrial mediante microscopía de agotamiento de emisiones estimuladas (STED).
Resumen
Las mitocondrias desempeñan muchas funciones esenciales en la célula, incluida la producción de energía, la regulación de la homeostasis de Ca2+ , la biosíntesis de lípidos y la producción de especies reactivas de oxígeno (ROS). Estos procesos mediados por mitocondrias asumen funciones especializadas en las neuronas, coordinando el metabolismo aeróbico para satisfacer las altas demandas de energía de estas células, modulando la señalización de Ca2+ , proporcionando lípidos para el crecimiento y la regeneración de axones y ajustando la producción de ROS para el desarrollo y la función neuronal. Por lo tanto, la disfunción mitocondrial es un factor central en las enfermedades neurodegenerativas. La estructura y la función mitocondrial están inextricablemente vinculadas. La membrana interna morfológicamente compleja con pliegues estructurales llamados crestas alberga muchos sistemas moleculares que realizan los procesos característicos de la mitocondria. Las características arquitectónicas de la membrana interna son ultraestructurales y, por lo tanto, demasiado pequeñas para ser visualizadas por la microscopía tradicional resuelta por difracción limitada. Por lo tanto, la mayoría de los conocimientos sobre la ultraestructura mitocondrial provienen de la microscopía electrónica en muestras fijas. Sin embargo, las tecnologías emergentes en microscopía de fluorescencia de superresolución ahora proporcionan una resolución de hasta decenas de nanómetros, lo que permite la visualización de características ultraestructurales en células vivas. Por lo tanto, las imágenes de superresolución ofrecen una capacidad sin precedentes para obtener imágenes directas de detalles finos de la estructura mitocondrial, las distribuciones de proteínas a nanoescala y la dinámica de las crestas, lo que proporciona nuevos conocimientos fundamentales que vinculan las mitocondrias con la salud y la enfermedad humanas. Este protocolo presenta el uso de microscopía de superresolución de agotamiento de emisiones estimuladas (STED) para visualizar la ultraestructura mitocondrial de células vivas de neuroblastoma humano y neuronas primarias de rata. Este procedimiento está organizado en cinco secciones: (1) crecimiento y diferenciación de la línea celular SH-SY5Y, (2) aislamiento, siembra y crecimiento de neuronas primarias del hipocampo de rata, (3) procedimientos para teñir células para obtener imágenes de STED vivas, (4) procedimientos para experimentos de STED de células vivas utilizando un microscopio de STED como referencia, y (5) orientación para la segmentación y el procesamiento de imágenes utilizando ejemplos para medir y cuantificar las características morfológicas de la membrana interna.
Introducción
Las mitocondrias son orgánulos eucariotas de origen endosimbiótico que se encargan de regular varios procesos celulares clave, como el metabolismo intermediario y la producción de ATP, la homeostasis iónica, la biosíntesis lipídica y la muerte celular programada (apoptosis). Estos orgánulos son topológicamente complejos, conteniendo un sistema de doble membrana que establece múltiples subcompartimentos1 (Figura 1A). La membrana mitocondrial externa (OMM) interactúa con el citosol y establece contactos directos entre orgánulos 2,3. La membrana mitocondrial interna (IMM) es una membrana que conserva la energía y mantiene los gradientes iónicos almacenados principalmente como un potencial de membrana eléctrica (ΔΨm) para impulsar la síntesis de ATP y otros procesos que requieren energía 4,5. El IMM se subdivide a su vez en la membrana límite interna (IBM), que está estrechamente adherida al OMM, y estructuras sobresalientes llamadas crestas que están unidas por la membrana de crestas (CM). Esta membrana delinea el compartimento más interno de la matriz del espacio intracristalino (ICS) y el espacio intermembrana (IMS).
Las mitocondrias tienen una morfología dinámica basada en procesos continuos y equilibrados de fisión y fusión que están gobernados por mecanoenzimas de la superfamilia de las dinaminas6. La fusión permite aumentar la conectividad y la formación de redes reticulares, mientras que la fisión conduce a la fragmentación mitocondrial y permite la eliminación de las mitocondrias dañadas por la mitofagia7. La morfología mitocondrial varía según el tipo de tejido8 y la etapa de desarrollo9 y está regulada para permitir que las células se adapten a factores que incluyen las necesidades energéticas 10,11 y los factores estresantes 12. Las características morfométricas estándar de las mitocondrias, como la extensión de la formación de la red (interconectada vs. fragmentada), el perímetro, el área, el volumen, la longitud (relación de aspecto), la redondez y el grado de ramificación, pueden medirse y cuantificarse mediante microscopía óptica estándar porque los tamaños de estas características son mayores que el límite de difracción de la luz (~200 nm)13.
La arquitectura de las crestas define la estructura interna de las mitocondrias (Figura 1B). La diversidad de morfologías de las crestas se puede clasificar ampliamente como planas (laminares o discoidales) o tubulares-vesiculares14. Todas las crestas se unen en el IBM a través de estructuras tubulares o en forma de ranura denominadas uniones de crestas (CJ) que pueden servir para compartimentar el IMS del ICS y el IBM del CM15. La morfología de las crestas está regulada por complejos proteicos clave de la IMM, que incluyen (1) el sitio de contacto mitocondrial y el sistema organizador de crestas (MICOS) que reside en los CJ y estabiliza los contactos IMM-OMM 16, (2) la GTPasa de atrofia óptica 1 (OPA1) que regula la remodelación de las crestas17,18,19, y (3) la F1FO ATP sintasa que forma ensamblajes oligoméricos estabilizadores en las puntas de las crestas (CT)20, Artículo 21. Además, el IMM está enriquecido en fosfolípidos no bicapa fosfatidiletanolamina y cardiolipina que estabilizan el IMM22 altamente curvado. Las crestas también son dinámicas, mostrando cambios morfológicos bajo diversas condiciones, como diferentes estados metabólicos 23,24, con diferentes sustratos respiratorios 25, bajo inanición y estrés oxidativo 26,27, con apoptosis 28,29 y con envejecimiento 30. Recientemente se ha demostrado que las crestas podrían sufrir grandes eventos de remodelación en una escala de tiempo de segundos, lo que subraya su naturaleza dinámica31. Se pueden cuantificar varias características de las crestas, incluidas las dimensiones de las estructuras dentro de las crestas individuales (p. ej., el ancho de CJ, la longitud y el ancho de las crestas) y los parámetros que relacionan las crestas individuales con otras estructuras (p. ej., el espaciamiento intra-crestas y el ángulo de incidencia de las crestas en relación con el OMM)32. Estos parámetros de crestas cuantificables muestran una correlación directa con la función. Por ejemplo, el grado de producción de ATP mitocondrial se relaciona positivamente con la abundancia de crestas, cuantificada como densidad de crestas o número de crestas normalizado a otra característica (p. ej., crestas por área OMM)33,34,35. Debido a que la morfología de IMM está definida por características a nanoescala, comprende la ultraestructura mitocondrial, lo que requiere técnicas de imagen que proporcionen una resolución mayor que el límite de difracción de luz. Como se describe a continuación, estas técnicas incluyen la microscopía electrónica y la microscopía de superresolución (nanoscopía).
Las células neurales y gliales del sistema nervioso central (SNC) dependen particularmente de la función mitocondrial. En promedio, el cerebro constituye solo el 2% del peso corporal total, pero utiliza el 25% de la glucosa corporal total y representa el 20% del consumo de oxígeno corporal, lo que lo hace vulnerable a alteraciones en el metabolismo energético36. Las enfermedades neurodegenerativas progresivas (ND), como la enfermedad de Alzheimer (EA), la esclerosis lateral amiotrófica (ELA), la enfermedad de Huntington (EH), la esclerosis múltiple (EM) y la enfermedad de Parkinson (EP), son algunas de las patologías más estudiadas hasta la fecha, con esfuerzos de investigación que van desde la comprensión de los fundamentos moleculares de estas enfermedades hasta la búsqueda de posibles intervenciones y prevención terapéuticas. Los ND se asocian con un aumento del estrés oxidativo originado en parte por las especies reactivas de oxígeno (ROS) generadas por la cadena de transporte de electrones mitocondrial (ETC)37, así como con la alteración del manejo del calcio mitocondrial 38 y del metabolismo de los lípidos mitocondriales39. Estas alteraciones fisiológicas se acompañan de defectos observados en la morfología mitocondrial que se asocian con EA 40,41,42,43,44, ELA45,46, EH47,48,49, EM50 y EP 51,52,53. Estos defectos estructurales y funcionales pueden ser acoplados por complejas relaciones causa-efecto. Por ejemplo, dado que la morfología de las crestas estabiliza las enzimas OXPHOS54, las ROS mitocondriales no solo son generadas por el ETC, sino que también actúan para dañar la infraestructura en la que reside el ETC, promoviendo un ciclo de ROS de retroalimentación que mejora la susceptibilidad al daño oxidativo. Además, se ha demostrado que la desorganización de las crestas desencadena procesos como la liberación de ADN mitocondrial (ADNmt) y las vías inflamatorias relacionadas con trastornos autoinmunes, metabólicos y relacionados con la edad55. Por lo tanto, el análisis de la estructura mitocondrial es clave para una comprensión completa de los ND y sus fundamentos moleculares.
Los métodos populares de visualización de crestas, como la microscopía electrónica de transmisión, la tomografía electrónica y la tomografía crioelectrónica (crio-ET), y la tomografía de rayos X, en particular la tomografía crioblanda de rayos X, han revelado importantes hallazgos y trabajan con una variedad de tipos de muestras 56,57,58,59,60 . A pesar de los avances recientes hacia una mejor observación de la ultraestructura de los orgánulos, estos métodos todavía vienen con la advertencia de requerir la fijación de la muestra y, por lo tanto, no pueden capturar directamente la dinámica en tiempo real de las crestas. La microscopía de fluorescencia de superresolución, particularmente en las formas de microscopía de iluminación estructurada (SIM), microscopía de reconstrucción óptica estocástica (STORM), microscopía de localización fotoactivada (PALM), microscopía de expansión (ExM) y microscopía de agotamiento de emisión estimulada (STED), se han convertido en formas populares de ver estructuras que requieren una resolución por debajo del límite de difracción que restringe los métodos clásicos de microscopía óptica. Cuando se utiliza ExM junto con otra técnica de superresolución, los resultados son impresionantes, pero la muestra debe fijarse y teñirse en un gel61. En comparación, SIM, PALM/STORM y STED se han utilizado con éxito con muestras vivas, y los colorantes nuevos y prometedores que generalmente tiñen el IMM proporcionan un enfoque novedoso y fácil para obtener imágenes en vivo de la dinámica de las mitocondrias crestas 62,63,64,65,66. Los avances recientes en los tintes vivos para la obtención de imágenes STED han mejorado el brillo y la fotoestabilidad del tinte, y estos tintes se dirigen al IMM con un mayor grado de especificidad que sus predecesores. Estos desarrollos permiten la recopilación de experimentos de lapso de tiempo y pila z a largo plazo con imágenes de superresolución, lo que abre la puerta a un mejor análisis de células vivas de la ultraestructura y la dinámica mitocondrial.
En este artículo, se proporcionan protocolos para la obtención de imágenes de células vivas de células SH-SY5Y indiferenciadas y diferenciadas teñidas con el colorante PKmito Orange (PKMO) utilizando STED63. La línea celular SH-SY5Y es un derivado tres veces subclonado de la línea celular parental, SK-N-SH, generada a partir de una biopsia de médula ósea de neuroblastoma metastásico67,68,69,70. Esta línea celular es un modelo in vitro comúnmente utilizado en la investigación de la EN, particularmente con enfermedades como la EA, la EH y la EP, en las que la disfunción mitocondrial está fuertemente implicada 10,43,71,72,73. La capacidad de diferenciar las células SH-SY5Y en células con un fenotipo similar al de las neuronas mediante la manipulación de medios de cultivo ha demostrado ser un modelo adecuado para la investigación en neurociencia sin depender de células neuronales primarias10,74. En este protocolo, se añadió ácido retinoico (AR) al medio de cultivo celular para inducir la diferenciación de las células SH-SY5Y. La AR es un derivado de la vitamina A y se ha demostrado que regula el ciclo celular y promueve la expresión de factores de transcripción que regulan la diferenciación neuronal75. También se proporciona un protocolo para el cultivo y la obtención de imágenes de células vivas de neuronas aisladas del hipocampo de la rata. Se ha demostrado que el hipocampo se ve afectado por la degeneración mitocondrial y, junto con la corteza, juega un papel importante en el envejecimiento y la ND 76,77,78,79,80.
Protocolo
1. Propagación y diferenciación de células SH-SY5Y
- Preparación de medios para el crecimiento y mantenimiento celular
- Prepare el medio de águila modificado de Dulbecco completo y con alto contenido de glucosa (DMEM, 4,5 g/L de D-glucosa, 4 mM de L-glutamina, 110 mg/L de piruvato de sodio) suplementado con un antibiótico-antimicótico final al 1% (v/v) (10.000 unidades/ml de penicilina, 10.000 μg/ml de estreptomicina y 25 μg/ml de anfotericina B) y cantidades variables de suero fetal bovino (FBS) (véase la Tabla de materiales). Las cantidades de FBS en los medios de diferenciación varían entre el 10%, 5% o 2% (v/v) final de FBS.
- Mantenimiento de la célula
- Mantener las células en DMEM suplementadas con 10% (v/v) de FBS y colocarlas a 37 °C y 5% de CO2, luego sembrar en DMEM que contenga 5% (v/v) de FBS para la diferenciación. Las existencias de células congeladas se almacenaron en FBS con 10% (v/v) de dimetilsulfóxido (DMSO) a 1-2 x 107 células/mL.
- Preparación de ácido retinoico (AR)
- Disolver 7,51 mg de all-trans-RA (ver Tabla de Materiales) en 5 mL de etanol al 95% recién preparado para obtener 5 mM de solución madre. Verificar la concentración con absorbancia a 350 nm (ɛ = 44.300 M-1 cm-1), obtenida de la ficha de información del producto del protocolo81 del fabricante, utilizando una dilución de la solución madre a 5 μM en etanol. Almacene 5 mM de existencias protegidas de la luz a 4 °C durante un máximo de 6 semanas.
- Preparación de poli-D-lisina para recubrimiento de cubreobjetos
NOTA: El protocolo de producto de poli-D-lisina (PDL), que se encuentra en la sección Documentos y descargas del sitio del proveedor82, proporciona información para recubrir una variedad de recipientes de cultivo.- Este protocolo incluye volúmenes basados en un recipiente de 2 pocillos con cámara con un área de 4cm2 por pocillo con fondos estériles de vidrio de borosilicato #1.5 (ver Tabla de Materiales). Diluir la solución madre de PDL dos veces a 50 μg/ml con PBS de Dulbecco (DPBS; sin calcio, sin magnesio).
NOTA: El cubreobjetos #1.5 o #1.5H son espesores aceptables, que son esenciales para la calidad de la imagen. Otros espesores inducirán aberración esférica y deben evitarse.
- Este protocolo incluye volúmenes basados en un recipiente de 2 pocillos con cámara con un área de 4cm2 por pocillo con fondos estériles de vidrio de borosilicato #1.5 (ver Tabla de Materiales). Diluir la solución madre de PDL dos veces a 50 μg/ml con PBS de Dulbecco (DPBS; sin calcio, sin magnesio).
- Recubrimiento de cubreobjetos con PDL
NOTA: Los cubreobjetos pueden exponerse a la luz ultravioleta (UV) durante 10-15 minutos en una cabina de bioseguridad para su posterior esterilización.- Aplique 1,2 ml de solución de PDL de 50 μg/ml a cada pocillo de cubreobjetos estériles en un armario de cultivo celular e incube a temperatura ambiente durante 1 h. Retire la solución de PDL y enjuague tres veces con 3,6 ml de agua destilada. Después de completar el lavado final, deje que la cámara recubierta se seque durante 2 h expuesta al aire antes de enjuagarla y usarla inmediatamente o almacenarla con un recipiente hermético a 4 °C durante un máximo de 2 semanas.
NOTA: Enjuague bien los cubreobjetos, ya que el exceso de PDL puede ser tóxico para las células.
- Aplique 1,2 ml de solución de PDL de 50 μg/ml a cada pocillo de cubreobjetos estériles en un armario de cultivo celular e incube a temperatura ambiente durante 1 h. Retire la solución de PDL y enjuague tres veces con 3,6 ml de agua destilada. Después de completar el lavado final, deje que la cámara recubierta se seque durante 2 h expuesta al aire antes de enjuagarla y usarla inmediatamente o almacenarla con un recipiente hermético a 4 °C durante un máximo de 2 semanas.
- Diferenciación de células SH-SY5Y con AR
NOTA: No utilice celdas por encima del pasaje 15. Las células se desplazan al 80%-90% de confluencia. Los procedimientos de diferenciación difieren, pero siguen pasos similares. La diferenciación adicional de los neuroblastomas a las neuronas maduras se obtiene con el tratamiento adicional con el factor neurotrófico derivado del cerebro (BDNF)68,83,84,85, pero no se realizó en este protocolo.
OPCIONAL: Establezca las celdas durante al menos 24 h antes de la siembra en el cubreobjetos. Para preparar células a partir de existencias congeladas, descongele rápidamente 1 ml de vial congelado de células y agréguelo a 9 ml de medios precalentados suplementados con FBS al 10%, luego centrifugue a 350 x g durante 10 minutos (a temperatura ambiente) y deseche el sobrenadante para eliminar el DMSO. Vuelva a suspender el gránulo de células en 5 ml de medio precalentado y las células semilla en un matraz T-25. Una vez que las células alcancen el 80%-90% de confluencia, pase las células contándolas y sembrándolas para su diferenciación cuando corresponda.- Día 0: Células semilla.
- Siembre las células en un cubreobjetos con cámara a partir de caldos congelados o en un matraz de trabajo. Utilice una densidad de siembra de 1,5 x 104 celdas/cm2.
NOTA: Un solo pocillo en un cubreobjetos estándar de 2 pocillos con 4cm2 de área de cultivo requerirá 6.0 x 104 celdas. Las células que permanecerán indiferenciadas deben sembrarse con DMEM suplementado con un 10% (v/v) de FBS, y las células que se diferenciarán deben sembrarse con DMEM suplementado con un 5% (v/v) de FBS.
- Siembre las células en un cubreobjetos con cámara a partir de caldos congelados o en un matraz de trabajo. Utilice una densidad de siembra de 1,5 x 104 celdas/cm2.
- Día 1: Iniciar el tratamiento de diferenciación de la AR.
- Preparar DMEM suplementado con 5% (v/v) de FBS, 1% (v/v) antibiótico-antimicótico y una concentración final de 10 μM de RA o etanol del mismo volumen de aditivo para que sirva como control del vehículo para este procedimiento de diferenciación. Retire el medio en el cubreobjetos con cámara utilizado para la siembra, enjuague con 1x PBS y agregue el nuevo DMEM a los pocillos.
- Día 3: Reemplace los medios con medios nuevos que contengan AR o etanol.
- Retire el medio del día 1 y reemplácelo con medio nuevo suplementado con FBS al 2 % (v/v), antibiótico antimicótico al 1 % (v/v) y 10 μM de RA o etanol al 95 % del mismo volumen de aditivo para que sirva como control del vehículo para este procedimiento de diferenciación. Retire el medio en el cubreobjetos con cámara utilizado para la siembra, enjuague con 1x PBS y agregue nuevos medios a los pocillos.
- Día 6: Realizar la toma de imágenes de las células.
NOTA: Los tiempos de diferenciación celular varían según el protocolo, pero seis días de exposición a la AR son suficientes para inducir un fenotipo similar al de una neurona en las células SH-SY5Y86.- Realice imágenes en vivo, con los detalles de las Secciones 3 y 4 (Figura 2).
- Día 0: Células semilla.
2. Cultivo primario de neuronas del hipocampo de rata
- Preparación de materiales para el aislamiento de neuronas del hipocampo de rata.
- Prepare DMEM recién suplementado.
- Filtrar y esterilizar una mezcla de DMEM (glucosa alta, sin piruvato de sodio) suplementada con FBS inactivado por calor al 10 % (v/v), solución de piruvato de sodio al 1 % (v/v) y penicilina-estreptomicina al 1 % (v/v) (10,000 U/mL). Conservar hasta 2 semanas a 4 °C.
- Prepare medios de crecimiento neuronal recién suplementados.
- Filtrar y esterilizar una mezcla de medios de crecimiento neuronal suplementados con un suplemento de B27 al 2% (v/v), un suplemento de glutamina al 0,25% (v/v) y un suplemento de penicilina-estreptomicina al 1% (v/v) (ver Tabla de materiales). Conservar hasta 2 semanas a 4 °C.
- Prepare DMEM recién suplementado.
- Preparar el cultivo primario de neuronas del hipocampo.
- Preparar el cultivo primario de neuronas del hipocampo siguiendo el trabajo publicado anteriormente87 y del protocolo del producto en el sitio del fabricante del que se obtiene el hipocampo de rata E18 disecado88 (ver Tabla de Materiales). Este protocolo da como resultado una población mayoritariamente neuronal con un <2% de astrocitos.
NOTA: El medio de hibernación con el que se envía este tejido se utilizará para los pasos futuros de este protocolo. No lo deseches. - Preparar materiales y medios para la disociación de tejidos.
- Flamee una pipeta Pasteur para disminuir el diámetro de apertura y guárdela en papel de aluminio para evitar la contaminación hasta su uso. Precaliente el DMEM preparado, la solución salina tamponada de Hank (HBSS) 1X y el medio de crecimiento de neuronas a 37 °C. Agregue 2 escamas de ADNasa con pinzas estériles a un tubo cónico de 15 ml.
- Realizar la disociación de tejidos.
- Extraer la mayor cantidad posible de medios de hibernación en los que se almacena el hipocampo de rata E18 diseccionado antes de colocar el tejido en el tubo cónico de 15 ml que contiene ADNasa e incubar brevemente a 37 °C. Añadir 900 μL de HBSS 1X seguido de 100 μL de tripsina al 0,5%. Incubar el tejido a 37 °C durante 15 min.
NOTA: Las placas recubiertas de PDL se pueden retirar del almacenamiento y colocarse en una incubadora hasta que se usen durante este tiempo de incubación.
- Extraer la mayor cantidad posible de medios de hibernación en los que se almacena el hipocampo de rata E18 diseccionado antes de colocar el tejido en el tubo cónico de 15 ml que contiene ADNasa e incubar brevemente a 37 °C. Añadir 900 μL de HBSS 1X seguido de 100 μL de tripsina al 0,5%. Incubar el tejido a 37 °C durante 15 min.
- Realizar la homogeneización de tejidos y el recuento celular.
- Después de la incubación con tripsina, retirar el medio y añadir 1 mL de medio de hibernación precalentado-ADNasa del paso anterior al tejido y homogeneizar con la pipeta Pasteur. Los medios se verán opacos y luego se aclararán gradualmente a medida que continúe la homogeneización.
- Agregue neuronas disociadas a un nuevo tubo con DMEM precalentado de 4 ml y luego cuente las células usando un contador de células.
- Realizar la siembra celular y el crecimiento de las células primarias.
- Celdas en placa a una densidad de aproximadamente 1,65 x 104 células/cm2 en DMEM. Para un cubreobjetos con cámara de 2 pocillos (4 cm2), siembre de 65.000 a 70.000 células por pocillo con 2 ml de DMEM. Incubar las células a 37 °C y 5% de CO 2 durante2 h antes de comprobar la adherencia. Una vez que las células comiencen a adherirse, retire 1 ml de medio y reemplácelo con el mismo volumen de medio de hibernación, luego agite suavemente para mezclar. Una vez que se mezclen los medios, repita este proceso y retire la mitad de los medios presentes y reemplácelos con el mismo volumen de medios de crecimiento de neuronas, luego mezcle suavemente.
NOTA: El día de la siembra se considera día in vitro (DIV) 0, y las células están listas para obtener imágenes en DIV 7 (Figura 2).
- Celdas en placa a una densidad de aproximadamente 1,65 x 104 células/cm2 en DMEM. Para un cubreobjetos con cámara de 2 pocillos (4 cm2), siembre de 65.000 a 70.000 células por pocillo con 2 ml de DMEM. Incubar las células a 37 °C y 5% de CO 2 durante2 h antes de comprobar la adherencia. Una vez que las células comiencen a adherirse, retire 1 ml de medio y reemplácelo con el mismo volumen de medio de hibernación, luego agite suavemente para mezclar. Una vez que se mezclen los medios, repita este proceso y retire la mitad de los medios presentes y reemplácelos con el mismo volumen de medios de crecimiento de neuronas, luego mezcle suavemente.
- Preparar el cultivo primario de neuronas del hipocampo siguiendo el trabajo publicado anteriormente87 y del protocolo del producto en el sitio del fabricante del que se obtiene el hipocampo de rata E18 disecado88 (ver Tabla de Materiales). Este protocolo da como resultado una población mayoritariamente neuronal con un <2% de astrocitos.
3. Preparación de células para la obtención de imágenes de células vivas
NOTA: Los tipos de células y el origen (es decir, células cultivadas y primarias) pueden diferir en los requisitos de tinción; Para más detalles, véanse los informes publicados62,63.
- Preparación de PKmito Orange
NOTA: Otros tintes que generalmente tiñen el IMM han sido reportados64,65,66 y están disponibles comercialmente. PKMO es el único que se utiliza en este protocolo.- Vuelva a suspender el polvo PKMO (consulte la tabla de materiales) en DMSO según las instrucciones del fabricante89. Aspire el medio de las células y lávelo en un medio precalentado sin rojo de fenol. Preparar un caldo de PKMO en DMEM precalentado y sin rojo de fenol suplementado con FBS al 2% (v/v) o al 10% (v/v) según el estado de diferenciación, HEPES a una concentración final de 20 mM y antibiótico-antimicótico al 1% (v/v) antes de teñir las células siguiendo las instrucciones del fabricante. Esta formulación, sin PKMO, es el medio de obtención de imágenes de células vivas.
- Tinción celular con PKMO
- Incubar las células con el colorante a 37 °C y 5% de CO2, durante 30 min. Lavar las células tres veces con medios de imagen de células vivas y, para el lavado final, incubar durante 30 min a 37 °C, 5% de CO2.
- Agregue medios de adquisición de imágenes de células vivas nuevos y precalentados. Las células ya están listas para la obtención de imágenes.
NOTA: Los tratamientos agudos (p. ej., medicamentos y/o factores estresantes), cuando se usan, se agregan antes de las imágenes en vivo; consulte la sección de Discusión y la Figura complementaria 1.
4. Obtención de imágenes de células vivas mediante microscopía STED
NOTA: Este protocolo utiliza un sistema STED construido alrededor de un microscopio invertido, con el sistema especificado en la Tabla de Materiales. Este sistema está equipado con láseres de excitación pulsada (láser de 561 nm con potencia nominal ~300 μW) y un láser de agotamiento STED pulsado de 775 nm (potencia nominal de 1,2 W), un escáner galvano continuamente ajustable y un detector de fotodiodos de avalancha (APD) basado en filtro de 615/20 nm. Aquí se utiliza una lente de inmersión en aceite de 100x/1,40 para STED. El software Lightbox se utiliza para la adquisición de imágenes. Todos los detalles proporcionados están relacionados directamente con este software y la configuración del sistema.
- Pautas generales y pasos para la obtención de imágenes
- Utilice una incubadora o una cámara ambiental para mantener la viabilidad celular, pero también son aceptables los experimentos a temperatura ambiente a corto plazo. Estos pasos son específicos de la configuración de STED descrita anteriormente.
- Seleccione los conjuntos de láser y filtros para la obtención de imágenes.
- Utilice los parámetros de un colorante naranja seleccionando los tintes utilizados en la tinción de la Lista de colorantes o el que tenga propiedades espectrales más cercanas a los tintes utilizados. Activa estos haciendo doble clic o arrastrándolos a la lista de muestras, donde dice "Arrastra los tintes aquí".
- Seleccione una región para crear una imagen.
- En una descripción general, cree una región de interés (ROI) alrededor de una mitocondria de interés seleccionando el botón ROI rectangular y haciendo clic y arrastrando para dar forma a la región. El ROI se puede cambiar de tamaño y rotar utilizando las esquinas ROI o los bordes curvos que aparecen al pasar el mouse sobre una esquina.
NOTA: En la Tabla 1 se puede encontrar un resumen de los parámetros de imagen sugeridos. Estos parámetros se ajustaron empíricamente utilizando los reportados previamente para esta configuración de STED y combinación de colorantes63.
- En una descripción general, cree una región de interés (ROI) alrededor de una mitocondria de interés seleccionando el botón ROI rectangular y haciendo clic y arrastrando para dar forma a la región. El ROI se puede cambiar de tamaño y rotar utilizando las esquinas ROI o los bordes curvos que aparecen al pasar el mouse sobre una esquina.
- Configurar la compuerta.
- Junto al menú General , seleccione el menú Puerta o haga clic y mantenga presionado para agregar el menú a la vista. Se recomienda que la compuerta del detector STED se ajuste a 1-1,05 a 7,8-7,85 ns, como se presenta aquí. Los tiempos de compuerta pueden variar y ser tan cortos como 1-1.05 a 7-7.05 ns.
- Ajuste la intensidad de acuerdo con la muestra.
NOTA: Generalmente, la potencia de excitación utilizada para STED es de 2 a 3 veces la potencia utilizada para el confocal, por lo que una muestra que requiera una potencia de láser de excitación del 5 % para confocal puede usar una potencia del láser de excitación del 10 % al 15 % durante la adquisición de STED.- Ajuste el láser de excitación para STED a 15%-20% y el láser de agotamiento STED a 20%-25% con 10 acumulaciones de línea. Utilice un tiempo de permanencia de píxeles de 4 μs con un tamaño de píxel de 20-25 nm. El agujero de alfiler se mantuvo a 1,0 UA para las células cultivadas y 0,7 UA para las células primarias del hipocampo de rata con el fin de mejorar el corte óptico en las mitocondrias más densamente empaquetadas.
NOTA: Las imágenes confocales y STED se pueden adquirir para la comparación lado a lado (Figura 3A, B, Figura 4A) o solo se pueden adquirir STED.
- Ajuste el láser de excitación para STED a 15%-20% y el láser de agotamiento STED a 20%-25% con 10 acumulaciones de línea. Utilice un tiempo de permanencia de píxeles de 4 μs con un tamaño de píxel de 20-25 nm. El agujero de alfiler se mantuvo a 1,0 UA para las células cultivadas y 0,7 UA para las células primarias del hipocampo de rata con el fin de mejorar el corte óptico en las mitocondrias más densamente empaquetadas.
- Información adicional para series temporales/series z
- Seleccione la serie temporal.
- Seleccione el menú desplegable Hora. Defina el número de iteraciones (5 utilizadas aquí) y el intervalo de tiempo (25 o 30 s utilizados aquí) deseado para un timelapse (Figura 3C, D, Figura 4B).
NOTA: Si el intervalo es más corto que el tiempo de adquisición, las iteraciones continuarán sin demora. Al realizar un timelapse, se recomienda encarecidamente utilizar una unidad de enfoque perfecta o un seguimiento de enfoque similar para evitar posibles desviaciones.
- Seleccione el menú desplegable Hora. Defina el número de iteraciones (5 utilizadas aquí) y el intervalo de tiempo (25 o 30 s utilizados aquí) deseado para un timelapse (Figura 3C, D, Figura 4B).
- Establezca el rango de volumen.
- Establezca el rango de volumen z como desee habilitando la opción Volume y ajustando los dos extremos del rango. Los tamaños de paso utilizados en este protocolo para la obtención de imágenes 2D fueron de 150 a 200 nm. El tamaño de paso recomendado con respecto al muestreo de Nyquist requerido para la deconvolución de STED en bruto se puede calcular con herramientas en línea90.
NOTA: La potencia del láser de agotamiento de STED y el número de planos fotografiados pueden aumentar el fotoblanqueo del tinte y la exposición a la luz de la célula a niveles dañinos. Compruebe si hay signos de fototoxicidad y fotodaño después de la adquisición.
- Establezca el rango de volumen z como desee habilitando la opción Volume y ajustando los dos extremos del rango. Los tamaños de paso utilizados en este protocolo para la obtención de imágenes 2D fueron de 150 a 200 nm. El tamaño de paso recomendado con respecto al muestreo de Nyquist requerido para la deconvolución de STED en bruto se puede calcular con herramientas en línea90.
- Seleccione la serie temporal.
5. Herramientas de procesamiento y analíticas para la ultraestructura mitocondrial
NOTA: El procesamiento de imágenes (es decir, la deconvolución) es opcional, pero normalmente se utiliza cuando se crean y analizan imágenes STED para su publicación. Se recomienda encarecidamente la deconvolución para mejorar el contraste y reducir el ruido para una segmentación óptima de las crestas individuales, como se describe a continuación (Figura 2).
- Deconvolución de imágenes STED
NOTA: El software utilizado para la deconvolución en este protocolo se proporciona en la Tabla de Materiales.- Establezca los parámetros microscópicos de la imagen.
NOTA: Asegúrese de que los metadatos del microscopio se introduzcan correctamente en la imagen Parámetros microscópicos. Esto incluye el índice de refracción del medio de montaje; medio de inmersión; tamaño de píxel; longitudes de onda de excitación, emisión y agotamiento; y cualquier otra información relevante. Las plantillas con estos parámetros se pueden guardar para su reutilización. - Deconvolucia imágenes STED sin procesar con el algoritmo de software.
- El acceso a algoritmos de deconvolución automatizados en el software de deconvolución permite el procesamiento de imágenes de deconvolución sin intervención. Seleccione el botón Exprés y establezca el tipo de deconvolución en Rápido, Estándar, Agresivo o Conservador para distintos grados de potencia de deconvolución. Se muestran imágenes representativas que utilizan la deconvolución rápida con ajustes agresivos (Figura 3, Figura 4 y Figura 5).
- Guarde las imágenes del software de deconvolución en formato ICS2.
- Para la deconvolución manual, realice los siguientes pasos.
- En resumen, al realizar la deconvolución manual, guarde las plantillas del asistente de deconvolución para mantener la coherencia y tenga la opción de cargar una plantilla al iniciar el asistente. Utilice la información de la función de dispersión de puntos medidos (PSF) si se genera con la configuración y los parámetros de adquisición.
- Recorte la imagen STED sin procesar, si es necesario, antes de que el software de deconvolución estabilice automáticamente la imagen. Los complementos de los paquetes de software de deconvolución permiten una compensación específica de posibles artefactos de imagen, como la deriva térmica y las aberraciones cromáticas.
- A continuación, genere un histograma logarítmico para realizar la resta de fondo de forma manual o automática. Seleccione el algoritmo clásico de deconvolución de estimación de máxima verosimilitud (CMLE).
NOTA: Para la deconvolución, los valores clave que se deben ajustar son el umbral de relación señal-ruido, el número de iteraciones y el umbral de calidad. Estos valores se pueden ajustar y se puede obtener una vista previa de la deconvolución para determinar la configuración óptima.
- Establezca los parámetros microscópicos de la imagen.
- Segmentación y análisis de partículas
NOTA: Este protocolo utiliza FIJI (Is Just ImageJ), un software de código abierto (consulte Tabla de materiales), para segmentación y análisis. Para estos fines se dispone de otro software comparable, como CellProfiler, Icy, Ilastik y QuPath.- Prepare las imágenes para la segmentación.
- Abra las imágenes STED sin procesar .obf o los archivos .ics2 desde el software de deconvolución yendo a Archivo → Abrir o haciendo clic y arrastrando los archivos a la barra de herramientas de ImageJ. A partir de aquí, cualquier procesamiento que facilite la segmentación de las imágenes se puede realizar antes de la segmentación.
- Para mantener la coherencia de los cambios, grabe funciones utilizando Plugins → Macros → Grabar y copie y pegue los comandos de teclado en una nueva macro, desde Plugins → Nueva → Macro. Asegúrese de seleccionar la imagen que desea procesar antes de ejecutar la macro.
NOTA: Las alteraciones comúnmente aceptables para la cuantificación del tamaño y la forma incluyen el recorte, la proyección de una pila z y la sustracción de fondo con el suavizado desactivado. Las alteraciones deben realizarse de forma coherente entre las imágenes dentro de un conjunto de datos y notificarse.
- Ajustar la configuración de segmentación de Weka entrenable
NOTA: Se han publicado detalles adicionales con instrucciones paso a paso para el uso de la herramienta de segmentación semiautomatizada y análisis posteriores para las mitocondrias91.- Abra las imágenes STED desconvolucionadas en el complemento Trainable Weka Segmentation (TWS)92 , ubicado en Plugins → Segmentation → Trainable Weka Segmentation. En la Configuración de segmentación, seleccione las funciones de desenfoque gaussiano, proyecciones de membrana y filtro Sobel . El valor predeterminado del grosor de la membrana es 1 y el valor predeterminado del tamaño del parche de la membrana es 19.
- Etiquete la clase 1 o 2 como las "Crestas" y la otra como el "Fondo" (Figura 2). Los modelos también se pueden guardar con el botón Guardar clasificador . Seleccione el clasificador de carga para reutilizar esta configuración para otras imágenes.
- Realice seguimientos de clase TWS.
- Usa la herramienta de línea u otras formas para resaltar algunas de las crestas o el fondo. Al menos algunas selecciones de fondo deben incluir espacios entre las crestas. Dibuje una línea sobre la estructura para asignarla a cualquiera de las clases y, a continuación, seleccione el botón Añadir a en el lado derecho para las crestas o el fondo. Haga doble clic en un trazado para eliminar la estructura de esa etiqueta.
- Realizar el entrenamiento de clasificación TWS.
- Seleccione el botón Entrenar clasificador en el lado izquierdo para generar un mapa basado en la información proporcionada al complemento. La superposición de clases segmentadas se puede activar y desactivar con el botón Alternar superposición, y la opacidad de la superposición se puede ajustar en Configuración. El clasificador se puede volver a entrenar con etiquetas adicionales. Una vez satisfecho, seleccione el botón Obtener probabilidad.
- Mide las partículas.
- Usando el mapa de probabilidad de crestas, umbral de la imagen para generar una máscara y luego vaya a Analizar → Analizar partículas. Por lo general, se puede utilizar el tipo de umbral predeterminado y ajustar el rango para garantizar que se tengan en cuenta todas las crestas. Las mediciones proporcionadas por el análisis de partículas se pueden ajustar mediante Analizar → establecer medidas.
NOTA: Se miden ejemplos de parámetros de tamaño y forma, como el área, el perímetro, la circularidad y la relación de aspecto de las crestas, y se muestran en función de las mediciones seleccionadas (Figura 2, Figura 5A, Tabla complementaria 1). - OPCIONAL: Seleccione la imagen STED sin procesar con las mismas dimensiones que la imagen convolucionada y aplique las ROI desde el administrador, luego seleccione Medir en el administrador de ROI para obtener valores de intensidad.
- Usando el mapa de probabilidad de crestas, umbral de la imagen para generar una máscara y luego vaya a Analizar → Analizar partículas. Por lo general, se puede utilizar el tipo de umbral predeterminado y ajustar el rango para garantizar que se tengan en cuenta todas las crestas. Las mediciones proporcionadas por el análisis de partículas se pueden ajustar mediante Analizar → establecer medidas.
- Obtener diagramas de líneas.
- Los diagramas de líneas se generaron a partir de las imágenes STED desconvolucionadas. Dibuje una línea de varios puntos, ajuste el grosor de la línea a varios píxeles de ancho para promediar el ruido y estrelle la línea para que se ajuste a las mitocondrias (Figura 2, Figura 5B). El diagrama de líneas resultante generado se utiliza para medir las distancias de pico a pico para informar la periodicidad y la distribución de las crestas en un rango determinado.
NOTA: En relación con esto, la densidad de crestas se puede informar como el recuento de crestas independientes dentro de un área determinada según lo determinado midiendo la parte externa de las mitocondrias. El área mitocondrial se puede determinar aplicando un filtro a la imagen STED desconvolucionada o sin procesar para generar una máscara. Asegúrese de que la máscara se ajuste con precisión al contorno de las mitocondrias antes de medir el área.
- Los diagramas de líneas se generaron a partir de las imágenes STED desconvolucionadas. Dibuje una línea de varios puntos, ajuste el grosor de la línea a varios píxeles de ancho para promediar el ruido y estrelle la línea para que se ajuste a las mitocondrias (Figura 2, Figura 5B). El diagrama de líneas resultante generado se utiliza para medir las distancias de pico a pico para informar la periodicidad y la distribución de las crestas en un rango determinado.
- Prepare las imágenes para la segmentación.
Resultados Representativos
Este protocolo describe las condiciones de crecimiento celular para células cultivadas y primarias, con un enfoque en la obtención de imágenes de células vivas de ETS y análisis posteriores de las crestas mitocondriales. Las proyecciones realizadas con ImageJ de mitocondrias a partir de células SH-SY5Y indiferenciadas (Figura 3A) y SH-SY5Y diferenciadas por AR (Figura 3B) se pueden recolectar como pilas z con confocal y STED tradicionales, y las imágenes STED sin procesar se pueden desconvolucionar. También se pueden realizar imágenes de lapso de tiempo y posteriormente desconvolucionarlas (Figura 3C, D). Utilizando parámetros de imagen ligeramente diferentes para las neuronas primarias del hipocampo de la rata (Tabla 1), las imágenes confocales y sin procesar de STED se pueden adquirir como pilas z, y las imágenes de STED sin procesar se pueden desconvolucionar (Figura 4A). También es posible obtener imágenes de lapso de tiempo de las mitocondrias de las neuronas primarias (Figura 4B). En general, las imágenes de lapso de tiempo deben ser capaces de mostrar eventos dinámicos mitocondriales.
Cuando las proyecciones de pila z STED sin procesar y STED desconvolucionadas de las muestras utilizadas para la segmentación parecen consistentes, se realizan mediciones cuantitativas. El complemento TWS utiliza la imagen STED desconvolucionada para segmentar y hacer una máscara de probabilidad, que luego se usa para crear una máscara binaria de las crestas para obtener parámetros de tamaño y forma (Figura 5A). Las regiones de esta máscara se guardan en el administrador de ROI y se pueden aplicar a la imagen STED sin procesar si se desea para medir las diferencias en la intensidad relativa. Las proyecciones STED desconvolucionadas también se pueden utilizar para determinar la periodicidad y la densidad de las crestas en un área determinada (Figura 5B).
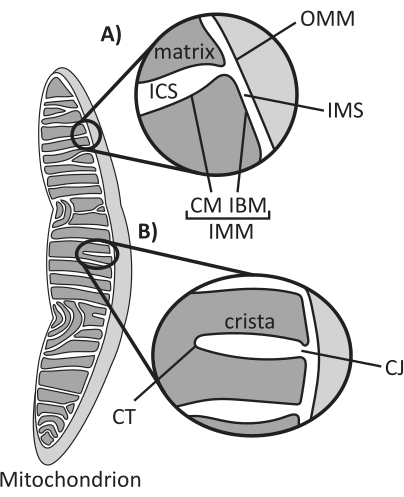
Figura 1: Morfología mitocondrial. Las mitocondrias tienen un sistema de dos membranas que define diferentes subcompartimentos (A). Las crestas son pliegues de la membrana interna con características definidas (B). Abreviaturas: OMM, membrana mitocondrial externa; ICS: espacio intracristalino; IMS: espacio intermembrana; CM: membrana crestista; IBM, membrana límite interna; IMM: membrana mitocondrial interna; TC: punta de cresta; CJ, cruce de crestas. Haga clic aquí para ver una versión más grande de esta figura.
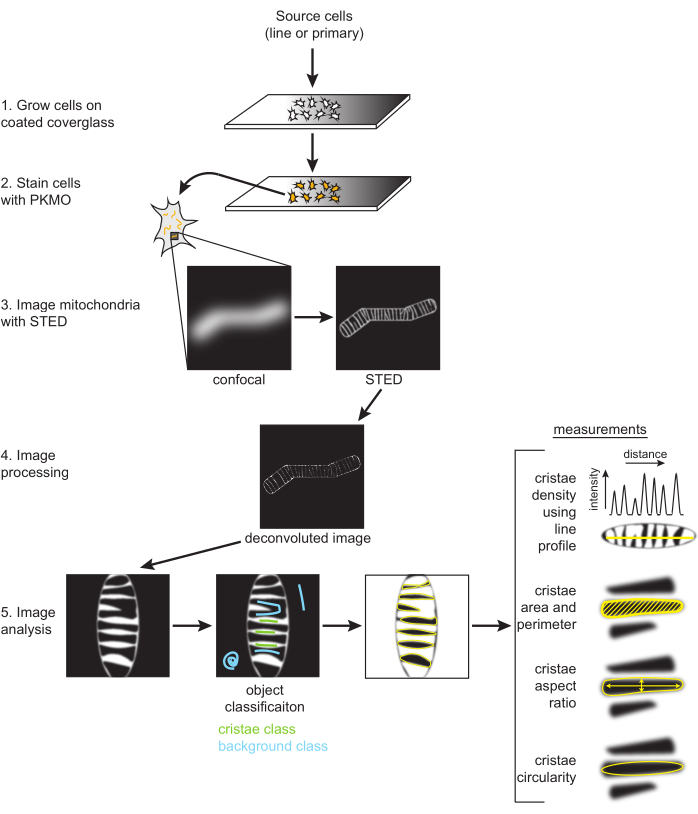
Figura 2: Esquema del flujo de trabajo. Las células SH-SY5Y o las neuronas primarias del hipocampo de rata se cultivan en un cubreobjetos recubierto de PDL. Las células SH-SY5Y se cultivan en paralelo para permanecer indiferenciadas o sometidas a diferenciación de AR en el transcurso de seis días. Las neuronas primarias del hipocampo de rata se cultivaron en un cubreobjetos recubierto de PDL después de ser aisladas de secciones del hipocampo durante siete días. Una vez que estaban listas para ser fotografiadas, las células se tiñeron con PKMO y se obtuvieron imágenes con STED. A continuación, las imágenes STED sin procesar se desconvolucionan y las imágenes desconvolucionadas se procesan en FIJI para obtener mediciones de tamaño y forma, como la densidad de crestas, el área, el perímetro, la circularidad y la relación de aspecto. Haga clic aquí para ver una versión más grande de esta figura.
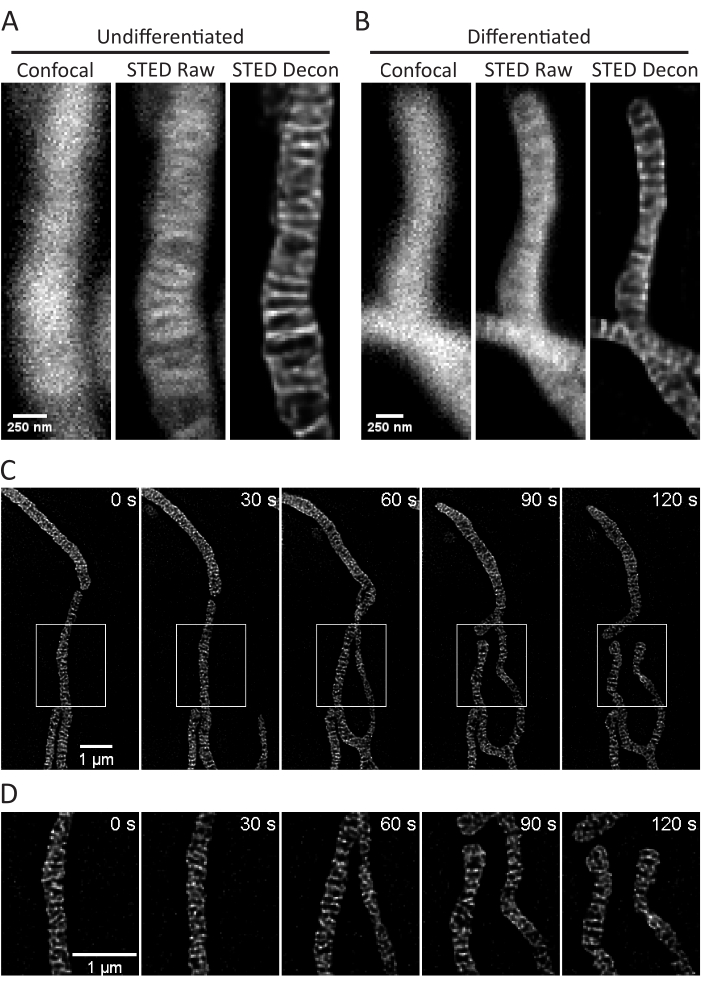
Figura 3: Imágenes de mitocondrias en células SH-SY5Y. Se muestran proyecciones representativas de la pila en Z de la imagen de STED confocal (izquierda), STED cruda (centro) y desconvolucionada de Huygens (derecha) de mitocondrias de células SH-SY5Y no diferenciadas (A) y diferenciadas por AR (B) con tinción de PKMO. Se muestra un timelapse con intervalos de 30 s y 5 iteraciones de celdas SH-SY5Y diferenciadas por AR (C) con regiones seleccionadas (cajas blancas) expandidas sobre (D) utilizando imágenes a escala de esas regiones sin interpolación. Barras de escala: A, B, 250 nm; C,D, 1 μm. Haga clic aquí para ver una versión más grande de esta figura.
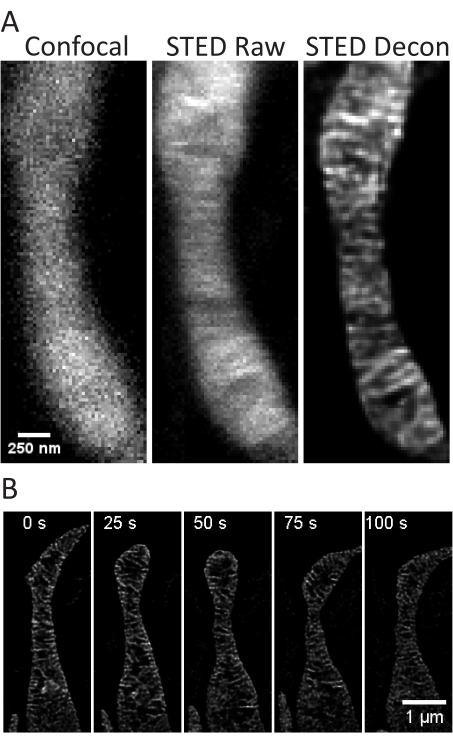
Figura 4: Imágenes de las mitocondrias en neuronas primarias del hipocampo de rata. Se muestran proyecciones representativas de la pila z de la imagen confocal (izquierda), STED cruda (centro) y STED desconvolucionada de Huygens (derecha) de mitocondrias de neuronas primarias del hipocampo de rata (A). Se muestra un timelapse con intervalos de 25 s y 5 iteraciones de mitocondrias en estas neuronas (B). Barras de escala: A, 250 nm; B, 1 μm. Haga clic aquí para ver una versión más grande de esta figura.
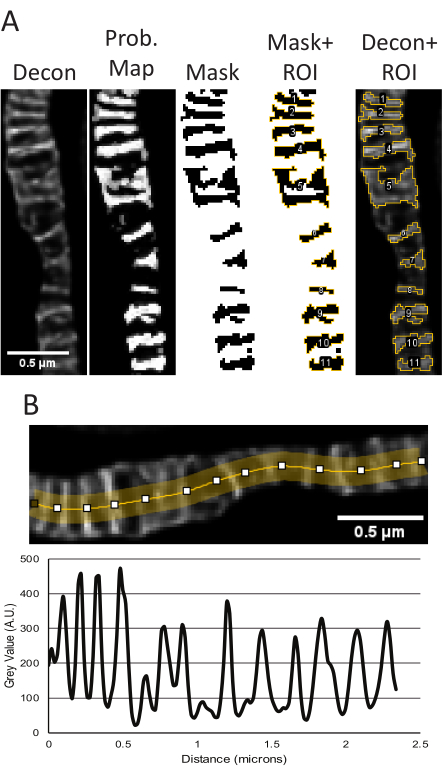
Figura 5: Procesamiento de imágenes STED desconvolucionadas en ImageJ. Se muestra el uso representativo del complemento Trainable Weka Segmentation para medir el tamaño y la forma de las crestas (A). De izquierda a derecha, se muestran las siguientes imágenes: la imagen STED desconvolucionada, el mapa de probabilidad basado en la segmentación del complemento TWS, la máscara del umbral en FIJI utilizando el mapa de probabilidad como entrada, la máscara con los ROI delineados y los ROI superpuestos en la imagen STED original desconvolucionada. Las mediciones resultantes del área, el perímetro, la circularidad y la relación de aspecto correspondientes a estos objetos se pueden encontrar en la Tabla Suplementaria 1. Se muestra un gráfico lineal que utiliza la imagen STED desconvolucionada para medir las distancias de pico a pico como lectura de la densidad de crestas (B). Barras de escala: 0,5 μm. Haga clic aquí para ver una versión más grande de esta figura.
| Tamaño de píxel (nm) | Tiempo de permanencia (μs) | Línea acc. | Excitación de 561 nm durante la adquisición de STED (%) | Potencia de agotamiento de STED de 775 nm (%) | Tamaño del paso (nm) | Estenopeico (AU) | Intervalo (s) de lapso de tiempo | Iteraciones de timelapse | |
| SH-SY5Y sin diferencias | 20 | 4 | 10 | 15 | 20 | 200 | 1.0 | 30 | 5 |
| SH-SY5Y diferenciado de RA | 20-25 | 4 | 10-12 | 15 | 20-22 | 150-200 | 1.0 | 30 | 5 |
| Neuronas primarias | 20-25 | 4 | 10 | 10 | 25 | 200 | 0.7 | 30 | 5 |
| NOTA: El tamaño de píxel puede variar en función de los requisitos de imagen y de la intención de desconvolucionar las imágenes. Se requiere un muestreo adecuado para la deconvolución. Los tamaños de píxel para imágenes STED sin procesar sin deconvolución pueden llegar hasta 30 nm. | |||||||||
Tabla 1: Resumen de los parámetros de adquisición de STED. Se muestran los ajustes utilizados para la obtención de imágenes STED 2D para cada tipo de célula, SH-SY5Y indiferenciada, SH-SY5Y diferenciada por AR y neuronas primarias del hipocampo de rata. Para todos los time-lapses, se tomaron 5 iteraciones con intervalos variables según el tamaño del ROI.
Figura complementaria 1: Imágenes de células SH-SY5Y con adición de amiloide-β (Aβ). Se muestran imágenes representativas confocales (izquierda), STED crudas (centro) y STED desconvolucionadas (derecha) de células SH-SY5Y diferenciadas por AR con tinción PKMO (arriba) y Aβ-HiLyte647 (abajo) (A). Se muestran proyecciones combinadas de la pila z de PKMO STED sin procesar (verde) con Aβ STED sin procesar (magenta) (B) o PKMO STED desconvolucionado (verde) con Aβ STED (magenta) (C) sin procesar. Barras de escala: 0,5 μm. Haga clic aquí para descargar este archivo.
Tabla complementaria 1: Medidas de tamaño y forma de las crestas segmentadas. Se muestran las medidas de tamaño y forma del área (μm2), el perímetro (μm), la circularidad y la relación de aspecto, correspondientes a los objetos descritos en la Figura 5A de mitocondrias segmentadas. Haga clic aquí para descargar este archivo.
Tabla complementaria 2: Resumen de los parámetros de adquisición con muestras de β amiloide. Se muestran los ajustes utilizados para la obtención de imágenes STED 2D de PKMO y Aβ-HiLyte647 en células SH-SY5Y indiferenciadas y diferenciadas por AR. El confocal de Aβ-HiLyte647 se puede usar solo, ya que no hay una estructura específica para resolver; Aquí se muestran imágenes STED de Aβ-HiLyte647 para tamaños de partícula más pequeños. Haga clic aquí para descargar este archivo.
Ficha complementaria 1: Protocolo de tratamiento de β amiloide. Haga clic aquí para descargar este archivo.
Discusión
Este protocolo presenta el uso de la línea celular de neuroblastoma humano SH-SY5Y y neuronas primarias del hipocampo de rata con el nuevo colorante PKMO dirigido a IMM para la obtención de imágenes de ETS de células vivas. Debido a la novedad de la PKMO, actualmente hay poco material publicado que utilice este colorante para la obtención de imágenes de ETS en vivo. El uso de estos tipos de células para la obtención de imágenes de ETS plantea desafíos, específicamente porque las células neuronales tienen mitocondrias más estrechas. Una limitación de este protocolo es el colorante PKMO utilizado, ya que puede ser tóxico para las células. Las diferentes células y líneas celulares responden de manera diferente al colorante, por lo tanto, es posible que se requieran ajustes en la concentración del colorante y el tiempo de incubación para optimizar los resultados de una señal fuerte sin dañar las células. Una solución sugerida es disminuir la concentración y aumentar el tiempo de tinción63; sin embargo, esto puede dar lugar a una tinción más deficiente sin aumentar la viabilidad celular.
Al igual que el PKMO, el colorante comercial Live Orange mito (Table of Materials) también presenta cierta toxicidad celular. Este colorante se utilizó para una variedad de células cultivadas, pero no pudo exhibir una tinción comparable en células SH-SY5Y diferenciadas por AR con éxito con los mismos parámetros que sus contrapartes indiferenciadas (nuestras observaciones no publicadas). Sin embargo, los protocolos de tinción susceptible pueden optimizarse para esta sonda y los tipos de células elegidos. Con este colorante, se utilizaron tiempos de activación del detector de 1-1,05 a 7-7,05 ns, permaneciendo todos los demás parámetros de la Tabla 1 iguales. En general, la tinción de células con 200-250 nM de mito de naranja vivo durante 45 min produjo resultados comparables a los que mostraron los resultados de PKMO. La tinción de mayor concentración durante menos tiempo o la tinción de menor concentración durante la misma cantidad de tiempo o un poco más puede producir resultados diferentes y puede ser favorable para otros tipos de células o condiciones de crecimiento.
La obtención de imágenes de las neuronas primarias del hipocampo de rata difiere de las células inmortalizadas debido a la naturaleza de las proyecciones de axones y dendritas, así como a la distribución mitocondrial en el momento de la obtención de imágenes. Una dificultad en esta parte del protocolo es que la densidad de siembra determina si los cultivos primarios podrán adherirse y crecer saludablemente, y a densidades más altas, las proyecciones tienden a crecer en exceso en DIV 10. Por lo tanto, es probable que las mitocondrias obtenidas de estas neuronas primarias provengan del cuerpo celular y no de las proyecciones; Sin embargo, el crecimiento exitoso a partir de una densidad celular inicial más baja produce mejores resultados de imagen en tiempos de crecimiento posteriores. La clave es garantizar un fondo bajo y una luz desenfocada para tener el mejor contraste para STED. Para abordar las preocupaciones relacionadas con la población celular, el cultivo de células primarias del hipocampo en medios de crecimiento neuronal suplementados con B27 previene el crecimiento de células gliales, y la fuente informa que el <5% de las células son astrocitos y la ausencia de suplemento de NbActiv1 en el medio de crecimiento reduce el número de astrocitos en cultivos al <2%87. Tanto para las células SH-SY5Y cultivadas como para las neuronas primarias del hipocampo de rata, el recubrimiento PDL utilizado para el crecimiento contribuye a la neblina de fondo en las imágenes. Se logra una relación señal/ruido suficiente con los ajustes indicados en el (Cuadro 1) y la deconvolución elimina la mayor parte del fondo observado.
Además de las imágenes que se tratan aquí, también es posible agregar tratamientos o estrés a las células antes o durante la obtención de imágenes. Por ejemplo, la adición de peróxido de hidrógeno terc-butilo (tBHP) induce estrés oxidativo, y es posible monitorear los cambios en las mitocondrias a lo largo del tiempo después de la adición. La adición de amiloide-β (Aβ) con una etiqueta fluorescente permite monitorizar la distribución de este péptido en relación con las mitocondrias, así como con la estructura mitocondrial a lo largo del tiempo. La salud mitocondrial ha sido fuertemente implicada en la EA y está ampliamente respaldada para desempeñar un papel en la toxicidad de Aβ 43,71,72. En particular, el estado de diferenciación de las células SH-SY5Y afecta a la localización del precursor de la proteína Aβ (AβPP)85, y los experimentos que utilizan AβPP deben construirse cuidadosamente.
Como ejemplo de cómo se puede adaptar este protocolo, se muestra que la variante fluorescente Aβ(1-42)-HiLyte 647 se puede añadir a las células teñidas con PKMO 15 minutos antes de la obtención de imágenes (Figura suplementaria 1). Los parámetros de imagen son similares (Tabla suplementaria 2), con la principal diferencia de que se necesita un orificio más pequeño cuando se obtienen imágenes de mitocondrias más estrechas. La obtención de imágenes de Aβ-HiLyte647 con STED requiere menos excitación general (6%-8%) y depleción de STED (10%-12%) de potencia láser y menos acumulaciones (seis). La compuerta del detector también se amplía de 0,1 a 10 ns. Aunque la resolución STED de Aβ no es necesaria, la relación señal-ruido general y el tamaño de partícula Aβ de STED sin procesar fueron mejores que los de las imágenes confocal, y también se puede realizar una deconvolución posterior. La recopilación de imágenes STED y la desconvolucionación de proyecciones STED en bruto de la pila z de Aβ parece particularmente útil cuando se fusionan con imágenes STED en bruto o STED desconvolucionadas de la tinción PKMO (Figura suplementaria 1B,C). Ambos canales se recopilaron en un solo paso de fotograma. Se pueden obtener mediciones de la localización dependiente del tiempo, similares a las enumeradas en la Figura 2 y mostradas en la Figura 5, cuando corresponda, y las diferencias en la arquitectura de las crestas después del tratamiento de estrés u otras adiciones.
Otros métodos posibles para el doble marcaje en células vivas de las mitocondrias de las ETS que no se informan aquí, pero que han sido informados por otros, incluyen el uso de proteínas93 marcadas con SNAP, proteínas marcadas con Halo y el uso de otros colorantes permeables a las células con objetivos genéricos, como el ADNmt63. En particular, la estrategia de marcaje de SNAP y el etiquetado de Halo influye en la intensidad de la señal de fluorescencia resultante y en la longevidad de la obtención de imágenes94. Además, aunque este protocolo presenta varios ejemplos de análisis que se pueden aplicar a mitocondrias segmentadas, hay muchos otros análisis que los paquetes de software pueden realizar en estas imágenes.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Las neuronas primarias del hipocampo de rata fueron suministradas por el Dr. George Lykotrafitis y Shiju Gu del Departamento de Ingeniería Biomédica de la Universidad de Connecticut (Storrs, CT, EE. UU.). El instrumento Abberior STED, alojado en la Instalación de Microscopía Óptica Avanzada en el Centro de Recursos y Equipos de Investigación Abierta, fue adquirido con una subvención de los NIH S10OD023618 otorgada a Christopher O'Connell. Esta investigación fue financiada por una subvención de los NIH R01AG065879 otorgada a Nathan N. Alder.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300054 | |
| 0.4% Trypan blue | Invitrogen | T10282 | |
| 0.5% Trypsin-EDTA, no phenol red | Gibco | 15400054 | |
| 100 X antibiotic-antimycotic | Gibco | 15240062 | |
| 100 X/1.40 UPlanSApo oil immersion lens | Olympus | Equipped in Olympus IX83 microscope for STED setup described in Section 4 | |
| All-trans-retinoic acid | Sigma | R2625 | |
| Amyloid-β (1-42, HiLyte Fluor647, 0.1 mg) | AnaSpec | AS-64161 | Other fluorescent conjugates available |
| B27 supplement (50 X), serum free | Gibco | 17504044 | |
| Cell Counter (Countess II FL) | Life Technologies | AMQAF1000 | |
| Centrifuge | Eppendorf | 5804-R | |
| Counter slides | Invitrogen | C10283 | |
| Conical tubes (15 mL) | Thermo Fisher Scientific | 339650 | |
| Cuvettes (Quartz Cells) | Starna Cells, Inc. | 9-Q-10 | Used with Spectrometer as described in Section 1.3 |
| DMEM (high glucose with sodium pyruvate) | Gibco | 11995073 | Used for SH-SY5Y cell materials as described in Section 1 |
| DMEM (high glucose no sodium pyruvate) | Gibco | 11965092 | Used for primary cell materials as described in Section 2 |
| DMEM (phenol red-free) | Gibco | 31053028 | Used for imaging as described in Section 3 |
| DMSO | Sigma | D8418 | |
| DNAase I from bovine pancreas | Sigma | DN25 | Used for primary cell materials as described in Section 2.2.1 and 2.2.2 |
| DPBS (no calcium, no magnesium) | Gibco | 14190144 | |
| E18 Rat Hippocampus | Transnetyx Tissue | SDEHP | |
| Ethanol (200 proof) | Fisher Bioreagents | BP28184 | |
| Fetal bovine serum (FBS), not heat-inactivated | Gibco | 26140079 | For cultured cells, in Section 1 |
| Fetal bovine serum (heat inactivated) | Gibco | 10082147 | For primary cell culture, Section 2 |
| Filter sterilization unit (0.1 µm, 500 mL) | Thermo Fisher Scientific | 5660010 | |
| FIJI (Is Just ImageJ) and Trainable Weka Segmentation (TWS) plug-in | -- | -- | Free, open-source image analysis software that includes plug-ins including Trainable Weka Segmentation described in Section 5; TWS plug-in from ref. 90 of the main text |
| GlutaMAX supplement (100 X) | Gibco | 35050061 | Glutamine supplement used for primary cell materials described in Section 2.1.2 |
| Hausser Scientific bright-Line and Hy-Lite Counting Chambers | Hausser Scientific | 267110 | |
| HBSS (no calcium, no magnesium) | Gibco | 14170120 | Used for primary cell materials described in Section 2.2.1 and 2.2.2 |
| HEPES | Gibco | 15630080 | |
| Huygens Professional deconvolution software (V. 20.10) | Scientific Volume Imaging (SVI) | -- | The deconvolution software used in this protocol and described in Section 5 |
| IX83 inverted microscope with Continuous Autofocus | Olympus | -- | This paper uses a STED Infinity Line system built around an Olympus IX83 inverted microscope, described in Section 4 |
| Lightbox software (V. 16.3.16118) | Abberior | -- | Vendor software used for STED image acquisition, described in Section 4 |
| Live Orange Mito dye | Abberior | LVORANGE-0146-30NMOL | Live cell imaging IMM-targeting dye described in Discussion |
| Neurobasal media | Gibco | 21103049 | Used for primary cell materials referred to in Section 2.1.2 |
| Nunc Lab-Tek II 2-well chambered coverglass | Nunc | 155379 | Can purchase a variety of chambers but make sure the coverglass is #1.5 |
| Pasteur Pipets (Fisherbrand) | Thermo Fisher Scientific | 22183632 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| PKmito Orange dye | Spirochrome | SC053 | |
| Poly-D-lysine | Gibco | A3890401 | |
| SH-SY5Y Cell line | ATCC | CRL2266 | |
| Sodium pyruvate (100 mM) | Gibco | 11360070 | Used for primary cell materials described in Section 2 |
| Spectrometer (GENESYS 180 UV-Vis) | Thermo Fisher Scientific | 840309000 | |
| STED Expert Line microscope | Abberior | -- | STED setup can be customized, but at time of purchase instrument was considered Abberior’s Expert Line; brief description of setup is available in Section 4 of protocol |
| T25 flask (TC-treated, filter cap) | Thermo Fisher Scientific | 156367 | Other culture vessels and sizes available |
Referencias
- Iovine, J. C., Claypool, S. M., Alder, N. N. Mitochondrial compartmentalization: emerging themes in structure and function. Trends in Biochemical Sciences. 46 (11), 902-917 (2021).
- Gupta, A., Becker, T. Mechanisms and pathways of mitochondrial outer membrane protein biogenesis. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1862 (1), 148323 (2021).
- Gordaliza-Alaguero, I., Cantó, C., Zorzano, A. Metabolic implications of organelle-mitochondria communication. EMBO Reports. 20 (9), e47928 (2019).
- Klecker, T., Westermann, B. Pathways shaping the mitochondrial inner membrane. Open Biology. 11 (12), 210238 (2021).
- Navarro, A., Boveris, A. The mitochondrial energy transduction system and the aging process. American Journal of Physiology-Cell Physiology. 292 (2), C670-C686 (2007).
- Yu, R., Lendahl, U., Nistér, M., Zhao, J. Regulation of mammalian mitochondrial dynamics: opportunities and challenges. Frontiers in Endocrinology. 11, 374 (2020).
- Horn, A., Raavicharla, S., Shah, S., Cox, D., Jaiswal, J. K. Mitochondrial fragmentation enables localized signaling required for cell repair. The Journal of Cell Biology. 219 (5), e201909154 (2020).
- Glancy, B., Kim, Y., Katti, P., Willingham, T. B. The functional impact of mitochondrial structure across subcellular scales. Frontiers in Physiology. 11, 541040 (2020).
- Bahat, A., et al. MTCH2-mediated mitochondrial fusion drives exit from naïve pluripotency in embryonic stem cells. Nature Communications. 9 (1), 5132 (2018).
- Detmer, S. A., Chan, D. C. Functions and dysfunctions of mitochondrial dynamics. Nature Reviews Molecular Cell Biology. 8, 870-879 (2007).
- Bertholet, A. M., et al. Mitochondrial fusion/fission dynamics in neurodegeneration and neuronal plasticity. Neurobiology of Disease. 90, 3-19 (2016).
- Zemirli, N., Morel, E., Molino, D. Mitochondrial dynamics in basal and stressful conditions. International Journal of Molecular Sciences. 19 (2), 564 (2018).
- Harwig, M. C., et al. Methods for imaging mammalian mitochondrial morphology: a prospective on mitograph. Analytical Biochemistry. 552, 81-99 (2018).
- Pánek, T., Eliáš, M., Vancová, M., Lukeš, J., Hashimi, H. Returning to the fold for lessons in mitochondrial crista diversity and evolution. Current Biology. 30 (10), R575-R588 (2020).
- Gottschalk, B., Madreiter-Sokolowski, C. T., Graier, W. F. Cristae junction as a fundamental switchboard for mitochondrial ion signaling and bioenergetics. Cell Calcium. 101, 102517 (2022).
- Khosravi, S., Harner, M. E. The MICOS complex, a structural element of mitochondria with versatile functions. Biological Chemistry. 401 (6-7), 765-778 (2020).
- Frezza, C., et al. OPA1 controls apoptotic cristae remodeling independently from mitochondrial fusion. Cell. 126 (1), 177-189 (2006).
- Meeusen, S., et al. Mitochondrial inner-membrane fusion and crista maintenance requires the dynamin-related GTPase Mgm1. Cell. 127 (2), 383-395 (2006).
- Patten, D. A., et al. OPA1-dependent cristae modulation is essential for cellular adaptation to metabolic demand. The EMBO Journal. 33 (22), 2676-2691 (2014).
- Paumard, P., et al. The ATP synthase is involved in generating mitochondrial cristae morphology. The EMBO Journal. 21 (3), 221-230 (2002).
- Strauss, M., Hofhaus, G., Schröder, R. R., Kühlbrandt, W. Dimer ribbons of ATP synthase shape the inner mitochondrial membrane. The EMBO Journal. 27 (7), 1154-1160 (2008).
- Basu Ball, W., Neff, J. K., Gohil, V. M. The role of nonbilayer phospholipids in mitochondrial structure and function. FEBS Letters. 592 (8), 1273-1290 (2018).
- Hackenbrock, C. R. Ultrastructural bases for metabolically linked mechanical activity in mitochondria. I. Reversible ultrastructural changes with change in metabolic steady state in isolated liver mitochondria. The Journal of Cell Biology. 30 (2), 269-297 (1966).
- Dlasková, A., et al. Mitochondrial cristae narrowing upon higher 2-oxoglutarate load. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1860 (8), 659-678 (2019).
- Pérez-Hernández, C. A., et al. Mitochondrial ultrastructure and activity are differentially regulated by glycolysis-, krebs cycle-, and microbiota-derived metabolites in monocytes. Biology. 11 (8), 1132 (2022).
- Mannella, C. A. Structural diversity of mitochondria: functional implications. Annals of the New York Academy of Sciences. 1147, 171-179 (2008).
- Plecitá-Hlavatá, L., Ježek, P. Integration of superoxide formation and cristae morphology for mitochondrial redox signaling. The International Journal of Biochemistry & Cell Biology. 80, 31-50 (2016).
- Scorrano, L., et al. A distinct pathway remodels mitochondrial cristae and mobilizes cytochrome c during apoptosis. Developmental Cell. 2 (1), 55-67 (2002).
- Heath-Engel, H. M., Shore, G. C. Mitochondrial membrane dynamics, cristae remodelling and apoptosis. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1763 (5-6), 549-560 (2006).
- Brandt, T., et al. Changes of mitochondrial ultrastructure and function during ageing in mice and Drosophilia. eLife. 6, e24662 (2017).
- Kondadi, A. K., et al. Cristae undergo continuous cycles of membrane remodelling in a MICOS-dependent manner. EMBO Reports. 21, e49776 (2020).
- Quintana-Cabrera, R., Mehrotra, A., Rigoni, G., Soriano, M. E. Who and how in the regulation of mitochondrial cristae shape and function. Biochemical and Biophysical Research Communications. 500 (1), 94-101 (2018).
- Nielsen, J., et al. Plasticity in mitochondrial cristae density allows metabolic capacity modulation in human skeletal muscle: Enlarged mitochondrial cristae density in athletes. The Journal of Physiology. 595 (9), 2839-2847 (2017).
- Afzal, N., Lederer, W. J., Jafri, M. S., Mannella, C. A. Effect of crista morphology on mitochondrial ATP output: A computational study. Current Research in Physiology. 4, 163-176 (2021).
- Heine, K. B., Parry, H. A., Hood, W. R. How does density of the inner mitochondrial membrane influence mitochondrial performance. American Journal of Physiology-Regulatory, Integrative and Comparative Physiology. 324 (2), R242-R248 (2023).
- Wang, W., Zhao, F., Ma, X., Perry, G., Zhu, X. Mitochondria dysfunction in the pathogenesis of Alzheimer's disease: recent advances. Molecular Neurodegeneration. 15, 30 (2020).
- Singh, A., Kukreti, R., Saso, L., Kukreti, S. Oxidative stress: a key modulator in neurodegenerative diseases. Molecules. 24, 1583 (2019).
- Pchitskaya, E., Popugaeva, E., Bezprozvanny, I. Calcium signaling and molecular mechanisms underlying neurodegenerative diseases. Cell Calcium. 70, 87-94 (2018).
- Estes, R. E., Lin, B., Khera, A., Davis, M. Y. Lipid metabolism influence on neurodegenerative disease progression: is the vehicle as important as the cargo. Frontiers in Molecular Neuroscience. 14, 788695 (2021).
- Calkins, M. J., Manczak, M., Mao, P., Shirendeb, U., Reddy, P. H. Impaired mitochondrial biogenesis, defective axonal transport of mitochondria, abnormal mitochondrial dynamics and synaptic degeneration in a mouse model of Alzheimer's disease. Human Molecular Genetics. 20 (23), 4515-4529 (2011).
- Petersen, C. A. H., et al. The amyloid beta-peptide is imported into mitochondria via the TOM import machinery and localized to mitochondrial cristae. Proceedings of the National Academy of Sciences of the United States of America. 105 (35), 13145-13150 (2008).
- Gan, X., et al. Inhibition of ERK-DLP1 signaling and mitochondrial division alleviates mitochondrial dysfunction in Alzheimer's disease cybrid cell. Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1842 (2), 220-231 (2014).
- Baloyannis, S. J., Costa, V., Michmizos, D. Mitochondrial alterations Alzheimer's disease. American Journal of Alzheimer's Disease & Other Dementias. 19 (2), 89-93 (2004).
- Tillement, L., Lecanu, L., Papadopoulos, V. Alzheimer's disease: Effects of β-amyloid on mitochondria. Mitochondrion. 11 (1), 13-21 (2011).
- Choi, S. Y., et al. C9ORF72-ALS/FTD-associated poly(GR) binds Atp5a1 and compromises mitochondrial function in vivo. Nature Neuroscience. 22 (6), 851-862 (2019).
- Smith, E. F., Shaw, P. J., De Vos, K. J. The role of mitochondria in amyotrophic lateral sclerosis. Neuroscience Letters. 710, 132933 (2019).
- Costa, V., et al. Mitochondrial fission and cristae disruption increase the response of cell models of Huntington's disease to apoptotic stimuli. EMBO Molecular Medicine. 2 (12), 490-503 (2010).
- Costa, V., Scorrano, L. Shaping the role of mitochondria in the pathogenesis of Huntington's disease: Mitochondrial and Huntington's disease. The EMBO Journal. 31 (8), 1853-1864 (2012).
- Vanisova, M., et al. Mitochondrial organization and structure are compromised in fibroblasts from patients with Huntington's disease. Ultrastructural Pathology. 46 (5), 462-475 (2022).
- de Barcelos, I. P., Troxell, R. M., Graves, J. S. Mitochondrial dysfunction and multiple sclerosis. Biology. 8 (2), 37 (2019).
- Park, J., et al. Mitochondrial dysfunction in Drosophila PINK1 mutants is complemented by parkin. Nature. 441, 1157-1161 (2006).
- Meng, H., et al. Loss of Parkinson's disease-associated protein CHCHD2 affects mitochondrial crista structure and destabilizes cytochrome c. Nature Communications. 8, 15500 (2017).
- Lu, L., et al. CHCHD2 maintains mitochondrial contact site and cristae organizing system stability and protects against mitochondrial dysfunction in an experimental model of Parkinson's disease. Chinese Medical Journal. 135 (13), 1588-1596 (2022).
- Cogliati, S., et al. Mitochondrial Cristae shape determines respiratory chain supercomplexes assembly and respiratory efficiency. Cell. 155 (1), 160-171 (2013).
- He, B., et al. Mitochondrial cristae architecture protects against mtDNA release and inflammation. Cell Reports. 41 (10), 111774 (2022).
- Polo, C. C., et al. Three-dimensional imaging of mitochondrial cristae complexity using cryo-soft X-ray tomography. Scientific Reports. 10, 21045 (2020).
- Rybka, V., et al. Transmission electron microscopy study of mitochondria in aging brain synapses. Antioxidants. 8 (6), 171 (2019).
- Mannella, C. A. Structure and dynamics of the mitochondrial inner membrane cristae. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1763 (5-6), 542-548 (2006).
- Fry, M. Y., et al. In situ architecture of Opa1-dependent mitochondrial cristae remodeling. biorxiv. , (2023).
- Barad, B. A., Medina, M., Fuentes, D., Wiseman, R. L., Grotjahn, D. A. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. The Journal of Cell Biology. 222 (4), 202204093 (2023).
- Kunz, T. C., Götz, R., Gao, S., Sauer, M., Kozjak-Pavlovic, V. Using expansion microscopy to visualize and characterize the morphology of mitochondrial cristae. Frontiers in Cell and Developmental Biology. 8, 617 (2020).
- Yang, Z., et al. Cyclooctatetraene-conjugated cyanine mitochondrial probes minimize phototoxicity in fluorescence and nanoscopic imaging. Chemical Science. 11 (32), 8506-8516 (2020).
- Liu, T., et al. Multi-color live-cell STED nanoscopy of mitochondria with a gentle inner membrane stain. Proceedings of the National Academy of Sciences. 119 (52), e2215799119 (2022).
- Yang, X., et al. Mitochondrial dynamics quantitatively revealed by STED nanoscopy with an enhanced squaraine variant probe. Nature Communications. 11, 3699 (2020).
- Zheng, S., et al. Long-term, super-resolution HIDE imaging of the inner mitochondrial membrane in live cells with a cell-permeant lipid probe. biorxiv. , (2022).
- Wang, C., et al. A photostable fluorescent marker for the superresolution live imaging of the dynamic structure of the mitochondrial cristae. Proceedings of the National Academy of Sciences of the United States of America. 116 (32), 15817-15822 (2019).
- Feles, S., et al. Streamlining culture conditions for the neuroblastoma cell line sh-sy5y: a prerequisite for functional studies. Methods and Protocols. 5 (4), 58 (2022).
- Shipley, M. M., Mangold, C. A., Szpara, M. L. Differentiation of the SH-SY5Y human neuroblastoma cell line. Journal of Visualized Experiments. (108), e53193 (2016).
- Kovalevich, J., Langford, D., Amini, S., White, M. K. Considerations for the use of SH-SY5Y neuroblastoma cells in neurobiology. Neuronal Cell Culture. 1078, 9-21 (2013).
- Biedler, J. L., Helson, L., Spengler, B. A. Morphology and growth, tumorigenicity, and cytogenetics of human neuroblastoma cells in continuous culture. Cancer Research. 33 (11), 2643-2652 (1973).
- Swerdlow, R. H. Mitochondria and mitochondrial cascades in alzheimer's disease. Journal of Alzheimer's Disease. 62 (3), 1403-1416 (2018).
- Wang, W., Zhao, F., Ma, X., Perry, G., Zhu, X. Mitochondria dysfunction in the pathogenesis of Alzheimer's disease: recent advances. Molecular Neurodegeneration. 15, 30 (2020).
- Reddy, P. H. Mitochondrial dysfunction in aging and alzheimer's disease: strategies to protect neurons. Antioxidants & Redox Signaling. 9 (10), 1647-1658 (2007).
- Horn, A., Raavicharla, S., Shah, S., Cox, D., Jaiswal, J. K. Mitochondrial fragmentation enables localized signaling required for cell repair. Journal of Cell Biology. 219 (5), e201909154 (2020).
- Korecka, J. A., et al. Phenotypic characterization of retinoic acid differentiated SH-SY5Y cells by transcriptional profiling. PLoS ONE. 8 (5), e63862 (2013).
- Baghel, M. S., Thakur, M. K. Vdac1 downregulation causes mitochondrial disintegration leading to hippocampal neurodegeneration in scopolamine-induced amnesic mice. Molecular Neurobiology. 56 (3), 1707-1718 (2019).
- Jiang, S., et al. Mfn2 ablation causes an oxidative stress response and eventual neuronal death in the hippocampus and cortex. Molecular Neurodegeneration. 13 (1), 5 (2018).
- Mu, Y., Gage, F. H. Adult hippocampal neurogenesis and its role in Alzheimer's disease. Molecular Neurodegeneration. 6, 85 (2011).
- Rao, Y. L., et al. Hippocampus and its involvement in Alzheimer's disease: a review. 3 Biotech. 12 (2), 55 (2022).
- Weerasinghe-Mudiyanselage, P. D. E., Ang, M. J., Kang, S., Kim, J. -. S., Moon, C. Structural Plasticity of the hippocampus in neurodegenerative diseases. International Journal of Molecular Sciences. 23 (6), 3349 (2022).
- . Poly-D-Lysine Available from: https://www.thermofisher.com/order/catalog/product/A3890401 (2023)
- Dravid, A., Raos, B., Svirskis, D., O'Carroll, S. J. Optimised techniques for high-throughput screening of differentiated SH-SY5Y cells and application for neurite outgrowth assays. Scientific Reports. 11, 23935 (2021).
- Hromadkova, L., et al. Brain-derived neurotrophic factor (BDNF) promotes molecular polarization and differentiation of immature neuroblastoma cells into definitive neurons. Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1867 (9), 118737 (2020).
- Riegerová, P., et al. Expression and Localization of AβPP in SH-SY5Y cells depends on differentiation state. Journal of Alzheimer's Disease. 82 (2), 485-491 (2021).
- Hoffmann, L. F., et al. Neural regeneration research model to be explored: SH-SY5Y human neuroblastoma cells. Neural Regeneration Research. 18 (6), 1265-1266 (2022).
- Abiraman, K., Tzingounis, A. V., Lykotrafitis, G. K. Ca 2 channel localization and regulation in the axon initial segment. The FASEB Journal. 32 (4), 1794-1805 (2018).
- E18 Rat Hippocampus. Transnetyx Tissue Available from: https://tissue.transnetyx.com/E18-Rat-Hippocampus_4 (2023)
- Kmito ORANGE - Probe for live cell imaging of mitochondria. Spirochrome Available from: https://spirochrome.com/product/pkmito_orange/ (2023)
- Nyquist Calculator. Scientific Volume Imaging Available from: https://svi.nl/Nyquist-Calculator (2023)
- Segawa, M., et al. Quantification of cristae architecture reveals time-dependent characteristics of individual mitochondria. Life Science Alliance. 3 (7), e2019000620 (2020).
- Arganda-Carreras, I., et al. Trainable Weka Segmentation: a machine learning tool for microscopy pixel classification. Bioinformatics. 33 (15), 2424-2426 (2017).
- Stephan, T., Roesch, A., Riedel, D., Jakobs, S. Live-cell STED nanoscopy of mitochondrial cristae. Scientific Reports. 9, 12419 (2019).
- Erdmann, R. S., et al. Labeling strategies matter for super-resolution microscopy: A comparison between HaloTags and SNAP-tags. Cell Chemical Biology. 26 (4), 584-592 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados