Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Modelado realista de membranas utilizando mezclas lipídicas complejas en estudios de simulación
En este artículo
Resumen
La diversidad lipídica de la membrana en estructura y composición es un importante contribuyente a los procesos celulares y puede ser un marcador de enfermedad. Las simulaciones de dinámica molecular nos permiten estudiar las membranas y sus interacciones con biomoléculas a resolución atomística. Aquí, proporcionamos un protocolo para construir, ejecutar y analizar sistemas de membranas complejos.
Resumen
Los lípidos son componentes estructurales de las membranas celulares; Las especies de lípidos varían entre los orgánulos celulares y entre los organismos. Esta variedad da como resultado diferentes propiedades mecánicas y estructurales en la membrana que impactan directamente en las moléculas y procesos que ocurren en esta interfaz. La composición lipídica es dinámica y puede servir para modular los procesos de señalización celular. Los enfoques computacionales se utilizan cada vez más para predecir las interacciones entre biomoléculas y proporcionar información molecular a los observables experimentales. La dinámica molecular (MD) es una técnica basada en la mecánica estadística que predice el movimiento de los átomos en función de las fuerzas que actúan sobre ellos. Las simulaciones de MD se pueden utilizar para caracterizar la interacción de biomoléculas. Aquí, presentamos brevemente la técnica, describimos los pasos prácticos para principiantes que estén interesados en simular bicapas lipídicas, demostramos el protocolo con un software amigable para principiantes y discutimos alternativas, desafíos y consideraciones importantes del proceso. En particular, enfatizamos la relevancia del uso de mezclas lipídicas complejas para modelar una membrana celular de interés para capturar los entornos hidrofóbicos y mecánicos apropiados en la simulación. También discutimos algunos ejemplos en los que la composición y las propiedades de la membrana modulan las interacciones de las bicapas con otras biomoléculas.
Introducción
Los lípidos son los principales constituyentes de las membranas, que proporcionan límites para las células y permiten la compartimentación intracelular 1,2,3. Los lípidos son anfifílicos, con un grupo de cabeza polar y dos colas de ácidos grasos hidrofóbicos; Estos se autoensamblan en una bicapa para minimizar el contacto de las cadenas hidrofóbicas con el agua 3,4. Varias combinaciones de grupos de cabezas hidrofílicas y colas hidrofóbicas dan como resultado diferentes clases de lípidos en las membranas biológicas, como glicerofosfolípidos, esfingolípidos y esteroles (Figura 1)1,5,6. Los glicerofosfolípidos son componentes primarios de las membranas celulares eucariotas compuestas de glicerofosfato, ácidos grasos de cadena larga y grupos principales de bajo peso molecular7. La nomenclatura lipídica se basa en las diferencias en los grupos de cabeza; algunos ejemplos son la fosfatidilcolina (PC), la fosfatidil-etanolamina (PE), la fosfatidil-serina (PS), el fosfatidilglicerol (PG), el fosfatidil-inositol (PI) o el ácido fosfatídico (PA) no modificado5,6. En cuanto a las colas hidrofóbicas, la longitud y el grado de saturación varían, junto con la estructura de la columna vertebral. Las combinaciones posibles son numerosas, resultando en miles de especies lipídicas en células de mamíferos6. Los cambios en la composición lipídica de la membrana conducen a diferentes propiedades mecánicas y estructurales de la membrana que afectan la actividad tanto de las proteínas integrales de membrana como de las proteínas periféricas 2,6.
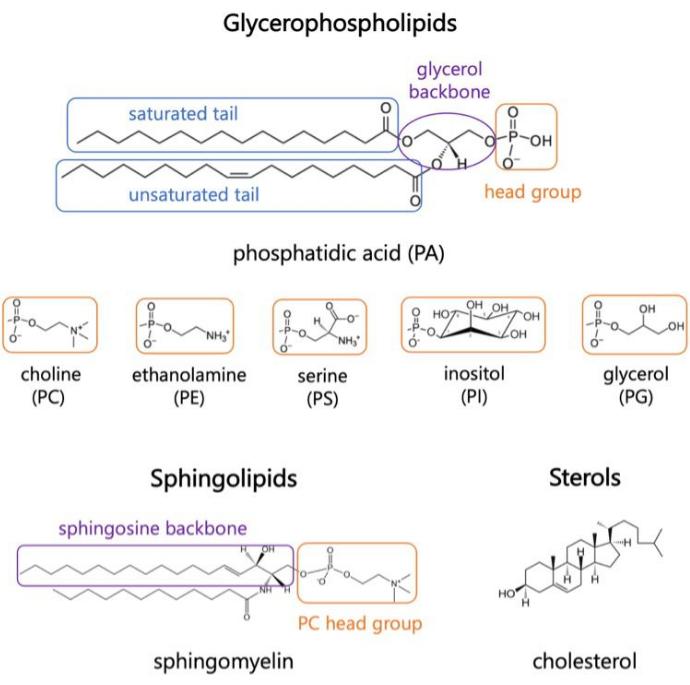
Figura 1. Estructuras lipídicas representativas. Las colas de ácidos grasos se muestran en cuadros azules, los grupos de cabezas de lípidos comunes en naranja y las columnas vertebrales de las muestras en púrpura. Haga clic aquí para ver una versión más grande de esta figura.
Los lípidos son actores activos en los procesos celulares, en la activación de proteínas en las cascadas de señalización y en la homeostasis de las células sanas 8,9. La alteración de la dinámica lipídica es el resultado de la infección o puede ser marcador de patogenia de la enfermedad 10,11,12,13,14,15. Como barreras para la célula, el estudio de los lípidos de membrana y su papel en la permeación de moléculas pequeñas es relevante para los sistemas de liberación de fármacos y los mecanismos de disrupción de la membrana16,17. La diversidad química y las diferentes proporciones de especies lipídicas en orgánulos, tejidos y organismos dan lugar a una dinámica compleja de membranas2. Por lo tanto, es importante mantener estas características en los estudios de modelización de bicapas lipídicas, especialmente cuando el objetivo de un estudio es examinar las interacciones de otras biomoléculas con la membrana. Las especies lipídicas a considerar en un modelo dependen del organismo y del compartimento celular de interés. Por ejemplo, los lípidos PG son importantes para la transferencia de electrones en la batería fotosintética18, mientras que los lípidos fosforilados de inositol (PIP) son actores importantes en la dinámica de la membrana plasmática (PM) y en las cascadas de señalización en las células de mamíferos19,20. Dentro de la célula, el PM, el retículo endoplásmico (RE), Golgi y las membranas mitocondriales contienen abundancias lipídicas únicas que influyen en su función. Por ejemplo, el RE es el centro de la biogénesis lipídica y transporta el colesterol hacia el PM y el Golgi; contiene una alta diversidad lipídica con abundancia de PC y PE, pero bajo contenido de esteroles, lo que promueve la fluidez de la membrana21,22,23,24. Por el contrario, el PM incorpora cientos e incluso miles de especies lipídicas dependiendo del organismo25, contiene altos niveles de esfingolípidos y colesterol que le confieren una rigidez característica en comparación con otras membranas de la célula24. Se debe considerar la asimetría de las valvas para membranas como la PM, que tiene una valva externa rica en esfingomielina, PC y colesterol, y una valva interna rica en PE, PI y PS que son importantes para las cascadas de señalización24. Finalmente, la diversidad lipídica también propicia la formación de microdominios que difieren en el empaquetamiento y el orden interno, conocidos como balsas lipídicas24,26; Estos exhiben asimetría lateral, se supone que juegan un papel importante en la señalización celular26 y son difíciles de estudiar debido a su naturaleza transitoria.
Se han utilizado técnicas experimentales como la fluoroscopia, la espectroscopia y sistemas de membranas modelo como las vesículas unilamelares gigantes (GUV) para investigar las interacciones de las biomoléculas con las membranas. Sin embargo, la naturaleza compleja y dinámica de los componentes involucrados es difícil de capturar solo con métodos experimentales. Por ejemplo, existen limitaciones en la obtención de imágenes de los dominios transmembrana de las proteínas, la complejidad de las membranas utilizadas en dichos estudios y la identificación de estados intermedios o transitorios durante el proceso de interés27,28,29. Desde el advenimiento de la simulación molecular de monocapas y bicapas lipídicas en la década de 198029, los sistemas lípido-proteína y sus interacciones pueden cuantificarse a nivel molecular. La simulación de dinámica molecular (MD) es una técnica computacional común que predice el movimiento de las partículas en función de sus fuerzas intermoleculares. Un potencial de interacción aditiva describe las interacciones enlazadas y no enlazadas entre las partículas del sistema30. El conjunto de parámetros utilizados para modelar estas interacciones se denomina campo de fuerza de simulación (FF). Estos parámetros se obtienen a partir de cálculos ab initio, semiempíricos y de mecánica cuántica, y se optimizan para reproducir datos de experimentos de rayos X y difracción de electrones, RMN, espectroscopía infrarroja, Raman y de neutrones, entre otros métodos31.
Las simulaciones de MD se pueden utilizar para estudiar sistemas en varios niveles de resolución32,33,34. Los sistemas que tienen como objetivo caracterizar interacciones biomoleculares específicas, enlaces de hidrógeno y otros detalles de alta resolución se estudian con simulaciones de todos los átomos (AA). Por el contrario, las simulaciones de grano grueso (CG) agrupan los átomos en grupos funcionales más grandes para reducir el costo computacional y examinarla dinámica a mayor escala. Entre estos dos se encuentran las simulaciones de átomos unidos (UA), donde los átomos de hidrógeno se combinan con sus respectivos átomos pesados para acelerar el cálculo33,35. Las simulaciones MD son una poderosa herramienta para la exploración de la dinámica de las membranas lipídicas y sus interacciones con otras moléculas y pueden servir para proporcionar mecanismos a nivel molecular para procesos de interés en la interfaz de la membrana. Además, las simulaciones de MD pueden servir para acotar objetivos experimentales y predecir las propiedades macromoleculares de un sistema determinado en función de las interacciones microscópicas.
En resumen, dado un conjunto de coordenadas iniciales, velocidades y un conjunto de condiciones como temperatura y presión constantes, las posiciones y velocidades de cada partícula se calculan a través de la integración numérica del potencial de interacción y la Ley del movimiento de Newton. Esto se repite de forma iterativa, generando así una trayectoria de simulación30. Estos cálculos se realizan con un motor MD; entre varios paquetes de código abierto, GROMACS36 es uno de los motores más utilizados y el que describimos aquí. También incluye herramientas para el análisis y la construcción de las coordenadas iniciales de los sistemas a simular37. Otros motores MD incluyen NAMD38; CHARMM39 y AMBER40, que el usuario puede seleccionar a su propia discreción en función del rendimiento computacional de un sistema determinado. Es fundamental visualizar las trayectorias durante la simulación, así como para el análisis e interpretación de los resultados. Hay una variedad de herramientas disponibles; aquí discutimos la dinámica molecular visual (VMD) que ofrece una amplia gama de características, incluida la visualización tridimensional (3-D) con métodos expansivos de dibujo y coloreado, la visualización de datos volumétricos, la construcción, preparación y análisis de trayectorias de sistemas de simulación MD y la creación de películas de trayectoria sin límites en el tamaño del sistema, si la memoria está disponible41,42,43.
La precisión de la dinámica predicha entre los componentes del sistema está directamente influenciada por el FF elegido para la propagación de la trayectoria. Los esfuerzos empíricos de parametrización de FF son llevados a cabo por pocos grupos de investigación. Los FF más establecidos y comunes para la distrofia muscular incluyen CHARMM39, AMBER 40, Martini44, OPLS45 y SIRAH46. El campo de fuerza47 del CHARMM36 aditivo de todos los átomos (C36) se usa ampliamente para AA MD de sistemas de membrana, ya que reproduce con precisión los datos estructurales experimentales. Fue desarrollado originalmente por la comunidad CHARMM y es compatible con múltiples motores MD como GROMACS y NAMD. A pesar de las mejoras en los FF comunes, existe un esfuerzo continuo para mejorar los conjuntos de parámetros para permitir predicciones que reproduzcan fielmente los observables experimentales, impulsados por el interés en sistemas particulares de estudio48,49.
Un reto a la hora de simular membranas lipídicas es determinar la longitud de la trayectoria de simulación. Esto depende en gran medida de las métricas a analizar y del proceso que se pretenda caracterizar. Típicamente, las mezclas lipídicas complejas requieren más tiempo para alcanzar el equilibrio, ya que más especies deben tener tiempo suficiente para difundirse en el plano de la membrana y alcanzar una organización lateral estable. Se dice que una simulación está en equilibrio cuando la propiedad de interés ha alcanzado una meseta y fluctúa alrededor de un valor constante. Es una práctica común obtener al menos 100-200 ns de trayectoria equilibrada para llevar a cabo un análisis estadístico adecuado sobre las propiedades e interacciones de interés. Es común ejecutar simulaciones solo de membrana entre 200 y 500 ns, dependiendo de la complejidad de la mezcla de lípidos y la pregunta de investigación. Las interacciones proteína-lípidos suelen requerir tiempos de simulación más largos, entre 500 y 2000 ns. Algunos enfoques para acelerar el muestreo y la dinámica observable con sistemas de membranas son: (i) el modelo mimético de membrana altamente móvil (HMMM), que reemplaza los carbonos finales de los lípidos en la membrana con solvente orgánico para acelerar el muestreo50; y ii) la repartición de la masa de hidrógeno (HMR), que combina una fracción de las masas de los átomos pesados dentro de un sistema con las de los átomos de hidrógeno para permitir el uso de un paso de tiempo de simulación más grande51.
En el siguiente protocolo se describe un enfoque fácil de usar para principiantes para crear, ejecutar y analizar modelos de membrana realistas mediante AA MD. Dada la naturaleza de las simulaciones de MD, se deben ejecutar múltiples trayectorias para tener en cuenta la reproducibilidad y el análisis estadístico adecuado de los resultados. Es una práctica actual ejecutar un mínimo de tres réplicas por sistema de interés. Una vez que se han seleccionado las especies lipídicas para el organismo y el proceso de interés, se describen y resumen en la Figura 2 los pasos básicos para construir, ejecutar y analizar una trayectoria de simulación de un sistema solo de membrana.
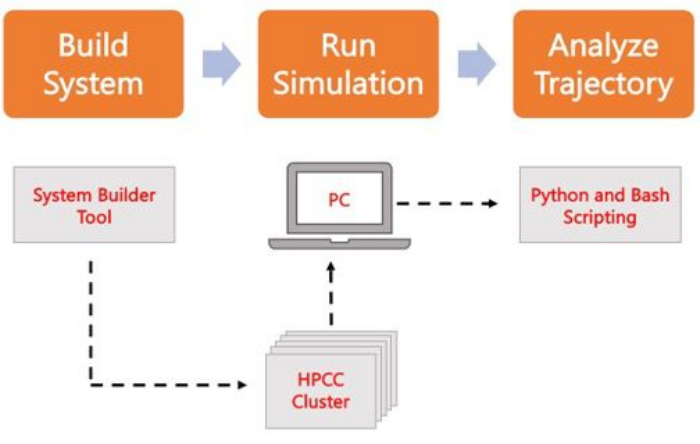
Figura 2. Esquema para ejecutar simulaciones de MD. Las casillas naranjas corresponden a los tres pasos principales descritos en el protocolo. Debajo está el flujo de trabajo del proceso de simulación. Durante la configuración del sistema, el sistema que contiene las coordenadas iniciales de un sistema de membrana solvatada se construye con un generador de entrada del sistema como CHARMM-GUI Membrane Builder. Después de transferir los archivos de entrada a un clúster informático de alto rendimiento, la trayectoria de simulación se propaga mediante un motor MD, como GROMACS. El análisis de la trayectoria se puede realizar en el clúster informático o en una estación de trabajo local junto con la visualización. A continuación, se lleva a cabo el análisis, ya sea utilizando paquetes con código de análisis incorporado, como GROMACS y VMD, o mediante scripts de Bash o varias bibliotecas de Python. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Construcción de las coordenadas del sistema
- Navegue hasta CHARMM-GUI.org (C-GUI) usando un navegador web. En el menú superior, vaya a Generador de entrada, luego seleccione Generador de membranas en las opciones verticales en el lado izquierdo de la pantalla.
- Para crear una bicapa, seleccione Bilayer Builder.
NOTA: Los usuarios nuevos deben activar su cuenta gratuita antes de crear su primer conjunto de coordenadas. - Seleccione Sistema de solo membrana. Guarde el ID de trabajo generado para recuperar el sistema y continuar desde donde lo dejó durante el proceso si es necesario.
- Visualice los sistemas durante cada paso del proceso de creación haciendo clic en Ver estructura en el cuadro situado en la parte superior de la página, o descargando el archivo PDB resultante. Esté atento a los componentes faltantes, errores en la especie lipídica de entrada seleccionada o en el tamaño del parche.
- Seleccione los componentes del sistema.
- Elija la opción Lípidos heterogéneos , incluso si construye una bicapa de un solo componente; y, a continuación, seleccione un tipo de caja rectangular .
- Seleccione 45 moléculas de agua por lípido para la opción de hidratación; Esto es suficiente para garantizar una bicapa completamente hidratada.
- Establezca la longitud de XY para que se base en el número de componentes lipídicos. A continuación, seleccione el número de lípidos que se incluirán para cada especie lipídica determinada antes del modelo. Para el caso de estudio que se discute en la siguiente sección, se construyó un modelo de membrana con 600 lípidos distribuidos simétricamente en dos valvas. Para modelar el RE de las células eucariotas se utilizó una mezcla de 336 lípidos DOPC, 132 DPPE, 60 CHOL, 72 POPI para el modelo PI; y 330 lípidos DOPC, 126 DPPE, 54 CHOL, 66 POPI y 24 DOPS para el modelo PI-PS.
NOTA: C-GUI proporciona una biblioteca de estructuras lipídicas para elegir; Haga clic en las imágenes junto al nombre de la especie por su estructura química. - Escriba el número deseado de moléculas en el folíolo superior e inferior en las dos casillas junto al nombre del lípido. Para el estudio de caso, se desea una composición de membrana simétrica: asegúrese de que no haya errores sobre el número inigualable de lípidos en el folíolo superior y el folíolo inferior. Si se desea asimetría, asegúrese de que el número total de lípidos en cada valva sea correcto. Para más detalles sobre la construcción de bicapas asimétricas, véase el trabajo de Park et al.52,53.
- Vaya a la parte superior de la lista de especies de lípidos y haga clic en el botón Mostrar información del sistema. Ensamble los componentes y complete el sistema.
- Seleccione la opción para incluir iones neutralizantes utilizando el algoritmo basado en la distancia para una convergencia más rápida54.
- Deje la concentración de solución predeterminada de KCl en 0,15 mM. Esta es una concentración de sal típica para hacer que la caja de simulación sea neutra para bicapas de membrana.
NOTA: Si se va a utilizar una concentración diferente, asegúrese de hacer clic en el botón Calcular composición de disolvente después de editarla.
- Seleccione las condiciones y la configuración de la simulación.
- Seleccione CHARMM36m como la opción FF; Se usa comúnmente para simulaciones de lípidos y proteínas, pero el usuario puede seleccionar otras opciones discutidas en la introducción.
- Seleccione GROMACS como motor MD para obtener archivos de entrada de muestra en el formato correspondiente.
NOTA: GROMACS se recomienda para nuevos usuarios porque tiene múltiples recursos en línea, tutoriales y foros de soporte. El usuario puede seleccionar entre varios motores MD para explorar opciones en términos de rendimiento de simulación y sintaxis de código. - Seleccione el conjunto Partícula-Presión-Temperatura Constante (NPT), con diferencia el conjunto dinámico más utilizado en la simulación de bicapas lipídicas.
- Establezca la temperatura y la presión en Kelvin y bares como 303 K y 1 bar, respectivamente. Es típico establecer la temperatura entre 298 K y 310 K para el estudio de procesos biológicos para asegurar una bicapa en estado de desorden líquido.
NOTA: La temperatura depende de las condiciones del proceso a simular y puede modificarse según sea necesario. En función de las especies lipídicas del modelo, establezca la temperatura para que esté por encima de la temperatura de transición de los componentes lipídicos puros antes de ejecutar la simulación.
- Descargue los archivos resultantes y transfiéralos al clúster de equipos.
- Visualice el sistema final en un software de su elección, como VMD o PyMol, e inspeccione la configuración adecuada.
NOTA: Es bueno verificar, por ejemplo, que haya suficiente agua alrededor de la membrana para que los lípidos no interactúen con los átomos de la imagen durante la simulación, y la configuración adecuada de la valva (una bicapa sin espacio ni agua en el medio).
- Visualice el sistema final en un software de su elección, como VMD o PyMol, e inspeccione la configuración adecuada.
2. Ejecución de simulaciones de MD
- Cargue y descomprima los archivos de C-GUI en su clúster informático. Vaya al directorio Gromacs . Cree un script de envío de relajación.
- Siga las directrices del clúster para el formato de un script de envío.
- Copie los comandos enumerados hasta justo encima del comentario # Production en el archivo README en el script de envío.
NOTA: Este valor predeterminado de C-GUI es un bucle que ejecuta una relajación de 6 pasos del sistema. Si se desea un protocolo diferente y bien establecido, edítelo para leer las coordenadas que acaba de construir y descargar de C-GUI.
- Copie los comandos enumerados hasta justo encima del comentario # Production en el archivo README en el script de envío.
- Envíe el script de relajación y confirme que se han descargado todos los archivos de salida para todos los pasos antes de pasar a la ejecución de producción. Al finalizar, compruebe los siguientes archivos de salida de GROMACS, generados durante la ejecución de 6 pasos: *..log, *.tpr, *.gro, *.edr, *.trr / *.xtc
- Cree un script de ejecución de producción.
- Utilice uno de los ejemplos de comandos gmx grompp y gmx mdrun de cualquiera de los pasos de relajación como plantilla.
- Antes de usar el script, asegúrese de crear un archivo *.mdp que contenga opciones de simulación similares a las del archivo step7_production.mdp proporcionado.
NOTA: Las opciones predeterminadas proporcionadas son estándar para las simulaciones de membrana; Las indicaciones se enumeran en NM y el tiempo se da en picosegundos o número de pasos (picosegundos / paso de tiempo de integración). Actualice los nsteps para que se ejecuten hasta la longitud de simulación deseada (que es igual a dt * nsteps) y nst[x,v,f]out para actualizar la frecuencia de guardado de datos en el número de pasos de integración. Para el caso práctico, establezca los nsteps en 250 000 000 para una longitud de simulación de 500 ns (tiempo de simulación/paso de integración = 500 000 ps / 0,002 ps) y nst[x,v,f]out en 50 000 para guardar datos cada 100 ps
- Antes de ejecutar la simulación real, ejecute estudios de referencia para determinar el mejor uso de los recursos.
- Ejecute el sistema durante 1-2 ns utilizando un número diferente de nodos informáticos.
NOTA: El estudio de caso de ER se presentó en el clúster de computación de alto rendimiento55 del Centro de Investigación Computacional (CCR) de la UB para 2 ns, donde se probó el rendimiento de 1 a 10 nodos. - Compare el rendimiento en ns/día para cada configuración para determinar los recursos óptimos para la ejecución. Es una práctica común seleccionar el número de nodos que dan como resultado un 75%-80% del rendimiento máximo.
- Ejecute el sistema durante 1-2 ns utilizando un número diferente de nodos informáticos.
- Ejecute la ejecución de producción.
- Ejecute cada sistema por triplicado para garantizar la reproducibilidad y realice análisis estadísticos de los datos.
- Amplíe la trayectoria en función de los puntos de referencia si se agota el tiempo de cola permitido para el envío en el clúster informático. Utilice los comandos gmx convert-tpr y, a continuación, gmx mdrun para continuar con la recopilación de trayectorias.
NOTA: Las opciones se describen en la documentación de GROMACS en línea (https://manual.gromacs.org/). - Para un sistema de membrana solamente, examine si el sistema ha alcanzado el equilibrio calculando el área por lípido a lo largo del tiempo. Si no es así, amplíe la trayectoria de simulación.
3. Analizar la trayectoria
- Visualice el sistema antes de ejecutar el análisis para determinar las moléculas de interés y la parte de la trayectoria que se pretende caracterizar.
- Comprima los archivos de trayectoria sin procesar (*.trr) cambiando el formato de archivo a *.xtc y/o omitiendo fotogramas para reducir el tamaño del archivo y facilitar una transferencia más eficiente a la estación local para su visualización y análisis.
NOTA: Para sistemas de membranas grandes, se puede optar por eliminar el agua de la trayectoria para reducir aún más el tamaño de la lima. Esto se puede hacer con archivos de índice en GROMACS, scripts TCL en VMD o bibliotecas de Python como MDAnalysis y MDTraj. - Realice los análisis elegidos durante la parte equilibrada de la trayectoria, según lo determinado a partir de la serie temporal de área por lípido.
NOTA: Consulte la discusión para obtener más detalles sobre los análisis típicos de membranas y cómo ejecutarlos.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para ilustrar el uso del protocolo y los resultados que se pueden obtener, se discute un estudio comparativo de modelos de membrana para el retículo endoplásmico (RE). Los dos modelos de este estudio fueron: (i) el modelo PI, que contiene las cuatro principales especies lipídicas encontradas en el RE, y (ii) el modelo PI-PS, que añadió las especies lipídicas aniónicas de fosfatidilserina (PS). Estos modelos fueron utilizados posteriormente en un estudio de una proteína viral y cómo interactúa con la membrana, e...
Access restricted. Please log in or start a trial to view this content.
Discusión
Las técnicas experimentales permiten visualizar biomoléculas de alta resolución utilizando criomicroscopía electrónica (crio-EM)58, técnicas de fluorescencia y microscopía de fuerza atómica (AFM)59. Sin embargo, es difícil captar la interacción y la dinámica de las interacciones moleculares que subyacen a las vías biológicas, la patogénesis de la enfermedad y la administración terapéutica a nivel atómico o de aminoácidos. Aquí, se discutieron las capacida...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen intereses contrapuestos que revelar.
Agradecimientos
Los autores agradecen a Jinhui Li y Ricardo X. Ramírez por sus trayectorias de simulación y discusiones durante la redacción de este manuscrito. O.C. contó con el apoyo de la Beca Presidencial de la Universidad de Buffalo y la Iniciativa para Maximizar la Capacitación en Desarrollo Estudiantil del Instituto Nacional de Salud 1T32GM144920-01 otorgada a Margarita L. Dubocovich (PI).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
Referencias
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M. Comprehensive Biophysics. Edward, H. . E. gelman , Elsevier. 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis,, Martin Raff,, Keith Roberts,, Peter Walter, Molecular Biology of the Cell. , Garland Science. (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, Ü, Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Biobased Surfactants (Second Edition) eds. Douglas G, H. ayes, Daniel, K. Y., Solaiman,, Richard, D. , AOCS Press. 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41(2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264(2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. Biochemistry. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5(2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897(2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167(2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441(2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S. Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. Scott Shell, M. , Cambridge University Press. 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational 'microscopy' of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901(2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B. Biogenesis of Fatty Acids, Lipids and Membranes. Otto Geiger, , Springer International Publishing. 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068(2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130(2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. Using VMD. , http://csbmb.beckman.illinois.edu/BIOP586C/vmd-tutorial-2011.pdf (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. CCR Facility Description. , https://ubir.buffalo.edu/xmlui/handle/10477/79221 (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841(2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38(2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- Hepatitis C. , https://www.who.int/news-room/fact-sheets/detail/hepatitis-c (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957(2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. Biochemistry. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. obert T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621(2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados