Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis espacial de alta resolución de los niveles de expresión génica en tendones
En este artículo
Resumen
Este artículo describe cómo realizar un protocolo in situ optimizado para tendones. Este método analiza la preparación de tejidos, la permeabilización de la sección, el diseño de la sonda y los métodos de amplificación de señales.
Resumen
En los últimos años, se han desarrollado muchos protocolos para la transcriptómica de alta resolución en muchos campos diferentes de la medicina y la biología. Sin embargo, los tejidos ricos en matriz, y específicamente, los tendones, se quedaron atrás debido a su bajo número de células, baja cantidad de ARN por célula y alto contenido de matriz, lo que los hizo complicados de analizar. Una de las herramientas unicelulares más recientes e importantes es el análisis espacial de los niveles de expresión génica en los tendones. Estas herramientas espaciales de ARN tienen una importancia específicamente alta en los tendones para localizar células específicas de poblaciones nuevas y desconocidas, validar los resultados de secuenciación de ARN de una sola célula y agregar contexto histológico a los datos de secuenciación de ARN de una sola célula. Estos nuevos métodos permitirán el análisis de ARN en células con una sensibilidad excepcional y la detección de dianas de ARN de una sola molécula a nivel de una sola célula, lo que ayudará a caracterizar molecularmente los tendones y a promover la investigación de los tendones.
En este artículo de método, nos centraremos en los métodos disponibles para analizar los niveles de expresión génica espacial en secciones histológicas mediante el uso de nuevos ensayos de hibridación in situ para detectar ARN objetivo dentro de células intactas a niveles de una sola célula. En primer lugar, nos centraremos en cómo preparar el tejido tendinoso para los diferentes ensayos disponibles y cómo amplificar las señales específicas del objetivo sin ruido de fondo, pero con alta sensibilidad y alta especificidad. A continuación, se describirán los métodos específicos de permeabilización, los diferentes diseños de sondas y las estrategias de amplificación de señales disponibles en la actualidad. Estos métodos únicos de análisis de los niveles de transcripción de diferentes genes en resolución de una sola célula permitirán la identificación y caracterización de las células del tejido tendinoso en poblaciones jóvenes y envejecidas de varios modelos animales y tejidos tendinosos humanos. Este método también ayudará a analizar los niveles de expresión génica en otros tejidos ricos en matriz, como huesos, cartílagos y ligamentos.
Introducción
Los tendones son tejidos conectivos que permiten la transmisión de fuerza entre el músculo y el hueso1. Desde el punto de vista del desarrollo, los tenocitos axiales se derivan de células mesenquimales dentro del esclerótomo de los somitas2; los tendones de las extremidades derivan del mesodermo de la placa lateral; y los tendones craneales surgen del linaje de la cresta neural craneal 3,4. El tendón se puede caracterizar por la expresión del factor de transcripción5 de la escleraxia, aunque varios marcadores también juegan un papel clave en el desarrollo del tendón, incluyendo la tenomodulina, el mohawk y la respuesta de crecimiento temprano 1/2 6,7,8,9.
A pesar de los pocos marcadores conocidos del tendón, en general, una caracterización más profunda sigue siendo un desafío porque el tendón contiene células que se extienden a través de un gradiente de propiedades biomecánicas. Desde la unión miotendinosa, la parte media del cuerpo del tendón y la entesis más calcificada, las células del tendón residen en matrices extracelulares que varían en propiedades de tracción. Dado que el tendón debe soportar la tensión de tracción impuesta por la diferencia de resistencia mecánica entre el tejido blando y el duro, la organización espacial de las células del tendón es particularmente importante para su función. Sin embargo, se sabe poco sobre estas subpoblaciones de tendones.
Se pueden utilizar muchas herramientas transcriptómicas espaciales de alta resolución para comenzar a dilucidar subpoblaciones celulares, incluidas, entre otras, la secuenciación de ARN de una sola célula o la hibridación in situ . Sin embargo, aunque estos ensayos de perfiles espaciales ayudan a descubrir la expresión de ARN en el tejido después de la microdisección o el seccionamiento, estos métodos pueden ser difíciles cuando se realizan en el tejido del tendón. Los tendones son tejidos ricos en matriz compuestos por casi el 86% de colágeno por masa seca10, lo que dificulta la extracción de las células para su secuenciación. Debido a las complicaciones en el aislamiento de las células de la matriz, la naturaleza hipocelular del tendón11 y el recuento relativamente bajo de ARN, el tendón es un tejido difícil de analizar.
En este artículo, presentamos un método para optimizar nuevos ensayos de hibridación in situ para aprovecharlos para los tendones al proporcionar métodos de preparación de tejidos, permeabilización y diseño de sondas. Junto con las tecnologías de secuenciación existentes, esto puede ayudar a los investigadores a caracterizar espacialmente las subpoblaciones de tendones en tendones en desarrollo, adultos o lesionados con una mayor sensibilidad y especificidad del ensayo.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos los experimentos con animales se realizaron de acuerdo con el Comité Institucional de Cuidado y Uso de Animales (IACUC) y las pautas de la AAALAC. Los experimentos se realizaron bajo el protocolo aprobado #2013N000062 en el Hospital General de Massachusetts. En este estudio se utilizaron ratones C57BL/J6 (5 semanas de edad y P0). Consulte la Tabla de materiales para obtener detalles relacionados con todos los materiales, reactivos e instrumentos utilizados en este protocolo.
1. Preparación y fijación de la muestra
- Eutanasia de ratones en una cámara de CO2 seguida de luxación cervical como método secundario de eutanasia. Luego, use unas tijeras para cortar las extremidades traseras del ratón a lo largo de la articulación de la cadera12. En un vial de centelleo, sumerja la extremidad posterior del ratón en una solución suficiente de paraformaldehído (PFA) al 4% para cubrir completamente la muestra y fije el tejido durante 24 horas.
NOTA: En lugar de 4% de PFA, también se puede usar formalina al 10% para la fijación. Después de la fijación, la muestra puede almacenarse en solución salina tamponada con fosfato (PBS) hasta su posterior procesamiento. Al fijar toda la extremidad posterior, podemos preservar la tensión en el tendón y tenemos la oportunidad de examinar la entesis, el cuerpo medio del tendón o la unión miotendinosa. - Después de retirar el PFA del vial de centelleo, lave la muestra durante 3 x 10 minutos con 1x PBS añadiendo suficiente PBS para cubrir la muestra. A continuación, sumergir las extremidades traseras en EDTA 0,5 M a 4 °C con agitación durante 1-2 semanas, sustituyendo por EDTA fresco cada 2-3 días.
- Después de retirar el EDTA, lavar la muestra durante 3 x 10 min con 1x PBS, deshidratar las muestras en EtOH al 70% e incrustarlas en parafina en la orientación deseada. Utilice técnicas convencionales de corte de FFPE para seccionar el tejido con un grosor de 3 μm y transferir las secciones a un portaobjetos de microscopio tratado y cargado (ver Tabla de Materiales)13.
NOTA: Las secciones se pueden almacenar a 4 °C antes de continuar con los siguientes pasos. - Desparafinación
- Hornee los portaobjetos en una placa calefactora a 60 °C durante 1 h y luego deje que los portaobjetos alcancen la temperatura ambiente (RT). Para quitar la cera de parafina de los portaobjetos, colóquelos en una rejilla para portaobjetos y bájelos en un plato para tinturas lleno de xileno. Repita en xileno fresco durante 3 x 5 min.
PRECAUCIÓN: El xileno es peligroso; Manéjelo con cuidado. - Para rehidratar las secciones, sumerja los portaobjetos en lo siguiente durante 5 minutos por lavado, todo en RT: lavar 2 veces en 100% EtOH, 1 vez en 75% de EtOH diluido en dH2O, 1 vez en 50% de EtOH diluido en PBS, 1 vez en 25% de EtOH diluido en PBS y 2 veces en PBS.
NOTA: Todos los pasos de rehidratación se realizan con los portaobjetos en una rejilla de portaobjetos y con las diversas soluciones de EtOH en platos de tinción.
- Hornee los portaobjetos en una placa calefactora a 60 °C durante 1 h y luego deje que los portaobjetos alcancen la temperatura ambiente (RT). Para quitar la cera de parafina de los portaobjetos, colóquelos en una rejilla para portaobjetos y bájelos en un plato para tinturas lleno de xileno. Repita en xileno fresco durante 3 x 5 min.
2. Adaptación del protocolo 14 de RNAscope (ISH comercializado)
- Preparación de soluciones y sondas
- Prepare la solución de TEG para el pretratamiento de muestras seccionadas combinando 25 mM de Tris-HCl a pH 8, 10 mM de EDTA y 50 mM de glucosa. Hacer un volumen suficiente para todas las muestras que se van a tratar o preparar el exceso; filtrar-esterilizar y almacenar en RT; Mezcle la solución correctamente antes de usarla.
- Prepare suficiente tampón de lavado 1x (consulte la Tabla de materiales) para todos los lavados y guárdelo en RT.
- En un baño de agua o incubadora a 40 °C, caliente las sondas de interés durante 10 minutos. A continuación, prepare las sondas de acuerdo con el protocolo ISH14.
- Pretratamiento de muestras
- En lugar de utilizar la solución de pretratamiento recomendada, sumerja los portaobjetos en una placa de tinción que contenga tampón TEG durante 4 h a 60 °C.
NOTA: La incubación del tampón TEG puede prolongarse hasta 6 h si el tejido es especialmente denso o si la recuperación del antígeno del motivo de unión al ARN de interés es un reto. En ese caso, se puede añadir un punto de parada, secar los portaobjetos y guardarlos en una nevera a 4 °C durante la noche. - Retire las muestras del tampón y déjelas secar. A continuación, extraiga las muestras con un rotulador de barrera hidrofóbico, sumérjalas en otra placa de tinción que contenga proteasa IV e incubelas en un horno durante 45 min a 40 °C.
NOTA: Recomendamos la proteasa IV para ratones adultos. Si se utilizan ratones más jóvenes, como P0, se recomienda utilizar una enzima más suave, como la proteasa III, durante 30 minutos. Al solucionar problemas para diferentes edades, observe la integridad del tendón y verifique si hay degradación. Si el tejido se está disociando o degradando, disminuya el tiempo de incubación o cambie la proteasa utilizada. - Lavar las muestras de nuevo en tampón TEG durante 30 min y luego hibridar las sondas de acuerdo con el protocolo ISH14.
- Añada 50-100 μL de reactivo de montaje sobre la muestra. Coloque un cubreobjetos encima de las muestras para la microscopía. Al tomar imágenes, utilice un aumento alto (objetivo de 40x o superior) para ver la señal.
- En lugar de utilizar la solución de pretratamiento recomendada, sumerja los portaobjetos en una placa de tinción que contenga tampón TEG durante 4 h a 60 °C.
3. Adaptación del protocolo HCR ISH15
- Postfijación
- Prepare PFA al 4% (en PBS), solución de PBT (0,1% Tween-20 en PBS) y proteinasa K en PBS (concentración de stock de 10 mg/mL, concentración final de 5 μg/mL).
- Para comenzar el primer paso del sufijo, sumerja los portaobjetos en un plato de tinción que contenga un 4% de PFA durante 5 minutos; luego, retire la solución de PFA al 4% y guárdela para usarla en pasos posteriores. Enjuague los portaobjetos durante 2 x 5 minutos en solución de PBT y retire la solución de PBT. Coloque los portaobjetos en una placa de tinción que contenga una solución de proteinasa K (5 μg/mL) durante 5 min y enjuáguelos en la solución de PBT 2 x 5 min.
- Comience el segundo paso del sufijo y sumerja los portaobjetos en el PFA al 4% durante 5 minutos.
NOTA: Este es el mismo 4% de PFA ahorrado del paso 3.1.2. - Enjuague los portaobjetos durante 3 x 5 minutos en la solución de PBT.
NOTA: Todos los pasos de postfijación se realizan con los portaobjetos en una gradilla de portaobjetos y en platos de tinción que contienen las soluciones respectivas.
- Acetilación
- Prepare la solución de acetilación que contiene 625 μL de anhídrido acético, 3,3 mL de tampón de trietanolamina (TEA) 1 M y 246 mL de ddH2O. Asegúrese de mezclar bien y usar inmediatamente después de la preparación.
- Coloque los portaobjetos en la solución de acetilación durante 10 minutos y luego enjuague 3 x 5 min en solución de PBT. Enjuague los portaobjetos en ddH2O y déjelos secar al aire durante 30 min. A continuación, dibuja alrededor de las muestras con un rotulador de barrera hidrofóbico.
NOTA: Todos los pasos de acetilación se realizan con los portaobjetos en una rejilla de portaobjetos y en platos de tinción que contienen las soluciones respectivas
- Hibridación
- Precalentar el tampón de hibridación a 37 °C y precalentar una cámara humidificada a 37 °C.
PRECAUCIÓN: El tampón contiene formamida, un material peligroso. Si los portaobjetos no están lo suficientemente secos, seque los bordes con una toallita de laboratorio para eliminar el exceso de dH2O. - Prepare las soluciones de sonda añadiendo 0,4 pmol de cada mezcla de sonda a 100 μL de tampón de hibridación.
NOTA: Si utiliza imágenes de dHCR, utilice una concentración más alta de sonda para mejorar la eficiencia de la hibridación de la sonda. - Prepare 4 L de tampón de citrato de sodio y cloruro de sodio (20x SSC) combinando 3 M de NaCl (701,1 g de NaCl en un volumen final de 4 L) y 0,3 M de Na,3CH,6H5O7•2H2O (352,8 g) y luego ajuste el pH final a 7 agregando HCl o 10 N NaOH. Agregue suficiente H2O para alcanzar 4 L. Prepare 5x SSCT diluyendo 20x SSC a 5x y agregando suficiente 10% Tween 20 para que sea el 0.1% del volumen total.
- Agregue 200 μL de tampón de hibridación a la muestra, coloque los portaobjetos en una cámara humidificada y deje que los portaobjetos se incuben durante 10 minutos. Retire la solución de hibridación y drene el exceso de tampón en el portaobjetos secando los bordes con una toallita de laboratorio.
- Añada 100 μL de la solución de sonda preparada sobre la muestra y coloque un cubreobjetos sobre la muestra. Incubar en un horno durante la noche o durante unas 12-16 h en una cámara humidificada con una solución que contenga 5x SSC y 50% de formamida. Ajuste la temperatura a 37 °C.
NOTA: Dado que la incubación es durante la noche, la adición del cubreobjetos es para minimizar la evaporación. - Deje una alícuota de 5 veces la solución de SSCT en un baño de agua a 37 °C y una segunda alícuota en RT. Anticipe los volúmenes de estas alícuotas necesarios en función del número de muestras y el número de lavados en el siguiente paso.
- Precalentar el tampón de hibridación a 37 °C y precalentar una cámara humidificada a 37 °C.
- Lavado
- Usando la solución SSCT 5x previamente precalentada, prepare soluciones de 75% tampón de lavado/25% 5x SSCT, 50% tampón de lavado/50% 5x SSCT y 25% tampón de lavado/75% 5x SSCT.
- Para eliminar el exceso de sondas, sumerja los portaobjetos en serie durante 15 minutos por lavado a 37 °C en 75% tampón de lavado/25% 5x SSCT, 50% tampón de lavado/50% 5x SSCT y 25% tampón de lavado/75% 5x SSCT. Deje que los cubreobjetos floten fuera de las muestras.
- Incubar los portaobjetos 2 x 15 min con 100% 5x SSCT y luego sumergirlos en 5x SSCT durante 5 min en RT.
NOTA: Los pasos de lavado se pueden realizar con los portaobjetos en una rejilla de portaobjetos y en platos de tinción que contengan las soluciones respectivas o con las soluciones añadidas como gotas.
- Amplificación
- Prepare 6 pmol de horquilla h1 y 6 pmol de horquilla h2 enfriando a presión 2 μL de un caldo de 3 μM (calentar a 95 °C durante 90 s y enfriar a RT). Protege las horquillas de la luz durante este proceso.
NOTA: Las horquillas HCR h1 y h2 se proporcionan en un búfer de almacenamiento de horquilla y están listas para el enfriamiento rápido. Encaje las horquillas frías h1 y h2 en tubos separados. - Seque los portaobjetos secando sus bordes con una toallita de laboratorio. A continuación, añada 200 μL de tampón de amplificación encima de la muestra y colóquela en una cámara humidificada durante 30 min en RT. Prepare la mezcla de horquillas añadiendo horquillas h1 y horquillas h2 enfriadas a presión hasta 100 μL de tampón de amplificación en RT.
- Retire el tampón de amplificación y drene el exceso de líquido en el portaobjetos secando los bordes con una toallita de laboratorio. Agregue 100 μL de la mezcla de horquilla previamente preparada y coloque el parafilm encima de la muestra. Incube los portaobjetos durante un mínimo de 4 h o toda la noche en una cámara oscura y humidificada en RT.
- Para eliminar el exceso de horquillas, sumerja los portaobjetos en 5x SSCT en una placa de tinción e incube a RT durante 30 min. Repita el lavado en SSCT fresco 5x durante 30 minutos y una tercera vez durante 5 minutos.
- Seque los portaobjetos secando los bordes con una toallita de laboratorio y añada 50-100 μL de reactivo de montaje encima de la muestra. Coloque un cubreobjetos encima de las muestras para la microscopía. Al tomar imágenes, utilice un aumento alto (objetivo de 40x o superior) para ver la señal.
- Prepare 6 pmol de horquilla h1 y 6 pmol de horquilla h2 enfriando a presión 2 μL de un caldo de 3 μM (calentar a 95 °C durante 90 s y enfriar a RT). Protege las horquillas de la luz durante este proceso.
Access restricted. Please log in or start a trial to view this content.
Resultados
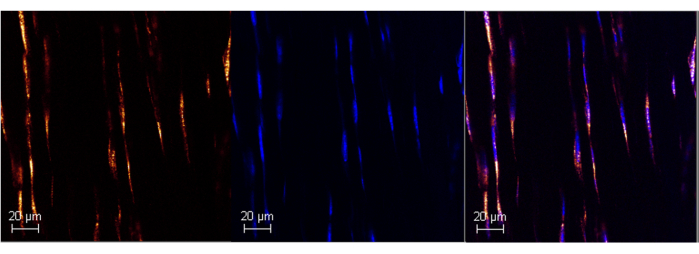
Figura 1: Expresión de ARN de Poly A en el tendón de Aquiles de ratón adulto utilizando RNAScope. Imagen representativa del etiquetado exitoso de Poly A en el tendón de Aquiles del ratón (panel izquierdo) utilizando el ensayo ISH comercializado. La colocalización con DAPI confirma la especificidad de la sonda...
Access restricted. Please log in or start a trial to view this content.
Discusión
En este artículo, describimos las modificaciones realizadas para aprovechar las herramientas de ISH existentes, de modo que puedan usarse en el tejido tendinoso con un alto grado de especificidad y sensibilidad. Dado que el tendón es un tejido altamente denso en matriz, a menudo se deben realizar ajustes en el protocolo para lograr grados similares de penetración y especificidad de la sonda. Estos métodos específicos de permeabilización y estrategias de amplificación de señales d...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Los autores agradecen a Jenna Galloway y a los miembros de Galloway Lab por su apoyo y aliento en el desarrollo y la resolución de problemas de estos protocolos.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 M triethanolamine buffer | |||
| 10% Formalin solution | |||
| 10% Tween-20 | |||
| 20x Saline Sodium Citrate buffer | |||
| 4% PFA | |||
| ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 | ACD | 323100 | |
| Acetic Anhydride | |||
| Axio Imager Microscope | ZEISS | ||
| C57BL/J6 mice | JAX ID: 000664 | ||
| Coverslips | Fisher | 12-541-042 | |
| ddH2O | |||
| ETDA | Thermofisher | AM9262 | |
| EtOH | |||
| Glucose | VWR Chemicals BDH | BDH9230-500G | |
| HCR RNA-FISH Bundle | Molecular Instruments Inc. | ||
| HybEZ II Hybridization System | ACD | ||
| Immedge Barrier Pen | Vector Laboratories | H4000 | |
| Leica SPE Confocal Microscope | Leica | ||
| Parafilm | Fisher | ||
| Phosphate-buffered saline (PBS, 1x) | Invitrogen | AM9625 | Dilute 10x PBS in milli-Q water to get 1x solution |
| Protease IV | |||
| Proteinase K | Roche | 3115836001 | |
| RNAscope H2O2 and Protease Reagents | ACD | PN 322381 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V3 |
| RNAscope Multiplex Fluorescent Detection Kit | ACD | PN 323110 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 |
| RNAscope Target Retrieval reagents | ACD | 322000 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V4 |
| RNAscope Wash Buffer | ACD | PN 310091 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V5 |
| RNAscope Probe Diluent | ACD | 300041 | |
| Slide holder | StatLab | 4465A | |
| Staining Dish with Lid | StatLab | LWS20WH | |
| Superfrost Plus Microscope slides | Fisher | 1255015 | treated, charged slides |
| Tris-HCl | |||
| Xylene | Sigma-Aldrich | 534056-4L |
Referencias
- Sharma, P., Maffulli, N. Tendon injury and tendinopathy: healing and repair. Journal of Bone and Joint Surgery. 87 (1), 187-202 (2005).
- Brent, A. E., Schweitzer, R., Tabin, C. J. A somitic compartment of tendon progenitors. Cell. 113 (2), 235-248 (2003).
- Noden, D. M. The role of the neural crest in patterning of avian cranial skeletal, connective, and muscle tissues. Developmental Biology. 96 (1), 144-165 (1983).
- Chen, J. W., Galloway, J. L. The development of zebrafish tendon and ligament progenitors. Development. 141 (10), 2035-2045 (2014).
- Schweitzer, R., et al. Analysis of the tendon cell fate using Scleraxis, a specific marker for tendons and ligaments. Development. 128 (19), 3855-3866 (2001).
- Tsai, S., Nödl, M., Galloway, J. Bringing tendon biology to heel: Leveraging mechanisms of tendon development, healing, and regeneration to advance therapeutic strategies. Developmental Dynamics. 250 (3), 393-413 (2021).
- Kannus, P., et al. Location and distribution of non-collagenous matrix proteins in musculoskeletal tissues of rat. The Histochemical Journal. 30 (11), 799-810 (1998).
- Kannus, P. Structure of the tendon connective tissue. Scandinavian Journal of Medicine & Science in Sports. 10 (6), 312-320 (2000).
- Thorpe, C. T., Birch, H. L., Clegg, P. D., Screen, H. R. C. The role of the non-collagenous matrix in tendon function. International Journal of Experimental Pathology. 94 (4), 248-259 (2013).
- Lin, T. W., Cardenas, L., Soslowsky, L. J. Biomechanics of tendon injury and repair. Journal of Biomechanics. 37 (6), 865-877 (2004).
- Grinstein, M., et al. A distinct transition from cell growth to physiological homeostasis in the tendon. eLife. 8, e48689(2019).
- Villaseñor, S., Grinstein, M. Two-photon microscopy for the study of tendons. Journal of Visualized Experiments. , e65853(2023).
- Qin, C., et al. The cutting and floating method for paraffin-embedded tissue for sectioning. Journal of Visualized Experiments. (139), e58288(2018).
- Wang, F., et al. RNAscope®: A Novel In Situ RNA Analysis Platform for Formalin-Fixed Paraffin-Embedded Tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Choi, H. M. T., et al. Third-generation situ hybridization chain reaction: multiplexed, quantitative, sensitive, versatile, robust. Development. 145 (12), 165753(2018).
- Jackson, D. J., Herlitze, I., Hohagen, J. A whole mount in situ hybridization method for the gastropod mollusc Lymnaea stagnalis. Journal of Visualized Experiments. (109), e53968(2016).
- Young, A. P., Jackson, D. J., Wyeth, R. C. A technical review and guide to RNA fluorescence in situ hybridization. PeerJ. 8, e8806(2020).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados