Automatización del cribado por criomicroscopía electrónica en múltiples redes con Smart Leginon
* Estos autores han contribuido por igual
En este artículo
Resumen
El cribado multirred de criomicroscopía electrónica (crioEM) suele ser un proceso tedioso que exige horas de atención. Este protocolo muestra cómo configurar una colección estándar de Leginon y Smart Leginon Autoscreen para automatizar este proceso. Este protocolo se puede aplicar a la mayoría de las rejillas de lámina con agujeros crioEM.
Resumen
Los avances en las técnicas de criomicroscopía electrónica (crioEM) en la última década han permitido a los biólogos estructurales resolver de forma rutinaria complejos de proteínas macromoleculares con una resolución casi atómica. El flujo de trabajo general de toda la línea de producción de cryoEM implica iterar entre la preparación de muestras, la preparación de la cuadrícula de cryoEM y el cribado de muestras/cuadrículas antes de pasar a la recopilación de datos de alta resolución. La iteración entre la preparación de la muestra/rejilla y el cribado suele ser un cuello de botella importante para los investigadores, ya que cada experimento iterativo debe optimizar la concentración de la muestra, las condiciones del tampón, el material de la rejilla, el tamaño del agujero de la rejilla, el grosor del hielo y el comportamiento de las partículas de proteínas en el hielo, entre otras variables. Además, una vez que estas variables se determinan satisfactoriamente, las cuadrículas preparadas en condiciones idénticas varían ampliamente en cuanto a si están listas para la recopilación de datos, por lo que se recomiendan sesiones de selección adicionales antes de seleccionar las cuadrículas óptimas para la recopilación de datos de alta resolución. Este proceso de preparación y cribado de muestras/rejillas a menudo consume varias docenas de rejillas y días de tiempo del operador en el microscopio. Además, el proceso de selección se limita a la disponibilidad del operador/microscopio y a la accesibilidad del microscopio. Aquí, demostramos cómo usar Leginon y Smart Leginon Autoscreen para automatizar la mayor parte del cribado de rejilla crioEM. Autoscreen combina el aprendizaje automático, los algoritmos de visión artificial y los algoritmos de manejo del microscopio para eliminar la necesidad de una intervención manual constante del operador. Autoscreen puede cargar e idear cuadrículas de forma autónoma con imágenes multiescala mediante un sistema automatizado de casetes de intercambio de muestras, lo que da como resultado una trama de cuadrícula desatendida para un casete completo. Como resultado, el tiempo del operador para el cribado de 12 rejillas puede reducirse a ~10 min con Autoscreen en comparación con ~6 h utilizando métodos anteriores que se ven obstaculizados por su incapacidad para tener en cuenta la alta variabilidad entre rejillas. Este protocolo primero presenta la configuración y la funcionalidad básicas de Leginon y, a continuación, demuestra la funcionalidad de Autoscreen paso a paso, desde la creación de una sesión de plantilla hasta el final de una sesión de cribado automatizada de 12 cuadrículas.
Introducción
La criomicroscopía electrónica de una sola partícula (crioEM) permite la determinación de la estructura de resolución casi atómica de complejos macromoleculares purificados. Un experimento crioEM de una sola partícula solo requiere una o dos cuadrículas bien elegidas seleccionadas de un conjunto mucho más grande de cuadrículas con diferentes condiciones de muestra y cuadrícula. El cribado microscópico para examinar estas rejillas implica la obtención de imágenes de cada rejilla con varios aumentos para determinar qué rejilla satisface la mayoría de los requisitos clave para la recopilación de datos de alta resolución, incluido el espesor del hielo, las áreas suficientes para la recopilación completa de datos, la pureza de las proteínas, la concentración de proteínas, la estabilidad de las proteínas y los problemas mínimos de orientación preferida1. La optimización de estos requisitos clave a menudo implica la retroalimentación entre el cribado al microscopio y las condiciones de preparación, como la producción de proteínas, la selección de tampones, los detergentes potenciales y el tipo de rejilla 2,3,4 (Figura 1). El cribado de rejilla convencional se realiza de forma manual o semimanual con software como Leginon5, SerialEM6 y EPU7. El cribado convencional requiere que el operador del microscopio pase horas frente al microscopio para cribar varias rejillas, lo que crea un cuello de botella significativo en el flujo de trabajo de una sola partícula de alta resolución al ocupar al operario con operaciones de memoria en lugar de la optimización de la muestra/rejilla.
Anteriormente, se han introducido Smart Leginon Autoscreen y el software de aprendizaje automático subyacente, Ptolemy, y se han descrito sus métodos y algoritmos subyacentes, junto con ejemplos 8,9. Varios otros paquetes de software son capaces o funcionan para el cribado multirredtotalmente automatizado 10, incluidos SmartScope11, Smart EPU12 y CryoRL13,14. Para abordar el cuello de botella de la detección, Smart Leginon permite al usuario configurar primero los parámetros de cribado en una sesión de microscopio de plantilla y, a continuación, utilizar los parámetros de esa sesión de plantilla como plantilla para cribar el casete completo de rejillas en el cargador automático del microscopio. Se elimina todo el trabajo manual durante el cribado de casetes, lo que permite que el bucle de retroalimentación de optimización proceda de manera significativamente más eficiente.
En este protocolo, se describe el flujo de trabajo completo de Smart Leginon Autoscreen para que el lector pueda realizar un cribado crioelectromagnético multirrejilla totalmente automatizado de forma independiente. Para aquellos que son nuevos en Leginon, la primera sección del protocolo describe el uso convencional de Leginon. Este conocimiento se compone de varios años de experiencia en varios microscopios de cargador automático, que luego se desarrolla en la siguiente sección Smart Leginon del protocolo. Se pueden encontrar videos tutoriales adicionales en https://memc.nysbc.org.
Protocolo
Para seguir este protocolo, representado en la Figura 2, Leginon 3.6+ debe instalarse en la computadora del microscopio y en una estación de trabajo Linux adicional, y Ptolemy debe instalarse en la estación de trabajo Linux. Este protocolo se ha desarrollado a lo largo de varios años utilizando microscopios Glacios y Krios de Thermo Fisher Scientific (TFS). Este protocolo asume que el lector ya ha configurado Leginon, Appion15, la base de datos asociada, las calibraciones del microscopio, ha realizado alineaciones directas en el microscopio y ha configurado dos aplicaciones Leginon: una para la recolección estándar de una sola partícula y otra para la recolección de una sola partícula con Ptolomeo. La información para la creación de Leginon está disponible aquí: https://emg.nysbc.org/redmine/projects/leginon/wiki/Leginon_Manual. La información para establecer a Ptolomeo dentro de Leginon está disponible aquí: https://emg.nysbc.org/redmine/projects/leginon/wiki/Multi-grid_autoscreening. Descarga Leginon de http://leginon.org y Ptolomeo de https://github.com/SMLC-NYSBC/ptolemy. Leginon está licenciado bajo la Licencia Apache, Versión 2.0, y Ptolemy está licenciado bajo CC BY-NC 4.0.
1. Uso de Leginon
- Iniciar Leginon
- En el microscopio de la computadora con Windows, cierre todos los clientes de Leginon y vuelva a abrirlos. En la estación de trabajo Linux, abra una ventana de terminal y escriba start-leginon.py o el alias apropiado del sistema para iniciar Leginon.
- En la nueva ventana Configuración de Leginon , seleccione Crear una nueva sesión y haga clic en Siguiente.
- Seleccione el proyecto de la lista desplegable y haga clic en Siguiente.
- Deje el nombre como está, seleccione el soporte correcto para la configuración del microscopio y haga clic en Siguiente.
- Para la descripción, introduzca la información relevante, como el nombre del microscopio, la descripción de la cuadrícula/muestra y la descripción del experimento y, a continuación, haga clic en Siguiente.
- Para el directorio de imágenes, asegúrese de que el sistema de archivos adecuado esté seleccionado y que la ruta completa sea adecuada para guardar imágenes y, a continuación, haga clic en Siguiente.
- En Conectar a clientes, haga clic en Editar. En el menú desplegable, seleccione todas las computadoras que deben conectarse y haga clic en el botón + para cada una, luego haga clic en Aceptar y Siguiente.
- Introduzca el tamaño de apertura C2 correcto y haga clic en Siguiente. Este valor se puede encontrar en la pestaña Aperturas del software TFS TUI.
- Interfaz de Leginon
- Seleccione Aplicación en la barra de herramientas y haga clic en Ejecutar.
- Seleccione la aplicación adecuada en el menú desplegable (haga clic en Mostrar todo si es necesario). Establezca main en el equipo Leginon y scope y camera en el equipo respectivo y, a continuación, haga clic en Ejecutar. El lado izquierdo de la ventana principal de Leginon se llenará con nodos.
NOTA: El panel izquierdo muestra todos los nodos de Leginon. Los nodos verdes del icono de la cámara son las imágenes que se guardarán: Cuadrícula, Cuadrado, Agujero y Exposición. Los nodos con el signo de destino son imágenes de menor aumento para apuntar a las imágenes de mayor aumento. Los nodos de cámara púrpura son los nodos que están programados para encontrar la altura z eucéntrica y el enfoque eucéntrico. Además, hay nodos para alinear el pico de pérdida cero, monitorear el ciclo de búfer, monitorear el llenado de nitrógeno líquido, recopilar imágenes de corrección de ganancia, calcular el espesor del hielo (IceT) y navegar por la cuadrícula a través de diferentes aumentos utilizando el desplazamiento de la etapa y la imagen. - Administrador de ajustes preestablecidos
- Haga clic en el nodo Presets_Manager. En ese nodo, haga clic en el icono inferior para importar ajustes preestablecidos o en el icono situado encima de ese para crear un nuevo ajuste preestablecido a partir del estado actual del microscopio.
- Si se hace clic en el icono inferior, se abrirá una ventana de importación de ajustes preestablecidos . Seleccione el TEM y la cámara digital correctos, luego haga clic en Buscar y seleccione la sesión más reciente con los ajustes preestablecidos deseados. Resalte todos los ajustes preestablecidos deseados y haga clic en Importar, luego haga clic en Listo.
NOTA: El nodo Administrador de ajustes preestablecidos ahora debería enumerar todos los ajustes preestablecidos importados y creados. Se recomienda tener ajustes preestablecidos para varios aumentos y enfoques, incluidos gr: Aumento de cuadrícula, sq: Aumento cuadrado, hln: Aumento de orificio, ventilador: Enfoque automático, fcn: Enfoque central, enn: Aumento de exposición (la 'n' final se refiere a la nanosonda). Los parámetros preestablecidos típicos para cada aumento se muestran en la Tabla 1, la Tabla 2 y la Tabla 3. Tenga en cuenta que este protocolo utiliza un tamaño de apertura C2 de 70 μm para el Glacios, 50 μm para el Krios con un Selectris X y un Falcon 4i, y 100 μm para el Krios con un BioQuantum con un K3.
- Navegación y altura excéntrica
- Para familiarizarse con el control del microscopio a través de Leginon y establecer la altura z de la cuadrícula, vaya al nodo Navegación, seleccione el ajuste preestablecido gr en la parte superior y haga clic en la flecha roja a la derecha para enviar los ajustes preestablecidos al microscopio. El microscopio debe actualizarse después de 1-2 s. Una vez actualizado, haga clic en el botón de la cámara a la derecha para adquirir una imagen.
- Con la herramienta de cursor, seleccione un cuadrado de cuadrícula para el lugar al que desea mover el escenario. Haga clic en el aumento cuadrado y luego en la flecha roja para enviarla al microscopio y haga clic en el botón de la cámara para adquirir una imagen.
- Vaya al nodo Z_Focus y haga clic en el botón Simular destino en la parte superior, cerca del centro de los botones. Mientras se recopilan imágenes para enfocar la inclinación del escenario, cambie a la vista de correlación y observe el pico para asegurarse de que está en la esquina de la imagen de correlación. Una vez finalizado el enfoque, asegúrese de que el escenario esté configurado a la altura z de la cuadrícula.
- Corrección de coma
NOTA: En esta subsección se asume que ya se han realizado alineaciones directas y que no se han realizado correcciones de coma.- Desplácese a un área de la cuadrícula que produzca anillos de Thon transparentes, como un sustrato de carbono.
NOTA: Se puede usar una rejilla transversal si la recolección se va a realizar en una rejilla dorada. - En la configuración de Beam_Tilt_Image, asegúrese de que el orden de los ajustes preestablecidos incluya solo fcn con cuatro direcciones de inclinación en un ángulo de 0,005 radianes.
- Haga clic en Simular destino para crear un cuadro de Zemlin. Haga clic en Tableau en el lado izquierdo de la ventana principal para ver el tableau.
- Corrija la coma comparando las transformadas de Fourier izquierda y derecha entre sí y las transformadas de Fourier superior e inferior entre sí. Si los pares de imágenes no son idénticos, primero haga clic en el icono del cursor a la derecha de los ajustes de imagen y, a continuación, haga clic ligeramente fuera del centro de la imagen de Tableau en la dirección de la diferencia y espere a que se recoja un nuevo conjunto de transformadas de Fourier. Repita hasta que las transformadas de Fourier sean idénticas.
NOTA: Cada clic en Tableau tarda varios segundos en completarse y no se deben realizar clics adicionales durante este tiempo.
- Desplácese a un área de la cuadrícula que produzca anillos de Thon transparentes, como un sustrato de carbono.
- Obtener referencias
NOTA: Omita esta sección si la cámara tiene referencias de hardware automáticas.- En el nodo Navegación, envíe un ajuste preestablecido de aumento bajo, como gr, y navegue hasta un área en la que no haya ninguna obstrucción a la trayectoria del haz.
- Confirme que la posición de la platina se encuentra en una ubicación no obstruida por la trayectoria del haz tomando una imagen de aumento medio utilizando el ajuste preestablecido sq o hln .
- Envíe el preajuste de enn de gran aumento al microscopio.
- En la configuración del nodo Corrección, seleccione la información adecuada del instrumento y configure la configuración de la cámara para que coincida con la configuración de la colección.
- Recopile una imagen de referencia oscura cerrando las válvulas de columna del microscopio y, a continuación, en el nodo Corrección, seleccione Oscuro y Ambos canales en los menús desplegables de la parte superior y haga clic en el botón Adquirir cámara a la derecha.
- Una vez completado, seleccione Bright en el menú desplegable y haga clic en Adquirir. Leginon abrirá las válvulas de columna automáticamente.
- Compruebe que la ganancia se ha recopilado correctamente seleccionando Corregido en el menú desplegable, haciendo clic en Adquirir y observando la imagen resultante.
- Imagen de referencia del espesor del hielo
- Si el microscopio tiene un filtro de energía, en la configuración del nodo IceT, marque Recopilar imagen de espesor de hielo, introduzca 395 para la ruta libre media y rellene el resto de los valores de la configuración.
- Si el microscopio no tiene un filtro de energía, en el nodo Navegación, envíe el ajuste preestablecido enn al microscopio y haga clic en Adquirir. Tome nota del valor medio del píxel en el lado izquierdo. En la configuración del nodo IceT, marque Calcular el espesor del hielo a partir de la dispersión limitada por apertura, introduzca 1055 para el coeficiente ALS y el valor medio de píxeles medido.
NOTA: Los valores 395 y 1055 se determinaron para el TFS Krios y Glacios, respectivamente, como se describió anteriormente16, y es posible que sea necesario recalibrarlos para diferentes configuraciones de microscopio.
- Calibración de la dosis por imagen
- En el Preset_Manager, seleccione el ajuste preestablecido enn y haga clic en el botón de la cámara (Adquirir imagen de dosis para el ajuste preestablecido seleccionado). Compruebe la dosis medida en la parte inferior. Si está cerca del valor esperado (normalmente entre 30 y 70), haga clic en SÍ.
- Alineaciones predefinidas
- En el Preset_Manager, compruebe todos los ajustes preestablecidos de gran aumento (enn, fcn y ventilador) para asegurarse de que el desplazamiento de la imagen y el desplazamiento del haz sean 0, 0.
- En la computadora del microscopio, navegue hasta un área de carbono.
- En el equipo Leginon en el nodo Navegación, adquiera una imagen con el valor preestablecido gr .
- Encuentre un objeto de interés y vaya a esa ubicación usando la herramienta de cursor.
- Adquiera una imagen con el ajuste preestablecido hln y reubique una parte única de ese objeto de interés en el centro utilizando la herramienta de cursor.
- Adquiera una imagen con el ajuste preestablecido enn y reubíquela en la misma parte única del objeto de interés utilizando la herramienta de cursor.
- Seleccione el desplazamiento de imagen en el menú desplegable y adquiera una imagen con el ajuste preestablecido hln. Reubique en la misma parte única del objeto de interés con la herramienta de cursor.
- En el Presets_Manager, seleccione el ajuste preestablecido hln , haga clic en elbotón s ettings e importe el desplazamiento de la imagen desde Navegación haciendo clic en la flecha verde izquierda junto a los valores de desplazamiento de imagen .
- Repita los pasos 2.9.7 y 2.9.8 para los ajustes preestablecidos sq y gr .
- Atlas de cuadrícula
- En la computadora del microscopio, cierre las válvulas de columna y retraiga la apertura del objetivo. Vaya al nodo Grid_Targeting. En la configuración, cambie la etiqueta de la cuadrícula. Elija el radio deseado del atlas (el radio máximo es de 0,0009 m). Haga clic en Aceptar. A continuación, haga clic en el botón Calcular calculadora de atlas en la parte superior y haga clic en el botón verde Reproducir ('Enviar objetivos').
- En el nodo Square_Targeting, las imágenes de cuadrícula se recopilarán y unirán para formar un atlas. Acércate y aléjate usando el menú desplegable y ajusta el contraste y el brillo. Utilice las barras de desplazamiento para desplazarse por la cuadrícula.
- Una vez recopilado el atlas, inserte la apertura del objetivo si lo desea.
- Si el microscopio tiene un filtro de energía, seleccione un objetivo de referencia en el centro de un cuadrado roto, presione el botón Reproducir y continúe con la alineación ZLP en la siguiente subsección. De lo contrario, omita el paso de alineación ZLP.
- Alineación ZLP
- En la configuración del nodo Align_ZLP, seleccione la posición de etapan para mover el destino de referencia y seleccione el administrador de ajustes preestablecidos como motor. Anule la selección del acondicionador de derivación y, a continuación, pulse OK.
NOTA: La alineación ZLP ahora debe configurarse de modo que el microscopio se mueva periódicamente al objetivo de referencia y ejecute la rutina de alineación ZLP de la cámara. Los tiempos de realineación ZLP de 30 min y 60 min suelen ser seguros para los filtros de energía Gatan BioQuantum y TFS Selectris X, respectivamente. Estos valores varían en función de las condiciones del filtro de energía, como la humedad constante, la temperatura constante, el aislamiento del campo electromagnético y el aislamiento de las vibraciones.
- En la configuración del nodo Align_ZLP, seleccione la posición de etapan para mover el destino de referencia y seleccione el administrador de ajustes preestablecidos como motor. Anule la selección del acondicionador de derivación y, a continuación, pulse OK.
- Configuración de la plantilla de perforación
- En el nodo Square_Targeting, seleccione varios destinos de adquisición y, a continuación, pulse Reproducir.
- En la configuración del nodo Hole_Targeting, asegúrese de que las opciones Permitir la verificación por parte del usuario de los destinos seleccionados y Poner en cola los objetivos estén activadas. Además, marque Omitir buscador de agujeros automatizado por ahora. Haga clic en Aplicar y, a continuación, en Aceptar.
- En la ventana principal, use Ctrl-Mayús-clic derecho para eliminar todos los objetivos. Seleccione el cursor de adquisición y coloque los objetivos. Seleccione el cursor de enfoque y coloque un objetivo de enfoque entre los objetivos de adquisición. Haz clic en Reproducir.
- Para la siguiente imagen Hole_Targeting , desmarque la opción Omitir buscador automático de agujeros en la configuración y, a continuación, haga clic en Aplicar y en Aceptar. Elimine los objetivos automáticos con Ctrl-Mayús-clic derecho.
- Seleccione la herramienta de regla y mida el diámetro a través de un agujero. En la configuración de la plantilla, cambie el Diámetro final de la plantilla al diámetro del agujero medido. No cambie el diámetro de la plantilla original. Haga clic en Probar. Si no hay picos brillantes en el centro de cada agujero, aumente el Diámetro final de la plantilla. Cuando haya terminado, haga clic en Aceptar.
- En la configuración de Umbral, elija un valor para A que segmente los agujeros individualmente cuando se haga clic en Prueba . Haga clic en Aceptar cuando esté satisfecho.
- En la configuración de blobs, introduzca los valores y haga clic en Probar. El valor máximo de blobs es 1, por lo que solo aparece un blob. Haga clic en Aceptar.
- En la configuración de Celosía, utilice la herramienta de regla para medir la distancia entre dos agujeros (de centro a centro). Introduzca el valor en Espaciado y haga clic en Probar. La mancha se convertirá en un punto de celosía. Haga clic en Aceptar.
- Vaya a la configuración de adquisición y optimice los objetivos de adquisición mediante umbrales de espesor de hielo y el botón Probar segmentación. Obtenga información sobre el espesor del hielo pasando el cursor sobre los puntos de celosía.
- Si los objetivos de adquisición no son satisfactorios, utilice la herramienta de regla para medir la distancia y el ángulo desde el punto de celosía hasta la ubicación deseada para un objetivo de adquisición. Elimine los puntos anteriores de la plantilla de destino de adquisición . Haga clic en Relleno automático, ponga 4 para el número de objetivos y cambie el radio y el ángulo a los valores medidos. Haga clic en Aceptar. Marque Aplicar umbral de espesor de hielo en objetivos de adquisición convolucionados por plantilla.
- Una vez que esté satisfecho con los puntos de celosía y los umbrales de espesor de hielo, haga clic en el botón Enviar objetivos .
- Repita cualquiera de los pasos anteriores según sea necesario para cada cuadrado seleccionado. Envíe toda la cola con el botón Enviar destinos en cola una vez que se hayan enviado todos los destinos cuadrados.
- Leginon comenzará a enfocar y obtener imágenes de cada conjunto de objetivos. En el nodo Z_Focus, asegúrese de que la altura eucéntrica se encuentre correctamente.
- Configuración de la segmentación de la plantilla de exposición
- En el nodo Objetivo de exposición, aparecerán imágenes de ampliación de agujeros. Use Ctrl-Mayús-clic derecho para eliminar los objetivos automáticos.
- Mida el diámetro de un agujero con la herramienta de regla. En la configuración de la plantilla, introduzca el diámetro en Diámetro final de la plantilla y haga clic en Probar. Ahora debe haber un pico en el centro de cada agujero. Ajuste los valores de Diámetro si es necesario.
- En la configuración de Umbral, ajuste el valor A hasta que la imagen de prueba binarizada muestre las áreas blancas solo donde se encuentran los agujeros.
- En la configuración de blobs, haga clic en Probar. Debería aparecer una mancha por agujero segmentado. Aumente el Borde para eliminar las manchas de los bordes de la imagen, si lo desea.
- En la configuración de Celosía, haga clic en Probar. Ajuste los parámetros hasta que todos los blobs se hayan convertido en puntos de celosía. Haga clic en Aceptar.
- Haga clic en la herramienta de regla y mida la distancia entre dos puntos de celosía. En la configuración de Celosía, cambie Espaciado a esa distancia.
- Pase el cursor sobre cada punto de celosía para ver la intensidad media, el grosor medio, la intensidad de la desviación estándar y el grosor de la desviación estándar. Tome nota de las intensidades de cada punto de celosía y utilícelas para establecer los parámetros de espesor de hielo deseados en la configuración de adquisición.
- Mida la distancia y el ángulo desde un punto de celosía hasta el centro de 4 agujeros con la herramienta de regla. En la configuración de adquisición, elimine los objetivos de enfoque actuales. Haga clic en Autorrelleno y cambie el radio y el ángulo a los valores medidos. Haga clic en Segmentación de prueba, haga clic en Aceptar y haga clic en Enviar destinos.
NOTA: Leginon encontrará el foco eucéntrico (nodo Enfoque) y recopilará exposiciones, que aparecerán en el nodo Exposición. - Una vez que se hayan obtenido imágenes de todos los objetivos, vaya al nodo Exposure_Targeting para ver la siguiente imagen del agujero. En la configuración, desmarque Permitir la verificación por parte del usuario de los destinos seleccionados. Además, desmarque Poner en cola los objetivos y Omitir el buscador de agujeros automatizado. Haga clic en Aceptar y haga clic en Enviar destinos.
NOTA: Leginon recopilará automáticamente imágenes en función de la configuración configurada anteriormente. Vea las imágenes y los metadatos en Appion.
- Los cambios se pueden realizar durante la recopilación automatizada. Por ejemplo, cambie el rango de desenfoque de la colección en cualquier momento editando el ajuste preestablecido enn en el Preset_Manager.
- Si es necesario detener la recopilación, finalice la cola haciendo clic en los botones Abortar y Abortar cola en los nodos Agujero y Exposición.
- Una vez finalizada la recopilación, vaya a Aplicación y haga clic en Eliminar, luego vaya a Archivo y haga clic en Salir.
2. Uso inteligente de la pantalla automática de Leginon
- Crear una sesión de plantilla de Smart Leginon
- Siga las instrucciones de la Sección 1 para iniciar Leginon.
- Vaya a Aplicación y haga clic en Ejecutar. En la ventana Ejecutar aplicación , seleccione la aplicación de Ptolomeo (seleccione Mostrar todo si es necesario). Establezca main en la computadora Leginon y scope y camera en esa computadora respectiva.
- En el Preset_Manager, importe los ajustes preestablecidos como se describe en el paso 1.2.3.
- Configure los ajustes del nodo.
- En la configuración del nodo Square_Targeting, asegúrese de que las opciones Ordenar destinos por la ruta más corta y Habilitar la segmentación automática estén activadas (Figura complementaria 1A).
- En la configuración del nodo cuadrado, asegúrese de que la opción Esperar a que el nodo procese la imagen esté marcada. Agregue el ajuste preestablecido cuadrado a la lista de la derecha del menú desplegable si aún no está allí. En Configuración avanzada, marque la configuración de estos diafragmas durante la obtención de imágenes y asegúrese de que los valores de los dos diafragmas sean correctos (Figura complementaria 1B).
- En la configuración del nodo Hole_Targeting, marque Permitir la verificación por parte del usuario de los destinos seleccionados. Desmarque Poner en cola los objetivos y Omitir el buscador de agujeros automatizado (Figura complementaria 2A).
- En la configuración del nodo Agujero, marque Esperar a que un nodo procese la imagen y el ajuste preestablecido Agujero estará en la lista de la derecha. En Configuración avanzada, marque la configuración de estos diafragmas durante la obtención de imágenes y asegúrese de que los valores de los dos diafragmas sean correctos (Figura complementaria 2B).
- En la configuración del nodo Exposure_Targeting, marque Permitir la verificación por parte del usuario de los destinos seleccionados. Desmarque Poner en cola los objetivos y Omitir el buscador de agujeros automatizado (Figura complementaria 3A).
- En los ajustes del nodo Exposición, asegúrese de que la opción Esperar a que un nodo procese la imagen esté desactivada, que el ajuste preestablecido de Exposición aparezca a la derecha y, en los ajustes avanzados, marque la opción Establecer estos diafragmas durante la creación de imágenes y asegurarse de que los valores de los dos diafragmas sean correctos (Figura complementaria 3B).
- En la configuración del nodo Enfoque, asegúrese de que la opción Esperar a que un nodo procese la imagen esté desactivada, que el ajuste preestablecido de enfoque automático aparezca a la derecha y que la precisión de enfoque automático deseada esté establecida en 4 x 10-6 m (Figura complementaria 4A).
- En el nodo Focus Sequence (Secuencia de enfoque ) (junto al botón de configuración), habilite solo dos pasos de enfoque automático de inclinación del haz (Figura complementaria 4B,C).
- En la configuración del nodo Z_Focus, asegúrese de que la opción Esperar a que un nodo procese la imagen esté desactivada, que el ajuste preestablecido de agujero aparezca a la derecha y que la precisión de enfoque automático deseada sea de 5 x 10-5 m (Figura complementaria 5A).
- En la secuencia de enfoque del nodo Z_Focus, habilite solo dos pasos de inclinación de platina de bajo aumento (Figura complementaria 5B,C).
- Determine la altura z de la cuadrícula como se describe en el paso 1.2.4.
- Recopile un atlas como se describe en el paso 1.2.10.
- Configure los parámetros del buscador de cuadrados.
- Una vez que se haya recopilado el atlas, Ptolomeo localizará cuadrados en el nodo Square_Targeting. Cada cuadrado mostrará un círculo azul, llamado mancha. Al pasar el cursor sobre cada mancha, Leginon informará de su tamaño calculado por Ptolomeo. Toma nota de las manchas más grandes y más pequeñas.
- En la configuración de Umbral, cambie el Rango de filtro mínimo y máximo para incluir cuadrados deseables y excluir cuadrados no deseados.
- Haga clic en el botón Buscar cuadrados en la barra de herramientas superior. Ajusta el Rango de Filtro hasta que Buscar Cuadrados apunte bien.
- En la configuración de adquisición, elija valores para el Número máximo de objetivos y el Número de grupos objetivo que se van a muestrear. Estos parámetros definirán cuántos cuadrados y grupos de cuadrados se dirigen.
- Una vez que esté satisfecho con los parámetros, haga clic en el botón Reproducir . En la Figura 6 complementaria se muestra un ejemplo de atlas después de la configuración.
- Configure los parámetros del buscador de agujeros.
- En el nodo Hole_Targeting, utilice la herramienta de regla para medir el diámetro de un agujero.
- En la configuración de la plantilla, introduzca el diámetro en Diámetro final de la plantilla y haga clic en Probar. Ajuste el diámetro hasta que todos los agujeros tengan picos blancos brillantes en el centro.
- En la configuración de umbral, haga clic en Probar. Ajuste el valor A hasta que la imagen binarizada muestre las áreas blancas solo donde se encuentran los agujeros.
- En la configuración de blobs, elija excluir los destinos de borde utilizando la regla para determinar una distancia mínima desde el borde e introduciendo ese valor. Los blobs se pueden filtrar por su tamaño, redondez y número deseados. Mantenga el puntero sobre los blobs para mostrar sus valores. Haga clic en Probar para inspeccionar los valores actuales.
- En la configuración de celosía, ingrese el radio de los orificios y el espaciado entre los orificios (use la herramienta de medición), luego haga clic en el botón 42 para medir el valor de Intensidad de referencia de un área de vacío (orificio vacío o película de soporte rota).
- En la configuración de adquisición, marque Usar subconjunto de los destinos de adquisición y establezca el valor máximo de muestra en un número pequeño, como 2. Establezca una amplia gama de medias y desviaciones estándar del espesor del hielo (mida estos valores pasando el cursor sobre los objetivos). Haga clic en Probar segmentación para aleatorizar la selección de segmentación en función de los valores anteriores.
- Haga clic en el botón Reproducir cuando esté satisfecho con todos los ajustes. Leginon realizará la etapa Z_Focus y recogerá el primer objetivo. En la Figura complementaria 7 se muestra una imagen de ejemplo después de la configuración.
- Configure los parámetros de segmentación de la exposición.
- En la configuración de Agujero, establezca la secuencia de comandos de shell en la ruta de secuencia de comandos hl_finding.sh de la instalación de Ptolomeo. Establezca la puntuación mínima que se aceptará para que sea ≤0. Introduzca el radio de los orificios (use la herramienta de medición) y, a continuación, haga clic en el botón 42 para medir el valor de Intensidad de referencia de un área de vacío (orificio vacío o película de soporte rota). Haga clic en Probar para encontrar la red de agujeros.
- En la configuración de adquisición, marque Usar subconjunto de los objetivos de adquisición y establezca el valor máximo de muestra en un número pequeño, como 4, para recolectar en un subconjunto de orificios para el cribado. Establezca una amplia gama de medias y desviaciones estándar del espesor del hielo (mida estos valores pasando el cursor sobre los objetivos).
- Haga clic en el botón Reproducir cuando esté satisfecho con todos los ajustes. Leginon realizará un enfoque eucéntrico y recopilará imágenes de gran aumento, que se pueden ver en el nodo Exposición . En la Figura complementaria 8 se muestra una imagen de ejemplo después de la configuración.
- Compruebe la siguiente imagen Exposure_Targeting para ver si los ajustes anteriores siguen siendo suficientes. Una vez satisfecho, desmarque Permitir la verificación por parte del usuario de los objetivos seleccionados en los ajustes de Orientación de exposición y Orientación de agujeros.
NOTA: La detección ahora debería ejecutarse sin supervisión para la cuadrícula actual. Esta sesión se utilizará como sesión de plantilla para todas las cuadrículas.
- Una vez que la cuadrícula haya terminado de filtrarse, haga clic en Archivo > Salir para cerrar Leginon.
- Configurar Smart Leginon Autoscreen
- En una ventana de terminal, ejecute el autoscreen.py de Smart Leginon.
- Seleccione la interfaz gráfica de usuario, ingrese una lista separada por comas de ranuras de cuadrícula en la pantalla, ingrese completa para el flujo de trabajo, ingrese el nombre de la sesión de plantilla en la que basar las nuevas sesiones (esto se puede encontrar en el visor de imágenes de Appion) e ingrese el valor de altura z de la sesión de plantilla (Figura complementaria 9).
- Se abrirá una interfaz gráfica de usuario que permite introducir el nombre de la sesión para cada cuadrícula y seleccionar sus respectivas asociaciones de proyecto (Figura complementaria 10).
NOTA: Smart Leginon Autoscreen ahora usará la configuración de sesión de la plantilla para filtrar automáticamente cada cuadrícula y cambiar entre cuadrículas desatendidas. - Siga durante la recolección en Leginon, Appion y la computadora del microscopio, o deje el microscopio completamente desatendido.
NOTA: Una vez que se hayan examinado todas las rejillas, Smart Leginon cerrará las válvulas de columna del microscopio.
Resultados Representativos
Siguiendo el protocolo, las sesiones de cribado crioEM pueden realizarse de forma automática y con éxito para la mayoría (80%-90%) de las rejillas y condiciones agujereadas. Anteriormente se han presentado varios ejemplos y experimentos 8,9 para demostrar los resultados esperados de las sesiones exitosas de Smart Leginon Autoscreen. Una sesión exitosa de Autoscreen comienza con ~ 10 minutos de configuración y comúnmente da como resultado un casete completo de 12 cuadrículas que se filtran automáticamente después de aproximadamente 6 h (30 minutos por cuadrícula) donde se obtienen imágenes de 3 a 5 cuadrados de diferentes tamaños y 3 a 5 orificios por cuadrado a gran aumento, lo que permite al usuario determinar rápidamente las características de la muestra en cada cuadrícula e iterar rápidamente a través de las condiciones de muestra / cuadrícula (Figura 3). Ocasionalmente, las sesiones no tienen éxito, generalmente debido a que Autoscreen apunta a cuadrados rotos, no interpreta correctamente grandes gradientes de espesor de hielo a través de la cuadrícula o a través de los cuadrados, o no identifica correctamente los agujeros en las cuadrículas de carbono. Además, las posibles fugas de memoria pueden hacer que Leginon se bloquee debido al uso excesivo de memoria, lo que puede resolverse liberando RAM o reiniciando la computadora, o mejorarse agregando más RAM a la computadora.
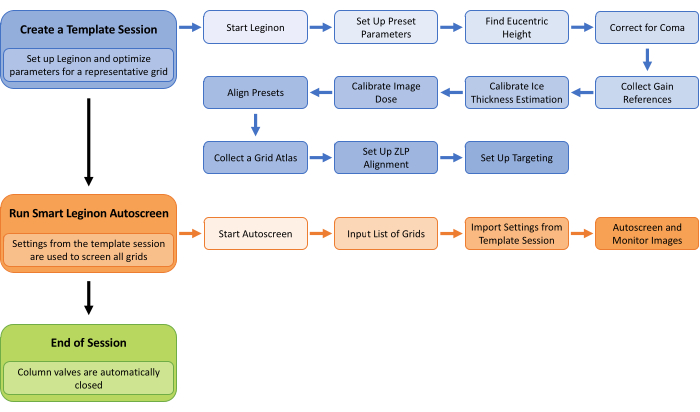
Figura 1: Flujo de trabajo de Smart Leginon Autoscreen. Una descripción general de alto nivel del flujo de trabajo de Smart Leginon Autoscreen. En primer lugar, se crea una sesión de plantilla seleccionando parámetros para una cuadrícula representativa en el lote de cuadrículas que se va a filtrar. La configuración de Leginon y la creación de una sesión de plantilla tardan menos de 45 minutos. En segundo lugar, Autoscreen está configurado para utilizar los parámetros de sesión de la plantilla para filtrar todas las cuadrículas del casete. La configuración de Autoscreen tarda menos de 10 minutos. Por último, Autoscreen finaliza la sesión de proyección. Haga clic aquí para ver una versión más grande de esta figura.
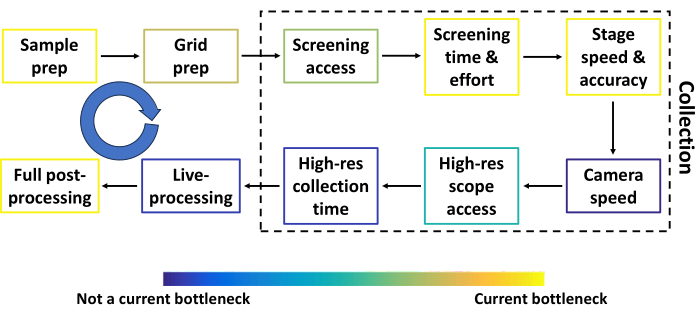
Figura 2: Tubería convencional de crioEM de una sola partícula antes del cribado automatizado. Los pasos más comunes en la línea de crioEM convencional de una sola partícula antes del cribado automatizado, junto con los componentes que se pueden mejorar. Cada paso está coloreado para aproximarse a la magnitud del cuello de botella que tiene el paso en relación con los demás. La flecha circular azul representa varios bucles de retroalimentación entre la mayoría de los pasos. El rendimiento en varios pasos depende en gran medida de la muestra, la financiación y la ubicación del investigador. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Resultados representativos de Smart Leginon Autoscreen. Imágenes representativas multiescala siguiendo el protocolo Smart Leginon Autoscreen recogidas en un TFS Krios cryoTEM con un filtro de energía BioQuantum y una cámara K3. (A) Una imagen compuesta de "atlas" que muestra una visión general de una rejilla crioEM. (B-F) Imágenes a varias escalas de las ubicaciones indicadas en el atlas de cuadrícula. Las imágenes de bajo aumento en la primera fila, las imágenes de aumento medio en la segunda fila y las imágenes de alto aumento en la tercera fila se seleccionaron automáticamente para obtener información sobre la muestra de cuadrados de hielo delgados a gruesos. El espesor del hielo estimado por Leginon se muestra en la parte inferior. Las barras de escala son de 500 μm en (A) y 10 μm para la primera fila, 5 μm para la segunda fila y 100 nm para la tercera fila para (B-F). Esta cifra ha sido modificada con permiso de Cheng et al.8. Haga clic aquí para ver una versión más grande de esta figura.
| gr: Cuadrícula | cuadrado: Cuadrado | hln: Agujero | ventilador: Enfoque automático | fcn: Foco central | enn: Exposición | |
| Magnificación | 210 | 2600 | 6700 | 120000 | 120000 | 120000 |
| Desenfoque | -0.0002 | -0.00015 | -0.00015 | -2 x 10-06 | -7 x 10-07 | -2.5 x 10-06 |
| Tamaño de punto | 5 | 5 | 4 | 2 | 2 | 2 |
| Intensidad | 1.1 | 0.83 | 0.65 | 0.44 | 0.44 | 0.45 |
| Dimensión | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 4096 x 4096 |
| Compensar | 0, 0 | 0, 0 | 0, 0 | 0, 0 | 512, 512 | 0, 0 |
| Binning | 4 x 4 | 4 x 4 | 4 x 4 | 4 x 4 | 2 x 2 | 1 x 1 |
| Tiempo de exposición (ms) | 200 | 500 | 500 | 500 | 500 | 1000 |
| Pre-Exposición(es) | 0 | 0 | 0 | 0 | 0 | 0 |
| Dosis (e/Å2) | -- | -- | -- | 36.5 | 36.5 | 64.7 |
| Guardar fotogramas RAW | No | No | No | No | No | Sí |
Tabla 1: Parámetros preestablecidos para el cribado de la rejilla crioEM en el Centro de Microscopía Electrónica Simons (SEMC) utilizando un crioTEM Glacios con una cámara Falcon 3EC. Se muestran los parámetros para cada preajuste comúnmente utilizado en un crioTEM Glacios con una cámara Falcon 3EC en SEMC. Los diferentes microscopios tendrán diferentes aumentos disponibles y los diferentes experimentos utilizarán diferentes parámetros, como el desenfoque y el tiempo de exposición.
| gr: Cuadrícula | cuadrado: Cuadrado | hln: Agujero | ventilador: Enfoque automático | fcn: Foco central | enn: Exposición | |
| Magnificación | 64 | 1700 | 2850 | 75000 | 75000 | 75000 |
| Desenfoque | 0 | -5 x 10-05 | -5 x 10-05 | -1 x 10-06 | -7 x 10-07 | -2 x 10-06 |
| Tamaño de punto | 6 | 9 | 9 | 6 | 6 | 7 |
| Intensidad | 0.001 | 1,65 x 10-05 | 1.5 x 10-05 | 4.3 x 10-07 | 4.3 x 10-07 | 5.5 x 10-07 |
| Anchura del filtro de energía | -- | -- | -- | 20 | 20 | 20 |
| Dimensión | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 2048 x 2048 | 4096 x 4096 |
| Compensar | 0, 0 | 0, 0 | 0, 0 | 0, 0 | 0, 0 | 0, 0 |
| Binning | 4 x 4 | 4 x 4 | 4 x 4 | 4 x 4 | 2 x 2 | 1 x 1 |
| Tiempo de exposición (ms) | 500 | 2000 | 1000 | 500 | 300 | 8700 |
| Pre-Exposición(es) | 0 | 0 | 0 | 0 | 0 | 0 |
| Dosis (e/Å2) | -- | -- | -- | -- | -- | 47.4 |
| Guardar fotogramas RAW | No | No | No | No | No | Sí |
Tabla 2: Parámetros preestablecidos para el cribado de rejilla crioEM en SEMC utilizando un crioTEM Krios con una cámara Selectris X y Falcon 4i. Se muestran los parámetros para cada preajuste comúnmente utilizado en un Krios con un filtro de energía Selectris X y una cámara Falcon 4i en SEMC. Los diferentes microscopios tendrán diferentes aumentos disponibles y los diferentes experimentos utilizarán diferentes parámetros, como el desenfoque y el tiempo de exposición.
| gr: Cuadrícula | cuadrado: Cuadrado | hln: Agujero | ventilador: Enfoque automático | fcn: Foco central | enn: Exposición | |
| Magnificación | 1550 | 940 | 2250 | 81000 | 81000 | 81000 |
| Desenfoque | 0 | -5 x 10-05 | -5 x 10-05 | -1 x 10-06 | -7 x 10-07 | -2 x 10-06 |
| Tamaño de punto | 4 | 8 | 7 | 6 | 6 | 6 |
| Intensidad | 0.0015 | 0.00017 | 7.3 x 10-05 | 1.3 x 10-06 | 1.3 x 10-06 | 9.2 x 10-07 |
| Anchura del filtro de energía | -- | -- | 50 | 20 | 20 | 20 |
| Dimensión | 1024 x 1024 | 1440 x 1024 | 1440 x 1024 | 1440 x 1024 | 1008 x 1008 | 5760 x 4092 |
| Compensar | 0, 0 | 0, 0 | 0, 0 | 0, 0 | 936, 519 | 0, 0 |
| Binning | 4 x 4 | 8 x 8 | 8 x 8 | 8 x 8 | 4 x 4 | 2 x 2 |
| Tiempo de exposición (ms) | 250 | 600 | 600 | 500 | 500 | 2100 |
| Pre-Exposición(es) | 0 | 0 | 0 | 0 | 0 | 0 |
| Dosis (e/Å2) | -- | -- | -- | -- | -- | 51 |
| Guardar fotogramas RAW | No | No | No | No | No | Sí |
Tabla 3: Parámetros preestablecidos para el cribado de rejilla crioEM en SEMC utilizando un crioTEM Krios con una cámara BioQuantum y K3. Se muestran los parámetros para cada preajuste comúnmente utilizado en un Krios con un filtro de energía BioQuantum y una cámara K3 en SEMC. Los diferentes microscopios tendrán diferentes aumentos disponibles y los diferentes experimentos utilizarán diferentes parámetros, como el desenfoque y el tiempo de exposición.
Figura complementaria 1: Configuración de Square Targeting y configuración de Square para Smart Leginon. (A) Configuración de Segmentación Square. (B) Ajustes cuadrados. Haga clic aquí para descargar este archivo.
Figura complementaria 2: Configuración de Hole Targeting y Configuración de Hole para Smart Leginon. (A) Configuración de Agujeros. (B) Ajustes de agujeros. Haga clic aquí para descargar este archivo.
Figura complementaria 3: Ajustes de Orientación de la exposición y ajustes de la exposición para Smart Leginon. (A) Configuración de segmentación por exposición. (B) Ajustes de exposición. Haga clic aquí para descargar este archivo.
Figura complementaria 4: Ajustes de enfoque y ajustes de secuencia de enfoque para Smart Leginon. (A) Ajustes de enfoque. (B) Ajustes de secuencia de enfoque (Desenfoque1). (C) Ajustes de secuencia de enfoque (Desenfoque2). Haga clic aquí para descargar este archivo.
Figura complementaria 5: Configuración de Z_Focus y configuración de secuencia de Z_Focus para Smart Leginon. (A) Z_Focus ajustes. (B) Z_Focus Ajustes de secuencia (Stage_Tilt_Rough). (C) Z_Focus Ajustes de secuencia (Stage_Tilt_Fine). Haga clic aquí para descargar este archivo.
Figura complementaria 6: Un ejemplo de atlas después de configurar los parámetros de Square_Targeting de Smart Leginon. Los círculos azules son manchas, los signos más verdes son ubicaciones de adquisición y la "x" marrón es la ubicación actual del escenario. Haga clic aquí para descargar este archivo.
Figura complementaria 7: Un ejemplo de atlas después de configurar los parámetros de Hole_Targeting de Smart Leginon. Los signos más morados son ubicaciones de celosía, los signos más verdes con casillas son ubicaciones de adquisición y el signo más azul es la ubicación de foco. Haga clic aquí para descargar este archivo.
Figura 8 complementaria: Un ejemplo de atlas después de configurar los parámetros de Exposure_Targeting de Smart Leginon. Los círculos azules son manchas, los signos más verdes son ubicaciones de adquisición y el signo más azul es la ubicación de foco. Haga clic aquí para descargar este archivo.
Figura complementaria 9: Configuración del terminal Smart Leginon Autoscreen. Haga clic aquí para descargar este archivo.
Figura complementaria 10: Configuración de la interfaz gráfica de usuario de Smart Leginon Autoscreen. Haga clic aquí para descargar este archivo.
Discusión
En este protocolo, describimos la canalización de Smart Leginon Autoscreen y, además, el uso básico de Leginon para aquellos que son nuevos en el software de recolección. La crioEM de una sola partícula está preparada para convertirse en la técnica de resolución de la estructura de proteínas tridimensional (3D) más productiva a finales de 202417. La canalización de crioEM de una sola partícula consta de varios pasos que se optimizan constantemente para aumentar la calidad y el rendimiento de los datos. La Figura 2 muestra los pasos más comunes (preparación de la muestra, preparación de la cuadrícula, tiempo y esfuerzo de detección, tiempo de recolección de alta resolución, procesamiento en vivo y posprocesamiento completo) junto con otros componentes de la tubería que se pueden mejorar (acceso al microscopio de detección, velocidad y precisión de la etapa, velocidad de la cámara y acceso al microscopio de alta resolución). Los resultados de la mayoría de los pasos se convierten en bucles de retroalimentación en los pasos anteriores (flechas azules en la figura 2), lo que hace que toda la canalización sea altamente interdependiente. Cada paso de la Figura 2 está coloreado para aproximarse a la magnitud del cuello de botella que tiene el paso en relación con otros. Smart Leginon Autoscreen reduce significativamente el tiempo y el esfuerzo del operador para el cribado de 12 rejillas de 6 h a menos de 10 min, lo que alivia ese cuello de botella y permite una retroalimentación más rápida a la preparación de la muestra/rejilla (Figura 3).
Hay varios pasos críticos en el Protocolo, que se muestran en la Figura 1. Es fundamental que la cuadrícula utilizada para crear la sesión de plantilla sea representativa de las cuadrículas restantes que se van a filtrar. Es importante destacar que Leginon recuerda todos los ajustes de todo el proceso de configuración para crear una sesión de plantilla (pasos azules en la Figura 1), lo que permite configurar sesiones de plantilla recurrentes cada vez más rápidamente. Al crear una sesión de plantilla, el paso más importante es configurar la segmentación en todos los aumentos para que los parámetros y umbrales reflejen la variación esperada en las cuadrículas que se van a examinar. Los distintos botones de "Prueba" permiten la eficiencia en este proceso de configuración. Durante una sesión de Autoscreen, es fundamental monitorear las primeras cuadrículas en Appion para detectar rápidamente cualquier problema y solucionarlo dentro de Leginon lo antes posible.
El flujo de trabajo típico en SEMC consiste en introducir datos de Autoscreen en CryoSPARC Live18 y utilizar esta información adicional para informar a los bucles de retroalimentación en la preparación de muestras/cuadrículas. Durante los días intensivos de optimización crioEM por parte del investigador-operador, la información sobre las condiciones de la muestra y la rejilla se retroalimenta en la preparación de la muestra y la rejilla, mientras que Autoscreen sigue cribando las rejillas. Esto permite congelar y cribar varias docenas de rejillas por semana8.
Smart Leginon Autoscreen funciona para la mayoría (80%-90%) de las rejillas y condiciones agujereadas observadas en SEMC. El 10%-20% restante de las rejillas incluye aquellas que a veces no funcionan bien: rejillas con una diferencia de contraste mínima entre los agujeros y el sustrato; cuadrículas con agujeros y espaciados más pequeños (por ejemplo, 0,6/0,8) - y cuadrículas en las que la orientación a través de múltiples cuadrículas a menudo no es práctica - Spotiton/Chameleon19,20 cuadrículas que consisten en franjas de muestra a lo largo de la cuadrícula; Rejillas de encaje. Tenga en cuenta que la colección de cuadrícula inclinada con Autoscreen está en desarrollo, pero aún no está disponible. Es posible modificar el protocolo para trabajar con las cuadrículas de Spotiton/Chameleon tomando primero imágenes manualmente de las áreas de la franja para determinar los umbrales de parámetros estrechos, luego intentando agrupar cuadrados más grandes y más pequeños, respectivamente, en el paso 2.1.7.4, y luego seleccionando objetivos del grupo con hielo. El objetivo de esta modificación es que Smart Leginon separe las casillas vacías y no vacías en dos grupos. Si se encuentran parámetros, es posible que no se extiendan bien a las rejillas restantes que se van a examinar. También es posible modificar el protocolo para que funcione con cuadrículas de encaje eliminando el script hl_finding.sh en el paso 2.1.9.1 y configurando los parámetros para apuntar a áreas más claras/oscuras según se desee. La tasa de éxito de esta modificación puede variar de una cuadrícula a otra en función del espesor del hielo y el material de la cuadrícula.
La solución de problemas durante una sesión de Autoscreen es posible y, a veces, apropiada. Los cambios en los parámetros preestablecidos (p. ej., desenfoque) y de segmentación (p. ej., umbrales de segmentación de agujeros) se pueden realizar durante la recopilación automatizada. Mientras se recopila una sesión de Autoscreen, no se puede cancelar una sesión de cuadrícula porque terminará autoscreen.py. Sin embargo, los botones Abortar de los nodos de segmentación se pueden utilizar para omitir cualquier parte de una cuadrícula o una cuadrícula completa. Ocasionalmente, autoscreen.py puede usar demasiada memoria y congelarse, ofreciendo dos opciones: 'forzar el cierre' o 'esperar'. Si se selecciona "forzar salida", todo el script finalizará, lo que requerirá que el usuario vuelva a ejecutar el script para aplicarlo a las cuadrículas restantes para su detección. Si se selecciona 'esperar', el script continuará y la configuración se puede modificar para evitar que se congele en el futuro, por ejemplo, apagando la visualización de la imagen en el nodo Exposición, disminuyendo el tamaño de píxel en el atlas o ejecutando un script de borrado de memoria. Si el programa se congela sin ofrecer las dos opciones, es posible que los errores de memoria no se resuelvan por sí solos, lo que provoca una pausa en la adquisición. La opción 'forzar salida' puede ser útil en este caso.
Smart Leginon Autoscreen se utiliza regularmente en SEMC. A medida que se sigan reduciendo los cuellos de botella en la cartera de crioEM de una sola partícula, la adopción de la crioEM seguirá aumentando para ayudar a responder a las preguntas biológicas. Este protocolo es un paso en la dirección de optimizar toda la canalización al proporcionar un camino claro para reducir significativamente los bucles de retroalimentación.
Divulgaciones
Los autores declaran que no tienen intereses financieros contrapuestos.
Agradecimientos
Parte de este trabajo se realizó en el Centro de Microscopía Electrónica Simons en el Centro de Biología Estructural de Nueva York, con el apoyo de la Fundación Simons (SF349247), los NIH (U24 GM129539) y la Asamblea del Estado de Nueva York.
Materiales
| Name | Company | Catalog Number | Comments |
| Glacios cryoTEM | Thermo Fisher Scientific | GLACIOSTEM | FEG, 200 keV, Falcon 3EC camera |
| Krios cryoTEM | Thermo Fisher Scientific | KRIOSG4TEM | XFEG, 300 keV, Gatan BioQuantum energy filter, Gatan K3 camera |
| Leginon | Simons Electron Microscopy Center | http://leginon.org | |
| Ptolemy | Simons Machine Learning Center | https://github.com/SMLC-NYSBC/ptolemy |
Referencias
- Noble, A. J., et al. Routine single particle CryoEM sample and grid characterization by tomography. eLife. 7, e34257 (2018).
- Weissenberger, G., Henderikx, R. J. M., Peters, P. J. Understanding the invisible hands of sample preparation for cryo-EM. Nature Methods. 18 (5), 463-471 (2021).
- Carragher, B., et al. Current outcomes when optimizing 'standard' sample preparation for single-particle cryo-EM. Journal of Microscopy. 276 (1), 39-45 (2019).
- Kampjut, D., Steiner, J., Sazanov, L. A. Cryo-EM grid optimization for membrane proteins. iScience. 24 (3), 102139 (2021).
- Suloway, C., et al. Automated molecular microscopy: The new Leginon system. Journal of Structural Biology. 151 (1), 41-60 (2005).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Koh, A., et al. Routine collection of high-resolution cryo-EM datasets using 200 KV transmission electron microscope. Journal of Visualized Experiments. 181, 63519 (2022).
- Cheng, A., et al. Fully automated multi-grid cryoEM screening using Smart Leginon. IUCrJ. 10 (1), 77-89 (2023).
- Kim, P. T., Noble, A. J., Cheng, A., Bepler, T. Learning to automate cryo-electron microscopy data collection with Ptolemy. IUCrJ. 10 (1), 90-102 (2023).
- Bepler, T., et al. Smart data collection for CryoEM. Journal of Structural Biology. 214 (4), 107913 (2022).
- Bouvette, J., Huang, Q., Riccio, A. A., Copeland, W. C., Bartesaghi, A., Borgnia, M. J. Automated systematic evaluation of cryo-EM specimens with SmartScope. eLife. 11, e80047 (2022).
- Deng, Y., Grollios, F., Kohr, H., van Knippenberg, B., Janus, M., Caglar, F. Smart EPU: SPA Getting Intelligent. Microscopy and Microanalysis. 27 (S1), 454-455 (2021).
- Fan, Q., et al. CryoRL: Reinforcement learning enables efficient cryo-EM data collection. arXiv. , (2022).
- Li, Y., et al. Optimized path planning surpasses human efficiency in cryo-EM imaging. bioRxiv. Biophysics. , (2022).
- Lander, G. C., et al. Appion: An integrated, database-driven pipeline to facilitate EM image processing. Journal of Structural Biology. 166 (1), 95-102 (2009).
- Rice, W. J., et al. Routine determination of ice thickness for cryo-EM grids. Journal of Structural Biology. 204 (1), 38-44 (2018).
- Russo, C. J. The potential for seeing molecules in cells, CZ Imaging Institute's Hardware Frontiers for CryoET Workshop. CZ Imaging Institute's Hardware Frontiers for CryoET Workshop. , (2023).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Budell, W. C., Allegri, L., Dandey, V., Potter, C. S., Carragher, B. Cryo-electron microscopic grid preparation for time-resolved studies using a novel robotic system, Spotiton. Journal of Visualized Experiments. 168, 62271 (2021).
- Darrow, M. C., Booth, T., Moore, J. P., Doering, K., Thaw, P., King, R. S. Enabling a paradigm shift in CryoEM sample preparation with chameleon. Microscopy and Microanalysis. 27 (S1), 524-525 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados