Method Article
PyDesigner v1.0: Una implementación Pythonic de la tubería DESIGNER para la difusión de imágenes de resonancia magnética
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí presentamos PyDesigner, una línea de procesamiento de imágenes de resonancia magnética de difusión (dMRI) basada en Python capaz de corregir artefactos típicos de dMRI y producir imágenes de tensor de difusión (DTI), imágenes de curtosis de difusión (DKI), imágenes de bolas de fibra (FBI), modelado de microestructuras (integridad de la materia blanca [WMTI] y materia blanca de bolas de fibra [FBWM]) y salidas de tractografía.
Resumen
PyDesigner es un paquete de software basado en Python basado en el parámetro de difusión original EStImation with Gibbs and NoisE Removal (DESIGNER) pipeline (Dv1) para el preprocesamiento de dMRI y la estimación de tensores. Este software se proporciona abiertamente para la investigación no comercial y no se puede utilizar para la atención clínica. PyDesigner combina herramientas de FSL y MRtrix3 para realizar la eliminación de ruido, la corrección de timbre de Gibbs, la corrección de movimiento de corrientes de Foucault, el enmascaramiento cerebral, el suavizado de imágenes y la corrección de sesgo de Rician para optimizar la estimación de múltiples medidas de difusión. Se puede utilizar en todas las plataformas de Windows, Mac y Linux para derivar con precisión las métricas de uso común de los conjuntos de datos DKI, DTI, WMTI, FBI y FBWM, así como los ODF de tractografía y los archivos .fib. También es independiente del formato de archivo, aceptando entradas en forma de formato .nii, .nii.gz, .mif y dicom. Fácil de usar y de instalar, este software también genera métricas de control de calidad que ilustran gráficos de relación señal-ruido, vóxeles atípicos y movimiento de la cabeza para evaluar la integridad de los datos. Además, esta canalización de procesamiento de dMRI admite el procesamiento de múltiples conjuntos de datos de tiempo de eco y presenta personalización de la canalización, lo que permite al usuario especificar qué procesos se emplean y qué salidas se producen para satisfacer una variedad de necesidades del usuario.
Introducción
La resonancia magnética de difusión (dMRI) se aplica ampliamente para el estudio no invasivo de las propiedades microestructurales en el cerebro. Si bien se han propuesto muchos métodos de resonancia magnética funcional, dos de los más utilizados son las imágenes de tensor de difusión (DTI) y las imágenes de curtosis por difusión (DKI). Estas técnicas están estrechamente relacionadas, siendo el DKI una extensión del DTI que incluye la cuantificación de la no gaussianidaddiffusional 1. Ambos proporcionan una variedad de medidas de difusión escalar y permiten la construcción de tractografía de fibra de sustancia blanca. Una ventaja importante de DTI y DKI es que tienen una base sólida en la física de la difusión, por lo que su validez no depende de suposiciones detalladas sobre la microestructura de los tejidos 2,3. Esto permite que DTI y DKI se apliquen en todo el cerebro y el cuerpo para participantes de cualquier edad y estado de enfermedad.
Debido a que las imágenes sin procesar ponderadas en difusión (DWI) se degradan por múltiples factores, incluidos el ruido de la señal, el movimiento, el timbre de Gibbs y la distorsión por corrientes de Foucault, se debe emplear un preprocesamiento antes del cálculo de cualquier cantidad de difusión4. El preprocesamiento de DWI es un campo activo de investigación que ahora está muy desarrollado. Sin embargo, combinar los pasos de procesamiento necesarios en una sola canalización que proporcione resultados coherentes es un desafío porque se deben ajustar varias configuraciones definidas por el usuario en función de los detalles de la adquisición de dMRI y porque el orden en que se realizan los pasos de preprocesamiento afecta el resultado. Por esta razón, en 2016 se propuso inicialmente la canalización Diffusion parameter EStImation with Gibbs and NoisE Removal (DESIGNER, GitHub: NYU-DiffusionMRI/DESIGNER) para optimizar, estandarizar y agilizar el preprocesamiento de DWI5. DESIGNER Dv1 se basa en herramientas de software integradas en FSL, MRtrix3, MATLAB y Python para crear un proceso DWI completo y sin interrupciones, que abarca la corrección de imágenes mediante el preprocesamiento y la difusión/estimación del tensor de curtosis5. Con indicadores de control para activar o desactivar los pasos de preprocesamiento, las correcciones de DWI se pueden realizar de forma selectiva. DESIGNER preprocesa en un orden específico: (i) análisis de componentes principales de Marchenko-Pastur (MP-PCA) eliminación de ruido6, (ii) corrección de timbre de Gibbs7, (iii) corrección de distorsión de imágenes ecoplanas (EPI)8, corrección de corrientes de Foucault 9, corrección de movimiento10 y reemplazo de valores atípicos11, (iv) corrección de campo de polarización B1, (v) generación de máscaras cerebrales, (vi) suavizado, (vii) corrección de sesgo de ruido de Rician y (viii) normalización b0. El preprocesamiento, en este orden específico, mejora tanto la precisión como la relación señal-ruido (SNR) efectiva5. Vale la pena señalar que cada paso de PyDesigner es opcional y se puede emplear u omitir según las preferencias del usuario. El suavizado, por ejemplo, puede no ser un paso de preprocesamiento necesario para algunos conjuntos de datos. Aunque ayuda a mitigar los errores de filtrado, es posible que no sea necesario para conjuntos de datos de ultra alta calidad. Como tal, los usuarios pueden optar por emplear solo los pasos necesarios para sus datos.
La implementación de DESIGNER en todas las plataformas es un desafío debido a las diferencias en los sistemas operativos y la configuración del entorno. En particular, el hecho de que DESIGNER Dv1 esté escrito principalmente en MATLAB crea importantes problemas de portabilidad derivados de los complicados requisitos de configuración necesarios para habilitar la interfaz Python-MATLAB. Además, las diferentes combinaciones de MATLAB, Python y versiones de dependencia comprometen la reproducibilidad de esta canalización. Por esta razón, hemos desarrollado PyDesigner, que está completamente basado en Python. Esta canalización de procesamiento de dMRI no solo permite un preprocesamiento sin problemas, sino que también permite que la canalización se incorpore a un contenedor Docker, lo que mejora en gran medida la portabilidad y la reproducibilidad. Además, al reemplazar el código de MATLAB, PyDesigner evita todas las tarifas de licencia y mejora la accesibilidad.
Esta canalización de procesamiento de dMRI aumenta el enfoque de manos libres introducido por DESIGNER, agrega varias características nuevas e incorpora herramientas de FSL y MRtrix3 para realizar el preprocesamiento. Se utilizaron bibliotecas matemáticas estándar de Python, como Numpy12, SciPy13 y CVXPY14,15, para reemplazar las partes de MATLAB de DESIGNER con código Python. Este software se proporciona abiertamente y está disponible en Github16. El objetivo de este documento no es validar o comparar nuestro software con software similar, sino más bien proporcionar a los usuarios una guía paso a paso para procesar sus datos con PyDesigner en caso de que decidan hacerlo.
Protocolo
Todos los datos utilizados para desarrollar y probar el software se recopilaron bajo un protocolo de estudio aprobado por la junta de revisión institucional.
NOTA: PyDesigner se creó en MacOS 10.14 y requiere un subsistema de Windows para Linux (WSL) para ejecutarse en Windows. Los mismos comandos se pueden utilizar para sistemas Linux/Mac.
1. Compatibilidad del sistema operativo
- Para sistemas Windows, siga el vínculo para instalar WSL17 y, a continuación, continúe con el paso 2.6.
- Para sistemas basados en Unix (como Linux y Mac), vaya a la sección 2.
NOTA: Los sistemas basados en Unix pueden instalar PyDesigner y sus dependencias de forma nativa.
2. Instalar FSL
NOTA: Todas las pruebas de PyDesigner se han realizado con FSL versión 6.0.2 y se recomienda usar v6.0.2 o superior.
- Ve a la wiki de FSL18 y selecciona Descargar/Instalar. Para los usuarios de Windows, vaya al paso 2.6.
- Elija Download FSL (Descargar FSL ) en el encabezado Installing FSL (Instalación de FSL). A continuación, seleccione la licencia de software de instalación de FSL, lea el acuerdo y seleccione aceptar.
- Rellene los campos obligatorios y seleccione Registrarse.
- Elija el enlace Descargar FSL (primer enlace de la página). A continuación, seleccione el enlace de instrucciones de instalación (segundo enlace de la página). Siga estas instrucciones para descargar FSL para cada sistema operativo.
NOTA: FSL tarda unos 10 minutos en instalarse. - Para verificar que la instalación se ha realizado correctamente, introduzca lo siguiente en una nueva ventana de terminal:
$ flirt -version. Esto debería devolver el número de versión. Continúe con el paso 3. - Para los usuarios de Windows, siga la guía de instalación de WSL para FSL19.
3. Crea un entorno de Conda
- Instale un paquete de Conda para administrar entornos de Python. Los usuarios pueden elegir entre Miniconda o Anaconda.
- Descargar Miniconda/Anaconda20. Los usuarios de Mac y Linux continúan con el paso 3.2.
- Para Windows, configure Conda dentro de WSL21.
NOTA: Las siguientes instrucciones son para Miniconda, pero los mismos comandos funcionan para Anaconda. Aunque Conda ya no es compatible con Python 3.7, todavía es posible crear un entorno de Conda con Python 3.7, lo que permite que este protocolo se implemente precisamente como se describe.
- Introduzca lo siguiente en el terminal para crear el entorno de Conda:
$ conda create -n mri python=3.7. Use este entorno de Conda para todas las actividades relacionadas con PyDesigner.- Cuando se le solicite con
Proceed ([y]/n)?Introy.
NOTA: Indicador -n: Utilícelo para indicar el nombre del entorno de Conda. En el vídeo, el entorno de Conda se denomina mri.
NOTA:python=Se recomienda la versión 3.7, ya que PyDesigner se construyó y probó en esta versión.
- Cuando se le solicite con
4. Instalar MRtrix3
- Para activar el entorno de Conda, escriba el siguiente comando:
$ conda activate mri - Abra la página de descarga de MRtrix322 y siga las instrucciones de descarga para el sistema operativo adecuado. El código de instalación solicitará la confirmación de ciertas dependencias. Entra
ypara continuar con el proceso de descarga.- Imprima la documentación de MRtrix3 para confirmar la instalación introduciendo lo siguiente en el terminal:
$ mrinfo -h
- Imprima la documentación de MRtrix3 para confirmar la instalación introduciendo lo siguiente en el terminal:
5. Instalar PyDesigner
- Abra una nueva ventana de terminal y active el entorno de Conda introduciendo lo siguiente en el terminal:
$ conda activate mri - Ejecute el comando:
$ pip install PyDesigner-DWI - Para verificar que la instalación se ha realizado correctamente, imprima el número de versión introduciendo lo siguiente en el terminal:
$ pydesigner -v- Si aparece el error "módulo no encontrado", consulte la sección Solución de problemas al final del protocolo (sección 10)
NOTA: $ pydesigner -h imprimirá documentación de ayuda que explica los comandos y la sintaxis del software. PyDesigner también se puede descargar a través de Github. Consulte las instrucciones de instalación en el sitio web23.
- Si aparece el error "módulo no encontrado", consulte la sección Solución de problemas al final del protocolo (sección 10)
6. Preparación de los datos
NOTA: PyDesigner funciona con varios tipos de archivos como .nii, .nii.gz, .mif y DICOM. En el vídeo, los DICOM se ordenan y se convierten a NIfTI antes de usar esta canalización de procesamiento de dMRI. Los archivos NIfTI son más fáciles de trabajar porque están desidentificados y hay un archivo por secuencia.
- Para ordenar los DICOM, utilice la herramienta DicomSort24.
NOTA: Se permite cualquier otra herramienta de clasificación DICOM disponible que realice la misma tarea. - Después de ordenar los DICOM, conviértalos en archivos NIfTI usando dcm2niix25.
- Instale dcm2niix ingresando el siguiente comando en el terminal:
$ sudo apt installdcm2niix - Una vez descargado, escriba lo siguiente en el terminal para verificar la instalación imprimiendo la documentación para dcm2nii:
$ dcm2niix -h
- Instale dcm2niix ingresando el siguiente comando en el terminal:
- Para ejecutar dcm2niix, introduzca el siguiente comando en el terminal:
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
NOTA: -f: Para especificar la estructura de nombre de archivo para el archivo de salida, siga esta etiqueta con cualquier estructura de nombre de archivo deseada. El video utiliza: -f %s_%p_%d . %s, %p y %d son notaciones para la serie, el número de protocolo y la descripción, respectivamente. Cualquier estructura de archivos funcionará. Las salidas esperadas de dcm2niix son los archivos .bval, .bvec, .json y .nii. Si se adquirieron datos de recarga, habrá archivos de recarga .json y .nii.
7. Uso básico de PyDesigner
NOTA: PyDesigner puede tardar hasta 1,5 h para un solo conjunto de datos, dependiendo de los parámetros de procesamiento y las capacidades de la computadora.
- Para ejecutar el procesamiento estándar, utilice el indicador
-so-standard. Este comando ejecutará la canalización de preprocesamiento recomendada en el siguiente orden: eliminación de ruido6, corrección de timbre de Gibbs7, corrección de distorsión EPI8, corrección de movimiento de corrientes de Foucault9, creación de máscaras de cerebro y líquido cefalorraquídeo (LCR), suavizado, corrección de sesgo de Rician, detección de valores atípicos11, estimación de tensores y creación de mapas paramétricos. - Para ejecutar el comando básico de PyDesigner, primero active el entorno de Conda introduciendo el siguiente comando en el terminal:
$ conda activate mri - Imprima la documentación de ayuda del software introduciendo el siguiente comando:
$ pydesigner -h. En esta documentación se explican todos los comandos y la sintaxis posibles para PyDesigner. - Utilice el siguiente ejemplo para estructurar el comando.
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - Tenga en cuenta los siguientes indicadores importantes:
--verbose: Utilícelo para ver la consola de PyDesigner a medida que procesa los datos (no es necesario).--rpe pairs #: Utilice esta opción para permitir que los usuarios con una secuencia TOPUP aceleren el proceso de corrección de la distorsión EPI8 mediante TOPUP. El # se refiere a la cantidad de volúmenes B0 de la secuencia TOPUP que se utilizarán. El uso de #=1 evita la sobreestimación del campo de distorsión y reduce el tiempo necesario para crear este campo. Los usuarios han utilizado rutinariamente #=1 con resultados confiables. Al usar #=1, PyDesigner tomará el primer volumen de forma predeterminada.
8. Uso avanzado de PyDesigner: Adaptación del software a las necesidades individuales de los usuarios
NOTA: Al adaptar PyDesigner, omita la -s marca; esta marca es el comando para el preprocesamiento automático.
- Active el entorno de Conda introduciendo el siguiente comando en el terminal:
$ conda activate mri - Consulte el sitio web del software23 para obtener una lista de indicadores; cada marca permite al usuario ejecutar cada parte de PyDesigner individualmente.
- Estructure el comando. Comience con las marcas de cada secuencia que el usuario desee realizar. A continuación, dirija PyDesigner a los archivos de entrada y salida. Por ejemplo:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - Ejemplo 1: Ejecute solo el ajuste de tensor (sin preprocesamiento adicional).
- Active el entorno de Conda introduciendo el siguiente comando en el terminal:
$ conda activate mri - Consulte el sitio web de PyDesigner23 para obtener una lista de banderas. En este ejemplo se utiliza
-m. -m: Agregue este indicador para limitar el ajuste del tensor solo dentro de los vóxeles cerebrales. Esto acelera el ajuste tensorial + detección de valores atípicos.- Utilice el siguiente ejemplo para estructurar el comando:
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
NOTA: La entrada para el ajuste del tensor es un archivo .nii preprocesado.
- Active el entorno de Conda introduciendo el siguiente comando en el terminal:
- Ejemplo 2: Ejecute solo la eliminación de ruido (sin preprocesamiento adicional).
- Active el entorno de Conda introduciendo lo siguiente en el terminal:
$ conda activate mri - Consulte el sitio web de PyDesigner23 para obtener una lista de banderas. En este ejemplo se utiliza
-n. - -n: Añade este indicador para completar el paso de eliminación de ruido. Ingrese el archivo DWI previamente preprocesado con la etiqueta DWI_preproccessed.nii, que se encuentra en la carpeta de salida de cada sujeto.
- Utilice el siguiente ejemplo para estructurar el comando:
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- Active el entorno de Conda introduciendo lo siguiente en el terminal:
9. Futuras actualizaciones de PyDesigner
NOTA: Las actualizaciones se pueden encontrar en el sitio web23.
- Para actualizar PyDesigner, introduzca lo siguiente en una nueva ventana de terminal:
$ pip install --upgrade PyDesigner-DWI
NOTA: Si PyDesigner se instaló a través de Github16, también debe actualizarse mediante git.
10. Solución de problemas de instalación
- Al descargar PyDesigner, aparecerá el código de error "módulo no encontrado" si falta un módulo de Python.
- Arregle un módulo faltante ingresando lo siguiente en el terminal:
$ pip install nameofmodule - A continuación, repita el primer comando de instalación introduciendo lo siguiente en el terminal:
$ pip install PyDesigner-DWI - PyDesigner ahora debería estar completamente instalado. Vuelva al paso 5.3. Si se produce el mismo error, repita este proceso hasta que se instalen todos los módulos de Python que faltan.
- Arregle un módulo faltante ingresando lo siguiente en el terminal:
11. Solución de problemas de errores de salida
- Si se produce algún problema, revise el archivo log_command.json para identificar cualquier error de preprocesamiento. Revise los archivos intermedios para determinar el error específico.
- Para obtener ayuda para solucionar problemas, envíe una solicitud en la página16 de GitHub.
12. Ejecutar PyDesigner con un conjunto de datos de ejemplo
- Descargue el conjunto de datos de ejemplo de OpenNeuro aquí26.
NOTA: Este conjunto de datos de ejemplo contiene datos sin procesar y preprocesados. Los datos sin procesar están pensados como entrada para el procesamiento de ejemplos de PyDesigner. Los datos preprocesados consisten en las salidas generadas después de que los datos sin procesar se procesaron mediante la canalización de procesamiento de dMRI en un iMac OS v12.4. Los usuarios pueden descargar los datos sin procesar, procesarlos mediante PyDesigner y comparar sus resultados con los resultados de ejemplo proporcionados para asegurarse de que PyDesigner se ejecuta correctamente. Los datos sin procesar se encuentran en la carpeta principal (/PyDesigner Example Dataset/sub-01) y los datos preprocesados se encuentran en la carpeta de derivados (/PyDesigner Example Dataset/derivatives/sub-01). A menos que se especifique lo contrario, el conjunto de datos de OpenNeuro se descargará en la carpeta de usuario predeterminada. - Abra un nuevo terminal en Mac/Linux o WSL en Windows.
NOTA: Para ejecutar la canalización de procesamiento estándar de PyDesigner, siga la estructura de comandos de ejemplo que se muestra a continuación. Introduzca los siguientes comandos a continuación en el terminal/WSL. En este ejemplo, donde " ./user_download_folder/" indica la ruta de acceso de la carpeta de datos de ejemplo. Esta ruta será específica del lugar donde se almacenan los datos de ejemplo en el equipo de los usuarios individuales. - Para crear una nueva carpeta para las salidas, introduzca lo siguiente en el terminal:
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - Para activar el entorno Conda, introduzca lo siguiente en el terminal:
$ conda activate mri - Para ejecutar PyDesigner en el conjunto de datos de ejemplo, escriba lo siguiente en el terminal:
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
NOTA: En este ejemplo, ./user_download_folder/ indica la ruta de acceso de la carpeta de datos de ejemplo. Esta ruta será específica para el equipo de cada usuario. Los comandos de los pasos 12.3 a 12.5 siguen la estructura del paso 8.3. Este comando emplea la marca -s, la marca de procesamiento estándar de PyDesigner, que ejecuta los siguientes pasos de procesamiento en este orden: eliminación de ruido6, corrección de timbre de Gibbs7, distorsión 8,9, enmascaramiento cerebral, suavizado y corrección de sesgo de Rician. - Si el software está instalado correctamente, todas las salidas se rellenarán en la carpeta PyDesigner_Outputs recién creada. Compare los archivos de preprocesamiento producidos por PyDesigner con la carpeta preprocesada del conjunto de datos de ejemplo que se encuentra en la carpeta "derivados" descargada de OpenNeuro.
- Consulte la sección de resultados representativos para comparar sus mapas de métricas con los de los resultados del conjunto de datos de ejemplo.
Resultados
El software PyDesigner aplica múltiples pasos de corrección de imagen a los datos de difusión sin procesar y genera salidas que se utilizan para mejorar la precisión de los archivos sin procesar al realizar análisis. Cada paso disponible en la tubería ha sido validado previamente a través de publicaciones revisadas por pares 5,6,7,8,9,10,11 como se discutió en la introducción. Los resultados del software se pueden utilizar en análisis como perfiles de tractografía, matrices de conectividad, análisis de vóxeles, análisis de ROI, análisis fODF, TBSS y análisis basados en fixel.
El sitio web del software23 enumera todos los archivos de salida generados durante el proceso de preprocesamiento. Después de ejecutar cada solicitud, la consola generará una descripción de todos los procesos completados. Hay 3 tipos de archivos de salida: archivos de procesamiento, métricas y control de calidad. La estructura del directorio de salida se muestra en la Figura 1. Estos archivos están disponibles cuando se utiliza el preprocesamiento estándar (consulte la sección 7 del protocolo). Si el usuario requiere un uso más avanzado (consulte la sección 8 del protocolo), los archivos de salida disponibles dependerán de los procesos que se hayan completado.
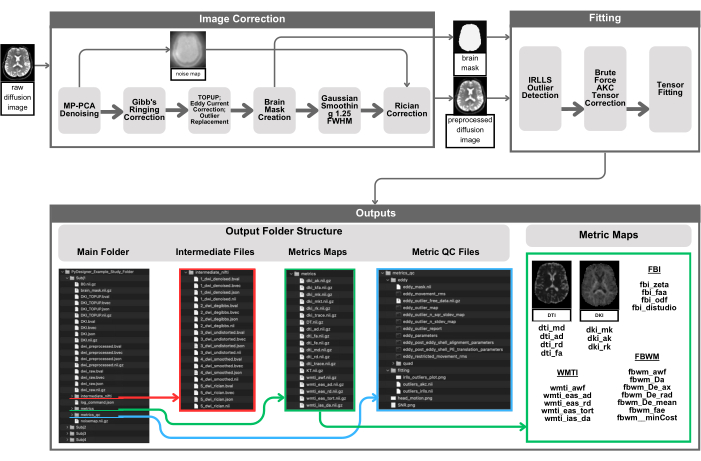
Figura 1: Representación visual de la canalización de PyDesigner. El preprocesamiento comienza proporcionando un DWI 4D de entrada a PyDesigner (arriba a la izquierda), que luego se somete a la eliminación de ruido MP-PCA para producir un DWI 4D sin ruido y un mapa de ruido 3D. A continuación, el DWI 4D sin ruido se somete a la corrección de timbre de Gibbs, la corrección de sesgo de Rician, el TOPUP, la corrección de corrientes de Foucault y la corrección de valores atípicos. A continuación, se calcula una máscara cerebral para los pasos siguientes, la detección de valores atípicos y el ajuste de tensores para acelerar los cálculos realizándolos solo dentro de la máscara cerebral. Los resultados se pueden encontrar en subdirectorios en la carpeta principal de procesamiento de asuntos: intermediate_nifti, métricas y metrics_qc. Tenga en cuenta que las capturas de pantalla de las salidas de PyDesigner no pretenden ser una lista exhaustiva de todas las salidas posibles, sino más bien proporcionar un ejemplo visual esquemático de lo que los usuarios pueden esperar. Las salidas, tanto las salidas finales como los archivos intermedios, variarán en función de los datos de entrada del usuario y los indicadores de procesamiento empleados. Haga clic aquí para ver una versión más grande de esta figura.
Tratamiento
Los archivos de procesamiento se usan durante la canalización de PyDesigner y se almacenan en el directorio de salida raíz. Para cada paso de preprocesamiento, los archivos DWI intermedios se guardan en la carpeta de salida "intermediate_nifti", como se muestra en la Figura 1. Se debe hacer referencia a estos archivos al abordar cualquier problema con el procesamiento o las salidas para evaluar cada paso de la canalización por separado.
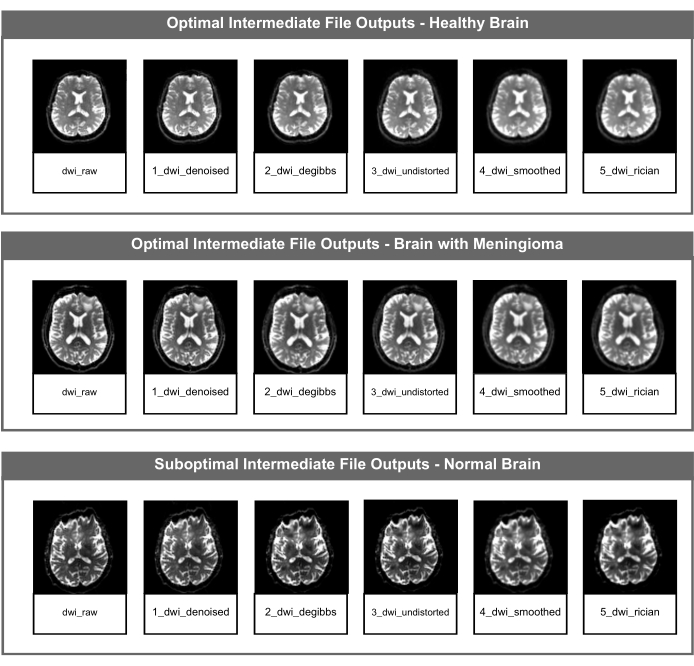
Figura 2: Archivos NifTI DWI intermedios óptimos y subóptimos. En la figura se muestra el archivo NIfTI intermedio para cada paso de corrección de imagen de la canalización de PyDesigner. La fila superior es un ejemplo de salidas de archivos intermedios óptimas utilizando datos de un cerebro adulto sano, la fila central es un ejemplo de salidas de archivos intermedios óptimas utilizando datos de un cerebro patológico (meningioma) y la fila inferior muestra salidas de archivos intermedios subóptimas utilizando datos de un cerebro adulto sano con un artefacto de susceptibilidad no relacionado con la estructura o la salud del cerebro. Haga clic aquí para ver una versión más grande de esta figura.
Métricas
Esta carpeta contiene todos los mapas paramétricos que PyDesigner calcula (consulte la Figura 1). Esto incluye mapas paramétricos para DTI/DKI, imágenes de bolas de fibra (FBI)/materia blanca de bolas de fibra (FBWM) y métricas de integridad del tracto de materia blanca (WMTI) (Tabla 1)16.

Tabla 1: Rango esperado de valores para las métricas DTI/DKI y FBI/FBWM. La tabla incluye una lista de métricas sólidas de DTI, DKI, FBI y FBWM generadas por PyDesigner y sus rangos de valores esperados. También se enumeran las secuencias necesarias y los valores b (s/mm2) para derivar cada métrica. Las métricas DTI enumeradas son FA, MD, AD y RD. Las métricas DKI enumeradas son MK, AK, RK y KFA. La métrica del FBI que aparece en la lista es FAA. Las métricas de FBWM enumeradas son AWF, DA, DE_AX, DE_RAD y FAE.
Los usuarios pueden llevar a cabo un control de calidad (QC) visual y de valor de las métricas de difusividad media (MD), anisotropía fraccional (FA) y curtosis media (MK) para identificar resultados subóptimos. Si estas métricas no son óptimas según los estándares que se describen a continuación, los usuarios deben examinar cada archivo intermedio descrito en la Figura 2 para determinar qué paso de preprocesamiento no se realizó correctamente.
El control de calidad visual se utiliza para identificar resultados subóptimos (por ejemplo, problemas de ajuste de tensores y artefactos). Recomendamos utilizar ImageJ para el control de calidad visual para garantizar que no se realice ninguna manipulación de la imagen a través de los valores predeterminados del software. La fila superior de la Figura 3 muestra los mapas métricos típicos de MD, FA y MK utilizando un umbral biológicamente plausible de 0-3 μm2/ms, 0-1 μm2/ms y 0-2 μm2/ms, respectivamente (Figura 3 [Fila superior]). El mapa de MD debe tener los valores más altos en los ventrículos y los valores más altos en la sustancia gris cortical (Figura 3A [fila superior]). El mapa de AF debe tener tractos aparentes de materia blanca que son claros en todo el cerebro (Figura 3B [fila superior]). El mapa MK debe tener valores altos en WM y valores más bajos en la materia gris y el CSF (Figura 3C [Fila superior]). La figura 3D [fila superior] es un ejemplo de un mapa métrico con problemas de ajuste de tensores, lo que da como resultado clústeres de vóxeles de valor cero. Si se produce algún problema, revise el archivo log_command.json para encontrar errores de preprocesamiento. Revise los archivos intermedios para determinar el error específico. Para obtener ayuda para solucionar problemas, envíe una solicitud en la página de GitHub de PyDesigner.
El control de calidad de valores se utiliza para identificar si los vóxeles de un mapa de métricas determinado son relativamente uniformes entre los sujetos de cada conjunto de datos. El rango de valores esperado para cada mapa y dataset depende de los datos y de los parámetros de PyDesigner. En nuestro conjunto de datos de prueba, tuvimos picos consistentes en los rangos de 8000 a 10 000, 2500 a 4000 y 5000 a 13 000 para MD, FA y MK, respectivamente, utilizando la agrupación de histogramas predeterminada de FSLeyes. La fila inferior de la Figura 3 proporciona ejemplos de la variabilidad del histograma. La tabla 1 contiene los valores esperados del eje x para estas métricas. Los vóxeles más altos o más bajos son característicos del conjunto de datos o indican un artefacto o un problema de preprocesamiento (Figura 3D [fila inferior]).
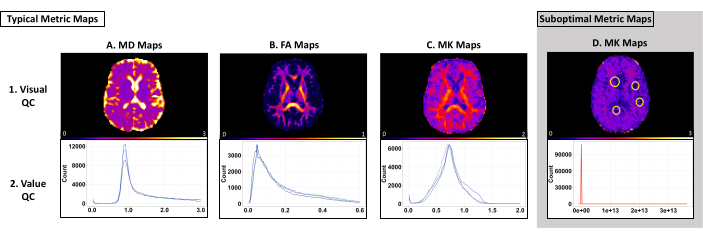
Figura 3: Ejemplos de mapas de métricas e histogramas de PyDesigner con resultados óptimos y subóptimos. La fila superior muestra ejemplos de mapas MD, FA y MK de un solo sujeto utilizados para el control de calidad visual. La fila inferior muestra los histogramas multisujeto utilizados para el control de calidad del valor. (A-C) Ejemplos típicos de mapas métricos e histogramas que superan el control de calidad visual y de valores. Cada línea azul de los histogramas de cada tipo de métrica representa un conjunto de datos individual. Tenga en cuenta que cada conjunto de datos sigue una curva similar y se encuentra dentro de un rango similar. (D) Un ejemplo de un mapa métrico que no pasa el control de calidad visual o de valores. Observe cómo la línea roja en el histograma muestra una curva que difiere de la de A-C. Los vóxeles cero marcados con un círculo en este mapa métrico se deben a problemas de ajuste del tensor durante el preprocesamiento (Panel D, fila superior). Este histograma es un ejemplo de vóxeles generalmente más altos o más bajos en un conjunto de datos de lo esperado (Panel D, fila inferior). Haga clic aquí para ver una versión más grande de esta figura.
Control de calidad
Una vez que PyDesigner ha procesado los datos, se debe usar la carpeta metrics_qc (consulte la Figura 1) para identificar conjuntos de datos subóptimos. Para cada conjunto de datos, PyDesigner genera tres gráficos que se utilizan para el control de calidad.
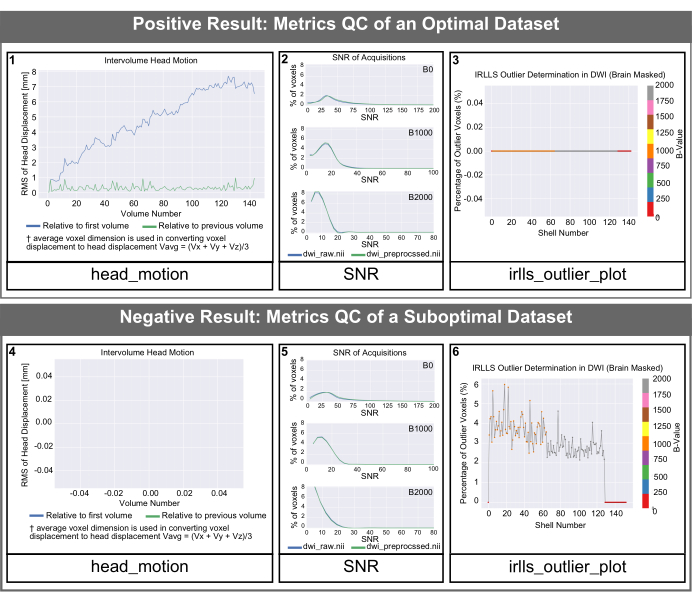
Figura 4: Histogramas de control de calidad generados por PyDesigner para conjuntos de datos óptimos y subóptimos. SNR, movimiento de la cabeza entre volúmenes e histogramas atípicos generados por PyDesigner. Ambas filas representan datos de un cerebro adulto sano. La fila superior es un ejemplo de histogramas de control de calidad para un conjunto de datos óptimo. La fila inferior muestra los resultados del control de calidad de un conjunto de datos subóptimo con un artefacto de susceptibilidad no relacionado con la estructura o la salud del cerebro. Tenga en cuenta que el tamaño del texto de las etiquetas en las salidas predeterminadas de PyDesigner es menor de lo que aparecerán en esta figura. Hemos aumentado el tamaño del texto en esta figura para facilitar la lectura. Haga clic aquí para ver una versión más grande de esta figura.
Los gráficos head_motion de la Figura 4 muestran el desplazamiento de la cabeza en relación con el primer volumen y el volumen anterior. Como se ve en la Figura 4 (panel 1), el desplazamiento de la cabeza suele ser pequeño y PyDesigner se ajusta a estos artefactos de movimiento en la tubería de procesamiento estándar utilizando el programa FSL Eddy junto con TOPUP para la corrección de movimiento y movimiento de corrientes de Foucault9. En el caso de conjuntos de datos subóptimos, el gráfico de head_motion puede aparecer vacío, como se muestra en la Figura 4 (panel 4). Esto indica que la corrección de movimiento por corrientes de Foucault no se realizó correctamente, por lo tanto, PyDesigner no pudo generar un gráfico. Los archivos de registro de corrección de corrientes de Foucault se pueden encontrar en la subcarpeta de Foucault dentro de la carpeta metrics_qc (consulte la Figura 1). El gráfico de relación señal-ruido (SNR) muestra 3 gráficos. Cada gráfico es para un valor b diferente y muestra tanto los datos preprocesados como los sin procesar. Para un conjunto de datos óptimo, el pico de SNR de datos brutos debe ser ≥5 (Figura 4 [panel 2]). Los conjuntos de datos subóptimos tendrán un pico de SNR de datos brutos de ≤3 (Figura 4 [panel 5]). Idealmente, los usuarios deberían ver que el pico de SNR para todos los valores b aumenta ligeramente, pero no drásticamente. El gráfico de valores atípicos se encuentra en la carpeta de ajuste dentro de metrics_qc y muestra el porcentaje de valores atípicos en el conjunto de datos (Figura 4 [paneles 3 y 6]). Un buen conjunto de datos debe tener un porcentaje bajo de valores atípicos, normalmente inferior al 5% (Figura 4 [panel 3]). Un conjunto de datos subóptimo tendrá un gran porcentaje de valores atípicos, como se muestra en la Figura 4 (panel 6).
Resultados de un conjunto de datos de ejemplo
Una vez que PyDesigner haya terminado de procesar el conjunto de datos de ejemplo, todas las salidas deben estar contenidas en la carpeta "PyDesigner_Outputs". Estos resultados se pueden comparar con los que se encuentran en la carpeta "derivados" empaquetada con el conjunto de datos de ejemplo descargado de OpenNeuro (procesado en MacOS 12.4). Si el software funciona correctamente, la estructura de archivos de "PyDesigner_Outputs" y "derivados" será exactamente la misma. Del mismo modo, los gráficos SNR, movimiento de la cabeza y valores atípicos que se encuentran en la subcarpeta "metrics_qc" deben coincidir con los de la Figura 5A. Los mapas métricos (que se encuentran en la subcarpeta "métricas") se pueden comparar a través de software de imágenes como FSLeyes, MRIcron, ImageJ, etc. Los histogramas de los valores FA, MD y MK de métricas preprocesadas se pueden ver en la Figura 5B. Tenga en cuenta que todos los histogramas métricos que se muestran en la Figura 5B se escalan en función de las escalas de valores métricos sugeridas en la Tabla 1.
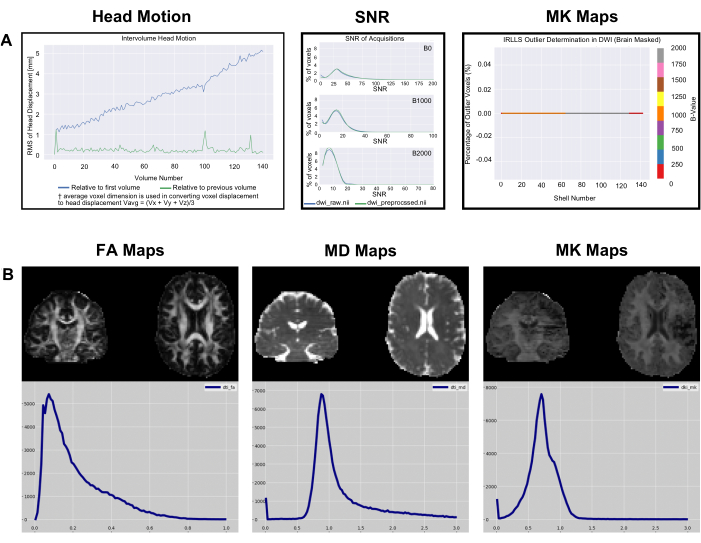
Figura 5: Histogramas métricos y métricos de control de calidad métricos de datos. (A) SNR, movimiento de la cabeza entre volúmenes e histogramas atípicos generados por PyDesigner para los datos de ejemplo descargados de OpenNeuro. Tenga en cuenta que el tamaño del texto se ha aumentado en los gráficos de esta figura para facilitar la lectura. (B) Histogramas de mapas métricos de un solo sujeto de recuentos de valores de vóxeles para FA, MD y MK para el mismo conjunto de datos, visualizados a través de FSLeyes v6.0. Haga clic aquí para ver una versión más grande de esta figura.
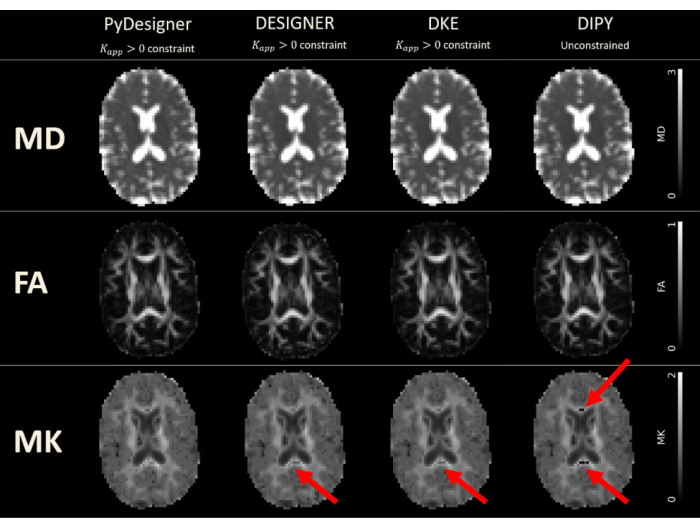
Figura 6: Mapas DTI y DKI derivados de PyDesigner, DESIGNER, DKE y DIPY. El ajuste tensorial se realizó con la restricción Kapp > 0 en PyDesigner, DESIGNER5 y Diffusional Kurtosis Estimator (DKE)27, mientras que el ajuste sin restricciones se utilizó en Diffusion Imaging in Python (DIPY)28 debido a limitaciones de software. Las unidades de MD son micrómetros cuadrados por milisegundo (μm2/ms), mientras que las otras métricas son adimensionales. Las interrupciones debidas a un problema genérico de la corrección del timbre de Gibbs se pueden ver en los mapas MK producidos por DESIGNER, DKE y DIPY. Esta figura ha sido reproducida con permiso de Dhiman et al.29. Haga clic aquí para ver una versión más grande de esta figura.
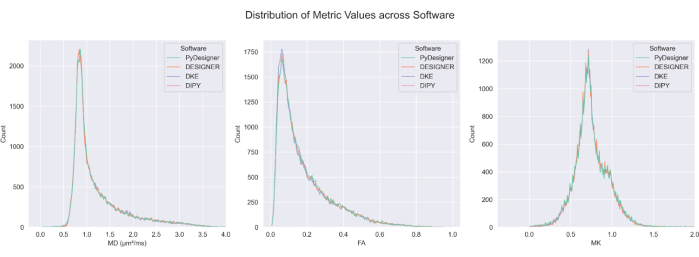
Figura 7: Comparación de FA, MD y MK entre tuberías. La distribución de los valores calculados para FA, MD y MK de PyDesigner, DESIGNER5, DKE27 y DIPY28 en cerebros excluidos de CSF es similar en la mayoría de los vóxeles. El cálculo del mapa métrico entre métodos es comparable. Esta figura ha sido reproducida con permiso de Dhiman et al.29. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La motivación principal para desarrollar PyDesigner fue implementar los elementos clave de DESIGNER mientras se reemplazaba todo el código de MATLAB con Python, lo que permitía una mayor portabilidad y accesibilidad. PyDesigner y DESIGNER Dv1 producen salidas casi idénticas29. No obstante, hay algunas opciones adicionales, configuraciones predeterminadas y correcciones de errores menores incluidas en PyDesigner. La documentación en línea de PyDesigner16 los describe en detalle.
PyDesigner también arroja resultados similares a las herramientas de análisis DKI Diffusional Kurtosis Estimator (DKE)27 y Diffusion Imaging in Python (DIPY)28 de uso común29 (ver Figura 6 y Figura 7), pero los mapas generados en base a la dMRI preprocesada por DESIGNER se consideran más precisos debido a su algoritmo de ajuste, como lo demuestran Ades-Aron et al.5. Consulte la Figura 6 para ver una comparación de los mapas de métricas MD, FA y MK de PyDesigner, DESIGNER5, DKE27 y DIPY28. En la figura 7 se muestra la comparación de los histogramas MD, FA y MK de cada canalización. La combinación de ajuste tensorial restringido, detección de valores atípicos y corrección del coeficiente de curtosis aparente produce un ajuste tensorial más robusto y preciso, como se ve en PyDesigner y DESIGNER5.
Una ventaja de PyDesigner sobre DESIGNER Dv1 es que está disponible a través del contenedor DockerDocker 30 de NeuroDock, lo que mejora en gran medida la portabilidad y simplifica la instalación. Este contenedor se ejecuta en todas las principales plataformas de sistemas operativos compatibles con Docker, incluidas Windows, Mac OS y varias distribuciones de Linux. En función de los comentarios de los usuarios, PyDesigner v2.0 incluirá actualizaciones del contenedor de Docker de PyDesigner. Las mejoras introducidas por la nueva versión de PyDesigner y la introducción de un Dockerfile de varias etapas resolverán todos los problemas existentes a los que se enfrentan los usuarios. Si los usuarios tienen problemas con la instalación, recomendamos enviar sus preguntas a la página de discusión16 de PyDesigner. La tecnología de contenedores de Docker también permite una implementación sencilla en clústeres de alto rendimiento (HPC) para el procesamiento por lotes de DWI rápidamente en clústeres locales compatibles con Docker.
PyDesigner también incluye cálculos de modelado microestructural que van más allá de DKI, incluidos WMTI3, FBI y FBWM. Para WMTI, un conjunto de datos DKI estándar es adecuado y los parámetros microestructurales asociados se calculan de forma predeterminada. Sin embargo, debe enfatizarse que la validez de WMTI está restringida a las regiones de sustancia blanca con altos AG (es decir, FA ≥ 0,4). Algunas métricas WMTI tienen una precisión limitada debido a la suposición de alineación paralela de los axones en cualquier vóxel31 dado. FBI 32,33,34 es un método distinto de dMRI aplicable en toda la sustancia blanca cerebral, que requiere un valor b alto (es decir, b ≥ 4000 s/mm 2) y datos de dMRI muestreados con un mínimo de 64 direcciones de codificación de difusión (junto con datos para b = 0). Las principales salidas del FBI son la función de densidad de orientación de la fibra (fODF) para cada vóxel de materia blanca, que se puede utilizar para la tractografía de la materia blanca y sirve como entrada para FBWM, así como la anisotropía fraccional intraaxonal (FAA). FBWM utiliza los datos de dMRI tanto de DKI como de FBI para estimar los mismos parámetros que WMTI, pero con mayor precisión, y se pueden aplicar en toda la materia blanca independientemente del valor de FA. Por lo tanto, si se dispone de estos datos adicionales, se prefieren las estimaciones de FBWM a las de WMTI35. Al igual que con el FBI, FBWM solo se ha validado en la sustancia blanca cerebral adulta.
Además de las invariantes rotacionales proporcionadas por PyDesigner (orientación RAS), los archivos .fib específicos de DSIstudio (orientación LPS) se producen por separado para DTI, DKI y FBI. Los archivos .fib contienen información direccional ODF para generar perfiles de tractografía para cada método. Los perfiles de tractografía resultantes se pueden utilizar para generar matrices de conectividad a nivel de sujeto y grupo. Los archivos .fib de DKI y FBI contienen información multidireccional que transmite información de fibra cruzada que no está disponible con la tractografía DTI. Además, dentro de cada fichero .fib se han incluido las distintas invariantes rotacionales de cada método, que pueden utilizarse en diversas combinaciones como criterio para sembrar, realizar y detener la tractografía. Más detalles sobre DSIstudio se pueden encontrar en su sitio web36.
Otra característica notable de PyDesigner es la entrada de múltiples archivos, que le permite manejar varias entradas de archivos: NifTi (.nii), NifTi comprimido (.nii.gz), DICOM (.dcm) y formato de archivo MRtrix (.mif). PyDesigner puede identificar automáticamente la información de adquisición de los metadatos del encabezado, independientemente del formato de entrada, y realizar las correcciones correspondientes, lo que permite un enfoque de no intervención. Independientemente de las diferencias en los protocolos, el mismo comando (ver arriba) se puede usar para procesar una amplia variedad de DWI. Por lo tanto, este software ahorra tiempo y esfuerzo al minimizar los pasos y comandos de preprocesamiento manual. En una actualización publicada recientemente (v1.0-RC10), esto se ha mejorado mediante la introducción de compatibilidad para múltiples conjuntos de datos de tiempo de eco (multi-TE). Esto permite a PyDesigner ejecutar pasos de preprocesamiento de imágenes, que son en gran medida independientes de TE, en un DWI multi-TE para producir una imagen con un ruido y artefactos mínimos. A continuación, se realizan cálculos de tensores dependientes de TE en cada TE por separado para producir métricas de difusión o curtosis.
Observamos que PyDesigner v1.0 no representa un punto final en el diseño de canalizaciones de preprocesamiento de imágenes. El desarrollo y la validación de herramientas de procesamiento de imágenes es un área activa de investigación. En particular, durante la preparación de este manuscrito, se propusieron nuevos desarrollos en la eliminación de ruido de imágenes y el zumbido de Gibbs, lo que resultó en el lanzamiento de una línea de Designer revisada, Designer Dv237, que incluye una eliminación de ruido mejorada y la corrección de artefactos de Gibbs para dMRI adquirida con adquisiciones parciales de Fourier35, y está disponible en el Github38 de DESIGNER. Del mismo modo, el cumplimiento organizacional de BIDS39 es una faceta importante de cualquier proceso de análisis de resonancia magnética. El formato BIDS permite un intercambio de datos y una implementación de canalizaciones más eficientes al establecer una estructura de conjunto específica para todos los tipos de datos de RMN. Aunque PyDesigner no es actualmente compatible con BIDS, una próxima versión de PyDesigner incluirá salidas compatibles con BIDS. Además, PyDesigner se escribió originalmente utilizando la versión 3.7 de Python. En el momento de esta publicación, Python 3.7 se considera obsoleto. Sin embargo, la información contenida en este manuscrito es independiente de la versión en Python. Es decir, los procedimientos descritos en este documento que son funciones importantes de preprocesamiento (por ejemplo, eliminación de ruido, corrección de timbre de Gibbs, etc.) seguirán el mismo flujo de trabajo operativo y conceptual independientemente de los cambios en los procedimientos de instalación. Es importante destacar que, al igual que con todo el software basado en Python, las futuras versiones de PyDesigner se actualizarán a una nueva versión de Python. Nos esforzamos por validar e integrar dichos desarrollos aún más a lo largo del tiempo.
Para obtener la versión más actualizada de PyDesigner, incluida cualquier nueva documentación relevante para las actualizaciones de la versión de Python, se recomienda a los lectores que consulten el sitio web antes de comenzar un nuevo análisis y participen en el foro de discusión donde se pueden enviar preguntas sobre PyDesigner16. La implementación de Docker para la portabilidad se denomina NeuroDock30, que contiene PyDesigner y sus dependencias para permitir el procesamiento en una amplia gama de plataformas.
Divulgaciones
Ninguno.
Agradecimientos
Estamos agradecidos por las útiles conversaciones con Olivia Horn, Daniel Linch y Graham Warner.
Las investigaciones reportadas en esta publicación fueron apoyadas, en parte, por las subvenciones de los Institutos Nacionales de la Salud R01AG054159, R01AG057602, R01AG055132, R01DC014021, R01NS110347, R21DA050085, F31NS108623, P20GM109040, P50DC000422, T32GM008716 y T32DC014435. El contenido es responsabilidad exclusiva de los autores y no representa necesariamente los puntos de vista oficiales de los Institutos Nacionales de Salud. La Fundación Litwin proporcionó fondos adicionales.
Materiales
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
Referencias
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados