Cuantificación de la muerte celular inducida por citocinas en organoides colónicos humanos mediante microscopía de fluorescencia en vivo
En este artículo
Resumen
Este protocolo describe un método simple y rentable para investigar y cuantificar la muerte celular en organoides colónicos humanos en respuesta a perturbaciones citotóxicas como las citocinas. El enfoque emplea un colorante fluorescente de muerte celular (SYTOX Green Nucleic Acid Stain), microscopía de fluorescencia en vivo y software de análisis de imágenes de código abierto para cuantificar las respuestas de un solo organoide a estímulos citotóxicos.
Resumen
La muerte de las células epiteliales intestinales (IEC) aumenta en pacientes con enfermedades inflamatorias intestinales (EII) como la colitis ulcerosa (CU) y la enfermedad de Crohn (EC). Esto puede contribuir a defectos en la función de la barrera intestinal, exacerbación de la inflamación e inmunopatogenia de la enfermedad. Las citocinas y los ligandos de los receptores de muerte son parcialmente responsables de este aumento en la muerte por IEC. Las citocinas relevantes para la EII, como el TNF-α y el IFN-γ, son citotóxicas para las IEC tanto de forma independiente como en combinación. Este protocolo describe un ensayo simple y práctico para cuantificar la citotoxicidad inducida por citocinas en organoides colónicos derivados de pacientes con EC utilizando un colorante fluorescente de muerte celular (SYTOX Green Nucleic Acid Stain), microscopía de fluorescencia en vivo y software de análisis de imágenes de código abierto. También demostramos cómo utilizar el modelo matemático de independencia de Bliss para calcular un coeficiente de interacción perturbagénica (CPI) basado en la citotoxicidad de los organoides. El CPI se puede utilizar para determinar si las interacciones entre combinaciones de citocinas u otros tipos de perturbágenos son antagónicas, aditivas o sinérgicas. Este protocolo se puede implementar para investigar la actividad citotóxica de citocinas y otros perturbágenos utilizando organoides colónicos derivados de pacientes.
Introducción
El epitelio intestinal crea una barrera física semipermeable entre el contenido de la luz intestinal y los tejidos subyacentes. Para mantener esta barrera de manera efectiva, las células epiteliales intestinales (IEC) se someten a un recambio celular extremadamente alto, con un ciclo continuo de muerte y regeneración celular. Sin embargo, durante los trastornos inflamatorios, como la enfermedad inflamatoria intestinal (EII), se producen niveles más altos de muerte celular aberrante1. Esto puede promover una ruptura en la función de barrera y la activación del sistema inmunológico, lo que desencadena una mayor inflamación. En la enfermedad de Crohn (EC), una forma de EII, se ha demostrado que la señalización de citocinas contribuye al aumento de los niveles de muerte por IEC2. Al estudiar cómo la señalización de citocinas induce la muerte celular de las IEC, se espera que se puedan desarrollar mejores tratamientos para los pacientes con EII y otros trastornos inflamatorios intestinales.
En biología y en la investigación de descubrimientos de dianas farmacológicas, se entiende generalmente que la sinergia se produce cuando un sistema biológico tratado con combinaciones de estímulos individuales muestra una respuesta a la combinación que es mayor que los efectos aditivos combinados de los estímulos individuales por sí solos. Las interacciones sinérgicas entre citocinas han sido bien documentadas en el impulso de las respuestas antivirales innatas3. También se sabe que las citocinas inducen la muerte celular de forma sinérgica, incluso en las IEC4. Sin embargo, el papel que desempeña la señalización sinérgica de citocinas citotóxicas en los trastornos inflamatorios intestinales como la EII está poco estudiado.
Los organoides intestinales humanos son microtejidos 3D producidos in vitro que se generan a partir de células madre epiteliales intestinales. Los organoides intestinales pueden cultivarse a partir de biopsias de la mucosa intestinal obtenidas de pacientes con EII y conservan muchas características de la enfermedad 5,6. Los organoides han demostrado ser un sistema modelo ideal para estudiar la citotoxicidad de las citocinas en el contexto de la inflamación intestinal 7,8. Previamente, nuestro grupo ha caracterizado los efectos matales sinérgicos de las citocinas IFN-γ y TNF-α relevantes para la EII en organoides colónicos (colonoides) derivados de pacientes con ECA9,10. Sin embargo, los mecanismos exactos implicados en la mediación de esta forma de muerte celular sinérgica siguen siendo difíciles de alcanzar. También hay potencialmente muchas más interacciones de citocinas citotóxicas no caracterizadas que son relevantes para los trastornos inflamatorios intestinales.
Existen varios protocolos para estudiar la muerte celular en organoides intestinales 10,11,12,13; Sin embargo, cada uno de ellos tiene inconvenientes. Algunas de estas técnicas solo miden la viabilidad celular y no miden la muerte celular directamente, son incapaces de evaluar las respuestas de un solo organoide o requieren equipos costosos y protocolos complejos. Se necesitan metodologías sólidas y sencillas para cuantificar la muerte celular de los organoides y las interacciones de las perturbaciones en los organoides intestinales. El protocolo que presentamos es un enfoque simple y económico para medir las respuestas de un solo organoide a citocinas citotóxicas, pero se puede utilizar para cualquier tipo de estímulo o perturbágeno. También demostramos cómo utilizar el modelo de sinergia de independencia de Bliss para calcular un coeficiente de interacción de perturbaciones (CPI) que describe las interacciones de citocinas citotóxicas.
Protocolo
Se recolectaron biopsias de la mucosa colónica de pacientes con EC sometidos a colonoscopia de rutina como parte del tratamiento estándar. La aprobación ética para el uso de muestras de tejido de pacientes y la generación de líneas de organoides colónicos a partir de estas muestras se obtuvo del Comité de Ética de Investigación Clínica de los Hospitales Universitarios de Cork (CREC). Se obtuvo el consentimiento informado por escrito de todos los pacientes de acuerdo con la Declaración de Helsinki. Todos los trabajos de cultivo de tejidos con biopsias de pacientes y colonoides deben realizarse dentro de una cabina de bioseguridad siguiendo los protocolos de seguridad BSL2. Asegúrese de que todo el uso de plástico sea estéril antes de usarlo. Consulte la Tabla de materiales para obtener detalles relacionados con todos los materiales, reactivos, instrumentos y software utilizados en este protocolo.
Los protocolos que utiliza nuestro grupo para el aislamiento de criptas y el cultivo de organoides fueron adaptados de los métodos establecidos 14,15,16 y han sido publicados previamente 9,10,17. Para el siguiente protocolo, los colonoides se cultivaron utilizando medios de proliferación de organoides (Tabla 1). Los colonoides cultivados con medios de proliferación de organoides son indiferenciados y están enriquecidos para obtener células madre colónicas. El componente principal de los medios de proliferación de organoides es un 50% de medios acondicionados con L-WRN, que contiene los factores de crecimiento del nicho de células madre intestinales Wnt-3A (W), R-espondina 3 (R) y Noggin (N)15. Los medios de proliferación de organoides se preparan combinando medios acondicionados con L-WRN y medios sin suero 1:1, seguidos de suplementación con nicotinamida e inhibidores químicos (Tabla 1).
1. Aislamiento de criptas colónicas y cultivo de colonoides
- Preincubar una placa de microtitulación de 48 pocillos a 37 °C, 5% de CO2 durante un mínimo de 72 h antes de sembrar con criptas.
NOTA: La preincubación de la placa acelera la polimerización del extracto de membrana basal (BME) durante la siembra. - Descongelar el BME en hielo a 4 °C la noche anterior al aislamiento de las criptas.
- Recoja las biopsias de colon en un tubo de recolección de muestras que contenga 15 mL de medio de recolección de biopsia (Tabla 1) y almacene a 4 °C hasta que esté listo para el procesamiento.
- Extraiga con cuidado la mayor cantidad posible de medio de recolección de biopsia con una pipeta. Lave las biopsias añadiendo 15 mL de DPBS helado suplementado con 2,5 μg/mL de anfotericina B y 100 μg/mL de gentamicina al tubo de muestra. Agite vigorosamente el tubo de muestra para disociar cualquier mucosa o residuo de las biopsias. Deje que las biopsias se asienten por gravedad y extraiga cuidadosamente la mayor cantidad posible de DPBS con una pipeta.
- Repita el procedimiento de lavado del paso 1.4 dos veces (2x).
- Añadir 10 mL de reactivo de disociación celular libre de enzimas suplementado con 2,5 μg/mL de anfotericina B y 200 μg/mL de gentamicina al tubo de muestra e incubar durante 15 min a temperatura ambiente con balanceo a 30 rpm.
- Después de la incubación, agite vigorosamente el tubo de muestra de lado a lado con la mano para liberar las criptas colónicas. Inspeccione el tubo con un microscopio óptico de baja potencia y busque las criptas liberadas y los fragmentos de criptas en suspensión. Si no es visible, agite el tubo y vuelva a comprobarlo; Repita hasta que las criptas se vean en suspensión.
- Conecte un filtro de células de 70 μm a un tubo de 50 ml y filtre la suspensión de la cripta a través del filtro. Agregue 10 ml de medio de lavado de organoides helado (Tabla 1) al tubo de muestra vacío, retire el medio y páselo a través del filtro de células.
- Transfiera las criptas filtradas a dos tubos de 15 ml (10 ml por tubo) y centrifugue a 4 °C durante 5 minutos a 150 × g.
- Retire el sobrenadante con cuidado de cada tubo de 15 ml, vuelva a suspender los gránulos de la cripta en 500 μL de medios de lavado de organoides helados, transfiera la solución de la cripta de ambos tubos de 15 ml a un solo tubo de microcentrífuga de 1,5 ml y centrifugue a 4 °C durante 3 minutos a 400 × g.
- Retire con cuidado el sobrenadante del tubo de microcentrífuga y vuelva a suspender el pellet de la cripta en 70 μL de BME (20 μL por pocillo y 10 μL de volumen muerto extra).
- Usando la placa preincubada de 48 pocillos (paso 1.1), siembre 20 μL de suspensión de BME/cripta en el centro de cada pocillo (1 domo BME por pocillo). Invierta la placa de forma suave y constante e incube a 37 °C, 5% de CO2 durante 20 min.
NOTA: La inversión de la placa evita que las células se adhieran a la superficie plástica del pocillo y asegura su distribución dentro del BME. - Retire la placa de la incubadora. Asegúrese de que el BME se haya polimerizado completamente y, a continuación, superponga los domos con 350 μL de medios de proliferación de organoides precalentados suplementados con 100 μg/mL de un reactivo antimicrobiano de amplio espectro. Incubar la placa a 37 °C, 5% de CO2.
NOTA: El reactivo antimicrobiano de amplio alcance es para prevenir la contaminación de los microbios de la mucosa asociados con las biopsias de colon. Solo se requiere durante la primera semana de cultivo después del aislamiento de la cripta. - Cambie el medio 2-3 veces en una semana utilizando medios de proliferación de organoides precalentados, complementando con el reactivo antimicrobiano de amplio espectro durante la primera semana de cultivo; Después de este período, retire el reactivo.
- Una vez que el cultivo de colonoides esté completamente establecido (1-2 semanas después del aislamiento), disocie los colonoides utilizando el reactivo de disociación enzimática suplementado con 10 μM del inhibidor ROCK-I/II Y-27632 (ver sección 2 para detalles completos sobre la disociación colonoide).
NOTA: Para los dos primeros pasajes (P0-1, P1-2), los colonoides deben expandirse usando una proporción de 1:1/2; después de P2, los colonoides se pueden pasar usando una proporción de 1:3/4. - Siembre y mantenga los colonoides siguiendo los pasos 1.11-1.13 (no complemente los medios de proliferación de organoides con el reactivo antimicrobiano de amplio rango).
2. Preparación de colonoides para el ensayo de muerte celular
NOTA: El protocolo de ensayo de muerte celular tarda 4 días en completarse (Figura 1A).
- Expanda los colonoides utilizando un formato de placa de 48 pocillos, sembrando 20 μL de suspensión de BME/cripta por pocillo, incubando a 37 °C, 5% de CO2 y cambiando el medio 2-3 veces por semana (350 μL por pocillo).
- Pase los colonoides aproximadamente 1 semana antes de la recolección para el ensayo de muerte celular. Antes de cosechar los colonoides, asegúrese de que se propaguen a una alta densidad (Figura 1Bi), que tengan aproximadamente 25-50 μm de diámetro y que estén proliferando activamente.
NOTA: Hemos utilizado colonoides para este ensayo desde el pasaje 3 hasta el pasaje 14 con resultados consistentes. Sin embargo, se ha demostrado que la respuesta transcripcional de los colonoides a las citocinas puede cambiar dependiendo de la duración del cultivo18. Sobre esta base, recomendamos no utilizar colonoides para este ensayo después del paso 15. - Preincubar placas de microtitulación de 96 pocillos a 37 °C, 5% de CO2 durante un mínimo de 72 h antes de sembrar con células colonoides.
- Descongele el BME en hielo a 4 °C la noche anterior al inicio del experimento.
- Prepare un volumen suficiente del reactivo de disociación enzimática (500 μL por pocillo de colonoides) suplementando con 10 μM del inhibidor ROCK-I/II Y-27632. Retire suavemente el medio de los pocillos que contienen colonoides; pipetea desde el borde del pocillo para evitar dañar la cúpula del colonoide. Añadir 300 μL de reactivo de disociación enzimática a cada pocillo.
- Para cada pocillo, rompa la cúpula del colonoide raspando la superficie del pocillo con la punta de una pipeta P1000 y pipeteando la suspensión celular hacia arriba y hacia abajo; Trate de evitar generar burbujas de aire. Recoja la suspensión celular en un tubo de 15 mL.
NOTA: Este tubo de 15 mL se utilizará para recolectar colonoides de 10 pocillos de la placa de 48 pocillos. - Lave el mismo pocillo con otros 200 μL del reactivo de disociación enzimática para asegurarse de que todo el material colonoide se recoja y transfiera al mismo tubo de 15 mL. Repita este proceso para cada pocillo de colonoides expandidos que se esté cosechando y recójalos en el mismo tubo de 15 ml.
NOTA: Para una disociación eficiente, se debe recolectar un máximo de 10 pocillos de colonoides por tubo. Si recolecta >10 pocillos, divida los colonoides recolectados en partes iguales en varios tubos de 15 ml.
- Para cada pocillo, rompa la cúpula del colonoide raspando la superficie del pocillo con la punta de una pipeta P1000 y pipeteando la suspensión celular hacia arriba y hacia abajo; Trate de evitar generar burbujas de aire. Recoja la suspensión celular en un tubo de 15 mL.
- Incubar el tubo de 15 ml con los colonoides recogidos del paso 2.5.2 en un baño de agua a 37 °C durante 5 min. Después de la incubación, centrifugar el tubo durante 3 min a 400 × g. Retire suavemente el sobrenadante, dejando aproximadamente 1,2 ml en el tubo.
NOTA: El siguiente paso requiere la disociación física de los colonoides mediante pipeteo rápido. Para que esto sea efectivo, debe tener un pequeño volumen de suspensión celular en el tubo: los 1,2 ml que quedan en el tubo de 15 ml son suficientes para este propósito. - Vuelva a suspender el gránulo de colonoide en los 1,2 mL restantes del reactivo de disociación enzimática. Con una pipeta P1000 ajustada a 1.000 μL, coloque la punta de la pipeta en la suspensión, sosteniéndola justo por encima de la parte inferior del tubo de 15 mL y, a continuación, pipetee rápidamente la suspensión dentro y fuera de la punta. Para pipetear rápidamente, presione rápidamente el botón del émbolo hasta el primer tope, suelte el botón hasta que esté aproximadamente a la mitad de la posición superior y repita. Pipetear rápidamente durante aproximadamente 10 s (30-40 depresiones), luego vuelva a suspender la suspensión por completo y repita el pipeteo. Realice 2-3 rondas de pipeteo rápido.
- Examinar la muestra al microscopio para confirmar que no quedan colonoides enteros y que la mayoría de los fragmentos de colonoides tienen un tamaño aproximado de 30-40 μm (Figura 1Bii).
- Si la muestra requiere una disociación adicional, incubar en el baño de agua a 37 °C durante otros 3 minutos, repetir la técnica de pipeteo en el paso 2.7 y examinar la muestra bajo el microscopio. Repita este proceso hasta que la mayoría de los fragmentos de colonoides tengan el tamaño óptimo.
NOTA: Tenga cuidado de no disociar demasiado los colonoides, ya que esto resultará en una muerte celular excesiva, baja eficiencia de siembra y colonoides de tamaño insuficiente. - Agregue 10 mL de Organoid Wash Media Helado (Tabla 1) al tubo de 15 mL. Centrifugar el tubo durante 3 min a 400 × g, retirar el sobrenadante, volver a suspender en 1 mL de medio de lavado de organoides helado y transferir a un tubo de microcentrífuga de 1,5 mL (denominado tubo de fragmento de colonoide).
- Mezclar el contenido del tubo del fragmento de colonoide mediante pipeteo y tomar una muestra de 50 μL; transfiera esta muestra de 50 μL a un nuevo tubo de microcentrífuga de 1,5 mL ( denominado tubo de recuento de células). Almacene el tubo de fragmento de colonoide en hielo desde este punto hasta la finalización del paso 2.15.
- Centrifugar el tubo de recuento de células durante 3 min a 400 × g, retirar el sobrenadante y resuspender en 500 μL de reactivo de disociación enzimática suplementado con 10 μM del inhibidor ROCK-I/II Y-27632.
- Incubar el tubo de recuento de células individuales en el baño de agua a 37 °C durante 5 min. Con una pipeta P1000 ajustada a 400 μL, pipetee la muestra como se describe en el paso 2.7 y compruebe la muestra al microscopio para asegurarse de que hay una suspensión unicelular. De lo contrario, repita este proceso hasta que los colonoides estén completamente disociados en una suspensión de una sola célula.
- Añada 1 ml de Organoid Wash Media al tubo de recuento de células individuales, centrifugue durante 3 min a 400 × g y retire con cuidado el sobrenadante. Vuelva a suspender las células en 50 μL de Organoid Wash Media y luego agregue 50 μL de azul de tripano.
- Cuente las células con un hemocitómetro. Calcule el número de células en la muestra de 50 μL y utilícelo para calcular la concentración de células en el tubo del fragmento de colonoide.
- Calcule el volumen de suspensión celular necesario para el experimento en el que se sembrarán 0,5 × 104 células colonoides por pocillo de una placa de microtitulación de 96 pocillos. Agregue aproximadamente un 15% adicional a este volumen calculado para tener en cuenta el volumen muerto. Transfiera este volumen total del tubo de fragmento de colonoide (paso 2.11) a un nuevo tubo de microcentrífuga de 1,5 mL.
- Centrifugar el nuevo tubo de microcentrífuga de 1,5 mL que contiene fragmentos de colonoides durante 3 min a 400 × g, retirar el sobrenadante y volver a suspender en BME (utilizar 10 μL de BME por 0,5 × 104 células).
NOTA: Debido a las características físicas del BME (polimerización de alta viscosidad y dependiente de la temperatura), se puede perder una cantidad significativa de material a través del pipeteo (volumen muerto).
- Centrifugar el nuevo tubo de microcentrífuga de 1,5 mL que contiene fragmentos de colonoides durante 3 min a 400 × g, retirar el sobrenadante y volver a suspender en BME (utilizar 10 μL de BME por 0,5 × 104 células).
- Coloque el tubo o depósito que contiene la suspensión de colonoide/BME en hielo en un recipiente estéril dentro del gabinete de bioseguridad.
NOTA: Mantener las células en hielo durante la siembra evita que el sustrato celular se polimerice prematuramente. - Utilizando una placa de microtitulación preincubada de 96 pocillos (del paso 2.3), pipetee inversamente 10 μL de la solución de colonoide/BME por pocillo. Asegúrese de colocar la punta justo por encima de la superficie del pocillo y pipetear en el centro para evitar golpear la pared del pocillo. Mezcle la suspensión de colonoide/BME con regularidad para evitar la siembra desigual.
NOTA: No siembre colonoides en los pocillos del borde exterior de la placa de microtitulación.- Para pipeta inversa:
- Coloque la pipeta y, a continuación, presione el botón del émbolo más allá de la primera parada hasta la segunda parada.
- Manteniendo esta posición, sumerja la punta en la suspensión de colonoide/BME y suelte lentamente el émbolo hacia la parte superior.
- Dispense la suspensión presionando el botón del émbolo suave y constantemente hasta el primer tope. Si siembra más pozos, mantenga esta posición y repita los pasos 2.18.1.2-2.18.1.3.
- Una vez terminado, expulse la pequeña cantidad de suspensión restante presionando el botón del émbolo hasta el segundo tope.
NOTA: Recomendamos utilizar la técnica de pipeteo inverso debido a la alta viscosidad del BME.
- Para pipeta inversa:
- Invierta la placa de forma suave y constante e incube a 37 °C, 5% de CO2 durante 20 min.
- Retire la placa de la incubadora. Asegúrese de que el BME se haya polimerizado completamente y, a continuación, superponga los domos con 200 μL de medios de proliferación de organoides precalentados. Si usa una placa de microtitulación de 96 pocillos con un foso circundante (que reduce la evaporación), llene cada depósito con 2 mL de medios de lavado de organoides.
- Incubar los colonoides a 37 °C, 5% CO2 durante 3 días e inspeccionar microscópicamente una vez al día para asegurarse de que los colonoides se han recuperado y están proliferando.
3. Tratamientos colonoides para el ensayo de muerte celular
- Prepare una solución de 2,5 μM de colorante de muerte celular fluorescente (tinción de ácido nucleico verde SYTOX) y solución de DMSO agregando colorante de muerte celular fluorescente o DMSO a los medios de proliferación de organoides precalentados (Tabla 2).
NOTA: Proteja de la luz el colorante de muerte celular fluorescente y la solución diluida. La tinción de ácido nucleico verde SYTOX se solubiliza en DMSO; la solución de DMSO se utiliza para preparar la condición de No Dye para controlar los efectos del solvente. - Utilice la solución de 2,5 μM de colorante de muerte celular fluorescente y la solución de DMSO del paso 3.1 para preparar los tratamientos como se muestra en la Tabla 2.
- Retire suavemente el medio de la placa de microtitulación de 96 pocillos sembrada con colonoides inclinando la placa y pipeteando desde el borde de los pocillos; luego, agregue 200 μL de medio de tratamiento por pocillo. Asegúrese de tener pozos de control PBS/BSA adicionales para las condiciones de toxicidad máxima (consulte la Tabla 2 para ver el mapa de placas).
- Incubar los colonoides a 37 °C, 5% de CO2 durante los puntos de tiempo de tratamiento requeridos hasta que estén listos para la obtención de imágenes.
- Al menos 2 h antes de la toma de imágenes, prepare una solución al 10% v/v de Triton-X 100 en agua estéril de grado de cultivo celular y agregue 22 μL del Triton-X 100 al 10% directamente al medio de los pocillos de control para las condiciones de toxicidad máxima hasta una concentración final de 1% v/v 1 h antes de la imagen.
NOTA: El uso de una solución al 10% reduce los errores de pipeteo que pueden ocurrir debido a la alta viscosidad del surfactante.
4. Adquisición de imágenes
- Retire la placa de microtitulación de 96 pocillos sembrada con colonoides de la incubadora y transfiérala a la etapa de un microscopio digital de epifluorescencia invertida. Deje que el plato alcance la temperatura ambiente.
- Confirme que los colonoides en condiciones de toxicidad máxima están completamente lisados examinándolos bajo el microscopio (Figura 1Biii).
- Seleccione un objetivo adecuado, como un objetivo de fluorescencia de 40x a larga distancia de trabajo. Optimice la configuración de imagen del microscopio antes de comenzar.
- Usando el canal de transmisión, concéntrese en un colonoide con células SYTOX positivas, cambie a la proteína verde fluorescente (GFP)
(488 nm) y ajuste la intensidad de la luz y el tiempo de exposición para maximizar la señal fluorescente y minimizar el fondo. Primero, pruebe una intensidad de luz baja, luego aumente gradualmente el tiempo de exposición; Si la duración de la exposición no es práctica, aumente ligeramente la intensidad de la luz.
NOTA: Aumentar el tiempo de exposición en lugar de la intensidad de la luz reducirá la fototoxicidad y el fotoblanqueo de las muestras19. Exponga las muestras a la luz fluorescente solo cuando sea necesario.
- Usando el canal de transmisión, concéntrese en un colonoide con células SYTOX positivas, cambie a la proteína verde fluorescente (GFP)
- Utilizando la configuración de imagen optimizada para el canal GFP, observe las condiciones Sin colorante y Toxicidad máxima para asegurarse de que las muestras no estén sobreexpuestas o subexpuestas. Una vez finalizado, mantenga la configuración de imágenes consistente entre las condiciones.
- Adquiera imágenes utilizando un enfoque de muestreo aleatorio: hágalo seleccionando campos de visión (FOV) que sigan un patrón de cuadrícula fija que cubra la cúpula del colonoide. Adquiera imágenes de colonoides de un mínimo de 10 FOV. Asegúrese de que el plano central del colonoide esté enfocado y adquiera imágenes tanto en los canales de transmisión como en los canales GFP.
- Aplique los siguientes criterios de exclusión.
- No adquiera imágenes si no hay colonoides presentes en el FOV.
- No adquiera imágenes si solo hay colonoides que se superponen en el mismo plano focal presente en el campo de visión (Figura 1Biv).
- No adquiera imágenes si solo hay restos de colonoides presentes en el campo de visión (Figura 1Bv). No incluya restos de colonoides en el análisis.
- Aplique los siguientes criterios de exclusión.
- Guarde imágenes en formato gráfico de red portátil y expórtelas.
- Si toma imágenes de puntos de tiempo adicionales, devuelva la placa de microtitulación de 96 pocillos con colonoides a la incubadora a 37 °C, 5% de CO2.
5. Análisis de imágenes
- Abra Fiji ImageJ e importe el conjunto de datos de imagen arrastrando y soltando los archivos en la barra de herramientas de ImageJ o navegando a Archivo | Abra y seleccione los archivos. Una vez abiertos, combine los archivos en una pila de imágenes haciendo clic en Imagen| Pilas| Imágenes para apilar. Convierta la pila de imágenes a un formato de archivo de 8 bits haciendo clic en Imagen| Tipo| 8 bits.
- Para cada conjunto de imágenes, haga clic en la herramienta Selecciones a mano alzada en la barra de herramientas de ImageJ y seleccione manualmente la región de interés (ROI) en la imagen de transmisión con el mouse de la computadora; el ROI es el perímetro del colonoide. A continuación, desplácese por la pila de imágenes a la imagen de canal GFP correspondiente.
- Haga clic en Analizar| Establecer medidas; en la ventana de diálogo Establecer mediciones , marque Valor medio de gris y deje todas las demás casillas sin marcar. Con la imagen GFP seleccionada, haga clic en Analizar | Medir. Repita este análisis para cada colonoide de la pila de imágenes. Una vez analizado el conjunto de datos, copie todos los datos de la ventana Resultados y péguelos en una aplicación de software de hoja de cálculo.
6. Cálculo del % de toxicidad máxima
- Calcule la media de los valores medios de gris (MGV) replicados técnicamente para cada condición utilizando la ecuación (1).
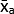 (media del tratamiento a) =
(media del tratamiento a) = 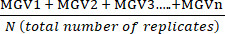 (1)
(1) - Exprese la media de cada condición como porcentaje en relación con la media de la condición de toxicidad máxima (MT) utilizando la ecuación (2).
%MTa =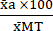 (2)
(2)
7. Cálculo del IPC
- Normalice (NORM) los datos de la siguiente manera, utilizando los valores medios calculados en el paso 6.1, reste la media de la condición no tratada (UT) de cada condición de tratamiento y la condición de toxicidad máxima (MT) (para eliminar la muerte celular de fondo que ocurre independientemente del tratamiento con citocinas). A continuación, divida cada condición de tratamiento por la media restada de fondo de la condición de toxicidad máxima (MT) como se muestra en la ecuación (3).
NORMAa =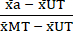 (3)
(3) - Reste los valores normalizados calculados en el paso 7.1 de 1; los valores resultantes representan la viabilidad celular (V) después del tratamiento (como en la ecuación (4) a continuación).
Va = 1 −NORMAa (4) - Calcule el coeficiente de interacción de perturbaciones (CPI) utilizando la ecuación (5):
IPC =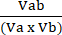 (5)
(5)
Donde a denota el primer tratamiento; b denota el segundo tratamiento; y AB es el tratamiento combinado. Los valores de CPI indican relaciones sinérgicas (<1), aditivas (=1) o antagónicas (>1).
Resultados Representativos
Utilizando este protocolo, demostramos cómo se pueden utilizar los colonoides de pacientes con EC para estudiar los efectos citotóxicos de las citocinas relevantes para la EII IFN-γ y TNF-α en el epitelio primario. Utilizamos un colorante fluorescente de muerte celular disponible en el mercado (SYTOX Green Nucleic Acid Stain), que solo puede entrar en las células que tienen una membrana celular comprometida, donde luego se activa uniéndose a los ácidos nucleicos. Cotratamos colonoides con citocinas y el colorante de muerte celular fluorescente y realizamos imágenes de células vivas a las 8 h y 24 h con un microscopio de epifluorescencia invertida. Las imágenes representativas de transmisión/superposición fluorescente a las 8 h indican que solo los colonoides tratados con IFN-γ + TNF-α son positivos para la señal fluorescente; sin embargo, solo hay un pequeño número de células fluorescentes (Figura 2A). La blebbing celular, un indicador morfológico de la muerte celular20, también se puede observar en la condición IFN-γ + TNF-α. A las 24 h, los colonoides tratados con IFN-γ + TNF-α muestran grandes regiones positivas para la señal fluorescente (Figura 2A). También hay una clara ruptura en la morfología del colonoide: la luz central ya no es visible y la barrera epitelial se ha interrumpido por completo.
Para cuantificar la señal del colorante de muerte celular, utilizamos un software de análisis de imágenes de código abierto para calcular la intensidad fluorescente de cada colonoide. A continuación, normalizamos los datos expresando la media de cada enfermedad como porcentaje del tratamiento con toxicidad máxima. A las 8 h, la muerte celular homeostática o de fondo en los colonoides control BSA fue relativamente baja (7,7% de toxicidad máxima) (Figura 2B). No hubo cambios estadísticamente significativos en los niveles de muerte celular en este momento; sin embargo, las condiciones tratadas con TNF-α mostraron un pequeño aumento en la citotoxicidad (Figura 2B). Después de 24 h, los niveles de muerte celular habían aumentado para todas las afecciones tratadas con citocinas. Sin embargo, hubo un cambio mínimo en la muerte celular para la condición de control de BSA entre puntos de tiempo (7,5% de toxicidad máxima a las 24 h). Los colonoides tratados con IFN-γ + TNF-α tuvieron el mayor aumento en los niveles de muerte celular en comparación con el control de BSA (29,4% de toxicidad máxima). La diferencia en los niveles de muerte celular entre el tratamiento combinado y los tratamientos con citocinas únicas (IFN-γ, TNF-α) fue altamente significativa. Estos resultados sugieren la posibilidad de una interacción sinérgica citotóxica entre el IFN-γ y el TNF-α a las 24 h.
Utilizamos el CPI para cuantificar las interacciones citotóxicas entre los tratamientos con citocinas y determinar si eran sinérgicas. Las interacciones entre citocinas se consideran sinérgicas cuando el valor de CPI es <1, aditivas cuando =1 o antagónicas cuando >1. Se calcularon los valores del IPC por punto de tiempo (Figura 2C). A las 8 h, el valor del IPC indicó un ligero sinergismo (0,99), disminuyendo sustancialmente a las 24 h (0,83). Este análisis confirmó que la interacción entre el IFN-γ y el TNF-α a las 24 h fue sinérgica. Además, ilustra cómo, en este contexto, la sinergia entre el IFN-γ y el TNF-α depende del tiempo.
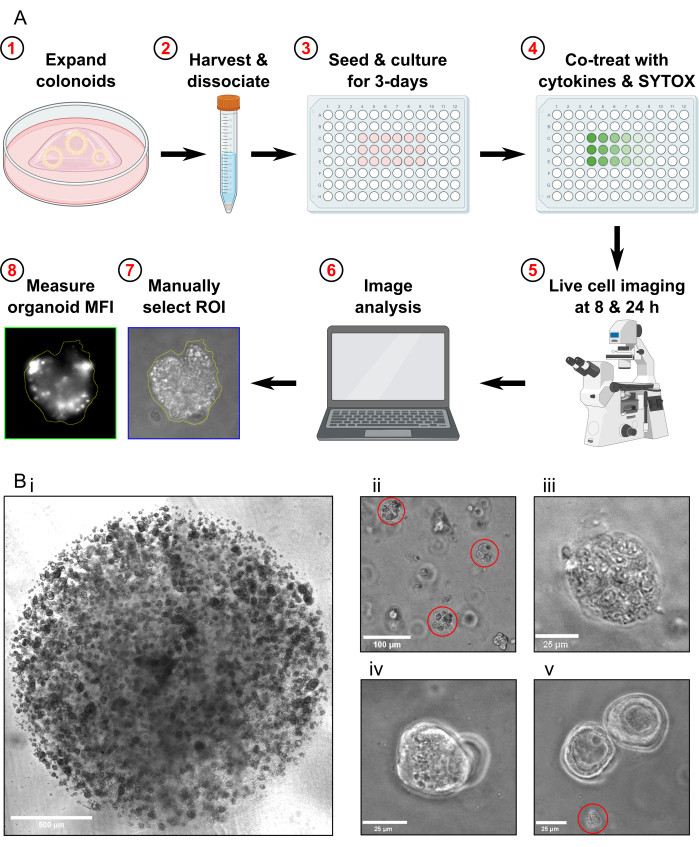
Figura 1: Esquema del flujo de trabajo experimental y resolución de problemas. (A) Descripción general esquemática del protocolo. (B) Imágenes representativas. (Bi) Imagen de microscopía óptica que ilustra la densidad óptima del cultivo y el tamaño óptimo de los colonoides antes de pasar para un ensayo. Barra de escala = 500 μm. (Bii) Imagen de microscopía óptica que ilustra el tamaño óptimo de los fragmentos de colonoides después de la disociación; Fragmentos resaltados en rojo. Barra de escala = 100 μm. (Biii) Imagen de microscopía óptica de la morfología del colonoide necrótico después del tratamiento con MT (con Triton X-100). Barra de escala = 25 μm. (Biv) Imagen de microscopía óptica de dos colonoides superpuestos en el mismo plano focal. Barra de escala = 25 μm. (Bv) Imagen de microscopía óptica de restos de células colonoides presentes después del paso; Escombros resaltados en rojo. Barra de escala = 25 μm. Abreviaturas: ROI = región de interés; MFI = intensidad media de fluorescencia; MT = Toxicidad máxima. Haga clic aquí para ver una versión más grande de esta figura.
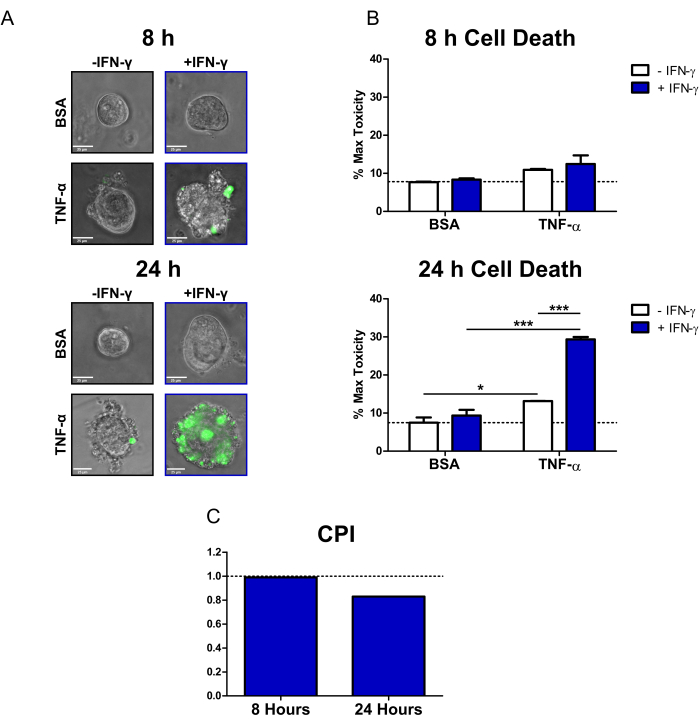
Figura 2: Análisis cuantitativo de la muerte celular inducida por citocinas en colonoides CD humanos. (A) Imágenes representativas de microscopía en vivo de colonoides CD tratados con tinción de ácido nucleico verde SYTOX (colorante fluorescente de muerte celular) y citocinas a las 8 h y 24 h; canales de transmisión y GFP (color verde) superpuestos. Los colonoides se trataron de la siguiente manera: 1) PBS/BSA, 2) 10 ng/mL de IFN-γ, 3) 10 ng/mL de TNF-α, 4) 10 ng/mL de IFN-γ + 10 ng/mL de TNF-α. Barras de escala = 25 μm. (B) Análisis cuantitativo de colonoides CD tratados con el colorante de muerte celular fluorescente y citocinas a las 8 y 24 h; los datos se expresan como un % de la condición MT. N = 2 líneas de colonoides CD, de 11 a 16 colonoides por afección. (C) IPC calculado por punto de tiempo utilizando el conjunto de datos de B, N = 2 líneas de colonoides CD. Los datos se expresan como medio ± SE. En B, se realizó un análisis de ANOVA de dos vías seguido de post-test de Bonferroni, *P < 0,05, ***P < 0,001 como se indicó. Abreviaturas: CD = enfermedad de Crohn; GFP = proteína verde fluorescente; CPI = coeficiente de interacción de perturbaciones; PBS = solución salina tamponada con fosfato; BSA = albúmina sérica bovina; TNF-α = factor de necrosis tumoral alfa; IFN-γ = interferón-gamma; MT = Toxicidad máxima. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Composición de los medios de cultivo para protocolo. Para preparar los medios de proliferación de organoides, combine los medios acondicionados con L-WRN y los medios sin suero 1:1, luego agregue suplementos. Los medios de proliferación de organoides deben usarse dentro de las 2 semanas posteriores a la preparación. Tenga en cuenta que todos los medios completos deben almacenarse a 4 °C. Haga clic aquí para descargar esta tabla.
Tabla 2: Diseño experimental de placas de 96 pocillos y tratamientos con citocinas. Haga clic aquí para descargar esta tabla.
Discusión
Se han desarrollado varios métodos para el análisis cuantitativo de la muerte celular en organoides intestinales. El examen de la alteración de la morfología de los organoides intestinales mediante microscopía óptica es un enfoque sencillo para cuantificar los efectos de las sustancias citotóxicas11. Sin embargo, los cambios morfológicos no son una medida directa de la muerte celular, y el método es solo semicuantitativo. Otro método es evaluar la actividad metabólica de los organoides mediante un ensayo MTT o ATP10,11. Es importante tener en cuenta que estos ensayos solo pueden determinar cambios en la viabilidad celular y deben validarse con un ensayo de muerte celular. Se han descrito otros ensayos fluorométricos de muerte celular utilizando colorantes de unión al ADN12,13. Es posible un enfoque sin imagen que utilice un lector de microplacas fluorescentes y permite un alto rendimiento12. Sin embargo, este método mide la señal promedio de un pozo completo, por lo que no es adecuado para poblaciones heterogéneas. También requiere el uso de un lector de microplacas con ajuste de altura Z. Las técnicas basadas en imágenes fluorescentes se pueden utilizar para el análisis de organoides individuales y la captura de datos celulares/subcelulares y morfológicos. Los sistemas automatizados de imágenes confocales de alto contenido (HCI) pueden generar grandes cantidades de datos con un alto rendimiento13. Desafortunadamente, la HCI confocal necesita equipos especializados, utiliza protocolos complejos, generalmente requiere software comercial de análisis de imágenes y es costosa.
Nuestro protocolo para el análisis cuantitativo de la muerte de células colonoides en múltiples puntos de tiempo es sencillo, sencillo y económico. Sin embargo, en comparación con los sistemas automatizados de HCI y lectores de placas, requiere mucho tiempo y ha reducido el rendimiento. Otra limitación de nuestro método es el uso de microscopía de campo amplio en lugar de confocal. La microscopía confocal es más adecuada para obtener imágenes de muestras 3D gruesas, como organoides, ya que reduce la señal desenfocada y puede adquirir secciones ópticas en serie (pilas Z). Sin embargo, las imágenes confocales suelen requerir tiempos de adquisición más largos y láseres de alta intensidad que aumentan la fototoxicidad/fotoblanqueo. Es fundamental tener en cuenta que los colorantes fluorescentes de muerte celular como SYTOX Green solo son adecuados para medir formas de muerte celular en las que hay pérdida de la integridad de la membrana celular, como la necrosis, la necrosis secundaria asociada a la apoptosis tardía, la necroptosis y la piroptosis21. Existen algunas formas de muerte celular regulada en las que la membrana celular permanece impermeable al menos durante las primeras fases de la muerte celular, como la apoptosis dependiente de caspasas. Sin embargo, este protocolo podría modificarse fácilmente para incorporar también la obtención de imágenes de un indicador fluorescente de actividad 3/7 de caspasa22. Esto proporcionaría datos adicionales para ayudar a caracterizar la modalidad específica de muerte celular.
Utilizamos nuestro protocolo para demostrar la interacción sinérgica citotóxica entre las citocinas IFN-γ y TNF-α (Figura 2C), que hemos descrito previamente en organoides derivados de pacientes con EC 9,10. La relevancia fisiológica de esta forma de sinergismo también ha sido demostrada en modelos murinos de linfohistiocitosis hemofagocítica y sepsis23. Se han implementado varios modelos y enfoques matemáticos de referencia para cuantificar la sinergia entre combinaciones de agentes biológicos24,25. Difieren en cuanto a su complejidad, el número de factores que consideran y el umbral para considerar que una interacción es sinérgica24,25. Algunos modelos requieren un conocimiento previo de los agentes biológicos ensayados, hacen ciertas suposiciones sobre la actividad de los agentes y pueden requerir curvas completas de dosis-respuesta para cada tratamiento único y combinado25. El método que seleccionamos para medir la sinergia es una modificación del modelo del coeficiente de interacción farmacológica (CDI), que se ha utilizado previamente para medir los efectos inhibidores de las combinaciones de fármacos quimioterápicos sobre la proliferación de líneas celularescancerosas 26. El CDI es un modelo de independencia de Bliss; al calcular el efecto combinado predicho de dos perturbaciones, la independencia de Bliss supone que se dirigen a vías separadas y tienen mecanismos de acción independientes27. Para que una interacción entre perturbaciones sea sinérgica, el efecto combinado real debe ser mayor que el efecto predicho. Este modelo es apropiado para nuestra configuración experimental, ya que se sabe que el IFN-γ y el TNF-α tienen diferentes receptores y componentes de señalización posteriores. Además, la independencia de Bliss permite el cálculo de un coeficiente de interacción para cuantificar la sinergia y no requiere conjuntos de datos de dosis-respuesta.
Hay algunos factores clave que deben tenerse en cuenta para garantizar resultados óptimos para este protocolo. Es importante que los colonoides se propaguen a una alta densidad (Figura 1Bi), que tengan aproximadamente 25-50 μm de diámetro y que proliferen activamente antes de intentar sembrar células. El uso de cultivos subóptimos de colonoides para ensayos puede dar lugar a un número insuficiente de células, una baja recuperación de colonoides y experimentos inconsistentes. Para obtener resultados reproducibles, también es importante sembrar la densidad de colonoides de manera consistente entre experimentos. Se ha demostrado previamente que la respuesta in vitro a las citocinas inflamatorias puede ser influenciada por la densidad de siembra celular28,29. Otro problema común es la formación de burbujas de aire en la cúpula BME, que pueden afectar a las imágenes. Esto se puede prevenir mediante el uso de la técnica de pipeteo inverso. Esta técnica también da como resultado una siembra más consistente.
Además, si toma imágenes de varios puntos de tiempo, prepare una condición de toxicidad máxima para cada punto de tiempo. Triton-X 100, un tensioactivo no iónico, se usa comúnmente como control positivo (condición de toxicidad máxima) para ensayos de citotoxicidad. La adición de Triton-X 100 lisa y mata los colonoides, lo que permite que el tinte fluorescente de muerte celular ingrese a las células. El uso de una condición de toxicidad máxima de un punto de tiempo anterior dará como resultado una normalización inexacta e inconsistente de los datos debido a que la señal fluorescente decae con el tiempo.
Un último punto a tener en cuenta es la elección de BME utilizado para el cultivo de colonoides. Hay varios productores comerciales de BME; sin embargo, para nuestro protocolo, solo hemos probado la marca incluida en la Tabla de Materiales. Un estudio reciente utilizando organoides de cáncer de páncreas derivados de pacientes encontró que la fuente comercial de BME alteró las tasas de proliferación celular, pero no tuvo un efecto significativo sobre la respuesta a los medicamentos de quimioterapia o la expresión génica30. Teniendo esto en cuenta, esperamos que la tendencia de los resultados sea similar entre las marcas de BME para nuestro protocolo, pero recomendamos utilizar la misma marca de forma constante.
Demostramos cómo se puede utilizar este protocolo para el análisis de la muerte celular inducida por IFN-γ y TNF-α utilizando colonoides derivados de pacientes con EC. Los organoides intestinales derivados de pacientes son una herramienta poderosa para estudiar la EC, ya que conservan muchas características de la enfermedad, incluida una mayor sensibilidad a los efectos citotóxicos del TNF-α31. Sin embargo, el protocolo podría modificarse fácilmente para investigar los efectos citotóxicos de los perturbágenos distintos de las citocinas o de los estados patológicos distintos de la EII, como el cáncer colorrectal (hemos probado con éxito el protocolo utilizando colonoides no EII). Creemos que este método es útil para cualquier área de investigación relacionada con los mecanismos de muerte celular, la función de barrera epitelial o la inmunología de la mucosa intestinal.
Divulgaciones
K.N. recibió fondos de investigación de AbbVie Inc. durante el tiempo en que se completó el trabajo. Esta financiación se realizó en el contexto de una adjudicación de un centro de investigación (SFI-14/SP/2710) a APC Microbiome Irlanda.
Agradecimientos
Los autores desean agradecer a los pacientes por su consentimiento informado y participación en el estudio de investigación, y al personal clínico por su excelente asistencia. La Figura 1A se creó con BioRender.com. Este trabajo contó con el apoyo de subvenciones de la Fundación de Ciencias de Irlanda, a saber, un premio de desarrollo profesional (CDA) para K.N. (SFI-13/CDA/2171), una subvención de centro de investigación (SFI-12/RC/2273) y un premio de radio para centro de investigación (SFI-14/SP/2710) para APC Microbiome Ireland. P.F. también recibió financiación del documento SFI/20/RP/9007.
Materiales
| Name | Company | Catalog Number | Comments |
| Advanced DMEM/F12 | Gibco | 12634010 | |
| Amphotericin B Solution | Sigma-Merck | A2942 | |
| A-83-01 | Sigma-Merck | SML0788 | |
| BioRender | Science Suite Inc. | N/A | Scientific illustration software |
| Bovine Serum Albumin | Sigma-Merck | A2058 | Essentially IgG-free, low endotoxin |
| B27 Supplement | Invitrogen | 17504-044 | |
| CHIR-99021 | Sigma-Merck | SML1046 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| Cultrex Basement Membrane Extract, Type 2, Pathclear | R&D Systems | 3532-010-02 | Basement membrane extract |
| Dimethyl sulfoxide | Sigma-Merck | D2650 | |
| Dulbecco′s Phosphate Buffered Saline | Sigma-Merck | D8537 | |
| EVOS FL Digital Inverted Fluorescence Microscope | Invitrogen | AMF4300 | Digital inverted epifluorescence microscope |
| EVOS 40x Objective, fluorite, LWD, phase-contrast | ThermoFisher Scientific | AMEP4683 | Long working distance 40x fluorescence objective |
| Fiji/ImageJ (Windows version) | Open-source software | N/A | Image analysis software |
| Foetal Bovine Serum | Sigma-Merck | F9665 | |
| Gentamicin Solution | Sigma-Merck | G1397 | |
| Gentle Cell Dissociation Reagent | STEMCELL Technologies | 100-0485 | Enzyme-free cell dissociation reagent |
| GlutaMAX-1 | Gibco | 35050061 | L-alanyl-L-glutamine dipeptide supplement |
| GraphPad Prism 5 (Windows version) | Dotmatics | N/A | Data graphics and statistics software |
| Greiner 15 mL Polypropylene Centrifuge Tube, Sterile with conical bottom & Screw Cap | Cruinn | 188261CI | |
| HEPES 1 M | Gibco | 15630080 | |
| Human recombinant EGF (animal free) | Peprotech | AF-100-15 | |
| N-Acetylcysteine | Sigma-Merck | A9165 | |
| Nicotinamide | Sigma-Merck | N0636 | |
| Normocin | InvivoGen | ant-nr-05 | Broad range antimicrobial reagent |
| Nunc Edge 96-Well, Nunclon Delta-Treated, Flat-Bottom Microplate | ThermoFisher Scientific | 15543115 | |
| N2 supplement | Invitrogen | 17502-048 | |
| Recombinant Human IFN-gamma Protein | R&D Systems | 285-IF | Resuspend in sterile filtered 0.1% PBS/BSA |
| Recombinant Human TNF-alpha Protein | R&D Systems | 210-TA | Resuspend in sterile filtered 0.1% PBS/BSA |
| SB202190 | Sigma-Merck | S7067 | |
| Snap Cap Low Retention Microcentrifuge Tubes | ThermoFisher Scientific | 3451 | |
| SYTOX Green Nucleic Acid Stain - 5 mM Solution in DMSO | Invitrogen | S7020 | Fluorescent cell death dye, protect from light |
| Triton X-100 | Sigma-Merck | 93420 | |
| Trypan Blue solution | Sigma-Merck | T8154 | |
| Tryple Express | Gibco | 12604013 | Enzymatic dissociation reagent |
| Y-27632 | MedChemExpress | HY-10071 | Inhibitor of ROCK-I and ROCK-II |
Referencias
- Patankar, J. V., Becker, C. Cell death in the gut epithelium and implications for chronic inflammation. Nat Rev Gastroenterol Hepatol. 17 (9), 543-556 (2020).
- Zeissig, S., et al. Downregulation of epithelial apoptosis and barrier repair in active Crohn's disease by tumour necrosis factor alpha antibody treatment. Gut. 53 (9), 1295-1302 (2004).
- Bartee, E., Mcfadden, G. Cytokine synergy: An underappreciated contributor to innate anti-viral immunity. Cytokine. 63 (3), 237-240 (2013).
- Fish, S. M., Proujansky, R., Reenstra, W. W. Synergistic effects of interferon γ and tumour necrosis factor α on T84 cell function. Gut. 45 (2), 191-198 (1999).
- Wakisaka, Y., Sugimoto, S., Sato, T. Organoid medicine for Inflammatory Bowel Disease. Stem Cells. 40 (2), 123-132 (2022).
- Flood, P., Hanrahan, N., Nally, K., Melgar, S. Human intestinal organoids: Modeling gastrointestinal physiology and immunopathology - current applications and limitations. Eur J Immunol. 54 (2), e2250248 (2024).
- Matsuzawa-Ishimoto, Y., et al. An intestinal organoid-based platform that recreates susceptibility to t-cell-mediated tissue injury. Blood. 135 (26), 2388-2401 (2020).
- Lee, C., et al. Intestinal epithelial responses to IL-17 in adult stem cell-derived human intestinal organoids. J Crohns Colitis. 16 (12), 1911-1923 (2022).
- Woznicki, J. A., et al. TNF-α synergises with IFN-γ to induce caspase-8-JAK1/2-STAT1-dependent death of intestinal epithelial cells. Cell Death Dis. 12 (10), 864 (2021).
- Flood, P., et al. DNA sensor-associated type I interferon signaling is increased in ulcerative colitis and induces jak-dependent inflammatory cell death in colonic organoids. Am J Physiol Gastrointest Liver Physiol. 323 (5), G439-G460 (2022).
- Grabinger, T., et al. Ex vivo culture of intestinal crypt organoids as a model system for assessing cell death induction in intestinal epithelial cells and enteropathy. Cell Death Dis. 5 (5), e1228 (2014).
- Bode, K. J., Mueller, S., Schweinlin, M., Metzger, M., Brunner, T. A fast and simple fluorometric method to detect cell death in 3D intestinal organoids. Biotechniques. 67 (1), 23-28 (2019).
- Mertens, S., et al. Drug-repurposing screen on patient-derived organoids identifies therapy-induced vulnerability in KRAS-mutant colon cancer. Cell Rep. 42 (4), 112324 (2023).
- Vandussen, K. L., et al. Development of an enhanced human gastrointestinal epithelial culture system to facilitate patient-based assays. Gut. 64 (6), 911-920 (2015).
- Miyoshi, H., Stappenbeck, T. S. In vitro expansion and genetic modification of gastrointestinal stem cells in spheroid culture. Nat Protoc. 8 (12), 2471-2482 (2013).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Woznicki, J. A., et al. Human BCL-G regulates secretion of inflammatory chemokines but is dispensable for induction of apoptosis by IFN-γ and TNF-α in intestinal epithelial cells. Cell Death Dis. 11 (1), 68 (2020).
- Edgar, R. D., et al. Culture-associated DNA methylation changes impact on cellular function of human intestinal organoids. Cell Mol Gastroenterol Hepatol. 14 (6), 1295-1310 (2022).
- Mubaid, F., Brown, C. M. Less is more: Longer exposure times with low light intensity is less photo-toxic. Microscopy Today. 25 (6), 26-35 (2017).
- Ziegler, U., Groscurth, P. Morphological features of cell death. News Physiol Sci. 19 (3), 124-128 (2004).
- Zhang, Y., Chen, X., Gueydan, C., Han, J. Plasma membrane changes during programmed cell deaths. Cell Res. 28 (1), 9-21 (2018).
- Tamura, H., et al. Evaluation of anticancer agents using patient-derived tumor organoids characteristically similar to source tissues. Oncol Rep. 40 (2), 635-646 (2018).
- Karki, R., et al. Synergism of TNF-α IFN-γ triggers inflammatory cell death, tissue damage, and mortality in SARS-CoV-2 infection and cytokine shock syndromes. Cell. 184 (1), 149-168 (2021).
- Geary, N. Understanding synergy. Am J Physiol Endocrinol Metab. 304 (3), E237-E253 (2013).
- Duarte, D., Vale, N. Evaluation of synergism in drug combinations and reference models for future orientations in oncology. Curr Res Pharmacol Drug Discov. 3, 100110 (2022).
- Wong, F. C., Woo, C. C., Hsu, A., Tan, B. K. The anti-cancer activities of Vernonia amygdalina extract in human breast cancer cell lines are mediated through caspase-dependent and p53-independent pathways. PLoS One. 8 (10), e78021 (2013).
- Ryall, K. A., Tan, A. C. Systems biology approaches for advancing the discovery of effective drug combinations. J Cheminform. 7, 7 (2015).
- Sukho, P., et al. Effect of cell seeding density and inflammatory cytokines on adipose tissue-derived stem cells: An in vitro study. Stem Cell Rev Rep. 13 (2), 267-277 (2017).
- Vaughan-Jackson, A., et al. Density dependent regulation of inflammatory responses in macrophages. Front Immunol. 13, 895488 (2022).
- Lumibao, J. C., et al. The impact of extracellular matrix on the precision medicine utility of pancreatic cancer patient-derived organoids. bioRxiv. , (2023).
- Lee, C., et al. TNF-α induces LGR5+ stem cell dysfunction in patients with Crohn's disease. Cell Mol Gastroenterol Hepatol. 13 (3), 789-808 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados