Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Extracción de ARN de un solo animal y un solo tubo para la comparación de niveles relativos de transcripción mediante qRT-PCR en el tardígrado Hypsibius exemplaris
En este artículo
Resumen
Este trabajo presenta un método de extracción rápida de ARN y comparación a nivel de transcripción para analizar la expresión génica en el tardígrado Hypsibius exemplaris. Mediante el uso de la lisis física, este método de alto rendimiento requiere un solo tardígrado como material de partida y da como resultado una producción robusta de ADNc para la reacción en cadena de la polimerasa con transcriptasa inversa cuantitativa (qRT-PCR).
Resumen
El tardígrado Hypsibius exemplaris es un organismo modelo emergente reconocido por su capacidad para sobrevivir a condiciones ambientales extremas. Para explorar los mecanismos moleculares y las bases genéticas de dicha extrematolerancia, muchos estudios se basan en la secuenciación de ARN (RNA-seq), que se puede realizar en poblaciones que van desde grandes cohortes hasta animales individuales. La reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR) y la interferencia de ARN (ARNi) se utilizan posteriormente para confirmar los hallazgos de RNA-seq y evaluar los requisitos genéticos de los genes candidatos, respectivamente. Dichos estudios requieren un método eficiente, preciso y asequible para la extracción de ARN y la medición de los niveles relativos de transcripción mediante RT-PCR cuantitativa (qRT-PCR). Este trabajo presenta un método eficiente de extracción de ARN de un solo tardígrado y un solo tubo (STST) que no solo aísla de manera confiable el ARN de los tardígrados individuales, sino que también reduce el tiempo y el costo requeridos para cada extracción. Este método de extracción de ARN produce cantidades de ADNc que se pueden utilizar para amplificar y detectar múltiples transcripciones mediante PCR cuantitativa (qRT-PCR). El método se valida mediante el análisis de los cambios dinámicos en la expresión de los genes que codifican dos proteínas reguladas por el choque térmico, la proteína de choque térmico 70 β2 (HSP70 β2) y la proteína de choque térmico 90α (HSP90α), lo que permite evaluar sus niveles relativos de expresión en individuos expuestos al calor mediante qRT-PCR. STST complementa eficazmente los métodos existentes de extracción de ARN de tardígrado a granel y de un solo tardígrado, lo que permite un examen rápido y asequible de los niveles transcripcionales individuales de tardígrados mediante qRT-PCR.
Introducción
Los tardígrados son pequeños animales pluricelulares reconocidos por su capacidad para sobrevivir a condiciones extremas que son letales para la mayoría de las otras formasde vida. Por ejemplo, estos animales pueden sobrevivir casi 1000 veces la dosis de radiación ionizante que es letal para los humanos 2,3,4,5,6,7,8,9,10, la desecación casi completa 11,12,13,14,15, la congelación en ausencia de adición crioprotectores 16,17,18, y, en su estado disecado, incluso el vacío del espacio 19,20. Debido a su capacidad única de supervivencia en ambientes extremos, estos animales se han convertido en modelos fundamentales para comprender la extrematolerancia en organismos complejos y pluricelulares 1,21,22,23.
La manipulación genética estable de estos notables animales, incluyendo la transgénesis y la modificación de genes de la línea germinal, ha sido difícil de alcanzar hasta hace poco24,25. Como tal, la mayoría de los experimentos para revelar los mecanismos moleculares de la extrematolerancia se realizan a través de perfiles transcripcionales a través de la secuenciación de ARN. Existen muchos conjuntos de datos de secuenciación de ARN valiosos e informativos para tardígrados en diversas condiciones extremas, que van desde la radiación 8,9,26,27,28, el estrés por calor 29, el estrés por congelación12 y la desecación 27,30,31,32,33. Algunos de estos estudios han utilizado métodos de extracción y purificación de ARN a granel para iluminar nuestra comprensión molecular de la extrematolerancia. Sin embargo, la extracción masiva de transcripciones de ARN de muchos animales impide el análisis de la variación en la expresión génica entre individuos, por lo que se pierde la riqueza potencial de conjuntos de datos más refinados. Es importante destacar que estos estudios a menudo analizan poblaciones heterogéneas de animales que incluyen tanto animales que sobreviven a los factores de estrés ambiental como aquellos que no lo hacen. Como tal, estos estudios se confunden al promediar los datos de expresión de múltiples estados de respuesta potencialmente drásticamente diferentes. Para abordar este problema, Arakawa et al., 201634 desarrollaron una elegante tubería de secuenciación de ARN de baja entrada que aplica un kit de extracción de ARN seguido de un paso de amplificación de PCR lineal utilizando un soloanimal 34,35,36 o varios 30,37,38 como entrada. Estos estudios han sido fundamentales para nuestra comprensión de la extrematolerancia a los tardígrados22. Curiosamente, este protocolo también se ha aplicado a la qRT-PCR utilizando siete animales como material de partida24.
En la mayoría de los organismos modelo, una vez identificados los objetivos potenciales a través de RNA-seq, se realiza qRT-PCR para confirmar los cambios transcripcionales identificados por RNA-seq y evaluar el curso del tiempo de expresión de los genes candidatos de una manera de alta resolución. Para probar la función de los genes identificados, estos estudios suelen ir seguidos de la eliminación mediada por ARNi de las dianas moleculares39,40 y el análisis de la capacidad de tolerancia extrema12,41. La eficacia de cada knockdown de ARNi se confirma normalmente mediante qRT-PCR mediante el seguimiento directo de la disminución de la abundancia de transcripciones. Sin embargo, el ARNi es un proceso laborioso en los tardígrados, ya que cada dsRNA debe administrarse mediante microinyección manual de individuos39,40. Debido a la naturaleza de bajo rendimiento de esta estrategia, un método de extracción de ARN rápido y de bajo costo adaptado para qRT-PCR de animales individuales sería muy valioso para la investigación de tardígrados. Aunque se han desarrollado métodos previos para extraer ARN de tardígrados individuales, estos protocolos no han combinado su extracción con qRT-PCR, sino que se han basado en métodos basados en la densidad óptica 12,40,41. Motivados por estos desafíos, buscamos desarrollar un protocolo que produjera de manera confiable ARN en cantidad y calidad que se pueda usar para qRT-PCR a partir de H. exemplaris.
Adaptado de un protocolo de extracción de ARN de un solo animal desarrollado para Caenorhabditis elegans42, STST está optimizado para H. exemplaris. El método de extracción consta de seis pasos rápidos de congelación-descongelación, que alteran físicamente la cutícula, lo que permite la extracción de ARN y la posterior síntesis de ADNc. El método STST disminuye el tiempo de extracción en más de 24 veces en comparación con los métodos de extracción de ARN a granel, como lo describe Boothby, 201843, y en un 30% en comparación con los kits de extracción de ARN de un solo tardígrado, como lo describe Arakawa et al., 201634. Además, el número de interacciones muestra-experimentador se reduce de 5 a solo 1 en comparación con las preparaciones del kit de extracción de ARN, lo que reduce el riesgo de contaminación por ribonucleasas exógenas. Al consultar genes altamente expresados, el método STST produce suficiente ADNc para 25 reacciones cuantitativas de RT-PCR por tardígrado, requiriendo solo 1 μL del volumen total de ADNc de 25 μL por reacción. Sin embargo, las concentraciones de molde deben determinarse empíricamente para transcripciones de menor abundancia.
La eficacia del método STST para analizar los cambios dinámicos en la expresión génica se evaluó mediante la investigación de la expresión diferencial de los genes que codifican la proteína de choque térmico-90α (HSP90α) y la proteína de choque térmico 70β2 (HSP70β2) en respuesta a un choque térmico a corto plazo a 35 °C durante 20 minutos. Tanto HSP70β2 como HSP90α en la mayoría de los organismos eucariotas se regulan rápidamente al alza después de una exposición a corto plazo al choque térmico (20 min)42. El análisis en H. exemplaris reveló que tanto los ARN codificantes de HSP70β2 como los de HSP90α extraídos de tardígrados individuales tratados térmicamente mostraron aumentos estadísticamente significativos en la expresión después de la exposición al calor a corto plazo. Estos hallazgos demuestran que el protocolo STST se puede utilizar para analizar los cambios dinámicos en la expresión génica en animales individuales a lo largo del tiempo.
El método de extracción STST debe complementar los métodos experimentales existentes, como RNA-seq, facilitando la extracción rápida y económica de ARN y la posterior comparación de los niveles de transcripción mediante qRT-PCR. Este método también será valioso para evaluar la eficiencia y la penetrancia del ARNi en individuos inyectados manualmente de manera más cuantitativa que la densidad óptica por sí sola. Finalmente, debido a sus estructuras cuticulares y características físicas similares, es probable que este método también sea efectivo para analizar la expresión génica en otras especies de tardígrados44.
Access restricted. Please log in or start a trial to view this content.
Protocolo
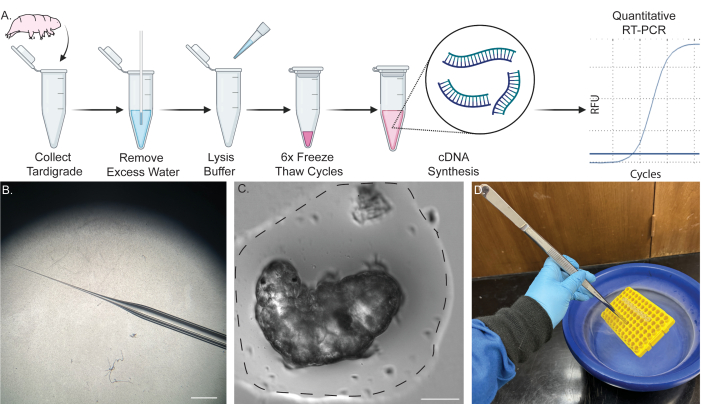
Figura 1: Tubería de un solo tubo para la extracción de ARN de un solo tardígrado. (A) Esquema que muestra el protocolo para la extracción de ARN de un solo tardígrado, incluidos seis ciclos de congelación-descongelación y la posterior síntesis de ADNc. Posteriormente, las muestras se pueden utilizar para RT-PCR y qRT-PCR. (B) Imagen del cono de la micropipeta utilizada para la eliminación del agua. Barra de escala: 2 mm. (C) Imagen de campo claro de un tardígrado en un pequeño volumen de agua (línea punteada). La eliminación de la mayor parte del agua en la medida mostrada es necesaria para una extracción exitosa y evita la dilución del tampón de lisis. Barra de escala: 50 μm. (D) Imagen que muestra la inmersión de muestras en nitrógeno líquido utilizando pinzas largas para congelar y descongelar rápidamente las muestras de manera segura. Parte del contenido fue creado en BioRender. Kirk, M. (2022) BioRender.com/d93s511 Haga clic aquí para ver una versión más grande de esta figura.
NOTA: La Figura 1A muestra un esquema del procedimiento. Para conocer los procedimientos detallados de cultivo de tardígrados y algas, consultar los informes publicados anteriormente 45,46,47.
1. Esterilización del agua de manantial
- Vierta 2 L de agua de manantial de una jarra de agua de 5 galones (consulte la Tabla de Materiales para conocer los detalles) en una botella de vidrio apta para autoclave de 2 L.
- Coloque la tapa en la botella apta para autoclave y selle con una pequeña cantidad de cinta adhesiva para autoclave. No apriete la botella; Coloque la tapa en la parte superior.
- Esterilizar el agua de manantial en autoclave durante 50 minutos en un ciclo húmedo sin paso de secado.
- Deje que el agua alcance la temperatura ambiente (RT) y selle la tapa firmemente antes de almacenarla en RT.
2. Extracción de micropipetas de vidrio (con un extractor de pipetas)
- Fije una micropipeta de vidrio (diámetro exterior: 1 mm, diámetro interior: 0,58 mm, longitud: 10 cm) en un extractor de micropipetas. Evite el contacto con el filamento calefactor, ya que esto alterará la forma de la pipeta y dañará el filamento.
- Determine empíricamente la tracción de la pipeta para cada filamento y extractor de pipeta. Sin embargo, para que sirva como punto de partida para la optimización, utilice 78 °C y un solo paso de tracción de peso de tracción de 182,2 g.
- Deje que el filamento se caliente y la gravedad separe la micropipeta de vidrio en dos micropipetas de vidrio con puntas afiladas (Figura 1B).
- Guarde estas micropipetas de vidrio estirado en una placa de Petri cerrada de 100 mm con cera o arcilla para mantenerlas en su lugar y evitar que se rompan las puntas afiladas.
3. Extracción de micropipetas de vidrio (sin extractor de pipetas)
- Encienda un mechero Bunsen u otra fuente de llama controlada a baja temperatura.
- Tome una micropipeta de vidrio con un extremo en cada mano.
- Sostenga el centro de la micropipeta de vidrio sobre la llama hasta que el vidrio comience a derretirse. Luego, separe rápidamente los dos extremos. Esto creará dos puntas afiladas muy delicadas.
- Rompa ligeramente la punta con un par de pinzas finas estériles.
- Guarde estas micropipetas de vidrio estirado en una placa de Petri cerrada de 100 mm con cera o arcilla para mantenerlas en su lugar y evitar que se rompan las puntas afiladas.
4. Extracción de ARN
- Obtenga 0,5 L de nitrógeno líquido en un recipiente crioseguro.
PRECAUCIÓN: El nitrógeno líquido es criogénico y puede causar quemaduras si se expone a la piel o los ojos. Al manipular, use ropa protectora, gafas protectoras, guantes de nitrilo, guantes criogénicos, una bata de laboratorio y zapatos cerrados. Asegúrese de que el recipiente sea seguro para el nitrógeno líquido antes de transportar el líquido. También puede ser posible usar un baño de hielo seco con etanol para este paso. - Prepare la mezcla maestra de síntesis de ADNc: una solución de 10 μL que contiene 1 μL de cebador de hexámero aleatorio, 2 μL de DNasa, 4 μL de tampón RT 5x, 1 μL de mezcla de enzimas, 1 μL de H2O y 1 μL de dNTP de 10 mM. Guarde esta solución en hielo.
- Prepare el tampón de lisis para tardígrados (5 mM Tris (pH = 8), 0,5% (v/v) Detergente 1, 0,5% (v/v) Detergente 2, 0,25 mM EDTA en agua estéril sin nucleasas).
NOTA: Esta solución se puede almacenar en la mesa de trabajo durante 6 meses. Sin embargo, mantenga la esterilidad y evite las posibles fuentes contaminantes de ARNasa. - Tampón de lisis alícuota suficiente para las extracciones (2 μL/tardígrado).
- Añadir inhibidor de ARNasa a la solución tampón de lisis tardígrada hasta una concentración final de 4 U/μL.
- Agite y centrifuga la solución en RT en una centrífuga de sobremesa a una velocidad de 2000 x g durante 5 s antes de almacenar la solución en hielo.
- Retire del cultivo tantos tardígrados como sean necesarios para el experimento con una pipeta P1000 estéril con punta de filtro y colóquelos en una placa de Petri estéril de 35 mm.
NOTA: Cualquier número de tardígrados puede ser procesado de esta manera. Por lo general, se procesan tres tardígrados por condición para la extracción. - Lave los tardígrados tres veces, utilizando 1 ml de agua de manantial estéril esterilizada en autoclave y una pipeta P1000 estéril con punta de filtro. Pipetearlos lentamente hacia arriba y hacia abajo ayuda a eliminar los contaminantes de las algas.
- Utilizando un microscopio de disección con un aumento de 25x a 50x, transfiera un solo tardígrado de este cultivo lavado a una nueva placa de Petri estéril de 35 mm utilizando una pipeta P10 estéril con punta de filtro.
- Utilice una pipeta P200 estéril con punta de filtro para lavar el tardígrado único en 100 μL de agua estéril sin nucleasas.
NOTA: Este paso de lavado se utiliza para eliminar aún más los contaminantes, incluidas las ribonucleasas. - Transfiera el tardígrado lavado al fondo de un tubo de PCR limpio y estéril en 1-2 μL de agua estéril sin nucleasas utilizando una pipeta P10 estéril con punta de filtro, asegurándose cuidadosamente de que el tardígrado no se pegue al costado de la punta.
- Visualice el tardígrado bajo un microscopio de disección con un aumento de 25x.
- Para facilitar la eliminación del agua, rompa ligeramente la punta de la micropipeta de vidrio estirado fuera del tubo. Asegúrese de que el orificio sea lo suficientemente grande como para sacar el agua, pero no el tardígrado.
- Utilizando la acción capilar de una micropipeta de vidrio estirado, extraiga el agua hasta que el animal esté rodeado por una pequeña burbuja de agua de aproximadamente dos longitudes tardígradas de diámetro.
- Supervise el proceso de eliminación de agua a través del endoscopio de disección para asegurarse de que el nivel del agua sea adecuado y que el tardígrado permanezca hidratado.
NOTA: La Figura 1C ofrece un ejemplo de la cantidad de agua que se debe eliminar. Este es un paso crítico. Una pequeña burbuja de agua rodeará al tardígrado para evitar que se seque, pero se debe eliminar la mayor cantidad posible de agua excesiva para evitar la dilución del tampón de lisis. Para ver un ejemplo de los niveles de agua restantes, consulte la Figura 1C. - Inmediatamente después de extraer el agua, añadir 2 μL de tampón de lisis tardígrado en el fondo del tubo, brevemente en vórtice, y centrifugar el tubo a RT durante 5 s a 2000 x g en una centrífuga de mesa.
- Coloque inmediatamente las muestras que contienen los tardígrados en una gradilla de tubos de PCR y asegúrese de que estén sujetas firmemente por la rejilla.
- Sujete la rejilla con un par de pinzas largas y gruesas y sumerja suavemente la rejilla que contiene las muestras en el nitrógeno líquido hasta que esté completamente congelada (Figura 1D).
- Retire la rejilla del nitrógeno líquido y colóquela inmediatamente sobre hielo. Deje que la muestra se descongele (tarda ~ 45 s a 1 min). Controle la muestra cada 15 s retirándola del hielo e inspeccionándola visiblemente. Una vez que la muestra esté visiblemente transparente, continúe con el siguiente paso.
- Repita los pasos 4.18-4.19 cinco veces más. Se requieren un total de seis ciclos de congelación-descongelación para obtener la máxima lisis y extracción (Figura 2A, B).
- Una vez que se complete la congelación-descongelación, coloque las muestras en hielo e inmediatamente continúe con el siguiente paso. No congele las muestras en este punto para almacenarlas, ya que esto disminuirá el ARN disponible para la preparación del ADNc.
5. Síntesis de ADNc
- Añada 2 μL de mezcla maestra de síntesis de ADNc al tubo de PCR que contiene lisado de tardígrado. Agite brevemente el tubo y gírelo a RT a 2000 x g durante 5 s con una centrífuga de mesa antes de reemplazar las muestras en hielo.
- Coloque las muestras en un termociclador e incube a 25 °C durante 10 min para recocer cebadores, a 55 °C durante 30 min para realizar la transcripción inversa y, finalmente, enzimas inactivas por calor a 85 °C durante 5 min.
- Después de la incubación, coloque inmediatamente el tubo en hielo y diluya la muestra hasta un volumen total de 25 μL añadiendo 21 μL de agua estéril sin nucleasa. En el caso de transcripciones con un número bajo de copias, modifique este paso de dilución según lo determine empíricamente.
6. qPCR
- Determine la temperatura de recocido del conjunto de cebadores utilizando ARN total preparado a partir de grandes cantidades de tardígrados, por ejemplo, el método de extracción a granel presentado en Boothby, 201843.
- Ejecute un gradiente de temperatura de PCR para determinar la temperatura óptima de recocido antes de ejecutar qRT-PCR (para todas las configuraciones de PCR utilizadas en este protocolo, consulte la Tabla 1 y la Tabla 2).
- Descongele un tubo de tinte indicador supermix en hielo y aísle de la luz. Coloque una placa de qPCR de 96 pocillos en hielo y coloque 5 μL de supermezcla, 2 μL de agua, 1 μL de cada cebador (10 μM) y 1 μL de producto de ADNc en el número de pocillos deseados.
- Selle la placa de PCR con un sello de placa y ejecute la qRT-PCR utilizando una temperatura de recocido adecuada para el conjunto de cebadores (para todas las configuraciones de qRT-PCR utilizadas en este documento, consulte la Tabla 3).
7. Cuantificación e interpretación de resultados
- Comparar los resultados cuantitativamente con uno o más genes de control, cuya expresión se espera que sea constante a lo largo de las condiciones impuestas. Para este estudio se utilizó el gen de la actina.
- Obtenga y compare los valores Ct o el umbral del ciclo para cada pocillo con los valores Ct de las reacciones génicas de mantenimiento de control. Calcule el cambio de pliegue en la expresión génica utilizando la siguiente ecuación:
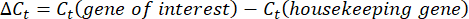
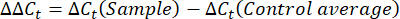
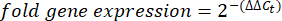
NOTA: La expresión génica de pliegue se representa gráficamente para cada transcripción y tardígrado como un 2-(ΔΔCt)48. - Para obtener una estimación aproximada del número de transcripción a partir del valorC t, utilice la siguiente ecuación:
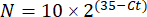
Donde N es el número de transcritos y 2 es la eficiencia de PCR asumida o el aumento de pliegues en la fluorescencia por ciclo de PCR48.
Access restricted. Please log in or start a trial to view this content.
Resultados
Desarrollo y optimización de la extracción de ARN de un solo tardígrado
Adaptando el protocolo de Ly et al., 201542 para la extracción de ARN en tardígrados, se optimiza el sistema STST para maximizar la cantidad y calidad de la preparación (Figura 1A). Se realizó RT-PCR para los transcritos de actina, cuantificando el rendimiento de la transcripción mediante la amplificación de una región de 527 pb que a...
Access restricted. Please log in or start a trial to view this content.
Discusión
Este estudio presenta un método eficiente para la extracción de ARN para qRT-PCR de un solo tardígrado. La comparación directa de la metodología STST con un kit de extracción de ARN de un solo tardígrado existente reveló que la extracción de ARN STST produce cantidades >200 veces mayores de transcripciones de ARN de actina, reduce el costo a menos de un dólar por muestra y reduce el tiempo requerido para la extracción en un 30%. Para aplicar la STST a una pregunta biológica r...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran no tener conflictos de intereses que divulgar.
Agradecimientos
Queremos agradecer a la Beca Ruth Kirschstein de los NIH # 5F32AG081056-02 y a la Beca Postdoctoral Errett Fisher, que apoyó a la Dra. Molly J. Kirk, a la Beca de la Familia Crowe, que apoyó a Chaoming Xu, y a una Beca del Senado Académico de la Universidad de California, Santa Bárbara, y a las subvenciones de los NIH #R01GM143771 y #2R01HD081266, que apoyaron estos esfuerzos de investigación. Los autores también reconocen el uso del Laboratorio de Nanoestructuras Biológicas dentro del Instituto de Nanosistemas de California, apoyado por la Universidad de California, Santa Bárbara, y la Universidad de California, Oficina del Presidente.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
Referencias
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890(2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884(2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333(2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621(2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330(2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495(2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245(2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, Ł The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983(2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522(2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120(2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298(2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405(2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143(2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266(2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663(2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168(2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063(2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24(2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888(2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- Goldstein, B. Hypsibius dujardini. collection notes and culture protocol from Bob McNuff. , At http://tardigrades.bio.unc.edu/protocols/CollectionCulture.pdf (2007).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508(2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424(2009).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados