Visualización en tiempo real de una sola molécula del desenrollado del ADN mediante CMG Helicase
En este artículo
Resumen
Este protocolo demuestra la realización de un ensayo de una sola molécula para la visualización en vivo del desenrollado del ADN por helicasa CMG. Describe (1) la preparación de un sustrato de ADN, (2) la purificación de la helicasa CMG de Drosophila melanogaster marcada con fluorescencia, (3) la preparación de una celda de flujo microfluídica para la microscopía de fluorescencia de reflexión interna total (TIRF) y (4) el ensayo de desenrollado de ADN de una sola molécula.
Resumen
La duplicación fiel del genoma es esencial para preservar la estabilidad genética de las células en división. La replicación del ADN se lleva a cabo durante la fase S por un complejo dinámico de proteínas denominado replisoma. En el corazón del replisma se encuentra la helicasa CDC45-MCM2-7-GINS (CMG), que separa las dos hebras de la doble hélice de ADN de modo que las ADN polimerasas puedan copiar cada hebra. Durante la duplicación del genoma, los replisomas deben superar una gran cantidad de obstáculos y desafíos. Cada uno de ellos amenaza la estabilidad del genoma, ya que la falta de replicación completa y precisa del ADN puede provocar mutaciones, enfermedades o muerte celular. Por lo tanto, es de gran interés comprender cómo funciona el CMG en el replisoma, tanto durante la replicación normal como durante el estrés de replicación. Aquí, describimos un ensayo de microscopía de fluorescencia de reflexión interna total (TIRF) que utiliza proteínas purificadas recombinantes, que permite la visualización en tiempo real de moléculas de ADN estiradas atadas a la superficie por complejos CMG individuales. Este ensayo proporciona una potente plataforma para investigar el comportamiento de CMG a nivel de una sola molécula, lo que permite observar directamente la dinámica de las helicasas con control en tiempo real de las condiciones de reacción.
Introducción
La replicación del ADN está estrictamente regulada, ya que una célula debe duplicar su genoma con precisión para evitar mutaciones, enfermedades o la muerte. La replicación del ADN eucariota se lleva a cabo mediante el complejo replisoma, que desenrolla el ADN parental y utiliza el ADN monocatenario (ssDNA) como molde para sintetizar nuevo ADN. En la fase G1, los hexámeros dobles catalíticamente inactivos de MCM2-7 se cargan en el ADN bicatenario (dsDNA) en los orígenes de replicación1. En la fase S, los complejos MCM2-7 se activan mediante la unión de CDC45 y GINS2 para formar complejos CMG de 11 subunidades (CDC45, MCM2-7, GINS). Cada CMG inicia el desenrollado del ADN en direcciones opuestas, formando la unidad central alrededor de la cual el replisoma se organiza alrededor de3.
Hace dos décadas, la helicasa CMG se identificó por primera vez como un complejo de 11 subunidades, esencial para la replicación del ADN4. Desde entonces, nuestra comprensión de la CMG ha avanzado considerablemente, desde la carga y activación 5,6, hasta el desenrollado y la terminación del ADN7. Las técnicas tradicionales de biología bioquímica y estructural han sido fundamentales para muchos de estos descubrimientos; sin embargo, estos métodos a menudo estaban limitados en su capacidad para estudiar los aspectos más dinámicos de la CMG. Los métodos de una sola molécula utilizan la manipulación física de biomoléculas individuales para medir o visualizar su actividad, una molécula a la vez. Esto se puede utilizar para proporcionar información sobre la dinámica en tiempo real de proteínas que a menudo se pasan por alto o son indetectables por otras técnicas 8,9.
Aquí, describimos un ensayo de microscopía de fluorescencia de reflexión interna total (TIRF) para visualizar el desenrollamiento del ADN por helicasa CMG en tiempo real. El CMG purificado y marcado con fluorescencia se carga en el extremo libre 3' del ADN largo que contiene una estructura de horquilla de ADN prefabricada. El ADN lineal se estira en un cubreobjetos de biotina-PEG en una celda de flujo microfluídico atando cada extremo del ADN secuencialmente a la superficie. Este enfoque permite un anclaje más uniforme del ADN, lo que reduce significativamente la variación que debe tenerse en cuenta durante el análisis de datos. En presencia de ATP-γ-s, la CMG se carga en el ADN monocatenario en el extremo 3' de la horquilla. El ATP-γ-s es un análogo de ATP hidrolizable lentamente, que permite que CMG se una al ADN pero no se desenrolle. La adición posterior de ATP, junto con RPA purificado y marcado con fluorescencia, activa CMG e inicia un extenso desenrollado del ADN. Visualmente, CMG se transloca a lo largo del ADN, dejando tras de sí un tracto creciente de ssDNA unido a RPA. El extremo de ADN sin ataduras viaja con CMG, formando una "bola apretada" debido a la compactación causada por la unión de RPA. El diseño de la celda de flujo permite que el tampón se intercambie en cualquier punto durante el desenrollado, lo que brinda un gran control durante y sobre cada experimento.
Este protocolo se divide en cuatro métodos, que se pueden realizar de forma independiente entre sí. En la sección 1 se describe la preparación de un sustrato de ADN bifurcado lineal de 20 kb para ensayos de una sola molécula. En la sección 2 se describe la purificación y el marcaje fluorescente de Drosophila melanogaster CMG (DmCMG). La información clave sobre la expresión de DmCMG se incluye en la sección de notas. La sección 3 cubre la preparación de una celda de flujo microfluídica que se puede usar en un microscopio TIRF. En la sección 4 se describe cómo llevar a cabo el ensayo de desenrollado del ADN de una sola molécula.
Protocolo
1. Preparación de ADN bifurcado lineal de 20 kb utilizado en ensayos de molécula única (Figura 1)
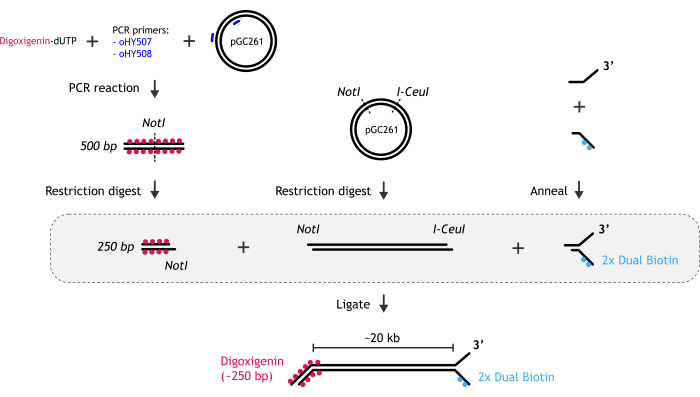
Figura 1: Representación gráfica de la preparación del sustrato de ADN. (A) El extremo de la horquilla de ADN biotinilado se crea mediante el recocido de dos oligonucleótidos parcialmente complementarios: uno biotinilado y otro no biotinilado. (B) El fragmento principal de dsDNA (~ 20 kb) se genera por digestión de restricción del plásmido pGC261 con dos enzimas para crear un ADN lineal con diferentes voladizos en cada extremo. (C) El extremo del ADN dúplex de digoxigenina se obtiene mediante una reacción de PCR realizada en presencia de digoxigenina-dUTP, seguida de una digestión de restricción. Haga clic aquí para ver una versión más grande de esta figura.
- Generación de extremo de horquilla de ADN biotinilado.
- Recocido el ADN de la horquilla.
- Mezcle dos oligonucleótidos (oHY502 y oHYbio85) en 1x tampón STE (100 mM de NaCl, 10 mM de Tris, pH 8,0; 1 mM de ácido etilendiaminotetraacético [EDTA]). La concentración final de oligonucleótidos es de 10 μM y el volumen de reacción final es de 100 μL.
- Incubar la reacción a 80 °C durante 5 min en un bloque de calor. Apague el bloque de calor y deje que la reacción se enfríe gradualmente, dejándolo en el bloque de calor hasta que la temperatura descienda por debajo de 30 °C.
- Valide la eficiencia de recocido en un gel de electroforesis en gel de tris-borato EDTA (TBE)-poliacrilamida (PAGE).
- Prepare un gel de acrilamida - TBE al 8% (para 4 geles: 32,8 mL deH2O; 4,8 mL de tampón TBE 10x (1 M de Tris, 1 M de ácido bórico, 20 mM de EDTA), 9,6 mL de solución de acrilamida/Bis al 40%, 37,5:1; 0,8 mL de persulfato de amonio al 10% [APS], 40 μL de tetrametiletilendiamina [TEMED]).
PRECAUCIÓN: TBE, acrilamida, APS, TEMED son tóxicos/dañinos. - Ejecute un pequeño volumen (1 μL) de oligonucleótidos recocidos junto con los oligonucleótidos individuales diluidos con colorante de carga 6x en el gel de acrilamida al 8% - TBE a 80 V, durante 1 h a RT.
- Remoje el gel en 1x TBE complementado con una tinción de ácido nucleico adecuada durante 30 minutos en un agitador oscilante, luego escanee el gel con un sistema de imágenes de gel.
- Prepare un gel de acrilamida - TBE al 8% (para 4 geles: 32,8 mL deH2O; 4,8 mL de tampón TBE 10x (1 M de Tris, 1 M de ácido bórico, 20 mM de EDTA), 9,6 mL de solución de acrilamida/Bis al 40%, 37,5:1; 0,8 mL de persulfato de amonio al 10% [APS], 40 μL de tetrametiletilendiamina [TEMED]).
- Resuelva el ADN recocido en el gel TBE-PAGE para la escisión en gel.
- Si los oligonucleótidos se recocen correctamente, resuelva el resto de la muestra en presencia de BSA (~100 μL de muestra recocida, 25 μL de colorante de carga 6x, 2 μL de 33 mg/mL de BSA). Para cargar el mayor volumen en el gel, combine varios pocillos con un bisturí estéril.
- Deje el gel en 1x TBE complementado con una tinción de ácido nucleico adecuada durante 30 min en un agitador oscilante.
- Transfiera el gel a una bandeja de vidrio y visualice el ADN en una habitación oscura bajo luz azul. Utilice una pantalla protectora adecuada o gafas protectoras cuando utilice la luz azul.
- Corta la banda deseada con un bisturí estéril. Retira el exceso de gel.
- Purificar la horquilla recocida por electroelución.
- Corte un trozo de tubo de diálisis lo suficientemente largo como para insertar el trozo de gel, humedézcalo en 1x TBE y cierre un extremo.
- Diluya BSA a 0,3 mg/ml en 500 μl de 1x TBE y pipetee todo el volumen en el tubo.
- Use la espátula de metal para deslizar la pieza de gel adentro. Mueva la pieza de gel a un lado del tubo para dejar espacio tampón para que el ADN migre durante la electroelución.
- Exprima el exceso de tampón y corte el segundo extremo del tubo.
NOTA: El volumen restante determinará el volumen y la concentración finales. - Coloque el tubo de diálisis en el tanque de electroforesis de agarosa en 1x tampón TBE, asegurándose de que el tubo esté completamente sumergido.
NOTA: Asegúrese de que la pieza de gel esté colocada en el lado más cercano al electrodo negativo para que el ADN pueda migrar fuera del gel a la bolsa de diálisis. - Haga funcionar la electroelución a 80 V durante 1-2 h.
- Diálisar el ADN electroeluido en un tampón Tris de 10 mM (pH 8).
- Después de la electroelución, saque la bolsa de diálisis del tanque y seque un extremo con un pañuelo de papel. Evite tocar la parte media del tubo con pañuelo de papel, donde se encuentran el gel y la muestra.
- Retire el clip del extremo seco y pipetee suavemente la muestra hacia arriba y hacia abajo dentro del tubo para mezclar el ADN con el tampón.
- Sumerja una espátula limpia en 1x tampón TBE y saque la pieza de gel del tubo.
NOTA: Mientras retira la pieza de gel, retire la menor cantidad de tampón posible. - Vuelva a cerrar el extremo abierto de la bolsa de diálisis, eliminando el aire del interior.
- Coloque la bolsa de diálisis en un vaso de precipitados de 2 L lleno de 1,5 L de tampón Tris de 10 mM (pH 8), 20 mM de NaCl y 2 mM de MgCl2. Envuelva los clips con parafilm para pegar el tubo al borde del vaso de precipitados mientras sumerge completamente el tubo en el tampón.
NOTA: La sal estabiliza los dúplex de oligo cortos. - Utilice una barra magnética para agitar suavemente el tampón y dializar la muestra a temperatura ambiente (RT) durante al menos 3-4 h o a 4 °C durante la noche.
- Después de la diálisis, seque un extremo de la bolsa de diálisis con un pañuelo desechable (para no diluir el ADN) y suelte este extremo.
- Pipetea suavemente la muestra hacia arriba y hacia abajo dentro del tubo y transfiérela a un tubo limpio de 1,5 ml.
- Mida la concentración del ADN utilizando un espectrofotómetro de microvolúmenes.
- Recocido el ADN de la horquilla.
- Generación de fragmento de 20 kb
- Realice la digestión de restricción del plásmido18 de pGC261 utilizando enzimas NotI/I-CeuI.
- Mezcle suavemente 8 μL de NotI-HF (20.000 U/mL), 8 μL de I-CeuI (5.000 U/mL) y el plásmido pGC261 (concentración final del plásmido ~40 ng/μL en una reacción de 200 μL) en un tampón recomendado por los productores de enzimas de restricción.
- Incubar la reacción a 37 °C durante la noche.
- Resuelva el ADN digerido en gel de agarosa para la escisión en gel.
- OPCIONAL: Ejecute primero una pequeña cantidad de la reacción en un gel de agarosa al 0,6% para probar la eficiencia de la digestión antes de cargar toda la reacción.
- Prepare el gel de agarosa al 0,6% siguiendo los pasos 1.2.2.3-1.2.2.4.
- Mezclar 0,48 g de agarosa en polvo con 80 mL de tampón 1x TBE. Calienta la solución en el microondas hasta que esté hirviendo. Revuelva para asegurarse de que la agarosa se derrita por completo. Deje enfriar durante unos minutos, luego vierta en el tanque designado.
- Para combinar los pocillos para acomodar el mayor volumen de la muestra, pegue un trozo de cinta adhesiva a través de varios pocillos del peine de antemano.
- Una vez que el gel se haya solidificado, retire suavemente el peine y rellene el tanque con 1x tampón TBE. Mezcle suavemente la muestra con el tinte de carga de ADN y haga funcionar el gel a 120 V durante 1 h.
- Deje el gel en 1x TBE complementado con una tinción de ácido nucleico adecuada en un agitador oscilante hasta que se manche.
- Transfiera el gel manchado a una bandeja de vidrio y visualice el ADN en una habitación oscura bajo luz azul.
- Corta la banda deseada con un bisturí estéril. Retira el exceso de gel.
- Opcional: Purificar el fragmento deseado por electroelución como se describe en los pasos 1.1.4 y 1.1.5. Omita la sal del tampón de diálisis aquí, ya que solo es necesaria para estabilizar el oligodúplex corto.
- Realice la digestión de restricción del plásmido18 de pGC261 utilizando enzimas NotI/I-CeuI.
- Generación del extremo del ADN dúplex de digoxigenina
- Realice una reacción de PCR con digoxigenina-dUTP (dig-dUTP) utilizando el ADN pGC261 como plantilla.
- Mezcla: 400 μL de agua, 8 μL de pGC261 (0,8 ng/μL), 3,5 μL de cebador oHY507 (100 μM), 3,5 μL de cebador oHY508 (100 μM), 8 μL de dig-dUTP (1 mM), 400 μL de 2x mezcla de PCR (preparada a partir de ADN polimerasa (20 μL), 10 mM de dNTPs (40 μL), 5 veces tampón de alta fidelidad (HF) (400 μL) y agua (540 μL).
- Ejecute la siguiente reacción en un termociclador:
98 °C - 1 min
30x: 98 °C - 20 s; 65 °C - 20 s; 72 °C - 30 s;
72 °C - 10 min
4 °C - Mantener
- Digiera y purifique el producto de PCR marcado con digoxigenina
- Purifique el producto de PCR utilizando un kit comercial de purificación de ADN.
- Mezcle 10 μL de NotI-HF (20.000 U/mL) con el producto de PCR en un tampón recomendado por el proveedor de la enzima (concentración final de ADN de ~50 ng/μL en una reacción de ~200 μL). Incubar la reacción a 37 °C durante la noche.
- Purifique el ADN digerido utilizando un kit comercial de purificación de ADN.
- Realice una reacción de PCR con digoxigenina-dUTP (dig-dUTP) utilizando el ADN pGC261 como plantilla.
- Ensambla los componentes para hacer el sustrato de ADN.
- Mezcle suavemente el extremo del tenedor biotinilado, el fragmento de ADN de 20 kb y el fragmento de PCR tratado con Not-I marcado con digoxigenina con 5 μL de ADN ligasa T4 (400.000 U/mL) en un tampón recomendado por el proveedor de la enzima en una reacción de 200 μL. Sin embargo, en función del fragmento de ADN de 20 kb añadido (1-5 μg), añada aproximadamente un exceso molar de aproximadamente 100 veces tanto el extremo de la horquilla biotinilado como el fragmento de PCR.
- Alícuota la reacción en tubos de PCR (50 μL cada uno) e incubar a 16 °C en un termociclador durante la noche.
- Resuelva la muestra ligada en un gel de agarosa al 0,6% y purifique el ADN ligado por electroelución como se describe en los pasos 1.2.2 y 1.2.3.
- Almacene el ADN congelado rápidamente a -80 °C.
2. Purificación de Drosophila melanogaster CMG (Figura 2)

Figura 2: Purificación de Drosophila melanogaster CMG a partir de 4 L de células Hi Five. Las proteínas se resolvieron en gel de poliacrilamida Bis-Tris al 4%-12% a menos de 200 V en presencia de tampón MOPS. La muestra se muestra en cada etapa de la purificación (lisado celular - 2 μL, elución FLAG - 10 μL, después de la primera columna de intercambio iónico - 10 μL, y después del etiquetado y la segunda columna de intercambio iónico - 1 μL. (A) La tinción de Coomassie confirma la presencia de las 11 subunidades del complejo CMG antes (10 μL) y después (1 μL) del marcaje fluorescente. (B) La eficiencia de etiquetado de la subunidad MCM3 se validó mediante el escaneo de Cy5 con un analizador de imágenes fluorescentes utilizando un filtro rojo de paso largo (LPR). Haga clic aquí para ver una versión más grande de esta figura.
NOTA: Para preparar el CMG de Drosophila melanogaster marcado con fluorescencia, se introdujo un sitio de escisión de TEV (ENLYFQG) seguido de cuatro residuos de Gly aguas abajo de la etiqueta FLAG N-terminal en la subunidad MCM3 (en el vector pFastBac1)10. Para expresar el complejo se utilizó el sistema de expresión de baculovirus. Para la transfección inicial, se utilizaron células Sf21 por separado para cada subunidad CMG (estadio del virus P1). Para amplificar aún más los virus, se utilizaron células Sf9 (estadio del virus P2). Posteriormente, los cultivos de células Sf9 (100 mL para cada subunidad CMG; 0,5 x 106 células/mL) se infectaron con 0,5 mL de virus P2 suplementado con suero fetal de ternero al 10% (estadio del virus P3). Para expresar todo el complejo CMG en 4 L de células Hi Five (1 x 106 células/mL), se utilizaron 200 mL de virus P3 para cada una de las subunidades. Después de cosechar las células Hi Five que expresan el complejo CMG, el pellet de la célula puede congelarse rápidamente en nitrógeno líquido y almacenarse a -80 °C. Realizar toda la purificación con hielo o a 4 °C. Los tampones pueden prepararse con antelación, siempre que los agentes reductores (DTT o 2-Mercaptoetanol) y los inhibidores de la proteasa (PRECAUCIÓN) se añadan justo antes de su uso. Asegúrese de que todos los tampones se preenfríen por adelantado, se filtren y se desgasifiquen.
- Prepare los siguientes búferes.
- Prepare el tampón de resuspensión A mezclando 25 mM de ácido 4-(2-hidroxietil)-1-piperazineetanosulfónico (HEPES) pH 7,5, 0,02% Tween-20, 10% glicerol, 15 mM KCl, 2 mM MgCl2, 2 mM 2-mercaptoetanol, 1 mM EDTA y 1 mM de etilenglicol-bis (β-aminoetílico éter)-N,N,N',N'-ácido tetraacético (EGTA).
PRECAUCIÓN: El 2-mercaptoetanol, el DTT y el EDTA son tóxicos/nocivos - Prepare tampón de solución salina tamponada con Tris (TBS; 0,1 M de glicina HCl, pH 3,5) para preparar el gel de afinidad ANTI-FLAG M2.
- Prepare el tampón A-100 mezclando 25 mM HEPES pH 7.5, 0.02% Tween-20, 10% glicerol, 100 mM KCl, 1 mM DTT, 1 mM EDTA y 1 mM EGTA.
- Prepare el tampón de diálisis añadiendo 25 mM de HEPES pH 7,5, 50 mM de acetato de sodio, 10 mM de acetato de magnesio, 10% de glicerol y 1 mM de DTT.
- Prepare el tampón TBS mezclando 50 mM de Tris-HCl, pH 7,5 y 150 mM de NaCl.
- Prepare el tampón de resuspensión A mezclando 25 mM de ácido 4-(2-hidroxietil)-1-piperazineetanosulfónico (HEPES) pH 7,5, 0,02% Tween-20, 10% glicerol, 15 mM KCl, 2 mM MgCl2, 2 mM 2-mercaptoetanol, 1 mM EDTA y 1 mM de etilenglicol-bis (β-aminoetílico éter)-N,N,N',N'-ácido tetraacético (EGTA).
- Resuspender pellet de cultivo de 4 L de células Hi Five en 200 mL de tampón de resuspensión en frío A suplementado con comprimidos de cóctel de inhibidores de la proteasa. Mezcle los tubos por inversión hasta que todo el pellet se vuelva a suspender por completo.
- Transfiera las células resuspendidas a un homogeneizador Dounce de 40 mL preenfriado. Para recuperar el mayor número posible de células, lave los tubos utilizados para la congelación de pellets de células con el mismo tipo de tampón y añádalos al homogeneizador Dounce. Asegúrese de que el volumen total del homogeneizador no supere el límite recomendado. Lise las células en hielo por 60-70 golpes.
NOTA: Asegúrese de que el émbolo llegue al fondo del tubo, pero no ejerza demasiada presión ya que el homogeneizador puede romperse. Tenga cuidado de no sacar el émbolo por encima del nivel del líquido, ya que esto introducirá burbujas. - Repita el paso anterior hasta homogeneizar toda la muestra y combínela en el vaso de precipitados preenfriado. Evalúe el volumen final de la muestra. Gota a gota, agregue KCl filtrado hasta la concentración final de 100 mM, mezclando suavemente.
- Enfríe previamente la centrífuga a 4 °C. Verter la muestra en tubos de centrifugación, asegurándose de alcanzar el volumen recomendado por el productor. Equilibra los tubos en una báscula.
NOTA: Los tubos con un volumen demasiado pequeño pueden romperse durante la centrifugación. - Centrifugar las células lisadas a 23.500 x g durante 15-30 min a 4 °C. Mientras tanto, prepare el gel de afinidad ANTI-FLAG M2 de acuerdo con el siguiente paso.
- Mezclar suavemente el frasco que contiene el gel de afinidad ANTI-FLAG M2 (50% de suspensión). Corte el extremo de una punta de pipeta P-100 (para ensanchar la punta) y utilícela para transferir inmediatamente 4 ml de suspensión (2 ml de perlas) a una columna de cromatografía de polipreparación de 20 ml. Para recuperar las cuentas atascadas en la punta, lave la punta con TBS y agréguela a la columna. Deje que todas las cuentas se asienten dentro de la columna, pero tenga cuidado de no secarlas.
- Lavar la resina con un volumen de perlas 3x (6 mL en total) de 0,1 M de Glycine HCl pH 3,5. Posteriormente, lavar las cuentas con un volumen de 3-5 cuentas de TBS (6-10 mL). A continuación, lave las perlas 3 veces (6 ml) con Buffer A-100.
NOTA: No deje las perlas en la solución de 0,1 M de glicina HCl pH 3,5 durante más de 20 minutos. Para aumentar la presión de flujo y la velocidad de lavado, cubra la columna con una tapa. No dejes que la resina se seque en ningún punto. - Después de preparar la resina, deje 2 mL de tampón A-100 por encima de la resina y cierre la salida de la columna.
- Después de la centrifugación, vierta suavemente el sobrenadante en tubos de 50 ml preenfriados. Tenga cuidado de no romper el pellet. Tome una pequeña alícuota (~10 μL) para validar mediante electroforesis SDS-PAGE más tarde.
- Agregue una cantidad igual de perlas ANTI-FLAG M2 (preparadas de acuerdo con el paso anterior) en cada uno de los tubos de 50 ml. Intenta recuperar todas las perlas de la columna resuspendiéndolas con un par de mililitros adicionales de Buffer A-100 y transfiriendo esta resuspensión a los tubos.
- Incubar la muestra (sobrenadante) con resina ANTI-FLAG M2, girando durante 2,5 h a 4 °C.
- Después de la incubación, centrifugar la muestra a 1.000 x g durante 5 min a 4 °C. Con una pipeta, elimine la mayor parte del sobrenadante. Con una punta cortada, vuelva a suspender las perlas en unos pocos mililitros del sobrenadante restante y transfiéralas a dos tubos de 15 ml. Para recuperar las perlas pegadas a las paredes de los tubos de 50 ml, agregue unos mililitros de tampón A-100 y transfiéralo también a los tubos de 15 ml.
- Girar los tubos de 15 ml a 1.000 x g durante 5 min a 4 °C. Retire el sobrenadante.
- Lave las perlas añadiendo ~14 mL de tampón A-100 a cada tubo, seguido de una rotación corta y suave a 4 °C y posterior centrifugado a 1000 x g durante 5 min a 4 °C. Repite los lavados dos veces. Tome pequeñas alícuotas de cada lavado (~10 μL) para validar posteriormente mediante electroforesis SDS-PAGE.
- Este paso (2.16) es un procedimiento alternativo a los pasos anteriores (2.13-2.15).
- Después de la incubación con resina ANTI-FLAG M2, vierta la muestra en dos columnas de cromatografía de poli-preparación de 20 mL.
- Si elige este método, deje que las cuentas se asienten en la parte inferior de la columna y la muestra no unida pase a través de la columna por el flujo de gravedad. A continuación, añada el tampón A-100 directamente a la columna y deje que lave la muestra mediante flujo por gravedad.
- Transfiera las perlas (resuspendidas en el tampón A-100) a dos columnas de cromatografía de polipreparación de 10 mL.
- Permita que el tampón A-100 pase a través de la resina, cierre la salida cuando el nivel del tampón alcance la parte superior de la resina y eluya la proteína de las perlas agregando tampón A-100 suplementado con 200 μg/mL (DYKDDDDK) péptido (tampón de elución FLAG).
- Para la primera elución, agregue 3 mL del tampón de elución FLAG y gire suavemente la columna cerrada durante 15 min en RT. Abra la salida para recoger la primera fracción de elución.
- Cierre la salida y añada 2 mL del tampón de elución FLAG a cada columna. Gire durante otros 10 minutos y recoja la segunda fracción de elución.
- Combine todas las fracciones eluidas juntas (~10 mL) y manténgalas a 4 °C. Tome una pequeña alícuota de la fracción eluida (~10 μL) para validar posteriormente mediante electroforesis SDS-PAGE.
- Filtre la muestra eluida con filtros de jeringa de 0,22 μm.
- Con anticipación, prepare la columna CaptoHiRes Q (5/50) (columna de cromatografía de intercambio iónico de alta resolución) conectada a un sistema de purificación de proteínas, de acuerdo con el protocolo del fabricante. A continuación, equilibre la columna con el tampón A-100 y cargue la muestra filtrada en la columna equilibrada.
- Lave la columna con 20 volúmenes de columna (CV) de tampón A-100 (~20 mL en total ya que el volumen de la columna es de ~1 mL).
- Para eluir la proteína, prepare dos tampones de antemano: el tampón A-100 y el tampón B con la misma composición que el tampón A-100, pero con 550 mM de KCl en lugar de 100 mM de KCl. Configure la elución en gradiente para 20 CV (~20 mL) con una concentración de sal creciente de 100 mM a 550 mM de KCl. Recoja eluciones de 0,3-0,5 mL en un colector de fracciones.
NOTA: El CMG debe eluir a una concentración de alrededor del 70%-75% del tampón B. Tome pequeñas alícuotas de las fracciones de lavado y eluido para la electroforesis SDS-PAGE. - Realice la electroforesis SDS-PAGE para confirmar que CMG está presente en las fracciones elegidas. Mientras tanto, limpie la columna y el sistema de purificación según el protocolo del fabricante.
- Incubar las fracciones elegidas con proteasa TEV durante la noche mezclando 50 μL de proteasa TEV (1 mg/mL) por 1 mL de muestra.
- Humedezca previamente un tubo de diálisis y asegúrese de que la membrana no se dañe. Añada la mezcla CMG/TEV en el tubo de diálisis y colóquela en un vaso de precipitados de 2 L con 1,5 L de tampón A-100 preenfriado y la barra magnética en su interior. Diálisis con agitación suave durante la noche a 4 °C.
- Mezcle la muestra con 50 μM de péptido marcado con LD655 y 10 μg/mL de enzima sortasa en presencia de 5 mM de CaCl2. Incubar la reacción a 4 °C, girando durante 30 minutos, asegurándose de que el tubo esté cubierto de luz. Tome una pequeña alícuota antes y después del etiquetado para la electroforesis SDS-PAGE.
- Filtre la muestra etiquetada con filtros centrífugos de 0,22 μm antes de cargarla en el sistema de purificación, ya que el péptido puede precipitar.
- Prepare la columna de cromatografía de intercambio iónico de alta resolución con anticipación, como se describió anteriormente (paso 2.20). Esta vez, cubra la columna y el sistema colector de fracciones con papel de aluminio para proteger la muestra de la luz.
- Para eliminar el péptido libre, cargue la muestra filtrada en la columna de cromatografía de intercambio iónico de alta resolución. Realice la purificación de la misma manera que se describió anteriormente (pasos 2.21-2.22). Asegúrese de que el CMG marcado eluya a una concentración de tampón-B similar a la anterior.
- Realizar electroforesis SDS-PAGE para validar la calidad de las fracciones elegidas. Para obtener imágenes de la fluorescencia, no hierva las muestras antes de cargarlas y asegúrese de que el tanque de electroforesis esté protegido de la luz. Visualice primero la fluorescencia de las proteínas utilizando un sistema de imágenes en gel, luego tiña el gel con el tinte Coomassie y vuelve a obtener imágenes para visualizar todas las proteínas.
- Diálisar las fracciones seleccionadas durante la noche a 4 °C, frente a 1,5 L de tampón de diálisis. Si es necesario, concentre la muestra.
- Congele rápidamente la proteína en nitrógeno líquido y guárdela a -80 °C hasta su uso posterior.
3. Preparación de la celda de flujo (Figura 3)
- Prepare los cubreobjetos de biotina-PEG siguiendo un protocolo11 descrito anteriormente, omitiendo el paso de horneado en horno. Los cubreobjetos de biotina-PEG son estables durante al menos 1 mes al vacío en RT. Para la preparación de la celda de flujo, corte el cubreobjetos de biotina-PEG (24 mm x 60 mm) por la mitad (aproximadamente 24 mm x 30 mm).
NOTA: Tenga cuidado de no tocar el área central del cubreobjetos donde estarán los canales de flujo. - Prepare pequeñas piezas de vidrio grabando y rompiendo un portaobjetos de vidrio en piezas de aproximadamente 2,4 cm x 1 cm.
- Taladre dos agujeros a 1,4 mm de distancia con una broca diamantada de 0,8 mm, uno ligeramente más ancho que el otro (0,043" = entrada, 0,048" = salida). Pruebe que los orificios sean del tamaño correcto tratando de insertar una pieza del tubo de entrada/salida, usando la misma broca para ensanchar el orificio hasta que el tubo encaje. Asegúrese de que el tubo esté lo suficientemente apretado como para que no se caiga fácilmente.
NOTA: Se utiliza un tubo de entrada más estrecho para minimizar el volumen muerto. - Corta cinta adhesiva de doble cara de la misma forma que las piezas de vidrio.
- Alinee la corredera en la cinta y apuñale una aguja a través de cada orificio para marcar sus posiciones en la cinta. Con una cuchilla de afeitar, corta un canal que abarque ambos agujeros.
NOTA: No corte el canal demasiado largo, ya que el exceso de espacio en el lado más alejado de cada orificio puede permanecer seco cuando fluye líquido y puede provocar problemas con las burbujas de aire durante el flujo. Al cortar, intente cortar los bordes largos en un solo corte limpio, ya que los bordes deshilachados de múltiples cortes pueden afectar la calidad del canal de flujo. Con un fin similar, no corte los bordes del canal de flujo, ya que los cortes en la pieza utilizable pueden afectar el flujo de líquido o incluso fugas en la celda de flujo ensamblada. - Limpie la pieza de vidrio con acetona y un pañuelo de papel hasta que se seque, luego colóquela sobre una superficie limpia. Retire un lado de la cinta adhesiva y péguela en la pieza de vidrio de modo que ambos orificios queden completamente dentro del canal.
- Selle la cinta a la pieza de vidrio utilizando una punta de pipeta p1000 para eliminar las burbujas presionando la superficie con una presión firme pero moderada. Repasa toda la superficie para sellar.
- Despega el segundo borde de la cinta de ambos pedazos de vidrio y colócalos sobre una superficie limpia con el lado adhesivo hacia arriba. Coloque los portaobjetos cerca unos de otros, pero sin tocarse en la posición en que se unirán al cubreobjetos (deje suficiente espacio entre ellos para que el epoxi cree un sello completo alrededor de cada pieza de vidrio).
- Recoja el cubreobjetos de medio biotina-PEG con pinzas de plástico, sosteniéndolo solo por el borde (no toque el área que formará el canal de flujo) y baje el lado funcionalizado de PEG sobre el adhesivo. Asegúrelo presionando hacia abajo con un dedo para fijarlo en su lugar, luego complete el sellado frotando la superficie moderadamente firmemente con la punta de una pipeta para eliminar las burbujas de aire (no en el área del canal de flujo, ya que puede romperse), luego voltee.
NOTA: Manipule el cubreobjetos con cuidado. - Para cada celda de flujo, corte ~ 10 cm de cada tipo de tubo de polietileno (PE20 y PE60). Utilice el tubo más estrecho como entrada a la cámara de flujo para reducir el volumen muerto.
- Inserte el tubo en los orificios del portaobjetos con la mano. Si el diámetro de los orificios es correcto, asegúrese de que el tubo se levante en el orificio por sí solo una vez insertado. Corte la punta del tubo en un ligero ángulo <45° para facilitar un poco la inserción del tubo y evitar que el tubo forme un sello contra el cubreobjetos, lo que puede hacer si el extremo es plano (esto puede obstruir el flujo).
NOTA: El ajuste preciso del tubo en la pieza de vidrio hace que la construcción de la celda de flujo sea más confiable y ayuda a evitar la introducción de burbujas en la celda de flujo. - Mezcle bien los componentes de epoxi, luego use una punta p200 para frotar epoxi para sellar el tubo y crear un sello alrededor de cada pieza de vidrio. Añade suficiente epoxi para que llegue hasta el borde de la funda redonda, ya que esto también reforzará la delicada cubreobjetos. Sin embargo, asegúrese de que no entre epoxi en la parte inferior del cubreobjetos, ya que puede evitar que quede plano en el escenario.
NOTA: Si es necesario, se pueden raspar pequeñas cantidades de exceso de epoxi de la parte inferior con una cuchilla de afeitar. - Déjalo durante al menos 30-60 minutos para que el epoxi se cure por completo.
NOTA: Si la celda de flujo no se va a utilizar de inmediato, guárdela al vacío en RT después de curarla por completo. - Después de usar la celda de flujo, recupere las piezas de vidrio y reutilícelas indefinidamente. Extraiga el tubo y coloque la celda de flujo en un frasco deslizante que contenga acetona durante al menos 24 horas para ablandar el epoxi y el adhesivo de doble cara, lo que permite que las piezas de vidrio se eliminen fácilmente. Antes de reutilizar para hacer otra celda de flujo, limpie las piezas de vidrio frotándolas con una esponja y agua jabonosa, séquelas y frótelas con acetona y papel de seda.

Figura 3: Representación gráfica de la preparación de la celda de flujo. (A) Corte cinta adhesiva de doble cara para que coincida con el tamaño de la pieza de vidrio. Alinee el portaobjetos en la parte superior de la cinta y marque la posición de cada orificio con una aguja. Con una cuchilla de afeitar, corta alrededor de cada agarre para crear un canal. (B) Pele un lado de la cinta y péguela en la pieza de vidrio. Asegúrese de que ambos orificios estén dentro del canal. Retire el segundo extremo de la cinta y pegue el cubreobjetos de biotina-PEG en la parte superior. (C) Inserte el tubo de polietileno en cada orificio y selle el tubo en su lugar con epoxi, sellando también cada pieza de vidrio al cubreobjetos. (D) Después de usar ambos canales, saque el tubo y coloque la celda de flujo en un frasco de tinción lleno de acetona. Después de aproximadamente 24 horas, el epoxi y la cinta se habrán ablandado, y las capas de la celda de flujo se pueden despegar. Las piezas de vidrio se pueden recuperar y almacenar en acetona para ser reutilizadas indefinidamente para hacer la siguiente celda de flujo. Haga clic aquí para ver una versión más grande de esta figura.
4. Ensayo TIRF de molécula única para visualizar el desenrollado del ADN mediado por CMG
- Prepare los siguientes búferes.
- Prepare el tampón de bloqueo (20 mM de Tris, pH 8,0; 50 mM de NaCl; 2 mM de EDTA; 0,2 mg/mL de BSA). Filtre el tampón con un filtro de jeringa de 0,22 μm y guárdelo a 4 °C.
- Prepare 10x Tampón de reacción 1 (250 mM HEPES pH 7.5; 50 mM NaCl; 100 mM de acetato de magnesio). Filtre el tampón con un filtro de jeringa de 0,22 μm y guárdelo a 4 °C.
- Prepare 10x Tampón de reacción 2 (250 mM Tris, pH 7,5; 100 mM de acetato de magnesio; 1,25 m de glutamato de potasio; 1 mM de EDTA; 0,025% Tween-20). Filtre el tampón con un filtro de jeringa de 0,22 μm y guárdelo a 4 °C.
- Desgasifique aproximadamente 2,5 mL de tampón de bloqueo y 5 mL de agua ultrapura colocando los tubos abiertos en un desecador y dejándolos al vacío durante 15 min.
NOTA: No llene los tubos hasta el borde, ya que las burbujas durante la desgasificación pueden hacer salpicaduras de líquido. - Coloque la celda de flujo en una platina de microscopio y asegúrela en su lugar con una masilla adhesiva en cada extremo. Es importante asegurar bien para evitar que la celda de flujo se mueva más tarde durante la adquisición.
- Conecte el tubo de salida de la celda de flujo a la bomba de jeringa, que tiene un tubo unido a la jeringa con una aguja en el otro extremo. Inserte esta aguja en el tubo de salida para conectar la celda de flujo.
- Encienda el calentador de objetivos a 30 °C.
- Asegure 1-2 mL de agua desgasificada en un tubo a una pieza separada de masilla adhesiva cerca de la celda de flujo de modo que el tubo de entrada pueda insertarse y llegar al fondo del tubo.
- Haga fluir el agua a través del canal, luego use un flujo más rápido para eliminar las burbujas atrapadas cerca de la tubería de entrada, agregando más agua si es necesario. De lo contrario, las burbujas grandes pueden desprenderse más adelante en los experimentos y pasar a través del canal. Deje actuar unos minutos para que el flujo se detenga por completo, ya que el flujo rápido puede causar una extracción residual de líquido durante un tiempo después de que se detenga, ya que la presión dentro de la jeringa se estabiliza.
NOTA: Cualquier ADN que entre en contacto con una burbuja se vuelve inutilizable, incluso una vez rehidratado. - Añadir 100 μL de tampón bloqueante desgasificado a una alícuota de 20 μL de 1 mg/mL de estreptavidina. Conecte el tubo abierto a la masilla adhesiva y transfiera el tubo de entrada del agua a la estreptavidina. Fluir a una velocidad de 40 μL/min durante 2 min (80 μL en total) e incubar durante 5 min.
- Lave el exceso de estreptavidina con el tampón de bloqueo (50 μL/min para 100 μL).
- Flujo en ADN biotinilado, diluido en tampón bloqueante con naranja SYTOX 25 nM. Imagen utilizando la vista en vivo con el láser de 532 nm para ver el ADN que se adhiere a la superficie en tiempo real.
- Cuando se alcanza la densidad aproximada de ADN en la superficie, se introduce un tampón de bloqueo, también con SYTOX de 25 nM (50 μL/min para 100 μL), para lavar el ADN libre.
- Flujo en anticuerpo anti-digoxigenina biotinilado (~10 μg/mL) diluido en tampón bloqueante que contiene 25 nM de naranja SYTOX (100 μL/min para 300 μL).
NOTA: La naranja SYTOX se intercala entre los pares de bases del ADN, extendiendo la longitud del contorno. Esto significa que la longitud de extremo a extremo del ADN aumenta en la naranja SYTOX, lo que permite que la segunda correa se adhiera a la superficie más lejos de la primera, lo que proporciona un ADN mejor estirado cuando la naranja SYTOX se lava más tarde. El uso de una fracción diferente en cada extremo del ADN permite una fijación más uniforme del ADN a la superficie del cubreobjetos (Figura 4). Cuando ambos extremos están biotinilados, se unen en el mismo paso, lo que resulta en una variación significativa en sus posiciones de fijación. El uso de digoxigenina en un extremo proporciona más control sobre la cobertura del ADN antes de atar el segundo extremo. Esto también permite aumentar la cobertura de ADN por incubación. - Lavar el anticuerpo anti-digoxigenina biotinilado y la naranja SYTOX con tampón bloqueante (50 μL/min para 100 μL).
- Prepare 120 μL de 'mezcla ATP-g-s' (1x Tampón de reacción 1, 0,75 mg/mL BSA, 1,25 mg/mL de caseína, 8 mM DTT, 0,33 mM ATP-g-s), agregue 30 μL a un nuevo tubo, luego fluya 50 μL desde el tubo original a la celda de flujo (50 μL/min para 50 μL). Esto ayuda a minimizar el efecto de la mezcla del tampón a través del reflujo o la difusión desde el tubo de salida.
- Agregue CMG purificado a ~ 100 nM final en 30 μL de mezcla de ATP-g-s, luego fluya a 20 μL/min durante 20 μL. Incube durante 15 min.
- Constituya 120 μL de "mezcla de ATP/RPA" (1x Tampón de reacción 1, 0,75 mg/mL BSA, 8 mM DTT, 3,3 mM ATP, 20 nM EGFP-hRPA) y fluya en la celda de flujo a 40 μL/min para 80 μL.
- Comience a adquirir imágenes de inmediato. Adquiera campos de visión de 6 x 6 para cada fotograma cada 30 s (o una velocidad de fotogramas adecuada para el experimento). Las velocidades de fotogramas más bajas pueden reducir la exposición al láser y el fotoblanqueo con fluoróforos. Visualice EGFP-hRPA con un láser de 488 nm al 1 % de potencia. Si CMG está etiquetado, por ejemplo, LD655, visualice CMG con láser de 640 nm (potencia máxima = 30 mW) al 10% de potencia. Visualice el ADN teñido de naranja de SYTOX con un láser de 532 nm al 0,5 % de potencia. Utilice cada láser con una exposición de 50-100 ms.
NOTA: Cada vez que el tubo de entrada de la celda de flujo se transfiera de un tubo de líquido a otro, frote la punta del tubo en la parte inferior del tubo varias veces antes de sacarlo del líquido. Esto ayuda a evitar que entren burbujas durante la transferencia. Minimice el tiempo que la punta del tubo de entrada se mantiene fuera del líquido y no apunte la punta hacia arriba ni la eleve significativamente, ya que esto hace que el líquido se drene hacia el canal y empuje el aire hacia la punta. Mientras transfiere el tubo de entrada, trate de no poner tensión en el tubo, ya que esto puede sacarlo del epoxi, asegurándolo a la celda de flujo. Para obtener imágenes del desenrollado del ADN, en presencia de naranja SYTOX para visualizar el ADN bicatenario, reemplace el tampón de reacción 10x 1 por el tampón de reacción 10x 2 para la combinación de ATP / RPA. La naranja SYTOX se une mejor al ADN en estas condiciones. Vacíe completamente la bomba de jeringa al final de cada experimento. Lavar de vez en cuando llenando con agua y vaciando varias veces o desmontando y lavando con agua jabonosa.

Figura 4: ADN anclado a la superficie. (A) Al atar sustratos de ADN con biotina en ambos extremos, la distancia entre los dos cables puede variar dependiendo de cómo los extremos entran en contacto con la superficie (i). Mediante el uso de digoxigenina en un extremo, el anclaje de cada extremo se puede separar temporalmente para obtener distancias de amarre más consistentes y un ADN estirado más uniformemente (ii). (B) Ejemplo de campo de visión que muestra ADN atado por ambos extremos (marcado con digoxigenina) y teñido con tinción de ácido nucleico intercalante fluorescente. El ADN, que está atado por ambos extremos, aparece como una línea, mientras que el ADN atado por un solo extremo aparece como manchas. Idealmente, el ADN debe estar atado lo más densamente posible sin superponerse a otros ADN. La imagen mide 512 x 512 píxeles (tamaño de píxel = 154,6 nm). Haga clic aquí para ver una versión más grande de esta figura.
Resultados Representativos
Cuando CMG desenrolla el ADN, un tracto RPA característico crecerá con el tiempo (Figura 5). El extremo 5' del ADN desenrollado está atado a la superficie; por lo tanto, se ve como un tramo lineal de señal RPA entre el cable y la bifurcación. El extremo 3' no está atado y, por lo tanto, se mueve con la horquilla y se observa como una señal EGFP-RPA compacta. La posición de la hebra de translocación sin enrollar compactada corresponde aproximadamente a la posición de la horquilla de replicación, que se mueve junto con LD655-CMG visualizada a través de un láser de 640 nm.
Es importante minimizar el daño al sustrato del ADN, ya que los daños como las muescas de ADN monocatenario reducen el número de eventos de desenrollado observables, lo que limita la cantidad de datos que se pueden recopilar (Figura 6).
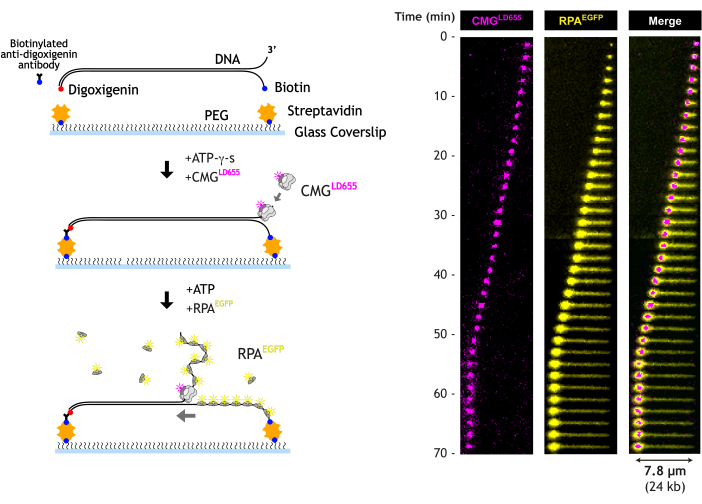
Figura 5: Ensayo de desenrollado de ADN de una sola molécula. El sustrato de ADN está atado a una superficie de cubreobjetos. El CMG purificado marcado con LD655 se incuba con el ADN durante 15 min en ATP-g-s. Se añaden ATP y RPA purificado marcado con EGFP, lo que inicia un extenso desenrollado del ADN por CMG. Se muestra un esquema de dibujos animados (izquierda) y un quimograma de datos representativos (derecha). Haga clic aquí para ver una versión más grande de esta figura.
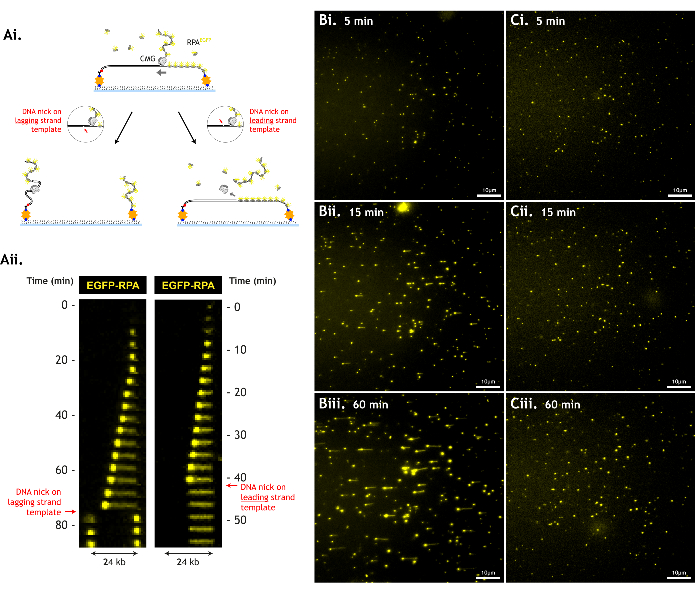
Figura 6: El daño en el ADN reduce el rendimiento del ensayo. (A) El CMG no puede desenrollar el ADN más allá de una ruptura en la columna vertebral del ADN (corte de ADN). Una muesca en la plantilla de hebra principal hace que CMG se deslice fuera del ADN, y tanto CMG como la plantilla de hebra principal se pierden. Una muesca en la plantilla de hebra rezagada hace que la molde de hebra rezagada se separe del resto del ADN, y cada pieza de ADN se retrae a su respectiva atadura. Esto se ilustra con (i) esquemas de dibujos animados y (ii) kymogramas de estos eventos (ii). Datos representativos con (B) un sustrato de ADN mínimamente dañado frente a (C) un sustrato de ADN más dañado a (i) 5 min, (ii) 15 min y (iii) 60 min en un solo campo de visión. El sustrato de ADN más dañado no genera largos tramos de desenrollado, ya que los CMG encuentran muescas antes, a pesar de niveles similares de actividad de desenrollado (densidad similar de puntos de RPA en crecimiento a 5 min, lo que indica una eficiencia de carga/desenrollado de CMG similar). El campo de visión es de 512 x 512 píxeles (tamaño de píxel = 154,6 nm). Obtención de imágenes EGFP-RPA con una potencia láser del 1% (488 nm). Barra de escala que muestra 10 μm. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este ensayo proporciona una plataforma para observar e investigar la dinámica en tiempo real de los CMG individuales, tanto de forma aislada como en el contexto de los factores adicionales deseados. Sin embargo, al igual que con muchas técnicas de fluorescencia de una sola molécula, existen algunos desafíos comunes que pueden requerir optimización para superarlos. Por lo general, se relacionan con la obtención de imágenes de fluoróforos durante largos períodos de tiempo (fotoblanqueo, brillo), la preparación del sustrato del ADN (daño en el ADN), la calidad de la superficie de la celda de flujo (ruido de fondo, interacciones no específicas) o la calidad de la preparación de la proteína purificada (contaminación por nucleasas, eficiencia del etiquetado).
Cada fluoróforo varía en fotoestabilidad y brillo, por lo que es importante elegir una molécula adecuada. Cuando se obtienen imágenes de proteínas oligoméricas marcadas con fluorescencia, como RPA, se puede utilizar una potencia láser más baja, ya que muchos fluoróforos se excitarán muy cerca, generando una señal visible. Para obtener imágenes de fluoróforos individuales, por ejemplo, CMG marcado en una sola subunidad, se necesita una mayor potencia láser para observar el fluoróforo con claridad. La vida útil del fluoróforo se puede prolongar minimizando la exposición al láser, por ejemplo, reduciendo la frecuencia a la que se toman las imágenes. Además, la excitación de un fluoróforo genera especies reactivas de oxígeno (ROS), que pueden contribuir al fotoblanqueo. La inclusión de un sistema de eliminación de oxígeno en el tampón de imágenes puede prolongar la vida útil de los fluoróforos al eliminar las ROS. Sin embargo, algunos sistemas de eliminación de oxígeno pueden afectar el pH12.
En cuanto a la preparación del sustrato del ADN, es crucial minimizar el daño al ADN, como muescas o huecos monocatenarios. El daño excesivo impide el desenrollado extenso del ADN, lo que limita la cantidad de datos que se pueden recopilar. El daño puede surgir del cizallamiento mecánico, el calentamiento excesivo, como resultado de la contaminación por nucleasas o las ROS generadas durante la toma de imágenes. El cizallamiento se puede minimizar manipulando la muestra de ADN con cuidado mediante el uso de puntas de diámetro ancho para el pipeteo, pipeteando lentamente y evitando mover la muestra. El efecto de las ROS se puede minimizar reduciendo la exposición al láser o incluyendo un sistema de eliminación de oxígeno en el tampón de imagen. Después de la preparación del sustrato de ADN, es posible utilizar kits comerciales de reparación de ADN para reparar el daño antes de realizar una reacción de desenrollado.
La eficiencia del desenrollado del ADN también depende de la pureza y la actividad de CMG. Es una buena práctica evaluar la pureza de la muestra después de cada paso de purificación mediante electroforesis SDS-PAGE para determinar dónde es necesaria la optimización. Si se observan demasiados contaminantes después del paso final, puede ser útil modificar los volúmenes de gradiente de sal utilizados para la elución de la columna CaptoHiRes Q (5/50). También es muy importante eliminar cualquier exceso de péptido fluorescente utilizado para el etiquetado de proteínas, ya que puede crear un fondo indeseable en la superficie del cubreobjetos. También es esencial evitar la contaminación por nucleasas, ya que puede degradar el sustrato del ADN. Después de un experimento, teñir el ADN restante con naranja SYTOX puede ser una buena manera de comprobar si el ADN se ha degradado significativamente o no. Un cierto nivel de daño en el ADN es inevitable en el transcurso de un experimento, pero un daño significativo a menudo indica una contaminación problemática de la nucleasa.
El ensayo también está inherentemente limitado por la resolución de los puntos limitados por difracción, lo que requiere que las proteínas fluorescentes estén a cientos de pares de bases de distancia (si no más) para distinguirlas como separadas. Esto limita el detalle con el que se pueden observar la progresión y las interacciones de CMG.
El número de eventos de desenredo que observamos para cada análisis varía. Para un experimento exitoso, esperamos ver al menos varios tramos de RPA de suficiente longitud por campo de visión de 512x512 píxeles (tamaño de píxel = 154,6 nm). Se pueden obtener imágenes de varios campos de visión en el mismo experimento, lo que permite una mayor recopilación de datos cuando sea necesario. No es necesario que los tractos tengan la misma longitud ni lleguen al final del ADN para ser útiles. Por ejemplo, la distancia media de amarre para cada experimento puede determinarse midiendo la longitud del ADN teñido con SYTOX antes de añadir CMG. Esto se puede utilizar para estimar la cantidad de ADN que se ha desenrollado para cualquier tracto RPA (siempre que se desenrolle suficiente ADN para mover visiblemente la horquilla) mediante la conversión de la distancia de 'μm recorrido' a 'kb desenrollado'.
CMG exhibe actividad de desenrollado en una variedad de sustratos de ADN, pero es esencial proporcionar un extremo de ADN 3' libre en un colgajo de polyT de al menos 30 nt para acomodar la huella de CMG10. La inclusión de múltiples restos de biotina en la horquilla garantiza un anclaje robusto de la superficie. El resto del sustrato de ADN se puede rediseñar de muchas maneras, como para incluir diferentes secuencias de ADN, longitudes y modificaciones químicas. La conformación del ADN puede alterarse mediante el uso de diferentes concentraciones de acetato de magnesio. A concentraciones más altas (≥10 mM) de acetato de magnesio, el filamento de ssDNA recubierto de RPA se compacta, lo que hace que el ADN sea arrastrado por la unión de RPA durante el desenrollado. Esto puede ser útil, ya que evita que el ADN se mueva excesivamente, lo que permite medir con mayor precisión la posición de la CMG y de la progresión del desenrollado. A bajas concentraciones (~3 mM) de acetato de magnesio, el RPA-ssDNA permanece relajado en todo momento.
El ensayo de una sola molécula descrito representa una plataforma sobre la que se puede construir y modificar para investigar más aspectos de la replicación del ADN. Durante la replicación del ADN, la CMG actúa como un núcleo alrededor del cual se ensamblan el replisma y sus componentes. Por lo tanto, se pueden agregar proteínas purificadas adicionales a este ensayo, incluidos factores accesorios como TIMELESS, TIPIN y CLASPIN, para estudiar su efecto en la dinámica del CMG. Se ha demostrado que estas proteínas afectan la tasa de replicación de las horquillas13, pero no está claro cómo afectan la tasa de desenrollado de CMG. Por lo tanto, sería interesante investigar cómo las diferentes proteínas replisómicas afectan a la CMG utilizando este ensayo. La adición de ADN polimerasas puede proporcionar una mejor comprensión de la replicación del ADN más allá del desenrollado del ADN por sí solo, como se describió anteriormente con las proteínas de levadura14. Además, la purificación de CMG modificada puede proporcionar una mejor comprensión de cómo ciertas mutaciones o modificaciones postraduccionales afectan la actividad de la helicasa15,16. Además, el diseño de diferentes sustratos de ADN puede permitir que el desenrollado del ADN por CMG se estudie en una variedad de condiciones que imitan el estrés de replicación17. Estas modificaciones incluyen obstáculos de ADN 9,18, enlaces cruzados entre hebras 19,20,2 1 y discontinuidades en las hebras de ADN22.
Divulgaciones
Los autores no tienen intereses financieros contrapuestos ni otros conflictos de intereses.
Agradecimientos
Agradecemos a Gheorghe Chistol por proporcionar el plásmido pGC261 y a la Instalación de Biología Química del Instituto Francis Crick para la síntesis y el etiquetado de péptidos. Este trabajo fue financiado por el Instituto Francis Crick, que recibe fondos básicos de Cancer Research UK, el Consejo de Investigación Médica del Reino Unido y The Wellcome Trust (CC2133).
Materiales
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma-Aldrich | M3148-25ML | |
| Acrylamide / Bis Solution 40% | BioRad | 1610148 | |
| Agarose UltraPure | Invitrogen | 16500-500 | Used to prepare Tris Buffer (pH 8) |
| ÄKTA Pure | GE Healthcare | - | Used with Fraction Collector F9-C; protein purification system |
| ANTI-FLAG M2 affinity gel | Sigma-Aldrich | A2220 | |
| APS 10% | Sigma-Aldrich | A3678-25G | |
| ATP (200 mM) | Sigma-Aldrich | A7699-5G | |
| ATP-g-s (100 mM) | Sigma-Aldrich | 11162306001 | |
| Biotin-PEG coverslip | N/A | N/A | Prepared as described: https://doi.org/10.1016/j.ymeth.2012.03.033 |
| Biotinylated anti-digoxigenin antibody | Perkin Elmer | N/A | |
| Blu Tack | Bostik | N/A | Adhesive putty |
| Boric acid | Thermo Fisher Scientific | B/3800/53 | |
| BSA; 33 mg/mL | SIGMA-ALDRICH | A3858-10G | Diluted from the stock |
| CaCl2 | Sigma-Aldrich | C7902 | |
| CaptoHiRes Q (5/50) | Cytiva | 29275878 | Connected to the AKTA protein purification system; high-resolution ion exchange chromatography column |
| Casein (5% in water, 50 mg/mL) | Sigma-Aldrich | C4765-10ML | |
| ChemiDoc system | Bio-Rad | N/A | |
| Clips | N/A | N/A | |
| Cutting board | N/A | N/A | |
| Dialysis tubing (3.5 kDa) | Fisher Scientific | 11425859 | |
| digoxigenin-11-dUTP | Roche | 11209256910 | |
| DNA loading dye 6x | NEB | B7024S | |
| dNTP 10 mM | NEB | N0447S | |
| Double-sided tape (0.14 mm thick) | N/A | N/A | |
| DTT | Fluorochem Limited | M02712-10G | |
| DYKDDDDK peptide | N/A | N/A | Synthetised by chemical biology STP of The Francis Crick Institute |
| EDTA | Thermo Fisher Scientific | D/0700/68 | |
| EGFP-RPA | N/A | N/A | Purified as desribed in ‘Single Molecule Analysis’, Series Methods in Molecular Biology, Vol. 783 (2011), Peterman, E. J. G. and Wuite G. J. L. editors, Humana Press. Protein information: 2 mM, human, expressed in E. coli. |
| EGTA | Sigma-Aldrich | 03779-10G | |
| Epoxy - 5 minute | Devcon | 20845 | |
| Fujifilm FLA-5000 | Fujifilm | FLA-5000 | Fluorescent image analyzer |
| GelRed Nucleic Acid Stain; 10000x | Cambridge Bioscience | 41003-BT | Example of nucleic acid stain which is a safer alternative to ethidium bromide. |
| Glass or quartz slide with holes for tubing | Dremel Model 395 | 1 mm thick slide, cut to 2.4 cm x 1cm, two holes drilled 1.4 mm apart. The holes should be drilled to different sizes to accommodate different tubing at each end. | |
| Glycerol | Fisher Scientific | G/0650/17 | |
| Glycine HCl, pH 3.5 | Sigma-Aldrich | G8898 | |
| Hamilton syringe, 1000 series GASTIGHT, PTFE luer lock 1010TLL, PTFE Luer lock (with slots), volume 10 mL | Merck | 26211-U | |
| HEPES pH 7.5 | Sigma-Aldrich | H4034 | |
| I-CeuI | NEB | R0699L | |
| KCl | Fisher Scientific | P/4280/53 | |
| Magnesium acetate | Sigma-Aldrich | M2545 | |
| Maxi GeBaFlex-tube Dialysis Kit; MWCO 14 kDa | Generon | D055 | |
| Metal spatula | N/A | N/A | |
| Metal tweezers | N/A | N/A | |
| MgCl2 | Sigma-Aldrich | M2393 | |
| Millex-GP (Syringe filters) 0.22 µm | Merck | SLGP033RS | |
| NaCl | Sigma-Aldrich | S7653 | |
| Needle | N/A | N/A | |
| NotI-HF | NEB | R3189L | |
| NuPAGE 4%–12% Bis-Tris Protein Gels | Thermo Fisher Scientific | 10247002; | |
| NuPAGE MOPS SDS Running Buffer | Thermo Fisher Scientific | NP0001 | |
| Objective heater | Okolab | N/A | |
| PCR mix - 2x | Prepared from Phusion DNA polymerase (20 µL), 10 mM dNTPs (40 µL), 5x Phusion HF buffer (400 µL) and water (540 µL). | ||
| Peptide NH2-CHHHHHHHHHHLPETGG-COOH, labelled with LD655-MAL | N/A | N/A | peptide NH2-CHHHHHHHHHHLPETGG-COOH, labeled with LD655-MAL (Lumidyne Technologies) on the cysteine residue, was synthetised and purified by the peptide chemistry STP of The Francis Crick Institute |
| pGC261 plasmid | N/A | N/A | https://doi.org/10.1016/j.cell.2018.10.053 |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Plastic tweezers | N/A | N/A | |
| Polyethylene tubing PE20 (inner diameter 0.015”, outer diameter 0.043”) | Becton Dickinson | 427406 | |
| Polyethylene tubing PE60 (inner diameter 0.03”, outer diameter 0.048”) | Becton Dickinson | 427416 | |
| Poly-Prep Chromatography Columns; 10 ml and 20 ml | Bio-Rad | 7311550 | |
| Potassium Glutamate (L-glutamic acid potassium salt monohydrate) | Sigma | G1501-1KG | |
| Protease inhibitor cocktail (cOmplete, EDTA free) | Roche | 5056489001 | |
| Pump 11 Elite Infusion/Withdrawal Programmable Single Syringe | Harvard Apparatus | 70-4504 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| Razor blade | VWR International Ltd | 233-0156 | |
| rCutSmart Buffer; 10x | NEB | B6004S | |
| Sodium acetate | Thermo Fisher Scientific | S/2120/53 | |
| Sortase (pentamutant) | N/A | N/A | Purified based on: https://doi.org/10.1073/pnas.1101046108 |
| Spin-X Centrifuge Tube Filters; 0.22 µm cellulose acetate | Fisher Scientific | 10310361 | |
| Sterile scalpel | N/A | N/A | |
| Steritop Vacuum Driven Disposable Filtration System; 0.22 um; PES; | Millipore | S2GPT05RE | |
| Streptavidin (1 mg/mL) in 1x PBS buffer | Sigma | S4762-10MG | 20 µL aliquotes |
| SYBR Gold | Thermo Fisher Scientific | S11494 | Highly sensitive nucleic acid stain we used to visualise DNA fork substrate. |
| SYTOX orange; 5 µM in DMSO | Thermo Fisher Scientific | S11368 | |
| T4 DNA ligase | NEB | M0202M | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | |
| TEMED | Sigma-Aldrich | T9281-25ML | |
| TEV protease (1 mg/mL); EZCut | Biovision | 7847-10000 | |
| Tissue grinders, Dounce type (40 mL, Wheaton) | DWK Life Sciences | 432-1273 | |
| Tris base | Sigma-Aldrich | T1503 | Used to prepare Tris Buffer (pH 8) |
| Tris HCl | Sigma-Aldrich | T3253 | |
| Tween-20 | Promega UK Ltd | H5152 |
Referencias
- Bell, S. P., Labib, K. Chromosome Duplication in Saccharomyces cerevisiae. Genetics. 203 (3), 1027-1067 (2016).
- Costa, A., et al. The structural basis for MCM2-7 helicase activation by GINS and Cdc45. Nat Struct Mol Biol. 18 (4), 471-477 (2011).
- Jones, M. L., Baris, Y., Taylor, M. R. G., Yeeles, J. T. P. Structure of a human replisome shows the organisation and interactions of a DNA replication machine. EMBO J. 40 (23), e108819 (2021).
- Moyer, S. E., Lewis, P. W., Botchan, M. R. Isolation of the Cdc45/Mcm2-7/GINS (CMG) complex, a candidate for the eukaryotic DNA replication fork helicase. Proc Natl Acad Sci U S A. 103 (27), 10236-10241 (2006).
- Douglas, M. E., Ali, F. A., Costa, A., Diffley, J. F. X. The mechanism of eukaryotic CMG helicase activation. Nature. 555 (7695), 265-268 (2018).
- Ilves, I., Petojevic, T., Pesavento, J. J., Botchan, M. R. Activation of the MCM2-7 helicase by association with Cdc45 and GINS proteins. Mol Cell. 37 (2), 247-258 (2010).
- Dewar, J. M., Walter, J. C. Mechanisms of DNA replication termination. Nat Rev Mol Cell Biol. 18 (8), 507-516 (2017).
- Burnham, D. R., Kose, H. B., Hoyle, R. B., Yardimci, H. The mechanism of DNA unwinding by the eukaryotic replicative helicase. Nat Commun. 10 (1), 2159 (2019).
- Kose, H. B., Larsen, N. B., Duxin, J. P., Yardimci, H. Dynamics of the eukaryotic replicative helicase at lagging-strand protein barriers support the steric exclusion model. Cell Rep. 26 (8), 2113-2125.e6 (2019).
- Kose, H. B., Xie, S., Cameron, G., Strycharska, M. S., Yardimci, H. Duplex DNA engagement and RPA oppositely regulate the DNA-unwinding rate of CMG helicase. Nat Commun. 11 (1), 3713 (2020).
- Tanner, N. A., Van Oijen, A. M. Visualizing DNA replication at the single-molecule level. Methods Enzymol. 475, 259-278 (2010).
- Swoboda, M., et al. Enzymatic oxygen scavenging for photostability without ph drop in single-molecule experiments. ACS Nano. 6 (7), 6364-6369 (2012).
- Baris, Y., Taylor, M. R. G., Aria, V., Yeeles, J. T. P. Fast and efficient DNA replication with purified human proteins. Nature. 606, 204-210 (2022).
- Lewis, J. S., et al. Tunability of DNA polymerase stability during eukaryotic DNA replication. Mol Cell. 77 (1), 17-25.e5 (2020).
- Li, Z., Xu, X. Post-translational modifications of the mini-chromosome maintenance proteins in DNA replication. Genes. 10 (5), 331 (2019).
- Lewis, J. S., et al. Mechanism of replication origin melting nucleated by CMG helicase assembly. Nature. 606 (7916), 1007-1014 (2022).
- Zeman, M. K., Cimprich, K. A. Causes and consequences of replication stress. Nat Cell Biol. 16 (1), 2-9 (2014).
- Sparks, J. L., et al. The CMG helicase bypasses DNA-protein cross-links to facilitate their repair. Cell. 176 (1-2), 167-181.e21 (2019).
- Taylor, M. R. G., Yeeles, J. T. P. The initial response of a eukaryotic replisome to DNA damage. Mol Cell. 70 (6), 1067-1080.e12 (2018).
- Amunugama, R., et al. Replication fork reversal during DNA interstrand crosslink repair requires CMG unloading. Cell Rep. 23 (12), 3419-3428 (2018).
- Wu, R. A., et al. TRAIP is a master regulator of DNA interstrand crosslink repair. Nature. 567 (7747), 267-272 (2019).
- Vrtis, K. B., Dewar, J. M., Chistol, G., Wu, R. A., Graham, T. G. W., Walter, J. C. Single-strand DNA breaks cause replisome disassembly. Mol Cell. 81 (6), 1309-1318.e6 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados