Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis electroforético de la replicación a través de repeticiones de ADN propensas a la estructura dentro del episoma humano basado en SV40
En este artículo
Resumen
Aquí, describimos el procedimiento para analizar la progresión de la replicación a través de repeticiones patógenas propensas a la estructura utilizando electroforesis en gel bidimensional.
Resumen
La electroforesis bidimensional en gel neutro/neutro (2DGE) surgió como una técnica de referencia para analizar la replicación del ADN a través de impedimentos naturales. Este protocolo describe cómo analizar la progresión de la horquilla de replicación a través de repeticiones de ADN expandibles y propensas a la estructura dentro del episoma basado en el virus del simio 40 (SV40) en células humanas. En resumen, tras la transfección de plásmidos en células humanas, los intermedios de replicación se aíslan mediante el protocolo Hirt modificado y se tratan con la enzima de restricción DpnI para eliminar el ADN no replicado. A continuación, los intermedios son digeridos por enzimas de restricción apropiadas para colocar la repetición de interés dentro de la mitad distal de origen de un fragmento de ADN de 3-5 kb de largo. Los intermedios de replicación se separan en dos dimensiones perpendiculares, primero por tamaño y luego por forma. Después de la hibridación de Southern blot, este enfoque permite a los investigadores observar el estancamiento de la horquilla en varias repeticiones de formación de estructuras en la mitad descendente del arco Y de replicación. Además, este posicionamiento del sitio de pérdida permite la visualización de varios resultados del estancamiento de la horquilla mediado por repetición, como la inversión de la horquilla, el advenimiento de una horquilla convergente y el reinicio recombinante de la horquilla.
Introducción
Las repeticiones cortas en tándem (STR) son secuencias repetitivas de ADN pequeñas, normalmente de 2 a 9 pares de bases (bp), que constituyen alrededor del 3%del genoma humano. Los STR juegan un papel importante en la regulación génica2; sin embargo, su composición repetitiva los hace propensos a la formación de estructuras secundarias de ADN no canónicas y a la posterior inestabilidad genética 3,4. Desde las hélices levógiras hasta las horquillas/cruciformes, pasando por las hélices de tres y cuatro hebras, estas estructuras alternativas de ADN causan desafíos intrínsecos para el replisoma. Un requisito natural para la formación de estructuras secundarias es el desenrollado del ADN, que es un requisito previo para la replicación del ADN. Esto presenta un enigma único para el funcionamiento del genoma, ya que muchas de estas estructuras pueden formarse durante la replicación, lo que dificulta la progresión del replicación y, en última instancia, causa el estancamiento de la horquilla de replicación 5,6,7 o, en casos graves, el colapso de la horquilla y la rotura del ADN 8,9. Se ha demostrado que tanto el reinicio de las horquillas estancadas como las vías de reparación del ADN conducen a inestabilidad repetida, como expansiones repetidas10,11 y reordenamientos complejos del genoma (CGR)12,13. Estos eventos pueden resultar en el desarrollo de aproximadamente 60 enfermedades humanas conocidas como trastornos de expansión repetida, incluyendo el síndrome de X frágil, la enfermedad de Huntington, la ataxia de Friedreich y otras14,15, así como enfermedades CGR, como el síndrome de Emmanuel16. Por lo tanto, para comprender mejor los mecanismos de las enfermedades humanas impulsadas por la inestabilidad de las repeticiones, es imperativo estudiar los detalles de la progresión de la horquilla de replicación a través de esas repeticiones.
Una técnica para estudiar la progresión de la replicación surgió a mediados de la década de 1980 cuando Brewer y Fangman buscaron proporcionar evidencia directa de que el inicio de la replicación en Saccharomyces cerevisiae ocurre en elementos de secuencia de replicación autónoma (comúnmente conocida como ARS)17. Al hacerlo, separaron las estructuras de los intermedios de replicación de la levadura en la agarosa, adaptando un método anterior de Bell y Byers conocido como electroforesis en gel neutra/neutro en 2 dimensiones (2DGE)18. Esta técnica utilizó el hecho de que el ADN no lineal viaja de manera diferente en gel de agarosa que su equivalente lineal de la misma masa. Más específicamente, en 2DGE, el ADN aislado se separa en dos dimensiones perpendiculares, primero principalmente por tamaño y luego principalmente por forma, para crear un mapa completo de la replicación en una región particular de interés. En su artículo original, Brewer y Fangman demostraron esto como un arco compuesto por estructuras "Y simples" o horquillas de replicación que unen el ADN no replicado con sus contrapartes replicadas. Además, describen otros intermedios observados como "burbujas" y "dobles Y", que representan orígenes de replicación y horquillas convergentes, respectivamente.
2DGE se puede utilizar para estudiar las poblaciones relativas de intermediarios de replicación del ADN en un momento dado. Por lo tanto, si una población de intermediarios es más prevalente que otra, esto sería evidente en la visualización. Esto hace que 2DGE sea una herramienta especialmente útil para estudiar la progresión de la replicación a través de secuencias desafiantes, como las repeticiones de formación de estructuras. Por ejemplo, si la región analizada contiene una secuencia capaz de inducir el estancamiento de la horquilla de replicación, esto se presentaría como una protuberancia en el arco (Figura 1A), lo que indica una acumulación de bifurcaciones de replicación en ese locus. Esto se puede ver con la replicación de las secuencias de repeticiones que forman horquillas en la levadura 19,20,21 y las repeticiones que forman triplex en células humanas 22,23,24. Además del estancamiento, el 2DGE se puede utilizar para observar estructuras de ADN que no se ajustan a los Ys simples estándar formados durante la replicación, como en el caso de los intermedios recombinantes25. Estos intermedios tienen una estructura en forma de X más pesada y ramificada y, por lo tanto, viajan más lentamente tanto en la primera como en la segunda dimensión que las horquillas de replicación estándar. También se pueden observar resultados similares con respecto a la inversión de la horquilla de replicación 20,24,26. En respuesta al fuerte estrés de replicación, se ha demostrado que las células eucariotas utilizan la inversión de la horquilla de replicación para rescatar las horquillas estancadas. Estas horquillas invertidas tienen un peso molecular similar al de las horquillas estancadas; sin embargo, su estructura de pata de gallina da como resultado una movilidad electroforética más lenta en la segunda dimensión en relación con sus complementos en forma de Y, lo que resulta en una extensión hacia arriba y hacia afuera del arco.
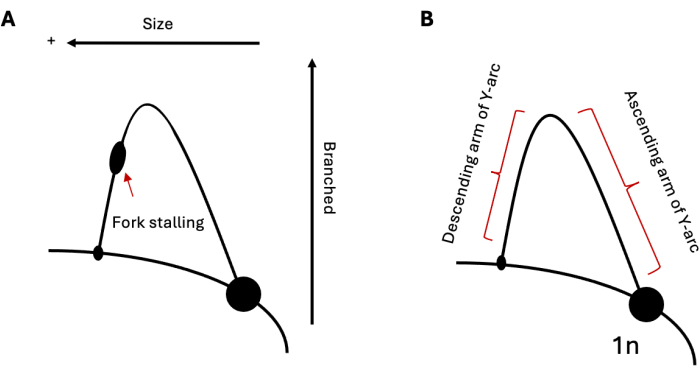
Figura 1: Análisis de electroforesis en gel 2D de la replicación del ADN. (A) Esquema de un 2DGE típico que representa la replicación a través de una repetición de formación de estructura capaz de inducir el estancamiento de la horquilla. El tamaño y la estructura intermedios influirán en la movilidad electroforética. (B) Arco Y de muestra con brazos ascendentes y descendentes respectivamente etiquetados. Abreviatura: 2DGE = Electroforesis bidimensional en gel neutro/neutro. Haga clic aquí para ver una versión más grande de esta figura.
Naturalmente, uno de los aspectos más importantes de 2DGE se refiere a la calidad y cantidad de los intermedios de replicación. Sin embargo, la resolución del análisis 2DGE de la replicación a través de loci endógenos en células de mamíferos es insuficiente para una secuencia diana de una sola copia dentro del genoma humano diploide de 6 × 109 pb, aunque se ha hecho para genes de múltiples copias, como el locusDHFR 27 fuertemente amplificado o el ARN ribosómico28. La replicación basada en SV40 es un medio eficiente y bien caracterizado para estudiar la replicación en células eucariotas29. Proporciona un modelo fiable de replicación eucariota que utiliza la mayor parte de la maquinaria de reploma del huésped para replicar el genoma viral, que se parcela en nucleosomas tras la infección30,31. Dos excepciones notables del replisma de mamíferos son que el antígeno T (Tag), en lugar del complejo CMG del huésped, sirve como helicasa de ADN replicante, y la ADN polimerasa delta sintetiza tanto las hebras de ADN principales como las rezagadas32. Hemos aprovechado este sistema colocando tramos patogénicos de repeticiones formadoras de estructuras aguas abajo de un origen de replicación SV40 dentro de un plásmido que se creó originalmente en el laboratorio de Massimo Lopes22. Es importante destacar que este plásmido también contiene el gen que codifica para el propio Tag, lo que resulta en su replicación constitutiva y extremadamente potente tras la transfección en una variedad de células humanas cultivadas. Esta característica da lugar a una gran cantidad de productos, ideales para el análisis 2DGE de los intermedios formados durante y en respuesta a la replicación de repeticiones patógenas en células humanas. Aquí, describimos un método detallado para visualizar la replicación de repeticiones formadoras de estructuras dentro del episoma humano basado en SV40 utilizando electroforesis en gel bidimensional.
Protocolo
NOTA: El plásmido diseñado para nuestro análisis 2DGE descrito en células de mamíferos debe contener un origen de replicación SV40 varios kb aguas arriba de las repeticiones propensas a la estructura (Figura 2). Se debe tener en cuenta la síntesis inicial y retrasada al elegir qué orientación en relación con el origen deben clonarse las repeticiones en el plásmido.

Figura 2: Digestión de plásmido que contiene repeticiones para el análisis 2DGE. Las repeticiones propensas a la estructura se representan varios kb aguas abajo de la bifurcación de replicación que se mueve a la derecha. La digestión con los cortadores únicos 1 y 2 colocará la secuencia de repetición en el brazo descendente del arco Y, dada la secuencia más allá del punto medio del fragmento digerido. Abreviatura: 2DGE = Electroforesis bidimensional en gel neutro/neutro. Haga clic aquí para ver una versión más grande de esta figura.
1. Transfección de plásmidos en células de mamíferos
- Siembrar 600.000 células HEK293T en una placa de cultivo de tejidos de 10 cm antes de la transfección. Deje que las células se recuperen a 37 °C durante la noche.
NOTA: Se pueden utilizar muchas líneas celulares para este experimento, aunque se recomiendan las celdas que contienen la etiqueta SV40 para obtener resultados óptimos. PRECAUCIÓN: HEK293T células se consideran BSL-2 y todo el trabajo de cultivo debe realizarse en una cabina de bioseguridad utilizando una técnica aséptica adecuada y un EPP adecuado. - Cuando las células alcancen el 60% de confluencia, transfecte 8 μg de ADN plásmido que contenga repeticiones en las células sembradas utilizando los reactivos de transfección adecuados de acuerdo con el protocolo del fabricante.
- Si no aísla los intermedios en este momento, aspire los medios viejos y reemplácelos con 10 ml de medios nuevos después de 24 h.
- Comience a recolectar células 24-48 h después de la transfección.
- Aspire los medios y lávelos cuidadosamente con 10 ml de solución salina tamponada con fosfato (PBS). Separe y recoja las células con 0,5 ml de tripsina y centrifuga a 340 × g durante 4 min.
- Aspirar el sobrenadante y lavar los pellets de la célula con PBS. Volver a centrifugar a 340 × g durante 4 min y aspirar el sobrenadante.
NOTA: El experimento puede detenerse aquí congelando gránulos de celda a -80 °C. Hemos encontrado los intermedios de replicación que aíslan la mejor resolución 48 h después de la transfección; sin embargo, 24 h ha dado resultados viables.
2. Aislamiento de intermedios de replicación
- Vuelva a suspender las células en 1,5 mL de tampón de lisis Hirt modificado [10 mM de tris-HCl (pH 7,5), 10 mM de ácido etilendiaminotetraacético (EDTA)] en tubos cónicos de 50 mL y comience la lisis celular.
- Añadir dodecil sulfato de sodio (SDS) a una concentración final de 0,6% (aproximadamente 650 μL de stock 2% SDS) y proteinasa K a una concentración final de 100 μg/mL (aproximadamente 10 μL de stock 20 mg/mL de proteinasa K) para eliminar las nucleasas.
- Mezclar suavemente pipeteando hasta que quede homogénea e incubar la mezcla a 37 °C durante al menos 90 min.
- Aumente la concentración de NaCl a 1 M (aproximadamente 540 μL de NaCl calzante 5 M) y mezcle suavemente hasta que quede homogéneo. Incubar durante la noche (18-24 h) a 4 °C para permitir la precipitación de restos celulares, ARN y proteínas mediante salado.
NOTA: La mezcla será muy viscosa, así que tenga cuidado y tenga paciencia mientras mezcla bien. - Al día siguiente, separe el ADN de los restos celulares, el ARN y la proteína.
- Centrifugar la mezcla a 29.500 × g durante 45 min a 4 °C.
- Transfiera el sobrenadante que contiene ADN, agregue un volumen de fenol:cloroformo:alcohol isoamílico 25:24:1 (v/v) y mezcle brevemente hasta que quede homogéneo.
PRECAUCIÓN: El fenol:cloroformo:alcohol isoamílico es un material peligroso y debe manipularse con el EPP adecuado en una campana extractora química. - Centrifugar de nuevo a 15.000 × g durante 5 min a temperatura ambiente. Transfiera la capa acuosa a un nuevo tubo cónico.
- Precipitar y lavar el ADN aislado.
- Agregue un volumen de isopropanol puro e incube a temperatura ambiente durante al menos 5 minutos. Centrifugar el ADN a 15.000 × g durante 30 min a 4 °C.
- Decantar el sobrenadante y lavar el pellet con etanol frío al 70% para eliminar el exceso de sal.
- Centrifugar una vez más a 15.000 × g durante 30 min a 4 °C, secar al aire y resuspender suavemente el pellet en el tampón Tris-EDTA (TE) (10 mM de Tris-HCl, 1 mM de EDTA).
NOTA: El experimento se puede pausar aquí y las muestras se pueden congelar a -20 °C; sin embargo, debe evitarse el ciclo de congelación/descongelación, ya que esto puede disminuir la calidad de los intermediarios de replicación del ADN.
3. Preparación de la muestra y electroforesis en gel bidimensional
- Digiere los intermedios aislados de la replicación de plásmidos.
- Agregue 100 unidades de las enzimas de restricción apropiadas a la muestra para digerir el ADN plasmídico, colocando específicamente la secuencia que contiene repeticiones en la mitad distal de origen del fragmento lineal (Figura 2). Además, agregue DpnI para cortar el ADN metilado, eliminando así cualquier ADN plásmido que no se haya replicado completamente en células humanas cultivadas.
NOTA: Para obtener los mejores resultados, las enzimas de restricción deben ser cortadores únicos que produzcan un fragmento de 3-5 kb que coloque la secuencia propensa a la estructura en el brazo descendente del arco Y. - Incubar las muestras a 37 °C durante 6-10 h para permitir la digestión completa del plásmido.
- Precipitar el ADN con 2,5 volúmenes de etanol puro frío e incubar a -20 °C durante la noche, o añadir un volumen de isopropanol e incubar a temperatura ambiente durante 5 min.
- Centrifugar las muestras digeridas y precipitadas a 15.000 × g durante 30 min a 4 °C.
- Decantar el sobrenadante y lavar la muestra con etanol frío al 70%. Volver a centrifugar a 15.000 × g durante 30 min a 4 °C.
- Decantar el sobrenadante, secar al aire durante 10 min y volver a suspender las muestras en 15 μL de tampón TE.
- Agregue 100 unidades de las enzimas de restricción apropiadas a la muestra para digerir el ADN plasmídico, colocando específicamente la secuencia que contiene repeticiones en la mitad distal de origen del fragmento lineal (Figura 2). Además, agregue DpnI para cortar el ADN metilado, eliminando así cualquier ADN plásmido que no se haya replicado completamente en células humanas cultivadas.
- Prepare el gel de agarosa de primera dimensión al 0,4-0,5% en 1x Tris-borato-EDTA (TBE) (89 mM de tris base, 89 mM de ácido bórico, 2 mM de EDTA). Deje que la solución se solidifique durante al menos 1 hora.
- Comience a cargar las muestras en la primera dimensión.
- Cargue la escalera dentro de los primeros 3 cm en relación con el borde más a la izquierda del gel. A continuación, cargue la totalidad de las muestras preparadas, dejando 3 cm entre cada par.
- Pasar el gel en 1x TBE durante 19-24 h a 0,85 V/cm para separar los intermedios con respecto a su tamaño. Asegúrese de que la cámara esté cubierta para proteger las muestras de la luz, que puede causar daños al ADN.
- Al día siguiente, retire el gel del tampón y estime la ubicación del fragmento lineal digerido con una regla.
- Extirpar los primeros 3 cm del gel que contiene la escalera y teñir el segmento de gel con 1x TBE que contenga 0,3 μg/mL de bromuro de etidio durante 10-15 min. Visualice la escalera utilizando un sistema de documentación de gel.
- Agregue 1,3 cm a la ubicación estimada, obteniendo el valor a. Luego, resta 7,5 cm del valor a, obteniendo el valor b. Alinee la regla contra el gel de primera dimensión y corte horizontalmente en los valores a y b. A continuación, corte verticalmente el espacio de 3 cm reservado para cada muestra. Consulte la Figura 3 para obtener un esquema visual.
- En una nueva bandeja de fundición, gire los segmentos en el sentido de las agujas del reloj y colóquelos en la posición de los pocillos de muestra (Figura 3).
- Prepare el gel de agarosa de segunda dimensión a una concentración de 1-1,3% en 1x TBE a 0,3 μg/mL de bromuro de etidio.
- Después de enfriar a aproximadamente 55 °C, vierta el gel de segunda dimensión sobre los segmentos de primera dimensión girados y deje que se solidifique durante al menos 1 h.
- Transfiera la segunda dimensión a una cámara con 1x TBE a 0,3 μg/mL de bromuro de etidio y deje que el gel se equilibre durante al menos 30 minutos.
- Pasar el gel, de nuevo tapado, durante 9-10 h a 4,23 V/cm a 4 °C para separar los intermedios con respecto a su forma.
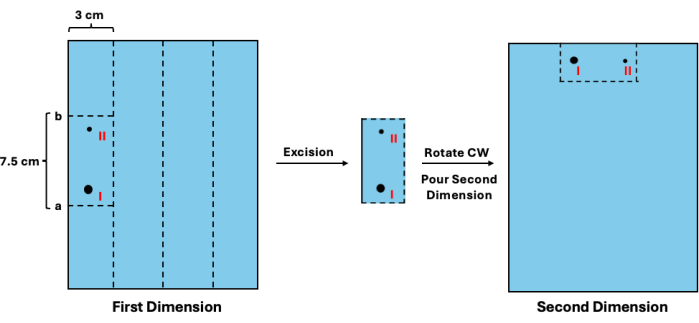
Figura 3: Escisión de intermedios de primera dimensión antes de la separación de segunda dimensión. Después de la visualización de la escalera, se puede estimar la movilidad de los fragmentos no replicados. (I) Este valor se puede utilizar para determinar los sitios de corte apropiados (a y b) para extirpar y sus contrapartes replicadas (II). A continuación, se debe rotar la sección del gel y colocarla en la posición de pocillos para la separación en segunda dimensión. Abreviatura: CW = en el sentido de las agujas del reloj. Haga clic aquí para ver una versión más grande de esta figura.
4. Southern blot e hibridación con sonda radiomarcada
- Retire el gel de segunda dimensión de la cámara y depurine los fragmentos de ADN durante 10 minutos en una solución de HCl de 0,24 M con un balanceo suave. Enjuague el gel con agua desionizada y sumérjalo en NaOH 0,4 M durante 10-15 min.
PRECAUCIÓN: El HCl y el NaOH son corrosivos y deben manipularse con el EPP adecuado en una campana de gases químicos. - Comience a ensamblar el Southern blot para facilitar la transferencia de los intermedios separados del gel a la membrana. Consulte la Figura 4 para obtener un esquema completo.
- Llene un recipiente de tamaño suficiente con 1 L de NaOH de 0,4 M.
- Alinee una hoja de vidrio larga a través del recipiente y doble (a lo largo) dos hojas largas de papel de cromatografía perpendiculares a la hoja de vidrio, extendiéndolas hacia el recipiente de NaOH.
- Humedece la parte superior del papel con NaOH y retira con cuidado las burbujas de aire que queden debajo de su superficie.
- Remoja tres hojas de papel de cromatografía con NaOH y colócalas sobre el papel doblado, eliminando de nuevo las burbujas.
- Dale la vuelta al gel de segunda dimensión y transfiérelo sobre los papeles.
- Humedece una membrana de nailon cargada positivamente (tamaño de poro de 0,45 μm) con agua desionizada y colócala sobre el gel.
- Finalmente, agregue tres hojas más de papel, mojadas con agua desionizada, sobre la membrana.
- Cubra cualquier NaOH expuesto en el recipiente inferior con una envoltura de plástico para evitar la evaporación. Coloque una pila de servilletas o toallas de papel sobre la mancha, asegurándose de que tenga entre 0,3 y 0,5 m de altura. Coloque un peso en la parte superior, comprimiendo toda la mancha para facilitar la acción capilar apretada. Espere al menos 2 días para que el ADN se transfiera a la membrana.
NOTA: La longitud y el ancho del papel de cromatografía y la membrana dependen del tamaño del gel de agarosa utilizado para la segunda dimensión. Para una transferencia más eficiente, utilice papel y membrana con las mismas dimensiones que las del gel.
- Después de la transferencia, entrecruza el ADN con la membrana utilizando un reticulante UV a 120 μJ/cm2 durante 1 min.
NOTA: El experimento puede detenerse aquí colocando la membrana en un protector de hoja hermético, seco y limpio a temperatura ambiente. - Lave la membrana 2 veces durante 5 minutos con 2 citrato de sodio salino (tampón SSC) (0,3 M de NaCl, 0,03 M de citrato de sodio).
- Prehibridar la membrana con 0,18 mL/cm2 de tampón de hibridación de Church & Gilbert [1 mM EDTA, 1% albúmina sérica bovina (BSA), 0,5 M fosfato sódico, 7% SDS] a 65 °C, rotando en una incubadora de hibridación durante al menos 2 h.
NOTA: La membrana puede prehibridarse durante varios días. - Prepare la sonda radiomarcada con α-32P dATP o dCTP y un kit de etiquetado de ADN de acuerdo con el protocolo del fabricante.
PRECAUCIÓN: Los dNTP radiomarcados son peligrosos y se debe usar el EPP adecuado al manipularlos. Todo el trabajo radiactivo debe realizarse detrás de un blindaje, y las personas capacitadas deben ser monitoreadas para determinar la absorción de radiación mediante dosímetros.- Diseñar un fragmento de ADN lineal de 400-900 pb complementario a la secuencia digerida de restricción (Figura 2) y amplificar el fragmento mediante reacción en cadena de la polimerasa (PCR).
NOTA: Recomendamos tener un fragmento de PCR en stock de 50-100 ng/μL - Combine 100 ng del fragmento de PCR complementario con ADN Pol I, tampón de fragmento de Klenow (3' 5' exo-) y oligonucleótidos de decanucleótidos aleatorios.
- Desnaturalizar el fragmento a 100 °C durante 10 min.
- Añadir a la muestra 5 unidades de ADN Pol I, fragmento de Klenow (3' 5' exo-), 50 μCi de dNTP α-32P y 30-50 μmol de mezcla de dNTP deficiente en el tipo dNTP radiomarcado.
- Incubar a 37 °C durante 10 min para permitir la polimerización y la incorporación de dNTP radiomarcado.
- Añada 30-50 μmol de tipo dNTP previamente ausente a la muestra e incube a 37 °C durante 10 min.
- Purifique el fragmento radiomarcado utilizando una columna de centrifugación a 3.000 × g durante 2 min.
- Diseñar un fragmento de ADN lineal de 400-900 pb complementario a la secuencia digerida de restricción (Figura 2) y amplificar el fragmento mediante reacción en cadena de la polimerasa (PCR).
- Añada la sonda radiomarcada a 50 mL de tampón de hibridación e incube con la membrana durante la noche a 65 °C rotando en una incubadora de hibridación.
- Al día siguiente, retire la sonda y lave la membrana 2 veces con tampón de lavado 1 (0,1x SSC, 0,1% SDS) a 42 °C y 2x con tampón de lavado 2 (2x SSC, 0,1% SDS) a 65 °C.
NOTA: Todos los lavados deben realizarse con rotaciones rápidas durante 15 min en la incubadora. - Seque la membrana durante 10 minutos y colóquela en un protector de lámina delgada y transparente. Guarde la membrana sellada en un casete controlado y resistente a la radiación con una pantalla sensible al fósforo. Deje que la membrana esté expuesta a la pantalla durante 1-10 días.
- Visualice los resultados utilizando un generador de imágenes biomoleculares configurado para imágenes de fósforo. Lavar y volver a exponer si es necesario.
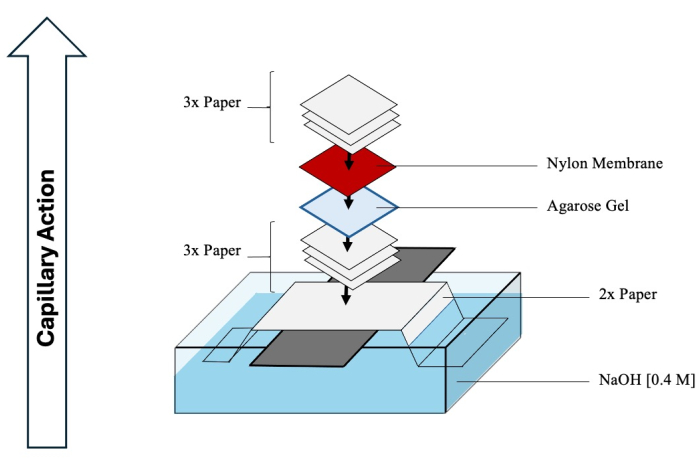
Figura 4: Ensamblaje de Southern blot. Esquema completo de un aparato típico utilizado para la transferencia de transferencia de intermedios de la segunda dimensión a una membrana de nailon. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Si tiene éxito, tras la visualización, se puede observar un arco agudo de horquillas de replicación que se extiende hacia arriba y hacia afuera desde el punto masivo 1n (Figura 5A). El tamaño de un fragmento, o el porcentaje que se replica, determina la movilidad del fragmento en la primera dimensión. A medida que los intermedios desarrollen una estructura más articulada, comenzarán a viajar más lentamente en la segunda dimensión. Por lo tanto, si u...
Discusión
2DGE proporciona una imagen semicuantitativa y completa de las poblaciones relativas de intermedios que surgen durante la replicación de una secuencia particular. Dado que las frágiles estructuras moleculares de las horquillas de replicación deben mantenerse durante todo este procedimiento, se debe tener mucho cuidado para evitar el cizallamiento físico y la desnaturalización química. Por lo tanto, se recomienda encarecidamente evitar cualquier tratamiento alcalino durante el aisla...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos a Jorge Cebrian y Anastasia Rastokina que comenzaron a desarrollar este enfoque en nuestro laboratorio, a Massimo Lopes por proporcionarnos el plásmido pML113 y consejos invaluables, a Ylli Doksani por las discusiones perspicaces y a los miembros del laboratorio Mirkin por su apoyo. El trabajo en el laboratorio de Mirkin cuenta con el apoyo del Instituto Nacional de Ciencias Médicas Generales [R35GM130322] y NSF-BSF [2153071].
Materiales
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
Referencias
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -. Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -. M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863 (2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23 (2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L., Adam, M. P. Emanuel Syndrome. GeneReviews®. , (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M., Kohwi, Y. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. , (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994 (2012).
- . BLAST: Basic Local Alignment Search Tool Available from: https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024)
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados