Marcaje final fluorescente y encapsulación de ARN largos para microscopía FRET-TIRF de molécula única
En este artículo
Resumen
Este artículo describe el marcaje de doble color de ARN largos en posiciones terminales y su inmovilización superficial mediante encapsulación en vesículas de fosfolípidos para aplicaciones de microscopía FRET TIRF de molécula única. La combinación de estas técnicas permite una visualización y un análisis precisos de la dinámica del ARN a nivel de una sola molécula.
Resumen
La transferencia de energía de resonancia de Förster de una sola molécula (smFRET) destaca en el estudio de biomoléculas dinámicas al permitir una observación precisa de sus cambios conformacionales a lo largo del tiempo. Para monitorear la dinámica del ARN con smFRET, desarrollamos un método para marcar covalentemente los ARN en su extremo con un par de fluoróforos FRET. Esta estrategia de marcaje final directo se dirige a la activación del 5'-fosfato por carbodiimida (EDC)/N-hidroxisuccinimida (NHS) y a la 3'-ribosa por oxidación periodada, que pueden adaptarse a otros ARN independientemente de su tamaño y secuencia para estudiarlos independientemente de las modificaciones artificiales. Además, la activación de 5'-EDC/NHS es de interés general para todos los ácidos nucleicos con un 5'-fosfato. El uso de productos químicos disponibles comercialmente elimina la necesidad de sintetizar sondas específicas de ARN.
La microscopía de fluorescencia de reflexión interna total (TIRF) requiere que las moléculas inmovilizadas en la superficie de interés estén dentro del campo evanescente que se va a iluminar. Una forma sofisticada de mantener las moléculas de ARN dentro del campo evanescente es encapsularlas en vesículas de fosfolípidos. La encapsulación se beneficia de lo mejor de ambos mundos, atando la molécula a la superficie al tiempo que permite la libre difusión de la molécula. Nos aseguramos de que cada vesícula contenga una sola molécula de ARN, lo que permite la obtención de imágenes de una sola molécula. Tras el marcaje y la encapsulación de doble extremo del ARN de interés, las mediciones de smFRET ofrecen una vista dinámica y detallada del comportamiento del ARN.
Introducción
La transferencia de energía por resonancia de Förster (FRET) es una técnica potente y sensible para estudiar las interacciones intermoleculares e intramoleculares de biomoléculas a nanoescala. Se basa en la transferencia de energía no radiativa de una molécula donante excitada a una molécula aceptora cercana, que se produce a distancias normalmente entre 1 nm y 10 nm. La distancia entre los colorantes donantes y aceptores determina la eficiencia de esta transferencia de energía, lo que convierte a FRET en una herramienta invaluable para estudiar la dinámica molecular, los cambios conformacionales y las interacciones en una amplia gama de sistemas biológicos 1,2, incluidos los ARN. La microscopía de fluorescencia de reflexión interna total (TIRF) ha demostrado ser una técnica poderosa para las investigaciones smFRET, ya que ilumina selectivamente las moléculas solo cerca de la superficie, lo que permite la dinámica FRET de moléculas individuales con una alta resolución espacial y temporal. Sin embargo, antes de realizar experimentos con smFRET-TIRF, la molécula de interés primero debe marcarse fluorescentemente con un par FRET apropiado y luego inmovilizarse en la superficie de la microscopía. El protocolo smFRET-TIRF descrito aquí se validó utilizando el intrón del grupo II de tipo salvaje Sc.ai5γ de Saccharomyces cerevisiae mitocondrias, flanqueado por sus dos secuencias exónicas (915 nucleótidos)3. Para obtener una visión más detallada de los intrones del grupo II de marcaje fluorescente y su inmovilización para la microscopía smFRET TIRF, consulte nuestra revisión4.
Una estrategia ideal de etiquetado de ARN específico del sitio permitiría la incorporación precisa de colorantes donantes y aceptores en posiciones predeterminadas sin alterar la estructura o función del ARN, lo que garantizaría mediciones FRET precisas y eficientes. Esto es un desafío debido a las similitudes químicas entre las cuatro nucleobases, lo que complica el etiquetado selectivo. El marcaje final une los tintes donantes y aceptores a los extremos del ARN dirigiéndose al fosfato 5' y a la ribosa 3'. Este enfoque ofrece un enfoque mínimamente invasivo y, al mismo tiempo, proporciona información valiosa sobre la dinámica estructural y las interacciones. La capacidad del intrón del grupo II para autoempalme en presencia de Mg2+ restringe el uso de enzimas dependientes de iones metálicos. Aquí, presentamos un enfoque para ARN duales largos y catalíticamente activos (ribozimas) que evitan la necesidad de enzimas o la síntesis de sondas especializadas.
Un enfoque común para atar moléculas de ARN a la superficie para la microscopía TIRF es unir covalentemente un fragmento de biotina directamente al ARN o hibridar un oligonucleótido antisentido (ASO) portador de biotina5,6,7. Sin embargo, este método de inmovilización directa puede introducir artefactos debido a las interacciones ARN-superficie, lo que puede dar lugar a ARN mal plegados8. Una solución elegante para mitigar estos artefactos de inmovilización es encapsular el ARN en vesículas de fosfolípidos a nanoescala adheridas a la superficie 9,10,11. Estas vesículas, que tienen aproximadamente 100 nm de diámetro, están ancladas a la superficie a través de un enlace biotina-estreptavidina 12,13,14, lo que permite que el ARN se difunda libremente en el interior y permite el intercambio de iones a través de la membrana lipídica 10. Después de marcar covalentemente un ARN funcionalgrande 3, presentamos un enfoque para encapsular dichos ARN en vesículas de fosfolípidos mediante la combinación de protocolos establecidos para la pasivación de superficie y la encapsulación de vesículas, adaptados para preservar la funcionalidad del ARN 10,11,14. Este enfoque de etiquetado y encapsulación de doble extremo logra una alta tasa de monoencapsulación de ARN funcionales para la microscopía smFRET TIRF.
Protocolo
1. Marcaje dual de ARN
NOTA: El siguiente protocolo describe el marcaje específico del sitio de ARN con un par FRET de fluoróforos mediante la unión covalente de un colorante donante (sCy3) al 5'-fosfato y un colorante aceptor (Cy5) a la 3'-ribosa. Un ARN largo catalíticamente activo, el intrón ribozima del grupo II, se elige como el ARN de interés. La Tabla 1 y la Figura 1 resumen este protocolo de etiquetado de doble extremo. Realice todos los pasos que involucren fluoróforos en condiciones de oscuridad.
| Día 0 | ▪ Alícuota 50-75 μg de ARN a un volumen total de 55 μL por tubo de 1,5 mL. | |||||
| Día 1 | Activación de 5′-fosfato | |||||
| ▪ Añadir 45 μL de solución EDC-NHS, pH 6.0 recién preparada al ARN en ddH2O hasta un volumen final de 100 μL, mezclar bien e incubar durante 4 h a 25 °C y 500 rpm. | ||||||
| ▪ Ronda de purificación 1: Precipitación nocturna de EtOH. | ||||||
| Día 2 | ▪ Precipitar el ARN activado 5', lavar y secar. | |||||
| Accesorio de tinte de 5′ | ||||||
| ▪ Resuspender en 95 μL de 100 mM MOPS, pH 7,5. | ||||||
| ▪ Añadir 5 μL de 2 mM de solución de colorante funcionalizado con amina. | ||||||
| ▪ Mezclar bien e incubar durante 16 h a 25 °C y 500 rpm. | ||||||
| Día 3 | ▪ Ronda de purificación 2: Precipitación de EtOH. | |||||
| Día 4 | ▪ Precipitar el ARN activado 5', lavar y secar. | |||||
| Paso de bloqueo | ||||||
| ▪ Resuspender en 100 μL de 100 mM de Tris-HCl, pH 7,5, e incubar durante 2 h a 25 °C y 500 rpm. | ||||||
| ▪ Ronda de purificación 3: Filtración centrífuga. | ||||||
| → eluir el ARN marcado con 5'. | ||||||
| Día 5 | Oxidación 3′-Periodate | |||||
| ▪ Incubar el ARN con 20 mM de NaIO4 en tampón de NaOAc de 50 mM, pH 5,5 en un volumen final de 100 μL durante 2 h a 25 °C y 500 rpm. | ||||||
| ▪ Apagar el exceso de periodato: Añadir 30 μL de glicerol al 50%, mezclar bien e incubar durante 30 min a 25 °C y 500 rpm. | ||||||
| ▪ Ronda de purificación 4: Precipitación nocturna de EtOH. | ||||||
| Día 6 | ▪ Precipitar el ARN 3′-oxidado, lavar y secar. | |||||
| Accesorio de tinte de 3′ | ||||||
| ▪ Resuspender en 95 μL de NaOAc de 50 mM, pH 6,0. | ||||||
| ▪ Añadir 5 μL de 2 mM de solución de colorante funcionalizado con hidrazida. Mezclar bien e incubar durante 16 h a 25 °C y 500 rpm. | ||||||
| Día 7 | ▪ Ronda de purificación 5: Precipitación de EtOH. | |||||
| Día 8 | ▪ Precipitar el ARN marcado, lavar y secar. | |||||
| ▪ Filtración centrífuga. | ||||||
| → Eluir el ARN marcado con doble extremo. | ||||||
Tabla 1: Resumen del protocolo para el marcaje de doble extremo de ARN. Haga clic aquí para descargar esta tabla.
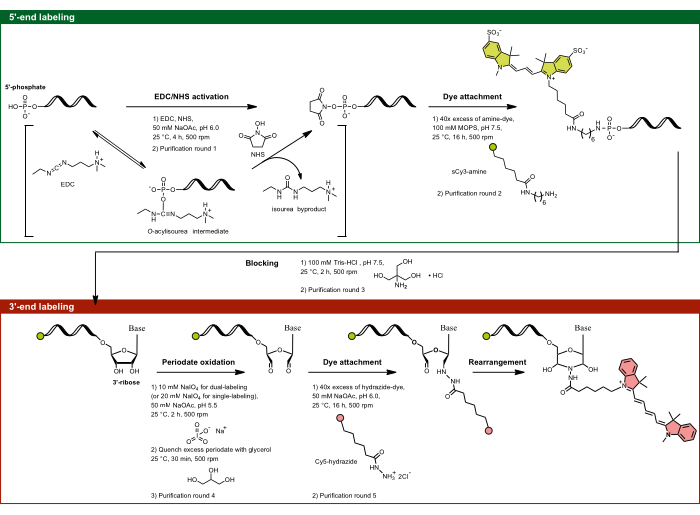
Figura 1: El flujo experimental de etiquetado de doble extremo dirigiéndose al 5'-fosfato y al azúcar 3'-ribosa. El fosfato 5' se activa mediante EDC en presencia de NHS y posteriormente se acopla con el colorante funcionalizado con aminas. La fracción de 3'-diol del ARN se oxida por la actividad periodada de dialdehído, que reacciona aún más con el colorante funcionalizado con hidrazida. Para el etiquetado de doble extremo, es importante comenzar con el etiquetado de 5' para evitar el etiquetado cruzado, seguido del etiquetado de 3' con un paso de bloqueo intermedio. Abreviaturas: EDC = carbodiimida; NHS = N-hidroxisuccinimida; MOPS = ácido 3-morfolinopropano-1-sulfónico; NaOAc = acetato de sodio. Haga clic aquí para ver una versión más grande de esta figura.
- Etiquetado de extremo 5': Dirigido al fosfato 5' mediante la activación de EDC/NHS
NOTA: Este método de marcado 5' se aplica no solo al ARN, sino a cualquier ácido nucleico monocatenario que contenga un fosfato 5'. La especificidad de la reacción se rige por la dependencia del pH, lo que permite que solo el fosfato 5' se dirija específicamente al sitio, a pesar de la presencia de múltiples fosfatos en la columna vertebral del ARN. A pH 6.0, la reactividad única del 5'-fosfato hacia las carbodiimidas se debe a su estado de protonación específico, donde dos oxígenos se desprotonan y uno permanece protonado. Esto hace que el fosfato 5' sea reactivo, mientras que los fosfatos de la columna vertebral totalmente desprotonados permanecen no reactivos, lo que permite el etiquetado selectivo del extremo 5' a través de la orientación EDC/NHS.- Prepare la solución EDC-NHS-NaOAc, pH 6.0. Mezclar 1,5 mg de EDC y 2,0 mg de NHS por alícuota en 35 μL deddH2O y 10 μL de NaOAc 0,5 M, pH 6,0 (pH ajustado con ácido acético glacial).
- Alícuota el ARN de interés para tener aproximadamente 50-75 μg de ARN por tubo en 55 μL de ddH2O. Agregue 45 μL de la mezcla EDC-NHS-NaOAc, pH 6.0 al ARN, alcanzando un volumen total de 100 μL con las concentraciones finales de 78 mM EDC, 174 mM NHS y 50 mM NaOAc, pH 6.0. Incubar durante 4 h a 25 °C agitando a 500 rpm.
NOTA: Aquí, 70 μg corresponden a 250 pmol de un ARN de 915 nt transcrito in vitro y 2,5 μM en el volumen de reacción final de 100 μL. - Ronda de purificación 1: Purificar el ARN activado por 5'-fosfato mediante precipitación de EtOH.
- Prepare una solución de 2 mM de cianinina3 sulfonada (sCy3-amina) en agua.
- Vuelva a suspender el pellet de ARN activado en 95 μL de tampón de 100 mM de ácido 3-morfolinopropano-1-sulfónico (MOPS), pH 7,5 (pH ajustado con NaOH). Añada 5 μL de solución de 2 mM de sCy3-amina al ARN activado. Acoplar el fluoróforo al fosfato 5' activado mediante incubación durante 16 h a 25 °C, mientras se agita a 500 rpm.
NOTA: Guarde el tampón MOPS a 4 °C en la oscuridad. El NaOH se elige para el ajuste del pH para mantener inactivo el intrón ribozima del grupo II. El fluoróforo debe ser al menos 40 veces superior al ARN. - Ronda de purificación 2: Aumentar el volumen añadiendo 200 μL deddH2O para mejorar la separación. Purifique el ARN marcado con 5' fosfato por precipitación de EtOH y repita hasta que el sobrenadante sea incoloro (generalmente se necesitan dos rondas).
- Paso de bloqueo: Resuspender el ARN en 100 μL de 100 mM de Tris-HCl, pH 7,5, e incubar durante 2 h a 25 °C y 500 rpm. Solo para el etiquetado único del extremo 5', omita el paso de bloqueo, vuelva a suspender el ARN en ddH2O y continúe con el paso 1.1.8. Sin embargo, para simplificar, si se trata de un etiquetado doble, separe el control de etiqueta única de 5' end solo después del paso 1.1.9.
NOTA: Este paso sirve para bloquear los 5'-fosfatos activados que no se han acoplado a un fluoróforo funcionalizado con amina al reaccionar con una fuente de amina primaria relativamente más pequeña (por ejemplo, Tris) para minimizar el riesgo de etiquetado cruzado con el fluoróforo funcionalizado con hidrazida que se utiliza para el protocolo de etiquetado del extremo 3'. - Ronda de purificación 3: Eliminar los colorantes libres lavando el ARN marcado por filtración centrífuga con un total de al menos 10 mL de ddH2O, luego eluir en ddH2O.
NOTA: El corte molecular del filtro debe ser inferior a la mitad del tamaño del ácido nucleico. Se puede usar el tampón de elección en lugar de ddH2O. La elución debe seguir las instrucciones del fabricante, asegurando que la muestra no se centrifuga hasta la sequedad completa. - Determinación de las concentraciones de ARN y colorante conjugado mediante espectroscopía UV-Vis.
- Etiquetado de extremo 3': Dirigido a la ribosa 3' mediante oxidación periodada
- Alícuota el ARN marcado con el extremo 5' en ddH2O para tener aproximadamente 50-75 μg de ARN por tubo en 90 μL. Si el volumen de elución de la etapa anterior fue alto, lo que resultó en una concentración baja, concentre el ARN precipitando y resuspendiendo el pellet en ddH2O. Agregue 5 μL de tampón NaOAc 1.0 M, pH 5.5 (correspondiente a 50 mM NaOAc, pH 5.5 para un volumen de reacción de 100 μL).
NOTA: Al igual que el ARN de marca simple de extremo 5', preparamos rutinariamente ARN de marca simple de extremo 3' como control. Para hacer esto, alícuota el ARN no marcado para tener aproximadamente 50-75 μg de ARN por tubo en 86 μL de ddH2O. - Añadir 4 μL de solución madre de metaperiodato sódico (NaIO4) de 500 mM recién preparada (correspondiente a 10 mM deNaIO4 para un volumen de reacción de 100 μL). Para el etiquetado único de 3', agregue 8 μL de solución madre de NaIO4 (correspondiente a 20 mM de NaIO4 para un volumen de reacción de 100 μL). Mezclar bien e incubar durante 2 h a 25 °C mientras se agita a 500 rpm en condiciones de oscuridad, ya que el NaIO4 es sensible a la luz.
NOTA: No cambie el orden de adición ni aumente aún más el tiempo de incubación, ya que esto puede causar fotoblanqueo del tinte donante adjunto. La concentración de NaIO4 utilizada se reduce a la mitad para el etiquetado de doble extremo para minimizar el enfriamiento del tinte ya adherido en el extremo 5'. - Detenga la reacción agregando 30 μL de glicerol al 50%. Incubar durante 30 min a 25 °C mientras se agita a 500 rpm.
NOTA: El glicerol sirve como diol para saciar el exceso de periodato. - Ronda de purificación 4: Agregue 400 μL de mezcla de precipitación de EtOH-NaOAc helada y realice la precipitación de etanol.
- Resuspender el pellet de ARN oxidado en 95 μL de 50 mM de NaOAc, pH 6,0.
- Accesorio de hidrazida-colorante: Prepare la solución de fluoróforo disolviendo unos pocos cristales de hidrazida de cianina 5 (Cy5-hidrazida) en DMSO y luego diluyendo con ddH2O a la concentración de 2 mM. Si el fluoróforo elegido es soluble en agua, prepare la solución en ddH2O. Agregue 5 μL de solución de 2 mM de Cy5-hidrazida al ARN oxidado. Mezclar bien e incubar durante 16 h a 25 °C agitando a 500 rpm.
NOTA: El fluoróforo debe ser al menos 40 veces superior al ARN. - Ronda de purificación 5: Purifique el ARN marcado con doble extremo (o ARN marcado con un solo extremo 3') mediante precipitación de EtOH y filtración centrífuga, similar a los pasos 1.2.6 y 1.2.8, respectivamente, y eluya en ddH2O.
- Cálculo de las concentraciones de ARN y colorante conjugado mediante espectroscopía UV-Vis.
- Caracterice el ARN marcado mediante ensayos analíticos basados en gel, espectroscopia de fluorescencia de conjunto (consulte la sección Resultados representativos ) y/o HPLC analítica como se muestra en otra parte3.
- Alícuota el ARN marcado con el extremo 5' en ddH2O para tener aproximadamente 50-75 μg de ARN por tubo en 90 μL. Si el volumen de elución de la etapa anterior fue alto, lo que resultó en una concentración baja, concentre el ARN precipitando y resuspendiendo el pellet en ddH2O. Agregue 5 μL de tampón NaOAc 1.0 M, pH 5.5 (correspondiente a 50 mM NaOAc, pH 5.5 para un volumen de reacción de 100 μL).
2. Preparación de la cámara microfluídica
NOTA: Recomendamos manejar seis u ocho cámaras a la vez. La sonicación se realiza a temperatura ambiente, a menos que se indique lo contrario. Este protocolo limita el uso de disolventes orgánicos, como la acetona, para evitar la solubilización de las trazas de impurezas. Para ver alternativas, véanse las referencias15,16.
- Limpieza
- Taladre cuatro agujeros en los portaobjetos de cuarzo con un perforador de diamante de acuerdo con el esquema dado en la Figura 2A para formar dos canales.
NOTA: Aunque pueden romperse más fácilmente durante la perforación, los portaobjetos de vidrio son una alternativa rentable a los portaobjetos de cuarzo para configuraciones de microscopía TIRF basadas en objetivos donde el cubreobjetos es la superficie de la imagen (Figura 2B). Esta no es una opción para configuraciones TIRF basadas en prismas, donde el portaobjetos de microscopía es la superficie de la imagen debido a la fluorescencia de fondo.
NOTA: Alternativamente, recicle las cámaras microfluídicas usadas16. Para ello, sumerja las cámaras usadas durante la noche en acetona (envuelva el tarro de tinción Coplin en papel de aluminio para minimizar la evaporación) en la campana extractora y luego sonique durante 5 minutos. Desmonte la cámara y deseche los cubreobjetos y las pegatinas. Si el desmontaje no funciona inmediatamente, sonique la cámara en acetona durante 10 minutos más. Continúe con el paso 2.1.2. - Coloque los portaobjetos de cuarzo perforados y aproximadamente el doble de cubreobjetos (ya que pueden romperse fácilmente) en un frasco de tinción Coplin de vidrio con tapa. Enjuague 3 veces con ddH2O. Sonicate en ddH2O durante 5 min, luego enjuague 3 veces con ddH2O.
- Sonicato en solución de limpieza de cristalería de laboratorio al 10% (ver Tabla de Materiales) durante 30 min a 50 °C. Enjuague al menos 3 veces con ddH2O hasta que desaparezcan las burbujas de detergente. Sonicato en ddH2O durante 5 min. Enjuague 3 veces con ddH2O.
- Sonicado en solución de KOH 1 M durante 30 min, luego dejar actuar toda la noche. Enjuague 3 veces con ddH2O. Sonicato en ddH2O durante 5 min. Enjuague 3 veces con ddH2O.
NOTA: A pesar de que la corrosión debida a una incubación excesiva es una preocupación previamente planteada por Chandradoss et al.15, recomendamos esta incubación larga que permite evitar el grabado de pirañas. - Seque los portaobjetos y cubreobjetos con N2(g).
- Trate los portaobjetos y cubreobjetos secos con un limpiador de plasma de oxígeno durante 30 minutos de acuerdo con las instrucciones del fabricante.
- Taladre cuatro agujeros en los portaobjetos de cuarzo con un perforador de diamante de acuerdo con el esquema dado en la Figura 2A para formar dos canales.
- Aminosilanización
- Prepare la solución de APTES-EtOH al 3% mezclando bien 288,5 mL de EtOH absoluto, 1,5 mL de ddH2O y 9 mL de (3-aminopropil)trietoxisilano (APTES) en un matraz Erlenmeyer de 500 mL.
- Coloque los portaobjetos y cubreobjetos limpios en un frasco de tinción Coplin, sumérjalos en la solución de APTES-EtOH al 3%, sonique durante 1 minuto e incube durante 30 minutos.
- Enjuague 3 veces con EtOH absoluto, luego 3 veces con ddH2O.
- Seque los portaobjetos y cubreobjetos bajo un flujo de N2 (g).
- Pasivación superficial y biotinilación
- Prepare una caja húmeda llenando una caja de punta de pipeta vacía hasta la mitad con ddH2O. Coloque los portaobjetos dentro de la caja, con el lado a tratar hacia arriba.
- Prepare la mezcla bPEG-mPEG en un tubo de microcentrífuga estéril de 1,5 mL mezclando suavemente 2 mg de biotina-polietilenglicol-valerato de succinimidilo 5000 (biotina-PEG-SVA) y 80 mg de valerato de metoxipolietilenglicol-succinimidilo (mPEG-SVA) en 640 μL de tampón de bicarbonato de sodio (NaHCO3) de 100 mM, pH 8,3. Centrifugar la mezcla bPEG-mPEG a 16.000 × g durante 1 minuto para eliminar las burbujas de aire.
- Retire con cuidado el sobrenadante y añada una gota de 30 μL en el centro del portaobjetos para cubrir ambos canales. Coloque una gota adicional en un lado del portaobjetos para tener un cubreobjetos pegilado adicional, ya que se rompen fácilmente, y es útil tener uno de repuesto. Finalmente, cubra la gota con un cubreobjetos limpio, cierre la caja húmeda y realice la pegilación durante la noche en condiciones de oscuridad.
- Enjuague a fondo los portaobjetos y cubreobjetos pasivados y biotinilados con ddH2O. Observe el cambio en la hidrofobicidad de las superficies tratadas. Secar con un caudal de N2(g).
- Montaje de la cámara microfluídica
- Corta una pegatina de doble cara para crear los canales. Pega la pegatina a la diapositiva, asegurándote de que cubra el área de interés. Coloque con cuidado el cubreobjetos en la parte superior, alineándolo de modo que las superficies pegiladas queden enfrentadas entre sí.
- Coloque cada cámara ensamblada en un tubo de centrífuga de 50 mL y llene los tubos con N2 (g), y almacene a -20 °C hasta por 1 mes.
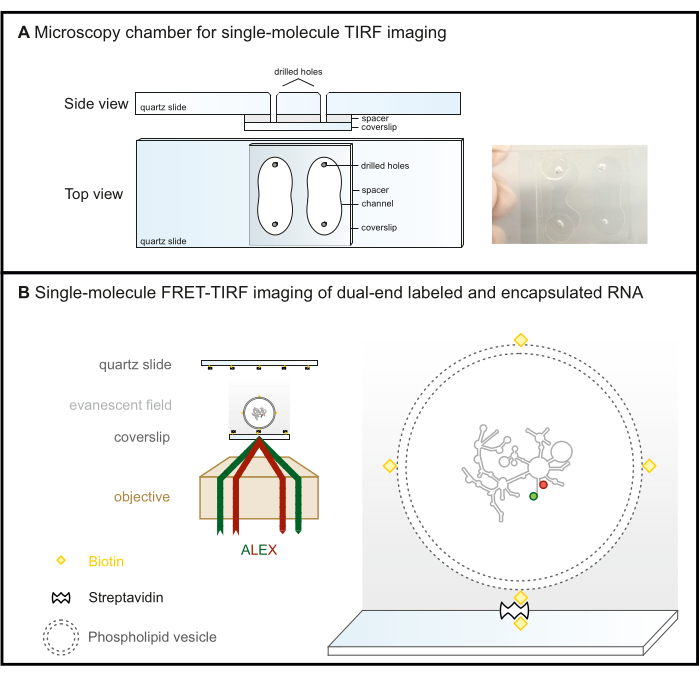
Figura 2: Microscopía FRET-TIRF de molécula única. (A) Cámara microfluídica para la obtención de imágenes TIRF. (B) El ARN marcado con FRET se encapsula en una vesícula de fosfolípidos biotinilados y se inmoviliza en una superficie de vidrio recubierta de steptavidina. Esto mantiene la molécula de interés dentro del campo evanescente (gradiente de grises), que es creado por la luz incidente que se refleja totalmente en el ángulo crítico en la microscopía TIRF. En este caso, ambos fluoróforos se excitan posteriormente con el esquema ALEX. Abreviaturas: FRET = Transferencia de energía de resonancia de Förster; TIRF = fluorescencia total de reflexión interna; ALEX = excitación láser alternativa. Haga clic aquí para ver una versión más grande de esta figura.
3. Encapsulación de vesículas de fosfolípidos
- Preparación de la torta de lípidos
NOTA: Siempre manipule el cloroformo debajo de una campana extractora.- Con una aguja estéril, haga agujeros en la tapa (de adentro hacia afuera) de los tubos de microcentrífuga de 2.0 mL estériles y limpios, como se muestra en la Figura 3A.
- Prepare la solución madre de biotinil (bPE) de 1,2-dipalmitoil-sn-glicero-3-fosfoetanolamina-N-cap disolviendo al menos 2 mg de bPE en la cantidad adecuada de cloroformo hasta una concentración final de 1 mg/ml.
- Preparar la solución madre de 1,2-dimiristoil-sn-glicero-3-fosfocolina (DMPC) disolviendo al menos 10 mg de DMPC en la cantidad adecuada de cloroformo hasta una concentración final de 10 mg/mL.
- Prepare la mezcla bPE-DMPC (manteniendo la relación 1:99 p/p) mezclando 100 μL de bPE y 990 μL de soluciones madre DMPC. Distribuya 109 μL de la mezcla bPE-DMPC por tubo de 2 mL.
- Coloque un inserto de tabique de celda de cartón de una caja de almacenamiento de microtubos en un matraz Schlenk, que sirva como soporte para tubos. Con unas pinzas largas, coloque los tubos que contienen la mezcla lipídica en el soporte del tubo en un matraz Schlenk de 500 ml (Figura 3B).
NOTA: Asegúrese de que estén en posición vertical y no inclinados. - Evapore el cloroformo a un flujo bajo de N2(g) durante la noche (o durante al menos 2 h hasta que el disolvente se evapore por completo).
- Selle los tubos con Parafilm para cubrir los agujeros. Almacene las tortas lipídicas a -20 °C durante un máximo de 1 mes.
- Encapsulación de ARN
- Prepare los siguientes tampones y soluciones ajustando las concentraciones de K+ y Mg2+ de acuerdo con el sistema biológico de interés.
- Prepare el tampón estándar 5x (5x SB): 2,5 M KCl, 400 mM MOPS; ajuste el pH a 6,9 con KOH, filtro estéril y almacene a 4 °C en la oscuridad (respectivamente 1x SB: 500 mM KCl, 80 mM MOPS, pH 6,9).
- Prepare el tampón antiparpadeo (AB): 100 mM MgCl2, Trolox, 1x SB, filtro estéril y almacene a 4 °C en la oscuridad durante un máximo de 1 semana.
NOTA: Utilice una punta de espátula de Trolox para obtener un volumen final de 10 mL, vórtice para mezclar y reajuste el pH.
- Ensamble el extrusor con una membrana de policarbonato (PC) de 100 nm y un disco de drenaje de poliéster (PE) de 10 mm, equilibre la jeringa y las membranas con AB y caliente a 30 °C (o esencialmente por encima de la temperatura de transición vítrea DMPC de 24 °C).
- Mezcle 5 μL de ARN marcado con doble extremo de 1 μM y 45 μL de AB (hasta un volumen total de 50 μL).
- Hidrata la torta lipídica con esta solución de ARN. A 30 °C, mezclar durante 5 minutos agitando a 1.400 rpm y luego durante 15 minutos a 700 rpm. Centrifugar durante 2 min a 13.000 g y transferir con cuidado el sobrenadante a un nuevo tubo.
- Diluir la muestra con 250 μL de AB.
- Llene la jeringa con la suspensión de ARN-lípidos. Extruya 35x a 30 °C en un bloque calefactor para encapsular el ARN marcado con doble extremo en vesículas de fosfolípidos de 100 nm de diámetro.
NOTA: La distribución del tamaño de las vesículas después de usar el extrusor se puede validar mediante dispersión dinámica de luz (DLS), como se muestra en la referencia11.
- Prepare los siguientes tampones y soluciones ajustando las concentraciones de K+ y Mg2+ de acuerdo con el sistema biológico de interés.

Figura 3: Preparación de la torta de lípidos. (A) Las tapas de los tubos se pinchan para permitir la evaporación del solvente. (B) Los tubos que contienen la mezcla de lípidos se colocan en un matraz Schlenk y el cloroformo se evapora para obtener una torta de lípidos. Haga clic aquí para ver una versión más grande de esta figura.
4. Microscopía smFRET-TIRF
- Inmovilización superficial
- Prepare los siguientes tampones y soluciones.
- Prepare el tampón antiparpadeo de azúcar (SAB): 1% de D-glucosa (p/v) en AB, filtro estéril y almacene a 4 °C en la oscuridad hasta por 1 semana.
- Prepare el 100x OSS: 100x Sistema de eliminación de oxígeno (OSS): 3 mg de glucosa oxidasa (correspondiente a 1,7 U), 10 μL de catalasa (correspondiente a 22 U) en 90 μL de 1x SB, almacenar a 4 °C en la oscuridad hasta por 1 semana.
- Prepare el tampón de imagen (IB) mezclando 198 μL de SAB y 2 μL de solución OSS 100x.
- Deje que la cámara microfluídica alcance la temperatura ambiente. Enjuague la cámara 2x con 200 μL de 1x SB.
- Llene la cámara con 50 μL de solución de estreptavidina de 20 μL/mL e incube durante 5 min.
- Enjuague la cámara con 200 μL de 1x SB y luego con 100 μL de AB.
- Inmovilizar el ARN encapsulado en la superficie añadiendo 75 μL de suspensión de vesícula e incubar durante 10 min.
- Enjuague la cámara con 200 μL de IB recién preparado e incube durante 5 min.
- La cámara ya está lista para la adquisición de datos (Figura 2B). Para evitar la evaporación durante mediciones largas, selle los orificios como se muestra en la Figura 2A. Si las fugas de un canal a otro son motivo de preocupación, selle el canal no utilizado antes de iniciar el protocolo de inmovilización de superficie.
NOTA: Recomendamos realizar mediciones en blanco como control de calidad para la limpieza de las cámaras microfluídicas y las vesículas de fosfolípidos contra las contaminaciones por fluorescencia.
- Prepare los siguientes tampones y soluciones.
Resultados Representativos
Presentamos el marcaje fluorescente simple y doble específico del ARN 915-nt de interés, el intrón mitocondrial Sc.ai5γ grupo II de levadura, flanqueado por secuencias exónicas. El par de fluoróforos FRET se posiciona en los extremos del ARN a través de la activación EDC/NHS del 5'-fosfato y la oxidación periodada de la 3'-ribosa, seguida de los respectivos aditivos al colorante. A continuación, verificamos los conjugados ARN-colorante mediante electroforesis en gel fluorescente, como se presenta en la Figura 4A. La comigración del ARN y los fluoróforos en el gel de agarosa confirma el éxito del etiquetado. A continuación, como se muestra en la Figura 4B, se utilizó espectroscopía de fluorescencia de conjunto para caracterizar el intrón del grupo II de doble marcado. La transferencia de energía, es decir, FRET, se observó tras la excitación del colorante donante, lo que demuestra el doble marcaje del ARN. En particular, en consonancia con la naturaleza dependiente de la distancia de FRET, el plegamiento del ARN del intrón del grupo II en presencia de iones metálicos condujo a un aumento en la eficiencia de FRET, como lo demuestra la disminución de la emisión del donante (flecha verde) y un aumento correspondiente en la emisión del aceptor (flecha roja). Esto indica que este etiquetado FRET rastrea los cambios conformacionales de la ribozima.
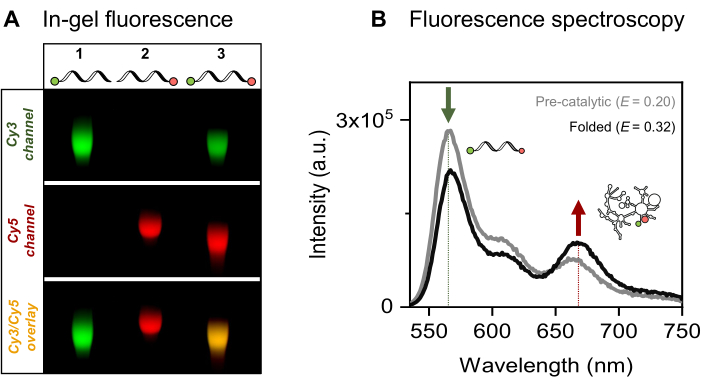
Figura 4: Caracterización de los conjugados ARN-colorante. (A) El análisis analítico basado en gel de ARN marcado con fluorescencia simple y doble muestra la comigración de los colorantes con el ARN en un gel de agarosa al 2%. La colocalización de los fluoróforos en la muestra de doble marcado se indica mediante la banda amarilla en la imagen fusionada (abajo) de los canales Cy3 (arriba, verde) y Cy5 (medio, rojo), visualizados bajo iluminación de 532 nm y 635 nm, respectivamente. Carril 1: 5'-sCy3 solo ARN marcado, carril 2: 3'-Cy5 solo ARN marcado y carril 3: ARN marcado de doble extremo (5'-sCy3 y 3'-Cy5). (B) La espectroscopia de fluorescencia de conjunto confirma el doble marcaje. La transferencia de energía tras la excitación del donante (λex = 515 nm, λem = 670 nm) verifica que ambos colorantes se acoplan con éxito al ARN. La curva gris representa el perfil de emisión del ARN precatalítico, mientras que la curva negra demuestra una mayor eficiencia de FRET en el ARN intrón del grupo II plegado (incubado con 500 mM de KCl a 70 °C durante 3 min, enfriado a 42 °C durante 5 min, seguido de la adición de 100 mM de MgCl2). Esta figura fue adaptada de Ahunbay et al.3 Haga clic aquí para ver una versión más grande de esta figura.
Con el intrón del grupo II de tipo salvaje de doble marca fluorescente en la mano, ahora estamos posicionados para explorar su dinámica a nivel de una sola molécula. Una vez encapsulado en vesículas de fosfolípidos, el ARN marcado se inmoviliza en una superficie de microscopía a densidades superficiales muy bajas para lograr una resolución de una sola molécula para smFRET-TIRF. Como se ve en la Figura 5A, se pueden rastrear varias moléculas individuales simultáneamente. La microscopía TIRF permite el monitoreo en tiempo real de la eficiencia de FRET y sus cambios a lo largo del tiempo. La Figura 5B ejemplifica las trazas FRET estáticas y dinámicas de una sola molécula del ARN marcado y encapsulado. Un rastro dinámico típico exhibe anticorrelación entre las señales del donante y del aceptor que fluctúan la eficiencia de FRET. Cuando la emisión del aceptor al excitar el donante aumenta, la emisión del donante disminuye correspondientemente, lo que indica un cambio dinámico en la distancia entre colorantes. Esta anticorrelación sugiere cambios conformacionales en la molécula de ARN.
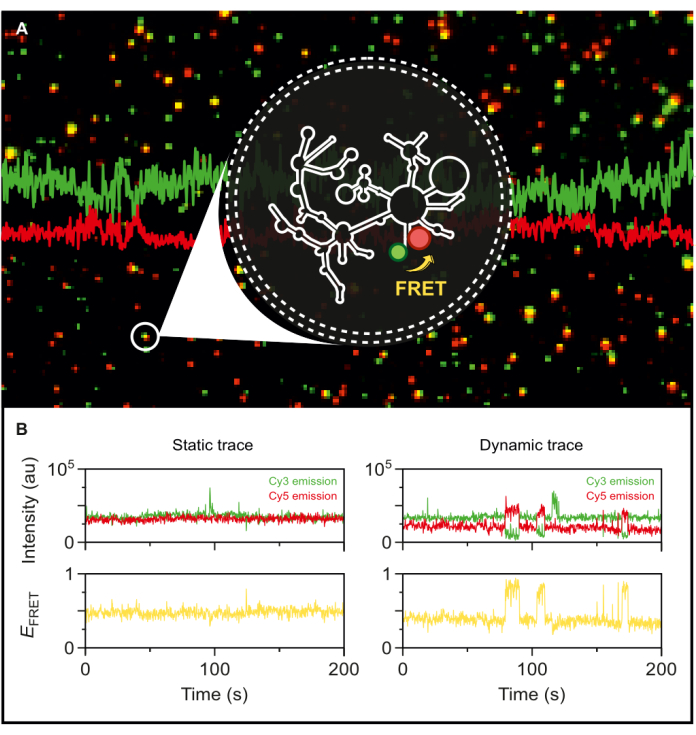
Figura 5: Comportamiento altamente dinámico del ARN del intrón del grupo II revelado por smFRET. El ARN del intrón del grupo II marcado con doble extremo se encapsula en una vesícula de fosfolípidos y se inmoviliza en la superficie para obtener imágenes con un microscopio TIRF basado en objetivos. (A) Imagen combinada de moléculas de ARN marcadas individuales que exhiben emisión donante (sCy3, verde) y emisión aceptora (Cy5, rojo) tras la excitación a 532 nm. (B) Las trayectorias típicas de smFRET de (izquierda) una molécula de ARN estática donde las intensidades del donante y del aceptor no fluctúan con el tiempo y (derecha) una molécula de ARN dinámica, donde las intensidades del donante y del aceptor se anticorrelacionan, con las eficiencias de FRET en amarillo. Las moléculas individuales se localizan y analizan utilizando MASH-FRET17. Se aplican correcciones de excitación directa, sangrado y factor de γ. Abreviaturas: smFRET = transferencia de energía de resonancia de Förster de molécula única. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El FRET a nivel de una sola molécula es único porque permite la observación y el análisis de moléculas individuales, revelando la heterogeneidad de la muestra y capturando estados transitorios que pueden oscurecerse en las mediciones de conjunto 1,2. La observación de moléculas de ARN individuales mediante smFRET proporciona información de alta resolución sobre sus vías de plegamiento y dinámica. Este protocolo describe el marcaje químico de doble extremo del ARN y su inmovilización superficial a través de la encapsulación de vesículas de fosfolípidos, que en conjunto permiten seguir cambios conformacionales dinámicos mediante microscopía smFRET-TIRF.
El estudio de la dinámica del ARN es un campo en constante crecimiento con la necesidad de nuevas estrategias de marcaje fluorescente específicas del sitio. Marcamos los extremos del ARN dirigiéndonos al 5'-fosfato con carbodiimidas y al azúcar 3'-ribosa con periodato. Estos enfoques se han descrito anteriormente (5' -extremo18,19 y 3' -extremo 19,20,21,22) pero no se han aplicado previamente a un ARN de tamaño similar al intrón Sc.ai5γ de tipo salvaje grupo II ribozyme, que requería optimización. La activación de la carbodiimida (por ejemplo, EDC) del 5'-fosfato es reversible. Por lo tanto, el imidazol se utilizó para reaccionar irreversiblemente con el intermedio O-acilisourea para formar un fosforimidazolido altamente reactivo19,20. Sin embargo, ahora se sabe que a pH más alto, las carbodiimidas pueden modificar las nucleobases, específicamente las guaninas y los uracilos, lo que ha llevado recientemente a su uso como agentes de sondeo estructural23,24.
Para evitar la reactividad cruzada, es crucial purificar el ARN activado del EDC antes de elevar el pH a 7,5 para la etapa de acoplamiento del colorante. Sin embargo, cuando introdujimos una etapa de purificación entre la activación y la fijación del colorante25, obtuvimos rendimientos muy bajos. De manera análoga, en el marcaje de proteínas, los residuos de lisina accesibles a la superficie pueden activarse con carbodiimidas. Sin embargo, en lugar de imidazol, que evita la reversión de la activación, se utiliza el NHS de forma rutinaria. También adoptamos esta estrategia, reemplazando así el intermediario de fosforimidazolido por un intermediario de fosfato NHS. De esta manera, logramos un control del pH, así como una mayor densidad de etiquetado a temperaturas más bajas y tiempos de incubación más cortos, es decir, 25 °C durante 4 h en comparación con 37 °C durante 16 h. Desarrollada para el ARN, esta estrategia de marcaje 5' se puede aplicar a cualquier otro ácido nucleico monocatenario con un fosfato 5'.
La activación del 5'-fosfato y la oxidación de la 3'-ribosa fueron mutuamente excluyentes, porque las químicas no son ortogonales. Para superar este desafío y evitar el etiquetado cruzado, comenzamos con el extremo 5', seguido de un paso de bloqueo para inhibir los sitios activados pero no etiquetados antes de continuar con el etiquetado del extremo 3'. Al oxidar el 3'-diol, el exceso de metaperiodato de sodio (NaIO4) podría apagar el fluoróforo ya adherido en el extremo 5'. Por lo tanto, redujimos la concentración de NaIO4 utilizada para el etiquetado único de 20 mM a 10 mM.
Se recomienda trabajar con varias alícuotas en paralelo en lugar de escalar verticalmente las reacciones. Este protocolo requiere múltiples pasos de precipitación de etanol (EtOH). Cuando se trabaja con varias alícuotas en paralelo, se prepare una mezcla de precipitación (30 mL de EtOH 100% absoluto y 1 mL de NaOAc 3 M, pH 5,2). El NaCl no se utiliza debido a su baja solubilidad en EtOH. Precipitar el ARN con 3,1 vol. de esta mezcla mediante incubación nocturna a -20 °C, seguida de centrifugación. Lave el gránulo de ARN dos veces con 500 μL de EtOH al 70% helado, centrifugue a 4 °C después de cada vez y seque al vacío. La precipitación de EtOH aprovecha la insolubilidad del ARN y la solubilidad de los colorantes libres en etanol al 70%. La filtración centrífuga elimina eficazmente los colorantes libres debido a su significativa diferencia de tamaño con respecto al ARN y facilita el intercambio de tampones, eliminando las sales. Además de los métodos de precipitación y filtración centrífuga de EtOH, los colorantes libres también se pueden eliminar mediante técnicas de extracción en gel y/o cromatografía (por ejemplo, HPLC); sin embargo, la escala debe adaptarse en consecuencia. No se deben utilizar los ARN largos en vórtice para resuspenderlos, ya que esto puede provocar un cizallamiento mecánico26. El momento óptimo para pausar el protocolo es cuando se granula el ARN. Utilizamos tubos de baja unión al ADN para mejorar la recuperación de ácidos nucleicos. Aunque el volumen de reacción final es de 100 μL, se prefieren los tubos de 1,5 mL (y no de menor volumen) para una mejor purificación por precipitación de EtOH.
Una vez eliminados los colorantes libres no reaccionados por precipitación y filtración centrífuga, confirmamos el marcaje mediante electroforesis en gel fluorescente (Figura 4A), espectroscopía UV-Vis, HPLCanalítica 3. Sin embargo, es importante tener en cuenta que estos métodos no pueden distinguir entre una molécula de ARN que transporta fluoróforos y una mezcla de ARN, cada uno marcado con un solo color. Del mismo modo, no se pueden utilizar para determinar si una molécula de ARN lleva varios fluoróforos del mismo color. La espectrometría de masas no se puede utilizar debido a las limitaciones de tamaño. Las espectroscopias Ensemble3 y FRET de molécula única corroboran el marcaje fluorescente dual, como se muestra en las Figuras 4B y 5B. La estequiometría de 0,5 (relación de sCy3 a Cy5 de 1:1) en los experimentos smFRET confirma la conjugación igual de los dos fluoróforos. Una de las preocupaciones fue el doble etiquetado en el extremo 3' al etiquetar ambos aldehídos en lugar de la ciclación propuesta. La ausencia de especies con una estequiometría de 0,25 (proporción de sCy3 a Cy5 de 1:2) en los experimentos de smFRET sugiere que la fijación del tinte dificulta y evita estéricamente la adhesión de un segundo tinte.
Con este marcaje fluorescente dual, los cambios en la señal FRET se pueden atribuir a reordenamientos estructurales a lo largo del plegamiento y la catálisis del ARN. Para mantener el ARN marcado con fluorescencia dentro del campo evanescente para la obtención de imágenes TIRF de una sola molécula, se prefiere la encapsulación sobre el anclaje directo de superficie. Este enfoque consiste en atrapar moléculas individuales de ARN dentro de las bicapas lipídicas de las vesículas, creando un entorno controlado propicio para observar su comportamiento dinámico. El protocolo descrito enriquece la monoencapsulación, como lo demuestra el fotoblanqueo en un solo paso11. Para comprender el plegamiento y la función del ARN, es esencial cerrar la brecha in vitro e in vivo 27. Los agentes de aglomeración molecular pueden imitar las condiciones dentro de las células para mejorar la catálisis del ARN por los intrones del grupoII 7,28. Alternativamente, la encapsulación crea microambientes restringidos que promueven el plegamiento del ARN29, acercando nuestra comprensión de la estructura y la dinámica del ARN a un contexto celular realista.
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Se agradece el apoyo financiero de la Fundación Nacional Suiza para la Ciencia [200020_192153 a RKOS], la UZH Forschungskredit [FK-20-081 a EA], la UZH Stiftung für wissenschaftliche Forschung [a RKOS y SZP], la Graduate Research Campus (GRC) Short Grant [2024__SG_092 a EA], la Escuela Superior de Ciencias Químicas y Moleculares de Zúrich (CMSZH) y la Universidad de Zúrich.
Materiales
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
Referencias
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22 (2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313 (2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152 (2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O., Farkas, D. L., Nicolau, D. V., Leif, R. C. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10 (2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados