Method Article
Nomograma de riesgo competitivo para predecir la supervivencia específica del cáncer en múltiples pacientes con cáncer colorrectal primario después de la cirugía
En este artículo
Resumen
El estudio encontró que el sexo masculino, el grado tumoral precario y el estadio avanzado de metástasis de ganglios tumorales se asociaron con una supervivencia específica del cáncer (CSS) más precaria en pacientes con cáncer colorrectal primario múltiple (MPCC) después de la cirugía. Desarrollamos un nomograma para predecir el CSS de los pacientes con MPCC y contribuir a la toma de decisiones clínicas sobre el tratamiento.
Resumen
La supervivencia específica del cáncer (CSS, por sus siglas en inglés) en pacientes con cáncer colorrectal primario múltiple (MPCC, por sus siglas en inglés) se ve afectada competitivamente por la muerte por otras causas. Este estudio tuvo como objetivo investigar el CSS y los factores de riesgo asociados mediante el análisis de riesgo competitivo en pacientes con MPCC. Los datos de este estudio provienen de la base de datos SEER. Utilizando el análisis univariable y multivariable del modelo de riesgo competitivo para debilitar el impacto de los eventos competitivos, explora los factores de riesgo de CSS y desarrolla un modelo de nomograma. Luego, el rendimiento del modelo se verifica mediante la curva ROC, la curva de calibración y el DCA. El estudio abarca un total de 8931 pacientes, de los cuales 6255 pertenecen a la cohorte de entrenamiento y 2676 a la cohorte de validación. En los análisis univariables y multivariados se observó que el sexo, el estadio de la metástasis de los ganglios tumorales (TNM) y el grado tumoral son factores de riesgo independientes para la supervivencia específica del cáncer en los pacientes con MPCC. Con base en los factores de riesgo, desarrollamos un modelo de diagrama para predecir CSS. La curva ROC, la curva de calibración y el DCA también muestran buenos resultados. En conclusión, se desarrolla un nomograma que sirve como una herramienta valiosa para predecir el CSS en pacientes con MPCC, proporcionando a los médicos información crucial para la planificación personalizada del tratamiento.
Introducción
El cáncer colorrectal, como uno de los tumores del sistema digestivo más comunes a nivel mundial, ha experimentado un aumento continuo en su incidencia en las últimas décadas. Las tasas de incidencia y mortalidad persistentemente altas han atraído una atención significativa. Según las últimas estadísticas, el cáncer colorrectal ocupa el tercer lugar entre los tumores malignos comunes en todo el mundo, y su tasa de mortalidad ocupa el segundo lugar a nivelmundial. El cáncer colorrectal primario múltiple (MPCC, por sus siglas en inglés) es un subtipo especial de cáncer colorrectal que ha atraído cada vez más la atención de los investigadores en los últimos años. Se define como el diagnóstico de dos o más lesiones independientes de cáncer colorrectal en un mismo paciente, con una distancia de más de 5 cm entre los bordes de las lesiones. En el MPCC, cuando se diagnostican múltiples tumores simultáneamente o en menos de 6 meses, se define como cáncer colorrectal sincrónico (SCRC), mientras que si el intervalo entre diagnósticos es mayor de 6 meses, se define como cáncer colorrectal metacrónico (MCRC)2,3,4,5.
La proporción de MPCC en todos los cánceres colorrectales es relativamente baja, y los informes sobre su incidencia varían según las diferentes regiones y estudios. Estudios recientes han reportado que el MPCC representa entre el 2% y el 10% de los cánceres colorrectales 2,3,4,5. En comparación con el cáncer colorrectal solitario, el MPCC tiene un peor pronóstico 3,6. En la actualidad, la evaluación clínica del pronóstico del MPCC se basa principalmente en el sistema de estadificación de la metástasis de los ganglios linfáticos tumorales (TNM) del American Joint Committee of Cancer, que utiliza el estadio más avanzado de las lesiones múltiples como el estadio real del MPCC. Sin embargo, basar las predicciones del pronóstico únicamente en la estadificación TNM es insuficiente. Todavía no existe una herramienta eficaz para predecir el pronóstico del CPMP después de la cirugía. En la era actual de la medicina de precisión, los modelos de predicción clínica para cuantificar el riesgo son ampliamente utilizados en la toma de decisiones clínicas y la evaluación del pronóstico de los pacientes 7,8. En los últimos años, los nomogramas basados en factores de riesgo pronóstico independientes han sido ampliamente aceptados para predecir el pronóstico tumoral9. Los nomogramas pueden visualizar modelos estadísticos complejos, lo que facilita su aplicación en entornos clínicos. En la evaluación del pronóstico tumoral, la supervivencia global (SG) y la supervivencia específica del cáncer (CSS) son indicadores de resultado comúnmente utilizados9. La SG se refiere al tiempo que transcurre desde la confirmación de un cáncer en un paciente hasta la muerte por cualquier causa. El CSS se refiere al tiempo transcurrido desde el diagnóstico de un tumor hasta la muerte causada específicamente por el cáncer, ofreciendo un reflejo más preciso del riesgo de muerte debido al cáncer. Cuando se utiliza el CSS como indicador de resultado, las muertes causadas por otros factores pueden afectar la probabilidad de mortalidad específica por cáncer, introduciendo una relación de riesgo competitiva entre los dos eventos10,11. Por lo tanto, los estudios de supervivencia específica del tumor deben usar modelos de riesgo competitivos para eliminar el impacto de los eventos competitivos. Estudios previos han construido modelos para predecir el pronóstico del MPCC, pero estos se han limitado al cáncer colorrectal sincrónico y no han utilizado modelos de riesgo competitivos para explicar el impacto de los eventos competitivos en el CSS 12,13,14.
En este estudio, exploramos los factores de riesgo independientes que compiten entre sí y que afectan la supervivencia específica del cáncer para el MPCC después de la cirugía. La justificación para emplear un modelo de riesgos competitivos se deriva de su capacidad para tener en cuenta la posibilidad de que los pacientes mueran por causas distintas al cáncer, lo cual es fundamental para obtener estimaciones de supervivencia no sesgadas15,16. Las técnicas tradicionales de análisis de supervivencia, como el modelo de Cox, pueden sobrestimar las probabilidades de supervivencia en presencia de eventos que compiten, lo que hace que el uso de modelos de riesgo competitivos sea más apropiado en estos escenarios17.
A partir de los factores de riesgo independientes identificados, construimos un nomograma para predecir la probabilidad de supervivencia y validamos su rendimiento. Los nomogramas han ganado popularidad en los entornos clínicos porque proporcionan una representación gráfica fácil de usar de modelos estadísticos complejos, lo que permite a los médicos calcular fácilmente las puntuaciones de riesgo individualizadas18,19. A diferencia de otras herramientas predictivas, los nomogramas incorporan múltiples factores de riesgo y proporcionan estimaciones de pronóstico más precisas y personalizadas. Este enfoque ha sido cada vez más validado en varios cánceres, demostrando un rendimiento superior a los sistemas de estadificación tradicionales 20,21,22. Nuestra herramienta tiene como objetivo ayudar a los médicos a realizar estimaciones de pronóstico personalizadas y más precisas durante el diagnóstico y el tratamiento, mejorando así la toma de decisiones en el manejo del MPCC.
Protocolo
Este estudio incluye dos etapas. En primer lugar, se obtuvieron datos clínicos y de supervivencia del MPCC de la base de datos SEER. A continuación, se utilizó el software R (versión 4.3.3) para analizar y construir un modelo de riesgo competitivo. El diagrama de flujo de trabajo del estudio se presenta en la Figura 1. Este estudio no requiere aprobación ética ni consentimiento para participar. Los datos utilizados en este estudio se obtuvieron de bases de datos.
1. Adquisición de datos
- Descargue el SEER. Software Stat 8.4.3 del sitio web de la base de datos SEER (http://seer.cancer.gov/about/overview.html). Regístrese e inicie sesión en SEER. Estadística 8.4.3 para obtener datos relevantes del paciente.
- Después de iniciar sesión en SEER. Estadística 8.4.3, haga clic en Datos de > de la sesión de listado de casos y seleccione la base de datos Incidence SEER Research Data, 17 Registries, Nov 2022 Sub (2000-2020).
- Haga clic en Selección > Editar y elija {Raza, Sexo, Año Dx. Año de diagnóstico} = '2004', '2005', '2006', '2007', '2008', '2009', '2010', '2011', '2012', '2013', '2014', '2015' Y {Sitio y Morfología. Recodificación del sitio ICD-O-3/OMS 2008} = 'Colon y recto' Y {Sitio y morfología. Confirmación diagnóstica} = 'histología positiva' Y {varios campos primarios. Número de secuencia}! = 'Solo una primaria'.
- Haga clic en Aceptar y guarde la selección. Haga clic en Tabla y, en la interfaz de variables disponibles, seleccione Recodificación por edad con edades únicas y 85+, Sexo, Recodificación del sitio ICD-O-3/OMS 2008, Tamaño del tumor CS, Recodificación de grado (hasta 2017), AJCC T derivado, 6ª ed (2004-2015), AJCC N derivado, 6ª ed (2004-2015), AJCC M derivado, 6ª ed (2004-2015), Recodificación de radiación, Recodificación de quimioterapia (sí, no/unk), Clasificación de muerte por causa específica de SEER, SEER, clasificación de otras causas de muerte, meses de supervivencia, identificación del paciente, RX Summ-Surg Prim Site (1998+), y haga clic en Columna.
- Haga clic en Salida, asigne un nombre a los datos y haga clic en Ejecutar para generar y guardar los datos.
- La base de datos de SEER no proporciona información directa sobre si un paciente tiene MPCC. Después de completar la descarga de datos como se describe anteriormente, use el ID del paciente para filtrar los pacientes con MPCC, es decir, aquellos diagnosticados con dos o más ocurrencias. Después de identificar a los pacientes con MPCC, calcule el intervalo entre los diagnósticos de tumores en función de sus tiempos de supervivencia. Usando 6 meses como límite, clasifique a los pacientes en SMPCC y MMPCC.
- En función de su edad en el momento del diagnóstico inicial, clasifique las edades de los pacientes como = 65 años y> 65 años. Para el grado tumoral, el grado I corresponde a una buena diferenciación, el grado II corresponde a una diferenciación moderada, el grado III corresponde a una diferenciación baja y el grado IV corresponde a la indiferenciación. Clasifique la ubicación del tumor en función de la distribución de múltiples tumores como colon derecho, colorrecto izquierdo y colorrecto completo.
- Existen diferencias evidentes en el desarrollo embrionario y las características biológicas entre el colorrecto proximal y distal, dividiendo la localización del tumor en función de la flexión esplénica23. El colon derecho se define como proximal a la flexión esplénica, incluyendo el ciego, el colon ascendente, la flexión hepática y el colon transverso, mientras que el colorrecto izquierdo incluye la flexión esplénica, el colon descendente, el colon sigmoide, la unión rectosigmoidea y el recto. Si los tumores de un paciente están completamente localizados en el colon derecho, defina la ubicación del tumor como colon derecho; Si todos los tumores se localizan en el colorrecto izquierdo, defina la ubicación como colorrecto izquierdo; Si hay tumores tanto en el colon derecho como en el colorrecto izquierdo, defina la ubicación como colorrecto completo.
- Determine el tamaño del tumor seleccionando el diámetro tumoral más grande entre varios tumores en el mismo paciente. Según el diámetro del tumor, clasifique el tamaño como = 5 cm o >5 cm. Un total de 8.931 pacientes fueron incluidos en el estudio. Para crear una cohorte de entrenamiento y una cohorte de validación, divida aleatoriamente todos los casos en una proporción de 7:3.
2. Construcción del modelo
- Descargue el software RStudio (2023.12.1+402) y R (4.3.3). Abra RStudio para ejecutar el software de R. Haga clic en Nuevo archivo y seleccione R Script para crear una nueva interfaz de programación de R. Introduzca el código correspondiente en el editor de código y haga clic en Ejecutar para ejecutar el código.
NOTA: El lenguaje R y sus funciones ofrecen una amplia variedad de parámetros. Agregar o modificar estos parámetros puede mejorar el análisis y la visualización de datos. - Utilice el siguiente código para realizar un análisis univariante y trazar la curva CIF.
Biblioteca(tidycmprsk)
biblioteca(gtresumen)
Biblioteca(ggsurvfit)
Biblioteca(ggprisma)
datos <-read.csv('data.csv')"
data$status<-factor(data$status, levels=c(0,1,2),labels=c("0","CSS","OCS"))
CIF <- tidycmprsk::cuminc(Surv(hora, estado) ~ Sexo, datos = datos)
ggcuminc(CIF,outcome= c("CSS", "OCS"),size=1.5)
donde data.csv son los datos obtenidos de la base de datos SEER. - Después de ejecutar el código anterior, haga clic en Exportar, luego haga clic en Guardar como imagen y, finalmente, haga clic en Guardar para guardar la imagen. El método para guardar las imágenes posteriores será el mismo que en este paso. Reemplace el sexo en el código anterior uno por uno con otros factores para realizar un análisis univariado para todos los factores.
- Utilice el siguiente código para realizar análisis y visualización BSR y multivariante.
Biblioteca(Saltos)
biblioteca(riskRegression)
Biblioteca(prodlim)
Biblioteca(parcela forestal)
leaps<-regsubsets(status==0~Sexo+Tamaño+Grado+T+N+M,datos = datos)
plot(saltos,escala="adjr2")
multi <- FGR(Hist(tiempo, estado)~Sexo + T + N + M + Grado + Tamaño, causa = 1, datos = datos)
Resumen (múltiple)
Multi <- read.csv("multi.csv",encabezado = T)
forestplot(labeltext=as.matrix(Multi [,1:4]), mean= Multi $HR_mean, ower= Multi $HR_1, upper= Multi $HR_2)
donde los datos de multi.csv provienen de los resultados del código anterior. - Siga el paso 2.3. para guardar la imagen.
- Utilice el siguiente código para trazar el nomograma, la curva ROC, la curva de calibración y la curva DCA.
biblioteca(QHScrnomo)
Biblioteca (RMS)
biblioteca(riskRegression)
Biblioteca(prodlim)
<- read.csv("data.csv")
D <- datadist(datos)
opciones(datadist = "d")
e <-cph(Surv(tiempo,estado==1)~Sexo + T + N + M +Grado,datos = datos,
x = T, y = T, surv = T, time.inc = 60)
nomo <- crr.fit(e,failcode=1,cencode = 0)
nomogram.crr(fit = nomo, lp = F, xfrac = 0.5, fun.at =seq(from=0, to=1, by= 0.1) , failtime =c(12,36,60),funlabel = c("Incidencia acumulativa de CSS de 1 año", "Incidencia acumulativa de CSS de 3 años","Incidencia acumulativa de CSS de 5 años"))
set.semilla(123)
data$pro <- tenf.crr(m3,tiempo = 60)
groupci(x=data$pro, ftime = data$time, fstatus = data$status, failcode = 1, cencode = 0, ci = TRUE)
f <- CSC(Hist(tiempo,estado)~Sexo + T + N + M +Grado,datos = datos)
x <- Puntuación(lista(modelo1=f), Hist(tiempo,estado)~1, datos=datos, causa=1, tiempos=c(12,36,60), se.fit=1L, plots="roc", metrics="auc")
g <- as.data.frame(x$AUC$puntuación)
h <- g[g$times %in% c(12,36,60),]
col = c("darkcyan","tomate","morado")
plotROC(x, xlab="1-Especificidad", ylab="Sensibilidad",col=col[1], cex=1.5, legend="", auc.in.legend = F, times = 12)
plotROC(x,col=col[2],legend = '', cex=1.5,times =36,auc.in.legend = F,add=T)
plotROC(x,col=col[3], times =60, add=T, cex=1.5, legend = '',auc.in.legend = F)
leg <- paste0(c("1-Año: ","3-Año: ","5-Año: "),substr(a$AUC,1,5)) - Siga el paso 2.3. para guardar la imagen.
- Utilice el paquete ggscidca para trazar la curva DCA.
Biblioteca(ggscidca)
df_surv<-read.csv("data.csv",encabezado = T)
cox_model <- coxph(Surv(tiempo,estado==1)~Sexo + T + N+ M +Grado, datos = df_surv)
cox_model1<-newcrr(cox_model)
scidca(cox_model1, newdata= df_surv,threshold.line = T,threshold.text = T)
donde data.csv son los datos obtenidos de la base de datos SEER. - Siga el paso 2.3. para guardar la imagen.
Resultados
Características del paciente
Un total de 8.931 pacientes fueron incluidos en el estudio. En cuanto a la distribución por sexo, hubo una mayor proporción de pacientes masculinos (56%) en comparación con pacientes femeninos (44%). En cuanto a la distribución de la localización tumoral, la mayoría de los tumores se distribuyeron en el colorrecto total, mientras que la menor cantidad de tumores se localizaron en el colon derecho (21%). En cuanto al grado tumoral, el grado más frecuente fue el grado II, con 6.251 pacientes, lo que representó el 70% del total; le siguió el grado III, con 2.026 pacientes (23%). Los grados I y IV tuvieron 355 y 299 pacientes, respectivamente, lo que representó el 4% y el 3,3% del total. En cuanto a la invasión tumoral, los estadios T3 y T4 fueron los más comunes. Más de la mitad de los pacientes no presentaron metástasis en los ganglios linfáticos, mientras que el 17% de los pacientes presentaron metástasis a distancia. 1.446 pacientes (16%) recibieron radioterapia y 3.843 pacientes (43%) recibieron quimioterapia. El CCE fue el más frecuente, con 5.327 pacientes, que representaron el 60% del total; Había 3.604 pacientes con cáncer colorrectal metacrónico, lo que representa el 40%. Los pacientes se dividieron aleatoriamente en grupos de entrenamiento y validación en una proporción de 7:3. No hubo diferencias estadísticamente significativas en los datos basales entre las cohortes de entrenamiento y validación, como se muestra en la Tabla 1.
Análisis univariable
Después de controlar la influencia de los eventos competitivos, los resultados del análisis univariado mostraron que el sexo, el grado y tamaño del tumor, el estadio TNM, la radiación, la quimioterapia, el estado sincrónico o metacrónico y la ubicación del tumor fueron los factores pronósticos que afectaron el CSS en los pacientes con MPCC. Solo la edad no es un factor pronóstico para el CSS en pacientes con MPCC. Observamos que hay una intersección significativa de la CIF en la radiación, la quimioterapia, la ubicación y el estado sincrónico o metacrónico, lo que indica que los efectos pronósticos a corto y largo plazo de la radiación en los pacientes con MPCC fueron diferentes, al igual que la quimioterapia, la ubicación y el estado sincrónico o metacrónico. La curva de riesgo acumulado de cada subgrupo se muestra en la Figura 2.
Análisis multivariable
Los factores pronósticos obtenidos por análisis univariado se incorporaron a la regresión de mejores subconjuntos (BSR) y análisis multivariado del modelo de riesgo competitivo. Entre ellos, se excluyen la radiación, la quimioterapia, la localización y el estado sincrónico o metacrónico debido a sus efectos duales en el pronóstico. Los resultados de la BSR y del análisis multivariado mostraron que el sexo, el estadio TNM y el grado tumoral fueron factores de riesgo independientes para CSS en pacientes con MPCC. (Figura 2)
Construcción y verificación de Nomogram
A partir de los factores de riesgo independientes obtenidos por análisis multivariante, construimos un nomograma de línea para predecir el CSS y verificar el rendimiento del modelo de predicción (Figura 3). A continuación, utilizamos la curva ROC, la curva de calibración y el DCA para evaluar el modelo. La curva ROC mostró que los AUC de 1 año, 3 años y 5 años de la cohorte de capacitación fueron 0,762, 0,742 y 0,734, y los AUC de 1 año, 3 años y 5 años de la cohorte de verificación fueron 0,801, 0,740 y 0,743. En la cohorte de entrenamiento y en la cohorte de verificación, la curva de calibración mostró una alta concordancia entre la probabilidad proyectada y los datos reales (Figura 4). Con el fin de verificar el rendimiento del modelo en la aplicación clínica, utilizamos DCA para evaluar el valor clínico del modelo. Los resultados muestran que el modelo muestra un buen beneficio neto (Figura 5).
Figura 1: Diagrama de flujo de trabajo del estudio. Este estudio consta de dos etapas: en primer lugar, los datos se obtuvieron mediante SEER. Stat, y luego el análisis y la visualización de datos se realizaron utilizando R. Haga clic aquí para ver una versión más grande de esta figura.
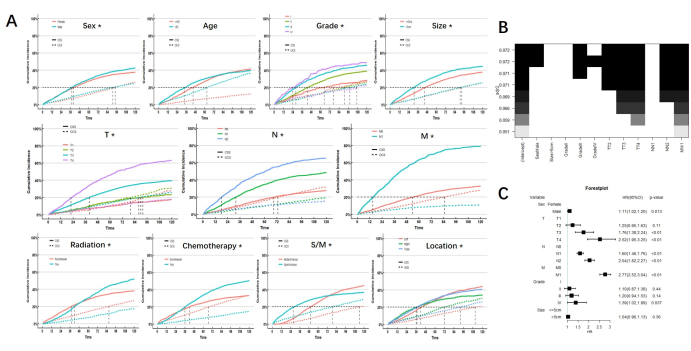
Figura 2: Resultados del análisis. (A) CIF de subgrupos. * indica P <0.05. (B) Regresión de los mejores subconjuntos. En la mejor bondad de ajuste, se consideraron para la inclusión el sexo, el grado y la etapa TNM (que se muestran como bloques negros en la parte superior). (C) El análisis multivariado también mostró que el sexo, el grado y el estadio TNM son factores de riesgo independientes. Haga clic aquí para ver una versión más grande de esta figura.
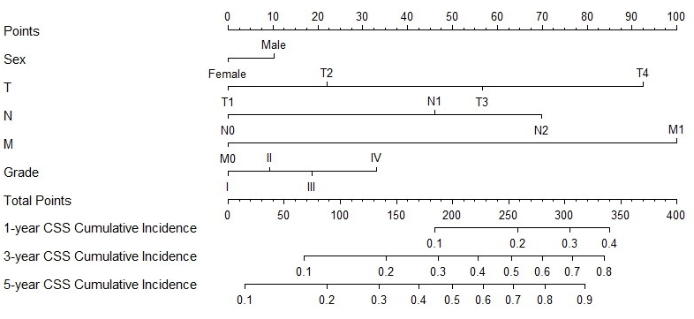
Figura 3: Nomograma de CSS en pacientes con MPCC. La puntuación total del paciente se puede calcular sumando las puntuaciones correspondientes a cada factor. Con base en la puntuación total, se puede predecir la probabilidad de muerte específica por cáncer a 1, 3 y 5 años . Haga clic aquí para ver una versión más grande de esta cifra.
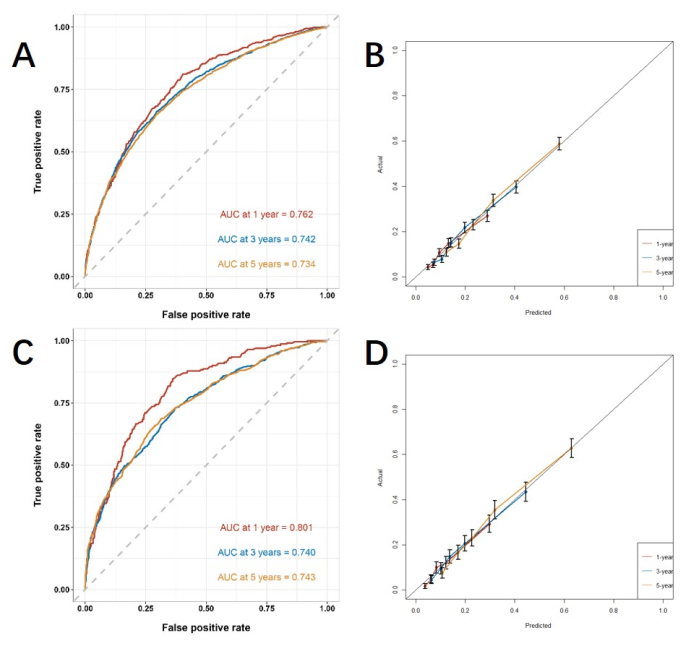
Figura 4: Curva ROC y curva de calibración. (A, B) Las curvas en la cohorte de entrenamiento (n = 6255) y la cohorte de validación (C, D) (n = 2676). Cuanto más cerca esté el valor de AUC de 1, mejor será el rendimiento de clasificación del modelo. Las barras de error muestran el intervalo de confianza del 95% para la probabilidad de que ocurra el evento real. Haga clic aquí para ver una versión más grande de esta figura.
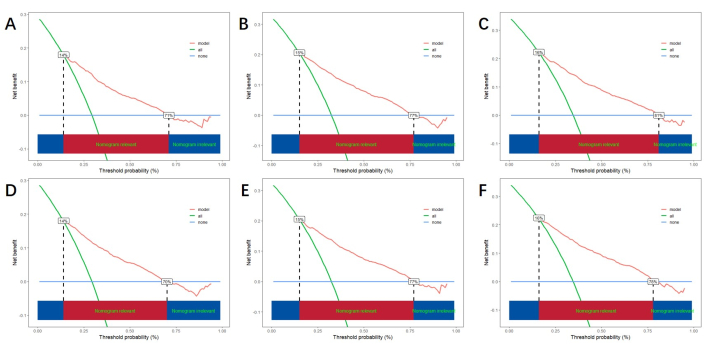
Figura 5: DCA para 1 año, 3 años y 5 años. (A, B, C) DCA en cohorte de capacitación y cohorte de validación (D, E, F). La línea verde representa el beneficio neto de todos los positivos, la línea azul representa el beneficio neto de ninguno positivo y la línea roja indica el beneficio neto del modelo. El área roja a continuación representa el beneficio del modelo que supera el de todos los positivos y ninguno positivo, lo que indica el rango de beneficio real del modelo. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Características clinicopatológicas y basales de los pacientes. Haga clic aquí para descargar esta tabla.
Discusión
Como tumor común del sistema digestivo, los factores que influyen en el pronóstico del cáncer colorrectal solitario han sido estudiados y confirmados en investigaciones previas24. Sin embargo, ha habido investigaciones limitadas sobre los factores pronósticos para los pacientes con MPCC. Este estudio incluyó a 8.931 pacientes con MPCC que se sometieron a cirugía de la base de datos SEER entre 2004 y 2015. Utilizamos un modelo de riesgo competitivo para investigar los factores de riesgo que afectan a CSS y construimos un modelo predictivo. En este estudio, el 39,1% de los pacientes fallecidos fallecieron por causas distintas al cáncer, lo que enfatiza la necesidad de utilizar un modelo de riesgo competitivo para analizar los factores de riesgo para el CSS.
Estudio previo ha encontrado que la edad avanzada es un factor de riesgo que afecta la SG de los pacientes con MPCC12. Sin embargo, en este estudio, encontramos que la edad no fue un factor de riesgo para CSS. Los pacientes de edad avanzada a menudo presentan condiciones de salud iniciales más precarias y más comorbilidades, lo que puede llevar a una SG más corta en comparación con los pacientes más jóvenes. Los ancianos también tienen más probabilidades de morir por otras causas, como eventos cardiovasculares e infecciones graves, lo que los hace más susceptibles a estos factores que los pacientes más jóvenes. En este estudio, nos centramos en la relación competitiva entre las muertes debidas a otras causas y las muertes debidas al propio cáncer. Mediante el uso de un modelo de riesgo competitivo y la exclusión de la interferencia de las muertes causadas por otras razones, encontramos que, en estas condiciones, la edad avanzada ya no era un factor de riesgo para el CSS de MPCC. Esto indica que los pacientes con MPCC de diferentes grupos de edad pueden enfrentarse a cargas tumorales similares. En general, los pacientes jóvenes y ancianos con esta enfermedad tienen un CSS similar, lo que proporciona una valiosa orientación para desarrollar estrategias de tratamiento personalizadas.
Este estudio también encontró que la incidencia y el pronóstico de MPCC exhiben tendencias relacionadas con el género, con una mayor proporción y peor CSS de pacientes masculinos. Esto es consistente con la situación observada en el cáncer colorrectal solitario. Estudios anteriores han demostrado que el cáncer colorrectal solitario es más común en los hombres, y los pacientes masculinos tienen un peor pronóstico que las mujeres. Esto puede deberse a la influencia de los estrógenos en la aparición y progresión del cáncer colorrectal25. Además, otras investigaciones han sugerido que la microbiota intestinal y los metabolitos intestinales en pacientes masculinos pueden ser una de las razones de las diferencias de género observadas en los pacientes con cáncer colorrectal26. Un estudio en Alemania encontró que la diabetes tipo 2 tiene un mayor impacto en el cáncer colorrectal en las mujeres27. Además, la vitamina D tiene un efecto protector contra la neoplasia colorrectal en las mujeres, pero no se encontraron hallazgos similares en los hombres28.
En un análisis univariado de la competencia, se encontró que los pacientes con MPCC con tumores de mayor tamaño tenían un CSS más precario. En estudios previos, el tamaño del tumor ha sido considerado a menudo como un indicador de la agresividad tumoral 29,30,31. Sin embargo, los resultados de los análisis multivariados que compitieron entre sí no respaldaron el tamaño del tumor como factor de riesgo independiente. Esto sugiere que en órganos huecos como el colorrectal, el tamaño del tumor puede tener una capacidad limitada para reflejar la agresividad del tumor. Este fenómeno puede deberse a varios factores, incluida la complejidad de la biología tumoral y los diferentes patrones de crecimiento y diseminación del tumor a través de diferentes órganos.
Un grado tumoral deficiente indica una mayor capacidad invasiva y migratoria de las células tumorales, lo que se alinea con las conclusiones de este estudio: un grado tumoral peor sugiere un CSS más pobre para los pacientes con MPCC. El estadio TNM es el método clínico más utilizado para guiar el tratamiento del paciente y predecir el pronóstico 32,33. En este estudio se encontró que cuanto más profunda era la infiltración tumoral, mayor era el número de ganglios linfáticos metastásicos y la presencia de metástasis en los órganos, peor era la supervivencia específica del paciente para el cáncer. Esto es totalmente coherente con el consenso clínico.
En el cáncer colorrectal, el pronóstico varía según la ubicación del tumor. Muchos estudios han encontrado que el colon derecho tiene un peor pronóstico en comparación con el colorrecto izquierdo 34,35,36,37. Sin embargo, algunas investigaciones sugieren que en el cáncer colorrectal resecable, el lado de la ubicación del tumor no afecta el pronóstico a largo plazo38. En este estudio, el MPCC localizado en el colon derecho tuvo un peor CSS a corto plazo, pero un mejor pronóstico a largo plazo. De manera similar, los pacientes con MPCC que recibieron radiación y quimioterapia tuvieron un mejor CSS a corto plazo, pero peor CSS a largo plazo. El SCRC tenía un CSS a corto plazo peor en comparación con el MCRC, pero un mejor CSS a largo plazo. Las razones de estos dos efectos sobre el pronóstico aún no están claras.
Este estudio tiene algunas limitaciones. Debido a las limitaciones inherentes a la base de datos SEER, no fue posible obtener o analizar algunos indicadores pronósticos conocidos, como los niveles de antígeno carcinoembrionario (CEA) y el estado de los microsatélites. Además, no fue posible excluir a los pacientes con enfermedad inflamatoria intestinal, cáncer colorrectal hereditario no poliposis y poliposis adenomatosa familiar que tienen mayor riesgo de MPCC39. Además, la inmunoterapia se ha convertido en un aspecto cada vez más importante del tratamiento del cáncer colorrectal, pero no pudimos obtener datos al respecto. La naturaleza retrospectiva de nuestro análisis y la dependencia de un único conjunto de datos pueden introducir sesgos inherentes.
El estudio basado en la base de datos SEER encontró que el sexo masculino, el grado tumoral deficiente y el estadio TNM avanzado se asociaron con un CSS más pobre en los pacientes con MPCC después de la cirugía. Es importante prestar mucha atención a los pacientes con estos factores de riesgo durante el diagnóstico y el tratamiento. Además, en este estudio se desarrolló un nomograma para predecir el CSS de los pacientes con MPCC, que puede predecir con precisión el pronóstico y contribuir a la toma de decisiones clínicas sobre el tratamiento.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Ninguno.
Materiales
| Name | Company | Catalog Number | Comments |
| forestplot package | Comprehensive R Archive Network (CRAN) | forest plot 3.1.3 | A forest plot that allows for multiple confidence intervals per row, custom fonts for each text element, custom confidence intervals, text mixed with expressions, and more. |
| ggprism package | Comprehensive R Archive Network (CRAN) | ggprism 1.0.5 | The ggprism package provides various themes, palettes, and other useful functions to customise ggplots and give them the ‘GraphPad Prism’ look. |
| ggscidca package | Comprehensive R Archive Network (CRAN) | ggscidca package | The 'ggscidca' package adds coloured bars of discriminant relevance to the traditional decision curve. Improved practicality and aesthetics. |
| ggsurvfit package | Comprehensive R Archive Network (CRAN) | ggsurvfit 1.1.0 | The ggsurvfit package eases the creation of time-to-event (aka survival) summary figures with ggplot2. The concise and modular code creates images that are ready for publication or sharing. |
| gtsummary package | Comprehensive R Archive Network (CRAN) | gtsummary 2.0.0 | The gtsummary package provides an elegant and flexible way to create publication-ready analytical and summary tables using the R programming language. The {gtsummary} package summarizes data sets, regression models, and more, using sensible defaults with highly customizable capabilities. |
| QHScrnomo package | Comprehensive R Archive Network (CRAN) | QHScrnomo 3.0.1 | The goal of QHScrnomo is to provide functionality to construct nomograms in the context of time-to-event (survival) analysis in the presence of competing risks. It also contains functions to build, validate, and summarize these models. |
| R Software | R Core Team | R 4.3.3 | Free software environment for statistical computing and graphics |
| riskRegression package | Comprehensive R Archive Network (CRAN) | riskRegression 1.3.7 | Risk Regression Models and Prediction Scores for Survival Analysis with Competing Risks |
| rms package | Comprehensive R Archive Network (CRAN) | rms 6.8-1 | rms does regression modeling, testing, estimation, validation, graphics, prediction, and typesetting by storing enhanced model design attributes in the fit. |
| RStudio | RStudio, Public Benefit Corporation(PBC) | Rstudio 2023.12.1+402 | Integrated Development Environment (IDE) used for running R scripts, data analysis, and model development. Provides a user-friendly interface for R programming with advanced features like script editing, debugging, and version control. |
| SEERstat | National Cancer Institute (NCI) | SEERstat 8.4.3 | Software for statistical analysis of SEER and other cancer-related databases |
| tidycmprsk package | Comprehensive R Archive Network (CRAN) | tidycmprsk 1.0.0 | The tidycmprsk package provides an intuitive interface for working with the competing risk endpoints |
Referencias
- Bray, F., et al. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 74 (3), 229-263 (2024).
- Derwinger, K., Gustavsson, B. A study of aspects on gender and prognosis in synchronous colorectal cancer. Clin Med Insights Oncol. 5, 259-264 (2011).
- Chin, C. C., Kuo, Y. H., Chiang, J. M. Synchronous colorectal carcinoma: predisposing factors and characteristics. Colorectal Dis. 21 (4), 432-440 (2019).
- Nikoloudis, N., et al. Synchronous colorectal cancer. Tech Coloproctol. 8 (Suppl 1), s177-s179 (2004).
- Huang, C. S., et al. Synchronous and metachronous colorectal cancers: Distinct disease entities or different disease courses. Hepatogastroenterology. 62 (140), 838-842 (2015).
- He, W., et al. Prognosis of synchronous colorectal carcinoma compared to solitary colorectal carcinoma: a matched pair analysis. Eur J Gastroenterol Hepatol. 31 (12), 1489-1495 (2019).
- Ranstam, J., Cook, J. A., Collins, G. S. Clinical prediction models. Br J Surg. 103 (13), 1886 (2016).
- Smith, T., et al. Comparison of prognostic models to predict the occurrence of colorectal cancer in asymptomatic individuals: a systematic literature review and external validation in the EPIC and UK Biobank prospective cohort studies. Gut. 68 (4), 672-683 (2019).
- Iasonos, A., Schrag, D., Raj, G. V., Panageas, K. S. How to build and interpret a nomogram for cancer prognosis. J Clin Oncol. 26 (8), 1364-1370 (2008).
- Conroy, T., et al. Five-year outcomes of FOLFIRINOX vs Gemcitabine as adjuvant therapy for pancreatic cancer: A randomized clinical trial. JAMA Oncol. 8 (11), 1571-1578 (2022).
- Zhong, M., et al. Impact of lung metastasis versus metastasis of bone, brain, or liver on overall survival and thyroid cancer-specific survival of thyroid cancer patients: A population-based study. Cancers. 14 (13), 3133 (2022).
- Xu, Y., Wang, X., Huang, Y., Ye, D., Chi, P. A LASSO-based survival prediction model for patients with synchronous colorectal carcinomas based on SEER. Transl Cancer Res. 11 (8), 2795-2809 (2022).
- Zhang, X., Zhao, L., Hu, Y., Deng, K., Ren, W. A novel risk prediction nomogram for early death in patients with resected synchronous multiple primary colorectal cancer based on the SEER database. Int J Colorectal Dis. 38 (1), 130 (2023).
- Zhang, X., et al. Developing prognostic nomograms to predict overall survival and cancer-specific survival in synchronous multiple primary colorectal cancer based on the SEER database. J Cancer Res Clin Oncol. 149 (15), 14057-14070 (2023).
- Austin, P. C., Lee, D. S., Fine, J. P. Introduction to the analysis of survival data in the presence of competing risks. Circulation. 133 (6), 601-609 (2016).
- Lau, B., Cole, S. R., Gange, S. J. Competing risk regression models for epidemiologic data. Am J Epidemiol. 170 (2), 244-256 (2009).
- Nolan, E. K., Chen, H. Y. A comparison of the Cox model to the Fine-Gray model for survival analyses of re-fracture rates. Arch Osteoporos. 15 (1), 86 (2020).
- Park, S. Y. Nomogram: An analogue tool to deliver digital knowledge. J Thorac Cardiovasc Surg. 155 (4), 1793 (2018).
- Balachandran, V. P., Gonen, M., Smith, J. J., DeMatteo, R. P. Nomograms in oncology: more than meets the eye. Lancet Oncol. 16 (4), e173-e180 (2015).
- Liang, W., et al. Development and validation of a nomogram for predicting survival in patients with resected non-small-cell lung cancer. J Clin Oncol. 33 (8), 861-869 (2015).
- Chen, D., et al. Predicting postoperative peritoneal metastasis in gastric cancer with serosal invasion using a collagen nomogram. Nat Commun. 12 (1), 179 (2021).
- Niu, X., et al. A prognostic nomogram for patients with newly diagnosed adult thalamic glioma in a surgical cohort. Neuro Oncol. 23 (2), 337-338 (2021).
- Luo, S., et al. Comparison of left- and right-sided colorectal cancer to explore prognostic signatures related to pyroptosis. Heliyon. 10 (7), e28091 (2024).
- Keum, N., Giovannucci, E. Global burden of colorectal cancer: emerging trends, risk factors and prevention strategies. Nat Rev Gastroenterol Hepatol. 16 (12), 713-732 (2019).
- Foster, P. A. Oestrogen and colorectal cancer: mechanisms and controversies. Int J Colorectal Dis. 28 (6), 737-749 (2013).
- Wang, L., et al. Male-biased gut microbiome and metabolites aggravate colorectal cancer development. Adv Sci. 10 (25), e2206238 (2023).
- Krämer, H. U., et al. Type 2 diabetes mellitus and gender-specific risk for colorectal neoplasia. Eur J Epidemiol. 27 (5), 341-347 (2012).
- Aigner, E., et al. Gender- and site-specific differences of colorectal neoplasia relate to vitamin D. Aliment Pharmacol Ther. 40 (11-12), 1341-1348 (2014).
- Dai, W., et al. Does tumor size have its prognostic role in colorectal cancer? Re-evaluating its value in colorectal adenocarcinoma with different macroscopic growth pattern. Int J Surg. 45, 105-112 (2017).
- Rössler, O., et al. Tumor size, tumor location, and antitumor inflammatory response are associated with lymph node size in colorectal cancer patients. Mod Pathol. 30 (6), 897-904 (2017).
- Zhang, Q., et al. Prognostic impact of tumor size on patients with metastatic colorectal cancer: a large SEER-based retrospective cohort study. Updates Surg. 75 (5), 1135-1147 (2023).
- Greene, F. L. TNM: our language of cancer. CA Cancer J Clin. 54 (3), 129-130 (2004).
- Ueno, H., et al. Optimal colorectal cancer staging criteria in TNM classification. J Clin Oncol. 30 (13), 1519-1526 (2012).
- Yahagi, M., Okabayashi, K., Hasegawa, H., Tsuruta, M., Kitagawa, Y. The worse prognosis of right-sided compared with left-sided colon cancers: a systematic review and meta-analysis. J Gastrointest Surg. 20 (3), 648-655 (2016).
- Holch, J. W., Ricard, I., Stintzing, S., Modest, D. P., Heinemann, V. The relevance of primary tumour location in patients with metastatic colorectal cancer: A meta-analysis of first-line clinical trials. Eur J Cancer. 70, 87-98 (2017).
- Petrelli, F., et al. Prognostic survival associated with left-sided vs right-sided colon cancer: A systematic review and meta-analysis. JAMA Oncol. 3 (2), 211-219 (2017).
- Zheng, C., Jiang, F., Lin, H., Li, S. Clinical characteristics and prognosis of different primary tumor location in colorectal cancer: a population-based cohort study. Clin Transl Oncol. 21 (11), 1524-1531 (2019).
- Karim, S., Brennan, K., Nanji, S., Berry, S. R., Booth, C. M. Association between prognosis and tumor laterality in early-stage colon cancer. JAMA Oncol. 3 (10), 1386-1392 (2017).
- Lindberg, L. J., et al. Risk of multiple colorectal cancer development depends on age and subgroup in individuals with hereditary predisposition. Fam Cancer. 18 (2), 183-191 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados