Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Double fluorescence In situ dans des coupes de cerveau frais
Dans cet article
Résumé
Ce protocole implique un non-radioactifs In-situ qui permet l'identification simultanée des deux espèces transcription, à une résolution seule cellule, dans les sections minces du cerveau des vertébrés.
Résumé
Nous décrivons ici une version modifiée d'une fluorescence à double
Protocole
Ce protocole a été développé et affiné basé sur le standard radioactifs et non radioactifs des méthodes d'hybridation in situ, développée précédemment par nous et les autres afin de détecter une ou deux espèces transcription dans 1-7 tissu cérébral. Le protocole décrit ci-dessous a une longueur totale de 2 ou 3 jours, selon le nombre d'interruptions de procédure choisie par l'utilisateur final. Toutes les étapes détaillées ci-dessous doivent être menées à température ambiante, à l'exception de l'étape d'hybridation ribosonde et post-hybridation lavages. Les solutions et les tampons requis pour toutes les étapes de cette méthode peut être trouvée à la fin de ce protocole.
1. Préparation des tissus et de sectionnement
- Décapiter et extrait de cerveau du sujet rapidement, et le placer dans un moule en plastique de taille suffisante.
- Couverture du cerveau avec Tissue-Tek milieu d'inclusion et de placer rapidement le moule en plastique dans un bain dry-ice/alcohol pour congélation rapide. Tissus congelés peuvent être conservés à -80 ° C jusqu'à utilisation.
- L'utilisation d'un cryostat, de recueillir 2 ou 3 coupes de cerveau par glisser sur Superfrost demandé, majoré des diapositives. Epaisseur des sections devrait être de 10 à 12 um. Les diapositives peuvent être conservés à -80 ° C jusqu'à utilisation.
2. Préparation des colonnes Sephadex G50 pour la purification de la sonde
«Colonnes Sephadex peuvent être achetés auprès de sources commerciales, cependant, nous fournissons ci-dessous une alternative à faible coût pour la génération de colonnes, ce qui sera nécessaire pour la purification de la sonde.
- Hydratez quantité adéquate de Sephadex G50 poudre avec RNase, traitée par DEPC eau (par exemple, 2 g de poudre dans 100 ml d'eau traitée au DEPC), brièvement solution de mélanger et conserver à température ambiante pour précipiter l'excès de Sephadex G50.
- Retirer le surnageant (couche supérieure de l'eau) à la suite de Sephadex G50 précipitations.
- Répétez le processus ci-dessus 3 - 5 fois.
- Après le lavage final, re-suspendre le Sephadex G50 solution dans un tampon TE (1:1), et conserver à 4 ° C jusqu'à utilisation.
- Placez autoclavé laine de verre dans une seringue de 1 ml stériles et le compresser avec le piston pour faire une couche compacte au fond, puis placez la seringue dans un tube Falcon de 15 ml.
- Mélangez bien la solution de Sephadex G50 dans TE. Remplir la seringue / colonne avec cette solution.
- Centrifuger la colonne pendant 30 sec à 1000 rpm.
- Répétez cette procédure jusqu'à ce que la colonne est presque complètement rempli de billes de Sephadex G50.
- Appliquer 200 pi de tampon de colonne de lavage à la colonne et on centrifuge pendant 2 min à 1000 rpm. Jeter l'éluat.
- Appliquer 200 pi de tampon de colonne de blocage à la colonne et la faire tourner pendant 2 min à 1000 rpm. Répétez cette étape fois 4-5 à équilibrer la colonne. Les colonnes peuvent être conservés à 4 ° C jusqu'à leur utilisation s'ils sont scellés avec du parafilm.
3. Étiquetage et purification de ribosondes
Ci-dessous nous détaillons la production et la purification d'une ribosonde unique. Pour dFISH, la préparation de chaque sonde impliquera la même méthodologie, sauf que l'une des sondes sera marquée à la digoxigénine (DIG)-UTP marqués tandis que l'autre avec de la biotine-UTP marqués.
- Préparer une solution concentrée (> 150 ng / pl) et solution purifiée d'ADNc linéarisées d'intérêt pour générer ou l'autre sens (de contrôle) ou ribosondes antisens.
- Dans un tube de 1,5 ml, ajouter 0,5 à 1 mg de protéine purifiée de matrice d'ADNc, 2 pl de tampon de la sonde étiquetage 5X, 1 pl de DIG 10X (ou biotine)-étiquetage mélanger, 0,5 pi de RNasine et 1 pl de la ARN polymerase appropriée , et porter le volume final de la solution à 10 ul avec RNAse free (traité au DEPC) d'eau.
- Incuber la solution dans un bain d'eau à 37 ° C pendant 2 heures.
- Ajouter 1 ul d'ARNt (stock; 20 pg / pl), et 39 pi de tampon de colonne de blocage à la solution.
- Préparer la colonne de Sephadex G50 pour la purification de la sonde. À cette fin, ajouter 50 ul de tampon de blocage à la colonne et la faire tourner pendant 10 sec à 1000 rpm.
Répétez cette étape 2-3 fois pour équilibrer la colonne (ie, le 50 ul appliquée à la colonne sont récupérés après cycle de centrifugation). - Appliquer la solution de sonde à l'Sephadex G50 colonne, la position d'un tube de centrifugeuse nouvelles au bas de la colonne, et la faire tourner pendant 3 min à 1000 rpm pour obtenir le purifiée 50 ul de solution ribosonde.
- Évaluer la qualité et le rendement de ribosonde marquée en utilisant soit une norme de formaldéhyde-agarose ARN gel, ou un spectrophotomètre.
4. Post-fixation, l'acétylation et hybridation
- Supprimez les parties du congélateur à -80 ° C et permettent une température ambiante de s'équilibrer. Par la suite, incuber sections dans un froid, fraîchement fait une solution de paraformaldéhyde 3% pendant 5 min.
- Rincer brièvement sections phosphate saline tamponnée (PBS) à deux reprises.
- Déshydrater sections à travers une série d'alcool type (70, 95 et 100%; 2 minutes chacun), et laissez-les sécher à l'air.
- Incuber sections dans une solution acétylation pendant 10 min.
- Rincer les sections 3 fois dans 2X SSPE.
- Déshydrater sections une fois de plus grâce à la série d'alcool standard décrit ci-dessus, et leur permettre de sécher à l'air.
- Préparer un volume adéquat de solution d'hybridation et d'ajouter deux ribosondes à la solution (concentration de la sonde: 1 ng / pl pour chaque sonde). Le volume total de la solution d'hybridation est déterminée en fonction du nombre de sections à hybrider (16 ul de solution d'hybridation par section).
- Appliquer un volume suffisant de la solution d'hybridation des lames de tissu et la lamelle veiller à ce qu'aucun des bulles d'air superposition des tissus.
- Placer les lames dans un support de diapositives de métal. Titulaire Plongez dans un bain d'huile minérale réglé à 65 ° C pendant la nuit. Assurez-vous que des lamelles sont face vers le haut (c'est à dire, la face latérale du support de diapositives doivent être en contact avec le fond du récipient d'huile minérale).
5. Post-hybridation Lave
- Le lendemain, retirez soigneusement le support de diapositives du bain d'huile minérale et brièvement le rincer dans du chloroforme pour éliminer l'excès d'huile à partir des diapositives.
- Placer les lames dans une solution de 2X SSPE pour les 5-10 min. Lamelles doit se détacher de diapositives tout en solution.
- Placer les lames dans une nouvelle solution 2X SSPE, et de les garder pendant 1 heure à température ambiante.
- Transfert des sections dans une solution contenant 2X SSPE, plus 50% de formamide. La température de cette solution doit correspondre à la température utilisée dans la procédure de l'hybridation pendant la nuit. Gardez diapositives dans cette solution pendant 1,5 heures.
- Transfert des sections d'une solution 0,1 X SSPE préchauffée à la même température de l'étape d'hybridation. Incuber sections dans cette solution pendant 30 min. Répétez cette étape pour 30 minutes supplémentaires.
6. Détection et visualisation de ribosondes
- Transfert diapositives tampon TNT à laquelle le peroxyde d'hydrogène de 0,3% a été ajoutée, pour 10 min.
- Lavez sections dans du tampon TNT, 3 fois (10 minutes chacun).
- Utiliser un stylo DAKO, dessiner un puits autour de la zone contenant les coupes de cerveau. Surtout, il faut être prudent afin d'assurer que les articles ne se dessèchent pas.
- Appliquer 150 pi de tampon TNB à chaque diapositive, et incuber sections dans cette solution pendant 30 min, dans une chambre humide.
- Retirer l'excès solution TNB par des diapositives basculement.
- Appliquer 150 ul de solution contenant TNB-peroxydase anticorps anti-DIG, et glisse stocker dans une chambre humide pendant 2 heures. Concentration d'anticorps doit être déterminée individuellement pour chaque transcription d'intérêt avant d'effectuer dFISH.
- Lavez sections dans du tampon TNT; 3 fois, 10 minutes chacun.
- Appliquer 150 ul d'Alexa 594-conjugué solution tyramide travail pour chaque diapositive, et les stocker dans une chambre humide pendant 1 heure. Cette solution doit être préparée selon les instructions du fabricant.
- Lavez sections dans du tampon TNT; 3 fois, 10 minutes chacun.
- Sections Incuber dans tampon TNT à laquelle le peroxyde d'hydrogène de 0,3% a été ajoutée pour 10 - 30 min.
- Lavez sections dans du tampon TNT; 3 fois, 10 minutes chacun.
- Appliquer 150 pi de tampon TNB par diapositive, et les garder dans une chambre humide pendant 30 min.
- Retirer l'excès solution TNB par des diapositives basculement.
- Ajouter 150 ul de la solution contenant TNB conjugué à la peroxydase d'anticorps anti-biotine, et glisse stocker dans une chambre humide pendant 2 heures.
- Lavez sections dans du tampon TNT; 3 fois, 10 minutes chacun.
- Appliquer 150 ul d'un Alexa 488-conjugué solution tyramide travail pour les diapositives et les sections incuber pendant 1 heure. Cette solution doit être préparée conformément aux recommandations du fabricant.
- Lavez sections dans du tampon TNT; 3 fois, 10 minutes chacun.
- Ajouter 150 pi de solution de Hoechst (1:1000 dans du tampon TNT) pour les sections et les garder dans la chambre humide pendant 2 min.
- Laver les coupes dans du tampon TNT; 3 fois, 5 minutes chacun.
- Sections lamelle avec un milieu de montage compatibles fluorescence (par exemple, ou de prolonger Vectashield Antifade).
7. Les résultats représentatifs
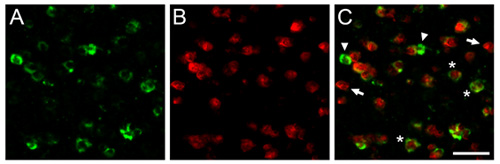
Figure 1. Nous montrons ici les résultats obtenus dFISH représentatives dans le cerveau diamant mandarin. Illustré sont des microphotographies obtenues à partir des caudomedial nidopallium (MR), l'analogue d'oiseaux chanteurs du cortex auditif des mammifères. Coupes de cerveau ont été hybridés avec une ribosonde biotinylé dirigé contre la parvalbumine (A), un marqueur d'une sous-population de neurones inhibiteurs, et une DIG marqué ribola sonde dirigée contre les Zenk dépendant de l'activité du gène (B), un marqueur fiable de la chanson axé sur les neurones. C) Superposition de (A) et (B) montre une population de neurones inhibiteurs qui sont activés par l'expérience auditive. Flèches et pointes de flèches indiquent les cellules marquées exclusivement avec chacun des deux ribosondes, et les astérisques indiquent les neurones représentant de co-exprimant les deux transcriptions d'intérêt. Barre d'échelle = 25 um.
Discussion
Nous avons utilisé ce protocole pour étudier comment le cerveau des vertébrés est neurochemically et fonctionnellement organisés, et de déterminer comment behaviorally pertinentes impact sensoriel des stimuli de la génomique des machines de neurones dans le cerveau adulte de 80 à 10. Nous avons utilisé avec succès cette méthode dans le tissu cérébral de souris, les rats et les oiseaux chanteurs, mais attendons à ce que ce protocole sera facilement adaptable à des sections de cerveau obtenue à ...
Déclarations de divulgation
Remerciements
Les travaux pris en charge par des subventions du NIH / NIDCD et la Fondation Schmitt à RP.
matériels
Solutions
- Tampon de colonne de lavage: 10 mM Tris-HCl, 0,15 M de NaCl, 0,05 mM EDTA, 50 ug / ul ARNt, SDS 0,1% dans 50 ml d'eau traitée par DEPC.
- Tampon de colonne de blocage: 10 mM Tris-HCl, 50 mM de NaCl, 0,1 mM d'EDTA dans 50 ml d'eau traitée par DEPC.
- Tampon TNT: 60 ml de 1 M Tris-HCl, 18 ml de NaCl 5 M et 1,8 ml de Triton X-100 dans 600 ml d'eau traitée au DEPC.
- TNB mémoire tampon: 100 mM Tris-HCl, 8,3 g / ul BSA, 0,15 M de NaCl et 3% de Triton X-100 dans l'eau traitée au DEPC.
- Tampon TE: 10 mM Tris-HCI, pH 7,5 et 1 mM EDTA, pH 7,5.
- L'acétylation solution: 2,7 ml de triéthanolamine majoré de 0,5 ml d'anhydride acétique dans 200 ml d'eau traitée au DEPC.
- La solution d'hybridation: 50% de formamide, 2X SSPE, 2 ug / ul ARNt, 1 ug / ul BSA et 1 ug / ul poly A dans l'eau traitée par DEPC. Il a besoin de 16 l par article.
Références
- Jin, L., Lloyd, R. V. In situ hybridization: methods and applications. J Clin Lab Anal. 11, 2-9 (1997).
- Stoler, M. H. In situ hybridization. Clin Lab Med. 10, 215-236 (1990).
- Komminoth, P., Werner, M. Target and signal amplification: approaches to increase the sensitivity of in situ hybridization. Histochem Cell Biol. 108, 325-3233 (1997).
- Kessler, C. The digoxigenin:anti-digoxigenin (DIG) technology--a survey on the concept and realization of a novel bioanalytical indicator system. Mol Cell Probes. 5, 161-205 (1991).
- Panoskaltsis-Mortari, A., Bucy, R. P. In situ hybridization with digoxigenin-labeled RNA probes: facts and artifacts. Biotechniques. 18, 300-307 (1995).
- Qian, X., Lloyd, R. V. Recent developments in signal amplification methods for in situ hybridization. Diagn Mol Pathol. 12, 1-13 (2003).
- Mello, C. V., Jarvis, E. D., Denisenko, N., Rivas, M. Isolation of song-regulated genes in the brain of songbirds. Methods Mol Biol. 85, 205-217 (1997).
- Pinaud, R. GABAergic neurons participate in the brain's response to birdsong auditory stimulation. Eur J Neurosci. 20, 1318-1330 (2004).
- Velho, T. A., Pinaud, R., Rodrigues, P. V., Mello, C. V. Co-induction of activity-dependent genes in songbirds. Eur J Neurosci. 22, 1667-1678 (2005).
- Tremere, L. A., Jeong, J. K., Pinaud, R. Estradiol shapes auditory processing in the adult brain by regulating inhibitory transmission and plasticity-associated gene expression. J Neurosci. 29, 5949-5963 (2009).
- Pinaud, R., Mello, C. V., Velho, T. A., Wynne, R. D., Tremere, L. A. Detection of two mRNA species at single-cell resolution by double-fluorescence in situ hybridization. Nat Protoc. 3, 1370-1379 (2008).
- Pinaud, R., Jeong, J. K. Duplex fluorescence in situ hybridization in the study of gene co-regulation in the vertebrate brain. Methods Mol Biol. 611, 115-129 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon