Un protocole simple, rapide et partiellement automatisé pour l’isolement de noyaux uniques à partir de tissus de mammifères congelés pour le séquençage d’un seul noyau
Dans cet article
Résumé
L’étude décrit un protocole simple, rapide et partiellement automatisé pour isoler des noyaux de haute qualité à partir de tissus de mammifères congelés pour le séquençage de l’ARN de noyaux uniques en aval.
Résumé
Le séquençage de l’ARN unicellulaire et mononoyau est devenu une application courante en laboratoire en raison de la richesse de l’information transcriptomique qu’il fournit. Le séquençage de l’ARN à noyau unique, en particulier, est utile pour étudier l’expression des gènes dans les tissus difficiles à dissocier. De plus, cette approche est également compatible avec les documents congelés (archives). Nous décrivons ici un protocole permettant d’isoler des noyaux uniques de haute qualité à partir de tissus de mammifères congelés pour le séquençage de l’ARN d’un seul noyau en aval de manière partiellement automatisée à l’aide d’instruments et de réactifs disponibles dans le commerce. Plus précisément, un dissociateur robotisé est utilisé pour automatiser et standardiser l’homogénéisation des tissus, suivie d’un gradient chimique optimisé pour filtrer les noyaux. Enfin, nous comptons les noyaux avec précision et automatiquement à l’aide d’un compteur automatisé de cellules fluorescentes. La performance de ce protocole est démontrée sur le cerveau de souris, les reins de rat et les tissus du foie et de la rate cynomolgus. Ce protocole est simple, rapide et facilement adaptable à divers tissus de mammifères sans nécessiter d’optimisation poussée et fournit des noyaux de bonne qualité pour le séquençage de l’ARN à noyau unique en aval.
Introduction
Le séquençage de l’ARN unicellulaire (sc) et mononoyau (sn) est devenu un protocole couramment utilisé en biologie moléculaire et cellulaire en raison de la résolution accrue de l’expression génique par rapport au séquençage de l’ARN en vrac. Cependant, l’isolement de préparations de cellules uniques et de noyaux uniques de bonne qualité à partir de tissus solides reste un défi et constitue souvent l’étape limitant la vitesse dans les expériences sc/sn-RNAseq. En effet, une pléthore de protocoles ont été développés qui utilisent diverses procédures chimiques et mécaniques pour obtenir des suspensions cellules/noyaux 1,2,3,4,5,6,7,8,9,10,11,12,13,14,15 . De plus, les stratégies de nettoyage de ces préparations des débris, des amas, etc., vont du tri par flux à la filtration en passant par le lavage. De tels protocoles sont souvent manuels (ce qui entraîne une variabilité liée à l’utilisateur), peuvent prendre beaucoup de temps (ce qui réduit la viabilité des cellules/noyaux) et/ou peuvent nécessiter l’accès à un cytomètre en flux pour le tri cellule/noyau. Cette étude s’est concentrée sur le développement d’un protocole simple, rapide et partiellement automatisé d’isolement de noyaux uniques à partir de tissus de mammifères congelés pour des applications de séquençage de l’ARN en aval. Nous nous sommes concentrés spécifiquement sur l’isolement des noyaux par opposition à l’isolement cellulaire, car il est compatible avec l’utilisation de tissus congelés, ce qui rend la collecte et le traitement des échantillons plus pratiques et permet un dosage non biaisé des échantillons, en particulier dans les expériences temporelles. De plus, bien que le transcriptome nucléaire ne reflète pas entièrement le transcriptome cellulaire, plusieurs études ont maintenant montré que les données de séquençage de l’ARN à noyau unique sont comparables aux données de séquençage de l’ARN d’une seule cellule pour l’identification du type cellulaire, même si les proportions de types cellulaires peuvent varier 6,16,17,18,19.
L’isolement des noyaux se compose de plusieurs étapes : 1) la perturbation mécanique ou chimique des tissus pour libérer les noyaux, 2) le nettoyage des débris et des amas, et 3) le comptage précis des noyaux pour la préparation des applications en aval. Dans un certain nombre de protocoles, l’étape 1 implique fréquemment l’utilisation d’un homogénéisateur Dounce afin de perturber le tissu 3,20. Alternativement, des méthodes chimiques peuvent être utilisées, bien que celles-ci doivent souvent être optimisées pour différents tissus 2,5,6. Nous avons constaté qu’une procédure manuelle de perturbation tissulaire est sujette à une variabilité associée à l’opérateur, ce qui entraîne une qualité et un rendement variables des noyaux. Afin de minimiser la variabilité technique et de disposer d’un protocole plus cohérent et reproductible qui fonctionne sur tous les tissus, un protocole qui utilise un dissociateur tissulaire robotisé disponible dans le commerce a été développé21. Pour l’étape 2, bien que l’échange de tampon soit généralement le moyen le plus simple de laver les noyaux, nous avons adopté l’utilisation d’une étape de centrifugation à gradient de saccharose relativement courte pour avoir une élimination plus complète des débris. Pour le tissu cérébral en particulier, nous utilisons un gradient colloïdal de silice au lieu d’un gradient de saccharose pour une élimination plus efficace de la myéline. Enfin, pour le comptage, l’utilisation d’un hémocytomètre est l’étalon-or pour le comptage et l’inspection visuelle des noyaux. Dans notre protocole, cette étape peut être automatisée de manière fiable à l’aide d’un compteur de cellules fluorescentes automatisé disponible dans le commerce22. Ce protocole a été testé et est compatible avec plusieurs tissus de mammifères congelés, y compris le cerveau, les reins, la rate et le foie, de différentes espèces de mammifères (rat, souris et primate non humain) et fournit des noyaux de bonne qualité pour le séquençage de l’ARN à noyau unique en aval avec une plate-forme commerciale à base de gouttelettes. Le protocole prend environ 75 minutes entre la préparation des tissus et le début du processus de séquençage de l’ARN à noyau unique.
Protocole
Toutes les études sur les animaux ont été menées avec l’approbation de l’autorité vétérinaire cantonale de Bâle-Ville dans le strict respect de la réglementation fédérale suisse sur la protection des animaux ou avec l’approbation de la Commission institutionnelle de protection et d’utilisation des animaux conformément à la loi allemande sur le bien-être des animaux.
1. Préparation des tissus et des réactifs/instruments
- Nettoyage et préparation des instruments
- Nettoyez les paillasses et les pinces à épiler avec une solution de décontamination à 70 % d’éthanol et de RNase. Pré-refroidir les centrifugeuses à 4 °C.
- Pré-refroidir les cartouches d’isolement des noyaux au réfrigérateur à 4 °C pendant au moins 30 min.
- Démarrez le dissociateur robotique et activez le refroidissement en réglant le curseur en haut à droite de l’écran pour refroidir et en cliquant dessus pour démarrer le refroidissement afin que le curseur apparaisse orange. Vérifiez que le flacon de réactif de stockage des noyaux (NSR) et le flacon de tampon d’isolement des noyaux (NIB) ont suffisamment de liquide et qu’ils sont correctement refroidis.
- Préparez une boîte en mousse de polystyrène remplie de glace carbonique et prérefroidissez les boîtes de Pétri et les lames de scalpel sur de la glace sèche.
- Préparation du tampon
- Préparez la solution de coussin de saccharose (SCS) de 1,5 M comme indiqué dans le tableau 1. Répartir le SCS en aliquotes de 500 μL dans des tubes de DNase/RNase de 2 mL pour obtenir quatre aliquotes de SCS de 500 μL par échantillon. Conservez les aliquotes sur de la glace jusqu’à leur prochaine utilisation.
- En cas de traitement du tissu cérébral, préparer à la place une solution colloïdale de silice à 18% comme décrit dans le tableau 2, en diluant la solution mère de colloïdes de silice dans NSR et en ajoutant un inhibiteur de la RNase. Préparez 3 mL de solution colloïdale de silice à 18 % par échantillon et conservez-la sur de la glace.
2. Homogénéisation des tissus et isolement des noyaux
- Retirez l’échantillon du congélateur à -80 °C et placez-le immédiatement sur de la glace carbonique.
- Coupez l’échantillon sur une boîte de Pétri pré-refroidie ou une plaque métallique sur de la glace carbonique avec un scalpel pré-refroidi en un morceau de 15 à 50 mg (s’il n’est pas déjà de la bonne taille). Assurez-vous de couper l’échantillon dans le bon sens afin qu’il soit toujours représentatif des structures d’organes qui vous intéressent.
REMARQUE : Avec ce protocole, la taille optimale de l’échantillon est de 15 à 50 mg pour l’extraction nucléaire. Pour obtenir un bon rendement après le nettoyage, une taille d’échantillon d’au moins 25 mg est recommandée. Pour les échantillons plus petits, des cartouches spéciales sont disponibles et sont optimisées pour le traitement de petites entrées avec le dissociateur robotisé. De petites cartouches d’isolement de noyaux d’entrée ont été utilisées pour dissocier des échantillons de tissus pesant aussi peu que 4 mg avec un rendement suffisant pour le séquençage de l’ARN à noyau unique en aval. Le tableau 3 donne un exemple de rendement des noyaux à l’aide de la cartouche à faible apport provenant de tissus hépatiques de rat. - Retirez la cartouche d’isolement des noyaux du réfrigérateur, déballez-la, retirez le broyeur et pipetez 15 μL d’inhibiteur de la RNase (40 U/μL) au fond de la cartouche.
REMARQUE : Pendant l’extraction des noyaux, le dissociateur robotisé ajoutera de la NIB et de la NSR à la cartouche jusqu’à un volume total de 3 mL (avec le protocole d’extraction de noyaux à faible volume). En ajoutant 15 μL d’inhibiteur de la RNase (40 U/μL) à la cartouche avant l’extraction, la suspension aura la concentration souhaitée d’inhibiteur de l’ARNase de 0,2 U/μL. - Placez l’échantillon de tissu au fond de la cartouche à l’aide d’une pince à épiler. Ne placez pas l’échantillon exactement au centre de la cartouche afin d’avoir une efficacité de perturbation optimale.
- Sélectionnez Exécuter un protocole sur l’instrument et cliquez sur l’option Noyaux dans le coin supérieur gauche.
- Dans le menu, sélectionnez le protocole Low Volume Nuclei Isolation et vérifiez que la vitesse de perturbation est réglée sur rapide en cliquant sur Modifier. Chargez la cartouche sur l’instrument en ouvrant la porte et en faisant glisser la platine vers l’extérieur en soulevant le bouton rouge.
- Insérez la cartouche à l’emplacement désigné, faites pivoter le verrou de la cartouche et faites glisser la platine jusqu’à ce que le bouton rouge s’enclenche. Fermez la porte et lancez l’extraction des noyaux sur l’instrument en cliquant sur Suivant. La course dure environ 7 min.
- Une fois le passage terminé, retirez la cellule de l’instrument en soulevant le bouton rouge et en tirant sur la platine. Placez immédiatement la cartouche sur de la glace.
- Pour tous les tissus, à l’exception du cerveau, passez à l’étape 3.1. Pour les échantillons de cerveau, passez directement à l’étape 3.2.
3. Nettoyage des noyaux
- Nettoyage du gradient de saccharose
REMARQUE : Pour les tissus cérébraux, sautez cette étape et passez directement à l’étape 3.2. Toutes les étapes de nettoyage sont effectuées sur de la glace afin de minimiser la dégradation de l’ARN. Les tampons et les tubes ainsi que les centrifugeuses doivent être pré-refroidis. Toutes les étapes de remise en suspension et de mélange sont effectuées uniquement par pipetage minutieux, car le vortex pourrait compromettre la qualité et l’intégrité des noyaux.- Percez délicatement la feuille ronde de la cartouche de dissociateur à l’aide d’une pointe de pipette.
REMARQUE : Après dissociation, la suspension de noyaux résultante a un volume approximatif de 2 mL. Pour faciliter le nettoyage du gradient de saccharose, diviser la suspension de noyaux en deux aliquotes de 900 μL, ce qui donne un volume total de 1,8 mL de suspension de noyaux utilisé pendant le nettoyage. - Retirer les 900 premiers μL d’aliquote de la suspension de noyaux de la cartouche et les ajouter à une aliquote SCS de 500 μL préalablement préparée dans un tube de 2 mL. Bien mélanger en pipetant jusqu’à ce que le mélange soit homogène.
- Retirez les 1400 μL de suspension de noyaux - mélange SCS et superposez-les soigneusement sur une nouvelle aliquote SCS de 500 μL en tenant le tube en biais et en ajoutant le mélange goutte à goutte, créant une séparation de phase clairement visible (voir Figure 1A).
- Fermez soigneusement le tube et replacez-le sur de la glace sans perturber la séparation des phases.
- Répéter les étapes 3.1.2 à 3.1.4 avec la deuxième aliquote de 900 μL de suspension et une nouvelle aliquote SCS pour avoir deux tubes de 2 mL par échantillon avec une séparation de phase clairement visible pour la centrifugation en gradient.
- Ajouter délicatement les tubes dans une centrifugeuse pré-refroidie et essorer à 13 000 x g pendant 15 min à 4 °C.
- Dans l’intervalle, préparez la NSR décrite dans le tableau 4, en ajoutant l’inhibiteur de la RNase à une aliquote de NSR. Préparer 1 mL de NSR par échantillon.
REMARQUE : À ce stade, les billes de gel de réactif d’expression génique unicellulaire peuvent être retirées du congélateur à -80 °C, ce qui leur permet de s’équilibrer à température ambiante (RT), et l’oligo du commutateur de modèle peut être remis en suspension dans un tampon à faible TE. - Après la centrifugation, retirez le surnageant des deux tubes sans perturber la pastille et remettez soigneusement la pastille en suspension dans 50 μL de NSR glacé selon les recommandations du fabricant. Regroupez les deux pastilles du même échantillon dans un nouveau tube de 1,5 mL et ajoutez 900 μL de NSR glacé à un volume total de 1 mL. Bien mélanger en pipetant.
- Centrifuger l’échantillon à 500 x g pendant 5 min à 4 °C à l’aide d’une centrifugeuse à rotor à godets oscillants.
REMARQUE : Il est fortement recommandé d’utiliser un rotor à godets oscillants pour minimiser la perte de noyaux, en particulier lorsque le rendement des noyaux est prévu pour être faible ou lorsque vous commencez avec de petites quantités de tissu. - Dans l’intervalle, préparer 500 μL par échantillon de 1x PBS (sans Ca2+ et Mg2+) avec 0,04 % d’albumine sérique bovine (BSA) et 0,2 U/μL d’inhibiteur de la RNase, comme décrit dans le tableau 5.
- Retirer délicatement le surnageant sans jeter la pastille et remettre la pastille en suspension dans 100 μL de solution de PBS comme préparé ci-dessus (1x PBS + 0,04 % BSA + inhibiteur de la RNase à 0,2 U/μL).
REMARQUE : Pour les petits échantillons de tissus, la concentration des noyaux peut être faible et, par conséquent, il est recommandé de remettre la pastille en suspension dans seulement 50 μL de solution de PBS afin d’assurer des concentrations suffisamment élevées pour le séquençage de l’ARN à noyau unique. - Passez directement à l’étape 4.
- Percez délicatement la feuille ronde de la cartouche de dissociateur à l’aide d’une pointe de pipette.
- Nettoyage du gradient colloïdal de silice
REMARQUE : Pour le tissu cérébral, un gradient colloïdal de silice est plus approprié qu’un gradient de saccharose pour éliminer la myéline et les débris de la suspension des noyaux. Toutes les étapes de nettoyage sont effectuées sur de la glace afin de minimiser la dégradation de l’ARN. Les tampons et les tubes, ainsi que les centrifugeuses, doivent être pré-refroidis. Toutes les étapes de remise en suspension et de mélange sont effectuées uniquement par pipetage minutieux, car le vortex pourrait compromettre la qualité et l’intégrité des noyaux.- Percez délicatement la feuille ronde de la cartouche de dissociateur à l’aide d’une pointe de pipette.
- Retirez la suspension de noyaux de la cartouche et ajoutez-la dans un tube de 5 ml.
- Centrifuger à 500 x g pendant 5 min à 4 °C dans une centrifugeuse pré-refroidie.
- Retirez délicatement le surnageant sans perturber la pastille et remettez la pastille en suspension dans 1 mL de solution colloïdale de silice glacée à 18 %.
- Ajouter 2 mL supplémentaires de solution colloïdale de silice à 18 % pour obtenir un volume total de 3 mL et bien mélanger par pipetage.
- Centrifuger l’échantillon à 700 x g pendant 5 min à 4 °C dans un rotor à godets oscillants avec le frein serré.
- Dans l’intervalle, préparez la NSR décrite dans le tableau 4 en ajoutant l’inhibiteur de la RNase à une aliquote de NSR. Préparer 1 mL de NSR par échantillon.
REMARQUE : À ce stade, les billes de gel de réactif d’expression génique unicellulaire peuvent être retirées du congélateur à -80 °C, ce qui leur permet de s’équilibrer à RT et l’oligo du commutateur de modèle peut être remis en suspension dans un tampon à faible TE. - Retirez délicatement l’échantillon de la centrifugeuse sans perturber la couche de myéline flottant sur le dessus.
- Tout d’abord, retirez la couche de myéline du haut et jetez-la ; Ensuite, retirez délicatement l’ensemble du surnageant sans perturber la pastille.
REMARQUE : La couche de myéline peut être facilement enlevée en enroulant une lingette stérile non pelucheuse autour d’un embout de pipette de 1 mL pour aspirer la couche de myéline avec 1 à 2 mL de surnageant. - Remettre la pastille en suspension dans 1 mL de NSR glacé selon les recommandations du fabricant.
- Centrifuger l’échantillon à 500 x g pendant 5 min à 4 °C dans une centrifugeuse à rotor à godets oscillants.
REMARQUE : Il est fortement recommandé d’utiliser un rotor à godets oscillants pour minimiser la perte de noyaux, en particulier lorsque le rendement des noyaux est prévu pour être faible ou lorsque vous commencez avec de petites quantités de tissu. - Dans l’intervalle, préparer 500 μL par échantillon de 1x PBS (sans Ca2+ et Mg2+) avec 0,04 % d’albumine sérique bovine (BSA) et 0,2 U/μL d’inhibiteur de la RNase, comme décrit dans le tableau 5.
- Retirer délicatement le surnageant sans perturber la pastille et remettre l’échantillon en suspension dans 100 μL de solution de PBS comme préparé ci-dessus (1x PBS + 0,04 % BSA + inhibiteur de la RNase à 0,2 U/μL).
REMARQUE : Pour les petits échantillons de tissus, la concentration des noyaux peut être faible et, par conséquent, il est recommandé de remettre la pastille en suspension dans seulement 50 μL de solution de PBS afin d’assurer des concentrations suffisamment élevées pour le séquençage de l’ARN à noyau unique.
4. Comptage
- Pour chaque échantillon à compter, diluer 10 μL de suspension de noyaux dans 20 μL de solution de PBS pour obtenir une dilution de 1 :3.
- Pour le comptage, ajouter 25 μL de solution de coloration à l’iodure de propidium (PI) dans un puits de mélange de la plaque de comptage fluorescente. Ajouter 25 μL de la suspension de noyaux dilués et bien mélanger par pipetage. Transférez l’échantillon coloré de 50 μL du puits de mélange au puits de chargement.
- Chargez la plaque de comptage sur le compteur de cellules et commencez le comptage.
REMARQUE : Le nombre de noyaux est prélevé dans le canal fluorescent rouge avec un temps d’exposition de 700 ms. Ce canal a été optimisé pour obtenir un comptage précis des noyaux par comparaison croisée avec le comptage manuel avec une chambre de Neubauer et la coloration au bleu de trypan au microscope. À des concentrations élevées, les noyaux sont très proches les uns des autres, ce qui rend difficile leur séparation par le logiciel. Dans ce cas, il est recommandé de recompter l’échantillon dans une dilution appropriée. L’intégrité des noyaux ainsi que la propreté peuvent être évaluées à partir de l’image en fond clair ou au microscope. - Diluer les échantillons avec du PBS (1x PBS + 0,04 % BSA + inhibiteur de la RNase à 0,2 U/μL) à la concentration souhaitée pour le séquençage de l’ARN à noyau unique.
REMARQUE : Des concentrations comprises entre 700 et 1 200 noyaux/μL sont considérées comme optimales pour le séquençage de l’ARN à noyau unique. Des concentrations cellulaires plus faibles, telles que 700 noyaux/μL, peuvent entraîner une réduction de la contamination de fond par l’ARN ambiant.
5. Préparation de la bibliothèque
- Effectuez le séquençage de l’ARN à noyau unique avec les réactifs d’expression génique unicellulaire en utilisant le protocole du fabricant ciblant une récupération de noyaux de 8000 à 10 000 noyaux par échantillon.
6. Séquençage
- Séquençez les bibliothèques avec la profondeur de séquençage souhaitée avec une indexation double à extrémité appariée et les lectures de séquençage suivantes : Lecture 1 : 28 cycles, Index i7 : 10 cycles, Index i5 : 10 cycles et Lecture 2 : 90 cycles.
Résultats Représentatifs
La performance et la polyvalence de ce protocole sont démontrées par la réalisation d’un séquençage de l’ARN à noyau unique sur du tissu du cortex occipital cérébral frais congelé de trois souris B6, du tissu rénal frais congelé coupé transversalement de trois rats Wistar, du foie et du tissu de rate d’archives (âgés de 11 ans) de trois macaques Cynomolgus mauriciens. Tous les animaux n’étaient pas perfusés.
Comme le montrent les figures 1B, C, on a obtenu des noyaux de bonne qualité qui présentaient des signes de saignement, de débris et d’agglutination. La filtration basée sur le gradient de saccharose a été optimisée pour éliminer la majorité des débris en testant différentes densités, vitesses et temps de rotation, et en évaluant la pureté et l’intégrité nucléaires au microscope, ainsi qu’en évaluant la distribution de la taille et le rendement des noyaux (Figure 1D). Cela nous a permis de choisir une densité de gradient de saccharose de 1,5 M et d’utiliser un temps d’essorage court de 15 min. Ensuite, pour évaluer plus en détail la qualité des noyaux, les données ont été prétraitées à l’aide de 10X Cell Ranger, et une analyse plus poussée des données en aval a été effectuée à l’aide de Besca23. Les noyaux avec >5% de contenu mitochondrial (car ils ont tendance à être des noyaux endommagés/stressés) ont été filtrés, et les noyaux avec 500 à 7 000 gènes (pour minimiser les gouttelettes vides et les multiplets) ont été conservés. Nous n’avons inclus que les gènes présents dans au moins 30 noyaux. Nous avons ciblé 8 000 noyaux par échantillon de cortex cérébral et 10 000 noyaux par échantillon de rein, de foie et de rate. Après filtration, 10 644 noyaux de haute qualité provenant des trois échantillons de cerveau, 14 960 noyaux de haute qualité provenant des trois échantillons de rein, 18 795 noyaux de haute qualité provenant des trois échantillons de foie et 13 882 noyaux de haute qualité provenant des trois échantillons de rate ont été obtenus. Les figures 2A, D, G et J montrent des diagrammes de violon représentant la distribution du nombre d’UMI, du nombre de gènes et du contenu mitochondrial dans chaque échantillon. Le nombre médian de comptages dans tous les échantillons de cerveau était de 7 563 UMI/noyau et de 3 208 gènes/noyau. Le nombre médian de numération dans tous les échantillons de rein était de 3 841 UMI/noyau et de 1 915 gènes/noyau. Le nombre médian de numération dans tous les échantillons de foie était de 2 649 UMI/noyaux et de 1 676 gènes/noyaux. Le nombre médian de numération dans tous les échantillons de rate était de 1 609 UMI/noyaux et de 1 138 gènes/noyaux. Nous avons ensuite généré des clusters à l’aide de gènes très variables et les avons annotés à l’aide des gènes marqueurs connus 17,24,25,26. Comme le montre la figure 2B, E, H, K, nous avons pu identifier les types de cellules attendus de chaque tissu. De plus, comme le montrent les figures 2B, E, H et K, tous les animaux ont contribué à tous les groupes, ce qui indique une faible variabilité technique introduite par le protocole. De plus, les proportions cellulaires étaient comparables dans les trois échantillons par type de tissu, tout comme l’UMI et le nombre de gènes (Figure 2A, C, D, F, G, I, J, L). Une exception notable est le foie, où les populations d’hépatocytes parmi les trois échantillons de foie étaient différentes en proportion et en profil. Cela est probablement dû à des différences biologiques entre les animaux (sexe, âge, statut métabolique).
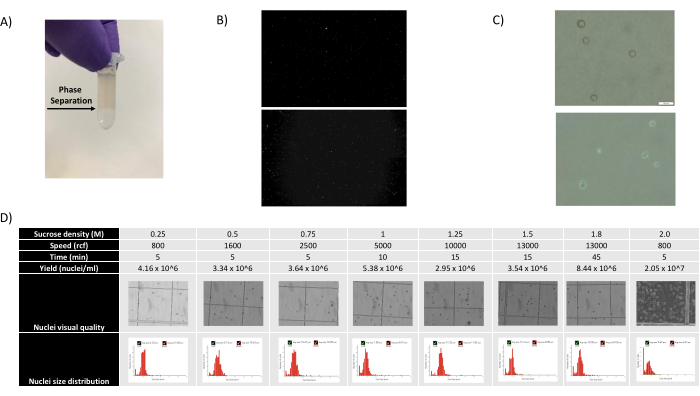
Figure 1 : Évaluation de la qualité des noyaux et optimisation du gradient de saccharose. (A) La séparation de phase attendue lors de la centrifugation par gradient de saccharose est indiquée par une flèche. (B) Images fluorescentes représentatives des noyaux de rein de rat (en haut) et de rate à cynomolgus (en bas) colorés à l’iodure de propidium obtenues avec le protocole. (C) Images représentatives de microscopie à fond clair de noyaux isolés du foie de souris (en haut) et du cerveau de souris (en bas), barre d’échelle de 500 μm. Notez la surface lisse et régulière des noyaux indiquant une bonne qualité nucléaire. (D) Optimisation du gradient de saccharose. Plusieurs densités de saccharose, vitesses d’essorage et temps d’essorage ont été testés. Des images microscopiques à fond clair des noyaux, de la distribution de la taille des noyaux et du rendement des noyaux sont présentées pour chaque condition. Veuillez cliquer ici pour voir une version agrandie de cette figure.
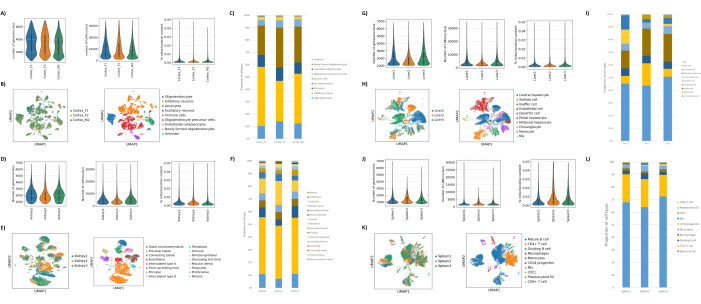
Figure 2 : Données représentatives de snRNAseq sur le cortex occipital cérébral de souris, le rein de rat (cortex et moelle) et le foie et la rate du macaque cynomolgus. (A) Diagrammes de violon montrant la distribution des gènes/noyau, des UMIs/noyau, et le pourcentage de contenu mitochondrial par échantillon de cerveau. (B) Panneau de gauche : tracé UMAP montrant la contribution de chaque échantillon aux amas identifiés dans le cerveau. Panneau de droite : UMAP montrant les identités des clusters annotés en fonction des gènes marqueurs dans les tissus cérébraux. (C) Proportions cellulaires observées dans les 3 échantillons de cerveau. (D) Diagrammes de violon montrant la distribution des gènes/noyau, des UMI/noyau, et le pourcentage de contenu mitochondrial par échantillon de rein. (E) Panneau de gauche : Graphique UMAP montrant la contribution de chaque échantillon aux grappes identifiées dans le rein. Panneau de droite : UMAP montrant l’identité des clusters annotés en fonction des gènes marqueurs dans le tissu rénal. (F) Proportions cellulaires observées dans les 3 échantillons de reins. (G) Diagrammes de violon montrant la distribution des gènes/noyau, des UMI/noyau et le pourcentage de contenu mitochondrial par échantillon de foie. (H) Panneau de gauche : Graphique UMAP montrant la contribution de chaque échantillon aux grappes identifiées dans le foie. Panneau de droite : UMAP montrant l’identité des clusters annotés en fonction des gènes marqueurs dans le tissu hépatique. (I) Proportions cellulaires observées dans les 3 échantillons de foie. (J) Diagrammes de violon montrant la distribution des gènes/noyau, des UMIs/noyau, et le pourcentage de contenu mitochondrial par échantillon de rate. (K) Panneau de gauche : Graphique UMAP montrant la contribution de chaque échantillon aux grappes identifiées dans la rate. Panneau de droite : UMAP montrant l’identité des clusters annotés en fonction des gènes marqueurs dans le tissu de la rate. (L) Proportions cellulaires observées dans les 3 échantillons de rate. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Composants | Concentration des stocks | Volume par échantillon | Concentration finale |
| Solution de coussin de saccharose | 2 millions | 1500 μL | 1,5 M |
| Coussin de saccharose Buffer | - | 500 μL | - |
| Dithiothréitol (DTT) | 1 M | 2 μL | 1 mM |
| Inhibiteur de l’ARNase | 40 U/μL | 10 μL | 0,2 U/μL |
Tableau 1 : Préparation d’une solution de coussin de saccharose à 1,5 M. Cette solution est utilisée pour la centrifugation du gradient de saccharose pendant le nettoyage de l’étape 3.1 et doit être fraîchement préparée à chaque fois avant de commencer le protocole. Gardez toujours le SCS sur la glace pendant le protocole. Les solutions mentionnées dans ce tableau sont référencées dans le tableau des matériaux.
| Composants | Concentration des stocks | Volume par échantillon | Concentration finale |
| Solution mère colloïdale de silice | 90% | 600 μL | 18% |
| Réactif de stockage des noyaux (génomique S2) | - | 2400 μL | - |
| Inhibiteur de l’ARNase | 40 U/μL | 15 μL | 0,2 U/μL |
Tableau 2 : Préparation d’une solution colloïdale de silice à 18 %. Cette solution est utilisée pour la centrifugation à gradient colloïdal de silice pendant le nettoyage de l’étape 3.2 et doit être fraîchement préparée à chaque fois avant de commencer le protocole. Gardez toujours la solution colloïdale de silice à 18 % sur de la glace pendant le protocole.
| Tissu | Poids de l’échantillon | Cartouche | Rendement |
| Foie de rat | 25 mg (en anglais seulement) | Cartouche d’isolement Nuclei | 65 000 noyaux par mg de tissu |
| Foie de rat | 4 mg (en anglais seulement) | Cartouche d’isolement de petits noyaux d’entrée | 32 000 noyaux par mg de tissu |
Tableau 3 : Rendement des noyaux de la cartouche d’isolement des noyaux à faible entrée par rapport à la cartouche d’isolement des noyaux après le nettoyage du gradient de saccharose.
| Composants | Concentration des stocks | Volume par échantillon | Concentration finale |
| Réactif de stockage des noyaux | - | 1000 μL | - |
| Inhibiteur de l’ARNase | 40 U/μL | 5 μL | 0,2 U/μL |
Tableau 4 : Préparation du réactif de stockage des noyaux (NSR). Cette solution est utilisée lors de l’isolement des noyaux aux étapes 3 à 5 ainsi que lors du nettoyage à l’étape 3.1.8. Il peut être conservé à 4 °C jusqu’à 4 mois. Préparez une aliquote fraîche avec un inhibiteur de la RNase pendant l’étape de centrifugation de l’étape de nettoyage 6. Les solutions mentionnées dans ce tableau sont référencées dans le tableau des matériaux.
| 1x PBS + 0,04 % BSA solution mère | |||
| Composants | Concentration des stocks | Volume pour le stock | Concentration finale |
| PBS (pas de Ca 2+, pas de Mg2+) | 1 fois | 30 ml | - |
| Albumine sérique bovine (BSA) | 30% | 40 μL | 0.04% |
| 1x PBS + 0,04 % BSA + 0,2 U/μL inhibiteur de l’ARNase | |||
| Composants | Concentration des stocks | Volume par échantillon | Concentration finale |
| 1x PBS + 0,04 % BSA Solution mère | - | 500 μL | - |
| Inhibiteur de l’ARNase | 40 U/μL | 2,5 μL | 0,2 U/μL |
Tableau 5 : Préparation du PBS + 0,04 % BSA. Cette solution est utilisée à la fin du nettoyage de l’étape 3.1.10 et après le comptage pour diluer la suspension de noyaux à la concentration souhaitée pour le séquençage de l’ARN à noyau unique 10X (étape de comptage 4.4). La solution mère peut être conservée à 4 °C jusqu’à 1 mois. Préparez une aliquote fraîche avec un inhibiteur de la RNase pendant l’étape de centrifugation de l’étape de nettoyage 6.
Discussion
Nous avons développé un protocole polyvalent et partiellement automatisé pour obtenir des noyaux uniques de haute qualité à partir de tissus de mammifères congelés et démontré le protocole sur le cerveau de souris, les reins de rat et les tissus de foie et de rate cynomolgus.
En comparant les performances de ce protocole à celles d’autres protocoles publiés pour le séquençage de l’ARN à noyau unique dans le cerveau, les reins, la rate et les tissus hépatiques 6,7,20,24,25,26, nous observons que nous sommes capables de détecter un nombre similaire de gènes et de nombres UMI par noyau et que nous sommes capables de récupérer les types de cellules attendus. Par rapport aux méthodes existantes, ce protocole présente plusieurs avantages. Tout d’abord, le protocole de cette étude automatise l’homogénéisation tissulaire et l’isolement de noyaux uniques. Ceci est réalisé grâce à l’utilisation d’un perturbateur tissulaire robotisé21. Dans la plupart des protocoles, le tissu est homogénéisé à l’aide d’un homogénéisateur Dounce afin de libérer des noyaux uniques 3,20. Cependant, nous avons remarqué que cette étape manuelle peut conduire à une variabilité expérimentale du rendement et de l’intégrité des noyaux en fonction de la quantité de force exercée lors de l’homogénéisation, compromettant la reproductibilité des expériences. Ici, en utilisant un broyeur de tissus automatisé avec des réglages fixes, une bonne qualité de noyaux et un rendement plus constant ont été obtenus à travers les expériences. De plus, l’automatisation de cette étape réduit également le temps de prise en main du protocole (l’étape de perturbation tissulaire prend environ 7 minutes), ce qui permet à l’utilisateur de se préparer aux étapes suivantes. Deuxièmement, le protocole décrit dans cette étude est polyvalent, c’est-à-dire qu’il est compatible avec différents tissus de différentes espèces. Cela nous permet d’éviter une longue optimisation du protocole, par exemple pour identifier des tampons/détergents d’homogénéisation pour différents tissus 2,5,6. Troisièmement, ce protocole ne dépend pas de l’accès à un trieur de flux pour obtenir des noyaux propres, ce qui le rend plus accessible aux laboratoires qui ne disposent pas de l’équipement/de l’expertise requis pour le tri de flux. Au lieu de cela, nous avons optimisé la filtration basée sur le gradient de saccharose pour éliminer la plupart des débris. Cependant, pour le tissu cérébral en particulier, l’utilisation d’un gradient colloïdal de silice au lieu d’un gradient de saccharose est recommandée pour une élimination plus efficace de la myéline. Nous avons également constaté que l’utilisation d’un rotor à godets oscillants à la fin de l’étape de centrifugation à gradient colloïdal saccharose/silice minimise la perte de noyaux. Par conséquent, l’utilisation d’un tel rotor est fortement recommandée. Quatrièmement, après avoir testé plusieurs méthodes de comptage des noyaux (comptage manuel au microscope, utilisation de plusieurs compteurs automatisés), l’utilisation d’un compteur automatisé de cellules fluorescentes22 est recommandée. L’utilisation d’un colorant intercalant l’ADN, tel que l’iodure de propidium, augmente la précision du comptage des noyaux. Cinquièmement, ce protocole prend environ 75 minutes entre le début et le chargement de la puce microfluidique. Cela permet de s’assurer que l’intégrité des noyaux reste élevée lors du traitement de plusieurs échantillons. Enfin, nous avons constaté que le protocole était également compatible avec les tissus incorporés dans un composé à température de coupe optimale (OCT). En cas d’utilisation d’un tel matériau, le tissu peut être retiré du bloc OCT à l’aide d’un scalpel avant l’homogénéisation.
L’un des défis fréquents dans les ensembles de données de séquençage de l’ARN à noyau unique est la présence d’ARN ambiant, qui peut être non nucléaire (par exemple, mitochondrial) ainsi que d’origine nucléaire27,28. Dans notre protocole, l’ARN mitochondrial (un indicateur de l’ARN ambiant non nucléaire) est faible même avant le filtrage (0,1-1,6% pour les tissus présentés). Cependant, à l’instar d’autres protocoles et ensembles de données, la contamination par l’ARN ambiant à partir de gènes hautement exprimés dans les noyaux de types cellulaires abondants (tels que les hépatocytes dans le foie, les neurones dans le cerveau, etc.) est toujours présente27. Il existe plusieurs outils bioinformatiques, tels que CellBender, SoupX, etc., qui permettent d’éliminer une telle contamination par l’ARN ambiant avant l’annotation des noyaux 29,30,31. Une autre limite de ce protocole est que, bien que les étapes de perturbation tissulaire et d’isolement des noyaux soient automatisées, le débit de cette étape est toujours limité car un seul échantillon peut être traité à la fois. Cependant, comme cette étape ne prend qu’environ 7 minutes par morceau de tissu, plusieurs échantillons peuvent toujours être traités dans un lot. Nous traitons généralement quatre échantillons par lot, mais nous avons fait jusqu’à six échantillons par lot avec de bons résultats. Les récentes améliorations apportées au dissociateur robotisé pour permettre le traitement parallèle de deux échantillons simultanément permettront le traitement de 8 à 12 échantillons par lot, ce qui est compatible avec le débit de la puce microfluidique utilisée pour l’encapsulation de noyaux uniques.
Bien que nous n’ayons pas utilisé les noyaux isolés par ce protocole pour d’autres applications en aval telles que l’ATAC-seq ou le snRNAseq en utilisant d’autres plateformes, sur la base de la qualité des données obtenues avec les réactifs d’expression génique utilisés ici, nous pensons que notre protocole devrait être compatible avec d’autres applications en aval. Cependant, les travaux futurs consisteront à tester ce protocole avec d’autres applications en aval, telles que ATAC-seq.
En conclusion, nous avons développé un protocole d’isolement de noyaux rapide, simple et partiellement automatisé pour le séquençage de l’ARN d’un seul noyau en aval qui s’est avéré compatible avec différents types de tissus de mammifères congelés.
Déclarations de divulgation
Tous les auteurs sont/étaient des employés de F. Hoffmann-La Roche lors de la réalisation de l’étude.
Remerciements
Les auteurs tiennent à remercier Filip Bochner, Marion Richardson, Petra Staeuble et Matthias Selhausen pour avoir fourni les tissus animaux qui ont été analysés dans ce manuscrit. Nous tenons également à remercier Petra Schwalie, Klas Hatje, Roland Schmucki et Martin Ebeling pour leur soutien bioinformatique.
matériels
| Name | Company | Catalog Number | Comments |
| 1 M DTT | Thermo Fisher Scientific | P2325 | |
| 10% Tween 20 | Bio-Rad | 1662404 | |
| 10x Magnetic Separator | 10x genomics | PN-120250 | |
| 10x Vortex Adapter | 10x genomics | PN-120251 | |
| 1x DPBS (10x), no calcium, no magnesium | Thermo Fisher Scientific | 14190144 | stored at 4°C |
| 30% Bovine Serum Albumin | Sigma-Aldrich | A9576_50ML | |
| 400 mM Tris-HCl, pH 8.0 | Thermo Fisher Scientific | 15568025 | |
| 40U/μl RNaseOUT Recombinant Ribonuclease Inhibitor | Thermo Fisher Scientific | 10777019 | Stored at -20 °C |
| Agilent High Sensitivity DNA Kit | Agilent | 5067-4626 | |
| Cellaca MX High-throughput Automated Cell Counter | Nexcelom Bioscience | CELMXSYSF2 | Automated fluorescent cell counter |
| Chromium Next GEM Chip G Single Cell Kit, 16 rxns | 10x genomics | PN-1000127 | Single cell gene expression reagent, stored at room temperature |
| Chromium Next GEM Secondary Holder | 10x genomics | PN-1000195 | |
| Chromium Next GEM Single Cell 3' Gel Bead Kit v3.1, 4 rxns | 10x genomics | PN-1000129 | Single cell gene expression reagent, stored at -80 °C |
| Chromium Next GEM Single Cell 3' GEM, Library & Gel Bead Kit v3.1, 4 rxns | 10x genomics | PN-1000128 | Single cell gene expression reagent |
| Chromium Next GEM Single Cell 3' Library Kit v3.1 4 rxns | 10x genomics | PN-1000158 | Single cell gene expression reagent, stored at -20 °C |
| Chromium Next GEM Single Cell 3'GEM Kit v3.1 4 rxns | 10x genomics | PN-1000130 | Single cell gene expression reagent, stored at -20 °C |
| Divided Polystyrene Reservoirs | VWR | 41428-958 | |
| DNA LoBind Tubes 1.5ml Eppendorf | Sigma-Aldrich | EP0030108051 | |
| DNA LoBind Tubes 2ml Eppendorf | Sigma-Aldrich | EP0030108078 | |
| Dry ice | - | - | |
| Dynabeads MyOne SILANE | 10x genomics | PN-2000048 | Single cell gene expression reagent, stored at 4 °C |
| Ethanol Pure | Sigma-Aldrich | E7023 | |
| Glycerin (Glycerol), 50% (v/v) | Ricca Chemical Company | 3290-16 | |
| Heatblock | |||
| High-Throughput Nexcelom Counting Plates | Nexcelom Bioscience | CHM24-A100-001 | Cell counter counting plate |
| Low TE Buffer (10 mM Tris-HCl pH 8.0, 0.1 mM EDTA) | Thermo Fisher Scientific | 12090015 | |
| Mini Centrifuge | - | - | |
| NovaSeq 6000 SP Reagent Kit v1.5 (100 cycles) | Illumina | 2002840 | |
| Nuclei Isolation Buffer | S2 Genomics | 100-063-396 | Stored at 4 °C |
| Nuclei Isolation Cartridge | S2 Genomics | 100-063-287 | Precooled at 4 °C before use |
| Nuclei PURE 2 M Sucrose Cushion Solution | Sigma-Aldrich | NUC201-1KT | Sucrose cushion solution |
| Nuclei PURE Sucrose Cushion Buffer | Sigma-Aldrich | NUC201-1KT | |
| Nuclei Storage Reagent | S2 Genomics | 100-063-405 | Stored at 4 °C |
| PCR Tubes 0.2 ml 8-tube strips | Eppendorf | 30124359 | |
| Percoll | GE Healthcare | 17-0891-02 | Silica colloid solution |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Qiagen Buffer EB | Qiagen | 19086 | |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32854 | |
| Refrigerated Centrifuge (Eppendorf 5804R) | Eppendorf | 5805000010 | |
| Refrigerated Centrifuge with Swinging-Bucket Rotor (Eppendorf 5810R) | Eppendorf | 5811000015 | |
| RNAseZap | Ambion | AM9780 | RNAse decontamination solution |
| Round cell culture petri dish | SPL | 330005 | |
| Scalpel disposable | Aesculap AG | BA210 | pre-cooled on dry ice before use |
| Single Index Kit T Set A, 96 rxns | 10x genomics | PN-1000213 | Single cell gene expression reagent, stored at -20 °C |
| Singulator 100 System | S2 Genomics | - | Commercially available robotic tissue dissociator |
| Sodium Hydroxide 1M | Sigma-Aldrich | 72068 | |
| SPRIselect Reagent Kit | Beckman Coulter | b23318 | |
| Sterile tweezers | - | - | |
| UltraPure DNase/RNase-Free Distilled Water | Thermo Fisher Scientific | 10977049 | |
| ViaStain PI Staining Solution | Nexcelom Bioscience | CS1-0109-5mL | Propidium iodide staining solution |
| Vortex Mixer+A2:D44 | VWR | - |
Références
- Burja, B., et al. An Optimized Tissue Dissociation Protocol for Single-Cell RNA Sequencing Analysis of Fresh and Cultured Human Skin Biopsies. Front Cell Dev Biol. 10, 872688 (2022).
- Kimbley, L. M., et al. Comparison of optimized methodologies for isolating nuclei from esophageal tissue. Biotechniques. 72 (3), 104-109 (2022).
- Maitra, M., et al. Extraction of nuclei from archived postmortem tissues for single-nucleus sequencing applications. Nature Protocols. 16 (6), 2788-2801 (2021).
- Nadelmann, E. R., et al. Isolation of nuclei from mammalian cells and tissues for single-nucleus molecular profiling. Current Protocols. 1 (5), e132 (2021).
- Rousselle, T. V., et al. An optimized protocol for single nuclei isolation from clinical biopsies for RNA-seq. Scientific Reports. 12, 9851 (2022).
- Slyper, M., et al. A single-cell and single-nucleus RNA-Seq toolbox for fresh and frozen human tumors. Nature Medicine. 26 (5), 792-802 (2020).
- Leiz, J., et al. Nuclei isolation from adult mouse kidney for single-nucleus RNA-sequencing. Journal of Visualized Experiments: JoVE. (175), 62901 (2021).
- Alvarez, M., et al. Isolation of nuclei from human snap-frozen liver tissue for single-nucleus RNA sequencing. Bio-Protocol. 13 (3), e4601 (2023).
- Ayhan, F., Douglas, C., Lega, B. C., Konopka, G. Nuclei isolation from surgically resected human hippocampus. STAR Protocols. 2 (4), 100844 (2021).
- Joshi, N., Misharin, A. Single-nucleus isolation from frozen human lung tissue for single-nucleus RNA-seq. Protocols.io. , (2019).
- Martelotto, L. G., Luciano Martelotto, L. 'Frankenstein' protocol for nuclei isolation from fresh and frozen tissue for snRNAseq. Protocols.io. , (2020).
- Masilionis, I., Chaudhary, O., Chaligne, R., Mazutis, L. Nuclei extraction for single-cell RNAseq from frozen tissue using Singulator™ 100. Protocols.io. , (2022).
- Matson, K. J. E., et al. Isolation of adult spinal cord nuclei for massively parallel single-nucleus RNA sequencing. Journal of Visualized Experiments: JoVE. (140), 58413 (2018).
- Mendelev, N., et al. Multi-omics profiling of single nuclei from frozen archived postmortem human pituitary tissue. STAR Protocols. 3 (2), 101446 (2022).
- Soule, T. G., et al. A protocol for single nucleus RNA-seq from frozen skeletal muscle. Life Science Alliance. 6 (5), e202201806 (2023).
- Bakken, T. E., et al. Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PLoS One. 13 (12), e0209648 (2018).
- Ding, J., et al. Systematic comparison of single-cell and single-nucleus RNA-sequencing methods. Nature Biotechnology. 38, 737-746 (2020).
- Hu, P., et al. Single-nucleus transcriptomic survey of cell diversity and functional maturation in postnatal mammalian hearts. Genes & Development. 32 (19-20), 1344-1357 (2018).
- Lake, B. B., et al. A comparative strategy for single-nucleus and single-cell transcriptomes confirms accuracy in predicted cell-type expression from nuclear RNA. Scientific Reports. 7, 6031 (2017).
- Narayanan, A., et al. Nuclei Isolation from Fresh Frozen Brain Tumors for Single-Nucleus RNA-seq and ATAC-seq. Journal of Visualized Experiments: JoVE. (162), 61542 (2020).
- Jovanovich, S., et al. . Automated processing of solid tissues into single cells or nuclei for genomics and cell biology applications with the Singulator™ 100 and 200 systems. , (2022).
- Bell, J., et al. Characterization of a novel high-throughput, high-speed and high-precision plate-based image cytometric cell counting method. Cell & Gene Therapy Insights. 7 (4), 427-447 (2021).
- Madler, S. C., et al. Besca, a single-cell transcriptomics analysis toolkit to accelerate translational research. NAR Genomics and Bioinformatics. 3 (4), lqab102 (2021).
- Wu, H., et al. Mapping the single-cell transcriptomic response of murine diabetic kidney disease to therapies. Cell Metabolism. 34 (7), 1064-1078 (2022).
- Han, L., et al. Cell transcriptomic atlas of the non-human primate Macaca fascicularis. Nature. 604 (7907), 723-731 (2022).
- Madissoon, E., et al. scRNA-seq assessment of the human lung, spleen, and esophagus tissue stability after cold preservation. Genome Biology. 21 (1), 1 (2019).
- Caglayan, E., Liu, Y., Konopka, G. Neuronal ambient RNA contamination causes misinterpreted and masked cell types in brain single-nuclei datasets. Neuron. 110 (24), 4043-4056 (2022).
- Luecken, M. D., Theis, F. J. Current best practices in single-cell RNA-seq analysis: a tutorial. Molecular Systems Biology. 15 (6), e8746 (2019).
- Fleming, S. J., et al. Unsupervised removal of systematic background noise from droplet-based single-cell experiments using CellBender. bioRxiv. , (2022).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biology. 21 (1), 57 (2020).
- Young, M. D., Behjati, S. SoupX removes ambient RNA contamination from droplet-based single-cell RNA sequencing data. Gigascience. 9 (12), giaa151 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon