Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Génération et analyse d’images histologiques à paramètres élevés avec la cytométrie en flux histologique
Dans cet article
Résumé
La description ici est une méthode qui peut être utilisée pour imager cinq paramètres fluorescents ou plus par microscopie immunofluorescente. Un pipeline d’analyse permettant d’extraire des cellules uniques à partir de ces images et d’effectuer des analyses de cellules uniques à l’aide de stratégies de type cytométrie en flux est décrit, qui permet d’identifier des sous-ensembles de cellules dans des coupes de tissus.
Résumé
L’utilisation de l’histologie pour étudier la diversité des cellules immunitaires dans des coupes de tissus telles que celles dérivées du système nerveux central (SNC) est limitée de manière critique par le nombre de paramètres fluorescents qui peuvent être imagés en une seule fois. La plupart des sous-ensembles de cellules immunitaires ont été définis à l’aide de la cytométrie en flux à l’aide de combinaisons complexes de marqueurs protéiques, nécessitant souvent quatre paramètres ou plus pour être identifiés de manière concluante, ce qui dépasse les capacités de la plupart des microscopes conventionnels. Comme la cytométrie en flux dissocie les tissus et perd des informations spatiales, il est nécessaire de disposer de techniques capables de conserver des informations spatiales tout en interrogeant les rôles de types cellulaires complexes. Ces problèmes sont résolus ici par la création d’une méthode permettant d’augmenter le nombre de paramètres fluorescents qui peuvent être imagés en collectant les signaux de fluorophores qui se chevauchent spectralement et en utilisant le démélange spectral pour séparer les signaux de chaque fluorophore individuel. Ces images sont ensuite traitées à l’aide d’un pipeline d’analyse pour prendre des images histologiques à paramètres élevés et extraire des cellules uniques de ces images afin que les propriétés fluorescentes uniques de chaque cellule puissent être analysées au niveau d’une seule cellule. À l’aide de stratégies de type cytométrie en flux, les cellules peuvent ensuite être profilées en sous-ensembles et cartographiées sur les coupes histologiques pour non seulement quantifier leur abondance, mais aussi établir comment elles interagissent avec l’environnement tissulaire. Dans l’ensemble, la simplicité et le potentiel de l’utilisation de la cytométrie histoflux pour étudier des populations immunitaires complexes dans des coupes histologiques sont démontrés.
Introduction
L’inflammation provoquée par les cellules du système immunitaire et les cellules gliales peut contribuer à des troubles chroniques du SNC où chaque population peut favoriser l’activité de l’autre 1,2,3. Comprendre comment le système immunitaire interagit avec ces éléments du SNC pour favoriser l’inflammation du SNC est actuellement un sujet d’intérêt majeur et a été grandement facilité par des techniques à paramètres élevés telles que le séquençage de l’ARN unicellulaire. Grâce au séquençage de l’ARN unicellulaire, nous avons découvert qu’il existe une communication étendue entre les cellules gliales et le système immunitaire dans plusieurs troubles du SNC 4,5,6. Comprendre comment ces interactions affectent ces troubles sera crucial pour élucider la biologie de ces maladies.
L’un des problèmes avec les analyses de séquençage de cellules uniques est que ces techniques nécessitent que le tissu soit perturbé pour obtenir des cellules ou des noyaux uniques, ce qui entraîne une perte complète d’informations spatiales. Savoir où se trouve une cellule dans un tissu est essentiel pour comprendre le rôle de la cellule dans l’inflammation. Par exemple, les cellules immunitaires telles que les lymphocytes B peuvent se concentrer dans le SNC pendant la neuroinflammation ; cependant, ils pénètrent rarement dans le parenchyme du SNC et se concentrent plutôt dans les barrières du SNC7. Compte tenu de leur localisation, il est peu probable que ces cellules contribuent à l’inflammation du SNC en interagissant physiquement avec les cellules gliales du parenchyme du SNC, ce qui suggère que toute interaction qu’elles pourraient avoir avec les cellules gliales se produirait par le biais de facteurs sécrétés. De plus, la pathologie qui se produit dans les troubles du SNC a souvent une structure 8,9 telle que la localisation d’une cellule dans le tissu pourrait déterminer de manière critique si elle contribue activement au trouble ou si elle est un spectateur. Ainsi, l’utilisation de l’orientation spatiale pour évaluer le rôle d’une cellule dans la pathologie est essentielle.
L’étude des cellules dans les tissus a généralement été réalisée à l’aide de l’immunohistochimie ou de la microscopie à fluorescence immunitaire. Un problème avec ces techniques est qu’elles ne peuvent généralement imager que jusqu’à quatre paramètres simultanément. Il s’agit d’une limitation majeure de ces techniques, car nous savons, grâce à la cytométrie en flux et aux analyses de séquençage de l’ARN unicellulaire, que de nombreuses populations cellulaires nécessitent deux paramètres ou plus pour leur identification ; En outre, le nombre de paramètres requis augmente généralement lors de la recherche de sous-ensembles spécifiques d’un typede cellule 10. Ainsi, il n’est pas pratique d’utiliser des techniques d’imagerie standard pour étudier comment des sous-ensembles de cellules peuvent interagir au sein d’un tissu.
Ce problème a été partiellement surmonté grâce à de nouvelles méthodes à paramètres élevés qui peuvent conserver des informations spatiales, telles que le séquençage spatial de l’ARN11 et l’imagerie par cytométrie de masse12. Bien que ces techniques soient précieuses, elles présentent plusieurs problèmes, tels que le fait qu’elles ne sont pas largement disponibles, qu’elles réduisent les données tridimensionnelles en deux dimensions et qu’elles nécessitent une expertise considérable pour être exécutées. Une autre technique connue sous le nom de coloration séquentielle, dans laquelle les tissus sont colorés avec un ensemble d’anticorps suivi de l’inactivation de l’ensemble précédent d’anticorps avant la coloration avec un autre ensemble d’anticorps, peut obtenir une histologie à paramètres élevés sans avoir besoin d’équipement spécialisé ou d’expertise 13,14,15 . Cependant, la coloration séquentielle peut être extrêmement laborieuse et nécessite beaucoup de temps de microscopie, ce qui peut être peu pratique pour les laboratoires qui ne possèdent pas de microscope personnel. Par conséquent, il est nécessaire de mettre au point des techniques capables d’augmenter le nombre de paramètres fluorescents pouvant être imagés en même temps sur des microscopes largement disponibles et en temps opportun.
Une fois les données à paramètres élevés acquises, un autre problème se pose : les méthodes d’analyse d’images conventionnelles ont peu de chances d’analyser avec succès les données. Des techniques telles que le comptage manuel ou le seuillage ne sont viables que si l’analyse consiste en un seul paramètre ou si plusieurs marqueurs ont la même localisation où seuls les signaux qui se chevauchent sont comptés. Cette limitation rend l’analyse traditionnelle inadéquate pour travailler avec des ensembles de données à paramètres élevés. L’analyse réussie de ces ensembles de données a été réalisée en segmentant des cellules uniques à partir d’images histologiques, puis en mettant en œuvre des stratégies de contrôle de type cytométrie en flux pour identifier les types de cellules16,17. Cependant, un autre problème qui affecte ces analyses est qu’elles ne fonctionnent que pour des ensembles de données dans lesquels toutes les cellules d’intérêt sont physiquement séparées les unes des autres, car ces techniques n’utilisent pas de méthodes capables de séparer avec précision les cellules en contact physique. Ainsi, une méthode plus récente est nécessaire pour effectuer des analyses de cellules uniques sur des coupes histologiques même si les cellules sont en contact physique.
Dans cet article, nous décrivons un protocole simple appelé cytométrie histoflux, qui a déjà été introduit18 et qui augmente le nombre de paramètres fluorescents pouvant être imagés simultanément à l’aide de microscopes largement disponibles. Ce protocole fonctionne en colorant les tissus avec des colorants qui se chevauchent spectralement, puis en utilisant la compensation spectrale pour éliminer le saignement des canaux qui se chevauchent afin d’obtenir des colorants uniques clairs. Pour faciliter l’analyse d’images histologiques à paramètres élevés, un pipeline d’analyse détaillé est décrit qui extrait des cellules uniques de coupes de tissus dans le but de trier les cellules en populations distinctes à l’aide de stratégies de type cytométrie en flux. Ce protocole fonctionne dans les tissus où les cellules sont présentes de manière diffuse et dans les tissus où les cellules sont étroitement compactées les unes contre les autres, ce qui rend cette technique polyvalente pour l’étude de tissus comme le SNC dans l’homéostasie et la neuroinflammation. La cytométrie histoflux est donc une technique utile pour étudier les interactions entre des types de cellules complexes qui nécessitent plusieurs marqueurs cellulaires pour définir les cellules tout en conservant des informations spatiales.
Protocole
Ce protocole ne couvre pas la section des tissus pour l’histologie ; veuillez consulter Jain et al.18 ou19 pour des descriptions sur la façon de sectionner les tissus pour l’histologie. Ce protocole peut être utilisé avec n’importe quel tissu sectionné sur des lames de verre. Cet article utilise des ganglions lymphatiques inguinaux isolés d’un animal immunisé comme décrit précédemment18. La procédure et le calendrier de ce protocole sont résumés à la figure 1. Les détails des réactifs et de l’équipement utilisés dans cette étude sont énumérés dans la table des matériaux.
1. Coloration de coupes de tissus
REMARQUE : En plus de colorer les tissus avec tous les marqueurs d’anticorps qui intéressent le chercheur, celui-ci doit également préparer des contrôles de couleur unique (un pour chaque paramètre fluorescent destiné à être utilisé) sur des lames de tissus adjacentes ou autrement identiques, dans lesquelles chaque contrôle de couleur unique est coloré avec les réactifs nécessaires pour produire le signal d’un paramètre fluorescent.
- Préparez la permanente et le tampon de blocage et le tampon de permanente et de coloration (voir le tableau 1 pour la composition).
- Décongelez des sections de tissu sur des lames de verre à température ambiante et essuyez la condensation à l’aide d’une lingette. Utilisez un marqueur hydrophobe pour délimiter le bord du ou des tissus à colorer et laissez sécher les marques.
- Réhydratez les tissus en trempant les sections de tissus dans une solution saline tamponnée au phosphate (PBS) pendant 1 min. Secouez le PBS et utilisez une lingette pour enlever l’excès de PBS sans toucher directement le tissu.
- Fixez les tissus en ajoutant suffisamment de paraformaldéhyde (PFA) à 4 % pour couvrir complètement le tissu. Incuber à température ambiante pendant 20 min.
ATTENTION : Le PFA peut être toxique et doit être manipulé à l’intérieur d’une hotte.
REMARQUE : Cette étape n’est requise que pour les tissus frais congelés. Si le tissu est déjà fixé, ignorez cette étape. La fixation du PFA est décrite ici, mais le tissu peut être fixé à l’aide d’autres méthodes en fonction des besoins de l’expérience. - Lavez les mouchoirs dans un bain PBS pendant 1 minute avec un léger balancement, puis retirez la lame du bain. Remplacez le PBS et répétez le lavage 3 fois de plus.
- Secouez l’excès de PBS et utilisez une lingette pour enlever l’excès de PBS sans toucher directement le tissu.
- Déterminer quels anticorps secondaires doivent être utilisés à l’étape 1.12 du protocole et l’espèce animale dont les anticorps secondaires sont dérivés. Enrichissez la permanente et le tampon de blocage en ajoutant du sérum de ces animaux à la réserve permanente et bloquante à une concentration finale de 5 % pour chaque espèce.
REMARQUE : L’utilisation d’anticorps secondaires permet d’amplifier l’intensité du signal. Ainsi, il est recommandé que les anticorps primaires qui produisent des signaux faibles soient associés à des anticorps secondaires pour amplifier leurs signaux. Si toutes les cibles d’anticorps colorées sont suffisamment brillantes pour ne pas nécessiter d’anticorps secondaires, les étapes 1.7 à 1.12 et 1.15 peuvent être sautées. - Bloquez la liaison non spécifique des anticorps en ajoutant suffisamment de Perm enrichi et de tampon de blocage pour couvrir chaque tissu. Incuber les lames pendant 1 à 8 h à température ambiante dans une chambre humidifiée à l’abri de la lumière.
- Ajouter des anticorps primaires non conjugués à la Perm et au tampon de coloration à des dilutions déterminées expérimentalement (les isotypes d’espèces ou d’immunoglobulines des anticorps primaires doivent tous être différents et reconnaissables aux anticorps secondaires que le chercheur a l’intention d’utiliser à l’étape 1.12).
- Videz la permanente et le tampon de blocage et utilisez une lingette pour enlever l’excès de tampon sans toucher directement le tissu. Ajoutez suffisamment de Perm et de tampon de coloration avec des anticorps primaires sur le tissu pour le couvrir et incubez la lame à 4 °C pendant 12 à 48 h.
REMARQUE : Le chercheur devra déterminer le moment optimal pour l’incubation des anticorps. - Préparez le tampon de blocage et le tampon de coloration (Tableau 1).
- Ajouter des anticorps secondaires au tampon de coloration préparé à l’étape 1.11 à des dilutions déterminées expérimentalement. Les anticorps secondaires utilisés doivent cibler les mêmes espèces ou isotypes d’anticorps que ceux utilisés à l’étape 1.9.
- Préparez le tampon d’extinction (comme dans le tableau 1).
REMARQUE : Cette étape aide à réduire l’autofluorescence des tissus. Si cela n’est pas nécessaire, les étapes 1.13 et 1.14 peuvent être ignorées. - Lavez les mouchoirs comme décrit aux étapes 1.5-1.6. Ensuite, ajoutez suffisamment de tampon de trempe pour couvrir les tissus sur les lames et incubez les lames à température ambiante pendant 1 à 2 minutes.
- Lavez le mouchoir comme décrit aux étapes 1.5-1.6. Ensuite, ajoutez les anticorps secondaires dilués dans le tampon de coloration préparé à l’étape 1.12 aux tissus de sorte que les tissus soient complètement recouverts du tampon de coloration. Incuber les lames à 4 °C pendant 2 à 8 h.
- Préparez la solution bloquante en ajoutant 50 μL de sérum normal de l’espèce d’anticorps qui sera utilisée à l’étape 1.18 (5 % de chaque espèce) au tampon de blocage préparé à l’étape 1.11.
- Lavez les mouchoirs comme décrit aux étapes 1.5-1.6. Ensuite, ajoutez la solution bloquante avec du sérum normal préparée à l’étape 1.16 aux tissus afin qu’ils soient complètement recouverts d’un tampon de coloration. Incuber les lames à température ambiante pendant 1 à 8 h.
- Préparez la solution de coloration en ajoutant des anticorps primaires conjugués aux fluorophores à des dilutions déterminées expérimentalement dans le tampon de coloration préparé à l’étape 1.11. Ajoutez également une coloration nucléaire, telle que le jaune nucléaire, à la solution de coloration à une dilution déterminée expérimentalement.
REMARQUE : Les étapes 1.18 et 1.19 ne sont requises que si le chercheur doit utiliser des marqueurs d’anticorps supplémentaires qui ne nécessiteront pas d’anticorps secondaires pour l’amplification du signal. Si vous n’utilisez pas d’anticorps conjugués primaires, des colorants de noyaux peuvent être ajoutés à l’étape 1.12. - Secouez le tampon bloquant et utilisez une lingette pour enlever l’excès de tampon sans toucher directement le tissu. Ajouter la quantité de solution de coloration préparée à l’étape 1.18 sur le tissu pour le couvrir et incuber à 4 °C pendant 12 à 48 h.
- Lavez les mouchoirs comme décrit aux étapes 1.5-1.6. Ajoutez 5 gouttes de collant sur chaque lame sans introduire de bulles, et basculez doucement et lentement une lamelle en verre sur le tissu pour couvrir complètement le tissu de montage et exclure toutes les bulles d’air. Stockez les lames à 4 °C dans l’obscurité jusqu’à ce qu’elles soient prêtes à être imagées.
2. Imagerie des coupes de tissus
REMARQUE : À ce stade, les coupes de tissus sont colorées avec tous les anticorps d’intérêt pour toutes les conditions qui doivent être imagées, et les contrôles unicolores sont colorés avec un paramètre fluorescent sur chaque section. L’imagerie doit être effectuée sur un microscope doté de plusieurs détecteurs qui peuvent être réglés pour détecter des gammes spécifiques de lumière, et idéalement, il devrait avoir accès à autant de lignes laser pour l’excitation que possible.
- Insérez l’une des lames entièrement colorées dans le microscope et configurez le microscope pour imager la coupe à l’aide de l’objectif destiné à être utilisé pour la collecte de données.
- Sur la base des fluorophores avec lesquels le tissu est coloré, identifiez les lignes laser d’excitation nécessaires pour stimuler ces fluorophores et identifiez les plages d’émission pour chaque fluorophore qui contiennent la plus grande quantité de signal.
REMARQUE : Les outils de visualisation des spectres de fluorescence en ligne (par exemple, voir le Tableau des matériaux) peuvent être utiles pour identifier les longueurs d’onde d’excitation optimales des fluorophores et identifier les plages optimales pour la détection des fluorophores. Les fluorophores utilisés pour la cytométrie histoflux doivent être choisis de manière à ce que chaque fluorophore diffère par la ligne laser optimale par laquelle le fluorophore est stimulé ou diffère par la gamme optimale de longueurs d’onde d’émission. Les fluorophores qui ont des longueurs d’onde d’excitation/émission plus proches seront plus difficiles à séparer. Les fluorophores utilisés pour cette étude sont détaillés dans la section des résultats. - Ouvrez le logiciel 1 (voir Table des matériaux) dans un microscope confocal et sélectionnez l’onglet de configuration en haut. Sélectionnez ensuite le bouton matériel sur le côté gauche et modifiez la profondeur de bits sur 16 bits.
REMARQUE : Il est recommandé d’utiliser des profondeurs de bits plus élevées, telles que des données de 16 bits, pour la collecte de données, car cela améliorera la précision de la compensation spectrale. De plus, des densités de pixels plus élevées, des nombres plus élevés pour les moyennes de lignes et plus d’étapes dans la pile z amélioreront la qualité de la séparation spectrale. - Sélectionnez l’onglet Acquérir en haut de l’écran et entrez le paramètre de format pour inclure la densité de pixels de l’image (généralement, des résolutions plus élevées produisent de meilleurs résultats).
- Si disponible, sélectionnez l’acquisition X bidirectionnelle pour accélérer le temps d’imagerie, à condition que cette fonction ait été correctement calibrée.
- Choisissez une valeur de sténopé pour le microscope confocal (généralement entre 400 et 600 nm) où des valeurs plus petites produiront généralement des résultats de cytométrie histoflux de meilleure qualité, bien que des valeurs de sténopé plus petites rendront plus difficile l’acquisition d’images et peuvent nécessiter un temps d’imagerie supplémentaire ou des intensités d’excitation plus élevées pour produire des images.
- Sélectionnez Afficher le panneau de balayage séquentiel en haut du menu de gauche pour ouvrir l’option de balayage séquentiel. Utilisez le signe « + » pour configurer des séquences supplémentaires, où chaque séquence contiendra une longueur d’onde d’excitation décidée à l’étape 2.2.
- Allumez tous les lasers nécessaires en cliquant sur les boutons ON associés aux lasers d’intérêt au centre de l’écran pour les mettre en état ON. Entrez dans chaque séquence spécifique et augmentez la puissance laser au centre de l’écran jusqu’à la valeur souhaitée, où chaque séquence aura une longueur d’onde d’excitation utilisée.
- Allumez autant de détecteurs que nécessaire dans la partie inférieure de l’écran dans chaque séquence pour imager tous les fluorophores qui seront imagés sur cette ligne laser. Ajustez les portées des détecteurs en double-cliquant sur leurs plages et en saisissant les plages supérieure et inférieure des longueurs d’onde qui doivent être collectées.
REMARQUE : Il est recommandé d’utiliser des détecteurs à haute sensibilité dans la mesure du possible et, idéalement, tous les canaux doivent être collectés sur les mêmes types de détecteurs, bien que cela ne soit pas obligatoire. - Appuyez sur le bouton live, puis concentrez-vous sur l’échantillon. Utilisez le bouton Auto Scale à gauche de l’image dans le logiciel pour évaluer si les canaux sont suffisamment saturés de signal. L’utilisateur doit ajuster les valeurs de gain et les intensités laser dans chaque séquence jusqu’à ce que la mise à l’échelle automatique n’atteigne pas la valeur maximale (65535 pour les données 16 bits) mais soit très proche de la saturation pour tous les canaux.
REMARQUE : Étant donné que la luminosité de différents fluorophores peut différer et que l’abondance de différentes cibles d’anticorps peut différer, la sensibilité des détecteurs devra être ajustée pour être de faible sensibilité pour les signaux lumineux et de sensibilité élevée pour les signaux faibles si les deux fluorophores sont stimulés par le même laser (l’intensité du laser ne peut pas changer dans ce scénario car les deux fluorophores seraient affectés). Pour de meilleurs résultats, le signal de fluorescence de chaque canal de l’expérience doit être équilibré de manière à ce que chaque canal ait des quantités de signal à peu près équivalentes, ce qui réduira le saignement spectral vers d’autres canaux et se traduira par des signaux plus forts. Étant donné que l’abondance des marqueurs d’intérêt colorés peut différer d’un échantillon à l’autre, il est recommandé de tester les réglages du microscope dans plusieurs conditions expérimentales pour confirmer qu’il n’y a pas de blanchiment dans l’une ou l’autre des conditions expérimentales. - Une fois les paramètres du microscope finalisés, chargez le microscope avec les lames de contrôle couleur uniques et trouvez une coloration représentative pour chaque fluorophore. Configurez une pile Z en vous concentrant sous l’échantillon et en appuyant sur le bouton de début dans le menu de la pile z, puis en vous concentrant sur l’échantillon et en appuyant sur le bouton de fin .
- Choisissez le nombre d’étapes et la distance entre les étapes (et utilisez le même nombre d’étapes et de paramètres tels que les densités de pixels et la profondeur de bits qui seront utilisés pour imager les diapositives entièrement colorées), puis appuyez sur le bouton Démarrer.
REMARQUE : Idéalement, ces images devraient inclure les signaux les plus brillants de la coloration réelle que l’on peut trouver sur la diapositive. - Une fois que les commandes monochromes ont été imagées, utilisez les mêmes paramètres de microscope pour imager tous les échantillons entièrement colorés.
3. Compensation de fluorescence
- Ouvrez les fichiers .lif dans le logiciel 1 et cliquez sur l’onglet processus.
- Choisissez le module Séparation des colorants , puis choisissez l’option Séparation automatique des colorants . Sélectionnez la méthode manuelle pour la séparation des colorants et choisissez aucune redimensionnement.
- Ouvrez l’une des images de contrôle monochromes. Inspectez manuellement les commandes monochromes pour détecter les fuites spectrales d’un canal à l’autre en observant les fenêtres en niveaux de gris pour chaque canal afin de détecter la présence d’un signal dans des canaux inappropriés. Utilisez le bouton Mise à l’échelle automatique si nécessaire pour voir un fond perdu faible.
- Commencez à supprimer le fond perdu en saisissant manuellement des nombres (généralement 0-1, où 0 représente aucun fond perdu et 1 représente un fond perdu de 100 %) dans la matrice à l’écran, puis appuyez sur Appliquer pour vérifier si le fond perdu est correctement supprimé. Cette opération doit être répétée jusqu’à ce que les signaux de tous les autres canaux, à l’exception du canal d’intérêt, aient été réduits aux niveaux de fond.
REMARQUE : Une surcompensation entraînera des résultats inexacts, de sorte que la fluorescence doit être réduite aux niveaux de fond mais pas en dessous de ceux-ci. Les données surcompensées apparaîtront comme un canal ayant un signal et d’autres canaux semblant avoir des trous noirs où se trouve le signal d’intérêt (Figure 2). - Une fois l’une des commandes monochromes terminée, enregistrez les valeurs dans la matrice, puis réinitialisez la matrice et passez à la commande monochrome suivante. Répétez ce processus pour chaque contrôle de couleur.
REMARQUE : Le premier colorant sera dans la rangée du colorant 1, et la quantité de fluorescence qui doit être retirée du colorant 1 sera dans les colonnes adjacentes. - Rassemblez toutes les valeurs acquises de chaque contrôle dans une matrice et appliquez cette matrice à un échantillon entièrement coloré. Si la séparation semble exacte, appliquez les paramètres à toutes les images de l’expérience, sinon, réévaluez les commandes de couleur unique.
REMARQUE : Une compensation efficace devrait éliminer la majeure partie du saignement entre les canaux, comme résumé et illustré à la figure 3. - Enregistrez tous les fichiers compensés dans un fichier .lif séparé.
4. Identifier les noyaux et les cellules à l’aide d’ilastik
- Ouvrez les fichiers .lif compensés dans FIJI20. Activez l’affichage des piles dans un hyperstack et sélectionnez une image.
- (Facultatif) Utilisez un outil de sélection tel que l’outil rectangle pour inclure les parties de l’image qui seront analysées et exclure les zones qui ne seront pas analysées. Recadrez l’image à l’aide de la fonction de recadrage de l’image >.
REMARQUE : la réduction de la taille de l’image est utile dans les étapes suivantes pour réduire la consommation de mémoire vive (RAM) et augmenter la vitesse de traitement. - Enregistrez l’image en tant que fichier .tif. Enregistrez également l’image en tant que fichier hdf5 en utilisant le plugin ilastik pour FIJI en cliquant sur Plugins > ilastik > Export hdf5. Enregistrez le fichier sous le nom « nom de votre choix ».h5 avec une compression de 0.
- Ouvrez Ilastik21 et créez un projet de classification de pixels. Utilisez la fonction Ajouter de nouvelles images > Ajouter des images distinctes pour ajouter au moins 3 images représentatives en ajoutant de nouvelles données d’entrée.
- Cliquez avec le bouton droit de la souris sur l’une des images, sélectionnez Modifier les propriétés et changez le mode d’affichage en niveaux de gris.
- Sur la gauche de l’écran, sélectionnez l’onglet de sélection de fonctionnalité. Cliquez sur Sélectionner les fonctionnalités et sélectionnez toutes les fonctionnalités jusqu’à la valeur sigma la plus élevée que le programme vous permettra d’utiliser, puis appuyez sur OK.
- Sélectionnez maintenant l’onglet Formation. À l’aide de l’outil pinceau (pendant que l’étiquette un est sélectionnée) pour mettre en évidence les cellules d’intérêt (figure 4) de sorte que l’intérieur (cytoplasme et noyau) et la membrane cellulaire soient inclus dans la mise en évidence.
- Répétez cette opération sur tous les échantillons d’apprentissage où tous les types de cellules d’intérêt sont sélectionnés, et où plusieurs cellules d’intensités, de morphologies et de localisations variables dans le tissu doivent être sélectionnées à partir de chaque échantillon.
REMARQUE : Selon les marqueurs choisis pour un ensemble individuel de colorants, il peut y avoir ou non des colorants intracellulaires ou des colorations de membrane cellulaire pour les cellules d’intérêt. Quels que soient les marqueurs utilisés, assurez-vous que la mise en évidence comprend la coloration la plus externe sur les cellules d’intérêt et inclut l’intérieur de la cellule, qu’une coloration intracellulaire soit utilisée ou non. - Ensuite, utilisez l’étiquette deux pour mettre en évidence tout ce qui n’est pas les cellules d’intérêt (Figure 4). Il peut s’agir d’espaces vides, de noyaux non marqués ou de tissus qui n’ont pas de marqueurs d’intérêt. Assurez-vous d’inclure l’espace immédiatement adjacent aux cellules d’intérêt pour favoriser l’apprentissage des limites exactes des cellules.
- Utilisez la fonction de mise à jour en direct pour évaluer la qualité de la formation sur les images qui vous intéressent. Si la formation est inadéquate, poursuivez la formation aux étapes 4.7 à 4.9. Sinon, si vous êtes satisfait, cliquez sur l’onglet Exportation de prédiction .
- Définissez la source comme étant les probabilités. Cliquez sur le bouton Choisir les paramètres d’exportation de l’image , puis définissez le paramètre c pour exporter 0-1 (en supposant que l’étiquette 1 met en évidence les paramètres d’intérêt).
- Convertissez le type de données en entier 16 bits et exportez-le en tant que fichier hdf5 dans le répertoire qui vous intéresse avec le nom qui vous intéresse (nous vous recommandons d’utiliser {nickname} mask.h5), puis appuyez sur OK.
- Cliquez sur l’onglet Traitement par lots . Appuyez sur le bouton Sélectionner les fichiers de données brutes , sélectionnez tous les fichiers .h5 d’intérêt qui nécessitent un traitement, puis appuyez sur Ouvrir. Cliquez sur Traiter tous les fichiers.
- Répétez les étapes 4.4 à 4.13, sauf qu’au lieu d’identifier les cellules, identifiez maintenant les noyaux et excluez tout le reste (Figure 4). Exportez-la en tant que prédiction distincte pour l’identification des noyaux cellulaires.
- Ouvrez Ilastik et créez un nouveau projet de conversion de données. Ajoutez un fichier hdf5 représentatif dans l’onglet Données d’entrée à l’aide de la fonction Ajouter un nouveau > Ajouter une image distincte .
- Cliquez sur l’onglet Exportation de données et choisissez le bouton Choisir les paramètres d’exportation de l’image avec la source définie à saisir. Changez le format en tiff de plusieurs pages, choisissez un emplacement d’enregistrement avec la fonction select et définissez le nom du fichier sur {nickname}. Appuyez sur OK.
- Cliquez sur l’onglet Traitement par lots et sélectionnez tous vos fichiers hdf5 à l’aide du bouton Sélectionner les fichiers de données d’entrée et appuyez sur le bouton Traiter tous les fichiers .
- Ouvrez le logiciel 2 (voir la table des matériaux) et ajoutez tous les fichiers tif/tiff générés aux étapes 4.3 et 4.17 à l’aide du bouton Ajouter des fichiers . Choisissez un emplacement pour les sorties de fichiers et appuyez sur le bouton Démarrer tout pour convertir tous les fichiers en fichiers .ims. Les fichiers sont maintenant prêts pour l’analyse dans le logiciel 3 (voir la table des matériaux).
5. L’analyse dans le logiciel 3
- Ouvrez l’une des images contenant toutes les taches fluorescentes. Sélectionnez Édition-> Ajouter des canaux et ajoutez les fichiers .ims correspondant aux masques créés sur les noyaux et les corps cellulaires. Répétez cette opération pour tous les échantillons.
- Sélectionnez le bouton Ajouter de nouvelles cellules . Sélectionnez l’option Détecter le noyau et la cellule et avancez.
- Sélectionnez le masque sur les noyaux qui sera le canal source de détection des noyaux et sélectionnez l’option avancée pour diviser les noyaux par points d’amorçage. Choisissez une valeur pour le diamètre du noyau) et avancez.
REMARQUE : La valeur optimale du diamètre du noyau devra être testée en exécutant la simulation et en évaluant manuellement si le programme ne divise pas suffisamment de noyaux (plusieurs noyaux sont regroupés) ou s’il y a sur-division (un noyau est divisé en plus d’un). - Choisissez une valeur pour l’évaluation de la qualité, puis poursuivez. En règle générale, il est préférable d’inclure tous les noyaux, car l’omission de noyaux peut entraîner la fusion de plusieurs cellules en une seule.
- Choisissez une valeur seuil pour la création de surfaces nucléaires, puis continuez. Assurez-vous que tous les noyaux, même ceux qui sont plus faibles, sont toujours inclus dans le seuil. Ne rendez pas le seuil trop bas, car cela pourrait surestimer la taille des noyaux ou potentiellement inclure des débris comme coloration nucléaire.
- Sélectionnez une valeur pour le nombre minimum de Voxels par noyau et continuez. En règle générale, il est préférable de tout conserver, à moins que les débris ou les non-noyaux ne puissent être exclus à l’aide de ce paramètre.
- Choisissez l’option Limite cellulaire et cytoplasme , sélectionnez le masque sur les cellules comme canal source pour la détection des corps cellulaires, puis continuez.
- Choisissez un seuil de cellule qui capture avec précision toutes les cellules d’intérêt, mais qui n’est pas si bas qu’il inclut la détection d’arrière-plan ou toute détection non spécifique des cellules. Sélectionnez l’option permettant de diviser les cellules en contact de sorte qu’il y ait un noyau par cellule et qu’elles soient séparées par la distance des noyaux. Choisissez également d’étendre les cellules sur les noyaux , puis de continuer.
- Sélectionnez une valeur pour le nombre minimum de voxels par cellule et continuez. En règle générale, il est préférable de tout conserver, à moins que les débris ou les non-cellules ne puissent être exclus à l’aide de ce paramètre.
- Évaluez si les surfaces créées sur les cellules sont précises en vérifiant si plusieurs cellules ont été fusionnées ou si des cellules sont manquantes. Si les surfaces sont inadéquates, revenez à l’étape 5.2 et essayez de nouveaux paramètres jusqu’à ce que les paramètres optimaux soient déterminés.
- Une fois les meilleurs paramètres établis, cliquez sur l’objet de cellules créé dans le menu de gauche, cliquez sur l’onglet de création, puis cliquez sur le bouton Stocker les paramètres pour le lot pour enregistrer les paramètres utilisés.
- Ouvrez plusieurs fenêtres du logiciel 3 et ouvrez des images distinctes dans chaque fenêtre. Appuyez sur le bouton nouvelles cellules dans chaque fenêtre, choisissez les paramètres enregistrés dans le menu Paramètres de création de favoris et demandez à chaque fenêtre de s’exécuter jusqu’à la fin. Répétez cette opération autant de fois que nécessaire jusqu’à ce que tous les échantillons soient analysés.
REMARQUE : L’exécution de plusieurs instances du logiciel 3 accélère l’analyse de l’analyse, mais le nombre de fenêtres pouvant être exécutées en parallèle dépend de la vitesse de traitement de l’unité centrale de traitement (CPU) et de la quantité de RAM sur laquelle l’analyse est exécutée. - Une fois tous les échantillons terminés, cliquez sur l’objet cellules créées dans le menu de gauche de chaque image et cliquez sur l’onglet Statistiques. Cliquez sur le bouton Configurer la liste des valeurs de statistiques visibles , désélectionnez toutes les statistiques à l’exception de la valeur « Moyenne de l’intensité de la cellule », puis appuyez sur OK.
- Pour chaque image à analyser, cliquez sur le bouton Exporter toutes les statistiques dans un fichier dans l’onglet statistiques et enregistrez les fichiers à un emplacement.
6. Analyse par cytométrie histoflow et cartographie des populations sur des coupes de tissus
- Ouvrez Anaconda et JupyterLab dans Anaconda. Ensuite, ouvrez le script fourni dans le Fichier de codage supplémentaire 1. Exécutez le code à l’aide du menu Exécuter et sélectionnez Exécuter toutes les cellules, ou exécutez les cellules individuellement dans l’ordre en sélectionnant Exécuter les cellules sélectionnées.
- Lorsque vous êtes invité à saisir le fichier source, entrez le répertoire de fichiers pour les valeurs fluorescentes exportées générées à l’étape 5.14. Le répertoire de fichiers peut être obtenu en examinant les propriétés de l’un des fichiers .csv exportés et en copiant le chemin d’accès au fichier dans l’invite.
- Ensuite, le programme demandera un emplacement de sortie pour les fichiers traités. Entrez le répertoire de fichiers pour n’importe quel emplacement approprié pour l’enregistrement des fichiers traités. Le programme détectera automatiquement le nombre de canaux fluorescents contenus dans le dossier.
- Exécutez la section suivante du code pour annoter les données avec les noms de chaque canal. Ce code peut être modifié si nécessaire pour changer le paramètre « numberOfChannels » afin qu’il corresponde au nombre de canaux détectés à l’étape 6.3. De plus, les noms des canaux peuvent être modifiés pour autant de canaux que nécessaire en répétant la ligne 'ChX' :'Marker' autant de fois que nécessaire et en modifiant les noms des marqueurs.
- L’exécution de la ligne de code suivante créera un fichier .csv à l’emplacement spécifié qui est annoté et contient tous les paramètres fluorescents de l’ensemble de données. Répétez les étapes 6.1 à 6.5 pour chaque ensemble d’exportations du logiciel 3 afin de créer .csv fichiers pour toutes les données.
- Convertissez les fichiers .csv en fichiers .fcs en ouvrant le logiciel 4 (voir la table des matériaux) et en faisant glisser les fichiers .csv dans la fenêtre du programme. Le logiciel 4 commencera automatiquement à convertir les fichiers en fichiers .fcs.
- Double-cliquez sur l’un des échantillons dans le logiciel 4 pour ouvrir une fenêtre sur cet échantillon. Utilisez les outils de contrôle du logiciel 4 pour créer une stratégie de contrôle. Il est recommandé de commencer par établir un marqueur de définition de lignée contenant toutes les cellules d’intérêt, puis de passer à des portes qui suppriment d’abord les sous-ensembles les plus rares, puis de terminer par les types de cellules les plus abondants.
- Une fois qu’une stratégie de contrôle est établie, testez la précision du contrôle en cliquant avec le bouton droit de la souris sur cette population dans le menu et en choisissant Exporter. Exportez tous les paramètres sous forme de fichier .csv avec en-têtes inclus.
- Ouvrez JupyterLab dans Anaconda. Ensuite, ouvrez le script fourni dans le Fichier de codage supplémentaire 2 et exécutez le code. Lorsque vous êtes invité à saisir l’emplacement du fichier Software 4, entrez le répertoire du fichier .csv exporté généré à l’étape précédente. Le répertoire de fichiers peut être obtenu en examinant les propriétés du fichier .csv exporté et en copiant le chemin d’accès au fichier dans l’invite.
- Lorsque vous êtes invité à ajouter l’emplacement de sortie, choisissez le répertoire de fichiers de votre choix et assurez-vous que le répertoire de fichiers inclut le nom de fichier à la fin ('file directory'\filename.txt). Exécutez le reste du code.
- Copiez le texte dans le fichier .txt et ouvrez le fichier .ims correspondant dans le logiciel 3 pour le même échantillon que celui sur lequel les données exportées sont basées. Cliquez sur l’objet Cell dans le fichier, passez à l’onglet statistiques, collez le texte dans la barre de recherche et lancez la recherche. Toutes les cellules d’intérêt seront mises en évidence.
- Vérifiez manuellement si toutes les cellules d’intérêt ont été capturées à l’aide de la stratégie de déclenchement. Si le nombre de cellules est sous-estimé ou surestimé (exemples illustrés à la figure 5), revenez à l’étape 6.7, ajustez la porte et réévaluez si les cellules sont capturées avec précision avec la nouvelle porte. Répétez cette opération avec tous les points de contrôle de la stratégie de contrôle et évaluez le point de contrôle sur plusieurs échantillons.
- Lorsque toutes les portes sont validées, exportez les données du logiciel 4 à l’aide de la fonction d’édition de table et analysez les données à l’aide de la méthode d’analyse préférée.
REMARQUE : Les populations cellulaires peuvent être cartographiées sur les coupes de tissus dans le logiciel 3 en identifiant la population contrôlée comme aux étapes 6.8 à 6.11, puis en créant une surface basée sur les cellules sélectionnées. Cela peut être fait pour chaque population fermée, comme le montrent les figures 6 et 7.
Résultats
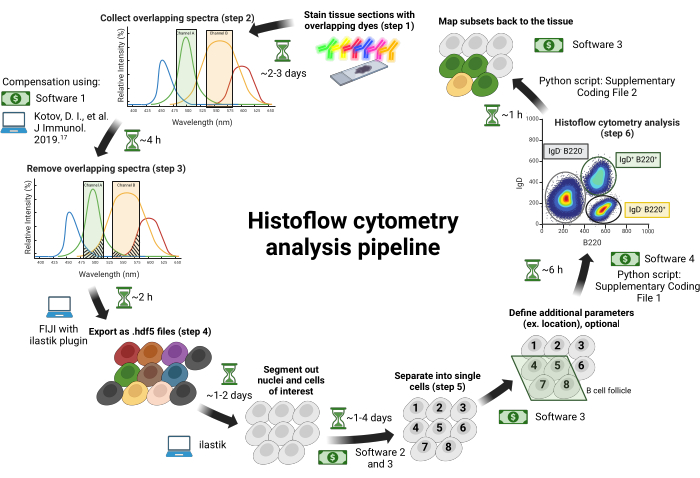
Figure 1 : Flux de travail de cytométrie histoflux. Des coupes de tissus sont colorées avec des colorants qui se chevauchent spectralement (étape 1). Les images sont collectées à travers des lasers d’excitation individuels associés à des filtres passe-bande réglables pour minimiser le saignement spectral entre les fluorophores (étape 2). ...
Discussion
Ici, l’utilisation de la cytométrie histoflux est décrite, une technique qui a été validée précédemment18. Il est démontré que lors de la coloration de coupes de tissus avec des colorants qui se chevauchent spectralement, cette fuite à travers les canaux peut être éliminée à l’aide de la compensation spectrale, ce qui permet de résoudre clairement un plus grand nombre de paramètres fluorescents que ce qui serait normalement possible avec les méthodes conventionnelles. Comme le...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts financier.
Remerciements
Nous remercions la plate-forme de microscopie avancée du Hotchkiss Brain Institute pour son infrastructure et son expertise en imagerie. RWJ a été soutenu par des bourses postdoctorales du programme Eyes High de l’Université de Calgary et par une bourse de formation sans restriction de la Société canadienne de la sclérose en plaques et de Roche Canada. VWY a reçu un soutien salarial du Programme des chaires de recherche du Canada de niveau 1. Ces travaux ont été financés par des fonds de fonctionnement provenant de la subvention 1049959 des Instituts de recherche en santé du Canada, de la subvention 3236 de la Société canadienne de la sclérose en plaques et du programme de recherche sur la sclérose en plaques dirigé par le Congrès du département de la Défense des États-Unis. La figure 1 est créée avec BioRender.com. Les chiffres adaptés dans cette publication ont été initialement publiés dans The Journal of Immunology. Rajiv W. Jain, David A. Elliott et V. Wee Yong. 2023. Analyse unicellulaire d’images histologiques à paramètres élevés à l’aide de la cytométrie histoflux. J. Immunol. 210: 2038-2049. Droit d’auteur © [2023]. L’Association américaine des immunologistes, Inc.
matériels
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
Références
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon