Method Article
PAR-Clip - שיטה לזהות Transcriptome רחב אתרי קישור של חלבונים RNA הכבילה
In This Article
Summary
תעתיקי רנ"א כפופים תקנה posttranscriptional נרחב כי הוא מתווך על ידי מספר רב של טרנס משחק RNA מחייב חלבונים (RBPs). כאן אנו מציגים שיטה להכליל לזהות במדויק בקנה מידה רחב transcriptome האתרים RNA מחייב של RBPs.
Abstract
תעתיקי רנ"א כפופים לרגולציה שלאחר תעתיק הגן על ידי אינטראקציה עם מאות RNA מחייב חלבונים (RBPs) ו microRNA המכילים קומפלקסים ribonucleoprotein (miRNPs), אשר באים לידי ביטוי לעתים קרובות סוג תא dependently. כדי להבין כיצד יחסי הגומלין הללו RNA מחייב גורמים משפיעים על ויסות תעתיקי אדם, מפות ברזולוציה גבוהה של חלבון in vivo-RNA אינטראקציות הכרחיים 1.
שילוב של גישות גנטיים ביוכימיים חישוביים מיושמים בדרך כלל לזהות RNA-RBP או RNA-RNP אינטראקציות. פרופיל microarray של RNAs הקשורים RBPs immunopurified (RIP-Chip) 2 מגדיר מטרות ברמה transcriptome, אבל היישום שלה מוגבל אפיון של אינטראקציות יציב kinetically ורק במקרים נדירים 3,4 מאפשר לזהות את מרכיב ההכרה RBP (RRE ) בתוך RNA היעד ארוך. עוד יעד ישיר RBP המידע באתר מתקבל על ידי שילוב של crosslinking UV vivo 5,6 עם immunoprecipitation 7-9 ואחריו הבידוד של crosslinked קטעי RNA ו רצפי cDNA (קליפ) 10. CLIP היה להשתמש בו כדי לזהות מטרות של מספר RBPs 11-17. עם זאת, CLIP הוא מוגבל על ידי היעילות הנמוכה של crosslinking UV 254 ננומטר RNA-חלבון, את המיקום של crosslink אינו לזיהוי בקלות בתוך שברי crosslinked רצף, ולכן קשה להפריד UV-crosslinked פלחי יעד RNA מרקע שאינו crosslinked RNA שברי נוכחת גם במדגם.
פיתחנו גישה חזקה מבוססי תאים crosslinking לקבוע ברזולוציה גבוהה transcriptome רחב של אתרי הקישור RBPs ו miRNPs הסלולר שאנחנו המונח PAR-Clip (Photoactivatable-Ribonucleoside משופרת Crosslinking ו immunoprecipitation) (ראה איור. 1A עבור מתאר של השיטה). השיטה מסתמכת על שילוב של אנלוגים ribonucleoside photoreactive, כגון 4-thiouridine (4-SU) ו - 6-thioguanosine (6-SG) לתוך המתהווה תעתיקי רנ"א על ידי תאים חיים. הקרנה של תאים על ידי אור UV של 365 ננומטר גורמת crosslinking יעיל של nucleoside שכותרתו RNAs photoreactive הסלולר RBPs אינטראקציה. Immunoprecipitation של RBP ריבית מלווה הבידוד של RNA crosslinked ו coimmunoprecipitated. RNA בודד מומר ספריית cDNA ו רצף עמוק באמצעות טכנולוגיית Solexa. אחד המאפיינים של ספריות cDNA שהוכן על ידי Clip-PAR היא כי מיקום מדויק של crosslinking ניתן לזהות על ידי מוטציות המתגוררים רצף cDNA. בעת שימוש 4-SU, רצפים crosslinked thymidine המעבר cytidine, ואילו באמצעות 6-SG תוצאות guanosine כדי אדנוזין מוטציות. הנוכחות של מוטציות ברצפי crosslinked מאפשרת להפריד אותם מהרקע של רצפי נגזר RNAs הסלולר בשפע.
יישום השיטה למספר מגוונים חלבונים RNA מחייב דווח Hafner et al. 18
Protocol
פרוטוקול להלן מתאר את הליך PAR-קליפ HEK293 תאים להביע הדגל / HA-tagged IGF2BP1 על אינדוקציה עם דוקסיציקלין. נשתמש נוגדן אנטי דגל immunoprecipitation.
PAR-Clip יעבוד עם כל שורת תאים להביע רמות לגילוי של אנדוגני, לא מתוייגת חלבון RNA מחייב (RBP) של עניין אם נוגדן יעיל immunoprecipitation זמין.
הרחבת תאים
- הרחב FlpIn-HEK293/TO/FLAG/HA-IGF2BP1 תאים במדיום הגידול. אנו ממליצים להשתמש בין 100-400 x 10 6 תאים (כ 10-40 ס"מ תא 15 צלחות תרבות) כנקודת מוצא. לגדל אותם כ 80% confluency.
- 14 שעות לפני crosslinking להוסיף) 4 thiouridine לריכוז סופי של 100 מיקרומטר (1:1000 v / v של 1 מ 4-thiouridine פתרון המלאי) ישירות אל המדיום תרבית תאים ו ב) להשרות ביטוי של דגל / HA מתויג IGF2BP1 על ידי תוספת של 1 מיקרוגרם / מ"ל של דוקסיציקלין (1:10,000 v / v פתרון דוקסיציקלין 10 מ"ג / מ"ל המלאי). הערה: במקום 4-thiouridine ניתן להשתמש גם 100 מיקרומטר של thioguanosine-6.
UV-Crosslinking
- שטפו תאים פעם אחת עם 10 מ"ל קר כקרח PBS לכל צלחת ולהסיר PBS לחלוטין.
- המקום הצלחות על מגש עם קרח מקרינים חשפו אור 0.15 J / cm 2 של 365 ננומטר UV ב Stratalinker 2400 (Stratagene) או מכשיר דומה.
- גרדו את התאים עם שוטר גומי 1 מ"ל PBS לכל העברה צלחת, עד 50 מ"ל צינורות צנטריפוגה ולאסוף ידי צנטריפוגה XG ב 500 דקות 5 ב 4 ° C ו להתעלמות supernatant. 100 x 10 6 HEK293 תאים (10 צלחות 15 ס"מ) תניב כ. 1 מ"ל של תא גלולה רטוב.
- (אופציונלי) אם ברצונך להמשיך ישירות עם תמוגה התא, הלם להקפיא את התא גלולה בחנקן נוזלי חנות ב -80 ° C. כדורי Cell יכול להיות מאוחסן במשך 12 חודשים לפחות.
תא תמוגה ו RNaseT1 לעכל
- קח את התא גלולה של תאים crosslinked ב 3 כרכים של חיץ NP40 1x תמוגה ו לדגור על קרח למשך 10 דקות.
- תא lysate נקה ידי צנטריפוגה ב XG 13,000 עבור 15 דקות ב 4 ° C.
- נקה את lysate נוספת היא על ידי סינון דרך ממברנה 0.2 מיקרומטר מזרק מסנן (Acrodisc מל או שווה ערך).
- הוסף RNase T1 (Fermentas, 10,000 U / μl) לריכוז סופי של 1 U / μl ו דגירה באמבט מים במשך 15 דקות על 22 ° C. תגובה Cool לאחר מכן במשך 5 דקות על הקרח לפני שתמשיך.
Immunoprecipitation והתאוששות של שברי היעד crosslinked RNA
שימוש ב מפריד מגנטי
פעל לפי ההנחיות ברחבי הכנת המדגם כדי למנוע חרוזים מגנטיים מהתייבשות.
- מניחים את הצינור המכיל את החרוזים על דוכן מגנטי עבור 1 2 דקות.
- מוסיפים את חיץ הצינור תוך הצינור הוא על מפריד מגנטי.
- שווי הצינור, להסירו מפריד מגנטיים, ועל resuspend את החרוזים. אתה יכול resuspend את החרוזים על ידי מצליף את הצינור בעזרת האצבע או להשתמש vortexer להגדיר 5 6.
- צנטריפוגה בקצרה לאסוף כל כך חרוזים עשויים להישאר בכובע הצינור.
- חזור על שלבים 1 עד 4 כנדרש.
הכנת חרוזים מגנטיים
- העברת 10 μl של Dynabeads חלקיקי חלבון G מגנטית (Invitrogen) לכל מ"ל lysate תא (עבור ניסוי טיפוסי יש כ. 40 50 μl של חרוזים) לצינור מ"ל 1.5 microfuge. שטפו פעמיים עם חרוזים מ"ל 1 של חיץ ציטרט, פוספט.
- Resuspend בהיקף של פעמיים חיץ ציטרט, פוספט יחסית לנפח המקורי של השעיה חרוז.
- הוסף 0.25 מיקרוגרם של נוגדנים חד שבטיים אנטי הדגל M2 (Sigma) לכל מ"ל ההשעיה ו לדגור על גלגל מסתובב במשך 40 דקות בטמפרטורת החדר.
- לשטוף חרוזים פעמיים מ"ל 1 של חיץ ציטרט, פוספט להסיר נוגדן מאוגד.
- Resuspend חרוזים בהיקף של פעמיים חיץ ציטרט, פוספט יחסית לנפח המקורי של השעיה חרוז.
Immunoprecipitation (IP), השני העיכול RNase T1 ו dephosphorylation
- הוסף 20 μl של נוגדנים מצומדות חרוזים מוכן טרי מגנטי לכל מ"ל של lysate חלקית RNase T1 תא מטופלים דגירה של 15 צינורות צנטריפוגה מ"ל על גלגל מסתובב במשך שעות 1 ב 4 ° C.
- איסוף חרוזים מגנטיים על אספן החלקיקים המגנטיים של 15 ו -50 צינורות צנטריפוגה מ"ל (Invitrogen) ולהעביר צינורות microfuge 1.5 מ"ל.
- לשטוף חרוזים 3 פעמים מ"ל 1 של חיץ לשטוף IP.
- הוסף RNaseT1 (Fermentas, 10,000 U / μl) לריכוז סופי של U 100 / μl ו דגירה ההשעיה חרוז באמבט מים במשך 15 דקות על 22 ° C. לאחר מכן מצננים על הקרחבמשך 5 דקות.
- לשטוף חרוזים 3 פעמים מ"ל 1 של חיץ גבוה מלח לשטוף.
- חרוזים Resuspend בנפח 1 של חיץ dephosphorylation
- הוסף phosphatase מעיים עגל אלקליין (CIAP, חוד) לריכוז סופי של 0.5 U / μl, ו דגירה ההשעיה על 10 דקות בשעה 37 ° C.
- לשטוף חרוזים פעמיים מ"ל 1 של חיץ לשטוף phosphatase
- לשטוף חרוזים פעמיים חיץ polynucleotide קינאז (PNK) ללא DTT (ריכוז DTT הכרחי התגובה האנזימטית גבוה מספיק כדי לגרום נזק חרוזים מגנטיים).
- חרוזים Resuspend בכרך אחד חרוז המקורי של PNK חיץ
Radiolabeling קטעי RNA crosslinked לחלבונים immunoprecipitated
- כדי השעיית חרוז שתואר לעיל, להוסיף Υ-32-P-ATP לריכוז סופי של PNK μCi / μl 0.5 ו-T4 (חוד) עד 1 U / μl בכרך אחד חרוז המקורי. דגירה ההשעיה למשך 30 דקות ב 37 ° C.
- הוסף הלא רדיואקטיבי ATP לקבל ריכוז סופי של 100 מיקרומטר ו דגירה של 5 דקות אחר על 37 ° C.
- שטפו את חרוזים מגנטיים 5 פעמים עם 800 μl של PNK חיץ ללא DTT.
- Resuspend את החרוזים μl 70 של חיץ SDS-PAGE הטעינה.
SDS-PAGE ו electroelution של RNA-חלבון מתחמי crosslinked מן פרוסות ג'ל
- לדגור על השעיית radiolabeled 5 דקות בתוך גוש חום 95 מעלות צלזיוס עד לפגל ולשחרר את RBP immunoprecipitated עם RNA crosslinked ו מערבולת.
- הסר את חרוזים מגנטיים על מפריד ולהעביר את supernatant אל צינור נקי מ"ל 1.5 microfuge.
- טען 40 μl של supernatant לכל היטב Novex Bis-טריס 4-12% (Invitrogen) ג'ל precast polyacrylamide ולהפעיל את ג'ל 55 דקות ב 200 V.
- לפרק את תא ג'ל ולפרק את הג'ל, והשאיר אותו רכוב על צלחת אחת. כדי להקל על יישור של הג'ל על תדפיס הנייר phosphorimager, אנו ממליצים על השתלת שלוש חתיכות זעירות ג'ל רדיואקטיבי סימטרית על שלושה מתוך ארבעת הפינות של הג'ל. חלקים רדיואקטיביים ג'ל ניתן שנאספו ג'לים אשר שימשו בעבר כדי לטהר oligonucleotides סינתטי radiolabeled. עטפו את ג'ל סרט פלסטי (למשל ניילון נצמד), כדי למנוע זיהום.
- לחשוף את הג'ל למסך phosphorimager אטומים עבור h 1 ולדמיין על phosphorimager.
- יישר את הג'ל על גבי התדפיס phosphorimager באמצעות חתיכות ג'ל מושתל להתמצאות. חותכים את רצועות המתאימים בגודלם הצפוי של RBP (IGF2BP1, כ. 75 KDA) והעברה Tube D-Tube Midi Dialyzer ולהוסיף 800 חיץ μl 1x SDS פועל.
- Electroelute crosslinked RNA-RBP מורכבים 1x SDS ריצה חוצץ ב V 100 ל 2 ח נכרת מן הג'ל ו electroeluted ב D-Tube Dialyzer Midi (Novagen) במאגר 800 SDS μl פועל על פי ההוראות של היצרן.
Proteinase העיכול K
- הוסף נפח שווה של הצפת 2x K proteinase ביחס electroeluate, ואחריו תוספת של proteinase K (Roche) לריכוז סופי של 1.2 מ"ג / מ"ל. דגירה למשך 30 דקות ב 55 ° C.
- שחזר את הרנ"א על ידי חומצי פנול / כלורופורם / רשות העתיקות החילוץ (25:24:1, pH 4.0) ואחריו החילוץ כלורופורם. הוסף 1 μl של גליקוגן (10 מ"ג / מ"ל המלאי) לזרז את הרנ"א על ידי הוספת 3 כרכים של אתנול. ממיסים את גלולה במים μl 10.5.
ספריית הכנה cDNA וסדר עמוק
קח את הרנ"א התאושש באמצעות פרוטוקול סטנדרטי ספריית cDNA הכנה תיאר במקור עבור שיבוט של RNAs הרגולציה קטן 19. הצעד הראשון, קשירת מתאם '3, בוצע כפי שתואר על סולם של 20 μl 10.5 באמצעות μl של ה-RNA התאושש. השתמש רצף Solexa מתאם מערכות תיאר. בהתאם לכמות של רנ"א התאושש, 5'-מתאם-3'-מתאם מוצרים ללא מוסיף עשוי להתגלות אחרי הגברה של cDNA להקות נוספות כמו PCR. במקרים כאלה, בלו המוצר PCR יותר של גודל הצפויה NuSieve 3% נקודת התכה נמוכה agarose ג'ל, elute המוצר PCR מן חתיכות ג'ל באמצעות ערכת GelElute (Qiagen) ורצף באמצעות טכנולוגיית Solexa. אחת Solexa רצף לרוץ בדרך כלל מאפשר רצף בין 6 ו - 10,000,000 נכתב כי הם מספיק לכיסוי רחב transcriptome של אתרי הקישור של חלבונים RNA מחייב.
Bioinformatic ניתוח
ניתוח bioinformatic זהירות של רצף קורא צריך לעשות כדי להשיג תובנות משמעותיות לתוך האתרים RNA ומחייבת את RBP שנבדקו, כגון אלמנט ההכרה RNA, באזורים מחייב המועדפת RBP יש (exonic לעומת intronic, קידוד רצף לעומת מתורגמים רצף). רצף קורא צריך להיות מתואם מול מסדי נתונים הגנום EST. אנחנו בדרך כלל להשתמש קורא mapping ייחודי בגנום עם עד הכניסה one אי התאמה, או מחיקה לבנות אשכולות של רצף נכתב כי אז יכול להיות מנותח נוספת. תדירות של מוטציות מאפיין את רצף באשכולות קורא, T למעברים C בעת שימוש 4-SU ו-G על מעברים בעת שימוש 6-SG, מעידים על רצפים crosslinked בהצלחה. מניסיוננו RNAs uncrosslinked שכותרתו עם 4-SU להראות מוטציה רקע שיעור של 20%. שיעור זה עולה ל כ. 50-80% על crosslinking.
תיאור מפורט של ניתוח bioinformatic ניתן למצוא חומר משלים לפרסום על ידי Hafner et al. 18
אופציונלי שלבים
קביעת רמות שילוב של 4-RNA thiouridine לתוך מסך
RNA בודד הכולל מקו ביציבות התא המבטא את RBP עניין לאחר גידול בינוני בתוספת 100 מיקרומטר 4SU 16 שעות לפני הקציר. כמו שליטה, תאים הקציר גדל ללא תוספת 4SU. RNA בודד הכולל על ידי תוספת של 3 כרכים של מגיב Trizol (Sigma) כדי לשטוף את כדורי תא ההנחיות של היצרן. בהמשך היה לטהר RNA סך באמצעות Qiagen RNeasy על פי פרוטוקול של היצרן. כדי למנוע חמצון של 4SU במהלך בידוד RNA וניתוח, להוסיף 0.1 מ"מ dithiothreitol (DTT) של מאגרים לרחוץ צעדים האנזימטית הבאים. תקציר ו-RNA סך dephosphorylated כדי נוקלאוזידים יחיד לניתוח HPLC כפי שתואר קודם לכן 20. בקצרה, בנפח 30 μl, דגירה 40 מיקרוגרם של מטוהרים RNA סה"כ היו 16 שעות ב 37 ° C עם phosphatase U 0.4 אלקליין חיידקי (וורטינגטון ביוכימיים) ו - 0.09 U ארס הנחש phosphodiesterase (וורטינגטון ביוכימיים). כסטנדרט התייחסות, להשתמש RNA 4SU שכותרתו סינתטיים, (אנחנו standardly להשתמש CGUACGCGGAAUACUUCGA (4SU) U) וגם נושא זה כדי להשלים עיכול אנזימטי. הפרד את תערובות כתוצאה של ribonucleosides ידי HPLC על גילוי Supelco C18 (סיליקה מלוכדות החלקיקים שלב 5 מיקרומטר, 250 x 4.6 מ"מ) טור שלב הפוכה (Bellefonte הרשות הפלסטינית, ארה"ב). מאגרים HPLC הם 0.1 M TEAA ב אצטוניטריל 3% (א) ו אצטוניטריל 90% במים (B). השתמש שיפוע isocratic: 0 B% במשך 15 דקות, 0-10-B% עבור 20 דקות, 10-100-B% למשך 30 דקות. החלת 5% ב 100 דקות לשטוף מיושם בין פועל כדי לנקות את הטור HPLC.
נציג תוצאות
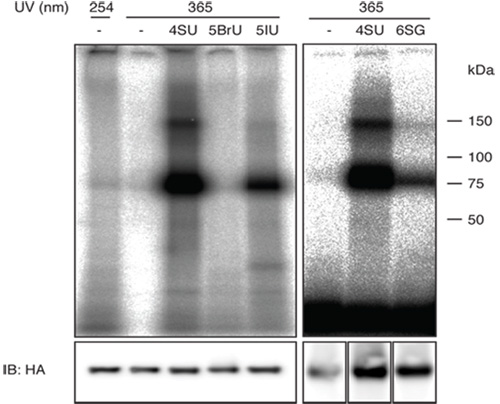
איור 1 (פאנל מימין) מראה תוצאה נציג של קליפ-PAR הופיע עם שורות תאים להביע את הדגל / HA-tagged IGF2BP1 עם 4-SU ו - 6-SG. ראוי לציין, כי יעילות crosslinking של 6-SG עבור IGF2BP1 נמוך יותר יעילות crosslinking עבור 4-SU. Crosslinking יעילות נמוכה תגרום רקע גבוהה יותר של רצפים נגזר שברי RNAs הסלולר בשפע ולכן כדאי לשקול קנה המידה את הניסוי בעת שימוש נוקלאוזידים photoreactive פחות יעיל.
הפאנל השמאלי של איור 1 מציג השוואה של שימוש שונה אנלוגים uridine photoreactive שיכול לשמש פוטנציאל עבור קליפ-PAR לעומת crosslinking המסורתית UV 254 ננומטר.
האינטנסיביות של הלהקה רדיואקטיבי באורך הנכון נותן לך רעיון טוב אם הניסוי PAR-Clip עבד ויש לך מבודדים רנ"א מספיק לבצע דרך קטן RNA רצף פרוטוקול (צעד אחר צעד תיאור להכנת ספריית cDNA של קטן רצף RNAs ניתן למצוא 19). תדירות של מוטציות אופייני קורא רצף, T למעברים C בעת שימוש 4-SU ו-G על מעברים בעת שימוש 6-SG, מעידים על רצפים crosslinked בהצלחה. מניסיוננו RNAs uncrosslinked שכותרתו עם 4-SU להראות מוטציה רקע שיעור של 20%. שיעור זה עולה ל כ. 50-80% על crosslinking.
Disclosures
Acknowledgements
אנו מודים לחברי במעבדה Tuschl לדיונים מועילים. MH נתמך על ידי Akademischer דויטשר Austauschdienst (DAAD). עבודה זו נתמכה על ידי הקרן הלאומית השוויצרית גרנט # 3100A0 114,001 ל-MZ: TT הוא חוקר HHMI, ולעבוד במעבדה שלו נתמך על ידי NIH מענקים GM073047 ו MH08442 וקרן סטאר.
Materials
Buffers ו ריאגנטים
מדיום גידול תאים HEK293
- DMEM
- 10% FBS
- 2 מ"מ L-גלוטמין
- 100 U / ml פניצילין
- 100 U / ml סטרפטומיצין
- 100 מיקרוגרם / מ"ל hygromycin
- 15 מיקרוגרם / מ"ל blasticidin
4-thiouridine פתרון מניות (1 ז)
- 260.27 מ"ג 4-thiouridine
- 1 מ"ל DMSO
Doxycyclin המניות (10 מ"ג / מ"ל)
- 10 מ"ג doxycyclin
- 1 מ"ל DMSO
1x NP40 חיץ תמוגה
הכינו מלאי של חיץ 5x ללא DTT ו מעכבי פרוטאז. הוסף DTT ו מעכבי פרוטאז ישירות לפני הניסוי.
- HEPES 50 מ"מ, ה-pH 7.5
- 150 מ"מ KCl
- 2 mM EDTA
- 1 mM NAF
- 0.5% (V / V) NP40
- 0.5 mM DTT
- מעכב להשלים EDTA ללא קוקטייל פרוטאז (Roche)
ציטראט פוספט-חיץ
- 4.7 גר '/ ליטר חומצת לימון
- 9.2 גר '/ ל Na 2 HPO 4
- pH 5.0
ה-IP לשטוף חיץ
- 50 mM HEPES-KOH, pH 7.5
- 300 mM KCl
- 0.05% (v / v) NP40
- 0.5 mM DTT (להוסיף ישירות לפני הניסוי)
- מעכב להשלים EDTA ללא קוקטייל פרוטאז (Roche) (להוסיף ישירות לפני הניסוי)
High-מלח לשטוף חיץ
- 50 mM HEPES-KOH, pH 7.5
- 500 mM KCl
- 0.05% (v / v) NP40
- 0.5 mM DTT (להוסיף ישירות לפני הניסוי)
- מעכב להשלים EDTA ללא קוקטייל פרוטאז (Roche) (להוסיף ישירות לפני הניסוי)
Dephosphorylation חיץ
- 50 מ"מ טריס-HCl, pH 7.9
- 100 mM NaCl
- 10 mM MgCl 2
- 1 mM DTT
Phosphatase חיץ לשטוף
- 50 מ"מ טריס-HCl, pH 7.5
- 20 מ"מ EGTA
- 0.5% (V / V) NP40
קינאז Polynucleotide (PNK) ללא חיץ DTT
- 50 מ"מ טריס-HCl, pH 7.5
- 50 mM NaCl
- 10 mM MgCl 2
PNK חיץ
- 50 מ"מ טריס-HCl, pH 7.5
- 50 mM NaCl
- 10 mM MgCl 2
- 5 מ"מ DTT
SDS PAGE חיץ העמסה
- גליצרול 10% (V / V)
- 50 מ"מ טריס-HCl, pH 6.8
- 2 mM EDTA
- 2% SDS (w / v)
- 100 מ"מ DTT
- 0.1% כחול bromophenol
2x proteinase K חיץ
- 100 מ"מ טריס-HCl, pH 7.5
- 150 mM NaCl
- 12.5 mM EDTA
- 2% (w / v) SDS
References
- Keene, J. D. RNA regulons: coordination of post-transcriptional events. Nat. Rev. Genet. 8 (7), 533-533 (2007).
- Tenenbaum, S. A. Identifying mRNA subsets in messenger ribonucleoprotein complexes by using cDNA arrays. Proc. Nat. Acad. Sci. 97 (26), 14085-14085 (2000).
- Gerber, A. P. Genome-wide identification of mRNAs associated with the translational regulator PUMILIO in Drosophila melanogaster. Proc. Nat. Acad. Sci. 103 (12), 4487-4487 (2006).
- Lopez de Silanes, I. Identification of a target RNA motif for RNA-binding protein HuR. Proc. Nat. Acad. Sci. 101 (9), 2987-2987 (2004).
- Greenberg, J. R. Ultraviolet light-induced crosslinking of mRNA to proteins. Nucl. Acids Res. 6 (2), 715-715 (1979).
- Wagenmakers, A. J. Cross-linking of mRNA to proteins by irradiation of intact cells with ultraviolet light. Eur. J. Biochem. 112 (2), 323-323 (1980).
- Mayrand, S. Structure of nuclear ribonucleoprotein: identification of proteins in contact with poly(A)+ heterogeneous nuclear RNA in living HeLa cells. The Journal of Cell Biology. 90 (2), 380-380 (1981).
- Dreyfuss, G. Characterization of heterogeneous nuclear RNA-protein complexes in vivo with monoclonal antibodies. Mol. Cell. Biol. 4 (6), 1104-11 (1984).
- Adam, S. A., Dreyfuss, G. Adenovirus proteins associated with mRNA and hnRNA in infected HeLa cells. J. Virol. 61 (10), 3276-3276 (1987).
- Ule, J. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1212 (2003).
- Licatalosi, D. D. HITS-CLIP yields genome-wide insights into brain alternative RNA processing. Nature. 456 (7221), 464-464 (2008).
- Yeo, G. W. An RNA code for the FOX2 splicing regulator revealed by mapping RNA-protein interactions in stem cells. Nat. Struct. Mol. Biol. 16 (2), 130-130 (2009).
- Sanford, J. R. Splicing factor SFRS1 recognizes a functionally diverse landscape of RNA transcripts. Genome Res. 19 (3), 381-381 (2009).
- Granneman, S. Identification of protein binding sites on U3 snoRNA and pre-rRNA by UV cross-linking and high-throughput analysis of cDNAs. Proc. Nat. Acad. Sci. , (2009).
- Guil, S., Caceres, J. F. The multifunctional RNA-binding protein hnRNP A1 is required for processing of miR-18a. Nat. Struct. Mol. Biol. 14 (7), 591-591 (2007).
- Chi, S. W. Argonaute HITS-CLIP decodes microRNA-mRNA interaction maps. Nature. 460 (7254), 479-479 (2009).
- Zisoulis, D. G. Comprehensive discovery of endogenous Argonaute binding sites in Caenorhabditis elegans. Nat. Struct. Mol. Biol. , (2010).
- Hafner, M. Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP. Cell. , (2010).
- Hafner, M. Identification of microRNAs and other small regulatory RNAs using cDNA library sequencing. Methods. 44 (1), 3-3 (2008).
- Andrus, A., Kuimelis, R. G. Base composition analysis of nucleosides using HPLC. Current Protocols in Nucleic Acid Chemistry. Chapter 10 (Unit 10.6), (2001).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved