A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מסך התפוקה גבוהה עבור Biomining פעילות cellulase מספריות Metagenomic
In This Article
Summary
פרוטוקול זה מתאר מסך התפוקה גבוהה לפעילות cellulolytic מספריית metagenomic לידי ביטוי Escherichia coli. המסך הוא פתרון מבוסס מאוד אוטומטיות, ומשתמש אחד סיר כימיה 384 microplates היטב עם readout הסופי כמו מדידת ספיגת.
Abstract
תאית, המקור הנפוץ ביותר של פחמן אורגני על פני כדור הארץ, יש טווח רחב יישומים תעשייתיים עם דגש הולך וגובר על ייצור דלק ביולוגי 1. שיטות כימיות לשנות או לבזות תאית בדרך כלל דורשים חומצות חזקות וטמפרטורות גבוהות. ככזה, שיטות אנזימטיות הפכו בולטים בתהליך bioconversion. בעוד זיהוי cellulases פעיל מן מבודד חיידקים ופטריות הייתה יעילה למדי, הרוב המכריע של חיידקים בטבע להתנגד טיפוח מעבדה. הגנומי הסביבה, הידוע גם גישות, הקרנת metagenomic יש הבטחה גדולה לגשר על הפער טיפוח בחיפוש אחר אנזימים bioconversion רומן. גישות ההקרנה Metagenomic התאוששו בהצלחה cellulases רומן מסביבות מגוונות כגון 2 קרקעות, תאו כרס פנימית 3 ו טרמיטים האחוריות בטן 4 משתמש (CMC) carboxymethylcellulose צלחות אגר מוכתם קונגו צבע אדום (מבוסס על שיטת Teather ועץ 5). עם זאת, השיטה CMC היא מוגבלת התפוקה, אינו מתבטא כמותית אות נמוך יחס רעש 6. שיטות אחרות דווחו 7,8 אבל כל אחד להשתמש בצלחת אגר מבוססי assay, אשר אינה רצויה להקרנה תפוקה גבוהה של ספריות גנטי גדול להוסיף. כאן אנו מציגים פתרון המבוסס על המסך לפעילות cellulase באמצעות dinitrophenol chromogenic (DNP)-cellobioside המצע 9. הספרייה שלנו משובטים לתוך השליטה pCC1 את העותק fosmid כדי להגביר את הרגישות assay באמצעות אינדוקציה עותק מספר 10. השיטה משתמשת בסיר אחד הכימיה-384 גם microplates עם readout הסופי כאמור מדידת ספיגת. Readout זה כמותי, רגיש אוטומטית עם תפוקה של עד 384-100x צלחות טוב ליום באמצעות המטפל נוזלי קורא צלחת עם מערכת לערום המצורפת.
Protocol
לפני שמתחילים בפרוטוקול זה, יהיה עליך בספרייה metagenomic שלך מאוחסנים בתבנית 384 צלחת היטב. במחקר שלנו, השתמשנו pCC1 לשלוט עותק וקטור fosmid בשילוב עם TransforMax phage T1 עמידים EPI300 T1-R א coli תאים כמארח הספרייה ומאוחסנים הצלחות שלנו ב -80 ° C 11.
1. שכפול של פלטות ספריית Metagenomic
- הפשירו צלחות המכיל את הספרייה ב 37 מעלות צלזיוס למשך כ 20 דקות, או עד בארות כל מופשר.
- השתמש בתכונת אור UV לחיטוי על qPix2 כדי לעקר את הרובוט במשך 15 דקות.
- הכינו מרק LB עם chloramphenicol בריכוז הסופי של 12.5ug/mL ו arabinose ב 100ug/mL בבקבוק 500ml מגיב. כל צלחת ישתמשו 20mL כ, בתוספת לבצע 50 מ"ל נוספים כדי לאפשר נפח מת.
- הגדרת qFill3 כפי הוראות היצרן לכל עם בקבוק התקשורת מחוברת סעפת דרך צינורות הסטרילי. תוכנית זאת עבור הסכום המתאים של מדיה, ולהגדיר את זה כדי למלא נפח 45uL היטב כל אחד.
- טהר את האוויר ואת צינורות סעפת באמצעות התכונה לטהר של הרובוט עד המדיה גלוי בא להצמיד לכל אחד סעפת.
- מלאו את המספר הרצוי של צלחות עם התקשורת LB באמצעות qFill3. צלחת לכל אורך כ 20 שניות כדי למלא.
- טען את צלחות ספריה צלחות טרי לאזורים המתאימים של הרובוט qPix2. מלאו את אמבטיות ניקוי עם ריאגנטים מתאימה; 2% Micro90 באמבטיה האחורי, autoclaved מים מזוקקים באמבטיה באמצע, 80% אתנול באמבטיה הקדמי.
- השתמש בתוכנית "שכפול" של qPix2. בשנת תוכנה, בחר את הראש המתאים, מספר סוגים של פלטות מקור וצלחות היעד. גם הקים את הראש כדי לנקות בין משוכפלים, בדרך כלל 6 מחזורים באמבטיה כל זה מספיק. קיבולת של המכונה הוא 10 צלחות בכל פעם, וזה לוקח בערך 15 דקות כדי לשכפל את כולם עם ראש סיכה 384 (או כ -50 דקות עם ראש סיכה 96).
הערה: אין אפשרות "לבחוש מקור" או "מערבבים יעד". מומלץ לא להשתמש באפשרויות אלה כבעיות התרחשו בעבר עם הרובוט שלנו להשתמש בהם. - לאחר הצלחות משוכפלים, לגדל את הצלחות על 37 מעלות צלזיוס למשך 24 שעות בתוך קופסת לחות. כי אנחנו גרימת מספר עותק גבוהה של fosmid עם תוספת של arabinose, שיבוטים נוטים לגדול לאט יותר מאשר אי - המושרה שיבוטים. החזר את הצלחות הספריה -80 ° C.
הערה: דגירה בקופסת לחות מבטיח אידוי של התקשורת אינה מתרחשת בארות בשולי הצלחות, שמירה על סביבה אחידה עבור כל בארות. - נקו את הרובוט qFill3 ידי הראשון טיהור אותו עם מים (~ 50 מ"ל) ולאחר מכן אתנול 80% (~ 50 מ"ל). לפרק את הרכיבים, ונקי החיטוי את הבקבוק, מכסה, צינורות עטופים בנייר אלומיניום.
2. מדידת צמיחה של E. coli המשובטים
- הסרת צלחות מ 37 ° C חממה. הסר להפריש את המכסים, וצלחות מקום על גבי פלטפורמת המגזין טוען המסופק עם RapidStak, עד מקסימום של 25 צלחות.
הערה: צלחת בתחתית הערימה יהיה הראשון לקרוא. הקפד לעקוב אחר הסדר של הצלחות, כמו התוכנה אינה רשומה זו. - הסר את מחסנית אחת מן Stak המהירה, ודא הוא ריק, ו לדחוף אותו על ערימת צלחות לקרוא. תפוס אותו בידית, ולוחות צריכים להיות טעון לתוך המגזין. טען את המחסנית לתוך RapidStak.
הערה: מגזין יש בורג קטן בפינה המציין אשר פינה את הבארות A1 של הצלחות צריך ללכת. המגזין רק הר של RapidStak בכיוון אחד. - פתח את התוכנית RE SkanIt במחשב המחובר למכונות. בחר "הפעלה חדשה" בשם כראוי. בחר "קורנינג קרקעית שטוחה-384 גם צלחת" כסוג צלחת.
- באזור "פריסה פלייט", בחר את "אשף" כפתור, לבחור בו כדי להוסיף 384 נעלמים אל הצלחת שלך.
- באזור "פרוטוקול", בחרו באפשרות "ובכן Loop", והזן עבור 384 בארות. סמל "ובכן Loop" יופיעו עץ הזרימה בצד שמאל של המסך. בחר את הסמל ולחץ כדי להוסיף "מדידה photometric". הגדר אותו לקרוא 600nm.
- שמור פרוטוקול שלך עם שם ייחודי, לסגור את התוכנית RE SkanIt.
- במחשב, פתח את התוכנית PolaraRS.
- בעמוד הראשי תהיה טבלה המפרטת את המכשירים המחוברים למכשיר RapidStak. VarioSkan צריך להיות בצד השמאלי של השולחן הזה. תחת הכותרת VarioSkan, יהיו שני קישורים: "RunSession" ו "לדגור". לחץ על האפשרות "RunSession".
- חלון חדש, החלון assay, יופיעו עם זרימה באמצע. בחר את "RunSession" פריט (זה צריך להיות הפריט הנוכחי בלבד). בצד ימין של המסך התפריט הנפתחיופיע. מצא את שמו של רי ששמרת SkanIt להפעיל בתפריט זה.
הערה: נראה שיש לא מזמין רציונלי לתפריט הזה. זה לא אלפביתי, או לפי תאריך נוסף, או הקשורים בו במחשב פרוטוקול נשמר. זה אומר שאתה צריך להסתכל דרך כל הפרוטוקולים לפני מציאת שלך, וזה כאב. - לאחר שבחרת פרוטוקול שלך, בפינה הימנית העליונה לחץ על "הפעל assay".
- תיבת יצוץ, תתבקש לציין אשר מגזין להשתמש בו כמקור, ואשר מגזין ריקה. תיבת נמוך יותר על המסך המתאים מגזין הקדמי. [אופציונלי: הוא מבקש ממך להזין את מספר צלחות עמוסות לקבוע אומדן בסכום של זמן זה ייקח. אומדן זה הוא בדרך כלל רחוק.]
- ודאו גם RapidStak ואת VarioSkan מופעלים, ולחץ על "אישור".
- RapidStak יטען אוטומטית צלחות שלך לתוך קורא את הצלחת, המאפשר מדידה רציפה של הצלחות. כדי לקרוא את 25 צלחות לוקח כ 50 דקות.
הערה: מומלץ לקיים את הטעינה הראשונה לתוך צלחת VarioSkan כדי להבטיח תיאום נאות של המכונות. אם RapidStak לא להעמיס על הקורא צלחת כראוי, להשתמש "זה assay השהה" כפתור בפינה הימנית העליונה של חלון PolaraRS. - לאחר VarioSkan נעשה לקרוא את כל הצלחות, להסיר את המגזין מלא ומניחים אותו על משטח הטעינה המגזין. הרימו את המלבן החיצוני של הפלטפורמה, ומגזין צריך להחליק משם, משאיר את הלוחות עומדים בערימה באמצע. החלף את המכסים על צלחות המתאים.
3. הוספת מערבבים את Assay על כל צלחת
- הכינו תערובת assay באמצעות מניות 10x premade של לערבב תמוגה (10% Triton X-100, RIS 100mm, 10mm EDTA) במאגר 50mm אצטט אשלגן ב-pH 5.5. הכינו מלאי של עד המצע DNP-cellobioside 75mg/mL ב DMSO, מוודא את המצע נמס לגמרי. הוסף DNP-cellobioside פתרון מניות לערבב assay לריכוז סופי של 0.1mg/mL. כל צלחת ישתמשו כ 20mL של פתרון, ובנוסף לעשות 50 מ"ל נוספים כדי לאפשר נפח מת.
הערה: DNP-Cellobioside אינו מסיס בקלות במים, כך מתמוססת DMSO מגביר את מסיסות. הנוכחות של DMSO בפתרון הסופי assay אין תופעות נצפות. - הגדרת qFill3 כפי הוראות היצרן לכל עם בקבוק התקשורת מחוברת סעפת דרך צינורות הסטרילי. תוכנית זאת עבור הסכום המתאים של מדיה, ולהגדיר את זה כדי למלא נפח 45uL היטב כל אחד.
- טהר את האוויר ואת צינורות סעפת באמצעות התכונה לטהר של הרובוט עד המדיה גלוי בא להצמיד לכל אחד סעפת.
- הוסף לערבב assay אל כל צלחת באמצעות qFill3. צלחת לכל אורך כ 20 שניות כדי למלא.
- לאחר לערבב assay הוא הוסיף, דגירה צלחות ב 37 ° C בקופסת לחות במשך 12-16 שעות.
הערה: Over-הדגירה של צלחות פעם לערבב את assay נוסף יגרום אידוי של בארות החיצוני של הצלחת, מניב קריאות ספיגת גבוה יותר מאשר לראות את הבארות בפנים. זה יכול להיות מקוזז על ידי דוגרים צלחות בתא לחות.
4. ספיגת קריאה של המשובטים assayed
- הסרת צלחות מ 37 ° C חממה. הסר להפריש את המכסים, וצלחות מקום על גבי פלטפורמת המגזין טוען המסופק עם RapidStak, עד מקסימום של 25 צלחות.
הערה: צלחת בתחתית הערימה יהיה הראשון לקרוא. הקפד לעקוב אחר הסדר של הצלחות, כמו התוכנה אינה רשומה זו. - הסר את מחסנית אחת מן Stak המהירה, ודא הוא ריק, ו לדחוף אותו על ערימת צלחות לקרוא. תפוס אותו בידית, ולוחות צריכים להיות טעון לתוך המגזין. טען את המחסנית לתוך RapidStak.
הערה: מגזין יש בורג קטן בפינה המציין אשר פינה את הבארות A1 של הצלחות צריך ללכת. המגזין רק הר של RapidStak בכיוון אחד. - פתח את התוכנית RE SkanIt במחשב המחובר למכונות. בחר "הפעלה חדשה" בשם כראוי. בחר "קורנינג קרקעית שטוחה-384 גם צלחת" כסוג צלחת.
- באזור "פריסה פלייט", בחר את "אשף" כפתור, לבחור בו כדי להוסיף 384 נעלמים אל הצלחת שלך.
- באזור "פרוטוקול", בחרו באפשרות "ובכן Loop", והזן עבור 384 בארות. סמל "ובכן Loop" יופיעו עץ הזרימה בצד שמאל של המסך. בחר את הסמל ולחץ כדי להוסיף "מדידה photometric". הגדר אותו לקרוא 400nm.
- שמור פרוטוקול שלך עם שם ייחודי, לסגור את התוכנית RE SkanIt.
- במחשב, פתח את התוכנית PolaraRS.
- בעמוד הראשי תהיה טבלה המפרטת את המכשירים המחוברים למכשיר RapidStak. VarioSkan צריך להיות בצד השמאלי של השולחן הזה. תחת הכותרת VarioSkan, יהיו שני קישורים: "RunSession" ו "Incubatדואר ". לחץ על" "אפשרות RunSession.
- חלון חדש, החלון assay, יופיעו עם זרימה באמצע. בחר את "RunSession" פריט (זה צריך להיות הפריט הנוכחי בלבד). בצד ימין של המסך התפריט הנפתח תופיע. מצא את שמו של רי ששמרת SkanIt להפעיל בתפריט זה.
- לאחר שבחרת פרוטוקול שלך, בפינה הימנית העליונה לחץ על "הפעל assay".
- תיבת יצוץ, תתבקש לציין אשר מגזין להשתמש בו כמקור, ואשר מגזין ריקה. תיבת נמוך יותר על המסך המתאים מגזין הקדמי. [אופציונלי: הוא מבקש ממך להזין את מספר צלחות עמוסות לקבוע אומדן בסכום של זמן זה ייקח. אומדן זה הוא בדרך כלל רחוק.]
- ודאו גם RapidStak ואת VarioSkan מופעלים, ולחץ על "אישור"
- RapidStak יטען אוטומטית צלחות שלך לתוך קורא את הצלחת, המאפשר מדידה רציפה של הצלחות.
הערה: מומלץ לקיים את הטעינה הראשונה לתוך צלחת VarioSkan כדי להבטיח תיאום נאות של המכונות. אם RapidStak לא להעמיס על הקורא צלחת כראוי, להשתמש "זה assay השהה" כפתור בפינה הימנית העליונה של חלון PolaraRS. - לאחר VarioSkan נעשה לקרוא את כל הצלחות, להסיר את המגזין מלא ומניחים אותו על משטח הטעינה המגזין. הרימו את המלבן החיצוני של הפלטפורמה, ומגזין צריך להחליק משם, משאיר את הלוחות עומדים בערימה באמצע. הצלחות יכול להיות מסולק על פי נהלי המעבדה שלך.
- כדי לייצא את ספיגת קריאות, לפתוח תוכנה SkanIt רי, ולבחור "פתח קובץ קיים".
- בחר את הספרייה בה שמרת ההפעלה שלך, פגע "+" סמל כדי להרחיב את הרשימה תחת הכותרת הכותרת יהיו אפשרויות שכותרתו "מיכל 1" ל "מיכל N" בהתאם למספר צלחות. בחר את הצלחת הראשונה להיות מנותח.
- בשורת התפריטים בחלקו העליון של הדף, בחר "עיבוד נתונים> דווח / ייצוא"
- בחר את האפשרויות הרצויות לייצוא.
הערה: אני בדרך כלל לבחור "פריסה פלייט" ו "photometric" הנתונים לייצוא. - לחץ על "הצג דוח" ולאחר מכן "File> Save". בחר את הספרייה הרצויה. תבנית הפלט הוא גיליון אלקטרוני של Microsoft Excel
5. נציג תוצאות
דוגמה קריאות ספיגת מהצלחת 384 יחיד היטב המכיל שיבוט חיובי מוצג באיור 2. שיבוטים חיובית להראות עלייה ניכרת ספיגת פני אלה שלא להביע את פעילות cellulase. הבדלים זמן assay, מיקום טוב על הצלחת, או ריכוז DNP (ניתן הציג ידי סינון בלתי מומס DNP) יכולה להשפיע על ספיגת קריאות מוחלטת. קריאות ספיגת יחסית, כגון ההבדל ספיגת מעל הממוצע צלחת או ממוצע העמודה, הם שיטה חזקים יותר לזהות שיבוטים חיובי cellulase.
בעקבות זיהוי של שיבוטים חיובי מהצלחות ספריה, מומלץ לשכפל כל שיבוטים חיובית לתוך צלחת חדשה לסינון משנית. זה מבטל תופעות הנובעות מיקום טוב או וריאציה צלחת ומאפשר השוואה ישירה בין שיבוטים יותר חיובי.

באיור 1. תרשים זרימה של assay תפוקה גבוהה עבור ספריית metagenomic משובטים לתוך E. coli ומאוחסנים ב -80 ° C.
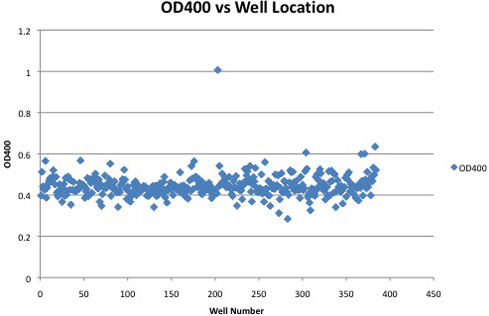
איור 2. ספיגת קריאות מתוך צלחת אחת 384-clone המכיל גם חיובית. שיבוטים חיובי cellulase ניתן לזהות על ידי ספיגת גדל באופן משמעותי על שיבוטים שלילית.
Discussion
מסך התפוקה גבוהה לגילוי המהיר של הפעילות cellulolytic מתוך ספרייה גדולה להכניס DNA הגנומי metagenomic לידי ביטוי E. coli מתואר בפרוטוקול זה. שיטה זו מהווה שיפור של assay האדום CMC / קונגו נפוץ בספרות. זהו פתרון המבוסס, ומאפשר הקרנה חד סיר הכימיה-384 גם צלחות, עם הפלט הסופי כמו קריאות ספ...
Disclosures
Acknowledgements
המחברים מבקשים להודות ד"ר סטיב Withers ו הונג מינג חן למתן DNP-Cellobioside המצע.
Materials
| Name | Company | Catalog Number | Comments |
| qPix2 | Genetix | With 384-pin gridding head | |
| qFill3 | Genetix | With 384-well manifold | |
| Varioskan | Thermo Fisher Scientific, Inc. | ||
| RapidStak | Thermo Fisher Scientific, Inc. | Connected to Varioskan | |
| Micro90 Detergent | Cole-Parmer | 18100-00 | Diluted to 2% in water |
| Ethanol | Major Lab Supplier | Diluted to 80% in water | |
| Chloramphenicol | Sigma-Aldrich | C0378 | 12.5mg/mL in ethanol |
| LB broth, Miller | Fisher Scientific | BP1426-2 | 25g/L, autoclaved |
| 384-well flat bottom plates | Corning | 3680 | |
| L-(+)-Arabinose | Sigma-Aldrich | A3256 | 100mg/mL in water |
| Potassium Acetate | Fisher Scientific | P171 | 50mM in water, autoclaved, adjusted to pH 5.5 with HCl |
| Triton X-100 | Fisher Scientific | BP151 | |
| Trizma hydrochloride | Sigma-Aldrich | T3253 | In TE buffer solution, 100mM |
| EDTA disodium salt | Sigma-Aldrich | E5134 | In TE buffer solution, 10mM |
| 2,4-dinitrophenyl cellobioside | Provided by Dr. Steve Withers, UBC | ||
| Dimethyl Sulfoxide | Sigma-Aldrich | D8418 |
References
- Rubin, E. M. Genomics of cellulosic biofuels. Nature. 454, 841-845 (2008).
- Voget, S., Steele, H. L., Streit, W. R. Characterization of a metagenome derived halotolerant cellulase. J. Biotechnol. 126, 26-36 (2006).
- Duan, C. J. Isolation and partial characterization of novel genes encoding acidic cellulases from metagenomes of buffalo rumens. J. Appl. Microbiol. 107, 245-256 (2009).
- Zhang, Y. H. P. Outlook for cellulase improvement: Screening and selection strategies. Biotech. Advances. 24, 452-481 (2006).
- Teather, R. M., Wood, P. J. Use of congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen. Appl. And Environ. Microbiol. 43, 777-780 (1982).
- Sharrock, K. R. Cellulase assay methods: a review. J. Biochem and Biophys Methods. 17, 81-106 (1988).
- Kasana, R. C., Salwan, R., Dhar, H., Dutt, S., Gulati, A. A rapid and easy method for detection of microbial cellulases on agar plates using Gram's Iodine. Curr. Microbiology. 57, 503-507 (2008).
- Huang, J. S., Tang, J. Sensitive assay for cellulase and dextranase. Anal. Biochem. 73, 369-377 (1976).
- Tull, D., Withers, S. G. Mechanisms of cellulases and xylanases: A detailed kinetic study of the exo-beta-1,4-glycanase from Cellulomonas fimi. Biochemistry. 33, 6363-6370 (1994).
- Martinez, A., Bradley, A. S., Waldbauer, J. R., Summons, R. E., DeLong, E. F. Proteorhodopsin photosystem gene expression enables photophosphorylation in a heterologous host. PNAS. 104, 5590-5595 (2007).
- Taupp, M., Lee, S., Hawley, A., Yang, J., Hallam, S. J. Large Insert Environmental Genomic Library Production. J Vis Exp. , (2009).
- Martinez, A., Tyson, G. W., DeLong, E. F. Widespread known and novel phosphonate utilization pathways in marine bacteria revealed by functional screening and metagenomic analyses. Environ Microbiol. 12, 222-238 (2010).
- Wild, J., Hradecna, Z., Szybalski, W. Conditionally amplifiable BACs: Switching from single-copy to high-copy vectors and genomic clones. Genome Research. 12, 1434-1444 (2002).
- Johnson, E. A. Sacchirification of complex cellulosic substrates by the cellulase system from Clostridium thermocellum. Appl Environ Microbiology. 43, 1125-1132 (1982).
- Stutzenberger, F. J. Cellulase Production by Thermomonospora curvata isolated from municipal solid waste compost. Appl Environ Microbiol. 22, 147-152 (1971).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved