Method Article
כרומוזום מלכודת הידיעות AP עבור זיהוי לטווח ארוך אינטראקציות DNA
In This Article
Summary
המלכודת קשורה כרומוזום (ACT) assay היא שיטה חדשנית לזיהוי משוחדת ארוכי טווח אינטראקציות-DNA. אפיון האינטראקציות DNA ארוך טווח תאפשר לנו לקבוע את הקשר של האדריכלות גרעינית ביטוי גנים בפיזיולוגיה הן נורמליות במדינות חולה.
Abstract
מידע גנטי המקודד על ידי ה-DNA מאורגן במבנה הכרומטין מורכב פיקוח הדוק. כל כרומוזום תופסת שטח ספציפי, זה עלול להשתנות בהתאם לשלב ההתפתחות או מחזור התא. התבטאות גנים יכולה להתרחש במפעלים תעתיק התמחה בו קטעים הכרומטין יכול לולאה החוצה מהשטחים כרומוזומים שונים, המוביל לוקליזציה שיתוף קטעי DNA אשר עשוי להתקיים על כרומוזומים שונים או רחוקים אחד מהשני על כרומוזום זהה. מלכודת Associated כרומוזום (ACT) assay מספק מתודולוגיה יעילה לזהות אלו לטווח ארוך עמותות DNA באופן בלתי משוחד על ידי הרחבת ושינוי קונפורמציה כרומוזום טכניקת לכידת. Assay ACT מאפשר לנו לחקור את מנגנוני הרגולציה תעתיק של טרנס, ויכול לעזור להסביר את היחס של האדריכלות גרעינית ביטוי גנים בפיזיולוגיה נורמלי במצבי מחלה.
Protocol
1. פורמלדהיד קיבעון של ארוכי טווח אינטראקציות הכרומטין
- התא האנושי והתרבות קו HL-60 במדיום RPMI1640 עם FBS 15% ו 1 x פניצילין / סטרפטומיצין כדי מפגש 80-90% באינקובטור מסופק עם 5% CO 2 ב 37 ° C.
- איסוף התאים לתוך צינור Nunc 50 מ"ל, צנטריפוגה בסל"ד 1200 למשך 15 דקות ולאחר מכן להסיר את המדיום על ידי שאיפה
- הוסף 5 מ"ל של מדיום תרבות resuspend את כדורי תא, לספור את התאים בעזרת hemocytometer. קח כ 1 x 10 7 תאים בהיקף של 40 מ"ל עם FBS RPMI1640 / 10%, ולאחר מכן להוסיף 1.7 מ"ל של פורמלדהיד 37% כדי לתקן את הכרומטין לדלל.
- דגירה בטמפרטורת החדר למשך 10 דקות עם רעד עדין, ולאחר מכן להרוות עם 2.4 מ"ל של גליצין 2M. צנטריפוגה במשך 15 דקות ב 1200 סל"ד ב 4 ° C, ולהסיר את supernatant. שטפו את הכדור פעם אחת עם 40 מ"ל קרח קר PBS, ואז לסובב את גלולה ולהסיר את PBS.
2. תא תמוגה לבודד גרעינים
- תאים Resuspend ב 40 מ"ל של חיץ קרים תמוגה (10 mM טריס-HCl, pH8.0, 10 mM NaCl, 0.2% NP-40) עם מעכבי פרוטאז הוסיף טרי (1:500 דילול) ו 0.1 מ"מ PMSF. דגירה בחדר קר עם סיבוב במשך 90 דקות, צנטריפוגה בסל"ד 2500 למשך 15 דקות, ולהסיר את supernatant.
3. אנזים הגבלת העיכול עם Bgl II
- Resuspend את הגרעינים ב 0.5 מ"ל של 1 x 3 חוד חיץ, ולהוסיף 15 μlof SDS 10%. לדגור על 37 מעלות צלזיוס במשך שעה 1 עם טלטולים, ולאחר מכן להוסיף 45 μl של 20% Triton X-100 כדי לעקל SDS. לדגור על 37 מעלות צלזיוס במשך שעה 1 עם רעד.
- השתמש aliquot של גרעינים 1 x 10 6 (כ -15 מיקרוגרם, 1 / 10 של התאים המקוריים) לעיכול אנזים הגבלה. קח את 55 μl של פתרון גרעינים משלב 3.1 ולעשות עד μl 500 עם 433 μl של חיץ 1 חוד x 3 ו 12 μl של Bgl II (50U/ul). לדגור על 37 מעלות צלזיוס למשך הלילה.
4. קשירת מקטעי דנ"א אינטראקציה
- להשבית את האנזים הגבלה על ידי הוספת 95 μl של 10% SDS, ו לפגל על ידי חימום על 65 מעלות צלזיוס במשך 20 דקות באמבט מים.
- הוסף 7 מ"ל של קשירת חיץ x 1 (30 מ"מ טריס-HCl, pH 8.0, 10 mM MgCl 2, 10 mM DTT, 1 mM ATP) ו - 360 μl של טריטון 20% X-100 ו לדגור על 37 מעלות צלזיוס במשך שעה 1 .
- מנמיכים את הטמפרטורה ל 16 מעלות צלזיוס ומוסיפים 50 μl של U 400 / μl האנזים T4 DNA. דגירה המדגם על 16 מעלות צלזיוס למשך 4 שעות ואז בטמפרטורת החדר למשך 30 דקות.
5. ה-DNA טיהור
- הוספת 300 מיקרוגרם של proteinase K, ו לדגור על 65 מעלות צלזיוס למשך הלילה.
- הוסף 5 מיקרוגרם של RNase A ו לדגור על 37 מעלות צלזיוס במשך 30 דקות
- לטהר DNA על ידי מיצוי פנול / כלורופורם, ו-DNA המשקע ב isopropanol. ממיסים-DNA של 150 μl מים מזוקקים סטריליים.
6. עיכול עם MSP אני ואת קשירת עם linkers oligonucleotide
- דגירה של 2μg מטוהרים DNA עם 5 יחידות של MSP אני ב 37 מעלות צלזיוס במשך 4-6 שעות. להשבית MSP אני על 65 מעלות צלזיוס למשך 10 דקות, ולאחר מכן להאיץ את ה-DNA של אתנול עם μl 1 של גליקוגן מ"ג / 5 מ"ל. ממיסים את גלולה דנ"א 50 μl של מים מזוקקים סטריליים.
- מערבבים 50 μl של ה-DNA אני שטופלו MSP עם 2 μl של L 20 oligonucleotide מקשר מיקרומטר (5'-gctgaccctgaattcgcacgtgcctgtcgttagcggacacagggcgattcac-3 "), 1 μl של 20 מיקרומטר oligonucleotide S (5'-cggtgaatc-3"), 1 μl של מים מזוקקים סטריליים 6 μl של 10 x למאגר דנ"א האנזים T4. מכסים את התערובת עם שעווה נוזלית.
- Oligonucleotides לפגל על 50 מעלות צלזיוס למשך דקה 1 ולאפשר להתקרר בהדרגה עד 10 מעלות שיפוע 0.5 C / דקה · ב Cycler תרמית.
- הוסף 1 μl של 400 U / DNA μl האנזים T4 ו לדגור על 15 מעלות צלזיוס למשך הלילה. לטהר את הדנ"א מקשר-ligated בעזרת ערכת ה-PCR QIAquick טיהור, elute ב 50 μl של מים מזוקקים סטריליים.
7. PCR הגברה רצף ניתוח
- בחר פריימר לאזור מסוים של הדנ"א שאתם רוצים לבחון את יחסי הגומלין ארוכי הטווח (כלומר, "פיתיון"). בדוגמה זו, אנו משתמשים ABL-1.
- קח 1 μl של ה-DNA מטוהרים כדי לבצע סיבוב הראשון באמצעות PCR 1 μl של 20 מיקרומטר פריימר ABL -1 ספציפי # 4656 (5'-gttcaagcgattctcctgcctcga-3 "), 1 μl של 20 מיקרומטר פריימר ספציפי מקשר # 2963 (5'- gctgaccctgaattcgcacgtgcct-3 "), 3μl של 3 x Klen DNA פולימראז תקי אני קוקטייל 3 μl של מים מזוקקים סטריליים. שני μl של 32 P-dCTP מתווסף μl 100 של 3 x תקי DNA פולימראז Klen אני קוקטייל לפני הגברה PCR. מחזור תרמית התחלה PCR חם היא 72 מעלות צלזיוס למשך 2 דקות, 95 ° Cfor 2 דקות, 18 מחזורים של 95 מעלות צלזיוס למשך 20 שניות, 65 מעלות צלזיוס למשך 40 שניות, 72 ° C למשך דקה 1: סיומת סופי מבוצע על 72 ° Cבמשך 5 דקות.
- לטהר את המוצרים הראשונים בסיבוב PCR בעזרת ערכת ה-PCR QIAquick טיהור, ו-DNA elute ב 30 μl של מים מזוקקים סטריליים.
- קח 1 μl של 100 x בדילול first מוצר PCR לבצע הסיבוב השני של ה-PCR בעזרת פריימרים מקוננים על ידי הוספת 1 μl של 20 מיקרומטר ABL-1 פריימר ספציפי # 4626 (5'-ggagaatttttatctgcctctgtga-3 ") (איור 1 א), 1 μl של 20 מיקרומטר של פריימר ספציפי מקשר # 2961 (5'-gtcgttagcggacacagggcgattc-3 "), 3 μl של 3 x Klen DNA פולימראז תקי אני קוקטייל 3 μl של מים מזוקקים סטריליים. לוח הזמנים של רכיבה תרמית היא 25 מחזורים של 95 מעלות צלזיוס למשך 20 שניות, 67 מעלות צלזיוס למשך 40 שניות, 72 ° C עבור 1 דקה, ואחריו סיומת על 72 מעלות צלזיוס למשך 5 דקות.
- דמיינו את מוצרי ה-PCR על ידי הפעלת 5% אוריאה-PAGE ג'ל וסריקה המסך נחשף PhosphoImager (איור 1b). להקה כל PCR ניתן למחזר מן הג'ל על ידי המסת את רצועות ג'ל בשפופרת Eppendorf המכיל 60 μl של מים מזוקקים סטרילי, דוגרים על 95 מעלות צלזיוס למשך 5 דקות. צנטריפוגה בקצרה בסל"ד 10,000 עבור 10 שניות לאסוף את כל דגימות. הסר 1 μl להשתמש כתבנית ה-DNA כדי לבצע PCR עם 2961/4626 תחל זוג באמצעות באותם תנאים כפי שתואר לעיל. ניתוח רצף יכול להתבצע לאחר טיהור באמצעות ערכת ה-PCR QIAquick טיהור.
- רצפי DNA מנותחים באמצעות כלי מקוון כדי לקבוע את המיקום הכרומוזומלי שלהם באתר האינטרנט: http://genome.ucsc.edu (איור 1 ג'). לחץ על "בלאט" להיכנס חלון חדש המאפשר להדביק רצף ה-DNA, חלון חדש מופיע לאחר לחיצה על 'שלח', ומראה את' תוצאות חיפוש בלאט ". לחץ על 'הדפדפן' של פגע עם זהות 100% כדי להיכנס לחלון הבא כדי להראות את מיקומו של רצף ה-DNA.
8. נציג תוצאות
1. ACT assay באמצעות ABL-1 באזור כפיתיון כדי לקבוע את ה-DNA שלה אינטראקציות ארוכות טווח
כמוצג באיור 1a, שני אתרי Bgl השנייה ואחד BamH אני באתר נבחרו assay את המעשה. בסיבוב השני של ה-PCR, תחל להגדיר 4626/2961 שימש להגביר ABL-M1, 4630/2961 שימש ABL-M2, ו 4636/2961 שימש ABL-M3. דפוס אופייני ג'ל מראה אחת כמה להקות (איור 1b). כל קטע מן assay ACT מורכב משני חלקים DNA משולב: קטע אחד נגזר באזור DNA הפיתיון, ואילו במגזר השני נובע השותף הקשורים, אשר הצטרפו למגזר באזור הפיתיון על ידי רצף הגבלה first אנזים הכרה. רצף second הכרה אנזים יופיעו בסוף רצף שותף הקשורים (איור 1 ג). משובטים ABL-M1 שבר מכיל DNA מאזור ABL1 הממוקם בכרומוזום 9q32.4 מ +133,592,306-133,592,399, והשותף הקשורים ממוקם בכרומוזום 3p13 מ +71,869,882-71,870,107. זהותם של השותפים הקשורים מתגלים על ידי פיצוץ רצפים שלהם באמצעות דפדפן הגנום UCSC (GRCh7/hg19 שוחרר בפברואר, 2009). PROK2 זוהה כשותף הקשורים ב מוקד chr3p13.
כמו כן, השותף ABL-M2 הקשורים היה מקומי כדי chr5q21.1, בעוד שיבוט ABL-M3 זוהה העמותה תוך כרומוזומלית ליד מוקד-1 ABL.
2. קביעת נפוצה פחות אינטראקציות ארוכות טווח באמצעות assay ACT
מבחני אחרים המבוססים על 3C דיווחו שותפים רבים יותר אינטראקציה מאשר שיש לנו שמוצג באיור 1 ובבית Igf2 / H19 מוקד 12. המתודולוגיה שיש לנו התווה יבחר את יחסי הגומלין הנפוצה ביותר לטווח ארוך. עם זאת, על ידי הגדלת מספר מחזורי ה-PCR, ניתן לזהות נוספות, עמותות פחות תכופים, כמו גם (איור 2).
3. הבדלים אינטראקציות ארוכות טווח בתאי סרטן בהשוואה לרקמות.
Assay ACT יכול לשמש גם כדי לזהות הבדלים בארכיטקטורה גרעיני אינטראקציות ארוכות טווח בין תאים נורמליים לתאים סרטניים (איור 3). הבדלים אלה עשויים לשקף שינויים בארכיטקטורה גרעיני המתרחשים במהלך שינוי התא, ובכך assay זה עשוי בסופו של דבר להיות ישימה למטרות אבחון. דפוסי ג'ל דומים המתרחשים הן נורמליות רקמות סרטן ציינו כי assay ACT הוא אמין לשחזור. בעוד assay ACT ניתן לזהות DNA ארוך טווח שותפים, ניתוחים נוספים, כגון דגים מבחני CHIP, נדרשים לאמת את נוכחותם של עמותות זיהו בין לוקוסים הרחוק. מחקרים גנטיים, פיזיולוגיים ביוכימיים אז יכול להיות מבוצע על מנת להבהיר את ההשלכות הביולוגיות של עמותות אלה DNA ארוך טווח.
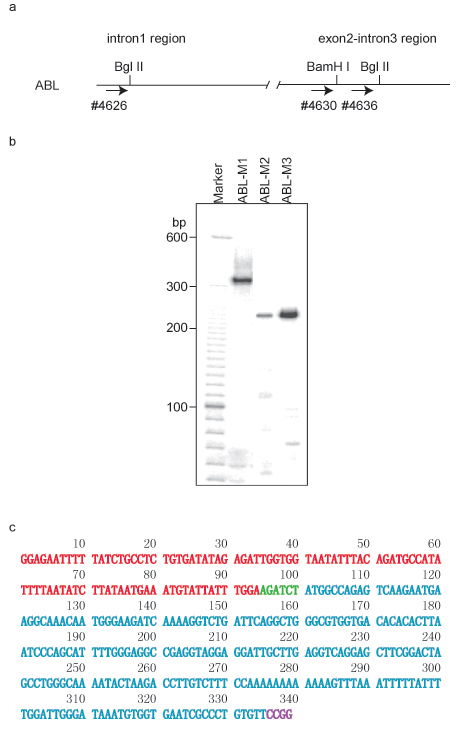
איור 1 ACT assay באמצעות ABL-1 אזור ה-DNA כמו פיתיון ב HL-60 תאים. א ה-DNA structיור באזור 1-ABL. אנזים ההגבלה הראשונה השתמשו בחוק assay היה Bgl השנייה. Primers לסיבוב השני של ה-PCR מסומנים גם על ידי חיצים ומספרים פריימר המקביל. ב ג'ל דפוס של assay לפעול 5% אוריאה עמודים. זוג פריימר 4626/2961 שימש הגברה שיבוט ABL-M1 ב PCR מקוננות, 4630/2961 שימש שיבוט ABL-M2, ו 4636/2961 שימש שיבוט ABL-M3. ג רצף ה-DNA של שיבוט ABL-M1. קטע ה-DNA מ-DNA 1-ABL הפיתיון מסומן באדום, ואת שותף DNA הקשורים מסומן ב ציאן. Bgl השנייה באתר (AGACTC) היה מסומן ירוק, MSP אני באתר (CCGG) מסומן בסגול.

איור 2 מחזורי PCR להשפיע על תוצאות ACT assay. שימוש בשלט החתמה האזור (ICR) בשעה מוקד Igf2/H19 כמו פיתיון DNA בתאים פיברובלסטים עכבר, תוכניות רכיבה שונים יושמו את הסיבוב הראשון והשני של ה-PCR ב assay את המעשה. 18-20 מחזורים בסיבוב הראשון של ה-PCR לא להגביר אות מספיק כדי להיות דמיינו. עשרים וחמישה מחזורים בתוצאות second-PCR בלהקות ברורה, תוך הגדלת מספר מחזורי לסיבוב השני של ה-PCR המושרה דפוס למרוח בנוסף להקות יותר.

איור 3 איתור של הבדלים בארכיטקטורה גרעיני בין רקמת המעי הגס נורמלי סרטן רקמות המעי הגס assay ACT. A. מבנה ה-DNA של האזור ICR בבית מוקד IGF2/H19 ואת הגן IGF2. DPN אתרים II עבור assay ACT מסומנים. Primers בשימוש השני בסיבוב PCR מסומנים על ידי חצים ומספרים בצבעים שונים אשר מתאימות כל נתיב ב פאנל של הדמות. ב ג'ל דפוס של assay לפעול 5% אוריאה עמודים. רקמת המעי הגס רגיל MAD03-1423 ו - סרטן המעי הגס רקמות MAD04-149 התקבל רשת שיתופית רקמה אנושית (CHTN) החטיבה המערבית. לאחר כל רקמת המעי הגס היה הומוגני, assay ACT בוצע בעקבות הליכים המתוארים להלן. ליין 1 מייצג את תוצאות ה-PCR באמצעות זוג פריימר # # 2961and 4161 (5'-tctgcgccatcagggcagtgagac-3 ") שכותרתו בוורוד בפאנל; מסלול 2 מייצג את תוצאות ה-PCR באמצעות זוג פריימר # # 2961 ו - 4163 (5'-3-gccgcgcggccacttccgattcc ') מסומן בכתום בפאנל: מסלול 3 מייצג את תוצאות ה-PCR באמצעות זוג פריימר # # 2961 ו - 5145 (5'-gccatgcaggtaggatttgagctg-3 ") שכותרתו בכחול בפאנל: מסלול 4 מייצג את תוצאות ה-PCR באמצעות זוג פריימר # 2961 ו # 5151 (5'-gtctcaaataggggccagctagcttgg-3 ") שכותרתו בירוק בלוח א להקות ייחודיות שמופיעות רק ברקמות המעי הגס נורמלי מסומנים על ידי חצים צהובים, ואלה הלהקות שהופיעו רק ברקמת סרטן המעי הגס מסומנים ב החצים האדומים. ICR, שליטה באזור החתמה, DMR, אזור מפוגל שונים.
Discussion
דקר et al. פיתח את הקונפורמציה כרומוזום ללכוד (3C) הגישה לזהות את התדר של אינטראקציה בין שני לוקוסים הגנומי 1, 3C נעשה שימוש נרחב לחקור עמותות התוך כרומוזומליות וכן בין הכרומוזומים בין שני אזורים הידועים DNA בתאי יונקים 2 -9. למרות פיתח מתודולוגיה Hi-C יכול להיות מיושם על המחקר של הגנום כולו DNA העמותה, assay ACT עדיין טכניקה יעילה ללימוד האינטראקציה מוקד ספציפי DNA 10-11. אנחנו צריכים לשנות את הגישה הזאת כדי לזהות אזורים DNA ידוע המשויכים באזור DNA ידוע עכבר בתרבית תאים אנושיים (איור 1). קראנו בשיטה זו מלכודת כרומוזום קשור (ACT) assay, כפי שהוא סיפק לנו שיטה אמינה לשעתקו לזהות רומן שותפים DNA ידוע כי לקשר עם אזור הידוע היעד DNA 12. Assay 3C מוצלח עם פקדי המתאים מבוצעת לפני ביצוע ההיבטים הרומן של assay ACT 13. על מנת tofind כמה אזורים DNA הקשורים ככל האפשר, יש צורך להשתמש שילובים שונים של אנזימי הגבלה הראשון והשני. חשוב במיוחד להשתמש אנזימי הגבלה, כי הם רגישים מתילציה CPG לבצע את הצעד הראשון קשירת 3C. מחייב חלבון מתילציה בדנ"א יכולים גם להשפיע על אנזים הגבלה יעילות העיכול עלולה להוביל לכישלון של קשירת האזורים DNA הקשורים עבור digestions אנזים מסוים הגבלה. הופעתם של קשירת פנים או בין כרומוזומליות תלוי-DNA חלבון cross-linking ומפות גופנית מתאימה של שני האזורים DNA הקשורים. לכן, כמה ניסויים ראשוניים חיוניים להקים ריכוז פורמאלדהיד מעשי יעיל בזמן הטיפול assay את המעשה. טווח של ריכוזי הסופי של פורמלדהיד (from1.5% עד 2%) היו בשימוש במעבדות רבות במהלך החלק 3C של assay 7,9. לחלופין, oligonucleotides לשמש linkers יכול להיות מתוכנן להתאים את סוף מלוכדת לחתוך על ידי אנזים ההגבלה השנייה. אמנם מצאנו כי 18-20 מחזורים בסיבוב הראשון של ה-PCR ו 20-25 מחזורים בסיבוב השני של ה-PCR יכולה לספק להקות ברור, יש צורך לקבוע את התנאים PCR הטוב ביותר עבור כל ניסוי (איור 2). תוך מולקולת חישול בין הרצף 5'-end 3'סוף מתאם משלימה של גדיל הדנ"א עלולה להתרחש PCR, זה מעכב מתאם ספציפי פריימר חישול עם מולקולת ה-DNA, וכתוצאה מכך יעילות הגברה נמוכה בהרבה את מספר מחזורי הראשון. אחרי ה-DNA היעד היה מוגבר על מחזורים, סכום שלה עשוי להיות הרבה יותר גדול מאשר התגובות האלה ספציפי, ויכול להקל על התחרות של פריימר חישול את מולקולות ה-DNA היעד. זו גם הסיבה שאנו עשויים לראות הגברה רקע, ומדוע המוצר הראשון בסיבוב PCR צריך להיות מדולל כדי להקטין amplification.It רקע חשוב להסיר primers עודף מן התגובה PCR על מנת להקטין את הרקע בסיבוב השני של ה-PCR. כמו בכל PCR מבוססי ניסויים, חיוני לתכנון primers שלא נמצאים ברצף אזורים חוזרים, המהווים את רוב הדנ"א האנושי ועכבר. למרות פיתח מתודולוגיה Hi-C יכול להיות מיושם על המחקר של הגנום כולו DNA העמותה, assay ACT עדיין טכניקה יעילה ללימוד האינטראקציה מוקד ספציפי DNA.
Disclosures
Acknowledgements
אנו בזכות אדל Murell ו וולף רייק מאוד לשיתוף פרוטוקול 3C שלהם. עבודה זו נתמכה על ידי מחלקת הביטחון של שירות המחקר של המחלקה לענייני יוצאי צבא.
Materials
| Name | Company | Catalog Number | Comments |
| RPMI1640 medium | Invitrogen | 22400-105 | |
| acrylamide | Invitrogen | 15512-023 | |
| ATP solution, 10mM | Invitrogen | AM8110G | |
| fetal bovine serum | Invitrogen | 16000-044 | |
| penicillin-streptomycin | Invitrogen | 15140-122 | |
| 1M Tris pH8.0 | Invitrogen | AM9856 | |
| RNase A | Invitrogen | 12091-039 | |
| SDS | Invitrogen | 15525-017 | |
| urea | Invitrogen | 15505-035 | |
| BamH I | New England Biolabs | R0136T | |
| Bgl II | New England Biolabs | R0144M | |
| Dpn II | New England Biolabs | R0543T | |
| Msp I | New England Biolabs | R0106S | |
| dNTPs | New England Biolabs | N0447L | |
| proteinase K | New England Biolabs | P8102S | |
| T4 DNA ligase | New England Biolabs | M0202T | |
| 37% formaldehyde | Sigma-Aldrich | F8775 | |
| Bis-acrylamide | Sigma-Aldrich | 146072 | |
| dithiothreitol | Sigma-Aldrich | 43815 | |
| glycine | Sigma-Aldrich | 50046 | |
| PMSF | Sigma-Aldrich | 93482 | |
| proteinase inhibitor | Sigma-Aldrich | S8830 | |
| Nonidet P-40 | Roche Group | 11754599001 | |
| KlenTaq1 | Ab peptides | 1001 | |
| dCTP alpha P32 | PerkinElmer, Inc. | BLU513H250UC | |
| PTC-100 Thermal Cycler | MJ Research | mjptc100 | |
| Power Supply | Bio-Rad | 164-5056 | |
| OmniPAGE Maxi | Aurogene Life Science | VS20D | |
| Typhoon 9400 | GE Healthcare | 63-0055-78 |
References
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295, 1306-1311 (2002).
- Apostolou, E., Thanos, D. Virus Infection Induces NF-kappaB-dependent interchromosomal associations mediating monoallelic IFN-beta gene expression. Cell. 134, 85-96 (2008).
- Duan, Z. A three-dimensional model of the yeast genome. Nature. 465, 363-367 (2010).
- Lomvardas, S. Interchromosomal interactions and olfactory receptor choice. Cell. 126, 403-413 (2006).
- Murrell, A., Heeson, S., Reik, W. Interaction between differentially methylated regions partitions the imprinted genes Igf2 and H19 into parent-specific chromatin loops. Nat Genet. 36, 889-893 (2004).
- Schoenfelder, S. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. Nat Genet. 42, 53-61 (2010).
- Spilianakis, C. G., Lalioti, M. D., Town, T., Lee, G. R., Flavell, R. A. Interchromosomal associations between alternatively expressed loci. Nature. 435, 637-645 (2005).
- Vakoc, C. R. Proximity among distant regulatory elements at the beta-globin locus requires GATA-1 and FOG-1. Mol Cell. 17, 453-462 (2005).
- Xu, N., Tsai, C. L., Lee, J. T. Transient homologous chromosome pairing marks the onset of X inactivation. Science. 311, 1149-1152 (2006).
- Lieberman-Aiden, E. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Berkum, N. L. van Hi-C: a method to study the three-dimensional architecture of genomes. J Vis Exp. , (2010).
- Ling, J. Q. CTCF mediates interchromosomal colocalization between Igf2/H19 and Wsb1/Nf1. Science. 312, 269-272 (2006).
- Dekker, J. The three 'C's of chromosome conformation capture: controls, controls, controls. Nat Methods. 3, 17-21 (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved