A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ReAsH / פלאש סימון ניתוח מקדימה של חלבונים בתאים חיישן Tetracysteine
In This Article
Summary
צבעים biarsenical פלאש ReAsH להיקשר באופן ספציפי מוטיבים tetracysteine בחלבונים והוא יכול סלקטיבי תווית חלבונים בתאים חיים. לאחרונה באסטרטגיה זו תיוג נעשה שימוש כדי לפתח חיישנים עבור תצורות חלבונים שונים או מדינות oligomeric. אנו מתארים את גישת התיוג ושיטות כמותית לנתח מחייב.
Abstract
חלבונים פלורסנט וצבעים הם כלים חיוניים לחקר לוקליזציה סחר, חלבונים לתפקד בתאים. בעוד חלבוני ניאון כגון חלבון פלואורסצנטי ירוק (GFP) נעשה שימוש נרחב כשותפים היתוך חלבונים כדי לעקוב אחר תכונות של חלבון של עניין 1, ההתפתחויות האחרונות עם תגיות קטנות מאפשרות פונקציות חדשות של חלבונים להיבחן בתאים כגון שינוי קונפורמציה וחלבון אסוציאציה 2, 3. אחת מערכת תג קטן כרוך מוטיב tetracysteine (CCXXCC) מוכנס לתוך גנטית חלבון מטרה, אשר נקשר צבעים biarsenical, ReAsH (אדום ניאון) ו-Flash (ירוק זרחני), עם סגוליות גבוהה אפילו בתאים חיים 2. מערכת TC / biarsenical לצבוע מציעה אילוצים סטרית הרבה פחות חלבון מאשר לארח חלבוני ניאון אשר אפשרה מספר גישות חדשות כדי למדוד שינוי קונפורמציה ו חלבונים אינטראקציות 4-7. אנחנו לאחרונה פיתחה יישום הרומן של תגיות TC כמו חיישנים של oligomerization בתאים לבטא huntingtin מוטנטים, אשר כאשר מוטציה אגרגטים נוירונים מחלת הנטינגטון 7. Huntingtin היה מתויג עם שני צבעי ניאון, אחד חלבון פלואורסצנטי לעקוב אחר המיקום חלבון, ואת תג שנייה TC שרק נקשר צבעים biarsenical ב מונומרים. לפיכך, שינויים colocalization בין חלבון תגובתיות לצבוע biarsenical מופעלת תוכן oligomer submicroscopic להיות ממופה מרחבית בתוך התאים. כאן אנו מתארים כיצד תווית TC-tagged חלבונים התמזגו חלבון פלואורסצנטי (שרי, GFP או CFP) עם פלאש או ReAsH בתאי יונקים לחיות וכיצד לכמת את הקרינה two צבע (שרי / Flash, CFP / Flash או ה-GFP / ReAsH שילובים).
Protocol
1. הכנת תאים עם תיוג ReAsH / פלאש
- בעזרת שיטות סטנדרטיות תרבות התא לקו הנייד של עניין, להכין התרבות של תאים חסיד ישירות שקופיות הדמיה לחיות התא מוכן transfection.
- Transfect פלסמיד המכיל שלך TC-tagged הגן של עניין על פי שיטת transfection בחירתך.
הערה חשוב להשתמש בפקדי חיוביים ושליליים כדי להעריך את מידת מחייב ספציפית את התגיות TC ולהעריך עבור bleedthrough של הקרינה בין הערוצים בעת איסוף micrographs confocal. לפיכך, עבור שני צבעים (לדוגמה Flash / או שרי ReAsH / או CFP ReAsH / GFP שילובים), להבטיח דגימות מוכנים צבעים בודד (חלבון פלואורסצנטי למשל לבד או אם אפשר חלבון TC-tagged חייב Flash / ReAsH אך ללא פלורסנט חלבון)
- יום או יומיים לאחר transfection, לשטוף בעדינות את התאים עם 300 μL מראש התחמם (ב 37 ° C) HBSS.
- בעדינות לטבול את התאים עם פלאש 1 מיקרומטר (או ReAsH) ב 300 μL של HBSS prewarmed ו -10 מיקרומטר 1,2-ethanedithiol (EDT).
חשוב להוסיף EDT הראשון לפני הוספת Flash / ReAsH ולהפוך את המאגר רק לפני הוספתו התאים. דגירה של 30 דקות בדיוק בשעה 37 ° C ברקמות תרבות חממה. כבר בשנת הניסיון שלנו פעמים הדגירה מגביר באופן משמעותי את הקרינה רקע. בונה חדש צריך להיות גם מותאם תיוג זמן Flash / ReAsH ריכוז (0.5-2 מיקרומטר).
- לשאוב בעדינות תיוג פתרון מתאי ולאחר מכן להחליף עם 300 HBSS prewarmed μL + 250 מיקרומטר 2,3-dimercaptopropanol (BAL) במשך 15 דקות על 37 ° C.
- הסר לשטוף פתרון על ידי שאיפה עדינה להחליף עם 300 HBSS prewarmed μL.
אחרי זה לשטוף, התאים עשויים להיות קבועים עם paraformaldehyde (15 דקות עם פתרון 3.2%), אם כי מצאנו כי זה מגביר את הלא ספציפית צבע פלואורסצנטי biarsenical. לכן אנחנו בדרך כלל תמונה תאים חיים בטמפרטורת החדר. (שים לב קיבעון של תאים לפני תיוג מונע מחייב לצבוע biarsenical).
2. דימות את התאים במיקרוסקופ confocal
- על מיקרוסקופ confocal, להגדיר פרמטרים עבור הדמיה fluorophores הפרט (ראה לוח 1), ולהבטיח שיש bleedthrough זניח בין הערוצים. זו יכולה להיות מושגת על ידי בדיקת fluorophores הפרט (בדגימות שליטה) נגד הגדרת הרכישה שונה בכל ניאון עבור פלואורסצנטי. התאמת טווח אורך גל הפליטה יכולים לעזור להקטין את bleedthrough (אם כי זה יכול גם להפחית את אות / רעש).
- גם להתאים את הגדרות מכפיל כך הקרינה המרבית במדגם לא להרוות את גלאי. (זה יכול להיות מזוהה באמצעות הגדרת Q-LUT על לייקה SP2 confocal). לאחר הגדרות הדמיה הטובים ביותר הם נחושים, לא לשנות כל אחד מהם בין דגימות.
- הגדרות אחרות אנחנו משתמשים בדרך כלל (אם כי אלה יכול להיות מותאם) הם קצב סריקה של 200 הרץ ולאסוף 4 ממוצעים קו בקוטר חריר של יחידה 1 אוורירי. קוטר חריר ניתן להרחיב אם האות / הרעש הוא בעיה, לעומת זאת, זה עלול לגרום לאובדן חלק הדמיה.
- איסוף התמונות confocal בפורמט (או יותר) 12-bit אם אפשר. 12-bit בפורמט לוכדת טווח דינמי גדול יותר של ערכים (0-4095) עבור עוצמת כל פיקסל מ 8 סיביות (0-255). זה חשוב כדי להבטיח את מערכת הנתונים העשיר ביותר נרשם, אשר ממקסם את האיכות של ניתוח נתונים כמותיים.
- איסוף התמונות של הערוץ חלבון פלואורסצנטי (שרי, GFP או CFP) וגם לערוץ צבע biarsenical (Flash או ReAsH) עבור כל המדגמים.
3. ניתוח הנתונים
- התקן את ImageJ התוכנה במחשב 8. http://rsbweb.nih.gov/ij/
- ודא כי גירסת ImageJ יש את התוספים הבאים:
- פתח ImageJ, ואם באמצעות גרסה ישנה יותר מ v1.32c, לחץ על האפשרויות הבאות על מנת לאפשר מספר רב של תמונות להיפתח בבת אחת (אשר יכול להיעשות על ידי החזקת לחצן בקרת בעת לחיצה על קבצים שונים):
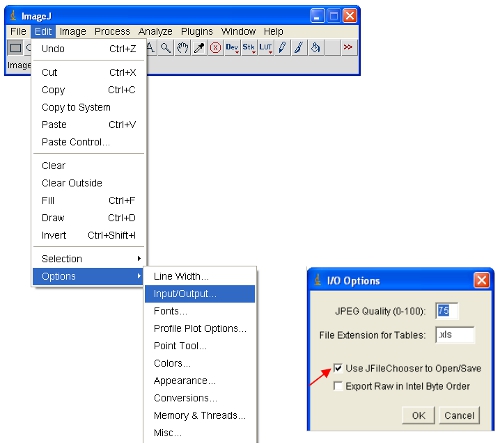
- פתח את התמונות עניין ImageJ.
ImageJ באופן אוטומטי מגדירים את עוצמות פיקסל מינימום ומקסימום המוצגות על המסך עבור כל תמונה בנפרד. בהתחשב בעובדה תמונות שונות יהיה דעוצמות פיקסל ifferent, זה אומר התמונות המוצגות לא ניתן להשוות כפי שנצפו. - כדי להבטיח את התמונות נפתח כולם באותו קנה מידה, אתה יכול להגדיר פיזית העליון פיקסל בעוצמות נמוכות כדי לראות (זה לא ישנה את תוכן הנתונים בפועל של התמונות כפי שניתן היה במקרה של תוכנה אחרת). ערכים אלה ניתן להגדיר תחת תפריט הפעולות הבאות:
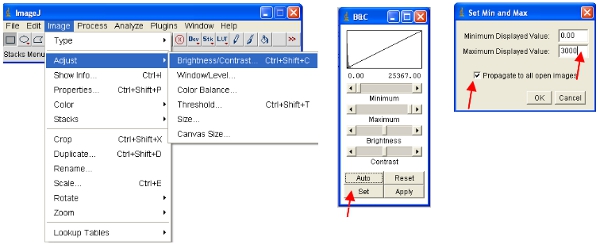
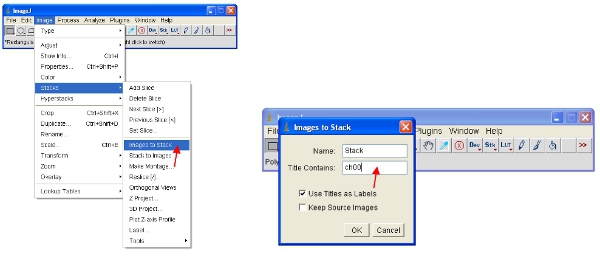
- גישה חלופית היא לעבוד עם ערימות, אשר מעמידה את כל התמונות יחד לתוך קובץ אחד וגם באופן אוטומטי בקנה מידה (לצפייה) כל התמונות בערימה בהיקף זהה. הדרך הקלה ביותר לעבוד עם הנתונים היא להמיר כל ערוץ בערימה. כך, ערוץ חלבון פלואורסצנטי להמיר את כל מחסנית כמוצג. באפשרותך לבחור בקלות ערוץ אחד על ידי הערמה המכיל את כל התמונות שם משותף (למשל, "ch00") בכותרת.
- עכשיו להמיר את כל התמונות פתוחה של 8 סיביות לצורך ניתוח. זה יהיה ממש rescale טווח שנצפו לתוך טווח 0-255 (המגדירה 8 סיביות).
חשוב: אל תשמור על 12-bit בפורמט המקורי (או יותר) או תאבד את תוכן המידע. שים לב כמה חבילות התוכנה ניתן להציג רק 8-bit תמונות בפורמט זה כל כך שימושי לביצוע דמויות וכו '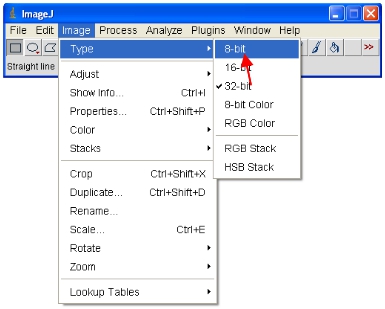
- שמור עותק בתיקייה חדשה בשם "8-bit המרה" באמצעות "שמירה בשם ..." אפשרות.
- אחת השיטות לבחון את מידת מחייב לצבוע biarsenical בתמונה כולה לבצע החלקה פיקסל מתאם אינטנסיביות. מגרשים זה כל ערך פיקסל בערוץ אחד ביחס לערך פיקסל המקבילה בערוץ השני. לפיכך עמדה פיקסל גבוה CFP הקרינה יהיה גם גבוה הקרינה ReAsH אם יש גבוה מחייב.
- כדי לנתח את פיקסל שיתוף המתאם, ודא 8-bit ReAsH Cerulean ו / GFP תמונות פתוחים ImageJ.
- פתח את "קורלטור תמונה" תוסף כמפורט להלן. בחר "Image1" כמו ערימה ReAsH או Flash ו "Image2" כמו ערימת חלבון פלואורסצנטי.
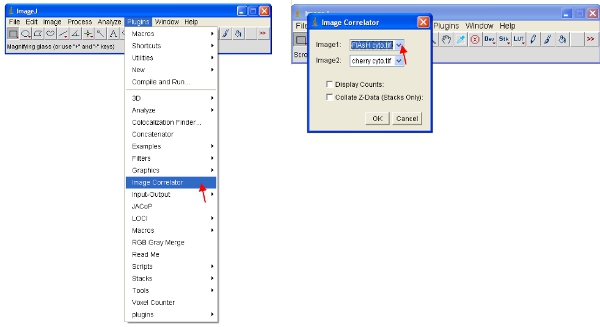
- מחסנית וכתוצאה מכך אין להציג מידע מפורט - זה נורמלי. שמור את מחסנית בתיקייה חדשה בשם "Scatterplots" ולתת לו את שם הקובץ בדיוק כמו המדגם הוא מתייחס (לדוגמה: "פלאש דובדבן העלילה מתאם")
- כדי להציג את הנתונים בצורה משמעותית אתה יכול לעשות אחת משתי אפשרויות. ראשית לשנות את הנתונים כך שיהיה בפורמט יומן. ואז חזותית rescale את הנתונים כדלקמן:
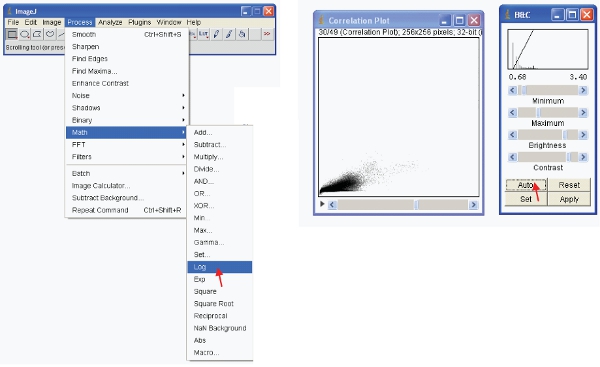
- לחלופין, ניתן rescale את הנתונים ויזואלית להציג רק ערכים נמוכים (למשל 1-255) ולהציג את הנתונים בעזרת LUT מיוחד. LUT (חפש טבלה) מייצג את טבלת הצבעים המוקצים ערך כל פיקסל בתמונה . זה יכול לשמש כדי להגדיר ערכת pseudocolor לתמונה והיא שימושית להגדרת תכונות מסוימות בתמונה. כדי ליצור LUT מותאם אישית לחץ על "עורך LUT" ולעשות אחד חדש כמו זה שמוצג (זה ניתן לשמור ולהשתמש בהם מאוחר יותר).
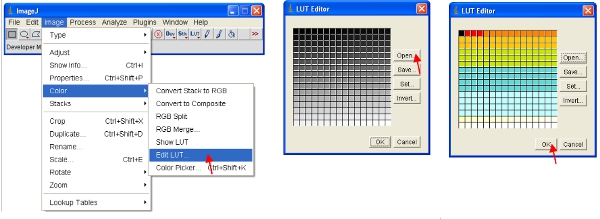
- הגדר את טווח בהירות / ניגודיות 0-255 (כמתואר בשלב 13 לעיל). כדי "לנעול" את התמונה כפי לזה המוצג על המסך, התמונה יכולה להיות המרה לפורמט RGB אשר חוסך ערך 8 סיביות עבור כל צבע, אדום, ירוק וכחול של כל פיקסל.
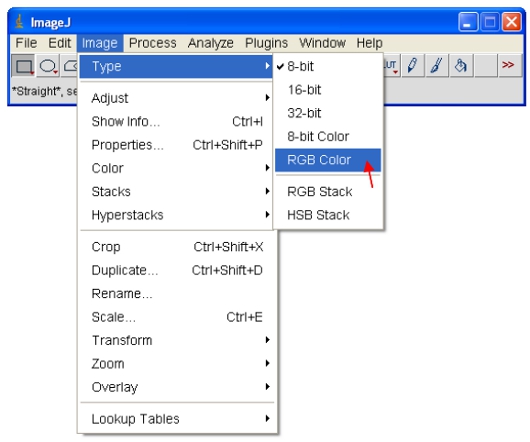
כדי להעתיק ולהדביק תמונות לתוכניות אחרות פותח לראשונה את התמונות 8-bit או 16-bit. כמתואר בשלב 14 לעיל, להקצות צבע LUT תכנית לתמונה. עבור CFP, להקצות את "כחול" LUT כמתואר להלן ...
- בחר את התמונה כדי להעתיק אותו ללוח, בחר ...
4. נציג התוצאות:
ההצלחה של תאים תיוג עם צבעים biarsenical תלוי כמה פרמטרים מרכזיים. ראשית, העיתוי של תיוג עם צבעים היא קריטית. מצאנו כי תקופות ממושכות של תיוג (יותר מ 30 דקות) תוצאות ברמה גבוהה של מכתים הלא ספציפית רקע. איור 1 מציג תוצאה אופיינית טופס wild-type של שבר huntingtin (25Q) התמזגו Cerulean נגזרת CFP המכיל תג TC כפי שתואר לעיל 7. מדגם זה היה מוכתם למשך 30 דקות עם ReAsH ויש רקע מינימלי במדגם חסרים את תג TC. מצאנו כי קיבוע תאים עם רקע עליות paraformaldehyde תוך קיבוע עם מתנול מבטלת את הקרינה של תג חלבון פלואורסצנטי. לפיכך במידת האפשר אנו התמונה תאים חיים. חשוב גם לציין כי הקיבעון מוקדמתעל תיוג עם צבעים biarsenical מונע, מחייב שלהם כנראה בשל שינויים של מוטיב TC.
גורם קריטי עבור תוצאות עקביות היא צפיפות של תאים. מצאנו זה קריטי תאים התמונה מופצים באופן רופף וגם כי clumping נרחב יכול להוביל מכתים אחידה של צבעי biarsenical בתאים שונים.
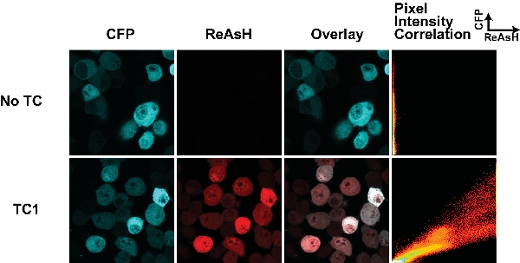
באיור 1. התגיות Tetracysteine והכתים ReAsH בתאים חיים transfected עם huntingtin (exon1-25Q)-Cerulean fusions. תג TC ממוקם בצומת של היתוך huntingtin-Cerulean (כפי שמתואר 7). העלילה פיקסל מתאם עוצמת מאפשר הערכה להבדלים ReAsH מחייב ברחבי התא והוא יכול לשמש כדי למפות שינויים ReAsH מחייב עקב שינוי קונפורמציה או אינטראקציות ליגנד.
Access restricted. Please log in or start a trial to view this content.
Discussion
הגישה לוקליזציה חלבון תווית עם חלבון פלואורסצנטי ומאפיינים קונפורמציה עם צבע שנייה מציע הרבה פוטנציאל למיפוי בו תצורות שונות של חלבונים בתאים לצבור ואירועים לשנות את הדינמיקה של קונפורמציה בחלבון. ReAsH / פלאש שימש לראשונה כחיישן ב-cell עבור קיפול חלבונים של חלבון יונק?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
אין ניגודי אינטרסים הכריז.
Acknowledgements
עבודה זו מומנה על ידי מענקים DMH ו TDM (NHMRC מענקים הפרוייקט). DMH הוא עמית Grimwade, שמומן על ידי הקרן Miegunyah.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| שם מגיב | חברה | מספר קטלוגי | תגובות |
|---|---|---|---|
| 8-μ היטב-שקופיות | Ibidi | 80826 | אנו מוצאים קאמרית אלה שקופיות להיות שימושי במיוחד עבור תאים culturing הדמיה. |
| TC-Flash השנייה בתוך תא איתור Tetracysteine תג קיט * * * פלואורסצנטי ירוק עבור לגור תאים הדמיה | Invitrogen | T34561 (Flash) או T34562 (ReAsH) | |
| הנקס "פתרון מאוזן מלח | Invitrogen | 14175-103 | |
| 2,3-Dimercapto-1-propanol | סיגמא אולדריץ | D1129-5 מ"ל | |
| 1,2-Ethanedithiol | סיגמא אולדריץ | 02390-25ML |
References
- Tsien, R. Y. The green fluorescent protein. Ann. Rev. Biochem. 67, 509-544 (1998).
- Griffin, B. A., Adams, S. R., Tsien, R. Y. Specific covalent labeling of recombinant protein molecules inside live cells. Science. 281, 269-2672 (1998).
- Uttamapinant, C. A fluorophore ligase for site-specific protein labeling inside living cells. Proc. Natl. Acad. Sci. USA. 107, 10914-10919 (2010).
- Ignatova, Z., Gierasch, L. M. Monitoring protein stability and aggregation in vivo by real-time fluorescent labeling. Proc. Natl. Acad. Sci. USA. 101, 523-528 (2004).
- Coleman, B. M. Conformational detection of prion protein with biarsenical labeling and FlAsH fluorescence. Biochem. Biophys. Res. Commun. 380, 564-568 (2009).
- Luedtke, N. W., Dexter, R. J., Fried, D. B., Schepartz, A. Surveying polypeptide and protein domain conformation and association with FlASH and ReAsH. Nat. Chem. Biol. 3, 779-784 (2007).
- Ramdzan, Y. M. Conformation sensors that distinguish monomeric proteins from oligomers in live cells. Chem. Biol. 17, 371-379 (2010).
- Abramoff, M. agelhaes, PJ, S. J. R. am Image processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Hearps, A. The biarsenical dye Lumio exhibits a reduced ability to specifically detect tetracysteine-containing proteins within live cells. J. Fluor. 17, 593-597 (2007).
- Adams, S. R. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. J. Am. Chem. Soc. 124, 6063-6076 (2002).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Meth. 2, 905-909 (2005).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved