A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
אתר ספציפי הנדסה כרומוזום בקטריאלי: ΦC31 מתווכת integrase קלטת Exchange (IMCE)
In This Article
Summary
שיטה מהירה ויעילה לשלב דנ"א זר של עניין תוך מוכנות זנים נוטלים, המכונות משטח נחיתה זנים, מתואר. השיטה מאפשרת לאתר ספציפי לשילוב של הקלטת ה-DNA לתוך לוקוס משטח מהונדסים הנחיתה של זן מסוים, דרך הצמידה וביטוי integrase ΦC31.
Abstract
כרומוזום חיידקי ניתן להשתמש כדי לשמור על דנ"א זר ביציבות בטווח מגה בסיס 1. אינטגרציה לתוך הכרומוזום עוקף בעיות כגון שכפול הפלסמיד, יציבות הפלסמיד, אי התאמה פלסמיד, ושונות פלסמיד מספר עותק. שיטה זו משתמשת integrase באתר ספציפי של הפאג Streptomyces (Φ) C31 2,3. Integrase ΦC31 מזרז רקומבינציה ישירה בין שני אתרי DNA ספציפיים: attB ו attP (34 ו - 39 נ"ב, בהתאמה) 4. רקומבינציה זו יציבה ולא לחזור 5. "משטח נחיתה" (LP) הרצף המורכב של הגן spectinomycin עמידות, aadA (SPR), וכן א ' SS-coli glucuronidase גן (uidA) מוקף אתרים attP שולב הכרומוזומים של Sinorhizobium meliloti, Ochrobactrum anthropi, ו tumefaciens Agrobacterium באזור intergenic, אניPC לוקוס, ואת מוקד תטא, בהתאמה. ס meliloti נעשה שימוש בפרוטוקול זה. וקטורים התורמות Mobilizable המכילים אתרי attB משני צדי אדום stuffer חלבון פלואורסצנטי (RFP) גנים גן עמידות לאנטיביוטיקה יש גם לבנות. בדוגמה זו pJH110 גנטמיצין פלסמיד עמיד משמש. הגן RFP 6 עשוי להחליף את המבנה הרצוי באמצעות SPH אני Pst א לחלופין מבנה סינתטי מוקף אתרים attB יכול להיות תת משובטים לתוך וקטור mobilizable כגון pK19mob 7. הביטוי של הגן ΦC31 integrase (משובטים מ pHS62 8) הוא מונע על ידי האמרגן לאק, על מגוון רחב מארח mobilizable פלסמיד pRK7813 9.
פרוטוקול ההזדווגות tetraparental משמש להעברת קלטת התורם אל זן LP ובכך להחליף את סמני ברצף LP עם קלטת התורם. תאים אלה הם טראןS-integrants. טרנס integrants נוצרים ביעילות אופיינית של 0.5%. טרנס integrants נמצאות בדרך כלל בתוך 500-1,000 מושבות הראשונים שהוקרנו על ידי רגישות לאנטיביוטיקה או כחול לבן ההקרנה באמצעות 5-bromo-4-כלורו-3-indolyl-beta-D-glucuronic חומצה (X-gluc). פרוטוקול זה מכיל את ההזדווגות ובחירת נהלים ליצירה בידוד טרנס integrants.
Protocol
1. הפקה של תרבות
- הכן התקשורת נוזלי סטריליים: TY במילה 10 (5 גר '/ ליטר tryptone, 3 גר' / ליטר תמצית שמרים, 0.44 גר '/ ליטר כלוריד מייבשים סידן), וכן 11 LB (10 גר' / ליטר tryptone, 5 גר '/ ליטר תמצית שמרים, 5 גר' / נתרן כלורי, pH 7).
- לחסן מהמושבה אחת: SmUW227 (ס 'meliloti LP-זן: בנייה יתוארו במקום אחר, להתאמץ פרטים בנייה זמינים לפי בקשה) () לתוך 5 מ"ל של TY התקשורת עם מיקרוגרם 50 / spectinomycin מ"ל. לחסן את הזנים הבאים לתוך 5 מ"ל של התקשורת נוזלי LB, להשלים עם אנטיביוטיקה שניתנה: E. coli DH5α 12 המכיל pJC2, הביטוי integrase פלסמיד, עם 10 מיקרוגרם / מ"ל טטרציקלין, א ' coli MT616 13, mobilizer, עם 10 מיקרוגרם / מ"ל chloramphenicol, וא ' coli DH5α המכיל pJH110, קלטת התורם פלסמיד, עם 5 מיקרוגרם / מ"ל גנטמיצין.
- דגירה א ' קולי זנים לילה ב 37 &מעלות: C עם הרעידות קבוע. דגירה ס meliloti מאמץ על 30 מעלות צלזיוס במשך יומיים עם רעד. אפשר לקבל ש meliloti לילה עם הבידוד גדול יותר (1:500 תת התרבות רווי) תרבות.
2. תרבות הכנה ערבוב
- לשטוף 1.5 מ"ל של תרבות זן זה על ידי איסוף תאים גלולה ידי צנטריפוגה ב XG 17,000 במשך 30 שניות resuspending ב 1 מ"ל NaCl 0.85% סטרילי, חוזר פעם אחת.
- Resuspend גלולה ב 100 μl של NaCl 0.85% סטרילית.
- בנפרד לזהות 10 μl של תרבות שטוף עבור זן אחד על צלחת אגר רגיל TY. נקודות אלו הן נקודות השליטה.
- הוסף 40 μl של זן זה כולל א coli DH5α 12 המכיל pJC2 בצינור תערובת סטרילי, ו 120 במקום μl של תערובת זו על צלחת אגר רגיל TY. נקודה זו היא שליטה ללא integrase שלילית.
- הוסף 40 μl של כל זן לא סטריליתאובה, לערבב, ו במקום 160 μl של תערובת זו על צלחת אגר רגיל TY. במקום זה המקום ההזדווגות IMCE.
- אפשר נקודות לייבוש מכסה המנוע זרימה למינרית.
- חותם צלחות דגירה על 30 מעלות צלזיוס למשך הלילה.
3. הבידוד של טרנס integrants
- בקרת פס כתמים ספוט IMCE על גבי צלחות אגר TY במילה השלימו עם 200 מיקרוגרם / מ"ל סטרפטומיצין (SmUW227 מבוסס על Rm1021 שנושא מוטציה המקנה ברמה גבוהה התנגדות סטרפטומיצין 14) ו - 30 מיקרוגרם / מ"ל גנטמיצין.
- לצורך חישוב יעילות IMCE, resuspend כ ¼ של המקום ההזדווגות IMCE ב 500 μl NaCl 0.85% סטרילית. תן של פי 10 עד 10 דילולים סדרתי -7. דילולים פלייט -2 10 עד 10 -5 על צלחות אגר TY במילה השלימו עם 200 מיקרוגרם / מ"ל ו סטרפטומיצין 30 מיקרוגרם / מ"ל גנטמיצין, כדי לבחור את טרנס integrants. צלחת דילולים 10 thr -3ough 10 -7 על צלחות אגר TY במילה השלימו עם 200 מיקרוגרם / מ"ל סטרפטומיצין, כדי לבחור הן טרנס integrants ומקבלי פוטנציאליים, מקבלי בסך הכל כלומר.
- עבור בידוד של טרנס integrants markerless resuspend כרבע במקום הזדווגות IMCE ב μl 500 של NaCl 0.85% סטרילית. תן של פי 10 דילולים סדרתי עד 10 -6. צלחת דילולים 10 -4 עד 10 -6 על ארבע צלחות אגר TY במילה השלימו עם 200 מיקרוגרם / מ"ל ו סטרפטומיצין 200 מיקרוגרם / מ"ל X-gluc.
- דגירה צלחות על 30 מעלות צלזיוס במשך 3 ימים.
- צפייה RFP טרנס integrants תחת אור ירוק (525 ננומטר) ודרך פילטר אדום (> 610 ננומטר) 15.
- לחשב את יעילות IMCE אחוזים CFU של טרנס integrants לעומת CFU מכלל המקבלים. כמחצית טרנס integrants לא עברו חילופי קלטת נכון מה שהופך אותם לבן spectinomycin רגיש.
- כדי לחפש את markerless טרנס integrants (Wכאן וקטור התורם אינו מכיל GM r כמו pJH110) מסך מושבה לבן (תקופת הדגירה ממושכת, 1-2 ימים נוספים בטמפרטורת החדר, ישפר את הבידול של המושבות באמצעות X-gluc, זה הכרחי ש ' meliloti SmUW227), לאשר את הרגישות spectinomycin של המושבה על ידי הקרנה על צלחת אגר TY ללא אנטיביוטיקה צלחת אגר TY המכיל 100 מיקרוגרם / מ"ל spectinomycin.
4. נציג תוצאות
אחרי שלושה ימים של דגירה על TY השלימו עם סטרפטומיצין ו גנטמיצין את הפסים שליטה צריך שום צמיחה. פס IMCE צריך הצמיחה confluent על פס הראש מושבות רבות על פס 2, כפי שניתן לראות באיור 2. את היעילות של שילוב, טראנס, מבוטא באחוזים של טרנס integrants לנמענים הכל, צריך להיות בטווח של 0.5%. כמחצית טרנס integrants יהיה spectinomycin רגיש לבןמראה שהם עברו חילופי קלטת נכון. טרנס integrants המכילים את הקלטת RFP הבוחן תורם pJH110 צריך להציג להבחין הקרינה RFP כאשר הוא מוצג באור ירוק (525 ננומטר) ועל דרך מסנן אדום (> 610 ננומטר) 15.
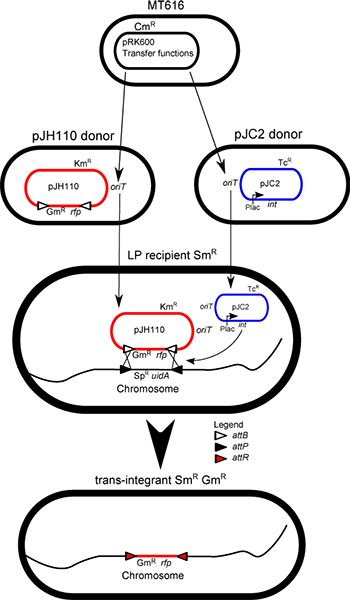
1. דמות הצמידה תערובת איור: בעזרת הביטוי של הגנים העברה pRK600, כל פלסמידים מועברים באופן אקראי מתא לתא. העברת התוצאה היא יצירה של טרנס integrants בתערובת, באמצעות רכישה של LP-זן של שני פלסמידים הנדרשים IMCE, integrase (int) pJC2 עוזר הפלסמיד ואת pJH110 התורם פלסמיד. קלטת התורם מתורם ללא שכפול הפלסמיד (pJH110) הוא החליף אגב פעילות ΦC31 integrase עם סמנים של קלטת LP-על כרומוזום, וכתוצאה מכך אובדן של LP-סמנים (פ"מR ו uidA) ושמירה על קלטת התורם (RFP ו-GM R) ב שהתקבל חוצה integrant.
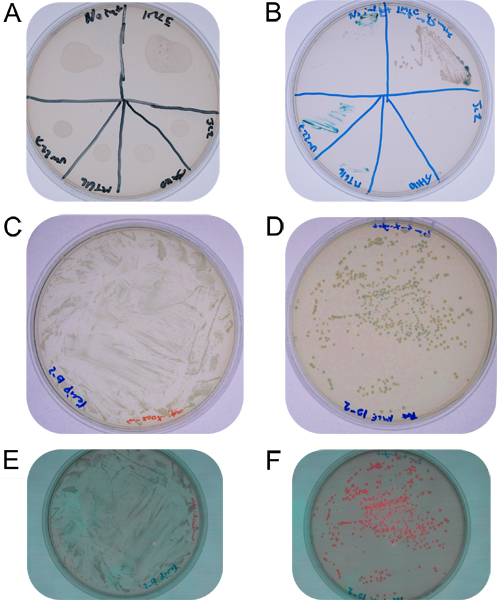
איור 2. : B במקום הזדווגות צלחת על צלחת TY הלא סלקטיבי מראה תערובות תאים יבשים על פני אגר:. נקודות הזדווגות מפוספסים בצלחת סטרפטומיצין גנטמיצין-X-gluc-, מלמעלה בכיוון השעון נשאר כל שליטה integrase, IMCE אל ס meliloti, א coli DH5α המכיל השליטה pJC2, א coli DH5a המכיל pJH110 שליטה, א ' MT616 השליטה coli, וס ' . meliloti UW227 השליטה C: 10 -2 דילול resuspension במקום הזדווגות על סטרפטומיצין TY-X-gluc אגר D:. 10 -2 דילול resuspension במקום הזדווגות על אגר סטרפטומיצין, גנטמיצין, X-gluc TY (מושבות כחולות הם recombinants בודדים, מושבות לבנים עברו האמיתי. החלפת קלטת) E: 10 -2 דילול resuspension ההזדווגות כתם על אגר סטרפטומיצין-X-gluc TY מראה חוסר הקרינה F:. 10 -2 דילול resuspension במקום הזדווגות על אגר סטרפטומיצין, גנטמיצין, X-gluc TY מראה שתי רמות של הקרינה (מושבות בהירים מתאימות מושבות כחולות יש יותר ביטוי RFP ככל הנראה בשל מקדם לקריאה אם כי מתוך רצף וקטור, שם מושבות שעברו נכון, הקלטת חילופי מכילים RFP עם האמרגן רק המיידית ללא לקריאה באמצעות האמרגן של לאק ב וקטור, אשר נעדר.).
Access restricted. Please log in or start a trial to view this content.
Discussion
הטכניקה IMCE מאפשר שילוב יעיל של קלטת ה-DNA בודד מוקף attB לתוך לוקוס-LP של זן מהונדס בעבר. ברגע מבנה הרצוי משובט במקום RFP ליצור את הקלטת התורם, הטכניקה אינה דורשת טיהור שינוי ה-DNA עתידיים, מה שהופך אותו מאוד חזקים. זה קריטי, כי בקרות מתאימות הצמיחה כלולות, כדי להיות ב?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
אין ניגוד עניינים הצהיר.
Acknowledgements
כדי מרגרט סי סמית עבור חביב מתן שיבוט integrase
גיוס תמיכה של:
הגנום קנדה / הגנום Prairie
NSERC גילוי מענקים פרויקט אסטרטגי
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| שם מגיב | חברה | מספר קטלוגי | תגובות |
| סטרפטומיצין | Bioshop קנדה בע"מ | STP101 | |
| Spectinomycin | Bioshop קנדה בע"מ | SPE201 | |
| גנטמיצין | Bioshop קנדה בע"מ | GTA202 | |
| Choramphenicol | Bioshop קנדה בע"מ | CLR201 | |
| טטרציקלין | Bioshop קנדה בע"מ | TET701 | |
| Kanamycin | Bioshop קנדה בע"מ | KAN201 | |
| אגר כיתה בקטריולוגית | Bioshop קנדה בע"מ | AGR001 | |
| Tryptone | Bioshop קנדה בע"מ | TRP402 | |
| תמצית שמרים | Bioshop קנדה בע"מ | YEX401 | |
| סודיום כלוריד | Bioshop קנדה בע"מ | SOD001 | |
| סידן כלוריד | Bioshop קנדה בע"מ | CCL444 | |
| X-gluc | גולד ביוטכנולוגיה בע"מ | G1281C1 | |
| E. coli MT616 זן | ניתן להזמין | משמש גם מחוץ למעבדה שלנו | |
| E. coli pJC2 זן | בבית, זמין לפי בקשה | ||
| E. coli pJH110 זן | בבית, זמין על ידי requesלא | ||
| SmUW227 זן | בבית, זמין לפי בקשה |
References
- Itaya, M., Tsuge, K., Koizumi, M., Fujita, K. Combining two genomes in one cell: Stable cloning of the Synechocystis PCC6803 genome in the Bacillus subtilis 168 genome. Proc. Natl. Acad. Sci. U.S.A. 102, 15971-15976 (2005).
- Kushtoss, S., Rao, R. N. Analysis of the Integration Function of the Streptomycete Bacteriophage FC31. J. Mol. Biol. 222, 897-908 (1991).
- Brown, W. R., Lee, N. C., Xu, Z., Smith, M. C. Serine recombinases as tools for genome engineering. Methods. 53, 372-379 (2011).
- Groth, A. C., Olivares, E. C., Thyagarajan, B., Calos, M. P. A phage integrase directs efficient site-specific integration in human cells. Proc. Natl. Acad. Sci. U.S.A. 97, 5995-6000 (2000).
- Rowley, P. A., Smith, M. C., Younger, E. A motif in the C-terminal domain of FC31 integrase controls the directionality of recombination. Nuc. Acid. Res. 36, 3879-3891 (2008).
- Campbell, R. E. A monomeric red fluorescent protein. Proc. Natl. Acad. Sci. U.S.A. 99, 7877-7882 (2002).
- Schafer, A. Small mobilizable multi-purpose cloning vectors derived from the Escherichia coli plasmids pK18 and pK19: selection of defined deletions in the chromosome of Corynebacterium glutumicum. Gene. 145, 69-73 (1994).
- Thorpe, H. M., Smith, M. C. In vitro site-specific integration of bacteriophage DNA catalyzed by a recombinase of the resolvase/invertase family. Proc. Natl. Acad. Sci. 95, 5505-5510 (1998).
- Jones, J. D., Gutterson, N. An efficient mobilizable cosmid vector, pRK7813, and its use in a rapid method for marker exchange in Pseudomonas fluorescens strain HV37a. Gene. 61, 299-306 (1987).
- Beringer, J. E. R Factor transfer in Rhizobium leguminosarum. J. Gen. Microbiol. 84, 188-198 (1974).
- Lennox, E. S. Transduction of linked genetic characters of the host by bacteriophage P1. Virology. 1, 190-206 (1955).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. J. Mol. Biol. 166, 557-580 (1983).
- Charles, T. C., Finan, T. M. Genetic map of Rhizobium meliloti megaplasmid pRmeSU47b. J. Bacteriol. 172, 2469-2476 (1990).
- Leigh, J. A., Signer, E. R., Walker, G. C. Exopolysaccharide-deficient mutants of Rhizobium meliloti that form ineffective nodules. Proc. Natl. Acad. Sci. U.S.A. 82, 6231-6235 (1985).
- Heil, J. R., Nordeste, R. F., Charles, T. C. The fluorescence theatre: a cost-effective device using theatre gels for fluorescent protein and dye screening. Can. J. Microbiol. 57, 339-342 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-6645 (2000).
- Lesic, B., Rahme, L. G. Use of the lambda Red recombinase system to rapidly generate mutants in Pseudomonas aeruginosa. BMC Mol. Biol.. 9, 20-20 (2008).
- Choi, K. -H., Schweizer, H. P. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nat. Protocols. 1, 153-161 (2006).
- Thomason, L. C., Calendar, R., Ow, D. W. Gene insertion and replacement in Schizosaccharomyces pombe mediated by the Streptomyces bacteriophage FC31 site-specific recombination system. Molecular Genetics and Genomics. 265, 1031-1038 (2001).
- Katzen, F. Gateway recombinational cloning: a biological operating system. Expert Opin. Drug Discovery. , 571-586 (2007).
- Charles, T. C., Doty, S. L., Nester, E. W. Construction of Agrobacterium strains by electroporation of genomic DNA and its utility in analysis of chromosomal virulence mutations. Appl. Environ. Microbiol. 60, 4192-4194 (1994).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved