A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הכרומטין immunoprecipitation (שבב) באמצעות תסיסנית רקמה
In This Article
Summary
לאחרונה תפוקה גבוהה טכנולוגיית ריצוף גדלה מאוד הרגישות של הכרומטין immunoprecipitation (שבב) הניסוי מתבקש יישומו באמצעות תאים או רקמות גזור מטוהרים. כאן אנו קובעים שיטה להשתמש בטכניקה שבב עם תסיסנית, אשר יכול להתמודד עם מצב הכרומטין אנדוגני במערכת הביולוגית היטב מאופיין.
Abstract
אפיגנטיקה נשאר בתחום המתפתח במהירות כי מחקרים איך המדינה הכרומטין תורם ביטוי ההפרש גנים סוגי תאים שונים בשלבים התפתחותיים שונים. תקנה epigenetic תורם ספקטרום רחב של תהליכים ביולוגיים, כולל התמיינות במהלך ההתפתחות העוברית הומאוסטזיס בבגרות. אסטרטגיה קריטית במחקרים אפיגנטים המשנים היא לבחון כיצד שינויים היסטון שונים וגורמים הכרומטין לווסת את ביטוי הגנים. לכתובת זו, הכרומטין immunoprecipitation (שבב) נעשה שימוש נרחב כדי לקבל תמונת מצב של הקשר של גורמים מסוימים עם ה-DNA בתאים של עניין.
הטכניקה שבב כלל משתמשת בתאי תרבית חומר המוצא, אשר ניתן להשיג בשפע והומוגניות להפיק נתונים לשחזור. עם זאת, ישנם מספר תנאים: ראשית, הסביבה לגדול תאים בצלחת פטרי שונה מזה in vivo, ולכן עשוייםלא משקפים את מצב הכרומטין אנדוגני של התאים בכל יצור חי. שנית, לא כל סוגי התאים יכולים להיות vivo לשעבר תרבותי. יש רק מספר מוגבל של שורות תאים, אשר אדם יכול להשיג מספיק חומר assay שבב.
כאן אנו מתארים שיטה לעשות ניסוי שבב באמצעות רקמות תסיסנית. חומר המוצא היא גזור רקמה מן החי, ובכך יכול לשקף במדויק את מצב הכרומטין אנדוגני. ההסתגלות של שיטה זו עם סוגים שונים של רקמות יאפשר לחוקרים להתייחס הרבה יותר מבחינה ביולוגית שאלות רלוונטיות לגבי רגולציה epigenetic ב 1 vivo, 2. שילוב שיטה זו עם רצף תפוקה גבוהה (שבב ואילך) יהיה עוד יותר לאפשר לחוקרים לקבל הנוף epigenomic.
Protocol
(הליך שבב כולו אורך כ 2 ימים. הכנת ספריות השבבים עבור רצף תפוקה גבוהה לוקח עוד 2-3 ימים.)
1. לנתח ולהכין רקמות לקראת ניסוי שבב (~~~HEAD=NNS 1 מיליון תאים)
- לנתח רקמות עניין (למשל 200 זוגות של תסיסנית האשכים) בקור מעכב פרוטאז PBS 1x + (1 לפרק גלולה של קוקטייל מעכבי פרוטאז ב 1.5 מ"ל 1x PBS להשיג פתרון המניות 7x) + PMSF (הריכוז הסופי 100 מיקרוגרם / מ"ל). יש לשטוף את רקמת פעמיים resuspend רקמות μL 200 הפתרון PBS גם עם מעכבי.
- כדי לתקן את המדגם, להוסיף 5.5 פורמלדהיד 37% μL (פורמלדהיד חם 37 ° C אמבט מים 1 דקות לפני השימוש). דגירה על 37 מעלות צלזיוס במשך 15 דקות, מערבולת כל 5 דקות ביניהן.
- מניחים את המדגם על קרח רקמות להתפשר על 2 דקות. יש לשטוף את פי 2 עם 450 PBS μL (מעכבי פרוטאז עם ו PMSF). לדוגמא יכול להיות בחנותD ב -20 ° C. חזור על פי הנתיחה מספר להשיג מספיק חומר על שבב.
2. הכן Supernatant עם נטיה חלבון-DNA על שבב Assay
- שלב מדגם מספיק צעד 1.3 ב צינור 1.75 eppendorf מ"ל.
- הסר PBS ממדגם רקמות. הוספת 200 μL מאגר תמוגה (50 מ"מ טריס-HCl, pH 7.6, 1 mM CaCl2, 0.2% טריטון X-100 או NP-40, 5 בוטיראט מ"מ, 1x מעכב קוקטייל proteinase). הוסף טרי פתרון PMSF המניות למאגר תמוגה (ריכוז PMSF הסופית של 0.5 מ"מ). Homogenize בכחול homogenizer (חתול פישר # 749521-1590) עד רקמות לנתק לחלוטין ללא צבירה כלשהו, דגירה על RT (טמפרטורת החדר) במשך 10 דקות.
- Sonication: microtip שימוש sonicator (Misonix HS-XL200) בהספק 20. Sonicate במשך 10 שניות ולנוח על הקרח עבור 50 שניות. חזור על 4-5 פעמים (בפעם sonication האופטימלי יש לקבוע באופן אמפירי, כמו sonication עוד תניב שברים קטנים יותר. לצ'יפ באמצעות נוגדנים נגד Histonשינויים אלקטרוניים, אנחנו בדרך כלל sonicate הכרומטין כ 200-300 נ"ב: כי גורם שעתוק או הרגולטור הכרומטין (כגון חלבונים Polycomb), אנו בדרך כלל sonicate הכרומטין כדי 200-1000 bp. עם זאת, גודל שבר אופטימלית יש לבדוק באופן אמפירי. הערה: תמיד מקיימים את TUBE ON ICE גם במהלך SONICATING למניעת מדגם חימום UP!
- לדלל מדגם ידי הוספת 1.8 מ"ל Ripa חיץ (10 mM Tris-HCl, pH 7.6, 1 mM EDTA, 0.1% SDS, 0.1% Na-Deoxycholate, טריטון 1% X-100, עם מעכבי פרוטאז ו PMSF בריכוז זהה, כמתואר קודם לכן). שמור 40 μL כמו בקרת קלט על ידי הוספת 2 5M NaCl μL ו דגירה על 65 מעלות צלזיוס למשך הלילה (O / N) כדי להפוך crosslink.
- כדי להטות נוגדנים חרוזים, להוסיף 40 חלבון μL כמה חרוזים כדי צינור 1.5 eppendorf מ"ל, ואז מוסיפים 600 PBS μL ו אבן על 4 מעלות צלזיוס למשך 2 דקות, כדי להחיל חרוזי מגנט ולהסיר supernatant. הוספת 100 μL של PBS ואת הנוגדנים של עניין (כמות הנוגדנים יש לקבוע empirically). על דגירה RT שעה 1 או 4 מעלות צלזיוס למשך 4 שעות.
- הסר supernatant מן החרוזים על ידי מגנט ולהוסיף תמצית הכרומטין 1mL משלב 2.4 על חרוזים. סיבוב ב 4 ° CO / נ
- החל מדגם צ'יפ מגנט ולהסיר supernatant. לשטוף את החרוזים עם למאגר הבאה ב 4 מעלות צלזיוס במשך 10 דקות כל אחד:
פי 2 עם 1 מ"ל של Ripa חיץ [1.89 "Ripa חיץ" 315 מ"ל + מעכבי פרוטאז μL 7x + 20 PMSF μL];
פי 2 עם 1 מ"ל של Ripa חיץ + 0.3 מ 'NaCl [1.89 מ"ל Ripa חיץ + 220 μL 3 מ' NaCl];
פי 2 עם 1 מ"ל של חיץ LiCl (0.25 מ 'LiCl, 0.5% NP40, NaDOC 0.5%);
1x עם 1 מ"ל של 1x TE + 0.2% טריטון X-100;
1x עם 1 מ"ל של 1x TE. - כדי להפוך crosslink, resuspend את החרוזים במאגר TE 100 μL + 3 μL 10% SDS + 5 μL של proteinase K (20 מ"ג / מ"ל). על דגירה 65 ° CO / נ
- החל חרוזים מגנט ולהעביר supernatant לתוך צינור חדש. SUPERNATANT מכיל דגימת DNA שלך. לשטוף עם חרוזים 100 μL TE + 0.5 מ ' NaCl ולשלב supernatant 2.
- פנול, כלורופורם לחלץ את דגימת DNA. הוספת 200 פנול μL: Chlorofrom: רשות העתיקות (25:24:1) לערבב מערבולת. מסובבים 14K במשך 5 דקות בכל RT. לחלופין, אפשר להפיק DNA באמצעות PCR Qiagen ערכת טיהור.
- מעבירים את שכבת supernatant / מימית לתוך צינור חדש, ולהוסיף acrylamide ליניארי לריכוז סופי של 20 מיקרוגרם / מ"ל (1 לחלופין μL של גליקוגן ב 20 מ"ג / מ"ל 1 מ"ל של כל supernatant יכול לשמש גם. גליקוגן משמש QPCR לאחר ניתוחים או ה-PCR בזמן ליניארי acrylamide משמש רצף מבחני.) הוסף 20 μL של 3M NaOAc ו 500 EtOH 100% μL. מערבבים היטב דגירה על 80 מעלות צלזיוס במשך 10 דקות. להסתובב במהירות מקסימלית במשך 20 דקות 4 ° C.
- הסר supernatant ולשטוף עם EtOH 300 70% μL. האוויר מדגם יבש resuspend במאגר 50 TE μL. לדוגמא כעת ניתן לאחסן ב -20 ° C. לדוגמה ניתן להשתמש assay כמותית (QPCR) PCR (תרשים 1).
= "Jove_title"> 3. נתח את שבב ה-DNA אד
3 א. נתח את שבב ה-DNA באמצעות עורך QPCR
- לדלל הדנ"א הגנומי בריכוז הבאה לעשות עקומת רגיל: חי, 1/10, 1/100, 1/1000, 1/5000.
- עבור כל זוג פריימר, להגדיר QPCR בשני עותקים בצלחת 96-היטב עם הדנ"א הגנומי של 3a.1 צעד, בקרת קלט שבב אד ה-DNA:
10 μL 2x SYBR PCR תערובת (Fermentas, K0222);
1 μL 10 מיקרומטר קדימה פריימר;
1 μL 10 מיקרומטר לאחור תחל;
X-DNA μL שבב אד (בקרת כניסה, או הדנ"א הגנומי);
y nuclease ללא μL H 2 O כדי לכוונן את עוצמת התגובה μL 20. - לסובב את הצלחת היטב עם 96-Mini צלחת הצנטריפוגה (Labnet).
- בצע QPCR עם ABI 7300 בזמן אמת PCR המערכת (או אחרים בזמן אמת למערכות ה-PCR), תוך שימוש בתנאי הבא:
שלב 1: 50 מעלות צלזיוס למשך 2 דקות, מחזור 1;
שלב 2: 95 מעלות צלזיוס במשך 10 דקות, 1 מחזור;
שלב 3: 95 מעלות צלזיוס למשך 15 שניות, 60 מעלות צלזיוס במשך דקות 1, 40 גycles;
שלב 4 (שלב דיסוציאציה): 95 מעלות צלזיוס במשך 15 S, 60 ° C עבור 1 דקות, 95 מעלות צלזיוס למשך 15 שניות. - בדוק את עקומת דיסוציאציה: אחת השיא ב העלילה ניתוק תרמי מציע amplicon אחד מתגובה PCR. יותר מ 1 שיא מציין שאינו ספציפי המוצר עם זוג פריימר, הנתונים QPCR אין להשתמש.
- לקבוע את שלב ליניארי של הגברה מעריכי התגובה PCR עבור כל קבוצה תחל, על ידי חישוב נוסחה פותר כמות ה-DNA על פי ערך ה-CT (סף מחזור), המבוססת על עקומת רגיל באמצעות שורה של הדנ"א הגנומי בדילול מלא 3 א. 1.
- אם הערך CT של ה-DNA שבב אד ובקרה קלט נמצאים בטווח הליניארי של הערך CT, לחשב את העשרת של ה-DNA שבב אד לעומת קלט, על פי גורם דילול של ה-DNA שבב אד ושליטה תשומה 2.4.
3 ב. להגביר את שבב ה-DNA של אד רצף קצב העברת נתונים גבוה.
- סוף תיקון ה-DNA שילוב שבב אד (שימושEpicentre DNA קצה ערכת תיקון), להקים על הקרח:
1-34 DNA הגנומי μl משלב 2.12 (0.1-5 מיקרוגרם)
5 תיקון μl 10x סוף חיץ
5 μl 2.5 dNTP תערובת mM
5 μl 10 mM ATP
μl x H 2 O כדי לכוונן את עוצמת התגובה 49 μl
1 אנזים μl קצה תיקון תערובת (DNA פולימראז T4 + קינאז Polynucleotide T4)
דגירה במשך 45 דקות בכל RT. לטהר את התגובה באמצעות תגובה Qiagen MinElute
ערכת ניקוי. Elute במאגר elution 30 μl (EB). - הוסף את A-overhangs עד הסוף '3:
30 μl של מוצר ה-DNA eluted מ 3b.1 צעד
מאגר μl 5 חוד 10x # 2
10 μl 1 mM dATP
2 μl DH 2 O
3 μl 5 U / μl שבר Klenow (3 '→ 5' exo-)
דגירה של 30 דקות בשעה 37 ° C. לטהר את התגובה באמצעות ערכת MinElute ReactionCleanup. Elute ב EB 10 μl. - Solexa מקשר קשירת:
10 μl של ה-DNA eluted מ 3b.2 צעד
10 μl DDH 2 O
2.5 &MU, 10x אני חיץ T4 DNA ligase
1 μl מתאם oligo תערובת מ Illumina (1/10-diluted ממלאי)
2.5 ligase μl DNA T4 (400 יחידות / μl)
דגירה שעה 1 ב RT. לטהר את התגובה באמצעות ערכת MinElute ReactionCleanup. Elute ב EB 20 μl. לדוגמא כעת ניתן לאחסן ב -20 ° C. - הפעל את דגימת DNA eluted מ 3b.3 צעד באמצעות מנגנון ה-ג'ל. לבודד את הלהקה 300-500 נקודות בסיס ב ג'ל ולטהר באמצעות ערכת ג'ל Qiagen הוצאה (צעד זה מבטל את ~~~HEAD=NNS 125-BP עצמית ligated linkers מ קשירת מתאם oligo). Elute ב EB 12 μl.
- להגביר באמצעות זיווג לקצה הספרייה (PE) primers מ Illumina:
10.5 μl של ה-DNA eluted מ 3b.4 צעד
12.5 μl של שילוב שני (2X Phusion HF, Finnzymes)
1 μl של פריימר PCR PE1 1
1 μl של פריימר PCR PE2 2
PCR מצב:
98 מעלות צלזיוס למשך 30 שניות
(98 ° C 10 שניות, 65 מעלות צלזיוס 30 שניות, 72 מעלות צלזיוס 30 שניות), חזור על 20 מחזורי
72 ° C במשך 5 דקות. - דגימות 3b.5 צעד ניתן להשתמש SEQ-Chip לאחר בחירת ה-DNA בגודל המתאים (300-500 BP) באמצעות תקן 2% agarose ג'ל. לדוגמא כעת ניתן לאחסן ב -20 ° C. עדיף לבודד כל מדגם שבב על ג'ל אחד על מנת למנוע זיהום. דוגמאות ניתן להגיש עבור סידור תפוקה גבוהה.
3 ג. Solexa ניתוח צינור
- 25-BP רצף קורא התקבלו הגנום Illumina Analyzer (GA) צינור.
- כניסות כולם מיושר הגנום תסיסנית (dm3) באמצעות בלאנד (יישור מקומי יעיל של נתונים נוקלאוטידים) תוכנה, המאפשר עד שתי חוסר התאמה עם רצף ההתייחסות.
- כניסות ממופה באופן ייחודי נשמרו לניתוח במורד הזרם. עבור כניסות זהים מרובים, עד 3 עותקים נשמרו כדי לצמצם את האפשרות של הטיות של הגברה PCR.
- התפוקה של צינור ה-GA הוסב להרחבה נתונים הדפדפן (מיטה) קבצים.
- כדי ליצוראת הקבצים הפאות עבור העלאת לדפדפן UCSC להדמיה, השתמשנו שטר פיתון אשר תוארה בפרסום הקודם 3 עם 4 נקודות בסיס כמו גודל החלון ו -160-BP כגודל שבר דנ"א.
- לסווג גנים תסיסנית לתוך גנים דוממים לידי ביטוי, השתמשנו במספר דיגיטלית בשם RPKM (קורא / דולר kilobase התמזגה באזור exonic / ממופה למיליון כניסות). גנים עם RPKM = 0 סווגו לקבוצה גנים שקט עם RPKM ≥ 1 סווגו לקבוצות לידי ביטוי, אשר ניתן לסווג לשלוש קבוצות כמו גנים עם רמות ביטוי נמוכות, בינוני וגבוה, וכל קבוצה יש על אותו מספר גנים.
- כדי להשוות את רמת שינוי היסטון ועל ביטוי גנים, השתמשנו סקריפט פייתון אשר תוארה ב 3 הפרסום הקודם.
4. נציג תוצאות
דוגמאות של שבב QPCR התוצאות באמצעות בום (שקית של גולות) מוטציה האשכים מוצגות באיור 1 4. ב בום האשכים, יש כשל במעבר spermatogonia שגשוג כדי להבדיל spermatocytes 5, 6. אנחנו משתמשים בם האשכים כמקור בתאי הנבט שלא עברו התמיינות, אשר מועשר זה סוג הרקמה. גנים בידול נדרש בידול הזרע, כגון זכר גורם ספציפי שעתוק 87 (mst87F), דון חואן (DJ), ובצל מטושטשת (fzo) לא באים לידי ביטוי בום האשכים. גנים אלה מועשר עם שינויים H3K27me3 הדיכוי היסטון 7 (איור 1 א), אבל הם ללא שינוי H3K4me3 פעיל היסטון 8 (איור 1 ב), חתימה הכרומטין קראנו 7 "חד ערכי". העשרת או H3K27me3 או H3K4me3 נקבע על ידי נורמליזציה כדי cyclin הביע constitutively (CycA) גן 4.
"תוכן> שבב seq ניתוח באמצעות אותה קבוצה של נוגדנים (כלומר H3K27me3 הדיכוי ואת H3K4me3 פעיל) עם בום האשכים מוטציה (איור 2) תוקף את התוצאות QPCR שמוצג באיור 1. לתקופה של שלושה גנים בידול הנבדקים מסוף mst87F, DJ ו fzo , אזורים הגנומי שלהם מועשר עם H3K27me3 אך לא H3K4me3 (איור 2 א, 2 ג). לעומת זאת, הגן הביעו constitutively CycA יש H3K4me3 משמעותי אך מעט H3K27me3 מחייב סמוך לאתר שלה תחילת השעתוק (TSS) (איור 2 ד).יתר על כן, את הפרופילים השבב של H3K27me3 ו H3K4me3 ליד TSSs של ארבע כיתות של גנים לידי ביטוי באופן דיפרנציאלי עולים בקנה אחד עם הפונקציה של שינוי כל היסטון. כפי שניתן לראות באיור 3 א, העשרת H3K27me3 במורד הזרם של TSSs קשורה ביחס הפוך עם רמת ביטוי גנים. הגנים שקטים יש H3K27me3 הגבוה ביותר ואילוביטוי גנים ביותר אין מחייב H3K27me3. נתונים אלה עולה בקנה אחד עם התפקיד המדכא של H3K27me3 על ביטוי גנים. לעומת זאת, העשרת H3K4me3 סביב TSSs הראו קורלציה הפוכה עם רמת ביטוי גנים (איור 3 ב), עולה בקנה אחד עם תפקיד פעיל של H3K4me3 על ביטוי גנים.
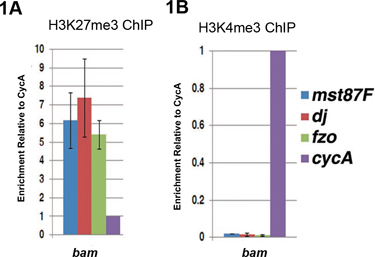
1. איור QPCR ניתוח ה-DNA שבב אד באמצעות נוגדנים נגד או שינוי H3K27me3 הדיכוי היסטון (א) או שינוי פעיל H3K4me3 היסטון (ב) האשכים מוטציה שלא עברו התמיינות התא מועשר בם. (א) בם האשכים, גנים בידול (Mst87F, DJ, fzo) מועשרים עם שינויים H3K27me3 היסטון הדיכוי. (ב) בידול הגנים מתרוקנים עם סימן H3K4me3 פעיל. ברמת ה-DNA שבב (DNA שבב / קלט) בשעה הגן היעד (Mst87F, DJ או fzo) היה מנורמל הראשון לב אל ה-DNA על שבב גנטי השליטה CycA. ברים שגיאה עולה סטיית התקן של 3 ביולוגית עצמאית משכפל.
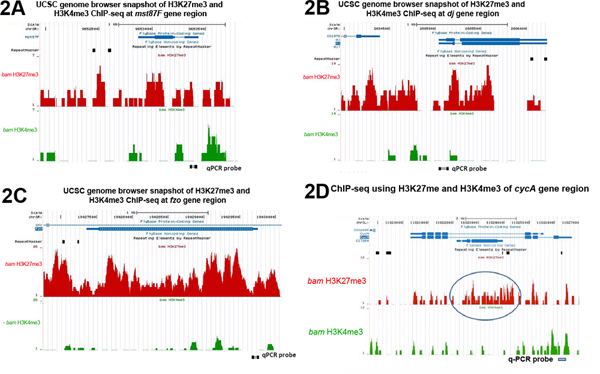
איור 2. UCSC תמונות הגנום של הדפדפן H3K27me3 ו H3K4me3 העשרה על פני אזורים שלמים של גנומית () Mst87F (ב ') DJ ו (ג) fzo, (ד) גנים CycA. הקיף H3K27me3 מועשר האזור (ד') משקף את מעמדו של הכרומטין הגן חופפים CG7264, אשר נחות לידי ביטוי בם האשכים (RPKM = 1) אך הביע מאוד wild-type האשכים (RPKM = 130) 8 (שבב seq נתונים 7). בדיקות PCR נעשה שימוש בניתוח כמותי של תוצאות שבב באיור 1 מסומנים בחלק התחתון של כל חלקה. לחץ כאן כדי להציג דמות גדולה .
tp_upload/3745/3745fig3.jpg "/>
איור 3. שבב seq פרופילי שימוש H3K27me3 ו H3K4me3 ב 7 בום האשכים. ארבע קבוצות גנטיות (7,509 גנים) סווגו לפי רמות ביטוי גנים שלהם על בסיס RNA-seq תוצאות 8. השיעורים מייצגים של גנים הם זממו להעשרה של שינוי היסטון בפרט, באמצעות רצפי מן הזרם כדי 3KB במורד 3KB מתחילת השעתוק שלהם באתרים (TSSs). זה יוצר פרופיל של העשרת () H3K27me3 (K27) ו (ב) H3K4me3 (K4) שבב seq ניתוחים בכל קבוצה. לחץ כאן כדי להציג דמות גדולה .
Access restricted. Please log in or start a trial to view this content.
Discussion
צדדיות של ניתוחים שבב שנדונו פרוטוקול זה יכול לשמש על רקמות שונות, אשר מספק הזדמנות לבחון את מצב הכרומטין במערכת רלוונטיות מבחינה ביולוגית. ניסויים שבב באמצעות תאים בתרבית ממערכות נוח לבצע בגלל כמות גדולה של תאים ניתן להשיג בקלות. עם זאת, תאים בתרבית אינן משקפות בהכר...
Access restricted. Please log in or start a trial to view this content.
Disclosures
אין לנו מה למסור.
Acknowledgements
המחברים מבקשים להודות מעבדה ד"ר Keji של ג'או (NIH / NHLBI) על עזרתם במתן תוצאות רצף. אנו רוצים גם להודות פרויקט הגנום UCSC לשימוש של דפדפן הגנום לדמיין רצף ממופה קורא.
עבודה זו נתמכה על ידי מסלול R00HD055052 NIH להעניק העצמאות R01HD065816 מ NICHD, Parkard לוסיל קרן, ואת ג 'ון הופקינס סטארט אפ מימון XC
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| שם מגיב | חברה | מספר קטלוגי | תגובות |
| מעכבי פרוטאז השלם מיני קוקטייל | רוש | 11836153001 | |
| פורמלדהיד (37%) | Supelco | 47083-U | |
| PMSF | סיגמא | 78830 | |
| Kontes גלולה העלי | פישר סיינטיפיק | K749521-1590 | |
| PCR ערכת טיהור | Qiagen | 28104 | |
| יישור קווי polyacrylamide | סיגמא | 56575-1ML | |
| גליקוגן | Qiagen | 158930 | |
| SYBR ירוק / רוקס QPCR מיקס מאסטר | Fermentas | K0223 | |
| מיני הצלחת טווה | Labnet | Z723533 | |
| בזמן אמת, מערכת ה-PCR | יישומי Biosystem | 4351101 | |
| נפח מעבד קטן Ultrasonic | Misonix | HS-XL2000 | דגם הופסק |
| Dynabeads, חלבון | Invitrogen | 100-01D | |
| Dynamag מגנט | Invitrogen | 123-21D | |
| פנול: Chlorofrom: רשות העתיקות | Invitrogen | 15593-049 | |
| Epicentre DNA קצה ערכת תיקון | Epicentre ביוטכנולוגיות | ER0720 | |
| MinElute Reaction ערכת ניקוי | Qiagen | 28204 | |
| Klenow שבר (3 '→ 5' exo-) | ניו אינגלנד Biolabs | M0212S | |
| T4 DNA ligase | Promega Corporation | M1794 | |
| מתאם oligonucleotides | Illumina | PE-400-1001 | |
| יחד סוף פריימר 1.0 ו 2.0 | Illumina | 1001783 1001 784 | |
| E-Gel Electorphoresis מערכת | Invitrogen | G6512ST | |
| 2X Phusion HF Mastermix | Finnzymes | F-531 |
References
- Kharchenko, P. V., Alekseyenko, A. A., Schwartz, Y. B., Minoda, A., Riddle, N. C., Ernst, J., Sabo, P. J., Larschan, E., Gorchakov, A. A., Gu, T. Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480-485 (2011).
- Filion, G. J., van Bemmel, J. G., Braunschweig, U., Talhout, W., Kind, J., Ward, L. D., Brugman, W., de Castro, I. J., Kerkhoven, R. M., Bussemaker, H. J., van Steensel, B. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Barski, A., Cuddapah, S., Cui, K., Roh, T. Y., Schones, D. E., Wang, Z., Wei, G., Chepelev, I., Zhao, K. High-resolution profiling of histone methylations in the human genome. Cell. 129, 823-837 (2007).
- Chen, X., Lu, C., Prado, J. R., Eun, S. H., Fuller, M. T. Sequential changes at differentiation gene promoters as they become active in a stem cell lineage. Development. 138, 2441-2450 (2011).
- Gonczy, P., Matunis, E., DiNardo, S. bag-of-marbles and benign gonial cell neoplasm act in the germline to restrict proliferation during Drosophila spermatogenesis. Development. 124, 4361-4371 (1997).
- McKearin, D. M., Spradling, A. C. bag-of-marbles: a Drosophila gene required to initiate both male and female gametogenesis. Genes Dev. 4, 2242-2251 (1990).
- Gan, Q., Schones, D. E., Eun, S. H., Wei, G., Cui, K., Zhao, K., Chen, X. Monovalent and unpoised status of most genes in undifferentiated cell-enriched Drosophila testis. Genome Biol. 11, 42-42 (2010).
- Gan, Q., Chepelev, I., Wei, G., Tarayrah, L., Cui, K., Zhao, K., Chen, X. Dynamic regulation of alternative splicing and chromatin structure in Drosophila gonads revealed by RNA-seq. Cell Res. 7, 763-783 (2010).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved