A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
כלי אנליטי שמכמת שינויי מורפולוגיה תאיים מתמונות פלואורסצנציה תלת ממדים
In This Article
Summary
פתחנו פלטפורמת תוכנה שמנצלת Imaris Neuroscience, ImarisXT וMATLAB כדי למדוד את השינויים במורפולוגיה של צורה מוגדרת נלקחה מפלואורסצנטי confocal תלת ממדי של תאים בודדים. זו גישה חדשנית יכולה לשמש כדי לכמת את השינויים בצורת התא בא הפעלה קולטנית ולכן מייצגת כלי נוסף אפשרי לגילוי סמים.
Abstract
The most common software analysis tools available for measuring fluorescence images are for two-dimensional (2D) data that rely on manual settings for inclusion and exclusion of data points, and computer-aided pattern recognition to support the interpretation and findings of the analysis. It has become increasingly important to be able to measure fluorescence images constructed from three-dimensional (3D) datasets in order to be able to capture the complexity of cellular dynamics and understand the basis of cellular plasticity within biological systems. Sophisticated microscopy instruments have permitted the visualization of 3D fluorescence images through the acquisition of multispectral fluorescence images and powerful analytical software that reconstructs the images from confocal stacks that then provide a 3D representation of the collected 2D images. Advanced design-based stereology methods have progressed from the approximation and assumptions of the original model-based stereology1 even in complex tissue sections2. Despite these scientific advances in microscopy, a need remains for an automated analytic method that fully exploits the intrinsic 3D data to allow for the analysis and quantification of the complex changes in cell morphology, protein localization and receptor trafficking.
Current techniques available to quantify fluorescence images include Meta-Morph (Molecular Devices, Sunnyvale, CA) and Image J (NIH) which provide manual analysis. Imaris (Andor Technology, Belfast, Northern Ireland) software provides the feature MeasurementPro, which allows the manual creation of measurement points that can be placed in a volume image or drawn on a series of 2D slices to create a 3D object. This method is useful for single-click point measurements to measure a line distance between two objects or to create a polygon that encloses a region of interest, but it is difficult to apply to complex cellular network structures. Filament Tracer (Andor) allows automatic detection of the 3D neuronal filament-like however, this module has been developed to measure defined structures such as neurons, which are comprised of dendrites, axons and spines (tree-like structure). This module has been ingeniously utilized to make morphological measurements to non-neuronal cells3, however, the output data provide information of an extended cellular network by using a software that depends on a defined cell shape rather than being an amorphous-shaped cellular model. To overcome the issue of analyzing amorphous-shaped cells and making the software more suitable to a biological application, Imaris developed Imaris Cell. This was a scientific project with the Eidgenössische Technische Hochschule, which has been developed to calculate the relationship between cells and organelles. While the software enables the detection of biological constraints, by forcing one nucleus per cell and using cell membranes to segment cells, it cannot be utilized to analyze fluorescence data that are not continuous because ideally it builds cell surface without void spaces. To our knowledge, at present no user-modifiable automated approach that provides morphometric information from 3D fluorescence images has been developed that achieves cellular spatial information of an undefined shape (Figure 1).
We have developed an analytical platform using the Imaris core software module and Imaris XT interfaced to MATLAB (Mat Works, Inc.). These tools allow the 3D measurement of cells without a pre-defined shape and with inconsistent fluorescence network components. Furthermore, this method will allow researchers who have extended expertise in biological systems, but not familiarity to computer applications, to perform quantification of morphological changes in cell dynamics.
Protocol
1. ניתוח מורפומטריות תלת ממדי של שינויים פנוטיפי תא בודד
- (HEK293) תאי כליה עובריים אנושיים היו transfected עם hemagglutinin (HA) מתויג-corticotropin משחרר factor receptor-2 (CRF-R2), הקולטן G-חלבון בשילוב (GPCR) כפי שתואר לעיל 4, 5.
- התאים היו מטופלת כראוי (ללא טיפול, NT), מגורה עם ליגנד CRF-R2 אנדוגני, גורם משחרר corticotropin, CRF (1 מיקרומטר, 30 דקות), או pretreated עם אנטגוניסט CRF-R2 סלקטיבית, 30 נגד sauvagine (AS -30, 1 מיקרומטר, 30 דקות) לפני טיפול אגוניסט.
- התאים אז היו נעוצים, permeabilized וטופלו באנטי HA. CRF-R2 היה דמיין באמצעות Alexa 594 ננומטר המצומד נגד עכבר (1 IgG) נוגדנים. DAPI שמש כדי להמחיש את השלב המיטוטי גרעינים.
- כדי להגביל את הסובייקטיביות הנסיין, את תנאי הניסוי לא היו ידועים עד לאחר התמונות נרכשו ונותחו.
- אנו רכשנו תמונהשל מHEK293 תאים קבועים באמצעות מיקרוסקופ Zeiss LSM 510 META confocal מטרת תכנית-apochromat 63x/1.4 שמן דסק"ש ומחובר למערכת קוהרנטית משולבת שני פוטוני ליזר המורכבת מורדים-V5 ליזר ומירה 900-F מערכת ליזר.
- במהלך תהליך רכישת הנתונים, התאים היו ממודרים הוא על ידי חתך multispectral, 488 ננומטר, ו790 ננומטר (ננומטר ~ Ex 350 2ph.) ומחיצות z (0.5 מרווחי מיקרומטר) כדי לכלול נתונים מקרום הגרעין לרצפטור התאי החיצוני גפיים.
- נתוני הקרינה עובדו ראשונים באמצעות Imaris, המאפשר הדמיה והפילוח של בסיס נתוני מיקרוסקופיה 3D, ומודל 3D המורכב מvoxels מעוקבים נוצר עבור ניתוח מורפומטריות.
ואז, Imaris XT מודול נוצל לImaris ממשק עם שפת תוכנת מחשב MATLAB כדי לקבוע את נקודת ציון של רחבות GPCR.
לקחת בחשבון השתנות סלולרית, השיג ונתחתי פלואורסצנטי תמונות takבדרכו מ22 תאים: ללא טיפול (NT) (n = 7), אגוניסט טיפול (CRF) (n = 8) וטיפול מראש עם יריב (AS-30) לפני טיפול אגוניסט (n = 7) (איור 2) .- האזור של עניין (ROI) צריך לכלול תא אחד שהוא לא בשלב המיטוטי פעיל ולא לסגור לתאים אחרים. באופן זה, הניתוח יכלול תאים בעלי גרעין אחד בלבד ושלוחות הקולטן אינם מוטרדים קרבה של תאים אחרים.
- המבנה שוחזר 3D הסלולרי ראשון מנתוני קרינת multispectral באמצעות Imaris (v.7.1.1).
- בעקבות האלגוריתם שתוכנן על ידי Imaris, 1 עיבוד פני השטח נוצל כדי לייצג את קרום הגרעין. Imaris יקבע אם יש יותר מגרעין אחד בהחזר על ההשקעה.
- אז אלגוריתם כתמי יצירה נוצל כדי לאתר את סיומת CRF-R2. זיהוי כתמים נוצל כי זה מפצה על רעשי רקע ועצמה חריגה שלרשת מורכבת של תאי אמורפי בצורה.
- כדי למקסם את ההכללה של כל יחידת איתור קרינה של CRF-R2, בקוטר של הכתמים נקבע ל 0.2 מיקרומטר, המהווה את היחידה הקטנה ביותר בתוך התמונה על מנת להסיק מידע ייחודי בצורה של עצמה נמדדת באמצעות גאוסי מסנן. סינון ספוט התאגד בתהליך יצירת הנקודות האוטומטיות. התוכנה, לעומת זאת, נותנת למשתמש את הגמישות להשתמש במסננים כדי להגדיר את הפרמטרים.
- כדי להימנע מחיתוך נתונים, ערכת הנתונים הוסבה מ8-bit (לא חתום) נקודה קבועה, לעשרוני 32-bit.
- נתוני עוצמות voxel הוחלפו לזהות נתוני קואורדינטות באמצעות מודול Imaris XT interfaced עם MATLAB ואת המיקום המרחבי המדויק של כל נקודה נקבע על ידי ביצוע שינוי מרחק באמצעות קרום הגרעין כנקודת התייחסות (איור 3).
- הנתונים שיתקבלו ניתן לכמת ומוצגים בפורמט גרפי ליםניתוח tatistical. השוואות בין קבוצות בוצעו באמצעות דו כיוונית ANOVA וBonferroni posttest. נתונים מוצגים כממוצע ± SD. הבדלים נחשבים משמעותי ב* p <0.05. חישובים נעשו עם פריזמה GraphPad 5.02 (איור 4).
2. נציג תוצאות
כדי להדגים את כוחה של הגישה שלנו, לכמת את השינויים התאיים הנובע מהאינטראקציה של קולטני G חלבון מצמידים (GPCRs) וגורם משחרר corticotropin הקולטן-2 (CRF-R2) עם CRF יגנד אנדוגני בHEK293 תאי transfected.
אנו מראים כי רצפטורים CRF-R2 ממוקמים בקרום הפלזמה ופרויקט מאזורים סופיים של הקרום של התאים (האיור 2A וסרט 1). באמצעות ניתוח 2D קונבנציונלי, ניתן לזהות קבוצת משנה של קולטני תאי CRF-R2 רק אם אנו מנתחים את הקולטן הידבקות points על הזכוכית הקדמית. כתוצאה מכך אנו מאבדים את כל מידע אחר הנגזר מZ-נערמו נתוני multispectral (איור 5).
כאשר תאי מטופלים עם CRF, הקולטנים התאיים מופחתים מאוד, כפי שמוצגים על ידי הירידה במרחק של כתמים מקרום הפלזמה. הם גם מחולקים מחדש ממקומות בעיקר סופיים למספר המקומות נפרדים (2B איור וסרט 2).
ההשפעה של CRF על חלוקת קרום קולט נמנעת על ידי טיפול מקדים באנטגוניסט CRF-R2 הספציפי, 30 antisavagine (AS-30) ואנו מוצאים כי הרחבות CRF-R2 לא משתנות (האיור 2C וסרט 3).
ההפצה הדיסטלי של כתמים, זממה ב 5 המרווחים המקודדים ספקטרום צבעי מיקרומטר, מנוצלת כדי להמחיש את המרחק מvoxels מקרום הגרעין. לא pretreatm טיפול ואנטגוניסטאף אוזן גרון (AS-30, 1 מיקרומטר, 30 דקות) לפני טיפול אגוניסט (CRF, 1 מיקרומטר, 30 דקות) לא הראה הבדל משמעותי (NS) בהתכווצות GPCR. טיפול בתאים עם אגוניסט (CRF, 1 מיקרומטר, 30 דקות) בהדרגה מפחית את מספר voxels-R2 המכיל CRF בהשוואה לכל טיפול, 0-5 מיקרומטר (NS), 6-15 מיקרומטר (** p < 0.01) ו> 15 מיקרומטר (*** p <0.005), או בהשוואה לכמות ש- 30 לטיפול, 0-10 מיקרומטר (NS), 11-15 מיקרומטר (** p <0.01) ו> 15 מיקרומטר (*** p <0.005) (איור 4).
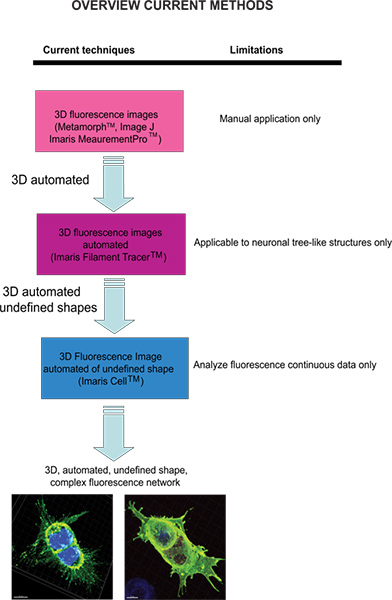
איור 1. תרשים של טכניקות הזמינות הנוכחיות והגבלתם לנתח תמונות פלואורסצנציה. לחץ כאן לצפייה בדמות גדולה.
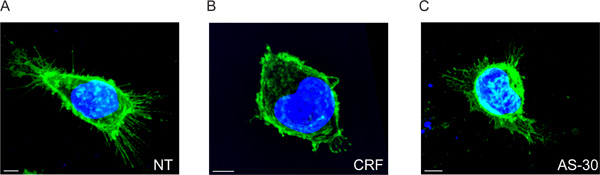
איור 2. תמונות פלואורסצנציה multispectral 3D של HEK293 תאי transfected עם HA-CRF-R2 לפני ואחרי אגוניסט אנטגוניסט וטיפול. תמונות ממוזגות מראות HA-CRF-R2-חקרו באמצעות אנטי HA ו מדמיינים באמצעות 488 אנטי עכבר (1 IgG) נוגדנים מצומדות אלקסה משניים; DAPI שמש כדי להמחיש את הגרעינים. התמונות שנרכשו עם ליזר confocal מיקרוסקופ סריקה (CLS). 5 מיקרומטר סרגל קנה מידה.
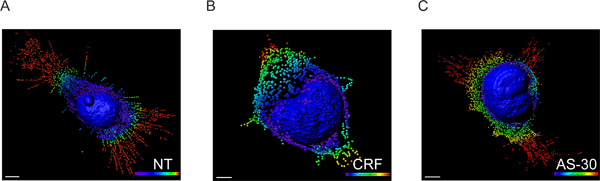
איור 3. מודל 3D של HEK293 תאי transfected עם HA-CRF-R2 שחזר מתמונות CLS באמצעות תוכנת Imaris. עיבוד פני שטח של הגרעין ויצירת כתמים מתאר רחבות GPCR הומרו לשלפוחית קטנה. נתוני הקרינה עובדו ראשון באמצעות Imaris המאפשר הדמיה והפילוח של נתונים להגדיר מיקרוסקופיה 3D. ואז, Imaris XT נוצל לImaris ממשק עם MATLAB. עוצמות voxel הוחלפו לתוךנקודת המרכזת. כתמי צבע קשת מקודד (0-5 מיקרומטר הכחול, ירוק, 6-10 מיקרומטר, מיקרומטר 11-15 צהוב והאדום> 15 מיקרומטר) מייצגים את צורת מרחק קרום הגרעין. הסולם של 5 מיקרומטר.
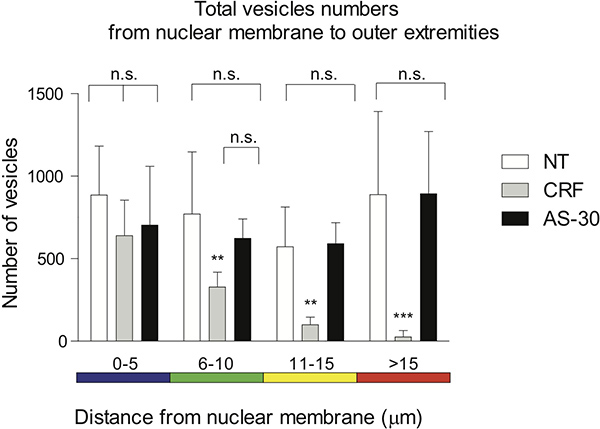
האיור 4. ייצוג גרפי של ההפצה הדיסטלי של כתמים זמם בספקטרום הצבעים המקודדים ב 5 מרווחי מיקרומטר מנוצל כדי להמחיש את המרחק מvoxels מקרום הגרעין. אין טיפול וטיפול מקדים ביריב (AS-30, 1 מיקרומטר, 30 דקות) לפני טיפול אגוניסט (CRF, 1 מיקרומטר, 30 דקות) לא הראה הבדל משמעותי (NS) בהתכווצות GPCR. טיפול בתאים עם אגוניסט (CRF, 1 מיקרומטר, 30 דקות) מפחית בהדרגה את המרחק של מספר voxels-R2 המכיל CRF בהשוואה לשום טיפול 0-5 מיקרומטר (NS), 6-15 מיקרומטר (עמ ** <0.01) ו> 15 מיקרומטר (*** p <0.005); או 0-10 מיקרומטר הטיפול AS-30 (NS), 11-15 מיקרומטר (** p <0.01) ו> 15 מיקרומטר (*** p <0.005).
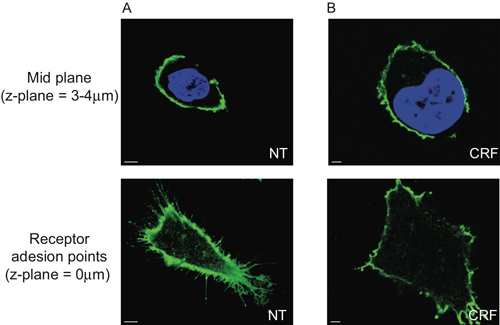
איור 5. הגבלת ניתוח מורפומטריות 2D של 293 תאי transfected HEK עם HA-CRF-R2. סעיף מטוס האמצע של תאים (3-4μm מעל זכוכית coverslip) מראה את מרכז הגרעינים דמיין בDAPI וHA-CRF-R2 חקר באמצעות אנטי HA ו דמיין באמצעות Alexa 488nm מצומדת אנטי עכבר (1 IgG) משני נוגדנים ורכש עם CLS מראים שום הבדל בין () ללא טיפול (NT) ו (ב) אגוניסט (CRF, 1 מיקרומטר, 30 דקות), ואילו את נקודתי ההדבקה הקולטן הן שונות באופן דרמטי.
סרט 1. הסתובב בחופשיות, מודל 3D במצב "לעלות" לHEK 293 תאי transfected עם HA-CRF-R2, ללא טיפול, כדי להעריך את ההבדלים בין פנוטיפ סלולריים חלבוני הקולטן. סרגל קנה מידה 5-20 מיקרומטר."Target =" / files/ftp_upload/4233/4233movie1.avi _blank "> לחץ כאן לצפייה בסרט.
סרט 2. הסתובב בחופשיות, מודל 3D במצב "לעלות" לHEK 293 תאי transfected עם HA-CRF-R2, טיפול אגוניסט, CRF (CRF, 1 מיקרומטר, 30 דקות) כדי להעריך הבדלי פנוטיפ סלולריים בין חלבוני הקולטן. סרגל קנה מידה 5-20 מיקרומטר. לחץ כאן לצפייה בסרט.
סרט 3. הסתובב בחופשיות במצב 3D "לעלות" לHEK 293 תאי transfected עם HA-CRF-R2, טיפול מקדים ביריב (AS-30, 1 מיקרומטר, 30 דקות) לפני טיפול אגוניסט (CRF, 1 מיקרומטר, 30 דקות ') כדי להעריך הבדלי פנוטיפ סלולריים בקרב חלבוני הקולטן. סרגל קנה מידה 5-20 מיקרומטר. לחץ כאן לצפייה בסרט.
Discussion
אנחנו הראינו כי טיפול CRF מושרה שינוי משמעותי במורפולוגיה ומיקום של CRF-R2. השינוי בCRF-R2 נבלם על ידי טיפול אנטגוניסט סלקטיבי. אנחנו הראינו כי שינויי הקולטן הם לא אותרו ולא ניתן למדוד באמצעות טכניקות multispectral 2D הסטנדרטיות. היכולת ללמוד תמונות 3D מורכבות היא קריטית כדי לשלב א?...
Disclosures
אין ניגודי האינטרסים הכריזו.
Acknowledgements
אנו מודים הביולוגית הדמית מרכז הפיתוח (BIDC) מאוניברסיטת קליפורניה, סן פרנסיסקו לשימוש בImaris, Imaris XT ו Matlab. אנו מודים V. Kharazia לסיוע הטכני ועל הנרי, LK פלורן, L. ךייטש על תרומתם לעריכה של כתב היד. עבודה זו נתמכה על ידי מימון ממדינת קליפורניה מחקר רפואי על אלכוהול התעללות וסמים דרך קליפורניה בסן פרנסיסקו לSEB, המכונים הלאומיים לבריאות: 1R21DA029966-01 ופרס NIH מסלול המהיר להקרין את אוסף MLSMR לSEB, קליפורניה בסן פרנסיסקו בית הספר לרוקחות ( משרד הדיקן ורוקחות קליניות) ובית הספר לרפואה (פרמקולוגיה קלינית & Therapeutics ניסיוני) לCLHK.
Materials
| Name | Company | Catalog Number | Comments |
| שם המגיב | חברה | מספר קטלוגים | תגובות (אופציונלי) |
| הכליה עוברית אנושית (HEK293) | אוסף תרבות סוג אמריקאי | CRL-1573 | |
| של Dulbecco שינוי הנשר הבינוני (DMEM) | Invitrogen | 11965118 | |
| עוברי בסרום שור (FBS) | Invitrogen | SH30070.03 | |
| AlexaFluor-488 (IgG2b) | Invitrogen | -11001 | |
| חד שבטי אנטי HA.11 (1 IgG) | Covance | 16B12 | |
| DAPI | מעבדות וקטור | H-1200 | ; |
| CRF | סיגמא | C2917 | |
| Antisauvagine-30 (AS-30) | סיגמא | A4727 |
References
- West, M. J. Design-based stereological methods for counting neurons. Prog, Brain Res. 135, 43-51 (2002).
- Burke, M., Zangenehpour, S., Mouton, P. R., Ptito, M. Knowing what counts: unbiased stereology in the non-human primate brain. J. Vis. Exp. (27), e1262 (2009).
- Sugawara, Y., Ando, R., Kamioka, H., Ishihara, Y., Honjo, T., Kawanabe, N., Kurosaka, H., Takano-Yamamoto, T., Yamashiro, T. The three-dimensional morphometry and cell-cell communication of the osteocyte network in chick and mouse embryonic calvaria. Calcif. Tissue Int. 88, 416-424 (2011).
- Vickery, R. G., von Zastrow, M. Distinct dynamin-dependent and -independent mechanisms target structurally homologous dopamine receptors to different endocytic membranes. J. Cell Biol. 144, 31-43 (1999).
- Bartlett, S. E., Enquist, J., Hopf, F. W., Lee, J. H., Gladher, F., Kharazia, V., Waldhoer, M., Mailliard, W. S., Armstrong, R., Bonci, A. Dopamine responsiveness is regulated by targeted sorting of D2 receptors. Pro.c Natl. Acad. Sci. U.S.A. 102, 11521-11526 (2005).
- Gordon, A., Colman-Lerner, A., Chin, T. E., Benjamin, K. R., Yu, R. C., Brent, R. Single-cell quantification of molecules and rates using open-source microscope-based cytometry. Nat. Methods. 4, 175-181 (2007).
- Schock, F., Perrimon, N. Molecular mechanisms of epithelial morphogenesis. Annu. Rev. Cell Dev. Biol. 18, 463-493 (2002).
- Pincus, Z., Theriot, J. A. Comparison of quantitative methods for cell-shape analysis. J. Microsc. 227, 140-156 (2007).
- Spiller, D. G., Wood, C. D., Rand, D. A., White, M. R. Measurement of single-cell dynamics. Nature. 465, 736-745 (2010).
- Loo, L. H., Wu, L. F., Altschuler, S. J. Image-based multivariate profiling of drug responses from single cells. Nat Methods. 4, 445-453 (2007).
- Yarrow, J. C., Totsukawa, G., Charras, G. T., Mitchison, T. J. Screening for cell migration inhibitors via automated microscopy reveals a Rho-kinase inhibitor. Chem. Biol. 12, 385-395 (2005).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved