A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
חיזוי מבנה המשני RNA באמצעות SHAPE תפוקה גבוהה
In This Article
Summary
acylation הידרוקסיל '2 סלקטיבית תפוקה גבוהה נותח על ידי הארכה תחל (SHAPE) מנצל כימי רומן חיטוט טכנולוגיה, שעתוק לאחור, אלקטרופורזה נימים ותוכנות לחיזוי מבנה משנית כדי לקבוע את המבנים של RNAs מכמה מאה עד כמה אלף נוקלאוטידים ברזולוציה נוקלאוטיד יחידה.
Abstract
הבנת תפקידו של רנ"א המעורב בתהליכים ביולוגיים דורשת ידע מעמיק של מבנה RNA. לשם כך, את המתודולוגיה המכונה "acylation הידרוקסיל 2 סלקטיבית תפוקה גבוהה 'נותח על ידי הארכה תחל", או צורה, מאפשרת חיזוי של מבנה המשני RNA עם רזולוציה נוקלאוטיד יחידה. גישה זו מנצלת סוכני חיטוט כימיים שמעדיפים acylate אזורים תקועים או גמישים בודדות של רנ"א בתמיסה מימית. אתרים של שינוי כימי מזוהים על ידי שעתוק הפוך של RNA השונה, ואת המוצרים של התגובה הזו הם מופרדים על ידי אלקטרופורזה נימים אוטומטית (CE). מאז רברס טרנסקריפטאז הפסקות באותם נוקלאוטידים RNA שונה על ידי חומרים כימיים צורה, ספריית cDNA כתוצאה בעקיפין ממפה האלה ribonucleotides שהם תקועים אחת בהקשר של RNA המקופל. באמצעות תוכנת ShapeFinder, המיוצר על ידי את electropherograms האוטומטית לספירה מעובד והוסב לנושולחנות תגובתיות cleotide שהם עצמם הפכו לאילוצים פסאודו אנרגיה בשימוש באלגוריתם חיזוי RNAStructure (V5.3). את מבני RNA דו ממדים המתקבלים על ידי שילוב של SHAPE חיטוט עם בחיזוי מבנה המשני סיליקון ורנ"א כבר נמצא להיות הרבה יותר מדויק מאשר מבנים המתקבלים בשני שיטות לבד.
Introduction
כדי להבין את הפונקציות של RNAs קטליטיים וללא קידוד מעורב בויסות של שחבור, תרגום, שכפול נגיף וסרטן, ידע מפורט של מבנה RNA נדרש 1,2. לרוע המזל, חיזוי מדויק של רנ"א מתקפל מציג אתגר עצום. סוכני חיטוט הקלסיים סובלים מחסרונות רבים כגון רעילות, כיסוי חלקי נוקליאוטידים ו / או תפוקה מוגבלת ל100-150 נוקלאוטידים לכל ניסוי. אלגוריתמים לחיזוי מבנה משניים בכוחות עצמו הם דומה נחות, בשל אי דיוקים הנובעים מחוסר היכולת שלהם להבחין ביעילות בין מבנים אנרגטי דומים. RNAs גדול בפרט הם לעתים קרובות גם עקשן לשיטות של קביעת מבנה 3D כגון קריסטלוגרפיה רנטגן ותהודה מגנטית גרעינית (NMR) ספקטרוסקופיה, בשל הגמישות שלהם קונפורמציה וכמויות גדולות של דגימות טהורות ביותר הנדרשות לטכניקות אלה.
HSHAPE igh תפוקה פותר רבים מהבעיות הללו על ידי מתן גישה יעילה, פשוטה לחיטוט המבנים של RNAs גדול ברזולוציה נוקלאוטיד יחידה. יתר על כן, את חומרים כימיים המשמשים עבור צורה בטוח, קל לטפל ו, בניגוד לרוב כימי אחרות חיטוט ריאגנטים, מגיבים עם כל ארבע ribonucleotides. ריאגנטים אלה יכולים גם לחדור ממברנות תאים, מה שמאפשר לחקור RNAs בהם בהקשר vivo (ים) 3. פותח במקור בשבועות המעבדה 4, צורה נעשתה שימוש כדי לנתח מגוון רחב של RNAs, הדוגמא הבולטת ביותר היא קביעת המבנה המשני המלא של ~ 9 KB HIV-1 RNA הגנום 5. הישגים בולטים אחרים בשימוש בצורה כוללים הבהרה של המבנים של viroids זיהומיות 6, RNAs אדם הארוך ללא קידוד 7, שמרי ריבוזומים 8, וriboswitches 9, כמו גם לזהות אתרי קישור בחלבון virion הקשורים RNA HIV-1 3. מה עלי לעשותאיל הווריאציות המקוריות ותפוקה גבוהה של פרוטוקול SHAPE כבר פורסם במקומות אחרים 10-12, העבודה הנוכחית מספקת תיאור מפורט של קביעת מבנה המשנית RNA על ידי צורת תפוקה גבוהה באמצעות oligonucleotides ניאון, מנתח Beckman Coulter CEQ 8000 הגנטי, ו SHAPEfinder וRNAStructure תוכנה (V5.3). פרטים טכניים שלא פורסמו בעבר ועצות לפתרון בעיות כלולות גם.
וריאציות של צורה
המהות של צורה והווריאציות שלה היא חשיפה של רנ"א בתמיסה מימית לפטאליק electrophilic כי סלקטיבי acylate קבוצות (2'-OH) 2'-הידרוקסיל ריבוז, ייצור adducts המגושם באתרים של שינוי. תגובה כימית זו משמשת כאמצעי לחקירת דינמיקה מבנית מקומית RNA, נוקלאוטידים כיחידים תקועים נוטים יותר לאמץ תצורות תורמות להתקפת electrophilic על ידי חומרים כימיים האלה, ואילו בסיס לזווג או ארכיטקטוני constrנוקלאוטידים ained הם פחות או unreactive 10. אתרים של היווצרות adduct מזוהים על ידי שעתוק לאחור ייזום מfluorescently או radiolabeled פריימרים הכלאה לאתר מסוים ברנ"א השונה ("(+)" תגובת ההארכה תחל). כאשר רברס טרנסקריפטאז (RT) לא מצליח לחצות את ribonucleotides acylated, בריכת של מוצרים מיוצרת cDNA שאורכי קנה אחד עם אתרים של שינוי. שליטה, "(-)" פריימר סיומת תגובת ניצול RNA שלא נחשף למגיב מבוצעת גם כדי שהסיום המוקדם של סינתזת דנ"א ("תחנות", כלומר) בשל מבנה RNA, שבר ספציפי RNA גדיל, וכו ', במאי. להבדיל ממשתהה מיוצר על ידי שינוי כימי. לבסוף, שתי תגובות dideoxy-רצף ייזום מאותם פריימרים משמשות כסמנים כדי לקשר בין נוקלאוטידים תגובתי עם רצף הרנ"א הראשוני בעקבות אלקטרופורזה.
ביישום המקורי של ShapE, אותו 32-P-שכותרתו סוף פריימר מנוצל ל( +), - ושתי תגובות רצף (). מוצרים של תגובות אלה נטענים לתוך בארות סמוכות בג'ל לוח polyacrylamide 5-8%, ומופרדים על ידי denaturing ג'ל אלקטרופורזה polyacrylamide (דף; איור 1). ניתוח כמותי של דימויי ג'ל מיוצרים על ידי צורה קונבנציונלית יכול להתבצע באמצעות סאפא, תוכנת ניתוח footprinting חצי אוטומטית 13.
לעומת זאת, צורת תפוקה גבוהה מעסיקה פריימרים שכותרתו fluorescently ואלקטרופורזה נימים אוטומטיות. באופן ספציפי, לכל אזור של רנ"א תחת חקירה, סט של ארבעה פריימרים DNA בעלי רצף משותף אך שונה 5 'תוויות ניאון חייבת להיות מסונתזים או נרכשו. oligonucleotides שונה שכותרתו אלה משמשים לשתי תגובות SHAPE הממשלה ושתי תגובות רצף, את המוצרים של אשר אספו ומופרד / זוהו על ידי אלקטרופורזה נימים אוטומטית (CE). WherEAS ניתן להשיג את פרופיל תגובתיות של 100-150 NT של רנ"א מתוך סדרה של ארבע תגובות המשתמשות בגישה המקורית, צורת תפוקה גבוהה מאפשרת רזולוציה של 300-600 NT ממדגם 3 חד נקווה. עד 8 קבוצות של תגובות עשויים להיות מופרד בו זמנית, ואילו רבים כמו 96 דגימות יכולות מוכנות לחלוקה במהלך 12 ריצות לספירה רצופות (איור 2). יתר על כן, תוכנת SHAPEfinder, שפותחה כדי לעבד ולנתח נתונים העולים מתוך CEQ ומנתחים גנטיים אחרים, היא אוטומטית יותר ודורשת התערבות משתמש הרבה פחות מסאפא 13 או חבילות ג'ל ניתוח אחרות.
מתודולוגיות תפוקה גבוהה יותר מתקדמות שהתפתחו לאחרונה כגון PARS (ניתוח מקביל של מבנה RNA) 14 ו-Frag Seq (שבר-רצף) 15, המשתמש באנזימי מבנה ספציפיים ולא מגיבים לאלקילציה בשיתוף עם הדור הבא רצף טכניקות כדי להשיג information על מבנה RNA. למרות האטרקטיביות של טכניקות אלה, המגבלות רבות הגלומות לnuclease חיטוט עדיין יישארו 16. בעיות אלה ניתן לעקוף ברצף הצורה (SHAPE-Seq) 17 פרוטוקול, שבו הרצף של הדור הבא הוא קדמה שינוי כימי ושעתוק לאחור של RNAs באופן דומה לזה שבוצע בצורה קונבנציונלית. בעוד שיטות אלה עשויות לייצג את העתיד של קביעת מבנה RNA, חשוב לזכור שהרצף של הדור הבא הוא יקר מאוד, ונשאר זמין למעבדות רבות.
ניתוח נתוני צורה
נתונים שהופקו במנתח הגנטי מוצגים בצורה של electropherogram, שבו עוצמת הקרינה של הדוגמא (ו) זורמת דרך גלאי הנימים זממו נגד מדד של זמן הגירה. עלילה זו לובשת הצורה של עקבות חופפות המקבילה לערוץ הקרינה ארבעהים משמש כדי לזהות את fluorophores השונים, ובו כל עקבות מורכבת מפסגות מתאימות למוצרי cDNA או רצף בודדים. נתוני electropherogram מיוצאים מהמנתח הגנטי כקובץ טקסט מופרד באמצעות טאבים ויובאו לשינוי ShapeFinder וניתוח תוכנה 18.
ShapeFinder משמש בתחילה לבצע סדרה של טרנספורמציות מתמטיות על הנתונים כדי לוודא שפעמי הגירה והיקפי שיא משקפים במדויק את זהותם וכמויות של מוצרי התגובה, בהתאמה. פסגות הן אז מיושרות ומשולבים, ואת התוצאות נספרו יחד עם רצף הרנ"א הראשוני. "פרופיל תגובתיות" למקטע הרלוונטי של רנ"א מתקבל על ידי החסרת ערכי בקרה מ( +) ערכים הקשורים לכל רנ"א נוקלאוטיד, ונרמול הנתונים כפי שיתואר להלן. פרופיל זה מיובא לRNAstructure (V5.3) 19,20 תוכנה, אשר ממירה val תגובתיות המנורמלues תוך אילוצים פסאודו אנרגיה שמשולבים באלגוריתם קיפול המבנה המשני RNA. שילוב כימי חיטוט וקיפול אלגוריתמים בדרך זו משפר באופן משמעותי את הדיוק של חיזוי מבנה בהשוואה לכל אחת מהשיטות לבד 12,21. הפלט של RNAstructure (V5.3) כולל תמונות של האנרגיה הנמוכה ביותר RNA מבנים משניים בצבעים עם צורת פרופיל תגובתיות (ים), כמו גם את אותם מבנים בסימון הדוט סוגר טקסטואלי. זו האחרונה עשויה להיות מיוצאות לתוכנה המיועדת לתצוגה הגרפית של מבנה המשני RNA כגון רנה 22 וPseudoViewer 23.

איור 1. תרשים זרימה של קביעת מבנה RNA באמצעות SHAPE 4,10. () RNA מ 'איי יתקבלו מדגימות ביולוגיות או על ידי שעתוק במבחנה. (ב) בהתאם למקור, RNA הוא מקופל או מעובד בדרך אחרת ושונים עם מגיב SHAPE. (ג) שעתוק הפוך באמצעות פריימרים שכותרתו fluorescently או רדיואקטיבית. (D) מוצרי cDNA הם מופרדים באמצעות שתי אלקטרופורזה מבוססת ג'ל נימים או לוח. (ה) ניתוח שבר. (F) ניבוי מבנה RNA. לחץ כאן לצפייה בדמות גדולה.
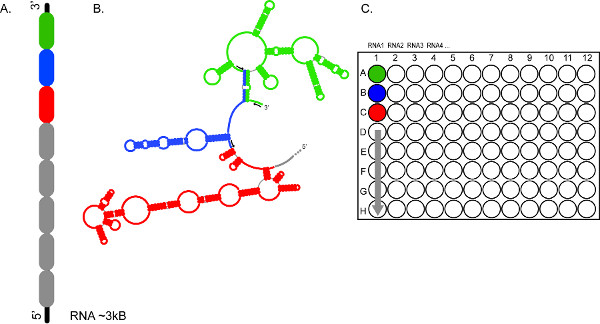
איור 2. אופי התפוקה גבוהה של צורה מבוססת CE מאפשר ניתוח מהיר של RNAs מרובים, ו / או מקטעים מרובים של אותם רנ"א. () מייצג כיצד ניתן לחלק RNA לסעיפי 300-600 NT (מקודד לפי צבע בירוק, כחול ואדום) (ב) חלקים של RNA הם חקרו באופן עצמאי באמצעות סטים שונים של פריימרים ניאון (חיצים שחורים) (ג) סטים תגובות הם אספו והעמיסו לתוך בארות A1, B1, C1, וכו ', בהתאמה, ומספקות כיסוי מלא ל~ 3 KB RNA1. תוצרי תגובה מRNAs 2, 3, 4, וכו 'יכול להיות מוכן באופן דומה לחלוקה בריצות electrophoretic רצופות. לחץ כאן לצפייה בדמות גדולה.
Protocol
עיצוב פריימר והרחבה של תחנה סופית 'רנ"א 3
כדי לנתח RNAs הארוך על ידי צורת תפוקה גבוהה, סדרה של אתרי הכלאה פריימר צריכה להיות כזה שהם בחרו (אני) מופרדים על ידי ~ 300 NT, (ii) הם 20-30 NT באורך, וכן (iii) כי RNA / הכלאיים DNA המיוצרים על ידי חישול DNA לאתרים האלה יש טמפרטורת התכה צפויה של> 50 ° C. בנוסף, מקטעים של RNA שהם חזו להיות מאוד מובנים יש להימנע, אם כי מה שהופך את קביעה כזו דורשת קצת ידיעה מוקדמת של מבנה רנ"א, אשר לעתים קרובות אינה זמינה. פריימרים DNA שלהכליא לאתרים אלה אז צריכים להיות מתוכננים, מקפידים לוודא שהם לא יהיו צפויים ליצירת הדימרים יציבים או מבנים משניים intrastrand.
ברגע שתוכנן, ערכות פריימר חייבים להיות גם נרכשו (למשל מDNA המשולב טכנולוגיות, איימס, איווה) או מסונתז 24,25. פריימרים 5'-שכותרתו עם Cy5, Cy5.5,WellRedD2 (Beckman Coulter) וIRDye800 (Lycor) / WellRedD1 (Beckman Coulter) הם מתאימים ביותר עבור Beckman Coulter 8000 CEQ, מתן עוצמת אות טובה, תוך מזעור crosstalk. oligonucleotides שכותרתו ניתן לאחסן לזמן בלתי מוגבל ב, 10 מיקרומטר aliquots הקטנים ב -20 ° C, להימנע מהקפאה / הפשרה מחזורים חוזרים ונשנים.
על ידי שימוש בפריימרים המיועדים באופן זה, ניתן לקבל נתונים צורה לכמעט כ RNA שלם בכל אורך. עם זאת, הרצף או בסמוך 3 "תחנה סופית של RNA הוא תמיד נגיש לצורה, אלא אם כן RNA מתוכנן להכיל 3 'סיומת מסוף (למשל" קלטת מבנה ") כדי שפריימר יכול להיות הכלאה 4.
הכנת RNA באמצעות אלקטרופורזה נימי
למרות RNAs מדגימות ביולוגיות עשוי להיות מנוצל עבור צורה של תפוקה גבוהה, בפרוטוקול המתפרסם כאן הוא מותאם לרנ"א מיוצר על ידי שעתוק במבחנה. מסחרי טרהערכות nscription כגון MegaShortScript (Ambion) הפועל בשיתוף עם עמודות טיהור RNA MegaClear (Ambion) מתאימות גם להפקת כמויות גדולות של RNA הטהור. RNAs צריך להיות מאוחסן בTE חיץ בין -20 ° C ו -80 ° C. לקבלת התוצאות הטובות ביותר, RNAs אמור להופיע הומוגנית על ידי שני ג'ל אלקטרופורזה polyacrylamide denaturing ולא denaturing.
1. רנ"א מתקפל
- בצינור microcentrifuge 0.5 מ"ל, לדלל 12 pmol של רנ"א ל18 μl עם מים ולהוסיף 2 μl של חיץ renaturation 10X. מערבבים היטב.
- חום עד 85 מעלות צלזיוס במשך 1 דקות, ולאחר מכן לצנן עד 4 מעלות צלזיוס בשיעור של 0.1 מעלות צלזיוס / שנייה.
- הוסף 100 μl מים ו30 μl של חיץ מתקפל 5X.
- לדגור על 37 מעלות צלזיוס במשך 30-60 דקות, תלוי ברנ"א ששלוב. באופן כללי, Mg 2 + תלוי קיפול של RNAs ארוך יותר, ודורש יותר מובנה פעמים דגירה ארוכות יותר.
- העבר aliquot μl 72 לכל אחד משני צינורות 0.5 מ"ל microcentrifuge: Modifiאד (+) ובקרה (-).
2. שינוי כימי של רנ"א
ובכן מאופיין, ריאגנטים SHAPE electrophilic כוללים אנהידריד isatoic (IA), אנהידריד N-methylisatoic (NMIA), אנהידריד 1-מתיל-7-ניטרו-isatoic (1M7) 26, ו( BzCN) 27 ציאניד נזואיל. מבין אלה, בשימוש הנפוץ ביותר עבור צורה של תפוקה גבוהה הוא 1M7 וNMIA, ורק האחרון הוא זמין באופן מסחרי (חיים טכנולוגיים). הריכוז הסופי של שינוי מגיב חייבת להיות מותאם לכל RNA להשיג קינטיקה שינוי "חד פגע", כלומר המצב שבו רוב RNAs בפתרון משתנים פעם אחת באזור של רנ"א שנתח 11. ריכוז אופטימלי זה יכול להיקבע על ידי ביצוע מספר רב של תגובות שבו הריכוז של מגיב מגוון על פני הטווח (ים) שצוין בטבלה שבסעיף 2.1 להלן. השתמש בריכוז של מגיב שמייצר אות זיהוי בקלות תוך דקותimizing ההבדל בעוצמת אות בין מוצרי סינתזת דנ"א ארוכים וקצרים (לדוגמא איור 3).
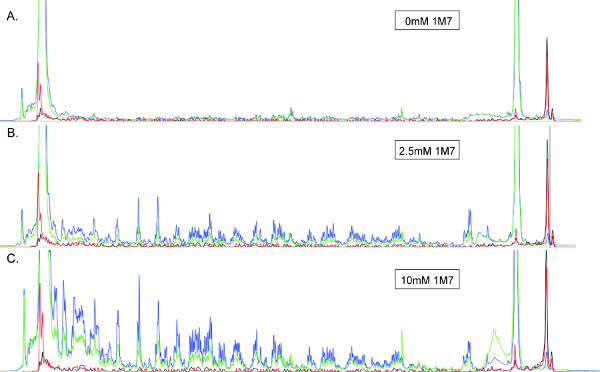
איור 3. electropherograms SHAPE המופק מ~ 360 NT טופל (א) (ב) 2.5 מ"מ 0 או (ג) 10 מ"מ 1M7 RNA. כל electropherograms מוצגים באותו קנה המידה. הכחול, ירוקות עקבות, אדומות ושחורות מתאימות למוצרים (+) תגובה (Cy5), (-) תוצרי תגובה (Cy5.5), ושני סולמות רצף (D2 WellRed וIRDye800), בהתאמה. כבר התייחס לRNA המשמש לייצור תמונה (ב ') עם הכמות האופטימלית של 1M7, הוכחת רזולוציה שיא טובה ועצמת, עם דעיכת אות מינימאלית לאורך כל העקבות (משמאל). קרא את האורך מקסימאלי הוא בתנאים אלה. לעומת זאת, היעדר int הבינוניensity, פסגות גם נפתרו ב() מצביעה על ריכוז תת אופטימלי של 1M7. לעומת זאת, באה לידי ביטוי בדעיכת האות (ג') עולה כי קינטיקה להיט אחד לא הוא ציין, וRNA הוא שונה-נגמר. במקרים כאלה, במיוחד כאשר RT לא היה צפוי להיתקל ב'5 תחנה סופית של תבנית RNA, לקרוא אורך יהיה הכי מוצלח.
- הכן את מניית 10X של מגיב צורה (NMIA או 1M7). זו מושגת הטובה ביותר על ידי הוספת כמות קטנה של מגיב לצינור 1.5 מיליליטר microfuge, ולאחר מכן הוספת DMSO כדי להשיג את הריכוז הרצוי שים לב:. פתרונות מגיב SHAPE חייבים להישאר נטול מים עד מעורבבים עם רנ"א. החנות DMSO בייבוש בטמפרטורת חדר, ופתרונות מניות להכין מייד לפני שימוש על מנת לצמצם את החשיפה לסביבת אדי מים.
מגיב ריכוז אופטימלי 10X (DMSO) זמןהשפלה של מגיב 27 מלאה NMIA 10-100 מ"מ ~ 20 דקות 1M7 10-50 מ"מ 70 שניות
טבלת מס '1. חומרים כימיים המשמשים לElectrophilic רנ"א שינוי. - הוסף 8 DMSO 10X NMIA/1M7 או נטול מים μl לעדכון (+) ושליטה (-) מתערבב, בהתאמה. הערה: 2.5 מ"מ הוכיח להיות יעיל לריכוז מתחיל גם NMIA ו1M7, ללא קשר לרנ"א להיות מנותח.
- לדגור על 37 מעלות צלזיוס למשך 50 דקות (NMIA) או 5 דקות (1M7), לפי עניין.
- RNA משקע על ידי הוספת 8 μl (0.1 בנפח) של 3 מ 'NaOAc (pH 5.2), 100 μl 8 מ"מ EDTA, 240 μl (3 כרך) של אתנול קר וגליקוגן מ"ג / מ"ל 1 μl 10. מכניס למקרר לשעה 2 ואז צנטריפוגות ב XG 14,000 עבור 30 דקות ב 4 ° C. שטוף את הכדור פעמיים עם 70% אתנול קר לב:. חשובלצמצם את זמן קירור, זמן צנטריפוגה ומהירות על מנת למזער שיתוף משקעים של מלח, כמו זה יכול להשפיע לרעה על רזולוציה שיא במהלך אלקטרופורזה.
- הסר supernatant עם micropipette, ואוויר יבש גלולה במשך 5 דקות בטמפרטורת חדר.
- ממיסים RNA זרז ב10 חיץ TE μl ודגירת 5 דקות בטמפרטורת חדר. זה מספיק מומס RNA לשתי תגובות שעתוק לאחור. לאחסן את החלק בשימוש ב -20 ° C. לב: resuspension מכאני של גלולה הוא בדרך כלל לא נחוץ, ועלול לגרום נזק לרנ"א.
3. היפוך תמלול
צעד זה מייצר את מוצרי fluorescently שכותרתו cDNA המשמשים כדי לזהות את המידה שבה נוקלאוטידים RNA שונו על ידי מגיב צורה עקיף. לצורה, ביצועים של עילי III (Invitrogen) RT היו מעולה לכל RTS האחר שנבדק, והוא האנזים שנבחר לשימוש עם זהפרוטוקול. Oligonucleotides שכותרתו עם Cy5 וCy5.5 משמשים לראש את (+) ו (-) תגובות, בהתאמה. לRNAs קצר יותר, הם הכלאה פריימרים ל3 'מסוף סיומת של רנ"א הילידים (למשל "קלטת מבנה") על מנת לקבל מידע על 3 "תחנה סופית 4 לב:. מנקודה זו ועד לספירה, צריכה להיות מוגנות מדגימות אור.
- הכן (+) ו (-) דוגמאות לשעתוק לאחור ב0.5 צינורות microfuge מ"ל. לתגובה (+) RT, לערבב 5 μl של עדכון רנ"א (+), 6 מים μl ופריימר 1 μl Cy5 שכותרתו (10 מיקרומטר); ל( -) תגובת RT, לערבב 5 שליטה RNA μl (-), 6. מים μl, וCy5 שכותרתו פריימר (10 מיקרומטר) μl 1 שים לב: Sarstedt PCR צינורות (REF 72.735.002) מומלצים ליישום זה.
- מקום צינורות בCycler תרמית, ולחשל פריימר לRNA ולהתכונן לשעתוק לאחור על ידי יישום התכנית הבאה: 85 ° C, 1 דקות, 60 מעלות צלזיוס, 5 דקות;35 מעלות צלזיוס, 5 דקות, 50 ° C, חזקה.
- במהלך שלב חישול, להכין תערובת RT מספיק 2.5x למספר התגובות שיש לבצע, בתוספת 50% (למשל לשניים (+) ושניים (-) תגובות, בקנה מידה של פי 4.5). תגובה אחת דורשת 8 μl, כדלקמן: 4 חיץ μl 5x RT, 1 μl 100 מ"מ DTT, 1.5 μl מים, 1 dNTPs 10 מ"מימ μl, עילי μl 0.5 III RT. שמור על קרח לב:. חיץ RT 5X וDTT 100 מ"מ מסופקים עם עילי III RT.
- ברגע שהטמפרטורה של תערובות חישול מגיעה ל -50 מעלות צלזיוס, להוסיף 8 μl תמהיל RT 2.5x ל( +) ואת (-) תגובות המלצה:. חם תמהיל RT עד 37 מעלות צלזיוס למשך 5 דקות לפני שמוסיף אותו לתגובות .
- דגירה במשך 50 דקות ב -50 מעלות צלזיוס, ולאחר מכן לצנן עד 4 מעלות צלזיוס ו / או מקום על קרח. הערה: דגירה מתגובות RT למשך זמן ארוך יותר מ -50 דקות עלולה לגרום למוצרי cDNA הסוטים.
- Hydrolyze הרנ"א על ידי הוספת 4 μl 1 M NaOH וחימום עד 95 מעלות צלזיוס במשך 3 דקות. תגובות מגניבים על קרח ולאחר מכן לנטרל אותם על ידי הוספת 2 μl של 2 M HCl לב:. השמטה של תוצאות שלב זה בהפרדה באיכות גרועה של מוצרי cDNA.
- לשלב (+) ו (-) ולזרז תגובות cDNA על ידי הוספת 0.1 כרך של 3 מ 'NaOAc, 0.1 כרך של 100 מ"מ EDTA, 1.5 כרך של אתנול קר וμl 1 של גליקוגן מ"ל 10 מ"ג /. מכניס למקרר לשעה 2, אז צנטריפוגות ב XG 14,000 עבור 30 דקות ב 4 ° C. שטוף את כדור פעמיים עם 70% אתנול קר לב:. צנטריפוגה בשיעורים גבוהים יותר או למשך תקופה ארוכה יותר בתוצאות קשיי resuspending גלולה (ים).
- Resuspend cDNA pelleted ב40 μl של פוראמיד deionized על ידי חימום עד 65 מעלות צלזיוס למשך 10 דקות, ואחריו vortexing הנמרץ במשך יותר מ 30 דקות לתשומת לבך:. פלטות עשויות להיות בלתי נראות. חוסר אות או אלקטרופורזה הבאה אות החלשה עשוי להיות התוצאה של כישלון לפזר את גלולה כראוי בשלב זה.
סולמות רצף לשמש כסמנים לקביעת עמדת נוקלאוטיד במהלך עיבוד הנתונים. אלה נוצרים באמצעות USB מחזור רצף ערכה (# 78,500), ה-DNA שיש אותו הרצף כמו שלמד את רנ"א, ופריימרים שכותרתו עם D2 WellRed או D1/Lycor 800. בדרך כלל, ה-DNA המועסק בתגובה הזאת תהיה שמשמשת כתבנית שעתוק לרנ"א בשאלה. אף על פי הפרוטוקול שהוצג כאן התגובה דומה מאוד לזו המומלצת על ידי יצרן הערכה, התגובה היא לשנותם פי כמה וכמה. בעוד DDA וDDT משמשים כTerminators שרשרת בתגובות שתוארו להלן, כל זוג Terminators יכול לשמש כדי ליצור את רצף סולמות.
- מערבבים 40 μl של תמהיל סיום DDA, 5 pmol של תבנית ה-DNA, 4.6 μl של חיץ Sequenase 10X, 10 μl של פריימר המסומן D2 WellRed, 4.6 μl של Sequenase ומים כדי להביא את הנפח הכולל עד 82 μl. הוסף Sequenase שעבר. Prepaתגובה מחדש רצף שני באותו אופן, תוך ניצול פריימר DDT וLicor IR800 כותרתו במקום.
- המשך להגברה באמצעות PCR ותנאים מומלצים USB שים לב:. תוספת של שמן מינרלים לא נדרשה ולא מומלץ לפרוטוקולים / cyclers התרמית לנצל מכסה מחומם.
- שלב DDA וDDT רצף תגובות לאחד צינור 1.5 מיליליטר microfuge (~ כוללת μl 164).
- ה-DNA המשקע כדלקמן: הוסף 3 μl 16 ז NaOAc (pH 5.2), 16 מ"מ EDTA μl 100, גליקוגן מ"ג / מ"ל 1 μl 10, ו480 95% אתנול μl. מערבבים היטב, דגירה על 4 מעלות צלזיוס למשך 30 דקות וצנטריפוגות ב XG 14,000 עבור 30 דקות ב 4 ° C.
- Resuspend cDNA pelleted ב100 μl של פוראמיד deionized על ידי חימום עד 65 מעלות צלזיוס למשך 10 דקות, ואחריו vortexing הנמרץ במשך לפחות 30 דקות.
5. חלוקה של מוצרי תגובה על ידי אלקטרופורזה נימי
אלקטרופורזה נימים מאפשרת בו זמניתהפרדה של מוצרי סינתזת cDNA מארבע תגובות ותקווינה למדגם יחיד. שמונה דגימות עשויות להיות מופרדים בו זמנית, ואילו רבים כמו 96 דגימות עשויות להיות מופרדים במהלך ריצה אחת (איור 2).
- מערבבים 40 μl של דגימות SHAPE אספו עם 10 μl של סולמות רצף אספו, ולהעביר לצלחות מדגם 96, גם לתשומת לבך:. זה הכרחי כי חומרים כימיים Beckman Coulter וצלחות (כולל LPA-ג'ל, ריצת חיץ, מוצרי נפט, מדגם פתרון טעינה ומדגם וצלחות חיץ) לשמש עם מנתח הגנטי בקמן קולטר CEQ 8000.
- תכנית ולהכין את מכשיר אלקטרופורזה נימים וליזום לפעול לפי הוראות יצרן. הערה: לקבלת ההחלטה הטובה ביותר של דגימות, השתמש בפרמטרים שפורסמו בעבר CAFA שיטת 28.
באופן אידיאלי, מחוץ לפריימר ופסגות חזקות הפסקה, אותות לכל שיא בכל הארבעה electropherogram לאגזעים צריכים להיות בטווח יניארי; ירידה-off הדרגתי באות מקובלת. לפעמים, לעומת זאת, פסגות גדולות (עצירות) ניכרות גם בתגובת השליטה, ואלה יכולים להפריע לעיבוד נתונים שלאחר מכן. cDNAs הקטוע המקנים לפסגות האלה יכולים להיות התוצאה של מכשול טבעי במהלך שעתוק לאחור (מבנה המשני למשל RNA), או השפלה RNA. במקרה הראשון, תוספים כגון betaine עשויים לשפר processivity RT ולהפחית RT השהיית סיום / מוקדם מדי.
עיבוד נתונים
תוכנה מאפשרת למשתמש ShapeFinder לדמיין ולהפוך עקבות CE ולהמיר אותם לפרופילי תגובתיות SHAPE 18. ברגע שערכי תגובתיות הם נספרו, הם תקין ומיובאים לRNAStructure (V5.3) כדי ליצור ולחדד את מודלים מבניים משניים.
6. ShapeFinder תוכנה
הארכת BaseFinder עקבות העיבוד פלאטטופס 29, הגרסה שפורסמה בShapeFinder הוא זמין באופן חופשי לשימוש לא מסחרי 18. הוראות מפורטות לטיפול בנתונים בShapeFinder מסופקות עם התיעוד של התוכנה.
- Electropherograms מיובא מCEQ לShapeFinder, שבו הם מותאמים לתקן ל( i) רקע ניאון, (ii) חפיפה ספקטרלית בין ערוצי ניאון, (iii) ניידות המשמרות הנחילה ידי פריימרים מתויגים באופן שונה, (iv) הבדלים בעוצמת פריחה של מוצרים נפוצים שכותרתו עם fluorophores שונה, ו( V) דעיכת אות כתוצאה מסיום המוקדם של שעתוק לאחור.
- פונקצית "ההגדרות" של "יישור ושלב את" הכלי בShapeFinder מקצה אוטומטית זהויות לפסגות בודדות וקושרת את זה לרצף הרנ"א כהגדרתו על ידי קלט המשתמש ושני סולמות רצף. למרות הקצאות ראשוניות הן בדרך כלל לא מושלמים, ניתן לתקן שגיאות באופן ידני באמצעות פונקצית "שינוי" שלבאותו הכלי. לבסוף, הפונקציה "Fit" מחשב את האזורים תחת מזדהה (+) ו (-) פסגות תגובה, ותגובתיות tabulates ערכים אלה יחד עם מספר נוקלאוטיד המקביל בקובץ טקסט מופרד באמצעות טאבים.
הערה: מניתוח הנתונים הוא קריטי לדיוק של צורה, וכמה שיקולים חשובים מאוד בניתוח זה, ובכלל זה:
- אות לרעש: יחס אות לרעש צריך להיות כזה שהפסגות הבודדות צריכים להיות קל לזיהוי אפילו לעמדות עם תגובתיות נמוכה. למרות ShapeFinder מספק אפשרות החלקת נתונים; אמורה לשמש חלופה זו מאוד בזהירות, כפי שהוא יכול להטות ניתוח שלאחר מכן.
- אזור של ניתוח: בדרך כלל, ניתן לקבל מcDNAs הארוך NT 300-600 נתונים מהימנים, החלו באזור 40-80 NT הוסרו מפריימר '3 תחנה סופית, ושהסתיים כדועך האות לרמות הקשה להבחין בין רעש רקע. שימוש רבסטי פריימר ple יידרשו לנתח מתיחות ממושכות יותר של רנ"א. במקרה זה, מומלץ שאת החפיפה באות אמינה בין סטי פריימר היא בטווח של 30-50 NT. על RNAs קצר יותר, שבו רברס טרנסקריפטאז לעתים קרובות מגיע לסוף תבנית RNA, יש להקפיד שלא לכלול אותן פסגות שאות יחס רעש מושפע DNA סינתזת התחנה החזקה.
- דעיכת אות: ריקבון האות קשור למידה של רנ"א שינוי במהלך הניסוי, כמו גם processivity מושלם של RT. באופן אידיאלי, קינטיקה יחידה פגיעה יחסית לאזור של רנ"א להיות מנותחים צריכה להיות מושגת על מנת למקסם לקרוא אורך. Shapefinder מכיל כלי שהוא יעיל בתיקון לדעיכת אות, עם זאת, כי זה נוטה להציג את השגיאה לניתוח - במיוחד כאשר קינטיקה להיט אחד לא ציינה, הוא משמש הטוב ביותר כאשר ריקבון אות הוא מינימאלי (כלומר, כאשר חלוקת פסגות היא בקנה אחד עם אחת פגע kineטיקים). לאחרונה, אלגוריתמים משופרים להפיכת דעיכת אות אות כבר פורסם 30 ויש לחקור אם ריקבון אות הוא דאגה מיוחדת בניסוי מסוים.
- לאותת קנה מידה. ניתן לטעון את הצעד השרירותי ביותר בעיבוד נתונים צורה, פרופיל השליטה צריך להיות מדורגים, כך שעוצמות השיא בין תגובתי מינימאלית (+) ו (-) עקבות שווה. קנה מידה של שליטת העקבות למידה גדולה מדי תגרום לשפע של ערכי תגובתיות שליליים ברבעון הראשון (ראה נורמליזציה נתונים בהמשך). במקרה זה, גורם קנה המידה צריך להיות מופחת בהתאם, ואת הנתונים משוחזרים.
- פסגות משימה. באופן כללי, את הגרסה האוטומטית של הקצאת השיא עובדת היטב. כאשר התהליך נכשל, עם זאת, זוהי החובה של המשתמש לוודא שכל הפסגות הוכרו על ידי התוכנה, במיוחד כאשר יחס אות לרעש נמוך. פסגות כתף, למשל, לא תמיד מזוהות, וה-G-עשיר sequences נדחסים לעתים קרובות.
7. נורמליזציה נתונים
לשלב פרופילי תגובתיות נוקלאוטיד לתוך אלגוריתם המבנה המשני בשימוש על ידי RNAStructure תוכנה (V5.3), ו / או להשוות את הפרופילים של RNAs הקשורים באופן הדוק, נתוני צורה חייבים להיות מנורמלים באופן סטנדרטי 12. זה כולל (i) למעט חריגים מחישובים שלאחר מכן, (ii) הקובעים את תגובתיות "היעילה המרבית" (כלומר, הממוצע של 8% הגבוהים ביותר של ערך תגובתיות, למעט חריגים), וכן (iii) נורמליזציה על ידי חלוקת כל ערכי תגובתיות על ידי "מקסימום היעיל", כמפורט להלן:
- פתח את קובץ הטקסט המופרד באמצעות טאבים שנוצר לאחר יישור ואינטגרציה ולהעתיק את התוכן שלו לתוך גיליון אלקטרוני Excel. העמודה הימנית ביותר של קובץ זה (RX.area-BG.area) מכילה את ערכי תגובתיות SHAPE המוחלטים מחושבים לכל נוקלאוטיד של רנ"א. עמודות השמאליות ביותר מתייחסות לתגובתיות זו sequ RNAence.
- לחשב ולאחסן את הרבעון הראשון ובשליש (כלומר, לאחוזון ה -25 וה -75) לערכים (RX.area-BG.area) באמצעות פונקצית אקסל "= רבעונים (מערך, ליטר)"
- לחשב ולאחסן את הבדל הרבעונים "= רבעונים (מערך, 3), רבעונים (מערך, 1)"
- לחשב ולאחסן את "ערך סף outlier" תוך שימוש בנוסחה "= (הרביעון, מערך, 3) * 1.5 ((אחוזון (מערך, 3), רבעונים (מערך, 1))". כל תגובתיות ערכים גדולה מערך זה שלא להיכלל בחישובים שלאחר מכן.
- להעתיק את הערכים מתגובתיות (RX.area-BG.area) ולהדביק אותם לעמודה סמוכה, ריקה, ואז למיין את הערכים הללו כאלה שהגדולים ביותר הם בחלק העליון של העמודה.
- ב" טור הערכים מסודרים "חדש שנוצר, מחק את הערכים גדולים מערך סף הנתון החריג.
- חישוב ואחסון הממוצע של 8% הגדולים ביותר של ערכי תגובתיות יתר ב" מסודרים על הערכים ColuMN ". ערך זה הוא" תגובתיות היעילה המרבית ".
- מחלקים את ממוין (RX.area-BG.area) של כל נוקלאוטיד (כולל חריגים) על ידי ערך תגובתיות "היעיל המרבי" כדי להשיג את "ערכי תגובתיות המנורמלים". לאחסן אלה בעמודה ריקה, והשאיר את עמודה ריקה בצד השמאל. לאחר מכן, העתק את מספרי נוקלאוטיד בצד השמאל של הטבלה ולהדביק אותם בעמודה הריקה ישירות משמאל את "ערכי תגובתיות המנורמלים".
- להעתיק ולהדביק את זוגות ערך תגובתיות עמדה-מנורמל נוקלאוטיד לתוך עורך טקסט.
- לחסל את הערכים מתחת -0.09 (כלומר, להשאיר ריק רווחים), כמו אלה הם ככל הנראה התוצאה של RT השהיית במהלך סינתזת cDNA מסיבות אחרות מאשר שינוי הכימי של התבנית. יתר על כן, כל ערכי תגובתיות לנוקלאוטידים שבו חזק השהיית הוא ציין בתבנית ללא שינוי (כפי שנקבע על ידי בדיקה ויזואלית של "יישור ושלב את" פרופיל ShapeFinder), צריכים להיות שליליים.
- Save את הקובץ עם סיומת. צורה לשימוש בניתוח מבני עם RNAstructure תוכנה (V5.3).
8. מידול נתונים
RNAstructure תוכנה (V5.3) משמשת כדי לחזות מבנה המשני בניסוי RNA-נתמך (ים) תוך שימוש באנרגיה ללא אילוצים פסאודו נגזרים מניתוח SHAPE 19. התוכנה מספקת ייצוג גרפי של המבנים הנמוכים האנרגיה 2D RNA, כמו גם ייצוג טקסטואלי של מבנים אלה בסימון הדוט סוגר. האחרון יכולה להיות מיובא בצופת מבנה RNA של ההעדפה של המשתמש, לדוגמה Pseudoviewer 23 או 22 ורנה, כדי להפיק תמונות באיכות פרסום.
הערה: יש להקפיד כאשר בוחנים את המבנים המיוצרים על ידי RNAstructure התוכנה (V5.3). לדוגמה, התוכנה לא יכולה לפתור את אינטראקציות שיישונים כגון pseudoknots ולולאות מתנשקות, וגם לא יכול להבחין אם זה חוסרתגובתיות באזור מסוים בשל הגנת basepairing או סטרית על ידי חלבונים מאוגדים. כתוצאה מכך, גורמים אלה, יחד עם האנרגיות שדווחו למבנים הבודדים, יש לקחת בחשבון בעת הצגת מודל מבני מוחלט.
תוצאות
RNA המכיל את רכיב ה-HIV-1 rev התגובה (RRE) ו'3 קלטת מבנה טרמינל 4 הוכנה מפלסמיד לינארית ידי שעתוק במבחנה, אחרי שהיא הייתה מקופלת על ידי חימום, קירור, ודגירה על 37 מעלות צלזיוס בנוכחות של MgCl 2. RNA נחשף לNMIA ולאחר מכן להפוך עיבד מ פריימר 5'-DNA הסוף שכותרתו הכלאה לקל?...
Discussion
אנו מציגים כאן פרוטוקול מפורט עבור צורה של תפוקה גבוהה, טכניקה המאפשרת קביעת מבנה משנית לרזולוצית נוקלאוטיד יחידה לRNAs בכל גודל. יתר על כן, צימוד נתוני צורה ניסיונית עם אלגוריתמים לחיזוי מבנה משניים מאפשר יצירת המודלים 2D RNA עם רמה גבוהה יותר של דיוק מאשר אפשרי עם כל אח?...
Disclosures
אין ניגודי האינטרסים הכריזו.
Acknowledgements
ס Lusvarghi, ג'Sztuba-סולינסקי, KJ Purzycka, ג"ו ראוש וSFJ Le גרייס נתמך על ידי תכנית המחקר העירונית של המכון הלאומי לסרטן, המכונים הלאומי לבריאות, ארה"ב.
Materials
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5' labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied - including any 3'terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies | ||
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 '-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 '-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2'-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues - A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved