A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
רזולוציה גבוהה ניתוח Spatiotemporal של קולטן Dynamics ידי המולקולה בודדת מיקרוסקופ פלואורסצנטי
In This Article
Summary
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
Abstract
מיקרוסקופיה המולקולה בודדת מתגלה כגישה רבת עוצמה כדי לנתח את ההתנהגות של מולקולות איתות, בפרט בנוגע לאלו היבט (למשל., קינטיקה, דו קיומן של מדינות שונות ובאוכלוסיות, אינטראקציות חולפות), אשר מוסתרות בדרך כלל במדידות הרכב, כגון אלו שהושגו עם שיטות ביוכימיות או במיקרוסקופ רגילה. לפיכך, אירועים דינמיים, כגון אינטראקציות רצפטור לרצפטור, יכולים להיות מיושמים בזמן אמת בתא חי עם רזולוציה spatiotemporal גבוהה. פרוטוקול זה מתאר שיטה המבוססת על תיוג עם fluorophores האורגני קטן ובהיר וקרינת השתקפות הפנימית מוחלטת מיקרוסקופיה (TIRF) ישירות לדמיין קולטנים בודדים על פני השטח של תאי חיים. גישה זו מאפשרת למקם במדויק קולטנים, למדוד את הגודל של מתחמי הקולטן, וללכוד אירועים דינמיים כגון אינטראקציות קולט קולט חולפות. הפרוטוקול מספק תיאור מפורט של איך perform ניסוי מולקולה בודדת, ובכלל זה הכנת מדגם, רכישת תמונה וניתוח תמונה. כדוגמא, היישום של שיטה זו כדי לנתח שני G-חלבון בשילוב קולטנים, כלומר., Β רצפטור סוג חומצת B-2 adrenergic וγ-aminobutyric (GABA-B), הוא דיווח. הפרוטוקול יכול להיות מותאם לחלבונים אחרים קרום ודגמים סלולריים שונים, שיטות transfection ואסטרטגיות תיוג.
Introduction
קולטנים הממוקמים על פני התא לחוש את הסביבה תאית ולהגיב למגוון של גירויים, כמו הניחוח, יונים, נוירוטרנסמיטורים והורמונים קטנים חלבון גדולים. טבע הנוזל של קרומים תאיים מאפשר תנועות של קולטנים וחלבונים קרום אחרים. זה חיוני להיווצרות של קומפלקסי חלבונים ואת המופע של אינטראקציות בין חלבונים חולפים, כגון אלו המשמשים על ידי קולטנים להרכיב ליחידות פונקציונליות וtransduce אותות לתוך התא הפנימי. לדוגמא, ה-G--חלבון בשילוב קולטנים (GPCRs), המהווה את המשפחה הגדולה ביותר של קולטני 1 על פני קרום התא, הוצעו להקים di-/oligomers, המופיע להיות מעורבים בוויסות מכויל של התמרה אות ו אולי יש השלכות פיסיולוגיות ותרופתיות חשובות 2-5.
יש מיקרוסקופיה המולקולה בודדת את הפוטנציאל הגדול של ישירות חזותי גבוה spatiotempרזולוציה אוראלית ההתנהגות הדינמית של קולטנים בודדים הממוקמים על פני השטח של תאי חיים, ובכלל זה הקשר שלהם ליצירת הדימרים וקומפלקסים מולקולריים מסדר גבוה יותר 6-10. זה מציע מספר יתרונות בהשוואה לשיטות ביוכימיות ומיקרוסקופיה רגילות, אשר בדרך כלל לדווח על ההתנהגות הממוצעת של אלפים או מיליון של מולקולות.
תיוג חלבון עם fluorophore מספיק בהיר וphotostable חיוני למיקרוסקופיה מולקולה בודדת. פרוטוקול זה מנצל את תג SNAP הציג לאחרונה 11 לצרף קוולנטית fluorophores האורגני קטן ובהיר לקולטנים על פני קרום תא. SNAP הוא 20 תג חלבון KD נגזר מalkyltransferase אדם תיקון DNA O אנזים 6-alkylguanine-DNA, אשר יכולה להיות מתויג באופן בלתי הפיך עם benzylguanine fluorophore מצומדות נגזרים (fluorophore-BG). CLIP, תג מהונדס נוסף הנגזר מSNAP, יכול להיות מתויג במקום עם fluorophore-cנגזרי benzylcytosine onjugated 12.
הפרוטוקול שפורסם בכתב היד הזה מסביר כיצד transfect והתווית SNAP-מתויג 11 קולטנים עם fluorophores האורגני הקטן ולהשתמש בקרינת השתקפות הפנימית מוחלטת מיקרוסקופיה (TIRF) כדי להמחיש קולטנים בודדים או מתחמי קולטן על פני השטח של תאי חיים 10. התוצאות שדווחו בפרוטוקול> 90% יעילות תיוג של חלבון על פני קרום תא 10 מתויג-SNAP extracellularly. מידע נוסף על אופן השימוש בנתוני מולקולה בודדת כדי לנתח את הגודל וניידות של מתחמי הקולטן, כמו גם כדי ללכוד אינטראקציות קולט קולט חולפות, מסופק. זרימת עבודה סכמטי של הפרוטוקול כולו היא נתון באיור 1. כדוגמא, transfection של סינית שחלות תאים (CHO) עם-G-חלבון בשילוב קולטנים מתויג-SNAP (GPCRs) ואחריו על ידי תיוג עם נגזר fluorophore-BG כמו גם היישום שלה לquantify וdi-/oligomerization קולט הצג מתוארים. פרוטוקול זה יכול להיות מורחב לחלבונים על פני קרום תא אחרים ותגי ניאון (למשל., CLIP), כמו גם לשיטות transfection וסימון אחרות.
Protocol
1. לדוגמא הכנה
- ניקוי coverslip
הערה: עבודה מתחת למכסת מנוע קטר.- להשתמש בפינצטה נקייה למקום coverslips זכוכית (קוטר 24 מ"מ) לבעל coverslip שמפריד coverslips פרט.
- שים את בעל עם coverslips לתוך מבחנה, ולהוסיף כלורופורם עד coverslips מכוסה. מכסה את הכוס בנייר אלומיניום כדי להפחית את האידוי וsonicate בsonicator אמבטיה במשך שעה 1 ב RT. קח את בעל coverslip מהכוס ולתת את coverslips יבש.
- חזור על שלב 1.1.2 עם פתרון 5M NaOH במקום כלורופורם.
- קח את בעל coverslip לתוך מבחנה חדשה ולשטוף שלוש פעמים במים מזוקקים. שים coverslips ניקה בצלחת תרבית תאי זכוכית מלאה 100% אתנול.
- הכנת דוגמאות כיול
- לפזר צבע פלואורסצנטי בממס מתאים.
- הכן דילול סדרה 1:10 של צבע פלואורסצנטי הנעמ13:00 ל1 ננומטר במסנן מעוקר מים (0.22 מיקרומטר).
- קח את coverslips ניקה מאוחסן ב100% אתנול ולשטוף עם מים מעוקרים מסנן. ספוט 20 μl של כל דילול צבע פלואורסצנטי על coverslip ניקה נפרד. בואו coverslips יבש מתחת למכסת מנוע סטרילית. הגן על coverslips מהאור והאבק עד לשימוש. השתמש בדוגמאות אלה כדי להעריך את עוצמתו של מולקולות ניאון יחידה (ראה שלב 3).
- Transfection
- תרבות תאי CHO ב1:01 תערובת הנשר בינוני / התזונתי שונה Dulbecco F-12 (DMEM/F12) בתוספת 10% בסרום שור העובר (FBS), 100 U / ml פניצילין ומיקרוגרם / מיליליטר סטרפטומיצין 100 על 37 מעלות צלזיוס, ב . 5% CO 2 הערה: השתמש בתקשורת חופשית פנול האדום לאורך כל הניסוי כדי למזער autofluorescence.
- קח את coverslips לנקות מ100% פתרון אתנול, לשטוף אותם עם פוספט בופר סליין סטרילי (PBS), ואת מקום coverslip אחד לבאר כל pl תרבית תאים גם 6אכלתי.
- Trypsinize, לספור ותאי CHO הזרע בצפיפות של 3 x 10 5 תאים / היטב בצלחת תרבית תאי 6 היטב המכילה את coverslips. בואו התאים לגדול בחממה (37 מעלות צלזיוס, CO 2 של 5%) ל24 שעות על מנת להשיג כ. confluency 80%, המהווה את צפיפות תאים האופטימלית עבור transfection.
- עבור כל טוב, לדלל 2 מיקרוגרם של ה-DNA פלסמיד הרצוי (למשל., SNAP-מתויג β רצפטור 2-adrenergic) ו6 Lipofectamine μl 2000 בשני צינורות נפרדים המכילים 500 בינוני OptiMEM μl. לדגור על RT במשך 5 דקות.
- לשלב את הפתרונות מהצעד 1.3.4 לתוך צינור אחד ומערבבים לקבלת תערובת transfection. דגירה את תערובת transfection ב RT עבור 20 דקות.
- במהלך הדגירה (1.3.5), לוקח את תאי CHO ולשטוף פעמיים עם מחומם מראש (37 מעלות צלזיוס) PBS. החלף PBS עם 1 מיליליטר / טוב של מדיום DMEM/F12 פנול האדום ללא תוספת 10% FBS אבל לא אנטיביוטיקה.
- מוסיף את כל transfectתערובת יונים (1 מיליליטר) מdropwise 1.3.5 שלב זה לזה היטב, ונער בעדינות את הצלחת קדימה ואחורה כדי להבטיח ערבוב מלא.
- דגירה עבור 2 עד 4 שעות על 37 מעלות צלזיוס, 5% CO 2 ולהמשיך מייד לאחר מכן לשלב הבא הערה:. תנאי transfection אלה כבר מותאמים להשגת צפיפות הקולטן <0.45 חלקיקים / מיקרומטר 2, אשר מתאימות ליחיד הדמיה של מולקולה. התאמות ייתכן שתידרשנה בעת השימוש בתאים, מבנים או חומרים כימיים שונים.
- תיוג חלבון
- לדלל 1 μl של פתרון מניות fluorophore-BG ב 1 מיליליטר בינוני DMEM/F12 בתוספת 10% FBS להשיג ריכוז סופי של 1 מיקרומטר. קח את תאי transfected מן החממה ולשטוף פעמיים עם prewarmed (37 ° C) PBS. החלף PBS עם 1 מיליליטר של תמיסת fluorophore-BG 1 מיקרומטר ודגירה של 20 דקות על 37 מעלות צלזיוס 5% CO 2 באינקובטור.
- לאחר הדגירה, לשטוף את התאים שלושהפעמים עם מדיום DMEM/F12 תוספת 10% FBS, בכל פעם ואחרי 5 דקות דגירה על 37 ° C. קח coverslip (עם תאים שכותרתו) עם פינצטה ולמקם אותו לחדר הדמיה.
- שטוף פעמיים עם 300 חיץ ההדמיה μl. הוסף 300 μl של חיץ הדמיה טרי ופנה מייד להדמיה (חלק 2).
2. רכישת תמונה
הערה: השתמש בקרינת השתקפות פנימית מוחלטת מיקרוסקופ (TIRF), מצויד במטרת נפט טבילה צמצם מספרי גבוה (למשל, צמצם המספרי magnification/1.46 100X.), לייזרים מתאימים (למשל, 405 ננומטר, 488 ננומטר, ננומטר 561 ו645 דיודות לייזר ננומטר), התקן צמוד מטען אלקטרון הכפלה (מצלמה EMCCD), חממה ובקרת טמפרטורה לדמיין מולקולות ניאון אחד.
- הגדר את הפרמטרים מיקרוסקופ הרצויים, כלומר., קו לייזר, זווית TIRF (פרמטר זה שולט penetraעומק tion של שדה חלוף), זמן חשיפה, במסגרת שיעור ומספר תמונות לכל סרט 10. שמור על שליטת דוד / חממה והטמפרטורה תמיד על להימנע מרחף טמפרטורה ועיבוי לחות.
- לשים טיפה של שמן טבילה במטרת 100X של המיקרוסקופ. מניחים את חדר ההדמיה עם התאים שכותרתו על בעל הדגימה של המיקרוסקופ, ולהביא את התאים בפוקוס באמצעות תאורת brightfield.
- לעבור לתאורת TIRF. שמור על כוח הלייזר נמוך ככל האפשר, כדי לאפשר מחפש את התא הרצוי, אך בה בעת מזעור photobleaching.
- בחר את התא הרצוי וקנס להתאים את הפוקוס. הגדר את כוח הלייזר לרמה שמאפשרת הדמיה של fluorophores אחת. רכישת תמונה ברצף ולשמור את קובץ תמונה ברצף גלם. TIFF.
3. כיול (fluorophores יחיד על הזכוכית וmonomeric / בקרת קולטן dimeric)
- להרכיב כל sampl כיולדואר מוכן כפי שתואר ב1.2 בחדר ההדמיה. מניחים כל דגימה על המיקרוסקופ ולבחור את המדגם המכיל כתמים עקיפה מוגבל מופרדים היטב שאקונומיקה בצעד הערה אחת:. כתמים אלה מייצגים מולקולות בודדות של צבע פלואורסצנטי.
- לרכוש רצפי תמונת TIRF כמתואר בשלב 2 חשוב:. אותו הפרמטרים ההדמיה חייבים לשמש את כל הניסויים, כולל אלה לכיול.
- לבצע זיהוי ומעקב וניתוח, כמפורט ב4.1-4.2. חלץ את העצמה של כל חלקיק, כמתואר ב4.2.6. מתוך נתונים אלה, לחשב את הממוצע (μ) וסטיית התקן (σ) של עוצמת fluorophores אחת.
- אופציונאלי: לבצע את אותו ניתוח על תאי transfected עם קולט monomeric על פני קרום תא (. למשל, CD86), N-סופני מתויג עם עותקים אחד או שתיים מSNAP 10 ומסומן עם נגזר fluorophore-BG. Follow ההליך המתואר לעיל עבור fluorophore היחיד על זכוכית. להעריך את יעילות התיוג כמתואר בCalebiro, ד 'ואח'. 10
4. ניתוח תמונה
- הכנת תמונה ברצף
- השתמש בתוכנת עיבוד תמונה (למשל., ImageJ) כדי לחתוך את התמונות.
- להציל את המסגרות בודדות כנפרדים. תמונות TIFF בתיקייה חדשה, מה שמצביע על כל תמונה את מספר המסגרת.
- למדוד את השטח פני תא על ידי ציור אזור של עניין (ROI) לאורך קווי המתאר של תא ושימוש בכלי המדידה בImageJ או כלי דומה בתוכנה אחרת. השתמש בערך זה כדי לחשב את צפיפות חלקיקים על ידי חלוקת המספר הכולל של חלקיקים בתחילת הסרט על ידי השטח פני התא.
- גילוי חלקיקים ומעקב
הערה: השתמש בתוכנה לא מסחרי כגון u-מסלול 13, עובד ב-Matlabסביבה, כדי לזהות באופן אוטומטי ולעקוב אחר חלקיקי קולטן אחד.
הערה: אלגוריתם u-המסלול מבוסס על גישת המעקב מרובה השערה. גישה זו מקשרת בין חלקיקי מסגרות ידי בניית מטריצות עלות, שבו ההסתברויות בודדות שחלקיקים ניתנים במסגרת אחת מתאימה לחלקיקים הניתנים במסגרת הבאה, מופיעים, נעלמים או מתמזגים / מפצל עם / מחלקיקים אחרים שהוקצו. הפתרון שברחבי עולם מצמצם את העלויות, כלומר., אחד עם ההסתברות הגבוהה ביותר, סוף סוף נבחר. זה גם מאפשר מעקב אחר חלקיק באופן זמני ונעלם, תופעה אופיינית הנגרמת על ידי מצמוץ fluorophore. הגרסה האחרונה של u-מסלול (2.1.0) יש ממשקי משתמש גרפיים המאפשרים ביצוע של ניתוחים אלו.- משורת הפקודה Matlab, "movieSelectorGUI" סוג כדי לפתוח את ממשק בחירת סרט. בצע את ההוראות ליצירהאתר סרט חדש החל מ התמונות נפרדות שנשמרו בעבר (ראה 4.1.2).
- לספק את גודל פיקסל בננומטר, מרווח זמן בשניות, צמצם מספרי, עומק מצלמה קצת ואורך גל פליטה של fluorophore, הנדרש לגילוי חלקיקים ומעקב. שמור את מסד הנתונים של הסרט.
- מממשק בחירת סרט, להפעיל את הניתוח, בחירה "חד חלקיקים" כסוג של אובייקטים. חלון חדש יופיע בו ניתן להגדיר את הפרמטרים המשמשים לגילוי חלקיקים ומעקב. בגין עם הפרמטרים ברירת המחדל. לאחר מכן, להתאים את הפרמטרים האלה אם איכות הגילוי ו / או המעקב אינה משביעה רצון (למשל, כמה חלקיקים אינם מזוהים או מסלולים מקוטעים) אופציונלית:. תחת הגדרות המעקב, לבדוק "תוצאות מעקב יצוא לפורמט מטריקס" לאחסון הקואורדינטות ואמפליטודות של כל החלקיקים במטריצה יחידה (שדה שנקראה "trackedFeaturedInfo"). לתיאור מפורט של פרמטרים אלה, עיינו בתיעוד של u-המסלול.
- הפעל את אלגוריתם זיהוי. אלגוריתם זה באופן אוטומטי לקבוע את המיקום ועוצמה מעל הרקע של כל נקודה עקיפה מוגבל (כלומר., מתחמי קולטנים יחיד / קולטן) על ידי התאמת פונקצית גאוס דו ממדים עם סטיית תקן שווה לפונקצית נקודת ההתפשטות של מיקרוסקופ סביב מקסימום עוצמה מקומי . לאחר מכן, הפעל את אלגוריתם המעקב. אחסן את התוצאות של הניתוחים בקובץ מחצלת..
- השתמש בשגרת "movieViewer", כלולה בחבילת u-המסלול, או אלה מותאמים אישית דומים כדי להמחיש את הרצועות ולבדוק את איכות זיהוי והמעקב.
- פתח את קובץ המחצלת. לראות את המיקום ואת המשרעת (כלומר., עוצמה) של החלקיקים במעקב בכל מסגרת. הנתונים שנוצרו בשלב 4.2.4 כלולים בתחום tracksCoordAmpCG של ar tracksFinalקרן ו / או בtrackedFeaturedInfo. מהמספר הכולל של חלקיקים זוהו לחשב את צפיפות חלקיקי חלוקת ערך זה על ידי שטח הפנים של תאים נמדד ב4.1.3.
- אופציונאלי: השתמש בחלקיקים מרכז לאורך זמן (ראה 4.2.6) כדי לנתח את התנועה של חלקיקי הקולטן. לחשב את התקות הממוצעים מרובעות (MSD) ומקדמי דיפוזיה (ד ') באמצעות Matlab או תוכנה דומה. לכל חלקיק וכל מרווח זמן (t Δ) נחשב, לחשב את העקירה מרובעת הממוצעת (MSD), תוך שימוש בנוסחה הבאה:
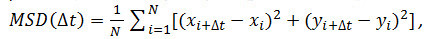
כאשר t Δ הוא מרווח הזמן במסגרות, N הוא מספר הצעדים ניתחו, x ו-y הוא x ו-y של חלקיק קואורדינטות במסגרת מצויינים על ידי האינדקס. השתמש MSD על חלקות זמן כדי להעריך את הסוג של תנועתו של חלקיק נתון: יחסים לינאריתמצביע על דיפוזיה חופשית (כלומר., תנועה בראונית), עקמומיות חיובית (כלומר., העקומה נראית כמו פרבולה) מציעה תנועה מכוונת, עקמומיות שלילית מעידה על תנועה מוגבלת 14. במקרה של חלקיקים לשדר באופן חופשי, לחשב את מקדם הדיפוזיה (D) של כל חלקיק על ידי התאמת נתוני MSD שהושגו עם המשוואה הבאה: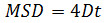
- חישוב של גודל חלקיקים בשיטת המדידה גאוס
הערה: ברגע שחלוקת עוצמת דגימות כיול (fluorophores אחת על זכוכית ו / או קולטנים monomeric שכותרתו fluorescently) ידועה, לבצע מעורב בכושר גאוס על חלוקת עוצמות חלקיקים בתחילת תמונה ברצף כדי לקבוע את הגודל של קומפלקסי רצפטור (כלומר., מספר הקולטנים לכל חלקיק) 10. לבצע ניתוחים אלה באמצעות Matlab או sתוכנת imilar.- לחשב את העצמה של כל חלקיק על ידי חישוב ממוצע עוצמת החלקיקים מהפריים הראשון למסגרת לפני השינוי הראשון בעוצמת התרחש (ברוב המקרים ירידה בשל photobleaching).
- לבצע התאמת גאוס מעורבת על פי המשוואה הבאה:
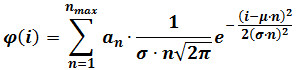
שבו φ (i) הוא התדר של חלקיקים שיש עוצמת i, n הוא מספר הרכיב, α n הוא פרמטר שתורם לגובה של n רכיב, μ וσ הם ממוצע וסטיית תקן של עוצמת fluorophores אחת ההתייחסות . (מחושב כמתואר בשלב 3) הערה: קבע tהוא מספר מקסימאלי של רכיבים (מקסימום n) עבור כל תמונה ברצף על ידי בהדרגה להגדיל את מקסימום n עד התוספת של מרכיב זה כבר לא מייצר סטטיסטי טוב יותר הולם, נשפט על ידי F-בדיקה. - אופציונאלי: (. למשל, 60 המסגרות האחרונות של רצף מסגרת 400) בצע מעורב בכושר גאוס על חלוקת העצמה שהושגה במסגרות האחרונות של הסרט. החלף μ וσ עם הערכים המתקבלים לאחר זה ראוי, שמספקים הערכות מעודנות לפרמטרים אלה, וחזרו על שלב 4.3.2.
- לחשב את השטח מתחת לעקומה (AUC) של כל מרכיב כושר גאוס מעורב. לחשב את השפע היחסי של חלקיקי הקולטן של גודל שונה (כלומר, מונומר, דימר, trimer, וכו ') על ידי חלוקת שווי AUC של כל רכיב על ידי AUC של כל ההפצה.
- אופציונאלי: השתמש בנתונים מהתאים שונים והצפיפויות המקבילה חלקיקים (מחושבים כפי שמתואר ב4.2.6) כדי ליצור חלקות שבו ההפצה של חלקיקים בעלי גודל שונה מתואמת עם חלקיקי צפיפות 10.
- חישוב של גודל חלקיקים בשיטת צעד הולם
הערה: השתמש בניתוח צעד לגוף כמו שיטה חלופית כדי לקבוע את הגודל של מתחמי קולט 10. הבסיס לניתוח זה הוא כי ההרס הנגרם האור (photobleaching) של תוצאות בודדות fluorophore בהיעלמותה מיידית - וכך חלקיקים המכילים fluorophores n צפוי בהדרגה אקונומיקה הפקת פרופיל עוצמת בצעדי n.- לחלץ פרופילי עוצמה של כל חלקיק מקובץ המחצלת. נוצר על ידי u-מסלול או בתוכנת איתור / מעקב דומה (ראה 4.2.6).
- השתמש באלגוריתם צעד הולם, כגון זו שהוצגהנ"צ. 10, כדי לספור את מספר צעדי הלבנת עבור כל חלקיק.
- אופציונאלי: להשתמש בתוצאות כדי ליצור הפצות המציגות את השפע היחסי של חלקיקי הקולטן של גודל שונה ולתאם אותם עם צפיפות חלקיקים כפי שתואר לעיל לתוצאות הראויה גאוס המעורבים (ראה 4.3).
תוצאות
הפרוטוקול המתואר ניתן ליישם במגוון רחב של חלבוני קרום שונים. כדוגמא, נציג תוצאות שהושגו עם β-adrenergic קולטנים 2 וGABA-B מדווחים 10. מאז אותות ניאון ממולקולות בודדות חלשים, מזעור הקרינה רקע הוא צעד המפתח הראשון לתוצאות מוצלחות. לכן, חשוב להשתמש coverslips ניקה בהרחבה
Discussion
הפרוטוקול המתואר מאפשר ניתוח של סידור המרחבי, הניידות וגודל של מתחמי קולטן על פני קרום התא ברמת מולקולה בודדת. בהשוואה לשימוש בחלבוני ניאון, תיוג עם fluorophores האורגני קטן, שהם בהירים וphotostable יותר, יש יתרון בכך שהתיר להדמיה ממושכת של חלקיקי קולטן אחד. מאז רמות ביטוי נמוכו?...
Disclosures
The authors declare that they have no competing financial interests.
Acknowledgements
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
Materials
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
References
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved