A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
טכניקת הארכת פריימר בהתבסס הקרינה לקביעת נקודות התחלת תעתיק ומחשוף אתרים של RNases
In This Article
Summary
We here describe a fluorescence based primer extension method to determine transcriptional starting points from bacterial transcripts and RNA processing in vivo using an automated gel sequencer.
Abstract
הארכת הקרינה מבוססת פריימר (FPE) היא שיטה מולקולרית כדי לקבוע נקודת התחלת שעתוק או אתרי עיבוד של מולקולות RNA. זו מושגת על ידי שעתוק לאחור של ה- RNA של עניין באמצעות פריימרים שכותרתו fluorescently ספציפיים וניתוח בדיעבד של שברי cDNA וכתוצאה מכך על ידי denaturing ג'ל אלקטרופורזה polyacrylamide. במקביל, תגובה סנגר רצף מסורתי מנוהלת על הג'ל למפות את הקצוות של שברי cDNA לבסיסים המקביל המדויקים שלהם. בניגוד ל5'-RACE (Rapid ההגברה של קצות cDNA), שבו המוצר חייב להיות משובט ומספר מועמדים רבים רצף, התפזורת של שברי cDNA נוצרו על ידי סיומת פריימר יכולה להתגלות בו זמנית בטווח ג'ל אחד. בנוסף, ההליך כולו (משעתוק לאחור לניתוח סופי של התוצאות) ניתן להשלים ביום עבודה אחד. על ידי שימוש בפריימרים שכותרתו fluorescently, השימוש באיזוטופ רדיואקטיבי מסוכן שכותרתו חומרים כימייםניתן להימנע וזמני עיבוד צומצמו ניתן לאתר מוצרים במהלך הליך אלקטרופורזה.
בפרוטוקול הבא, אנו מתארים שיטת סיומת פריימר ניאון in vivo באופן מהיר כדי לזהות קצוות '5 של RNAs להסיק תעתיק נקודות התחלה ואתרי עיבוד RNA (למשל, על ידי רכיבי מערכת רעלן-אַנְטִיטוֹקסִין) בס aureus, E. coli וחיידקים אחרים.
Introduction
סיומת פריימר 1 היא שיטה מולקולרית כדי לקבוע קצוות '5 של מולקולות RNA ספציפיות עד רזולוציה בסיסית אחד. היתרון לשיטות אחרות כגון 5'-RACE (הגברה מהירה של cDNA מסתיימת) הוא זמן אספקה המהיר והיכולת לנתח תערובת של אורכים שונים של מולקולות RNA בקלות.
שיטה זו פועלת על ידי חשיפת מולקולות RNA להפוך תגובות שעתוק באמצעות פריימרים ספציפיים ניאון, יצירה שברי cDNA של אורכים מסוימים. מולקולות cDNA אלה לרוץ לצד תגובות סנגר רצף מסורתי 2 על ג'לים polyacrylamide denaturing ויכולות להיות מזוהות על ידי הקרינה שלהם בשל השימוש בפריימרים שכותרתו fluorescently. האורכים של ברי cDNA מוערכים על ידי השוואה לסולם הרצף, המאפשרים המיפוי של 5 קצות RNA ".
באופן מסורתי, תגובות סיומת פריימר משמשות בשילובעם איזוטופים רדיואקטיביים לזיהוי תרכובות cDNA על צילומי רנטגן. בשל סכנות בריאותיות, בעיות סילוק פסולת ולהקל על טיפול, פרוטוקולים חדשים יותר לנצל הקרינה לגילוי סיומת פריימר עם sequencers האוטומטי, אם כי הרגישות שלהם נמוכה במקצת. באמצעות פריימרים שכותרתו fluorescently, ניתן להשמיט את הליך רדיו תיוג החוזר, כצבעי יסוד ניאון הם יציבים במשך זמן רב (יותר משנה בידיים שלנו).
השיטה שאנו מתארים כאן מנצלת רצף ג'ל אוטומטי, אך עם שינויים קלים, sequencers נימים יכול לשמש גם להפרדת cDNA וגילוי 3. הטבע המקביל של ניתוח ג'ל מאפשר לזהות אפילו כמות קטנה של מחשוף RNA או עיבוד. יתרון נוסף הוא ברזולוציה הגבוהה של שיטה זו, כמחשוף או עיבוד של אף אחד בסיס מסוף ניתן לאתרם.
בהקשר לזיהוי של מחשוף RNA או עיבוד, typically שני סוגים שונים של הרחבות פריימר הם מכובדים. במקרה אחד, הטיפול אנזימטי נעשה במבחנה באמצעות RNA מטוהר ואנזים מטוהר, ואילו במקרה האחר, העיבוד נעשה in vivo ו- RNA וכתוצאה מכך הוא מטוהרים. בשני המקרים RNA הוא נתון לרחבת פריימר בוצעה במבחנה, עם זאת, בהתאם למקור של RNA, השיטה היא גם נקראת סיומת פריימר vivo במבחנה או ב. בפרוטוקול אנו מציגים כאן, אנו מתמקדים אך ורק על in vivo סיומת פריימר, בגלל קלות שימוש (אין חלבונים מטוהרים צורך) ואת האפשרות לקבוע נקודות התחלת תעתיק ועיבוד באותו הזמן. עם זאת, במבחנה הרחבות פריימר באופן עקרוני להגדיר באותה הצורה ופרוטוקול זה יכול לשמש כנקודת התחלה.
השיטה מאוירת כאן יכולה להיות מיושמת על מינים רבים של חיידקים, כל עוד הם ניתנים לגבוהיםהכנת טוהר ותשואה גבוהה של חומצות גרעין.
המחקר במעבדה שלנו מתמקד בהיקף הרגולציה של מערכות רעלן-נסיוב (ת א) 4,5, תחום שבו נעשתה שימוש נרחב בשיטת הרחבה פריימר. מדד ת"א מערכות אלמנטים גנטיים קטנים הנמצאים בגנומים פרוקריוטים שמורכבים מחלבון רעיל יציב ואופן אנדוגני פעיל וחיסון אנטי-חלבון או RNA בעיקר לא יציב שמנטרל רעילות 6,7. לפעמים פעילות רעלן מופעלת על ידי עיכוב של שכפול, סינתזת דופן תא או מנגנונים אחרים, אבל לרוב על ידי פעילות RNase 8,9. בדרך כלל, סגוליות RNase נקבע על ידי ביצוע בדיקות שונות, שאחד מהם היא שיטת סיומת פריימר. תגובות סיומת פריימר הם גם מתאימות ליישום זה, כתערובת של שברי אורך בקעו ומלאים ניתן לנתח בו זמנית כדי לקבוע את '5 מסתיים. באמצעות שילוב של במבחנה וברחבות פריימר vivo,מחשוף RNase רעלן ספציפי, לדוגמא, ניתן לקבוע סגוליות רצף 10-13.

איור 1. סקירה כללית של הליך סיומת פריימר. תרבויות חיידקים מודגרת וטופלו בהתאם לצרכי הניסוי. RNA סה"כ מופק מהתאים, שטופל בDNase אני כדי להסיר עקבות DNA ונתונים לתגובת שעתוק לאחור באמצעות פריימרים יעד ספציפיים ניאון DNA מניב cDNA. DNA או פלסמידים הגנומי מופקים ולאחר מכן משמש לתגובות סנגר רצף ניאון להשוואת גודל עם שברי cDNA. מוצרי סיומת פריימר מנוהלים על צד מוצרים סנגר רצף על ג'ל polyacrylamide האוריאה denaturing וניתחו עם לייזר ומיקרוסקופ אוטומטיים. בסיס הרצף ששורות עם להקת cDNA הוא להרח בסיס של 5 סוף cDNA '(החץ הכחול). מידע נוסף ב Fekete, et al. 3 אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
ניתן למצוא סקירה של ההליך כולו סיומת פריימר באיור 1. בקצרה, תאי חיידקים בתרבית, שנקטפו, התא גלולה lysed וRNA חילוץ. RNA המטוהר לאחר מכן טופל בDNase אני כדי להסיר עקבות של מולקולות DNA אשר יכול לפעול כתבניות לטרנסקריפטאז ההפוך. פריימרים ניאון ספציפיים מתווספים ל- RNA, כלאה אל האזור של עניין, ולאחר מכן להפוך את תמלילי עדויות, וכתוצאה מכך דנ"א יחיד שנתקע משלים (cDNA). סולם רצף נוצר על ידי סנגר רצף מסורתי העסקת פריימרים ניאון ומופרד על ג'ל polyacrylamide denaturing צד של שברי cDNA סיומת פריימר. וכתוצאה מכךג'ל מנותח על ידי השוואת להקות ניאון, המאפשרת זיהוי של קצוות '5 של עניין. נקודות התחלת תעתיק ואתרי עיבוד מוערכים בנפרד על-ידי השוואות רצף.
Protocol
1. תשואה גבוהה RNA הכנה
- בידוד RNA
הערה: ריכוזים גבוהים של רנ"א הכל יש צורך בתגובת סיומת פריימר. ערכות טור ספין בדרך כלל לא תניב את כמות הרנ"א צורך (~ 5-16 מיקרוגרם ב5 μl נפח). לכן טיהור תוך שימוש בשיטת חילוץ thiocyanate-פנול, כלורופורם guanidinium החומצה מומלצת, המפורטים להלן.
הערה: פנול הוא מסרטנת, רעיל ומאכל. אנא קראו את גיליונות נתוני בטיחות חומרים ושימוש במנדף עם הגנה מתאימה!- לגדול או לטפל בתאי החיידקים (ס aureus או E. coli בדוגמא זו) כרצוי וקציר על ידי צנטריפוגה 10 דקות ב 4,600 × גרם ו -4 מעלות צלזיוס. הערה: בדרך כלל אנחנו קוצרי OD כולל של 600 20 - 70. כדורי תא יכולים להיות מאוחסנים במשך כמה שבועות ב -20 מעלות צלזיוס.
- Resuspend התא גלולה ב 1 מיליליטר של תמיסת thiocyanate-פנול, כלורופורם guanidinium החומצה ולהעביר ל2 מיליליטר לדפוק כוס המכילה 0.5 מיליליטר של חרוזים זירקוניום / סיליקה זכוכית 0.1 מ"מ.
- Lyse התאים שלוש פעמים במקצף הכנה מהירה / חרוז על 6.5 מ '/ שנייה למשך 30 שניות לשלושה סיבובים, קירור דגימות קרח למשך 5 דקות אחרי כל הסיבוב. הערה: מדגם הומוגני יכול להיות מאוחסן ב -80 מעלות צלזיוס למשך מספר שבועות.
- דגירה lysate למשך 5 דקות ב RT ולאחר מכן להוסיף 200 μl של כלורופורם.
- במרץ לנער או מערבולת במדגם למשך 30 שניות כדי לחלץ RNA.
- לדגור על RT במשך 3 דקות אז צנטריפוגות במשך 15 דקות ב13,000 - 15,000 × גרם ו -4 מעלות צלזיוס. הערה: ממסים מופרדים לשלב אורגני נמוך יותר (ורוד, מכיל חלבונים), שלבי ביניים (לבנים, מכילים DNA) ושלב מימי עליון (ברור, מכיל RNA).
הערה: משלב זה על שימוש רק RNase ריאגנטים חופשיים וכלי פלסטיק! - הכן טרי RNase ללא צינורות 1.5 מיליליטר תגובה, תווית כראוי ולהוסיף כ -500 μl של 100% isopropanol RNase ללא כל (השתמש בערך באותו VOlume כשלב המימי בצינור הקודם).
- החזק את הצינור בזווית ולהעביר את השלב המימי (כ -500 μl) לצינורות שהוכנו באמצעות RNase עצות חינם. נא לא להפריע הביניים.
- לזרז את הרנ"א על ידי היפוך כמה פעמים ודוגרים במשך 10 דקות ב RT.
- צנטריפוגה הדגימות במשך 15 דקות ב13,000 - 15,000 × גרם ו 4 מעלות צלזיוס ולהסיר supernatant ידי pipetting או שאיפה (משאבת סילון מים ובקבוק האכלה). נא לא להפריע גלולה RNA השקופה הלבנה בתחתית.
- הוסף 1 מיליליטר של 70-80% אתנול RNase חינם (לא מערבולת) כדי לשטוף. הערה: RNA באתנול יכול להיות מאוחסן במשך כמה שבועות ב -20 מעלות צלזיוס.
- צנטריפוגה במשך 5 דקות בg 7,500 × ו -4 ° C וזורקים supernatant ידי pipetting או רצוי שאיפה.
- גלולה אוויר יבשה RNA במשך 15 - 30 דקות מתחת למכסת מנוע קטר. אל overdry, אחרת כדורים עלולים להיות קשים לredissolve.
- Resuspend גלולה ב 50 μl של DDH RNase חינם 2 O או חיץ אחסון RNA.
- למדוד את ריכוז RNA עם ספקטרופוטומטר microvolume UV-Vis או קובט קוורץ (ופוֹטוֹמֶטֶר הקונבנציונלי) ולהמשיך DNase אני עיכול.
- אני DNase עיכול של RNA כדי להסיר עקבות של ה- DNA
הערה: מאחר שה- DNA יכול לשמש כתבנית מזויפת בתגובת שעתוק לאחור (סיומת פריימר), יש להרחיקה מהמדגם. שיטות שונות להסרת DNA מפתרונות RNA זמינות אשר בדרך כלל מסתמכים על עיכול DNase. שיטה יעילה פשוטה אך יעילה ועלות להסרת DNA מפורטת להלן.- אמבט מים מחממים עד 37 מעלות צלזיוס.
- מערבבים את החומרים המפורטים בטבלה המס '1 בצינור 1.5 מיליליטר תגובה.
- דגירה את התערובת למשך שעה 1 על 37 מעלות צלזיוס באמבט מים, ולאחר מכן להמשיך ישירות למיצוי פנול / כלורופורם. הערה: איון חום שלי DNase אינו מומלץ, שכן הדבר עלול לבזות את RNA.
- חילוץ פנול / כלורופורם של RNA לאחר DNase אני עיכול
הערה: RNA חייב להיות מטוהר כדי להסיר נוקלאוטידים חופשיים, שברי DNA ורכיבי חיץ מעיכול DNase אני. חילוץ פנול / כלורופורם מאפשר התאוששות גבוהה וריכוז של מדגם RNA ולכן מפורט להלן. גם שיטות אחרות לטיהור RNA עשויות לשמש, אם הם עומדים בדרישות האלה.- לפצל את DNase 500 μl אני עיכול לערבב לתוך שתי 250 דגימות μl ב 2 מיליליטר צינורות תגובה.
- הוסף 1 נפח (250 μl) של P חומצי / C / פתרון (במים רוויים פנול, כלורופורם וisopentanol, יחס של 25: 24: 1, pH 4.5 - 5).
הערה: P / C / פתרון הוא מסרטנת, רעיל ומאכל. אנא קראו את גיליונות נתוני בטיחות חומרים ושימוש במנדף עם הגנה מתאימה! - מרץ מערבולת או מקום בפלטפורמת vortexing 1 - 3 דקות.
- צנטריפוגה למשך 30 דקות ב 13,000 - 15,000 × גרם ו -4 מעלות צלזיוס.
- אסוף את השלב העליון (המימי) ולהעביר לצינור טרי (250 μl).
- להוסיף 1/9 נפח (28 μl) של 3 M pH נתרן אצטט 5.2.
- להוסיף 2.5-3 כרכים של אתנול טהור (700 μl).
- מערבבים על ידי vortexing זמן קצר והמקום ב -80 מעלות צלזיוס למשך 30 דקות או ב -20 מעלות צלזיוס 2 - 3 שעות. במידת הצורך, לאחסן RNA O / N ב -20 מעלות צלזיוס.
- צנטריפוגה במשך 30-60 דקות ב13,000 - 15,000 × גרם ו -4 מעלות צלזיוס.
- הסר supernatant ידי pipetting או שאיפה.
- לשטוף גלולה ידי הוספת 1 מיליליטר של אתנול 70% על גלולה. האם לא מדגם מערבולת.
- מדגם צנטריפוגה במשך 5 דקות ב13,000 - 15,000 × גרם ו -4 מעלות צלזיוס.
- הסר supernatant ידי pipetting או שאיפה.
- גלולה האוויר יבשה מתחת למכסת מנוע קטר. אחסן את גלולה ב -20 CO ° / N במידת צורך.
- ממיסים את גלולה בDEPC 30 μl טופל H 2 O על ידי vortexing למשך 2 דקות ולהשתמש בפתרון זה כדי לפזר את PEllet של הצינור השני המתאים לדגימה (30 פתרון μl לכל זוג חילוץ אחד).
- למדוד את ריכוז RNA, ולוודא שהיא עולה על מיקרוגרם / μl 1 לשימוש בסיומת פריימר של mRNAs הביע בממוצע.
- במידת הצורך, לאחסן RNA ב -20 מעלות צלזיוס במשך כמה שבועות עד כמה חודשים.
תגובת 2. פריימר Extension
- עיצוב פריימר
הערה: בעת תכנון פריימרים לניסוי סיומת פריימר, לציית להנחיות כלליות של עיצוב פריימר PCR (ראה המדריך המלווה את רצף ג'ל האוטומטי במשך יותר מדור מידע ודיון במאמר זה).- באופן ספציפי, להבטיח שפריימרים (i) אינם מכילים ריצות של בסיסים, (ii) יש G או C בסוף '3, (iii) יש GC מאוזן: AT יחס, (iv) יש חישול בטמפרטורה של כ 55-60 מעלות צלזיוס ו( v) נ"ב לאגד לפחות 50, טובים יותר 100 נ"ב במורד הזרם של האזור של עניין לקבל תמונות ברורות.
- תגובת סיומת פריימר
הערה: תגובת סיומת פריימר (סינתזת cDNA) דורשת כמויות גבוהות של RNA תבנית. אם הכמויות של RNA המשמשות נבחרות לנמוך, האות עלולה להיות נמוכה מדי כדי לזהות! לכן אנו ממליצים לטיהור של RNA כמתואר לעיל.
הערה: שים לב: כלי ריאגנטים ופלסטיק חופשיים השימוש RNase !!!- מחממים את התרמו-Cycler לטמפרטורה של 95 מעלות צלזיוס ולבצע את כל שלבי דגירה נוספים בתרם-Cycler עבור קלות שימוש ושחזור.
- מערבבים את החומרים מטבלה 2 בצינור PCR עבור כל דגימת RNA.
- לפגל דגימות דקות 1 על 95 מעלות צלזיוס.
- מניחים את הצינורות על קרח וקור במשך 5 דקות כדי להכליא RNAs ופריימרים.
- להגדיר את מכשיר ה- PCR ל- 47 מעלות צלזיוס.
- בינתיים מכין את תערובת מאסטר השעתוק לאחור כמתואר בטבלה 3.
- הוסף 4 μl של תמהיל אב שעתוק לאחור לכל RNA הכלאהמדגם.
- דגירה הצינורות עבור השעה 1 ב 47 מעלות צלזיוס. הערה: הטמפרטורה האופטימלית לAMV RT היא 42 מעלות צלזיוס, טמפרטורה גבוהה יותר לעומת זאת לעזור להתגבר על מבנים משניים של מולקולות RNA.
- לעצור את התגובה על ידי חימום הדגימות עד 95 מעלות צלזיוס למשך 2 דקות.
הערה: Formamide הוא מאכל, רעיל ויכול להזיק לעובר. אנא קראו את גיליונות נתוני בטיחות חומרים, להתמודד עם טיפול וללבוש הגנה ראויה! - הוסף 6 μl של טעינת צבע פוראמיד (95% (v / v) פוראמיד deionized, 10 mM EDTA, 0.05% (w / v) bromophenol הכחול) והחנות לO / N לשבועיים ב -20 מעלות צלזיוס בחושך.
3. הכנת סולם הרצף
הערה: תגובת סולם הרצף דורשת גם כמויות מתונות של פלסמידים או כמויות גבוהות של הדנ"א הגנומי. במידת האפשר, השימוש בפלסמידים בתגובת הרצף מומלץ בשל הקלות של בידוד וגבוה sigעוצמה סופית. במקרים אחרים, אנו משתמשים באופן שיגרתי בשיטה שאומצה ממרמור 5,14 להכין הדנ"א הגנומי מא coli וס תאי aureus ללא צורך בשימוש בפנול. בעיקרון ניתן להשתמש בכל שיטה שמניבה סכומים ובטוהר של הדנ"א הגנומי גבוהים.
- בידוד הדנ"א הגנומי
- לגדול 10 מיליליטר של א ' coli או S. תאי aureus O / N ב LB, BM 5 או בינוני TSB.
- קציר תאים על ידי צנטריפוגה במשך 10 דקות בגובה של 4,600 × גרם בצינור בז 15 מיליליטר.
- גלולה Resuspended ב 2 P1 מיליליטר החיץ כפי שנמצא בכמה ערכות הכנת מיני (50 טריס-HCl pH 8.0 מ"מ, 10 מ"מ EDTA, 100 מיקרוגרם / מיליליטר RNase).
- תאי lyse 45 - 60 דקות עם 20-40 lysostaphin μl (0.5 מ"ג / מיליליטר, אחסון ב -20 ° C). הערה: E. קולי תאים האנזימטית מראש הטיפול יכול גם להיות מושמט או יזוזים משמשים.
- הוספת 100 μl של SDS-פתרון רווי (ב -45% אתנול) על השעיית וincubatדואר במשך 5 דקות על 37 מעלות צלזיוס.
- להוסיף 650 μl 5 M NaClO 4 והתאים בקצרה מערבולת.
הערה: כלורופורם הוא מסרטן פוטנציאלי. אנא קראו את גיליונות נתוני בטיחות חומרים ושימוש במנדף עם הגנה מתאימה !!! - הוסף 3 מיליליטר של כלורופורם / isopentanol (24: 1 יחס) לתערובת ולנער במשך לפחות 60 שניות. הערה: הנוזל צריך להפוך לתחליב לבן אחיד.
- מדגם צנטריפוגות במשך 10 דקות ב 4,600 × גרם וRT להפריד שלבים.
- להעביר בזהירות את השלב ברור העליון (המימי) לצינור טרי. אם הפתרון הוא עכור, לחזור על מיצוי כלורופורם / isopentanol. מדוד את עוצמת הקול של פתרון ה- DNA ולהכין צינור טרי עם 2 כרכים של אתנול (100%).
- לאט למזוג או פיפטה פתרון ה- DNA לתוך אתנול המכיל צינור. הערה: ה- DNA צריך לזרז סלילים שקופים, צפופים בתחתית או כאשר לחלוטין מיובש כאשכול לבן צף.
- אחזר את DNA באמצעות קרסים עשויים טפטפות פסטר זכוכית (איור 2) ולשטוף כל דגימה פעמיים על ידי טבילה לתוך צינור בודד של 1 מיליליטר של אתנול 70%.
- הנח את הווים זקופים במעמד ואוויר יבש גלולה במשך 60 דקות. במידת צורך, לאחסן את ה- DNA המיובש במשך כמה ימים על RT.
- לפזר DNA על ידי ניתוק ווי זכוכית DNA מכוסה והצבה בצינור 2.0 מיליליטר תגובה מכיל 100-500 μl DDH 2 O. התאם את עוצמת קול לריכוז ה- DNA של 1,000 - 1,500 ng / μl. לתגובת סידור אחד, להשתמש 10-18 מיקרוגרם של ה- DNA הגנומי.

איור 2. הדרכה על איך ליצור חכת DNA. החזק את קצה פיפטה פסטר זכוכית לתוך הלהבה של מבער בונזן. זה גורם לזכוכית כדי להמס לאחר מספר שניות, יצירת וו קטן בtהוא בסופו של. להסיר במהירות מהאש ומצנן דקות 1.
- בידוד פלסמיד
- הכן פלסמידים באמצעות ערכות הכנת מיני סטנדרטית ולפזר במאגר elution (10 מ"מ טריס-Cl, pH 8.5). בהתאם לגודל פלסמיד, להשתמש 100 - של פלסמיד 500 ng לסולם רצף אחד.
- תגובה סנגר רצף
הערה: מצא מתחת פרוטוקול פשוט שמשתמש בערכת רצף פריימר שכותרתו fluorescently עם 7-deaza-dGTP שפועל היטב לצורך הרחבות פריימר. עיין במדריך לערכת הרצף לקבלת מידע מפורט. שים לב שתגובת סידור חייבת להשתמש באותו צבע היסוד כתגובת סיומת פריימר כדי ליצור מוצרים באותו האורך.- מערבבים 12 μl של הדנ"א הגנומי (~ 10-15 מיקרוגרם) עם μl DMSO 1 ו 1 פריימר μl שכותרתו fluorescently (2 pmol / μl).
- לכל μl 1 של ארבע תערובות תגובת סידור (A, C, G או T), להוסיף 3 μl של ה- DNA / DMSO / תערובת פריימר.
- מניחים את הדגימות למכונת PCR, ולהפעיל את תכנית ה- PCR הבאה: 95 מעלות צלזיוס למשך 2 דקות; 35 מחזורים של 95 מעלות צלזיוס למשך 20 שניות, 54 מעלות צלזיוס למשך 20 שניות, 70 מעלות צלזיוס למשך 30 שניות; לשמור על 4 מעלות צלזיוס לנצח.
- לאחר הריצה, להסיר את הדגימות מהמכונה, להוסיף 6 μl של צבע טעינה ולאחסן על קרח (לטווח קצר) או ב -20 מעלות צלזיוס במשך כמה ימים עד שבועות.
4. התקנת ג'ל ומכשירי הפעלה
הערה: מידע מפורט על איך מנגנון ג'ל הרצף מורכב, ג'ל מוכן וניתן למצוא כמה ג'ל מנוהל בפרוטוקול היצרן.
- הכנות
- הכן 10x TBE כמצוין בטבלה 4.
- ביום של ריצת ג'ל להכין 1 ליטר של חיץ 1x TBE עם ultrapure DDH 2 O.
- הכן 10% (w / v) APS. הערה: ניתן לאחסן 200 aliquots μl ב -20 מעלות צלזיוס במשך כמה חודשים, אך הפעילות עלולה לגרום לירידה לאורך זמן <./ Li>
- הרכבה של תא ליהוק ג'ל
- הימנע מאבק ומוך בין לוחות הזכוכית. משטחי עבודה ולכן יש לנקות ביסודיות באמצעות מגבונים לחים.
- נקה זוג לוחות זכוכית 25 סנטימטר באמצעות מגבות נייר חד פעמיות ומים מזוקקים בשני הצדדים ולאחר מכן isopropanol לצד הפנימי של לוחות הזכוכית.
- הנח 0.25 מ"מ מפרידי על צלחת הזכוכית האחורית ולהפחית את צלחת זכוכית מחורצת על גבי (איור 3).
- הצמיד את פסי ג'ל לשני הצדדים של לוחות הזכוכית עם המגרעת וטייסי כניסת הרכבת יפנו כלפי מעלה ולהדק ידיות קלות.
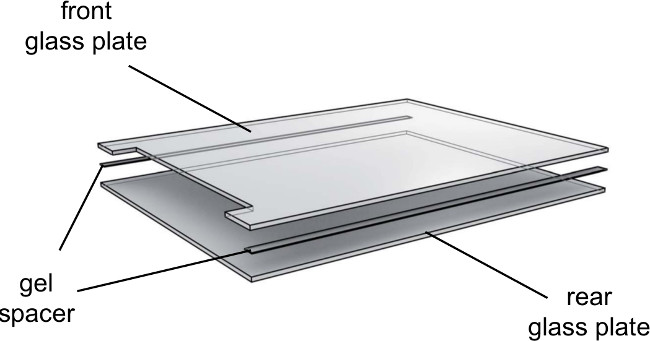
איור 3. התפוצץ נוכח לוחות זכוכית ג'ל אלקטרופורזה. יש להשתמש בצלחות זכוכית directionally. תשמור על עצמך כדי להתמודד עם הצד הפנימי של לוחות הזכוכית פנימה וחיצוניצד כלפי חוץ.
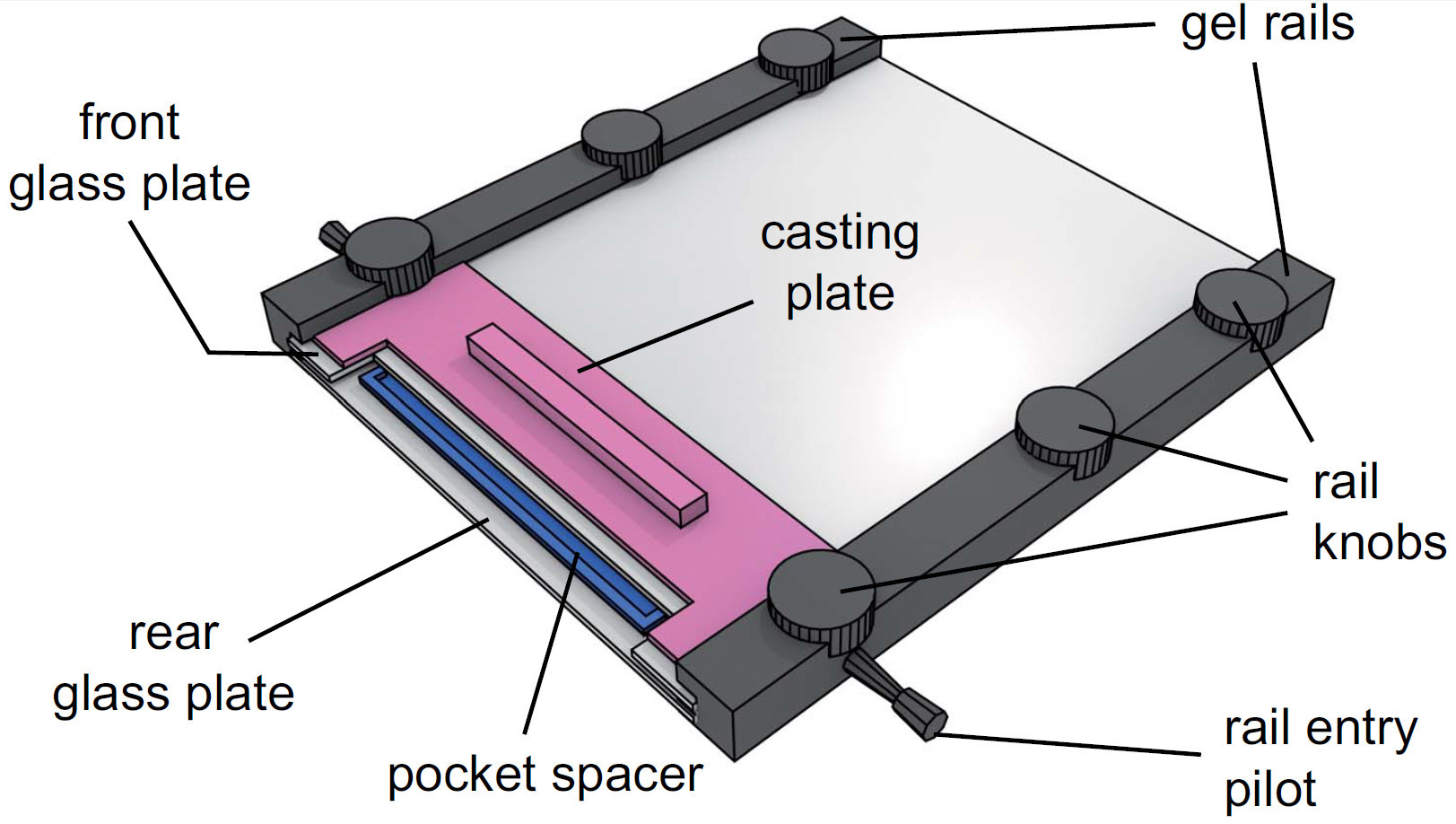
איור 4. מבט של מנגנון ג'ל התאסף. לאחר שהזריק פתרון ג'ל, spacer הכיס ממוקם בפתרון בין לוחות הזכוכית. צלחת הליהוק החליקה אז בין צלחת הזכוכית הקדמית ומסילות ג'ל ומאובטחת על ידי הידוק ידיות הרכבת.
- יציקת ג'ל
הערה: acrylamide פולימר שאינו רעיל למערכת עצבים היא! אנא קרא את גיליונות נתוני בטיחות חומרים ולהשתמש בהגנה מתאימה !!!- הוסף את התרכובות המופיעות בטבלה 5 לתוך כוס ומערבבים באמצעות בר ומערבב ובוחש מגנטי.
- מייד לאחר הוספת APS וTEMED, לקחת את פתרון ג'ל במזרק 50 מ"ל ולמקם את מסנן 0.45 ננומטר על הקצה.
- כך או להחזיק את הקצה העליון של צלחת הזכוכית ביד אחת או הנח את sandwich בליהוק ג'ל לעמוד ליצור מדרון בזווית 10 - 20 מעלות.
- לאט לאט לוותר פתרון ג'ל בין לוחות הזכוכית תוך כדי התנועה ברציפות קצה המזרק מצד אחד למשנהו ולהפסיק ברגע שפתרון ג'ל פוגש את הקצה התחתון.
- לעבור לצד או להסיר לחלוטין את כל בועות שנוצרו באמצעות וו בועה.
- חלק את spacer כיס ג'ל (0.25 מ"מ) בין לוחות הזכוכית בסוף מחורצים, לצלול לתוך תמיסת ג'ל ולתקן על ידי הצמדת צלחת הליהוק.
- ברגים עליון רכבת לְכַפתֵר קל (ראו תרשים 4 למכשירים מורכבים בשלמות).
- בואו סט ג'ל ל1 - 2 שעות.
- הסר את spacer כיס הצלחת והליהוק ולנקות את כיס משאריות מלח וג'ל.
- לשטוף עם DDH 2 O ומנגב את הפתרון עודף עם ניירות רקמה.
- פועל באופן חזותי את הג'ל
הערה: ג'לים הרצף ישירות נתונים לאלקטרופורזה בג'ל תרמי, תוךהקרינה מזוהה בו זמנית על ידי מיקרוסקופ לייזר. בניגוד לג'ל אלקטרופורזה הקונבנציונלית, שבו ג 'מנוהל ראשון ולאחר מכן מוכתמת ומדמיין, יחידת האיתור קבועה וסורקת את הלהקות בזמן אמת כפי שהם עוברים את הלייזר. להלן נוהל לתוכנת אוסף ImagIR נתונים על OS / 2 המתואר, אשר יכול להיות מאומץ לגרסות מאוחרות יותר. לקבלת מידע נוסף, ראה מדריך למשתמש.- חלק את מחזיק טנק חיץ לפסי ג'ל על לוחות הזכוכית הקדמיים ולהדק את הידיות.
- הנח ג'ל לתוך טנק ג'ל התחתון של תרמי ג'ל האוטומטי נגד צלחת החימום ולתקן על ידי הזזה טייס כניסת הרכבת לסוגר המנגנון.
- מלא חיץ TBE 1x לתאי חיץ ג'ל התחתונים ועליונים, לסגור את תא החיץ הנמוך ולחבר את תא החיץ העליון לכח באמצעות כבל החשמל.
- אם קיימים, לנקות את כיס ג'ל ממלח-שאריות על ידי pipetting שוב ושוב חיץ לכיס.
- סגור את מיכל חיץ התא העליון באמצעות מכסה החיץ העליון.
- סגור את דלת המכונה ולעבור על תרמי והמחשב ולהפעיל את תוכנת אוסף Base ImagIR נתונים.
- צור קובץ פרויקט חדש (File-> New ...), הזן שם קובץ פרויקט, לבחור את טווחי הלייזר המתאימים (700 או 800 ננומטר) ואשר בלחיצה על אישור.
- בחר רווח אפשרויות-> אוטומטי ... מתפריט התמונה בחלק העליון, לחץ על Auto להתחיל מדידת רווח אוטומטית וקיבלתי את הגדרות על ידי לחיצה על אישור.
- פוקוס הלייזר על ידי בחירת אפשרויות-> פוקוס ... מתפריט שליטת סורק, לחיצה על לחצן האוטומטי ולקבל את ההגדרות על ידי לחיצה על אישור.
- חזור על תהליך העלייה האוטומטית כדי להתאים לאזור ממוקד החדש.
- הגדרת שליטת הסורק בהתאם להגדרות הבאות: 2,000 V, 35 מילי-אמפר, 45 W, 45 ° C, מסנן סריקה: 3, מהירות סריקה: 3.
- Prerun את הג'ל הריק במשך 20 דקות (במתח בחר ולחצו על ).
- בינתיים, מחמם את סולם הרצף ומוצרי סיומת פריימר במכונה PCR במשך 2 דקות עד 90 מעלות צלזיוס, ולאחר מכן לצנן על קרח.
- עצור את אלקטרופורזה, לפתוח את רצף ג'ל האוטומטי ולהסיר את מיכל חיץ העפעף העליון.
- הכנס את השיניים מסרק הכריש בין לוחות הזכוכית ומעט לנקב את הג'ל עם שיני הכריש (ראה תרשים 5).

איור 5. תצוגת תקריב של ג'ל עם מסרק שן כריש. לדוגמא (סגולה) מוחלת בין שיני הכריש.
- פיפטה או 1 - 2 μl של מוצרי המשך האו תגובות סולם רצף פריימר לכל כיס ג'ל (שהוקם על ידי שיני הכריש).
- אם יש צורך לא כל הכיסים, למלא את הכיסים ריקים עם צבע טעינה כדי למנוע בעקבית ריצת התנהגות.
- סגור את מיכל החיץ ודלת של רצף ג'ל.
- התחל אלקטרופורזה ולהדליק לייזר (בחר מתח ובליזר ובעיתונות ).
- להפסיק אלקטרופורזה פעם אחת באזור של העניין עבר את הלייזר.
תוצאות
כפי שמתואר באיור 6, תגובת סיומת פריימר ניתן להשתמש כדי לקבוע את נקודות התחלת תעתיק של תמלילים של עניין ויכולה לעזור להסיק אזורי אמרגן (מזוהה בדרך כלל על ידי -10 ו-35 אלמנטים). בר cDNA העליון (הארוך ביותר) מייצג את הקצה 5 'של ה- mRNA, ולכן יכול להיות ממופה בקלות בהשוואה...
Discussion
סיומת פריימר ניאון היא שיטה פשוטה ומהירה לקביעת קצוות "5 של RNA, או לTSP- או הזדהות עיבוד RNA משנית. בשל השימוש בצבעי יסוד ניאון, ניתן להגדיר את התגובות ולרוץ ללא אמצעי אבטחה נוסף (שלא כמו במקרה של פריימרים שכותרתו רדיואקטיבית). כדגימות זוהו על ידי הקרינה, הם יכולים להיות ?...
Disclosures
The authors have nothing to disclose that would present a conflict of interest.
Acknowledgements
We thank Anne Wochele for her assistance in the laboratory and Vera Augsburger for help with the automated gel sequencer. We thank the Deutsche Forschungsgemeinschaft for funding by grants BE4038/2 and BE4038/5 within the “priority programmes” SPP1316 and SPP1617.
Materials
| Name | Company | Catalog Number | Comments |
| AMV Reverse Transcriptase (20-25 U/µl) | NEB / Roche | NEB: M0277-T / Roche: 10109118001 | |
| DNase I (RNase free) | Ambion (life technologies) | AM2222 | |
| FastPrep-24 Instrument | MPBio | 116004500 | |
| Fluorescently labeled primers | Biomers | n/a | 5’ DY-681 modification of “ordinary” DNA oligonucleotides. Compatible dyes such as the LICOR IRDye 700/800 are also available from other suppliers such as IDTdna. |
| Li-Cor 4200 Sequencer incl. ImagIR Data collection software | Li-Cor | Product discontinued | |
| NanoDrop 2000 | Thermo Scientific | ||
| Nuclease free water | Ambion (life technologies) | AM9915G | |
| Plasmid mini preparation kit | QIAGEN | 12125 | |
| RapidGel-XL-40% Concentrate | USB | US75863 | |
| RNA STORAGE BUFFER | Ambion (life technologies) | AM7000 | |
| Roti-Aqua-P/C/I | Carl Roth | X985.3 | Alternative: “Acid-Phenol:Chloroform, pH 4.5 (with IAA, 125:24:1)” from Ambion (AM9720) |
| SUPERase•In RNase Inhibitor | Ambion (life technologies) | AM2696 | |
| Thermo Sequenase fluorescently labelled primer cycle sequencing kit with 7-deaza-dGTP | GE Healthcare | RPN2538 | |
| TRIzol reagent | life technologies | 15596-026 | |
| Zirconia/Silica Beads 0.1 mm | BioSpec | 11079101z |
References
- Simpson, C. G., Brown, J. W. Primer extension assay. Methods Mol. Biol. 49, 249-256 (1995).
- Sanger, F., Nicklen, S., Coulson, A. R. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74, 5463-5467 (1977).
- Fekete, R. A., Miller, M. J., Chattoraj, D. K. Fluorescently labeled oligonucleotide extension: a rapid and quantitative protocol for primer extension. Biotechniques. 35, 90-94 (2003).
- Schuster, C. F., et al. Characterization of a mazEF Toxin-Antitoxin Homologue from Staphylococcus equorum. J. Bacteriol. 195, 115-125 (2013).
- Nolle, N., Schuster, C. F., Bertram, R. Two paralogous yefM-yoeB loci from Staphylococcus equorum encode functional toxin-antitoxin systems. Microbiology. 159, 1575-1585 (2013).
- Yamaguchi, Y., Park, J. H., Inouye, M. Toxin-antitoxin systems in bacteria and archaea. Annu. Rev. Genet. 45, 61-79 (2011).
- Schuster, C. F., Bertram, R. Toxin-antitoxin systems are ubiquitous and versatile modulators of prokaryotic cell fate. FEMS Microbiol. Lett. 340, 73-85 (2013).
- Goeders, N., Van Melderen, L. Toxin-antitoxin systems as multilevel interaction systems. Toxins. 6, 304-324 (2014).
- Yamaguchi, Y., Inouye, M. mRNA interferases, sequence-specific endoribonucleases from the toxin-antitoxin systems. Progress in molecular biology and translational science. 85, 467-500 (2009).
- Park, J. H., Yamaguchi, Y., Inouye, M. Bacillus subtilis MazF-bs (EndoA) is a UACAU-specific mRNA interferase. FEBS Lett. 585, 2526-2532 (2011).
- Zhu, L., et al. et al.Staphylococcus aureus MazF specifically cleaves a pentad sequence, UACAU, which is unusually abundant in the mRNA for pathogenic adhesive factor SraP. J. Bacteriol. 191, 3248-3255 (2009).
- Zhu, L., et al. The mRNA interferases, MazF-mt3 and MazF-mt7 from Mycobacterium tuberculosis target unique pentad sequences in single-stranded RNA. Mol. Microbiol. 69, 559-569 (2008).
- Fu, Z., Donegan, N. P., Memmi, G., Cheung, A. L. Characterization of MazFSa, an endoribonuclease from Staphylococcus aureus. J. Bacteriol. 189, 8871-8879 (2007).
- Marmur, J. A procedure for the isolation of deoxyribonucleic acid from micro-organisms. J. Mol. Biol. 3, 208-218 (1961).
- Emory, S. A., Belasco, J. G. The ompA 5' untranslated RNA segment functions in Escherichia coli as a growth-rate-regulated mRNA stabilizer whose activity is unrelated to translational efficiency. J. Bacteriol. 172, 4472-4481 (1990).
- Cole, S. T., Bremer, E., Hindennach, I., Henning, U. Characterisation of the promoters for the ompA gene which encodes a major outer membrane protein of Escherichia coli. Mol. Gen. Genet. 188, 472-479 (1982).
- Schleifer, K. H., Kilpper-Bälz, R., Devriese, L. Staphylococcus arlettae sp. nov., S. equorum sp. nov. and S. kloosii sp. nov.: Three New Coagulase-Negative, Novobiocin-Resistant Species from Animals. Syst. Appl. Microbiol. 5, 501-509 .
- Yu, H., Goodman, M. F. Comparison of HIV-1 and avian myeloblastosis virus reverse transcriptase fidelity on RNA and DNA templates. J. Biol. Chem. 267, 10888-10896 (1992).
- Ying, B. W., Fourmy, D., Yoshizawa, S. Substitution of the use of radioactivity by fluorescence for biochemical studies of RNA. RNA. 13, 2042-2050 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved