A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
שיטה פשוטה DNA הקרינה
In This Article
Summary
Here, we present a simple method for performing fluorescence DNA in situ hybridization (DNA ISH) to visualize repetitive heterochromatic sequences on slide-mounted chromosomes. The method requires minimal reagents and it is versatile for use with short or long probes, different tissues, and detection with fluorescence or non-fluorescence-based signals.
Abstract
DNA הכלאה באתר (DNA ISH) הוא שיטה נפוצות לרצפי מיפוי לאזורים של כרומוזום מסוימים. גישה זו היא יעילה במיוחד במיפוי רצפים חוזרים מאוד לאזורי heterochromatic, שבו גישות חישוביות להתמודד עם אתגרי אוסרני. כאן אנו מתארים פרוטוקול יעיל עבור DNA ISH שעוקף שוטף פוראמיד שהם צעדים סטנדרטיים בפרוטוקולי DNA ISH אחרים. הפרוטוקול שלנו מותאם להכלאה עם בדיקות יחידים קצרות גדיל DNA שנושאים צבעי ניאון, אשר למעשה לסמן רצפי DNA חוזר על עצמו באזורי כרומוזומים heterochromatic על פני מספר סוגים שונים של רקמות חרקים של. עם זאת, יישומים ניתן להאריך להשתמש עם בדיקות ויזואליזציה של רצפי DNA עותק יחיד (לא חוזר על עצמו) גדולים יותר. אנו מדגימים שיטה זו על ידי מיפוי כמה רצפים חוזרים שונים לכרומוזומים מעוכים מדרוזופילה melanogaster תאים עצביים וNasoniaspermatocytes vitripennis. אנחנו מראים דפוסי הכלאה של שני חלליות קטנות, מסונתזים באופן מסחרי ולבדיקה גדולה יותר להשוואה. הליך זה משתמש ציוד מעבדה פשוט וריאגנטים, והוא אידיאלי לחוקרים שיש להם ניסיון מועט עם ביצוע DNA ISH.
Introduction
DNA הכלאה באתר (DNA ISH) הוא שיטה נפוצות לרצפי מיפוי לאזורים של כרומוזום מסוימים. יכולות להיות שנוצרו בדיקות לאזורי עותק יחידים בתוך euchromatin באמצעות קומץ של גישות, כולל nick-תרגום או בסופו תיוג של מוצרי DNA ארוכים 1,2 ושילוב של deoxygenin (DIG) נוקלאוטידים -attached וההכרה שלהם באמצעות מגוון רחב של נוגדנים-מצומדות קבוצה 1-3. ויזואליזציה של רצפי euchromatic בעותק מספר מעטים או בודד מחייבת השימוש בשני בדיקות יחידים, גדולות עם פעילות ספציפית גבוהה או קוקטייל של בדיקות מרובות, קטנות יותר, כי באופן קולקטיבי לשפר אות.
בניגוד לכך, חוזר על עצמו מאוד רצפים נמצאו בheterochromatin, כגון DNAs לווין, הם מטרות קלים יותר לDNA ISH מכיוון שהם קיימים בדרך כלל כעשרות אלפי חזרות התקבצו באזורי כרומוזום יחידים הידועים כלוקים. אלמנטים ניידים גם יכולים להיותנמצא במספרים גבוהים בעותק לוקוסי כרומוזומליות שונים 2. במקרים אלה, בדיקות יחידים עם פעילות ספציפית נמוכה יכולות למעשה תווית רצפי heterochromatic בשל ההכלאה שלהם באתרים מרובים. בדיקות רצפים חוזרים יכולות להיות מסונתזת oligonucleotides הקצר מסחרי כ( 30-50 נ"ב) וכימיות מצומדות עם כל קבוצות ניאון שונות מרובות. מיפוי רצפים חוזרים בתוך heterochromatin על ידי שימוש בטכנולוגיות הגנום רצף קשה בשל אתגרים נתקלו בפיגומי בניין בלוקי לווין חוזר על עצמו מאוד 4-6,7. נכון לעכשיו, איש עומד כדרך היעילה ביותר למיפוי רצפים אלה ברמה תת-כרומוזום. אסטרטגיה זו היא חשובה למיפוי מספרים גדולים של רצפים חוזרים הנחשפים על ידי מחקרי הגנום ורצף transcriptome מתמשכים.
היעילות וקלות רצפים חוזרים מיפוי על כרומוזומים רכוב שקופיות תהיה GREמשופר atly על ידי פרוטוקול פשוט לDNA ISH. לדוגמא, פרוטוקולים קיימים לDNA ISH כרוכים שטיפות מרובות של רקמות הכלאה בפתרון פוראמיד 2,8, ובכך להוסיף משמעותי את הזמן הנדרש לרצפי מיפוי וגם לייצר כמויות גדולות של פסולת כימית למגיב יקר זה. כאן אנו מתארים שיטת DNA ISH מתוקנת שעוקפת את הצורך בשטיפת פוראמיד ומשתמש בציוד מעבדה בסיסי וחומרים כימיים. שיטה זו תוכננה במקור למיפוי המהיר של רצפי DNA חוזר על עצמו מאוד באזורי heterochromatic של neuroblasts זחל תסיסנית באמצעות oligos מסונתז מסחרי המצומדות עם צבעי הקרינה. עם זאת, שיטה זו פועלת גם למיפוי רצפים חוזרים באמצעות בדיקות גדולות יותר מסונתזים באמצעים אחרים 9,10 ועל פני סוגי רקמות וכרומוזום שונים מרובים. בנוסף, שיטה זו יכולה לשמש כדי למפות רצפי euchromatic באמצעות יותר או multiple, בדיקות קצרות בתוך רצף euchromatic של עניין.
Protocol
1. רקמות Dissection וקיבוע (60 דקות)
- למוח של דרוזופילה, מקום זחלי instar 3 rd בירידה של 1x PBS (נאגר מלוח פוספט). בחר זחלי instar 3 rd גדולים שבאופן פעיל בזחילה מבקבוקונים או בקבוקים שאינם צפופים.
- השתמש זוג פינצטה ultrafine אחד לאחוז ווי הפה וזוג פינצטה אחרת כדי לתפוס 2/3 לאורך הגוף (איור 1 א ', ב'). משוך בעדינות על הפה הווים לחשוף את המוח, גרעיני הגחון, בלוטות רוק וחלק של מערכת עיכול הזחל. השתמש בפינצטה כדי להפריד את גרעיני המוח וגחון (איור 1 א ', ב') מהרקמות האחרות והמקום בטיפה של 1x PBT (נאגר מלוח פוספט עם Tween) על צלחת פטרי פלסטיק.
- ולניתוחי אשך Nasonia, לבחור גלמי 3 יום בן זכר (גופים צהובים עם עיניים אדומות). יש לי זכר Nasonia אורכי כרית כנף קטנים יחסית לfemales בשלב גלמים (איור 1 ג).
- החזק את הגולם בחלק העליון של הבטן ליד אזור בית החזה עם זוג אחד פינצטה, ובאמצעות פינצטה הזוג האחר, לתפוס את הקצה קצהו המרוחק של הבטן ולשלוף את האשכים המדמיע בצורת הטיפה (הם יהיו מוקפים בשומן גוף שיכול להיות מזועזע משם בעדינות; 1D איור). לנתק את כל חלקי גוף חיצוניים מהאשכים, מה שימנעו מְעִיכָה הנכונה, ומקום בטיפה של 1x PBT על צלחת פטרי פלסטיק.
- עבור כל שקופית, לנתח כולל של ארבעה או חמש דגימות רקמה (כלומר., המוח של זחל תסיסנית או אשכים Nasonia).
הערה: יותר מחמש דגימות תוביל לגרעינים וכרומוזומים צפופים. - לחלופין, כדי להשיג כמה הפרדת chromatids אחות באזורי euchromatic של כרומוזומים mitotic, טיפול ברקמה עם פתרון hypotonic: להעביר את המוח מ1x PBT לירידה של 0.5% נתרן ציטרט למשך 5-10 (לא יותר מ 10) דקות.
הערה: קולכיצין (מעכב mitotic) יכולה להיות מועילה עבור הגדלת מספר דמויות mitotic בmetaphase 11. עם זאת, השימוש בו הוא מיותר אם משמשים זחלים בריאים מאוד גדולים, ואפילו רצויים משום שהיא עלולה להשפיע לרעה על החלטת כרומוזום, הפצה ומורפולוגיה. - מקום טיפה (~ 20 μl) של פתרון מקבע (paraformaldehyde 2.5% ב- 45% חומצה אצטית) על פני השטח של להחליק את המכסה שטופל Sigmacote נקי.
הערה: הפוך פתרון מקבע טרי עבור כל יום של שימוש. פתרונות מקבע החל paraformaldehyde 1.8-3.7% ב- 45% חומצה אצטית יניבו את התוצאות הטובות ביותר לדגים. שימוש ברקמות אחרות מאשר מוח או אשכים, או התאמת פרוטוקול זה לאימונו-FISH עשויה לדרוש להתנסות עם fixatives שונה (לרשימה של fixatives שונה, לראות 12). - להעביר בזהירות כל דגימת רקמה מהמאגר לנתח (1x PBT) לתוך אגל מקבע עם פינצטה ultrafine, minimiזיע העברת לנתח חיץ לפתרון מקבע. מקם את דגימות הרקמה, כך שהם מחולקים באופן שווה זה מזה בטיפה מקבע. דגירה הרקמות במקבעות במשך 4 דקות בטמפרטורת חדר.
- מקום פני שקופית poly-ליזין מצופה בזהירות כלפי מטה על תלוש הרקמה וכיסוי. אל תלחץ כלפי מטה, בשלב זה, אך במקום לאפשר לשני לפנות באורח קל, כך שלהחליק את מכסה מקלות על החלק התחתון של השקופית. הפוך את השקופיות כך שלהחליק את המכסה הוא על העליונה.
- כריך שקופיות / רקמה / כיסוי להחליק בתוך פיסת נייר סינון מקופלת. על משטח יציב, באמצעות האגודל, לחץ בחוזקה מאוד היישר בעמדה ישירות מעל להחליק את המכסה. להיות זהיר, כדי למנוע החלקה לרוחב של להחליק את המכסה (זה יגרום מריחה של הרקמה).
- להטביע את השקופיות / רקמה / כיסוי להחליק לתוך חנקן נוזלי (ראה מנגנון באיור 1E, F), ולתת לעמוד עד החנקן מפסיק רותח (יותר הוא f ine). הסר את השקופית ומייד להצמיד את הכיסוי להחליק בסכין גילוח טרי ידי מצליף פינה להחליק את המכסה בכיוון מעלה (להימנע מגרד את הרקמה קבועה עם סכין הגילוח).
- טרום קירור השקופיות / רקמה / כיסוי להחליק על גוש קרח יבש, הכיסוי לפשל, במשך 1-2 דקות לפני הצללה בחנקן נוזלי יסייע למנוע את השקופיות מהפיצוח.
- מייד למקם את השקופית עם רקמה לתוך צנצנת Coplin מלא אתנול 100% בטמפרטורת חדר ולתת לעמוד במשך 5 דקות לפחות (הפעם יכול להיות ארוך יותר במידת הצורך).
יכולה לשמש גם אתנול 100% קר: הערה. - הסר את השקופית עם רקמה, פתיל משם אתנול עודף עם Kimwipe (בלי לגעת ברקמה קבועה), ולהשאיר את השקופיות לייבוש באוויר במשך שעה 1.
- להמשיך ישירות לשלב 2 או לשמור שקופיות יבשות באוויר לחות נמוך או בתא התייבשות במשך שבועות לחודשים לפני הביצוע אפילו הכלאה.
= "Jove_content" fo: לשמור-together.within-page = "תמיד"> 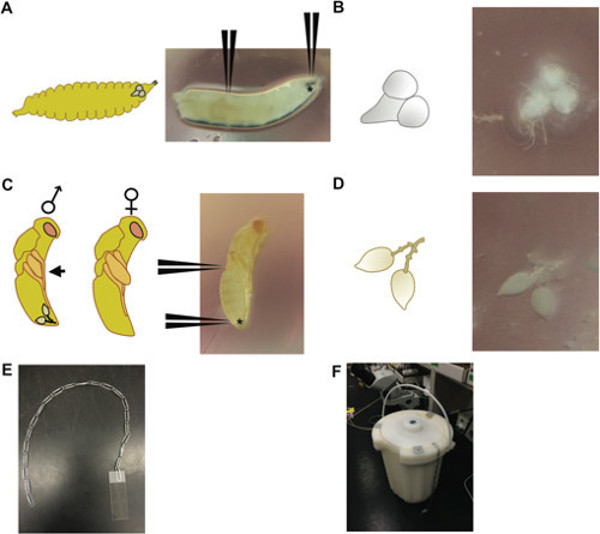
איור 1: זחל תסיסנית instar 3 rd (מימין) (א), עם עמדות שצוינו לשם כדי לתפוס את הפה הווים (מסומן ב- *) ו2/3 של הדרך למטה הזחלים לנתח את המוח; (משמאל) סכמטי של זחל באותו שלב התפתחותי, המתאר את המיקום היחסי של המוח בתוך ראש הזחל. (ב) במוח וגרעיני הגחון גזור מזחל instar 3 rd דרוזופילה (מימין) וסכמטי של זה רקמה (משמאל). גולם Nasonia (C) 3-בן יומו בשלב עין הגוף-אדום הצהוב. (ד) זוג אשכים גזורים מגולם 3 ימים ישן Nasonia זכר (מימין) עם עמדות המציין היכן לתפוס את הגולם באחורי של הבטן (מסומן ב- *) ובאמצע על הגוף; (משמאל) זכר מתאר סכמטי ונקבה היהגלמי p; גלמי זכר ניתן להבחין על ידי כנפיים שלא להאריך עבר פרופיל saggital (חץ שחור), בניגוד לנשים, שיש להם כנפיים שמרחיבות בעבר הפרופיל; המיקום היחסי של זוג האשכים מוצג בגולם הזכר. (E) המנגנון-מחרוזת של מהדקים-ושיטה (F) משמשים כדי לטבול שקופיות בכלי של חנקן נוזלי. מחרוזות מהדק מרובות יכולות לשמש לטבילה בו זמני של מספר שקופיות.
2. באתרו הכלאה (30 דקות ביום 1; 1 hr-2.5 שעות לבדיקות ארוכות-ביום 2)
- הוסף 1 μl (100 ng) של כל בדיקה עד 20 μl של חיץ הכלאת 1.1x. בדיקה Pipet / מאגר הכלאה על פני השטח של הרקמה קבועה (להימנע ממגע ברקמות).
- בזהירות במקום להחליק את המכסה ישירות על גבי חיץ בדיקה / הכלאה, ולוודא כי להחליק את המכסה מרוכז ישירות על הרקמה. החיץ צריך להעביר הדואר קצה חיצוני של להחליק את המכסה, לא משאיר בועות אוויר.
הערה: בועות קטנות שלא לפנות אל רקמות אינם מהווה כל בעיה להליך. הסר בועות גדולות על ידי ההרמה בזהירות פינה להחליק את המכסה והזהירות הפילה אותו חזרה לשקופית. - מניחים את תלוש שקופיות / רקמה / כיסוי על פני השטח של בלוק שחומם מראש עד 95 ° C (כיסוי לפשל). לכסות בפיסת נייר אלומיניום גדולה כדי למנוע חשיפה לאור. בואו השקופיות לדגור על 95 מעלות צלזיוס למשך 5 דקות.
הערה: גוש חום טיפוסי עם חורים לצינורות ניתן התהפכה לספק משטח שטוח שעליו במקום להחליק / רקמה / להחליק לכסות. - הסר את השקופית, לתת לה להתקרר מעט עד שהוא חם למגע. לעטוף בזהירות חתיכת Parafilm המתוח סביב להחליק את המכסה לאטום את הנוזל מתחתיו.
- מניחים את השקף האטום בתוך תא לחות ולמקם את החדר לחממה שחומם מראש ל 30 מעלות צלזיוס. לדגור על 30 מעלות צלזיוס למשך 4 שעות ללילה.
- ליצור תא לחות מתיבת קצה ריק או קופסא פלסטיק עפעפיים עם Kimwipes הלח או במגבות נייר ממוקם בתחתית.
הערה: בדיקות אוליגו DNA גדילים יחידה תוכננה להיות 28-33 בסיסים כדי להשיג טמפרטורת התכה תיאורטית (T מ ') של 45-47 ° C. טווחים מ 'אורך ו- T אלה משקפים את העובדה שרצפים חוזרים רבים שיש לנו למדנו הם AT-עשיר, ולכן יש להם תוכן GC נמוך מאוד. סביר להניח שיש לי בדיקות ארוכות יותר ערכי T מ 'גבוהים יותר; זה עלול לגרום להכלאת רקע גבוהה יותר בטמפרטורת הכלאה סטנדרטית של 30 מעלות צלזיוס. לפיכך, ייתכן שיידרשו לפתרון בעיות מסוימים עם טמפרטורות הכלאה כדי להשיג את התוצאות הטובות ביותר. כדי למצוא את טמפרטורת ההכלאה הטובה ביותר, העלייה (או ירידה) הטמפרטורה של 5 מעלות צלזיוס, באופן הדרגתי.
- ליצור תא לחות מתיבת קצה ריק או קופסא פלסטיק עפעפיים עם Kimwipes הלח או במגבות נייר ממוקם בתחתית.
- מוציא בזהירות את Parafilm מהשקופיות ולאחר מכן הסר את תלוש לכסות בזהירות על ידי הרמת פינה אחת באיטיות. ווהsh השקופיות שלוש פעמים במשך 15 דקות כל אחד לשטוף במאגר SSC 0.1x. מכסה את צנצנת Coplin עם רדיד אלומיניום במהלך שוטף כדי למזער את החשיפה לאור.
- אם לא באמצעות בדיקה biotinylated ארוכה, המשך לשלב 2.8.
- אם באמצעות בדיקה ארוכה biotinylated, לייבש סביב הרקמה עם Kimwipe האזור, נזהר שלא לגעת ברקמה עצמה. מקום חסימת פתרון על הרקמה 100 μl ולכסות בעדינות עם coverslip, לטפל כדי למנוע בועות השמנה. עטוף את השקופיות על coverslip עם Parafilm והמקום על 37 מעלות צלזיוס למשך 30 דקות.
- מוציא בזהירות את coverslip ולמחוק סביב הרקמה עם Kimwipe. Pipet של rhodamine-avidin מדולל 1 100 μl: טיפול 1,000 בSBT על הרקמות ולכסות בעדינות עם coverslip, לוקח כדי למנוע השמנה בועות. עטוף את השקופיות על coverslip עם Parafilm והמקום על 37 מעלות צלזיוס למשך 30 דקות.
- מוציא בזהירות את coverslip ולשטוף את השקופית 3 פעמים במשך 5 דקות כל אחד ב4x SSCT ולאחר מכן3 פעמים במשך 5 דקות כל אחד בSSC 0.1x.
הערה: ניתן לכבס שקופיות לתקופות זמן ארוכות יותר.
- הסר את השקופית ולמחוק סביב הרקמה עם Kimwipe יבש כדי להסיר עודפי חיץ (להימנע ממגע ברקמות). הנח את צד רקמת שקופית במקום חשוך למשך 10-15 דקות או עד שהלחות מתפוגגת לחלוטין.
- 11 μl Pipet של הרכבה בינונית Vectashield (עם 4 ', 6-diamidino-2-phenylindole-DAPI) על גבי הרקמה. בזהירות במקום להחליק נקי כיסוי (שלא טופל בSigmacote) ישירות מעל המרכז של המדיום ורקמת הרכבה. ההרכבה בינונית צריכים להעביר לאט החוצה לעבר הקצוות של להחליק את המכסה.
הערה: אם ההרכבה בינונית לא מצליח להגיע לקצה להחליק את המכסה מכל הצדדים, ואז 1-2 μl נוסף של הרכבה בינונית יכול להיות מיושם על עמדה אחת בקצה להחליק את המכסה למלא את הנפח הדרוש. במקרה זה, כדי להיות בטוח כדי למחות כל מדיום עודף ממשטח השקופיות לפניאיטום. - לאטום את הקצוות של להחליק לכסות עם לק. הימנע צובע את הציפורניים על דגימת הרקמה.
- מניחים את השקף זקוף במקום חשוך ולתת לק היבש עד קשה (בדרך כלל 30 דקות או יותר) לחלוטין. בשלב זה, תמונת הרקמה או בחנות ב -20 ° C למשך שבוע להדמיה מאוחר יותר של עד 1.
/ מתכוני פתרון חיץ
10x PBS
- 80 גרם NaCl
- 2.0 גרם KCl
- 14.4 גרם Na 2 4 HPO
- KH 2.4 גרם 2 PO 4
- pH 7.4, H 2 O L 1
1x PBT
- 5 מיליליטר 10x PBS
- 45 מיליליטר H 2 O
- 0.1% Tween 20
20x SSC
- 175.3 g NaCl
- 88.2 g Na ציטראט
- בשנת 800 מיליליטר H 2 O
- pH 7, H 2 O L 1
4x SSCT
- 200 מיליליטר 20x SSC
- 799 מיליליטר H 2 O
- 0.1% Tween 20
0.1x SSC
- 5 מיליליטר 20x SSC
- 995 מיליליטר H 2 O
תערובת הכלאה (20 μl; שונתה מ -11)
- 10 μl פוראמיד
- 4 μl 50% סולפט dextran
- 2 μl 20x SSC
- 4 μl H 2 O
SBT 8 (10 מיליליטר)
- 2 מיליליטר 20x SSC
- 0.01 g בסרום שור אלבומין (BSA)
- 10 μl Tween 20
- 7.9 מיליליטר H 2 O
פתרון חסימה 8 (10 מיליליטר)
- BSA 0.3 g
- 10 μl Tween 20
- 2 מיליליטר 20x SSC
- 8 מיליליטר H 2 O
פתרון מקבע עם paraformaldehyde (1 מיליליטר)
- 393.75 H μl 2 O (ראשון להוסיף מים)
- 450 חומצה אצטית קרחונית μl
- paraformaldehyde 156.25 μl 16%
תוצאות
כדי להדגים שיטה זו, אנו הכלאה סט של oligos המסונתזת מסחרי הקטנה ששונו כימיים עם conjugates ניאון (איור 2) ובדיקה ארוכה יותר biotinylated (שנעשתה באמצעות תרגום ניק של מוצר ה- PCR; איור 2), לכרומוזומים מכמה רקמות שונות סוגים (ראה טבלה 1). רצפי היעד כללו חזרות לווי...
Discussion
DNA ISH משמש לעתים קרובות כדי למפות רצפים ספציפיים לכרומוזומים. שתארנו שיטה פשוטה לDNA ISH מותאם לגבוה עותק מספר, רצפי heterochromatic. במקום להשתמש בשטיפות בתמיסת פוראמיד, אשר היא דרישה בפרוטוקולי ISH DNA קיימים אחרים, אנו שמים שקופיות רכוב רקמה ישירות על בלוק מחומם מראש ללפגל DNA. שי?...
Disclosures
The authors declare that they have no competing financial or any other conflict of interest.
Acknowledgements
We thank Zhaohua Irene Tang in the W. M. Keck Science Department for the use of her epifluorescence microscope and the Werren lab for donating Nasonia for dissections. This work was supported in part by an NIH-NRSA fellowship (5F32GM105317-02) to AML.
Materials
| Name | Company | Catalog Number | Comments |
| Poly-L-lysine coated slides (regular slides also can be used) | Sigma Aldrich | ||
| Ultrafine tweezers (5 gauge) | Dumont | ||
| 22 x 22 mm cover slips | Fisher | Sigmacote-treated by immersion for 15 sec, blotting dry, and wiping away all traces of Sigmacote so that cover slip is clear | |
| Sigmacote | Sigma | ||
| Filter paper | 75 - 150 mm | ||
| Paraffin wax paper | |||
| Heat block with thermometer | |||
| Dry incubator | |||
| Razor blades | |||
| Humidity chamber | empty pipette tip box or Tupperware, lined with moistened paper towels or Kimwipes | ||
| Coplin jars | with slide grooves | ||
| Aluminum foil | |||
| Pasteur pipettes | |||
| 1.5 ml microfuge tubes | |||
| Nail polish | clear or colored | ||
| P20 micropipette and plastic tips | |||
| Paperclips | 20 - 25 standard metal paperclips linked to form a chain | ||
| Reagents | |||
| 16% EM grade paraformaldehyde | Electron Microscopy Reagents | ||
| Acetic acid | Sigma | ||
| Liquid nitrogen | |||
| 100% Ethanol, chemical grade | |||
| Commercially synthesized, fluorescently labeled oligos | |||
| Long biotinylated probe | Invitrogen; Alternative steps 2.7.1-2.7.3 | e.g., nick translated and biotinylated with BioNick from Invitrogen | |
| Rhodamine-Avidin | Roche; Alternative steps 2.7.1-2.7.3 | for detection of long biotinylated probe | |
| Hybridization buffer | Recipe above | ||
| 4x SSCT | Recipe above | saline-sodium citrate + Tween | |
| 0.1x SSC | Recipe above | saline-sodium citrate | |
| Blocking solution | Recipe above | ||
| SBT | Recipe above | SSC, bovine serum albumin, Tween | |
| 1x PBT | Recipe above | phosphate-buffered saline + Tween | |
| 1x PBS | phosphate-buffered saline | ||
| Hypotonic solution | 0.5% sodium citrate in H2O | ||
| Formamide | Sigma Aldrich | ||
| Vectashield mounting medium with DAPI | Vector laboratories | ||
References
- Blattes, R., Kas, E. Fluorescent in situ hybridization (FISH) on diploid nuclei and mitotic chromosomes from Drosophila melanogaster larval tissues. Cold Spring Harbor Protocols. 2009 (9), (2009).
- Dimitri, P. Fluorescent in situ hybridization with transposable element probes to mitotic chromosomal heterochromatin of Drosophila. Methods in Molecular Biology. 260, 29-39 (2004).
- Pardue, M. L. In situ hybridization to polytene chromosomes in Drosophila using digoxigenin-labeled probes. Cold Spring Harbor Protocols. 2011 (8), 1003-1006 (2011).
- Hoskins, R. A., et al. Heterochromatic sequences in a Drosophila whole-genome shotgun assembly. Genome Biology. 3 (12), (2002).
- Hoskins, R. A., et al. Sequence finishing and mapping of Drosophila melanogaster heterochromatin. Science. 316 (58331), 1625-1628 (2007).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nature Reviews Genetics. 13 (1), 36-46 (2012).
- He, B., et al. Mapping the pericentric heterochromatin by comparative genomic hybridization analysis and chromosome deletions in Drosophila melanogaster. Genome Research. 22 (12), 2507-2519 (2012).
- Pimpinelli, S., Bonaccorsi, S., Fanti, L., Gatti, M. Fluorescent in situ hybridization (FISH) of mitotic chromosomes from Drosophila larval brain. Cold Spring Harbor Protocols. 2010 (3), (2010).
- Larracuente, A. M., Noor, M. A., Clark, A. G. Translocation of Y-linked genes to the dot chromosome in Drosophila pseudoobscura. Molecular Biology and Evolution. 27 (7), 1612-1620 (2010).
- Ferree, P. M., Barbash, D. A. Species-specific heterochromatin prevents mitotic chromosome segregation to cause hybrid lethality in Drosophila. PLoS Biology. 7 (10), e1000234 (2009).
- Williams, B. C., Karr, T. L., Montgomery, J. M., Goldberg, M. L. The Drosophila l(1)zw10 gene product, required for accurate mitotic chromosome segregation, is redistributed at anaphase onset. The Journal of Cell Biology. 118 (4), 759-773 (1992).
- Gatti, M., Bonaccorsi, S., Pimpinelli, S. Looking at Drosophila Mitotic Chromosomes. Methods in Cell Biology. 44, 371-391 (1994).
- Werren, J. H., Stouthamer, R. PSR (paternal sex ratio) chromosomes: the ultimate selfish genetic elements. Genetica. 117 (1), 85-101 (2003).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved