A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מערכת ג'ל קונצנטריים כדי ללמוד את תפקיד Biophysical של מטריקס מייקרו-סביבה על נדידת תאי 3D
In This Article
Summary
הנכסים ומייקרו של המטריצה תאית מכאניים מאוד משפיעים על הגירת 3D של תאים. שיטה במבחנה ללמוד spatiotemporal התנהגות נדידת תאים בסביבות biophysically משתנים, בשתי רמות אוכלוסייה ותא בודדות, מתואר.
Abstract
היכולת של תאים לנדוד היא חיונית במגוון רחב של פונקציות תא לאורך כל חיים מהתפתחות עוברית וריפוי פצעים לגידול וגרור סרטני. למרות מאמצי מחקר אינטנסיביים, העקרונות ביוכימיים וbiophysical הבסיסיים של נדידת תאים עדיין לא הבינו באופן מלא, במיוחד במייקרו-הסביבות הרלוונטיות מבחינה פיזיולוגית תלת ממדי (3D). כאן אנו מתארים assay במבחנה נועד לאפשר בחינה הכמותית של התנהגויות נדידת תאי 3D. השיטה מנצלת את יכולתו של התא mechanosensing ונטייה לנדוד לתוך מטריצה פנויה בעבר תאי (ECM). אנו משתמשים בפלישה של תאי סרטן השד פולשני מאוד, מד"א MB-231, ג'ל קולגן כמערכת מודל. התפשטות אוכלוסיית תא ודינמיקת ההגירה של תאים בודדים מעל שבועות של התרבות ניתן לנטר באמצעות Live- תא הדמיה וניתחו כדי לחלץ נתונים spatiotemporally נפתרו. יתר על כן, השיטה היא להתאמה בקלות למטריצות תאית מגוונות, ובכך להציע דרך פשוטה אך רבת עוצמה כדי לחקור את תפקידם של גורמי biophysical במייקרו-הסביבה על נדידת תאים.
Introduction
נדידה של תאים ממלאים תפקיד מרכזי בתגובות פיסיולוגיות שונות כגון התפתחות עוברית, haemostasis, ותגובה חיסונית, כמו גם בתהליכים פתולוגיים כגון מחלות לב וכלי דם, דלקת, וסרטן 1. לנתח את הגורמים ביוכימיים וbiophysical יסוד נדידת תאים ולכן חשוב ביסודו לא רק כדי להבין את העקרונות הבסיסיים של פונקציות סלולריות, אלא גם לקידום יישומים ביו-רפואיים שונים, כגון בהנדסת רקמות, אנטי-גרורות ופיתוח תרופות אנטי-דלקתי. מאז תצפית in vivo היא מאתגרת מבחינה טכנית, הרבה מאמצים התמקד בסיכום במבחנה של נדידת תאים.
בשיטות מבחנה ללמוד נדידת תאים במידה רבה תוכננו עבור מבחני על שני משטחים ממדיים (2D), שהבולטים בם השריטה או פצע ריפוי assay 2. מבחני כאלה מציעים התקנה ניסיונית פשוטה, קל live-הדמיה תא, ולספק תובנות שימושיות למנגנונים ביוכימיים שונים שבבסיס נדידת תאים. עם זאת, מבחני אלו אינם מהווים מטריצת ארכיטקטורה תאית (ECM) ושיפוץ, שהם היבטים קריטיים בהבנה בהגירה vivo. לאחרונה, זה כבר מוערך יותר ויותר כי מודל תרבות 3D, לעתים קרובות במטריצות מבוססות קולגן 3, מספק פלטפורמה שיותר דומה למצב בvivo. ואכן, תאי תערוכת דינמיקת migrational כי הם שונים מאלה על משטחי 2D, בעיקר בשל ממדית של הסביבה 4 השונים. יתר על כן, את מאפייני biophysical ומכאניים של מטריקס ברגישות משפיעים תא הגירה 5, כוללים בהקשר של תאים סרטניים פלישה 6.
כאן, אנו מציגים שיטה ללמוד 3D נדידת תאי התנהגות בECM עם מאפייני biophysical שיכול להיות בקלות מגוונת עם תנאי הכנה. התאיםזורעים ב" ג'ל הפנימי "ולאפשר להם להימלט וללפלוש" ג'ל החיצוני "בתחילה acellular. השיטה מסתמכת על יכולתו של התא להכיר בנוכחותו של, ונטייה להגר ל, אזורי תא ללא בג'ל החיצוני, אשר קשור באופן הדוק לתא mechanosensing 7. במחקר זה, אנו מעסיקים רשתות קולגן כECMS פלש על ידי תאי סרטן השד פולשני מאוד, MD-MBA-231. יכולים להיות מכוון הנכסים ומייקרו של שני ג'לים הפנימיים וחיצוניים מכאניים 8 ומאופיינות 9 כדי להשיג תנאים רלוונטיים מבחינה פיזיולוגית. שחזור וניתוח של מסלולי התא מאפשרים בדיקה כמותית מפורטת של הגירת התנהגות spatiotemporal בשתי רמת האוכלוסייה ורמת תא בודדת. חשוב לציין, ההתקנה של מערכת ג'ל קונצנטריים מחקה את in vivo טופולוגיה הרקמה מתמודדת על ידי תאים נודדים, במיוחד פולשים תאים סרטניים, ובכך להציע תובנות חשובות בלמנגנונים הפיזיים של נדידת תאים וגרורות.
Protocol
קציר 1. תא
- להשיג MD-MBA-231 תאים מ37 ° C, החממה 5% CO 2. ניתוק תאים מצלחת תרבית רקמה באמצעות 0.5% פתרון טריפסין- EDTA. השתמש 1 מיליליטר של תמיסת טריפסין-EDTA לתאים בתרבית בבקבוק T25.
- תאים גלולה בצינור חרוטי 15 מיליליטר על ידי צנטריפוגה ב × 200 גרם במשך 4 דקות, לשאוב supernatant, ותאי resuspend ב 5 מיליליטר של תקשורת בתרבות.
- לספור צפיפות תאים, ρ, באמצעות hemocytometer.
הערה: כדי להכין את הג'ל הפנימי התא-זרע, ההשעיה התא תהיה בהמשך להיות מדולל 10 × להגיע צפיפות זריעת תא הסופית. לכן, נדרש 10 × השעיה תא מרוכז. - חשב את נפח הבינוני הנדרש כדי להשיג ריכוז 10 × תא:
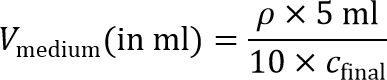
הערה: צפיפות זריעת תא סופית, ג סופי, של כ 215; 10 6 תאים / מיליליטר מומלץ לMD-MBA-231 תאים ומשמש בפרוטוקול זה. צפיפות זריעה אחרת גם יכולה להיות חוקרים לסוגי תאים אחרים. - תאים גלולה עוד פעם אחת בצינור חרוטי 15 מיליליטר על ידי צנטריפוגה ב × 200 גרם במשך 4 דקות, ולשאוב supernatant.
- Resuspend התאים בכמות הנדרשת (מדיום V מחושבים בשלב 1.3) של מדיום תרבית תאים ללא סרום ביסודיות כדי למזער clumping תא.
הערה: פנול אדום הוא auto-ניאון, ויכולים להפריע להדמית הקרינה / החזרה. שימוש במדיום ללא פנול אדום עשוי להיחשב להשגת איכות תמונה הטובה ביותר.
2. הכנת פתרונות קולגן
- השג את פתרון קולגן המניות, 10 × חיץ PBS, מילי-Q H 2 O, 0.1 M NaOH, וכמה צינורות microcentrifuge. שמור את כל על קרח כדי למנוע פילמור קולגן המוקדם, ולשמור על מצב סטרילי.
- לאזן זכוכית סטריליתצלחת תחתית על ידי ההתחממות מראש בחממת 37 ° C.
הערה: כל הכרכים בפרוטוקול זה כבר מותאם למנת זכוכית תחתונה עם 12 מ"מ גם. אם נעשה שימוש בסוג מנה אחר, להתאים את הכרכים בהתאם. - חשב את הנפח הנדרש הדרוש כדי להכין 50 μl של 2.4 מ"ג / מיליליטר פתרון קולגן לג'ל הפנימי (אני פתרון) המבוסס על מניית קולגן הריכוז.
הערה: ניתן להשתמש גם בריכוזי קולגן אחרים לג'ל הפנימי. - בסביבת סטרילית (בדרך כלל מכסה המנוע בטיחות ביולוגי), להוסיף באיטיות 5 μl של 10 × חיץ PBS לכמות הנדרשת של מניות פתרון קולגן (מחושבת בשלב 2.3) עם מתערבל עדין. תשמור על עצמך, כדי למנוע היווצרות בועת אוויר.
- התאם את ה- pH של התערובת ל -7.4 באמצעות 0.1 M NaOH באמצעות pH מטר מכויל. כמדריך גס, השתמש כ -5 μl להביא קרוב pH 7.4 (הסכום משתנה בהתאם לריכוז המניות וpH).
הערה: שים לב שהכרכים מעורבים בזההצעד קטן מדי לשימוש במד pH סטנדרטי. השתמש באחד הטריקים הבאים:- הכן פתרונות קולגן לדוגמאות רבות. התאם את ה- pH בכמות גדולה באמצעות מד pH סטנדרטי ולהפיץ את פתרונות קולגן פני הדגימות.
- לחלופין, לשנות את ה- pH של פתרון קולגן בנפח גדול יותר (i. דואר., נפח המאפשר שימוש במד pH סטנדרטי). שים לב לכמות של NaOH הדרוש כדי להביא את ה- pH ל pH הסופי. להקטין את הכמויות ולהשתמש בכמות המתאימה של NaOH לניסוי. אשר את ערך ה- pH משתמש בנייר לקמוס.
- אחרת, להשתמש אלקטרודת מיקרו כדי להתאים את ה- pH של כמויות קטנות באופן מדויק יותר.
- להביא את הפתרון להיקף של 45 μl באמצעות H 2 O. לבצע את כל הצעדים על קרח כדי למנוע פילמור קולגן המוקדם.
3. כינונה של קונצנטריים ג'ל תרבות
- קח את צלחת הזכוכית תחתונה המחוממת מראש (ראה שלב 2.2) מהחממת דואר.
- הוסף 5 μl של ההשעיה 10 × המרוכז התא (מוכנה בשלב 1.5) לפתרון I. Resuspend ביסודיות. תשמור על עצמך, כדי למנוע היווצרות בועת אוויר. התערובת עכשיו יש נפח של 50 μl ומכילה את הריכוז הסופי קולגן (2.4 מ"ג / מיליליטר) וצפיפות תאים (c = 2 × 10 6 תאים / מיליליטר סופי).
- הוסף 20 μl של הפתרון המכיל תאים שאני לאט לאט למרכז היטב, כך שהיא מהווה טיפה (איור 1 א) בצורה כיפה. תשמור על עצמך כדי למנוע היווצרות בועה בשלב זה. אם צורות בועה, בזהירות אבל במהירות מנסה גם לפגוע בו או למצוץ אותו החוצה באמצעות קצה פיפטה. מניח בעדינות את הצלחת בחזרה בחממה כדי לאפשר את הג'ל הפנימי פלמר במשך 45 דקות.
- הכן O פתרון (לג'ל קולגן החיצוני, acellular) כ -15 דקות לפני תום שלב דגירה זה.
הערה: מצב ג'ל החיצוני יכול להיות מגוון במונחים של ריכוז קולגן וpH פילמור ללהשיג ברשתות עם microstructures שונה 10. בפרוטוקול זה, יתמקד ב1.5-4.0 מ"ג / מיליליטר ג'ל קולגן polymerized בpH של 7.4.- בהתבסס על מניית קולגן הריכוז, לחשב את הנפח הנדרש הדרוש כדי להכין 200 μl של O פתרון קולגן בריכוז הסופי.
- הוסף 20 μl של 10 × PBS חיץ לאט את הכמות הנדרשת של מניות פתרון קולגן (מחושב בשלב 3.4) עם מתערבל עדין. התאם את ה- pH של התערובת לpH הסופי באמצעות 0.1 M NaOH עם השימוש של pH מטר מכויל. ראה הערה לצעד 2.5 לגבי התאמת pH.
- להביא את הפתרון לנפח סופי של 200 μl באמצעות H 2 O. לבצע את כל הצעדים על קרח כדי למנוע פילמור קולגן המוקדם.
- קח את הצלחת מן החממה לאחר 45 דקות של פילמור ג'ל הפנימי (ראה שלב 3.3). בעדינות להוסיף 180 μl של O פתרון על גבי ג'ל הפנימי, כך שהפתרון מכסה לחלוטין פנימיג 'ומתמלא היטב (איור 1).
- לבצע שלב זה בזהירות מבלי ערבוב הפתרון, אשר יכול להוביל לכיווני סיבים לא אחידים בג'ל החיצוני. תשמור על עצמך כדי להימנע מלגעת בג'ל הפנימי עם קצה פיפטה, וכדי למנוע את היווצרותן של בועות או כיסי אוויר. אם צורות בועה, בזהירות אבל במהירות מנסה גם לפגוע בו או למצוץ אותו החוצה באמצעות קצה פיפטה. מניח בעדינות את הצלחת בחזרה בחממה כדי לאפשר לפלמר ג'ל החיצוני.
- קח את הצלחת מן החממה לאחר 45 דקות של פילמור. ג'ל צריך להיות כבר התגבש למדי בשלב זה, למרות שהוא עדיין יכול להתנתק מהמשטח התחתון אם טופל בגסות.
- בעדינות יוצק 2 מיליליטר של מדיום תרבית תאים חימם לצלחת (איור 1 ג). לוודא כי ג'ל הוא שקוע לחלוטין בטווח הבינוני. רענן הבינוני כל 2-3 ימים לכל אורכה של התרבות.
4. Live- תא הדמיה
- לבצע הדמיה באמצעות מיקרוסקופ confocal הפוך מצוידים ביכולת Live- תא הדמיה לטווח ארוך. כולל מובנה בתא דגירה עם טמפרטורה (37 ° C) ובקרת CO 2 (5%). לעבור על המיקרוסקופ ולחמם את הבמה שעה לפחות 1 לפני תחילת הניסוי.
הערה: עדשה אובייקטיבית שימוש עם מרחק עבודה ארוך כדי לייעל את התצפית ולוקליזציה של תאים בג'לים 3D. - דגירה ג'ל במדיום המכיל 5 μl של צבע גשש תא ניאון למשך 30 דקות כדי לאפשר לוקליזציה מדויקת של התאים במערכת 3D. בהמשך לכך, להסיר צבע מאוגד על ידי שטיפה שלוש פעמים עם 1 × PBS. לאחר מכן, להוסיף בינוני תרבות תאים לצלחת.
- קח את הצלחת מן החממה ולמקם אותו על הבמה מיקרוסקופ (1D איור).
הערה: הדמיה תא חי יכולה להתחיל בעיקרון מייד לאחר פילמור של ג'ל החיצוני. עם זאת, בשלב זה, התאים in ג'ל הפנימי לא התפשט עדיין. כדי לבחון את ההגירה של התאים הראשונים שפלשו ג'ל החיצוני, Live- תא הדמיה יכולה להתחיל 24 שעות (תלוי בסוג התא) לאחר תחילתה של התרבות. כמדריך גס, סביב 12 - 14 ימים של התרבות יש צורך ברוב של התאים בג'ל הפנימי להיכנס לג'ל החיצוני. - כרכים נבחרים של צפה (vov של) באזורים בג'ל החיצוני המקיף את הג'ל הפנימי. לאחר 24 שעות של דגירה, אוכלוסיית התא התפשטה, חצה את הממשק בין ג'לי הפנימי וחיצוני, והתחיל לפלוש ג'ל החיצוני.
- לvov של, כוללים אזורי ג'ל מייד ליד ממשק ג'ל, אזורי ביניים, והאזורים הקרובים לoutskirt של ג'ל החיצוני 7. לא לכלול אזורים קרובים מ -50 מיקרומטר מהמשטחים התחתון וצד, כמו גם מהחלק העליון של ג'ל, כדי למנוע תופעות קצה אפשריות. כל vov בדרך כלל מודד 647 × 647 × 100 מיקרומטר 3 (בx, Y, z וכיוונים, בהתאמה), עם מרווח של 5 מיקרומטר -stack z.
- ודאו מצבי הדמיה, ערוצים / מסננים, זמני חשיפה, והחלטות תמונה נבחרות בצורה נכונה. עבור הדמיה ללא תווית של רשת קולגן, להשתמש במיקרוסקופ ההחזרה confocal בו-זמנית בLive- תא ההדמיה הזמן לשגות.
- קח תמונות לדוגמא, העלילה היסטוגרמות העצמה, ולהתאים את הרווחים וקיזוזים להתבונן אות מספיק ולמנוע הרוויה על ידי הבטחה כי היסטוגרמה נמצאת בין אפס והעוצמה מקסימלית. אין לשנות הגדרות אלה יותר לאורך כל תקופת הניסוי.
- קח תמונות של vov של נבחר זמן לשגות, עם מרווח זמן, t Δ, של 10 דקות במשך 8 שעות (או יותר במידת צורך).
5. מעקב סלולארי וניתוח נתונים
- לבצע לאחר עיבוד תמונה כמותי מתמונות -stack z באמצעות appropria תוכנת עיבוד תמונת te.
- קטע הזמן לשגות תמונות כדי לבחור את עמדות תא 3D ב( x, y, z, t) באופן אוטומטי.
- לכל מסגרת, לבדוק באופן ידני את דיוק הלוקליזציה ולהסיר חיוביים שגוי בשל פסולת תא ובליטות סלולריות שעשויים להיחשב בטעות לתאים. הסרת תאי שגשוג מהניתוח והתאים חופפים או קשורים פיצול לאובייקטים שונים.
- ליצור מסלולי תא הזמן לשגות 3D מקואורדינטות התא (x, y, z, t) שהושגו בשלב הקודם על ידי קישור למיקום של כל תא ברצף הזמן.
- לחסל את הרעש אקראי ומערכת על ידי הסרת מסלולים קצרים יותר מאורך מסלול סף (בדרך כלל 20 דקות).
- לתקן את סחיפת מדגם על ידי הפחתת ההתקות נטו הכולל מהמסלולים במידת צורך.
- חישוב תזוזת תא,Iles / ftp_upload / 52,735 / 52735eq2.jpg "width =" 80 "/>, ומרחק נדידת תאים,
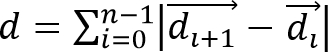 , ממסלולי התא הנצפים, שבו הוא וקטור המייצג את מיקום 3D של תא בזמן והוא המספר הכולל של נקודות זמן.
, ממסלולי התא הנצפים, שבו הוא וקטור המייצג את מיקום 3D של תא בזמן והוא המספר הכולל של נקודות זמן. - לחשב את מהירות תא כS = ד / (t Δ • n), כאשר t Δ הוא את מרווח הזמן בין מסגרות. לחשב יווני נדידת תאים (או התמדה) באמצעות P = Δd / ד. מדד פשוט זה של התמדה מרמז כי לP = 0 העקירה נטו היא אפס ולP = 1 המסלול הוא קו ישר כיוונית.
תוצאות
Assay ג'ל קונצנטריים מוצג כאן בוצע באמצעות תאי סרטן השד פולשני מאוד, מד"א MB-231, עם 2.4 מ"ג / מיליליטר ג'ל קולגן הפנימי וצפיפות זריעת תא של = 2 × 10 6 תאים / מיליליטר, כדוגמא. כפי שניתן לראות באיור 2, בדרך כלל אחרי כמה ימים של תרבות, תאי הפר ממשק ג'ל הפנימי-ח...
Discussion
In this protocol we describe an in vitro assay to study the 3D migrational behavior of cells in matrix environments that topologically resemble ECMs encountered in vivo. There are three main strengths of this assay as compared to other currently available methods. First, this assay allows one to simultaneously examine the cell migration mechanisms at both population level and individual cell level. This opens up possibilities of studying collective cell migration13, which has to date been lar...
Disclosures
יש לי המחברים אין לחשוף.
Acknowledgements
המחברים מודים W. שמש וק Jansen לדיונים הביקורתיים, ומכירים תמיכה על ידי מעבדת ביומכניקה Nano באוניברסיטה הלאומית של סינגפור. NAK מודה תמיכה על ידי מארי קירי IIF מלגה.
Materials
| Name | Company | Catalog Number | Comments |
| Cell culture incubator | Fisher Scientific Pte Ltd | Model: 371, S/No 318854-6055 | |
| Confocal microscope | Nikon A1R | Inverted confocal laser scanning microscope equipped with incubator chamber | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Life Technologies | 11965-092 | |
| Fetal Bovine Serum (FBS) | Life Technologies | 10082-147 | |
| Fluorescent CellTracker dye CMTMR | Life Technologies | C2927 | |
| Glass-bottom dish | IWAKI Cell Biology | 3931-035 | 35 mm diameter dish with 12 mm diameter glass-bottom well |
| Hemocytometer | iN CYTO | DHC-N01 (Neubauer Improved) | |
| Microprocessor pH meter | Hanna Instruments | pH 211 | |
| Nutragen Collagen | Advanced BioMatrix | #5010-D | Acid-solubilized bovine collagen type I (stock pH ~ 2) |
| Objective lens | Nikon | CFI Super Plan Fluor ELWD ADM 20XC, W.D. 8.2-6.9mm, NA 0.45. | |
| Penicillin-Streptomycin | Life Technologies | 15140-122 | |
| pH meter | Sartorius | S/No 29153352 | Basic pH Meter PB-11 |
| Trypsin-EDTA | Life Technologies | 15400-054 |
References
- Horwitz, R., Webb, D. Cell migration. Curr Biol. 13 (19), R756-R759 (2003).
- Liang, C. C., Park, A. Y., Guan, J. L. In vitro scratch assay: a convenient and inexpensive method for analysis of cell migration in vitro. Nat. Protoc. 2 (2), 329-333 (2007).
- Provenzano, P. P., Eliceiri, K. W., Inman, D. R., Keely, P. J. Engineering three-dimensional collagen matrices to provide contact guidance during 3D cell migration. Curr. Prot. Cell Biol. 10, 10-17 (2010).
- Friedl, P., Sahai, E., Weiss, S., Yamada, K. M. New dimensions in cell migration. Nat. Rev. Mol. Cell Biol. 13 (11), 743-747 (2012).
- Grinnell, F., Petroll, W. M. Cell motility and mechanics in three-dimensional collagen matrices. Annu. Rev. Cell. Dev. Biol. 26, 335-361 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nat. Rev. Cancer. 11 (7), 512-522 (2011).
- Sun, W., Kurniawan, N. A., Kumar, A. P., Rajagopalan, R., Lim, C. T. Effects of migrating cell-induced matrix reorganization on 3D cancer cell migration. Cell. Mol. Bioeng. 7 (2), 205-217 (2014).
- Achilli, M., Mantovani, D. Tailoring mechanical properties of collagen-based scaffolds for vascular tissue engineering: the effects of pH, temperature and ionic strength on gelation. Polymers. 2 (4), 664-680 (2010).
- Kurniawan, N. A., Wong, L. H., Rajagopalan, R. Early stiffening and softening of collagen: interplay of deformation mechanisms in biopolymer networks. Biomacromolecules. 13 (3), 691-698 (2012).
- Sun, W., Lim, C. T., Kurniawan, N. A. Mechanistic adaptability of cancer cells strongly affects anti-migratory drug efficacy. J. R. Soc. Interface. 11 (99), 20140638 (2014).
- Guzman, A., Ziperstein, M. J., Kaufman, L. J. The effect of fibrillar matrix architecture on tumor cell invasion of physically challenging environments. Biomaterials. 35 (25), 6954-6963 (2014).
- Wolf, K., et al. Physical limits of cell migration: control by ECM space and nuclear deformation and tuning by proteolysis and traction force. J. Cell Biol. 201 (7), 1069-1084 (2013).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol. Cell Biol. 10 (7), 445-457 (2009).
- Vedula, S. R. K., Ravasio, A., Lim, C. T., Ladoux, B. Collective cell migration: a mechanistic perspective. Physiology. 28 (6), 370-379 (2013).
- Petrie, R. J., Doyle, A. D., Yamada, K. M. Random versus directionally persistent cell migration. Nat. Rev. Mol. Cell Biol. 10 (8), 538-549 (2009).
- Miron-Mendoza, M., Seemann, J., Grinnell, F. The differential regulation of cell motile activity through matrix stiffness and porosity in three dimensional collagen matrices. Biomaterials. 31 (25), 6425-6435 (2010).
- Mouw, J. K., et al. Tissue mechanics modulate microRNA-dependent PTEN expression to regulate malignant progression. Nat. Med. 20 (4), 360-367 (2014).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin. Cell Dev. Biol. 20 (8), 931-941 (2009).
- Wong, L. H., Kurniawan, N. A., Too, H. -. P., Rajagopalan, R. Spatially resolved microrheology of heterogeneous biopolymer hydrogels using covalently bound microspheres. Biomech. Model. Mechanobiol. 13 (4), 839-849 (2014).
- Gupton, S. L., Waterman-Storer, C. M. Spatiotemporal feedback between actomyosin and focal-adhesion systems optimizes rapid cell migration. Cell. 125 (7), 1361-1374 (2006).
- Provenzano, P. P., Inman, D. R., Eliceiri, K. W., Trier, S. M., Keely, P. J. Contact guidance mediated three-dimensional cell migration is regulated by Rho/ROCK-dependent matrix reorganization. Biophys. J. 95 (11), 5374-5384 (2008).
- Wolf, K., et al. Multi-step pericellular proteolysis controls the transition from individual to collective cancer cell invasion. Nat. Cell Biol. 9 (8), 893-904 (2007).
- Jansen, K. A., Bacabac, R. G., Piechocka, I. K., Koenderink, G. H. Cells actively stiffen fibrin networks by generating contractile stress. Biophys. J. 105 (10), 2240-2251 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved