A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
חלבונים Immunofluorescence ניתוח של אנדוגני אקסוגניים centromere-קינטוכור
In This Article
Summary
Here we report protocols to detect endogenous and exogenous centromere-kinetochore proteins in human cells and quantify these protein levels at centromeres-kinetochores by indirect immunofluorescent staining through the use of fixation (paraformaldehyde, acetone, or methanol fixation).
Abstract
"Centromeres" and "kinetochores" refer to the site where chromosomes associate with the spindle during cell division. Direct visualization of centromere-kinetochore proteins during the cell cycle remains a fundamental tool in investigating the mechanism(s) of these proteins. Advanced imaging methods in fluorescence microscopy provide remarkable resolution of centromere-kinetochore components and allow direct observation of specific molecular components of the centromeres and kinetochores. In addition, methods of indirect immunofluorescent (IIF) staining using specific antibodies are crucial to these observations. However, despite numerous reports about IIF protocols, few discussed in detail problems of specific centromere-kinetochore proteins.1-4 Here we report optimized protocols to stain endogenous centromere-kinetochore proteins in human cells by using paraformaldehyde fixation and IIF staining. Furthermore, we report protocols to detect Flag-tagged exogenous CENP-A proteins in human cells subjected to acetone or methanol fixation. These methods are useful in detecting and quantifying endogenous centromere-kinetochore proteins and Flag-tagged CENP-A proteins, including those in human cells.
Introduction
"צנטרומר" הוגדר קלסי כאזורים של רקומבינציה meiotic מורחקת בגנטיקה ומאוחר יותר הוכר ההתכווצות העיקרית של הכרומוזומים mitotic, אשר ממלאת תפקיד חיוני פרדת כרומוזום מדויקת במהלך מיטוזה. "קינטוכור" תואר מבנים הרבים השכבתיים אשר נקלטים על ידי microtubules על פני השטח של צנטרומר, כפי שהיא מתגלה על ידי מיקרוסקופי אלקטרונים; "קינטוכור" הוגדרו מאוחר כמתחם macromolecular שמתאימה בבית centromere של הכרומוזומים mitotic. למרות הבדלים דרמטיים של רצפי DNA centromeric בין חוליות, מבנה קינטוכור ורכב הם שמורים ביותר. אינטראקציה דינמית בין microtubules ציר ואת קינטוכור נדרשת הפרדה נאמן של הכרומוזומים במהלך מיטוזה, ופגמים בעופרת פונקציה centromere-קינטוכור כדי aneuploidy ובכך סרטן.
Centromere ברוב אאוקריוטיםאין רצף DNA מוגדר, אבל מורכב מערכים גדולים (0.3-5 Mb) של DNA alphoid החוזר מורכב 171-נ"ב DNA α-הלווין. למעט ניצני שמרים, זהות centromere מושגת לא על ידי רצף ה- DNA אלא על ידי נוכחות של הנוקלאוזום מיוחד המכיל את גרסה H3 היסטון CenH3 (חלבון centromere [CENP-א] בבני אדם). 5 נוקלאוזום CENP-A למקם אל צלחת פנימית של קינטוכור יונק 7 ו נקשר לדנ"א 171-נ"ב α-הלווין. צנטרומר Active דורש CENP-A-המכיל נוקלאוזום לכוון את הגיוס של רשת centromere קשור מכוננת (CCAN) והחלבונים קינטוכור, שביחד להסדיר את הקובץ המצורף של הכרומוזומים אל ציר mitotic והתקדמות מחזור לאחר מכן במחסום הכישור.
לאור הראיות הנ"ל, CENP-A הוצע להיות סימן אפיגנטיים של centromere 8; עם זאת, התהליך שבאמצעותו-A CENP משולב iDNA centromeric n כדי וגורמי אחראי התאגדות זה טרם מאופיין היטב. תחום קצר מיקוד-centromere (CATD) מתגורר היסטון לקפל באזור CENP-A, והחלפת באזור המקביל H3 עם CATD מספיקה כדי H3 ישירה centromere. 9 מספר מחקרים הציעו תפקידים פונקציונאליים-translational פוסט שינוי (PTM) של CENP-A 12-16; עם זאת, המנגנונים המולקולריים של PTMs אלה של-A CENP בגיוס כדי צנטרומר טרם הובהר. דיווחנו בעבר כי CUL4A-RBX1-COPS8 פעילות האנזים E3 נדרש CENP-A K124 ubiquitylation ולוקליזציה של CENP-א 'עד צנטרומר. 17
התגלית ואפיון של חלבונים קינטוכור הובילו לתובנה חדשה לגבי הפרדה כרומוזום. 18 יותר מ -100 רכיבים קינטוכור זוהו בתאים חוליות ידי גישות שונות. 19,20 תחתמעמדו של איך קינטוכור להרכיב ותפקוד מגיע גם מן האפיון של התפקודים התאיים של כל חלבוני centromere-קינטוכור ורשת החלבונים בתוך תאים. 19 להדמיה ישירה ושיטות הדמיה מתקדמת מיקרוסקופ פלואורסצנטי לספק רזולוציה מרשימה של רכיבים-קינטוכור centromere ולאפשר תצפית ישירה של רכיבים מולקולריים ייחודיים של צנטרומר ו קינטוכור. בנוסף, שיטות immunofluorescent העקיף (IIF) מכתים באמצעות נוגדנים ספציפיים הם קריטיים תצפיות אלה. עם זאת, למרות דיווחים רבים על פרוטוקולים IIF, כמה דנו בבעיות פרט חלבונים centromere-קינטוכור ספציפיים. 1-4 לפיכך, פיתוח ודיווח שיטות מכתים IIF וכן assay IIF כמותית לנתח באופן ספציפי כל חלבון centromere-קינטוכור חשוב מאוד. בשנת מכתים IIF, אחד צריך להמשיך עם הפרוטוקול המכתים כדי למנוע אובדן של החלבון של עניין אושאר התא. עם זאת, קיבעון הורס אתרים אנטיגני מדי פעם, ושילובי נוגדן-אנטיגן שונים לעבוד היטב עם מקבע אחד, אבל טוב מאוד עם אחר, 21 ובחירה מקבע תלויים במידה רבה על החלבון (ים) של עניין. לכן, שיטות מקבעים שונות הן קריטיות מכתים IIF של חלבונים-קינטוכור centromere.
הנה שיטות אופטימיזציה של immunofluorescent עקיף (IIF) מכתים ו assay להתייחס לוקליזציה של חלבונים centromere-קינטוכור אנדוגני, כולל CENP-A ודגל מתוייגים אקסוגניים חלבונים CENP-A, ו לכמת את החלבונים האלה בתאים אנושיים פותחו. ניתן ליישם שיטות אלה כדי הניתוח של חלבונים-קינטוכור centromere במינים אחרים.
Protocol
1. תרבות Transfection ניידות
- שים כוס כיסוי (22 מ"מ x 22 מ"מ) בצלחת קלקר 6 באר. מעיל לחלופין כוס כיסוי עם פולי- L- ליזין, 0.1% w / v, במים (ראה רשימה של חומרים / ציוד) כדי לשמור על התאים mitotic על הזכוכית המכסה ביצוע השלבים הבאים:
הערה: תנאים אופטימליים צריכים להיקבע עבור כל קו תא ויישום.- משטח תרבות מעיל בסביבה נקייה מחיידקים עם פולי- L- ליזין, 0.1% w / v, במים (0.4 מ"ל / גם צלחת 6-גם קלקר). רוק בעדינות כדי להבטיח אפילו ציפוי של משטח התרבות.
- לאחר 5 דקות, להסיר פתרון על ידי שאיפה לשטוף ביסודיות משטחת במי כיתת רקמת סטרילית התרבות.
- יבש לפחות שעה 2 לפני החדרת התאים ובינוניים.
- תאי זרע הלה 17 או הלה ט-Off תאים 17,22 על כוס כיסוי (22 מ"מ x 22 מ"מ) להכניס צלחת קלקר 6 באר. בדוק כי צפיפות התאים הוא 5.4 x 10 5 לכל טוב. תרבות תאיםב DMEM גבוהה גלוקוז עם FBS 10% ו -1%, סטרפטומיצין פניצילין.
הערה: לקבלת תוצאות אופטימליות, באופן אמפירי לקבוע את צפיפות התאים לשימוש ב זריעה. - דגירת התאים ב 37 מעלות צלזיוס אווירה של 5% CO 2 עבור 18 שעות.
הערה: במקרה של תאים הלה ט-Off, שעתוק של הגן אקסוגניים פעילה בהעדר inducer (כלומר, טטרציקלין / דוקסיציקלין) ולכן התרבות תאים ללא טטרציקלין / דוקסיציקלין ו transfect זמני עם וקטור ביטוי יתר pTRM4 (טבלה 2 ), מוסדר שעתוק אשר על ידי אמרגן TRE (ראה להלן). - שמונה עשר שעות לאחר הזריעה, תאי transfect כדלקמן:
- בצע פתרון על ידי ערבוב אוליגו siRNA 1.5 μl (20 מיקרומטר המניות annealed; טבלה 1) ו / או 2.0 מיקרוגרם פלסמיד (טבלה 2) ב 50 μl בינוני סרום מופחת (ראה רשימת חומרים / ציוד), דגירה על RT במשך 5 דקות .
הערה: בניתוח זה,CA-UTR siRNAs (תערובת של 5 'ו 3' UTR siRNA; טבלה 1) היו-טרנספקציה שיתוף לשימוש הפרוטוקולים 3 ו -4 (ראה דיון). - הפוך B פתרון על ידי ערבוב 0.75 מגיב transfection μl לי (ראה רשימה של חומרים / ציוד) ב 50 μl המופחתים בינוני בסרום, לדגור על RT במשך 5 דקות.
הערה: צעד אופציונלי הוא תוספת של 1.0 מיקרוליטר מגיב transfection השנייה (ראה רשימה של חומרים / ציוד). - מערבבים פתרונות A ו- B יחד, לדגור על RT במשך 15 דקות.
- שוטפים את התאים בתרבית פעם עם PBS, ולאחר מכן להוסיף 500 μl מופחת בינוני בסרום היטב בכל צלחת קלקר 6 באר. מוסיפים את תערובת של פתרונות ו- B (כלומר, RNA ו / או מורכבות השומנים-DNA) ישירות זה של הפרט היטב.
הערה: הריכוז הסופי הוא 3.3 מיקרוגרם / מ"ל (פלסמיד); 50 ננומטר (siRNA). - דגירת התאים ב 37 מעלות צלזיוס אווירה של 5% CO 2 עבור 4.5 שעות. שינוי בינוני עד שנינות DMEM גלוקוז גבוההh 10% FBS ו -1%, סטרפטומיצין פניצילין.
- דגירה התאים ב 37 מעלות צלזיוס אווירה של 5% CO 2 עבור 48-72 שעות לאחר transfection.
הערה: לקבלת תוצאות אופטימליות, תקופת הדגירה של צמיחת תאים לפני הקיבוע צריכה להיקבע באופן אמפירי, ודלדול חלבון ו / או ביטוי חייב להיות מאושר על ידי ניתוח כתם מערבי (ראה פרוטוקול 6). - אם בניתוח תא mitotic הוא עניין, להוסיף פקליטקסל (10 ננומטר) תאים בתרבית 24 שעות לפני קיבעון, או להוסיף TN16 (0.5 מיקרומטר) אל hr 2.5 תאים בתרבית לפני קיבעון.
- בצע פתרון על ידי ערבוב אוליגו siRNA 1.5 μl (20 מיקרומטר המניות annealed; טבלה 1) ו / או 2.0 מיקרוגרם פלסמיד (טבלה 2) ב 50 μl בינוני סרום מופחת (ראה רשימת חומרים / ציוד), דגירה על RT במשך 5 דקות .
2. תא קיבוע Immunofluorescent מכתים כדי זיהוי חלבונים אנדוגניים centromere-קינטוכור (קיבוע Paraformaldehyde)
- הכנה מקבע, מאגרים, וכן חומרים כימיים.
- טרי להכין 50 מ"ל של תמיסת paraformaldehyde 4% ב- PBS, pH 7.4.
- הוסף 40 מ"ל של 1 × PBS על כוס זכוכית על צלחת ומערבבים במנדף מאוורר. מחממים תוך בערבובגרם לכ -60 מעלות צלזיוס. הוסף 2 גרם של אבקת paraformaldehyde אל PBS המחומם.
- הרם לאט את ה- pH על ידי הוספת 1 מ"ל של 1 N NaOH בסך הכל, כי האבקה לא מיד לפזר.
הערה: הפתרון מנקה לאחר תוספת של NaOH. - לאחר paraformaldehyde נמס, קריר לסנן את הפתרון.
- התאם את ה- pH עם 1 N HCl pH 7.4 והיקף הפתרון עם 1 × PBS 50 מ"ל.
הערה: Aliquots של הפתרון אפשר להקפיא או מאוחסן ב 2-8 מעלות צלזיוס למשך כל עוד 1 בשבוע. הפתרון צריך להיות קר כקרח או לאחסן 4 ° C עד שהוא לשמש.
- הכן 50 מ"ל של חיץ KB1, אשר מורכב של 10 mM Tris-HCl, pH 7.5; 150 מ"מ NaCl; 0.5% BSA; ו -0.5% Triton X-100.
הערה: BSA יש להוסיף טרי.
הערה: סדרת חיץ KB מבוססת על KB חיץ המתוארים בדוחות קודמים 1,2,4. - הכן 50 מ"ל של חיץ KB2, אשר מורכב של 10 mM Tris-HCl,pH 7.5; 150 מ"מ NaCl; ו -0.5% BSA.
הערה: BSA יש להוסיף טרי. - הכן 1 מ"ל של חיץ KB3 המכיל DAPI (50 ng / ml).
- הכן 100 מ"ל של מדיום הרכבה, אשר מורכב של 1 מ"ג / מ"ל p-phenylenediamine; 10% PBS, ו -90% גליצרול.
- התאם את ה- pH של 1 × PBS ל 9.8 עם 1 N NaOH, לפזר p-phenylenediamine בתמיסה, ולאחר מכן להוסיף גליצרול.
- חנות 1 aliquots מ"ל ב -80 ° C. להגן מפני אור.
- טרי להכין 50 מ"ל של תמיסת paraformaldehyde 4% ב- PBS, pH 7.4.
- הסר את מדיום התרבות על ידי שאיפה ב transfection 48-72 שעות שלאחר (ראה פרוטוקול 1) עבור קיבוע תא. תאים לשטוף פעם עם PBS. החל PBS לצד של בארות התרבות להימנע משיבוש פני השטח של התאים.
הערה: נקודת הזמן האופטימלית עבור קיבוע תא צריכה להיקבע באופן אמפירי. - תקן את התאים paraformaldehyde 4% ב PBS במשך 30 דקות ב 4 °. שוטפים את התאים פעמיים עם חיץ KB2 להסיר paraformaldehyde 4% שיורית מספיק.
- PermeabiLize הדגימות במאגר KB1 למשך 30 דקות ב RT. שוטפים את התאים פעם עם חיץ KB2, ולהוסיף חיץ KB2 במשך 5 דקות ב RT לאתרים גוש נוסף של מחייב ספציפי.
הערה: הצפת KB1 גם תורם משמעותי חסימה. - הסרת מכסה זכוכית (22 מ"מ x 22 מ"מ) מתוך צלחת קלקר 6-גם באמצעות מלקחיים. באמצעות עט מחסום הידרופובי (ראה רשימה של חומרים / ציוד), לצייר ריבוע ירוק חיוור או מעגל כדי ליצור מחסום הידרופובי סביב מדגם מכסה זכוכית. אין לגעת או להתקרב יותר מדי אל התאים עם עט מחסום הידרופובי. תניח את כוס כיסוי צלחת קלקר 6-באר חדשה.
- לדלל נוגדן ראשוני כדי centromere או חלבון קינטוכור (יחס דילול של 1: 100 עד 1: 200; ראו רשימה גם של חומרים / ציוד) ואו נוגדן אנטי CENP-B (יחס דילול של 1: 400) או אנטי centromere נוגדן (ACA) (יחס דילול של 1: 2,000) כסמן מיקום centromere במאגר KB2.
הערה: לקבלת תוצאות אופטימליות, את Concentra הסופיtion של הנוגדן הראשוני בפתרון זה צריך להיקבע באופן אמפירי. - החל נפח מספיק (בערך 30 μl) של הנוגדן הראשוני המדולל לטבול מדגם התא. דגירת מדגם התא עבור שעה 1 ב 37 מעלות צלזיוס. שוטפים את התאים 3 פעמים עם חיץ KB2.
- באמצעות חיץ KB2, לדלל נוגדנים משני fluorophore מצומדות (יחס דילול של 1: 100 עד 1: 200) המכוונים נגד נוגדן ראשוני.
הערה: לקבלת תוצאות אופטימליות, את הריכוז הסופי של נוגדנים משני בפתרון זה צריך להיקבע באופן אמפירי. - החל נפח מספיק (ירידה של μl בקירוב 30 על הזכוכית המכסה) של נוגדנים משני בדילול לטבול מדגם התא. דגירת מדגם התא עבור שעה 1 ב 37 מעלות צלזיוס. שוטפים את התאים 5 פעמים עם חיץ KB2 במהלך תקופה של 30 דקות (חמש 6 שטיפות דקות).
הערה: לקבלת תוצאות אופטימליות (כלומר, עבור הפסד מינימאלי של תאים), תנאי הכביסה האופטימלי צריך להיקבע Empirically. - החל נפח מספיק של חיץ KB3 המכיל DAPI (50 ng / ml) לטבול את המדגם התא. דגירת מדגם התא במשך 5 דקות ב RT. יש לשטוף את התאים 1-2 פעמים עם חיץ KB2.
- הר זכוכית המכסה המכיל מדגם התא לשקופית מייקרו.
- מניחים ירידה של הרכבה בינונית במרכז השקופית מיקרו.
- הסר נוזל מן המדגם התא, באמצעות הידיים או מלקחיים, מקם את המדגם במרכז השקופית מיקרו. הימנע בועות אוויר.
- הסר בינוני הרכבה עודף עם מגבת נייר.
3. תא קיבוע Immunofluorescent מכתים של בטרמינל C-דגל-tagged CENP-A חלבונים (קיבוע אצטון)
- הכנה
- כן אצטון 75% קרים כקרח.
- טרי להכין 50 מ"ל של PBS (pH 7.4) המכיל חלב 0.5% דל שומן 0.5% BSA.
- טרי להכין 50 מ"ל של PBS (pH 7.4) המכיל חלב 0.1% דל שומן 0.1% BSA.
- כן 1 מיליליטר של PBS (pH 7.4) המכיל DAPI (50-100 ng / ml).
- הכן 100 מ"ל של מדיום הרכבה כמתואר 2.1.5.
- הסר את מדיום התרבות על ידי שאיפה ב transfection 48-72 שעות שלאחר (ראה פרוטוקול 1) עבור קיבוע תא. תאים לשטוף פעם עם PBS. החל PBS לצד באר התרבות להימנע משיבוש פני השטח של התאים.
הערה: נקודת הזמן האופטימלית עבור קיבוע תא צריכה להיקבע באופן אמפירי. - תקנו את תאי אצטון קר כקרח 75%, ו דגירת התאים במשך 10 דקות ב -20 מעלות צלזיוס. תאי ניקוי על הזכוכית המכסה במנדף 30-60 דקות ב RT.
הערה: לקבלת תוצאות אופטימליות, את משך הזמן הדרוש לייבוש קיבעון תא צריך להיקבע באופן אמפירי. - באמצעות עט מחסום הידרופובי (ראה רשימה של חומרים / ציוד), לצייר ריבוע ירוק חיוור או מעגל כדי ליצור מחסום הידרופובי סביב מדגם מכסה זכוכית. אין לגעת או להתקרב יותר מדי אל התאים עם עט מחסום הידרופובי.
- בלוק nonspecific אתרי הקישור שבין התאים על ידי הוספת PBS המכיל חלב 0.5% דל שומן BSA 0.5% במשך 5 דקות ב RT.
- באמצעות PBS המכיל חלב 0.1% דל שומן BSA 0.1%, לדלל נוגדן אנטי הדגל (1: 1,000 יחס דילול) ואו נוגדן אנטי CENP-B (יחס דילול של 1: 200) או ACA (1: יחס דילול 2,000 כמו) סמן centromere מיקום.
הערה: לקבלת תוצאות אופטימליות, את הריכוז הסופי של הנוגדן הראשוני בפתרון זה צריך להיקבע באופן אמפירי. - החל נפח מספיק (בערך 30 μl) של הנוגדן הראשוני המדולל לטבול מדגם התא. דגירת מדגם התא עבור שעה 1 ב 37 מעלות צלזיוס. שוטף את התאים 5 פעמים עם חיץ החסימה במהלך תקופה של 30 דקות.
הערה: תאים ניתן לשטוף עם PBS. לקבלת תוצאות אופטימליות (כלומר, כדי למנוע אובדן של תאים), תנאי הכביסה האופטימליים צריכים להיקבע באופן אמפירי. - לדלל נוגדנים משני fluorophore מצומדות (יחס דילול של 1: 100 עד 1: 200) מכוונים נגדכל נוגדן ראשוני PBS המכיל חלב 0.1% דל שומן 0.1% BSA.
הערה: לקבלת תוצאות אופטימליות, את הריכוז הסופי של נוגדנים משני בפתרון זה צריך להיקבע באופן אמפירי. - החל נפח מספיק (בערך 30 μl) של נוגדנים משני בדילול לטבול מדגם התא. דגירת מדגם התא עבור שעה 1 ב 37 מעלות צלזיוס. יש לשטוף את התאים 2 פעמים עם PBS המכיל 0.1% חלב דל שומן 0.1% BSA.
הערה: PBS לבד יכול לשמש גם כדי לשטוף את התאים. - החל נפח מספיק של PBS המכיל DAPI (50-100 ng / ml) לטבול את המדגם התא. דגירת מדגם התא במשך 5 דקות ב RT. יש לשטוף את התאים 1-2 פעמים עם PBS.
- הר זכוכית המכסה המכיל מדגם התא לשקופית המייקר כמתואר 2.11.
4. תא קיבוע Immunofluorescent מכתים של N-terminal דגל-tagged CENP-A חלבונים (קיבוע מתנול)
- הכנה
- כן מתנול קר כקרח.
- כן TBS (pH 7.4) המכיל 4% נסיוב עז.
- הכן 1 מ"ל של TBS (pH 7.4) המכיל DAPI (50-100 ng / ml).
- הכן 100 מ"ל של מדיום הרכבה כמתואר 2.1.5.
- הסר את מדיום התרבות על ידי שאיפה ב transfection 48-72 שעות שלאחר (ראה פרוטוקול 1) עבור קיבוע תא. תאים לשטוף פעם עם TBS. החל TBS לצד של בארות התרבות להימנע משיבוש פני השטח של תאים.
הערה: נקודת הזמן האופטימלית עבור קיבוע תא צריכה להיקבע באופן אמפירי. - תקן את תאי מתנול קר כקרח, דגירת התאים למשך 6 דקות ב -20 מעלות צלזיוס. שוטפים את התאים פעמיים עם TBS להסיר מתנול שיורית מספיק.
- באמצעות עט מחסום הידרופובי (ראה רשימה של חומרים / ציוד), לצייר ריבוע ירוק חיוור או מעגל ליצור חיץ הידרופובי סביב מדגם מכסה זכוכית. אין לגעת או להתקרב יותר מדי אל התאים עם עט מחסום הידרופובי.
- חסום ספציפי אתרי קישור על cells ידי TBS הוספה המכיל 4% נסיוב עז. דגירה של 10 דקות ב RT.
- לדלל נוגדן אנטי הדגל (1: 1,000 דילול) ואו נוגדן אנטי CENP-B (יחס דילול של 1: 200) או ACA (1: 2,000 יחס דילול) כסמן מיקום centromere ב TBS המכילה סרום עיזים 4% .
הערה: לקבלת תוצאות אופטימליות, את הריכוז הסופי של הנוגדן הראשוני בפתרון זה צריך להיקבע באופן אמפירי. - החל נפח מספיק (בערך 30 μl) של הנוגדן הראשוני המדולל לטבול מדגם התא. דגירת מדגם התא עבור שעה 1 ב 37 מעלות צלזיוס. שוטף את התאים 5 פעמים עם חיץ החסימה במהלך תקופה של 30 דקות. לקבלת תוצאות אופטימליות (כלומר, ההפסד המינימאלי של תאים), תנאי הכביסה האופטימלי צריך להיקבע באופן אמפירי.
- לדלל נוגדנים משני fluorophore מצומדות (יחס דילול של 1: 100 עד 1: 200) המכוונים נגד נוגדן ראשוני ב TBS המכילה סרום עיזים 4%.
הערה: לקבלת תוצאות אופטימליות, את Finaריכוז l של נוגדנים משני בפתרון זה צריך להיקבע באופן אמפירי. - החל נפח מספיק (בערך 30 μl) של נוגדנים משני בדילול לטבול מדגם התא. דגירת מדגם התא עבור שעה 1 ב 37 מעלות צלזיוס. שוטפים את התאים 3 פעמים עם חיץ חסימה.
- החל נפח מספיק של TBS המכיל DAPI (50-100 ng / ml) לטבול את המדגם התא. דגירת מדגם התא במשך 5 דקות ב RT. יש לשטוף את התאים 1-2 פעמים עם כפות.
- הר זכוכית המכסה המכיל מדגם התא לשקופית המייקר כמתואר 2.11.
5. תצפית תמונה Immunofluorescence, רכישה, כימות, וניתוח
- שים מדגם התא דרך מיקרוסקופ פלואורסצנטי ממונע מצויד עדשת טבילת שמן 63X ו 100X, מקור אור קומפקטי חיצוני, ומצלמת CCD דיגיטלית.
- בצע רכישת תמונה ועיבוד, כולל deconvolution, באמצעות תוכנהA, או תוכנות B1 ו- B2 (רואים רשימה של חומרים / ציוד). עיין קבצי קוד משלימה (5.2.1) עבור כל הפקודות הנמצאות בשימוש ב א תוכנה עבור כל הפקודות בשימוש תוכנות B1 ו- B2, עיין (5.2.2) ב משלים קוד קבצים.
- השתמש שיטה שתואר לעיל 23-25 לכמת אותות של חלבונים-קינטוכור centromere (למשל, הנותרים אותות של-A CENP בבית centromere) עם שינויים קלים הבאים:
- בחירת האזור של חלבונים-קינטוכור centromere וכי הרקע כדלקמן:
- תא mitotic: (אזור קינטוכור-centromere) בחירת האזור חופף עם כרומוזומים מוכתמים DAPI; (אזור ברקע) ואת האזור שמחוץ הכרומוזומים אבל בתוך התא הבודד הזהה (כלומר, cytosolic באזור).
- שלבי ביניים תא: (אזור-קינטוכור centromere) בחירת האזור חופף עם הכרומטין מוכתם DAPI; (אזור ברקע) ואת האזור שמחוץ הכרומטין אבל בתוך התא הבודד הזהה ( כלומר, באזור cytosolic).
הערה: ראה גם קבצי קוד משלימה (5.2.1.4.3) או (5.2.2.6.4) לבחירה באזור עם תוכנה או תוכנת B2, בהתאמה.
- לכמת אחוז אותות הנותרים צנטרומר באמצעות תוכנה או B. עבור משימה זו, השתמש בנוסחה הבאה:
הנותרים אותות של חלבון centromere-קינטוכור בבית-קינטוכור centromere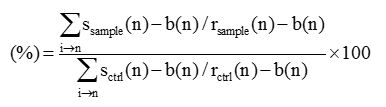
כאשר s בוהק האות של האזור הנבחר, אשר אושר על ידי ACA או מכתים CENP-B; ב בוהקת ברקע אות; מדגם r הוא ACA התייחסות או CENP-B אותות עבור siRNA (הים) תאי -treated; ו r ctrl הוא ACA או CENP-B אותות הייחוס עבור תאי-טרנספקציה siRNA לוק.
הערה: בניתוח זה, אותות CENP-B שימשו אותות הייחוס עבור CUL4A ו RBX1 כמתואר בדוחות קודמים. 17- השתמש בקובץ אקסל לחישוב זה אחרי שאתה מעתיק ומדביק את הנתונים הגולמיים מתוכנת quantitation האות שתוארה לעיל. ראה גם קבצי קוד משלימה: (5.2.1.4) ו (5.2.2.6) לפרטים. דוגמא החישוב אחת מוצגת בטבלה 3.
- לנתח לפחות 20 תאים לחסל וריאציה מכתימה רכישת תמונה עבור כל רמת מדידה. לחלופין להשתמש "ערך בממוצע" של הנותרים אותות-קינטוכור centromere לכל מנותח תא להשוות ערכים אלה בקרב חלבונים centromere-קינטוכור שונים.
- בחירת האזור של חלבונים-קינטוכור centromere וכי הרקע כדלקמן:
6. ניתוח כתם המערבי של חלבון סה"כ
- Resuspend התאים denaturing הצפת (20 mM Tris-HCl, pH 7.4, 50 mM NaCl; 0.5% Nonidet P-40; 0.5% deoxycholate; 0.5% SDS; 1 mM EDTA; קוקטייל ומלא-EDTA חינם מעכבי פרוטאז), נושא 26 את ההשעיה sonication ותהליך להקפיא להפשיר, ולמדודריכוזי חלבון כדלקמן:
- לקבלת מידע אודות תהליך sonication, להוסיף 50 μl של חיץ אל התאים שנאספו משתי הבארות של צלחת קלקר 6-היטב transfection 48-72 שעות שלאחר (ראה פרוטוקול 1). להפעיל sonicator מצויד צופר משבש ו microtip (ראה רשימה של חומרים / ציוד) למשך לסירוגין דופק הכולל 15 שניות (מחזור העבודה 50%) לדגימה אחד.
- כדי שתהליך להקפיא להפשיר, להקפיא תאים עם חנקן נוזלי להפשיר תאים ב RT.
- מדוד ריכוז חלבון באמצעות שאני מגיב assay החלבון מסחרי או שני (ראה רשימה של חומרים / ציוד).
הערה: Lysates מדולל ביחס של 1:10 במדידת ריכוזי חלבון או עם אני מגיב או שני. בדילול זה, בהווה SDS חיץ תערוכות מעט או ללא הפרעה במדידה זו.
- מערבבים את lysate המכיל 20-30 מיקרוגרם של החלבון הכולל עם 2 × או 4 × חיץ טעינת SDS-PAGE. 27 מרתיחים את הדגימות למשך5 דקות ולאחר מכן לטעון אותם על% 12.0% -15.0 denaturing ג'ל SDS-polyacrylamide אלקטרופורזה.
- מעביר את החלבונים מופרדים על ידי SDS-PAGE על קרום PVDF באמצעות שיטה מערבית סופגת שתוארה לעיל. 17,24,27-31
- חסום את אתרי הקישור ספציפי על הממברנה עם 5% חלב דל שומן ב 1 × PBS, ולאחר מכן דגירה הממברנה עם פתרונות של נוגדנים ראשוניים בדילול עבור שעה 1 ב RT. ראה רשימה של חומרים / ציוד עבור מידע מפורט (כדוגמת: יחס דילול) של כל נוגדן ראשוני.
- לאחר שטיפת פעמי קרום 3-4 (כל דגירת 3 עד 5 דקות עם רעד) עם חיץ PBS-T (1 × PBS ו -0.1% Tween-20), הדגירה הממברנה בתערובת של קרוב אינפרא אדום (IR) צבען מצומדות פלורסנט נוגדנים משני (יחס דילול של 1: 20,000), נוגדנים משני DyLight מצומדות (יחס דילול של 1: 20,000), ו / או peroxidase חזרת (HRP), מצומדות נוגדנים משני (בדילול מלא PBS-T; dilutiעל יחס של 1: 10,000) במשך שעה 1 ב RT. ראה רשימה של חומרים / ציוד עבור מידע מפורט (כדוגמת: יחס דילול) של נוגדן זה משני.
- לשטוף את הממברנה 3 פעמים, ולאחר מכן לסרוק את קרום לנתח את החלבונים עם מערכת הדמיית אינפרא אדום ו / או תרמי chemiluminescence לגילוי immunoblot (ראה רשימה של חומרים / ציוד).
- עבור שימוש במערכת ההדמיה אינפרא האדומה, ראה (6.4.1) ב קבצי קוד משלימה.
- לקבלה באמצעות תרמי chemiluminescence, ראה (6.4.2) ב קבצי קוד משלימה. השתמש chemiluminescent משופרת אולטרה רגיש (ECL) מצע (ראה רשימה של חומרים / ציוד) עבור מערכת זו.
תוצאות
ניתוח Immunofluorescence של CENP-A אנדוגני תומך בהשערה כי האנזים CUL4A-E3 נדרש עבור לוקליזציה של CENP-א 'עד צנטרומר
המחקרים האחרונים שלנו הראו כי CUL4A-RBX1-COPS8 פעילות אנזים E3 נדרשה ubiquitylation של ליזין 124 (K124) על CENP-A ולוקליזציה של CENP-א 'עד צנטרומר. 17 בתחי?...
Discussion
בשנים האחרונות מחקרים רבים פתחו מבחני מיקרוסקופיה כמותיים שונים עבור תאים קבועים. 42 התקדמות בביולוגיה-קינטוכור centromere לעתים קרובות דורש הבנה של קינטוכור ספציפי הספציפי centromere או תפקודם של חלבונים אשר מרחבית-טמפורלית subcellular תקנה משקפת את הפונקציות המשתנות של חל...
Disclosures
החוקרים מצהירים כי אין להם אינטרסים כלכליים מתחרים.
Acknowledgements
עבודה זו נתמכה על ידי GM68418 מענק NIH.
Materials
| Name | Company | Catalog Number | Comments |
| Lipofectamin 2000 | Life Technologies/Invitrogen | 11668 | transfection reagent I |
| Lipofectamin RNAiMAX | Life Technologies/Invitrogen | 13778 | transfection reagent II |
| Opti-MEM I | Life Technologies/Invitrogen | 31985 | Reduced serum media, warm in 37 °C water bath before use |
| High-glucose DMEM (Dulbecco’s modified Eagle’s medium) | Life Technologies/BioWhittaker | 12-604 | high-glucose DMEM, warm in 37 °C water bath before use |
| Fetal Bovine Serum, certified, heat inactivated, US origin | Life Technologies/Gibco | 10082 | FBS (fetal bovine serum) |
| Poly-L-Lysine SOLUTION | SIGMA-SLDRICH | P 8920 | Poly-L-Lysine, 0.1% w/v, in water |
| UltraPure Distilled Water | Life Technologies/Invitrogen/Gibco | 10977 | Sterile tissue culture grade water |
| Micro Cover glass (22 mm x 22 mm) | Surgipath | 105 | Cover glass (22 mm x 22 mm) |
| 6 Well Cell Culture Cluster | Fisher/Corning Incorporated | 07-200-83 | 6-well polystyrene plate |
| Penicillin, Streptomycin; Liquid | Fisher/Gibco | 15-140 | Penicillin-streptomycin |
| PAP PEN | Binding Site | AD100.1 | Hydrophobic barrier pen (for a water repellant barrier in immunofluorescent staining) |
| Paclitaxel (Taxol) | SIGMA-SLDRICH | T7402 | Taxol for mitotic cell analysis |
| TN-16, microtubule inhibitor (TN16) | Enzo Life Sciences | BML-T120 | TN16 for mitotic cell analysis |
| BSA (bovine serum albumin) | SIGMA-SLDRICH | A7906 | Blocking reagent |
| Triton X-100 | SIGMA-SLDRICH | T8787 | Detergent for permeabilization |
| Paraformaldehyde | SIGMA-SLDRICH | P6148 | Fixation reagant |
| DAPI | SIGMA-SLDRICH | D9542 | For nuclear staining |
| p-phenylenediamine | SIGMA-SLDRICH | P6001 | For mounting medium |
| VWR Micro Slides, Frosted | VWR International | 48312-013 | Micro slides |
| Anti-CENP-A antibody | Stressgen/Enzo Life Sciences | KAM-CC006 | Mouse monoclonal antibody; dilution ratio of 1:100 (IIF), 1:5000 (WB) |
| Anti-CENP-B antibody | Novus Biologicals | H00001059-B01P | Mouse monoclonal antibody; dilution ratio of 1:200 (IIF, methanol/acetone fixation)-1:400 (IIF, paraformaldehyde fixation) |
| Anti-CENP-B antibody | abcam | ab25734 | Rabbit polyclonal antibody; dilution ratio of 1:200 (IIF, methanol/acetone fixation)-1:400 (IIF, paraformaldehyde fixation) |
| Anti-centromere antibody (ACA) | Fitzgerald Industries International, Inc. | 90C-CS1058 | Human centromere antiserum; dilution ratio of 1:2000 (IIF) |
| Anti-CENP-H antibody | Bethyl Laboratories | BL1112 (A400-007A) | Rabbit polyclonal antibody; dilution ratio of 1:200 (IIF) |
| Anti-CENP-H antibody | BD | 612142 | Mouse monoclonal antibody; dilution ratio of 1:200 (IIF) |
| Anti-CENP-I antibody | N/A, Dr. Katusmi Kitagawa | N/A, Dr. Katusmi Kitagawa | Rabbit polyclonal antibody; dilution ratio of 1:1000 (IIF); Niikura et al., Oncogene, 4133-4146 (2006) |
| Anti-KNL1 antibody | Novus Biologicals | NBP1-89223 | Rabbit polyclonal antibody; dilution ratio of 1:100 (IIF) |
| Anti-Hec1 antibody | Novus Biologicals / GeneTex | NB 100-338 / GTX70268 | Mouse monoclonal antibody; dilution ratio of 1:100 (IIF) |
| Anti-Hec1 antibody | GeneTex | GTX110735 | Rabbit polyclonal antibody; dilution ratio of 1:100 (IIF) |
| Anti-Ska1 antibody | abcam | ab46826 | Rabbit polyclonal antibody; dilution ratio of 1:100 (IIF) |
| Anti-Flag antibody | SIGMA-ALDRICH | F3165 | Mouse monoclonal antibody; dilution ratio of 1:1000 (IIF), 1:5000 (WB) |
| Anti-Flag antibody | SIGMA-ALDRICH | F7425 | Rabbit polyclonal antibody; dilution ratio of 1:1000 (IIF), 1:5000 (WB) |
| Anti-CUL4A antibody | N/A, Dr. Pradip Raychaudhuri | N/A, Dr. Pradip Raychaudhuri | Rabbit polyclonal antibody; dilution ratio of 1:3000 (WB); Shiyanov et al., The Journal of biological chemistry, 35309-35312 (1999) |
| Anti-RBX1 antibody | Cell Signaling | 4397 | Rabbit polyclonal antibody; dilution ratio of 1:2000 (WB) |
| Anti-GAPDH antibody | Chemicon | MAB374 | Mouse monoclonal antibody; dilution ratio of 1:5000 (WB) |
| Alexa Fluor 488 Goat Anti-Mouse IgG | Life Technologies/Invitrogen | A11001 | fluorophore-conjugated secondary antibody (Affinity-purified secondary antibody) |
| Alexa Fluor 594 Goat Anti-Mouse IgG | Life Technologies/Invitrogen | A11005 | fluorophore-conjugated secondary antibody (Affinity-purified secondary antibody) |
| Alexa Fluor 488 Goat Anti-Rabbit IgG | Life Technologies/Invitrogen | A11008 | fluorophore-conjugated secondary antibody (Affinity-purified secondary antibody) |
| Alexa Fluor 594 Goat Anti-Rabbit IgG | Life Technologies/Invitrogen | A11012 | fluorophore-conjugated secondary antibody (Affinity-purified secondary antibody) |
| Non fat powdered milk (approved substitution for carnation powdered milk) | Fisher Scientific | NC9255871 (Reorder No. 190915; Lot# 90629) | Skim milk |
| Leica DM IRE2 motorized fluorescence microscope | Leica | motorized fluorescence microscope | |
| HCX PL APO 63x oil immersion lens | Leica | LEICA HCX PL APO NA 1.40 OIL PH 3 CS | 63X oil immersion lens |
| HCX PL APO 100x oil immersion lens | Leica | LEICA HCX PL APO NA 1.40 OIL PHE | 100X oil immersion lens |
| Leica EL6000 compact light source | Leica | External compact light source for fluorescent excitation | |
| ORCA-R2 Digital CCD camera | Hamamatsu | C10600-10B | digital CCD camera |
| Openlab version 5.5.2 Scientific Imaging Software | Perkin Elmer/Improvision | For image observation, acquisition, quantification, and analysis | |
| Velocity version 6.1.1 3D Image Analysis Software | Perkin Elmer/Improvision | For image observation, acquisition, quantification, and analysis | |
| Complete EDTA-free protease inhibitor cocktail | Roche | 11873580001/11836170001 | Protease inhibitor cocktail tablets |
| PlusOne 2-D Quant Kit | Amersham Biosciences | 80-6483-56 | Commercial protein assay reagent I for measurement of protein concentration (compatible with 2% SDS) |
| Bio-Rad Protein Assay | Bio-Rad | 500-0006 | Commercial protein assay reagent II for measurement of protein concentration (compatible with 0.1% SDS) |
| Immobilon-FL | EMD Millipore | IPFL00010 | PVDF membrane for transferring |
| IRDye 800CW Goat Anti-Mouse IgG | LI-COR Biosciences | 926-32210 | IR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:20000 (IIF) |
| IRDye 680 Goat Anti-Rabbit IgG | LI-COR Biosciences | 926-32221 | IR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:20000 (IIF) |
| Goat anti-Mouse IgG DyLight 549 | Fisher Scientific | PI35507 | DyLight-conjugated secondary antibodyIR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:20000 (IIF) |
| Goat anti-Rabbit DyLight 649 | Fisher Scientific | PI35565 | DyLight-conjugated secondary antibodyIR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:20000 (IIF) |
| Goat anti-mouse IgG-HRP | Santa Cruz | SC-2005 | HRP-conjugated secondary antibodyDyLight-conjugated secondary antibodyIR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:10000 (IIF) |
| Goat anti-rabbit IgG-HRP | Santa Cruz | SC-2004 | HRP-conjugated secondary antibodyDyLight-conjugated secondary antibodyIR fluorescent dye-conjugated secondary antibody (Affinity-purified secondary antibody); dilution ratio of 1:10000 (IIF) |
| Openlab version 5.5.2. Scientific Imaging Software | Improvision/PerkinElmer | Software A | |
| Volocity version 6.3 3D Image Analysis Software (Volocity Acquisition) | PerkinElmer | Software B1 | |
| Volocity version 6.3 3D Image Analysis Software (Volocity Quantification) | PerkinElmer | Software B2 | |
| Branson SONIFIER 450 | Sonicator | ||
| Branson Ultrasonics sonicator Microtip Step, Solid, Threaded 9.5 mm | VWR Scientific Products Inc. | 33995-325 | Disruptor horn for sonication |
| Branson Ultrasonics sonicator Microtip Tapered 6.5 mm | VWR Scientific Products Inc. | 33996-185 | Microtip for sonication |
| Odyssey CLx Infrared imaging System | LI-COR Biosciences | Infrared imaging system for immunoblot detection | |
| Image Studio Analysis Software Ver 4.0 | LI-COR Biosciences | Software C | |
| Molecular Imager Versadoc MP4000 System | Bio-Rad | Chemiluminescence imager for immunoblot detection | |
| Quantity One 1-D analysis software | Bio-Rad | Software D | |
| SuperSignal West Femto Maximum Sensitivity Substrate | Thermo | 34095 | Ultra-sensitive enhanced chemiluminescent (ECL) substrate |
References
- DeLuca, J. G., et al. Hec1 and nuf2 are core components of the kinetochore outer plate essential for organizing microtubule attachment sites. Molecular biology of the cell. 16, 519-531 (2005).
- Earnshaw, W. C., Halligan, N., Cooke, C., Rothfield, N. The kinetochore is part of the metaphase chromosome scaffold. J Cell Biol. 98, 352-357 (1984).
- Hoffman, D. B., Pearson, C. G., Yen, T. J., Howell, B. J., Salmon, E. D. Microtubule-dependent changes in assembly of microtubule motor proteins and mitotic spindle checkpoint proteins at PtK1 kinetochores. Molecular biology of the cell. 12, 1995-2009 (2001).
- Regnier, V., et al. CENP-A is required for accurate chromosome segregation and sustained kinetochore association of BubR1. Mol Cell Biol. 25, 3967-3981 (2005).
- Bernad, R., Sanchez, P., Losada, A. Epigenetic specification of centromeres by CENP-A. Exp Cell Res. 315, 3233-3241 (2009).
- Black, B. E., Cleveland, D. W. Epigenetic centromere propagation and the nature of CENP-a nucleosomes. Cell. 144, 471-479 (2011).
- Warburton, P. E., et al. Immunolocalization of CENP-A suggests a distinct nucleosome structure at the inner kinetochore plate of active centromeres. Curr Biol. 7, 901-904 (1997).
- Karpen, G. H., Allshire, R. C. The case for epigenetic effects on centromere identity and function. Trends Genet. 13, 489-496 (1997).
- Black, B. E., et al. Structural determinants for generating centromeric chromatin. Nature. 430, 578-582 (2004).
- Black, B. E., et al. Centromere identity maintained by nucleosomes assembled with histone H3 containing the CENP-A targeting domain. Mol Cell. 25, 309-322 (2007).
- Fachinetti, D., et al. A two-step mechanism for epigenetic specification of centromere identity and function. Nat Cell Biol. 15, 1056-1066 (2013).
- Zeitlin, S. G., Shelby, R. D., Sullivan, K. F. CENP-A is phosphorylated by Aurora B kinase and plays an unexpected role in completion of cytokinesis. The Journal of cell biology. 155, 1147-1157 (2001).
- Zhang, X., Li, X., Marshall, J. B., Zhong, C. X., Dawe, R. K. Phosphoserines on maize CENTROMERIC HISTONE H3 and histone H3 demarcate the centromere and pericentromere during chromosome segregation. The Plant cell. 17, 572-583 (2005).
- Goutte-Gattat, D., et al. Phosphorylation of the CENP-A amino-terminus in mitotic centromeric chromatin is required for kinetochore function. Proc Natl Acad Sci U S A. 110, 8579-8584 (2013).
- Bailey, A. O., et al. Posttranslational modification of CENP-A influences the conformation of centromeric chromatin. Proc Natl Acad Sci U S A. 110, 11827-11832 (2013).
- Samel, A., Cuomo, A., Bonaldi, T., Ehrenhofer-Murray, A. E. Methylation of CenH3 arginine 37 regulates kinetochore integrity and chromosome segregation. Proc Natl Acad Sci U S A. 109, 9029-9034 (2012).
- Niikura, Y., et al. CENP-A K124 Ubiquitylation Is Required for CENP-A Deposition at the Centromere. Dev Cell. , (2015).
- Chan, G. K., Liu, S. T., Yen, T. J. Kinetochore structure and function. Trends in cell biology. 15, 589-598 (2005).
- Hori, T., Okada, M., Maenaka, K., Fukagawa, T. CENP-O class proteins form a stable complex and are required for proper kinetochore function. Molecular biology of the cell. 19, 843-854 (2008).
- Fukagawa, T., Earnshaw, W. C. The centromere: chromatin foundation for the kinetochore machinery. Developmental cell. 30, 496-508 (2014).
- Kedersha, N., Grainger, D. . The Proteintech Blog.Proteintech. , (2012).
- Clontech Laboratories, Inc. . HeLa Tet-Off Advanced Cell Line. , (2012).
- Meraldi, P., Sorger, P. K. A dual role for Bub1 in the spindle checkpoint and chromosome congression. EMBO J. 24, 1621-1633 (2005).
- Niikura, Y., et al. 17-AAG, an Hsp90 inhibitor, causes kinetochore defects: a novel mechanism by which 17-AAG inhibits cell proliferation. Oncogene. 25, 4133-4146 (2006).
- Yang, Z., et al. Silencing mitosin induces misaligned chromosomes, premature chromosome decondensation before anaphase onset, and mitotic cell death. Mol Cell Biol. 25, 4062-4074 (2005).
- Wang, H., et al. Histone H3 and H4 ubiquitylation by the CUL4-DDB-ROC1 ubiquitin ligase facilitates cellular response to DNA damage. Mol Cell. 22, 383-394 (2006).
- Lamb, J. R., Tugendreich, S., Hieter, P. Tetratrico peptide repeat interactions: to TPR or not to TPR?. Trends Biochem Sci. 20, 257-259 (1995).
- Kitagawa, K., Skowyra, D., Elledge, S. J., Harper, J. W., Hieter, P. SGT1 encodes an essential component of the yeast kinetochore assembly pathway and a novel subunit of the SCF ubiquitin ligase complex. Mol Cell. 4, 21-33 (1999).
- Niikura, Y., Dixit, A., Scott, R., Perkins, G., Kitagawa, K. BUB1 mediation of caspase-independent mitotic death determines cell fate. J Cell Biol. 178, 283-296 (2007).
- Niikura, Y., Kitagawa, K. Identification of a novel splice variant: human SGT1B (SUGT1B). DNA Seq. 14, 436-441 (2003).
- Niikura, Y., Ogi, H., Kikuchi, K., Kitagawa, K. BUB3 that dissociates from BUB1 activates caspase-independent mitotic death (CIMD). Cell Death Differ. 17, 1011-1024 (2010).
- Ando, S., Yang, H., Nozaki, N., Okazaki, T., Yoda, K. CENP-A, -B, and -C chromatin complex that contains the I-type alpha-satellite array constitutes the prekinetochore in HeLa cells. Mol Cell Biol. 22, 2229-2241 (2002).
- Izuta, H., et al. Comprehensive analysis of the ICEN (Interphase Centromere Complex) components enriched in the CENP-A chromatin of human cells. Genes to cells : devoted to molecular & cellular mechanisms. 11, 673-684 (2006).
- Obuse, C., et al. Proteomics analysis of the centromere complex from HeLa interphase cells: UV-damaged DNA binding protein 1 (DDB-1) is a component of the CEN-complex, while BMI-1 is transiently co-localized with the centromeric region in interphase. Genes Cells. 9, 105-120 (2004).
- Merlet, J., Burger, J., Gomes, J. E., Pintard, L. Regulation of cullin-RING E3 ubiquitin-ligases by neddylation and dimerization. Cell Mol Life Sci. 66, 1924-1938 (2009).
- Bennett, E. J., Rush, J., Gygi, S. P., Harper, J. W. Dynamics of cullin-RING ubiquitin ligase network revealed by systematic quantitative proteomics. Cell. 143, 951-965 (2010).
- Antonelli, A., et al. Efficient inhibition of macrophage TNF-alpha production upon targeted delivery of K48R ubiquitin. Br J Haematol. 104, 475-481 (1999).
- Codomo, C. A., Furuyama, T., Henikoff, S. CENP-A octamers do not confer a reduction in nucleosome height by AFM. Nat Struct Mol Biol. 21, 4-5 (2014).
- Thrower, J. S., Hoffman, L., Rechsteiner, M., Pickart, C. M. Recognition of the polyubiquitin proteolytic signal. The EMBO journal. 19, 94-102 (2000).
- Yoda, K., et al. Human centromere protein A (CENP-A) can replace histone H3 in nucleosome reconstitution in vitro. Proc Natl Acad Sci U S A. 97, 7266-7271 (2000).
- Shelby, R. D., Vafa, O., Sullivan, K. F. Assembly of CENP-A into centromeric chromatin requires a cooperative array of nucleosomal DNA contact sites. J Cell Biol. 136, 501-513 (1997).
- Majumder, S., Fisk, H. A. Quantitative immunofluorescence assay to measure the variation in protein levels at centrosomes. J Vis Exp. , (2014).
- Masumoto, H., Masukata, H., Muro, Y., Nozaki, N., Okazaki, T. A human centromere antigen (CENP-B) interacts with a short specific sequence in alphoid DNA, a human centromeric satellite. J Cell Biol. 109, 1963-1973 (1989).
- Yoda, K., Kitagawa, K., Masumoto, H., Muro, Y., Okazaki, T. A human centromere protein, CENP-B, has a DNA binding domain containing four potential alpha helices at the NH2 terminus, which is separable from dimerizing activity. J Cell Biol. 119, 1413-1427 (1992).
- Sugata, N., et al. Human CENP-H multimers colocalize with CENP-A and CENP-C at active centromere--kinetochore complexes. Hum Mol Genet. 9, 2919-2926 (2000).
- Earnshaw, W. C., Ratrie, H., Stetten, G. Visualization of centromere proteins CENP-B and CENP-C on a stable dicentric chromosome in cytological spreads. Chromosoma. 98, 1-12 (1989).
- Goshima, G., Kiyomitsu, T., Yoda, K., Yanagida, M. Human centromere chromatin protein hMis12, essential for equal segregation, is independent of CENP-A loading pathway. J Cell Biol. 160, 25-39 (2003).
- Liu, S. T., Rattner, J. B., Jablonski, S. A., Yen, T. J. Mapping the assembly pathways that specify formation of the trilaminar kinetochore plates in human cells. J Cell Biol. 175, 41-53 (2006).
- Gascoigne, K. E., Cheeseman, I. M. T time for point centromeres. Nat Cell Biol. 14, 559-561 (2012).
- Gascoigne, K. E., et al. Induced ectopic kinetochore assembly bypasses the requirement for CENP-A nucleosomes. Cell. 145, 410-422 (2011).
- Nishino, T., et al. CENP-T provides a structural platform for outer kinetochore assembly. EMBO J. 32, 424-436 (2013).
- Malvezzi, F., et al. A structural basis for kinetochore recruitment of the Ndc80 complex via two distinct centromere receptors. EMBO J. 32, 409-423 (2013).
- Schleiffer, A., et al. CENP-T proteins are conserved centromere receptors of the Ndc80 complex. Nat Cell Biol. 14, 604-613 (2012).
- Rago, F., Gascoigne, K. E., Cheeseman, I. M. Distinct organization and regulation of the outer kinetochore KMN network downstream of CENP-C and CENP-T. Curr Biol. 25, 671-677 (2015).
- Nishino, T., et al. CENP-T-W-S-X forms a unique centromeric chromatin structure with a histone-like fold. Cell. 148, 487-501 (2012).
- Bodor, D. L., et al. The quantitative architecture of centromeric chromatin. Elife. 3, e02137 (2014).
- Oliver, C., Rapely, R., Walker, J. M. Ch 58. Molecular Biomethods Handbook. , 1063-1079 (2008).
- Terasima, T., Tolmach, L. J. Changes in x-ray sensitivity of HeLa cells during the division cycle. Nature. 190, 1210-1211 (1961).
- Levenson, G. B. R., Vo-Dinh, T. u. a. n. Ch 8. Biomedical Photonics Handbook. , 8-19 (2003).
- Sanderson, J. . Fluorescence bleed-though. , (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved