Method Article
רצף עמוק משוחד של RNA וירוסי ממדגמים קליניים
In This Article
Summary
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Abstract
כאן אנו מתארים פרוטוקול רצף RNA הדור הבא המאפשר דה נובו אסיפות ושיחות גרסת תוך שורה של הגנום נגיפי שנאסף ממקורות קליניים וביולוגיים. השיטה היא משוחדת ואוניברסלית; היא משתמשת פריימרים אקראיים לסינתזת cDNA ואינה דורשת ידע מוקדם של תוכן רצף ויראלי. לפני בניית ספרייה, סלקטיבית RNase H מבוסס עיכול משמש כדי לרוקן RNA רצוי - כולל פולים (ע"ר) מוביל ו- RNA ריבוזומלי - מן מדגם RNA ויראלי. דלדול סלקטיבי משפר הן את איכות הנתונים ומספר ייחודי קורא בספריות רצף RNA ויראלי. יתר על כן, הצעד מבוסס transposase 'tagmentation' משמש בפרוטוקול כפי שהוא מקטין את זמן בניית ספרייה הכולל. הפרוטוקול אפשר רצף עמוק מהיר של מעל 600 לסה דגימות-כולל נגיף האבולה אוסף משני דם ורקמות מבודדות-והוא החלים רחב ללימודים הגנומיקה חיידקים אחרים.
Introduction
רצף הדור הבא של וירוסים ממקורות קליניים יכול להודיע הולכת האפידמיולוגיה של זיהומים, כמו גם רומן תמיכת עזרת אבחון, חיסון ופיתוח טיפולי. סינתזה cDNA באמצעות פריימרים אקראיים אפשרה זיהוי והרכבה של הגנום מ-מדביק שיתוף מסתעף, או אפילו וירוסים רומן 1,2. כמו שיטות משוחדות אחרות, מזהמים לא רצויים לכבוש רצף רב קורא על תוצאותיהן רצף שלילי. מארח פולי RNA המוביל (ע"ר) הם מזהמים באוספי מדגם ויראלי רבים קיימים.
הפרוטוקול מתאר דרך יעילה וחסכונית של הגנום וירוס RNA רצף עמוק מבוסס על-seq רנ"א הכל משוחדת. השיטה משתמשת צעד דלדול סלקטיבית RNase H 3 להסיר ריבוזומלי מארח רצוי ו- RNA המוביל. דלדול סלקטיבי מעשיר עבור תוכן ויראלי (איור 1) ומשפר את האיכות הכוללת של נתוני רצף(איור 2) מדגים קליני. יתר על כן, tagmentation מוחל על הפרוטוקול כמו שהיא מפחיתה באופן משמעותי את זמן בניית ספרייה. שיטות אלו שימשו להפקת מערכי נתונים גדולים במהירות של הגנום וירוס אבולה לסה 2,4,5 וניתן להשתמש בו כדי ללמוד מגוון רחב של וירוסים RNA. לבסוף, הגישה אינה מוגבלת דגימות אנושיות; השירות של דלדול סלקטיבית הודגם על דגימות רקמה שנאספו מכרסמים נגועים לסה ומודלי מחל הפרימטים לא אנושי 5,6.

תוכן איור 1. סך RNA משקף העשרת תוכן וירוס לסה שימוש סלקטיבי אזילה. החל תוכן הכולל (קלט RNA) והעשרה של נגיף לסה ייחודי (LASV) קורא (תוכן הספריה) על דלדול rRNA מתשע מבודד קליניים שונים. נתון זה יש הבדל בין תמונה 6 . אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
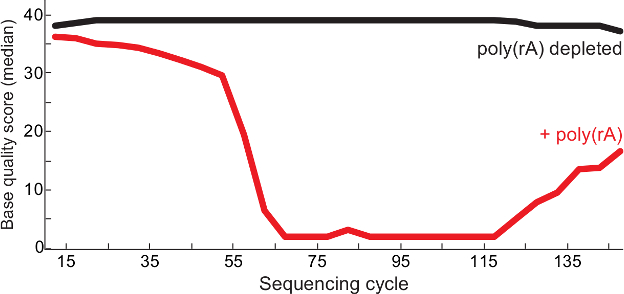
איור 2. איכות גבוהה יותר רצף לאחר Carrier RNA אזילה. איכויות בסיס חציון מחזור רצף של פולי (ע"ר) -contaminated נגיף לסה ספריות (אדום) ובקרה (לא מוביל נצפה בספרייה, שחור) מדווח QC 13. שניהם לקרוא 1 ולקרוא 2 של סוף לזווג קורא ימוזגו בקובץ BAM הספרייה ואת ציוני איכות מוצגים בכל הבסיס. נתון זה יש הבדל בין 6. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
בניית פרטי פרוטוקול RNA-seq ויראלי של ספריות ישירות חילוץRNA שנאסף דגימות קליניות וביולוגיות. כדי להבטיח ביטחונם אישי, כל בסרום ויראלי, דגימות הפלזמה ורקמות צריכות להיות מובטלות מאגרים מתאימים לפני מיצוי RNA. בחלק ערכות איון והפקה, פולי מוביל (ע"ר) RNA כלול; זו תוסר במהלך שלב דלדול סלקטיבית הראשוני RNase H. בהתבסס על התאוששות מלאה, הריכוז הצפוי של RNA המוביל הוא 100 ng / μl. בפרוטוקול, 110 ng / אוליגו μl RNA dT (ריכוז המוביל 1.1x) משמש דלדול. אם פולי (ע"ר) ספק אינו נוכחי במדגם, אז אוליגו (DT) לא צריך להתווסף לפני הדלדול.
הפרוטוקול הבא מיועד 24 תגובות בפורמט צלחת PCR (עד 250 נפח μl). גרסה קודמת של פרוטוקול כך מדווחת Matranga, et al. 6.
Protocol
הצהרה אתיקה: חולי קדחת לסה גויסו למחקר זה באמצעות הפרוטוקולים שאושרו על ידי ועדות בבני אדם ב אוניברסיטת טוליין, אוניברסיטת הרווארד, Broad Institute, בית חולים אוניברסיטאי מומחה Irrua (ISTH), בית חולים ממשלתי Kenema (KGH), אויו המדינה משרד הבריאות, Ibadan , ניגריה משרד סיירה לאונה הבריאות. כל החולים טופלו עם תקן דומה של טיפול שהוצעו התרופה Ribavirin, אם הם החליטו להשתתף במחקר. עבור קדחת לסה (LF) חולים, טיפול עם Ribavirin בעקבות ההנחיות המומלצות כיום הוצע כלל בהקדם LF היה חשד מוצק.
בשל ההתפרצות החמורה עבור אבולה וירוס המחלות (EVD), חולים לא ניתן הסכימו באמצעות הפרוטוקולים הסטנדרטיים שלנו. במקום שימוש במדגמים קליניים עודף מחולים EVD הוערך ואושרה על ידי לוחות סקירה מוסדיים בסיירה ליאון באוניברסיטת הרווארד. במשרהce של סקירה אתיקה מדעית סיירה לאונה הוועדה, משרד סיירה לאונה הבריאות והתברואה, וועדת הרווארד על השימוש בבני אדם העניקו ויתור על הסכמה שממפה להעמיד לרשות רצפים ויראלי בפומבי המתקבל דגימות המטופל ומגע שנאספו במהלך התפרצות אבולה ב סיירה לאונה. גופים אלו כמובן מאליו גם שימוש בנתונים קליניים ואפידמיולוגיים, דגימות-מזוהה דה שנאספו מכל חולי EVD חשד טיפול במהלך התגובה הפרוצה. משרד סיירה לאונה הבריאות ותברואה גם אישרה משלוחי שאינם מדבקים, שאינם ביולוגיים דגימות מסיירה לאון למכון ברוד ואוניברסיטת הרווארד ללימודי הגנומי של דגימות פרוץ.
1. DNase-טיפול של RNA לדוגמא (עד 55 μl שחולצו RNA סה"כ, ~ 4 שעות)
- הגדר את התגובה DNase בצלחת 96-היטב PCR על הקרח בתוך ארון בטיחות ביולוגית כמתואר בטבלה 1 , שלב 1.1 (הנפח הכולל, 70 μl / טוב). הערה: תערובת אמן יכולה להיות מוכנה.
- וורטקס בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות.
- לדגור על 37 מעלות צלזיוס למשך 30 דקות.
- ניקוי באמצעות RNA מוצק שלב הפיך קיבוע (SPRI) חרוזים.
- חרוזי RNA חמים RT למשך 30 דקות.
- נער בעדינות RNA חרוזי בקבוק resuspend כל חלקיקים מגנטיים שייתכן התיישבו. הוספת נפח 1.8x (126 μl) של חרוזים RNA ל RNA שטופלו DNase (70 μl), ומערבבים על ידי פיפטה 10 פעמים דגירה במשך 5 דקות ב RT (הנפח הכולל גם, 196 μl).
- מניחים לתערובת בתחנת מגנטי. חכה הפתרון לנקות (5 - 10 דקות).
- סור פתרון פינה ואילו בתחנה ידי פיפטה וזורק. בעוד על תחנה, לשטוף חרוזים על ידי כיסוי גלול עם אתנול 70% ו דגירת דקות 1. הסר אתנול עם פיפטה וזורק. חזור עבור סכום כולל של שני שוטף.
הערה: שימוש טרי 70% בדיוק et מוכןhanol הוא קריטי, כמו אחוז גבוה יותר יגרום כביסה יעילה של מולקולות קטנות יותר, ואילו <70% אתנול יכול לגרום לאובדן מדגם 7. - שמור הצליחו על התחנה ולהשאיר פתוח-אוויר יבש. הערה: הקפד לאפשר חרוזים להתייבש לחלוטין עד חרוזים להתחיל לפצח.
- הוסף 55 μl מים nuclease חינם לצלחת כדי elute רנ"א. הסר את הצלחת מהתחנה לערבב את החרוזים ומים ידי pipetting ביסודיות. הערה: לחילופין, להשתמש פחות מים (≤ 10 μl) על מנת לרכז את RNA מוחלט.
- מניחים צלחת בחזרה על התחנה. חכה עד פתרון מנקה להעביר על ידי פיפטה צינור בורג מכסה חדש עבור אחסון לטווח ארוך (-80 ° C). מניחים 5 RNA μl בניו 96-גם צלחת PCR עבור דלדול (שלב. 2.4).
- אופציונלי:. שמור לדלל 1 μl ב 19 מים μl (1:20) עבור qRT-PCR של rRNA (למשל, 18S, 28S rRNA) (לוח 2) וסמנים ויראלי 5
2. דלדול סלקטיבי של RNA ריבוזומלי והספק ממדגם RNA ויראלי (~ 4 שעות)
- הפוך הכלאה 5x ו 10x מאגרים התגובה RNase H, ו nuclease ללא מים עם הספק acrylamide ליניארית כמתואר בטבלה 1.
- הגדרת התגובה הכלאה ידי שילוב RNA עם oligos דלדול rRNA (טבלה 3) ו אוליגו (DT) על קרח בצלחת 96-היטב PCR כמתואר בטבלה 1.
הערה: תערובת אמן יכולה להיות מוכנה. 50 femtograms (FG) של רנ"א ייחודי (ERCCs 8) ניתן להוסיף למעקב הן את תהליך רצף ויראלי זיהום צולב מדד פוטנציאל לקרוא.- וורטקס בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות.
לדגור על 95 מעלות צלזיוס למשך 2 דקות, ramping האיטי עד 45 מעלות צלזיוס ב -0.1 מעלות צלזיוס לכל שניות. השהה את thermocycler על 45 מעלות צלזיוס.
- וורטקס בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות.
- הגדרת תערובת התגובה RNase H על הקרח כמו לתארד בטבלה 1, אז מחממים על 45 מעלות צלזיוס למשך 2 דקות. הערה: תערובת אמן יכולה להיות מוכנה.
- מוסיף את התערובת המחוממת מראש RNase H תגובת ההכלאה בצלחת תוך שמירה על צלחת thermocycler ב 45 מעלות צלזיוס.
- מערבבים היטב על ידי pipetting עדין 6 - 8 פעמים. לדגור על 45 מעלות צלזיוס במשך 30 דקות אחרות. מניחים על קרח.
- הגדר את תערובת התגובה DNase על הקרח כמתואר בטבלה 1 הערה:. תערובת אמן יכול להיות מוכן.
- הוסף את התגובה H RNase בצלחת, מערבולת בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות. לדגור על 37 מעלות צלזיוס למשך 30 דקות.
- בקצות DNase ידי הוספת 5 μl 0.5 M EDTA. וורטקס בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות.
- ניקוי באמצעות חרוזים RNA (ראה שלב 1.3) באמצעות נפח 1.8x (144 μl) חרוזים. Elute ב 11 μl של מים nuclease חינם. הערה: בקירור בטוח, חנות מדולדלת RNדגימה ב -80 ° C O / N.
3. סינתזה cDNA (~ 6 שעות)
- מערבבים rRNA / RNA מדולדל המוביל עם פריימרים אקראיים על קרח בצלחת 96-היטב PCR כמתואר בטבלה 1, מערבולת בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות.
- מחממים את התערובת עד 70 מעלות צלזיוס למשך 10 דקות ב thermocycler. מיד לאחר denaturation חום, למקם את הרנ"א על קרח 1 - 5 דקות. אל תאפשרו RNA לעמוד (אפילו על הקרח) למשך זמן ארוך יותר מ -5 דקות לפני התגובה הראשונה גדיל.
- הגדרת גדיל הראשונה תערובת התגובה סינתזה על הקרח כמתואר בטבלה 1.
הערה: לערבב מנהל עשוי להיות מוכן.- הוסף RNA / תערובת פריימר אקראית בצלחת, מערבולת בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות. לדגור על 22 - C ° 25 במשך 10 דקות.
- לדגור על 55 מעלות צלזיוס באינקובטור באוויר במשך 60 דקות. מניחים את הצלחת על הקרח כדי לסיים את התגובה. לֹאדואר: השימוש באינקובטור אוויר מומלץ ליצור התחממות הדרגתית של התגובה הראשונה גדיל שבמהלכו לחשל פריימרים ואת הגדיל הראשון מתחיל להאריך.
- הגדרת תערובת תגובת הסינתזה שניית גדיל על קרח כמתואר בטבלה 1.
הערה: לערבב מנהל עשוי להיות מוכן.- הוסף סינתזת התגובה הראשונה גדיל בצלחת, מערבולת בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות. דגירה עבור שעה 2 ב 16 ° C (לשמור מכסה של 25 מעלות צלזיוס). אל תאפשר לטמפרטורה לעלות מעל 16 מעלות צלזיוס.
- מניחים את הצלחת על הקרח, אז להשבית התגובה על ידי הוספת 5 μl של 0.5 EDTA M, ומערבבים בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות.
- ניקוי עם חרוזים DNA (ראה שלב 1.3 עבור פרוטוקול) באמצעות נפח 1.8x (153 μl) של חרוזים. Elute ב 9 μl של חיץ elution (EB). שמור 1 μl כימות. השתמש 1 ננוגרם של cDNA עבור subsequeצעדי NT. אם ריכוז cDNA הוא נמוך מדי כדי לזהות, השתמש 4 μl של cDNA עבור tagmentation (ראה שלב 4.1).
- לקבלת בקירור בטוח, לאחסן פעמיים תקועים cDNA ב 4 ° CO / N או -20 ° C עבור אחסון לטווח ארוך.
4. הכנת הספרייה - בניית ספריית DNA (~ 4 שעות)
- העברת 4 μl של cDNA לצלחת 96-היטב ולשמור את cDNA הנותר עבור ניסיון שני במידת הצורך.
- הגדר את התגובה tagmentation על הקרח כמתואר בטבלה 1.
הערה: לערבב מנהל עשוי להיות מוכן. כדי להפחית את הרקע ואת העלות הכוללת, את הנפח הכולל של התגובה tagmentation מצטמצם מ -20 ל -10 μl. כפי cDNA הוא הגורם המגביל, בסך של ATM (כלומר, transposome) המשמש התגובה מצטמצם גם כדי להקטין את מספר אתרי אינטגרציה.- להוסיף תערובת tagmentation כדי cDNA בצלחת, מערבולת בעדינות וביסודיות צנטריפוגות ב 280 XG (ב RT) עבור 1 דקות.לדגור על 55 מעלות צלזיוס למשך 5 דקות, להחזיק ב 10 ° C.
- פעם ב 10 ° C, מיד להוסיף 2.5 μl של לנטרל Tagment הצפת (NT) לסיים את התגובה. מערבבים על ידי pipetting למעלה ולמטה, ואז צנטריפוגות ב 280 XG (ב RT) עבור 1 דקות.
- לדגור על RT במשך 5 דקות.
- הגדרת תגובת הגברת PCR על קרח כמתואר בטבלה 1.
- וורטקס בעדינות וביסודיות, אז צנטריפוגות ב 280 XG ב RT עבור 1 דקות.
- בצע PCR על thermocycler באמצעות בתנאים המתוארים בטבלה 1.
הערה: 12 מחזורים של PCR הם הציעו עבור 1 ננוגרם של cDNA tagmented; עם זאת, דגימות קליניות ויראלי לעתים קרובות יש כמויות בלתי מורגשות של cDNA. עבור סכומים נמוכים של cDNA (<1 נ"ג), השתמש עד 18 מחזורים של PCR כדי ליצור ספריית מספיק עבור סידור.
- ניקוי ואיגום עבור סידור - הכנה הספרייה
- תביא דגימה של עד 50 μl עם EB.
- ניקוי עםחרוזי DNA (ראה שלב 1.3 עבור פרוטוקול) באמצעות נפח 0.6x חרוזים (30 μl). Elute ב 15 μl EB.
- קביעת ריכוז של הספרייה (איור 3) על ידי ביצוע ניתוח באזור (150 עד 1,000 נ"ב) באמצעות bioanalyzer התוכנה 9, למעט הדימרים פריימר (~ 120 נ"ב) מניתוח באזור. הערה: לחילופין, qPCR יכול לשמש כדי לכמת ספריות 10.
- ספריות ברכה בריכוז הטוחן הנמוך ביותר של 1 ננומטר או יותר. אם ספרייה היא מתחת ל -1 ננומטר, להוסיף נפח קטן של ספריית ברכה (~ נפח 1x של ספריות אחרות) כדי ללכוד מידע רצף מספריות אלה.
- ניקוי בריכה עם חרוזים DNA 0.7x כפי שתואר לעיל (ראה שלב 2). Elute ב 15 μl EB. הערה: נפח של חרוזים יהיה תלוי בנפח הסופי של הברכה.
- לנתח ברכה 9. קביעת ריכוז טוחנת ידי ביצוע ניתוח באזור (150 עד 1,000 נ"ב) 9. הערה: לחילופין, qPCR יכול לשמש כדי לכמת בריכת הספרייה 10 </ Sup>.
- ברצף טען בריכוז הספרייה 10 בערב עד לייצר 101 נ"ב, לזווג סוף קורא עם ברקוד כפול קורא 11.
איור 3. נבנו ספריות ממדגמים קליניים וירוס האבולה. תמונה ג'ל של 4 וירוס האבולה יציג ספריות (EBOV). אזורים של הדימרים ספרייה פריימר מוצגים. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
תוצאות
הפרוטוקול המתואר מאפשר את הדור של רצף באיכות גבוהה קורא ממדגם רנ"א נגיפיים נמוך קלט תוך העשרת תוכן ויראלי ייחודי. כפי שניתן לראות בתרשים 1, הפרוטוקול מועשר תוכן לסה וירוס ייחודי לפחות פי חמש בכל הדגימות (בהשוואה לקבוצת ביקורת הלא-מדולדל) עם לפחות ממיליון עותקים של 18S rRNA (~ 100 pg RNA סה"כ). כמו כן, הצלחת רצף מתואמת גם עם כמות הנגיף בתוך מדגם נתון. שימוש qRT-PCR כתחליף כמות ויראלי, דגימות שהכילו ~ 1,000 או יותר עותקים הגנום נגיפיים לרוב נוצרו מכלולים מלאים (מידע לא מוצג). יתר על כן, דלדול של פולי (ע"ר) מוביל מפחית רצפי homopolymer של A ו- T בספריות, וכתוצאה מכך הכנות מנקות והבטחת רצף באיכות טובה יותר קוראת (איור 2). ספריות סופיות ממדגמים קליניים ויראלי קלט נמוך בדרך כלל יש אורך קטע רחב בין 150 ל -1,000 נקודות בסיס(איור 3).
לאחר רצף, כדי להפחית מדגם מזיהוי שגוי ו crosstalk בין ספריות בתוך ברכה 12, רק מדד קורא עם ציון איכות בסיס של 25 (שאלה 25) ולהבטיח אפס התאמות נשמרות בתהליך demultiplexing. הגנום נגיפי מורכב באמצעות צינור ביואינפורמטיקה ספציפי לאיתור וירוסים מסתעפים 2,4-6. כלים אלה זמינים ב https://github.com/broadinstitute/viral-ngs או באמצעות פלטפורמות ענן מסחריות 4.
| שלב 1.1: תגובת DNase | |
| מֵגִיב | נפח לכל תגובה (μl) |
| חיץ 10x DNase | 7 |
| nuclease ללא מים | 6 |
| רנ"א נגיפי שחולצו | 55 |
| DNase (2 U / & #181; l) | 2 |
| נפח כולל | 70 |
| שלב 2.1: חיץ הכלאה 5x | |
| מֵגִיב | נפח עבור 1 מ"ל (μl) |
| 5 M NaCl | 200 |
| 1 M טריס- HCl (pH 7.4) | 500 |
| nuclease ללא מים | 300 |
| נפח כולל | 1,000 |
| שלב 2.1: 10x RNase H התגובה חיץ | |
| מֵגִיב | נפח עבור 1 מ"ל (μl) |
| 5 M NaCl | 200 |
| 1 M טריס- HCl (pH 7.5) | 500 |
| 1 M MgCl 2 | 200 |
| nuclease ללא מים | 500 |
| נפח כולל | 1,000 |
| שלב 2.1: מי wacrylamide ליניארי ה- i | |
| מֵגִיב | נפח עבור 1 מ"ל חיץ (μl) |
| nuclease ללא מים | 992 |
| acrylamide ליניארית (5 מ"ג / מ"ל) | 8 |
| נפח כולל | 1,000 |
| שלב 2.2: תגובת הכלאת דלדול סלקטיבית | |
| מֵגִיב | נפח לכל תגובה (μl) |
| מאגר הכלאה 5x | 2 |
| rRNA-דלדול תמהיל אוליגו (100 מיקרומטר) | 1.22 |
| אוליגו (ד) T (550 ng / μl) | 1 |
| שטופלו DNase רנ"א הכל | עד 5 |
| ספייק-ב RNA (זה הוא אופציונלי) | 0.5 |
| מים (עם acrylamide ליניארי) | להביא עד 10 סה"כ |
| volu סה"כלִי | 10 |
| שלב 2.3: תגובת H RNase דלדול סלקטיבית | |
| מֵגִיב | נפח לכל תגובה (μl) |
| מאגר התגובה 10x RNase H | 2 |
| מים (עם acrylamide ליניארי) | 5 |
| RNase H Thermostable (5 U / μl) | 3 |
| נפח כולל | 10 |
| שלב 2.4: דלדול סלקטיבית פוסט התגובה DNase | |
| מֵגִיב | נפח לכל תגובה (μl) |
| 10x DNase הצפה | 7.5 |
| מים (עם acrylamide ליניארי) | 44.5 |
| מעכב RNase (20 U / μl) | 1 |
| RNase ללא DNase אני (2.72 U / μl) | 2 |
| נפח כולל (עם תגובת RNase H) | 75 |
| שלב 3.1: סינתזת cDNA, כלאה פריימר אקראי | |
| מֵגִיב | נפח לכל תגובה (μl) |
| rRNA / מדולדל המוביל RNA | 10 |
| 3 מיקרוגרם פריימר אקראי | 1 |
| נפח כולל | 11 |
| שלב 3.2: סינתזת תגובת cDNA גדיל הראשון | |
| מֵגִיב | נפח (μl) |
| מאגר התגובה 5x ראשון סטרנד | 4 |
| 0.1 M DTT | 2 |
| לערבב 10 מ"מ dNTP | 1 |
| מעכב RNase (20 U / μl) | 1 |
| טרנסקריפטאז הפוך (להוסיף אחרון) | 1 |
| נפח כולל (עם RNA לעיל) | 20 |
| שלב 3.3: סינתזת התגובה cDNA גדיל שנית | |
| מֵגִיב | נפח (μl) |
| RNase ללא מים | 43 |
| 10x הצפת התגובה השנייה סטרנד | 8 |
| לערבב 10 מ"מ dNTP | 3 |
| E. coli DNA האנזים (10 U / μl) | 1 |
| E. coli DNA פולימראז אני (10 U / μl) | 4 |
| E. coli RNase H (2 U / μl) | 1 |
| נפח כולל (עם 1 st גדיל תגובה) | 80 |
| שלב 4.2: תגובת Tagmentation | |
| מֵגִיב | נפח (μl) |
| Amplicon Tagment Mix (ATM) | 1 |
| Tagment DNA הצפה (TD) ד> | 5 |
| נפח כולל (עם cDNA) | 10 |
| שלב 4.3: תגובת הספרייה PCR | |
| מֵגִיב | נפח (μl) |
| מיקס מאסטר PCR (NPM) | 7.5 |
| פריימר מדד 1 (i7) | 2.5 |
| פריימר מדד 2 (i5) | 2.5 |
| הנפח הכולל (עם tagmented cDNA) | 25 |
| שלב 4.3.2: תנאי PCR ספרייה | |
| 72 ° C, 3 דקות | |
| 95 מעלות צלזיוס, 30 שניות | |
| עד 18 מחזורי 10 שניות על 95 מעלות צלזיוס, 30 שניות על 55 מעלות צלזיוס, 30 שניות על 72 מעלות צלזיוס | |
| 72 מעלות צלזיוס, 5 דקות | |
| 10 ° C, לנצח | |
FO: keep-together.within-page = "1"> טבלה 1:. עד שנקבע התגובה מאגרים שלב-אחר-צעד שולחנות עם התוכן של כל מאגרים ותערובות התגובה.
טבלה 2: qRT-PCR Primers רצפים Primers המשמש למדידת המארח (18S rRNA) ויראלי (אבולה לסה) תוכן.. 'KGH' הוא בית חולים ממשלתי Kenema בסיירה לאונה, שבו פריימרים אבולה נבדקו 2. 'Kulesh' הוא החוקר שעיצב את פריימר להגדיר 14.
טבלה 3: RNA ריבוזומלי (rRNA) דלדול oligos 195 רצפים ארוכים 50-נוקלאוטיד משלים rRNA האדם דלדול סלקטיבית בשלב 6.. אנא לחץ כאן להורדת הקובץ הזה.
| שם אוליגו | רצף (5 'ל 3') |
| אבולה KGH FW | GTCGTTCCAACAATCGAGCG |
| RV אבולה KGH | CGTCCCGTAGCTTTRGCCAT |
| אבולה KULESH FW | TCTGACATGGATTACCACAAGATC |
| RV אבולה KULESH | GGATGACTCTTTGCCGAACAATC |
| לסה SL FW | CC AAB GTA AGC CCA GCD GYA |
| RV לסה SL | AAG CCA CAG AAA RCT GGS AGC |
| 18S rRNA FW | TCCTTTAACGAGGATCCATTGG |
| RV 18S rRNA | CGAGCTTTTTAACTGCAGCAACT |
Discussion
שהגישה המתוארת מאפשרת חזק, אוניוורסלי, רצף מהיר שמשה רצף נגיף האבולה במהלך 2,4 פרוץ 2014. על ידי צימוד דלדול סלקטיבית סינתזת cDNA עם בניית ספריית tagmentation, הפעם התהליך הכולל הופחת על ידי ~ 2 ימי משיטות קשירת מתאם קודמות. לאחרונה, פרוטוקול זה הועסק על ידי משתפי פעולה בינלאומיים ואחרים בהצלחה רבה 15,16 ו ייפרס מעבדות במערב אפריקה לתמוך מחקרים מבוססי הגנומיקה מקומיים ואבחון 17.
הפרוטוקול המתואר כאן משתמש פריימרים אקראיים להכין cDNA עבור ספריות RNA-seq ויראלי. שלא כמו גישות RNA-seq ויראלי קודמות, זה לא דורש ידע אפריורי של נתוני רצף או משוכלל ונדרש זמן רב עיצוב פריימר עבור וירוס או clade ספציפי. השיטה יכולה להיות מיושמת על כל מדגם רנ"א נגיפי. לדוגמה, שימש אותו כדי ליצור תוכן ויראלי משני אבולהו לסה דגימות 6. הפרוטוקול יכול לשמש גם עבור transcriptomic מארח, פרויקטי רצף metagenomic ו הפתוגן גילוי 1.
שלב קריטי של הפרוטוקול הוא ממוקד עיכול H RNase, א-תפוקה גבוהה, שיטת עלות נמוכה להסרת ספק רצוי ו- RNA מארח ממדגם ויראלי. צעד הדלדול סלקטיבית של הפרוטוקול עושה שימוש מרכיבים רבים ודורש מיומנות ודיוק. זמן טיפול נוסף יש לנקוט במהלך ההתקנה הראשונית.
כמו רוב דגימות סרום ופלזמה קליניות לעתים קרובות יש מעט מאוד חומר חומצות גרעין, זיהום ואובדן מדגם נפוץ. כדי למנוע בעיות אלה, טיפול מיוחד יש לנקוט בעת שימוש בפרוטוקול זה. ראשית, RNA הוא מאוד רגיש שפל; ולכן בכל התחומים צריכים להיות נקיים של nucleases. שנית, לזהות דגימות מתאימות לשימוש בפרוטוקול זה, מבחני qRT-PCR עבור שניהם RNA מארח הווירוס אמורים לשמש עבור כימות 5,6 . בהשוואת קלט מסתכמת עם תוצאות רצף מן הפרוטוקול, הצלחת רצף (כלומר, דור נתונים המספיקים להרכבת ויראלי מלאה) בקורלציה עם דגימות שהכילו לפחות 100 pg RNA סך 1,000 עותקים של נגיף. יש להימנע מחשיפה שלישית, מקורות סביבתיים של חומצות גרעין. הפרוטוקול המתואר כאן נעשה בתוך ארון בטיחות ביולוג הוראות בטיחות להגבלת מזהמים סביבתיים. יתר על כן, הקבוצה ואחרים שלנו הבחינו כי אנזימים מסחריים עשויים להיות מקור נוסף של זיהום חיידקי חומצות גרעין בדגימות קלט נמוך 6,18. שימוש סביבת עבודה נקיה (למשל, מכסה מנוע PCR, ארון בטיחות ביולוגי) ובקרות שליליות (למשל, מים או חיץ) יעזרו להקל ולעקוב אחר זיהום, בהתאמה. לקבלת דוגמיות עם <100 pg של רנ"א הכל, רק פולי RNA המוביל (RA), לא rRNA, צריך להיות מדולדל על מנת להבטיח תוצאות רצף באיכות גבוהה תוך הגבלת הפסד של החומר. לקבלת מאודדגימות קלט נמוכות, שיטות הגברת cDNA יכולות להיות יותר מתאימות 19, למרות פולי (ע"ר) מנשא להסירו לפני סינתזת cDNA.
דלדול של המארח rRNA מעשירה עבור תוכן ויראלי בספריות רצף שלכם והיא חלה על אוספים דגימה שונים כולל סרום או פלזמה, וסוגים שונים של רקמות מן מכרסמים פרימטים לא אנושיים 5,6. באורגניזמים שאינם בני אדם, קריאות יישור כדי 28S rRNA שנותר לאחר דלדול, דבר המצביע 28S rRNA הוא פחות משומר בין בני האדם ומינים אחרים 6,20. בעת השימוש בשיטה זו עם בידודים שאינם בני אדם, זה עשוי להיות נחוץ כדי להשלים עם oligos DNA משלים רצפי rRNA מסתעפים של 3,21 המארח הספציפי.
מאז הפרוטוקול הוא משוחד, ויראלי קורא עשוי לייצג רק חלק קטן של תוכן הספריה. למרות rRNA הוא המין הנפוץ ביותר של רנ"א מארח ורק אחוז קטן של rRNA קורא (0; 1%) נמצאים לאחר דלדול סלקטיבית, כל RNA המארח האחר (למשל, mRNA) יישאר לאחר הדלדול עשוי להסביר רצף רב קורא מן המדגם. לכן "oversampling" (כלומר, oversequencing) ספריות פרט נדרשה על מנת לקבל כיסוי מספיק לשיחות הרכבת גרסת ויראלי. עבור מחקרים שלנו, אנו מנסים רצף ~ 20 מיליון קורא לדגימה יש מספיק עומק לניתוח של וריאנטים גנומי הקשורים ויראלי וכן תכנים metagenomic 2,5. עבור מחקרים גילוי metagenomic ו הפתוגן, חשוב לציין כי זיהום DNA המארח הוא הוסר על ידי מערכת העיכול DNase. לכן נגיפים ופתוגנים אחרים המכילים הגנום DNA עלולים ללכת לאיבוד בתהליך, אולם ביניים RNA עשויים עדיין להיות רצף.
Disclosures
The authors have no competing financial interests.
Acknowledgements
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
Materials
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
References
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved