A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
זרימה מיון ו Exome רצף של תאים ריד סטרנברג של לימפומה קלאסית הודג'קין
In This Article
Summary
כאן, אנו מתארים זרם תא cytometric תזרים מיון נמוך קלט, הדור הבא של ספריית פרוטוקול בנייה שנועד לייצר באיכות גבוהה, נתונים exome מלא של Hodgkin ריד- Sternberg (HRS) תאים של לימפומה הודגקין קלאסית (CHL).
Abstract
תאים Hodgkin Reed-Sternberg של לימפומה הודג 'קין קלאסית מופצים בדלילות ברקע של לימפוציטים דלקתיים בדרך כלל מהווים פחות מ 1% של מסת הגידול. חומר נגזר הגידול בתכולה מכיל תוכן הגידול על ריכוז מספיק לאפיון. לכן, פלואורסצנטי מופעלת תא מיון באמצעות שמונה נוגדנים, כמו גם בצד ופיזור קדימה, מתואר כאן כשיטה של הפרדה במהירות וריכוז עם אלפי טוהר גבוה של תאים HRS מן הגידול למחקר הבא. במקביל, בגלל פרוטוקולים סטנדרטיים עבור רצף exome בדרך כלל דורשים 100-1,000 ng של קלט DNA, אשר לעתים קרובות גבוה מדי, אפילו עם מיון זרימה, אנו מספקים גם אופטימיזציה, נמוך קלט ספריה פרוטוקול מסוגל לייצר באיכות גבוהה נתונים של פחות מ 10 ng של קלט DNA. שילוב זה מסוגל לייצר את הדור הבא ספריות מתאים ללכידת הכלאה של כל eXome baits או ממוקד יותר ממוקד לוחות, לפי הצורך. Exome רצף של תאים HRS, כאשר לעומת intratumor בריאים T או B תאים, יכול לזהות שינויים סומטיים, כולל מוטציות, הוספות ומחיקות, ושינויים מספר עותק. ממצאים אלה מבהירים את הביולוגיה המולקולרית של תאי ה- HRS ועשויים לחשוף אפיקים לטיפולים ממוקדים.
Introduction
התקדמות בגנום הסרטן כתוצאה מרצף הדור הבא הובילה לפריצות דרך משמעותיות בזיהוי מטרות טיפוליות ובפרוגנוזה עבור גידולים המטולוגיים ולא המטולוגיים רבים. אסטרטגיות טיפול ייחודיות חדשות, המבוססות על שינויים גנומיים ספציפיים, מועברות במהירות לסוגים רבים של גידולים (נבדקו בהמלצות 1 , 2 ). למרות התקדמות משמעותית בגנומיקה הלימפומה, הגנום של תאי ה- HRS הניאופלסטיים בלימפומה קלאסית של הודג'קין (CHL) לא אובחן. החקירות כבר הקשו על ידי מחסור של תאים ניאופלסטיים HRS בתוך microenvironment תגובתי, מה שהופך אותו קשה לבודד מטוהרים אוכלוסיות תאים HRS 3 .
השיטה לבידוד תאי HRS בת קיימא מגידולים ראשוניים פותחה על ידי פרום ואחרים. 4 ,5 , 6. השיטה משתמשת בקוקטייל של שמונה נוגדן, המורכב מתקליטורים מסוג CD30, CD15, CD40, CD95, CD45 CD20, CD5 ו- CD64, כדי לזהות באופן חד משמעי תאי HRS מתליית גידול של CHL. מסוגלים לבודד לפחות 1,000 תאים בת קיימא HRS מן המתלים טריים או קפואים התא מ ביופסיות הגידול המורכב של לפחות 10 7 תאים (כ 10 מ"ג של רקמה) .הטוהר הוא גדול מ -90% על ידי זרימת cytometric זרימה מוערך להיות לפחות 80% על ידי ניתוח גנומי exome של עשרה מקרים רצופים.
יש לנו מעודן הזרימה cytometric תא בידוד טכניקה כי יש להקל מאוד את התהליך, המאפשר בידוד מהיר של אלפי תאים HRS קיימא של גידולי CHL העיקרי 7 . השתמשנו בטכניקה כדי לייצר את מה שהוא האמין להיות רצף שלם הראשון exome של תאים סרטניים במקרים העיקריים של לימפומה הודג'קין. המחקרים שלנו מדגימיםE היתכנות של תפוקה גבוהה, גנום רחב מחקרים של מקרים CHL הפרט כבר הובילו לזיהוי של שינויים גנומי חדש עם פוטנציאל להסביר היבטים של פתוגנזה CHL.
אנחנו עוד פיתחה צינור כדי לנצל את ה- DNA שחולצו עבור תפוקה גבוהה מחקרים גנומיים. על מנת להשיג תוצאות אמינות מעטים כמו 1,000 תאים HRS מיון (מינימום המתקבל מקרים רציף), אנו עוד פיתחו שינוי הדור הבא של דנ"א בניית הספרייה 8 שאיפשר לנו להגדיל את קשירת קשירת יעילות וליצור ספריות שבר DNA ללא הגברה מופרזת. שיטה זו מאפשרת ניתוח של דגימות קליניות שגרתיות וזיהוי של מוטציות חוזרות ושינויים כרומוזומליים 7 .
Access restricted. Please log in or start a trial to view this content.
Protocol
1. עיבוד רקמות והקפאה
- איסוף רקמת הצומת הלימפה פוספט שנאגרו מלוחים (PBS) או Roswell פארק הזיכרון בינוני (RPMI) בינוני בתהליך בתוך 24 שעות של אוסף. העברה נחקרת רקמת הצומת הלימפה 9 לצלחת פטרי המכיל 10 מ"ל של RPMI עם 2% עגל בסרום בסרום (FCS) ו דק דק את זה עם להב אזמל טרי. השתמש האחורי של 10 מ"ל הבוכנה הבוכנה כדי לטחון נוסף / לנתק את הרקמה.
- מעבירים את הנוזל לצינור חרוטי 50 מ"ל דרך מסננת 100 מיקרומטר התא. שוטפים את צלחת פטרי ואת המסנן עם 10 מ"ל נוספת של FCMI 2 RPMI.
- השג ספירת תאים באמצעות מונה תאים אוטומטי או hemocytometer.
הערה: בדרך כלל, לפחות 2 x 10 7 תאים יהיה צפוי מ כ 5 מ"מ 3 של רקמת הצומת לימפה CHL. הכדאיות של מעל 80% צפוי, אבל זה עשוי להשתנות לפי המדגם. - ספין את התאים ב XG 400 במשך 10 דקות ו aspiraTe supernatant. מניחים את הצינור המכיל תא גלולה על הקרח.
- טרום צמרמורת הקפאת בינוני על הקרח. Resuspend התאים במדיום הקפאה קר בריכוז של 2 x 10 7 / mL ו resuspend אותו pipetting. אל מערבולת. דגירה ההשעיה על הקרח במשך 10 דקות.
- Aliquot המדגם 1 מ"ל / בקבוקון cryo (מראש צונן על הקרח). מעבירים את צלוחיות למקפיא -80 מעלות צלזיוס במשך 1-2 ימים ולהעביר אותם שוב חנקן נוזלי.
הכנת תרחישי תא למיון תאים
הערה: כל הרבה נוגדן חייב להיות titered כראוי באמצעות 10 מיליון תאים בהיקף מכתים 300- μΛ. דם היקפי ניתן להשתמש עבור כל הנוגדנים למעט CD30, שעבורו קו תאים KMH2 spiked לתוך הדם ההיקפי עשוי לשמש טיטרציה 10 . אנחנו בדרך כלל מתחילים עם נפח היצרן המומלץ של נוגדנים ולבצע שני דילולים כפולה פי שניים עלייה פי שניים (ארבע נקודות נתונים)עבור כל מידה של טיטרציה נוגדנים. לדוגמה, אם היצרן ממליץ על נפח 10 μL, אנו מבצעים את טיטרציה באמצעות 2, 5, 10, ו 20 כרכים μL.
- הגדר אמבט מים ל 37 מעלות צלזיוס. Premix כמות titered של נוגדנים בבקבוקון זכוכית כהה ולהוסיף PBS + 2% BSA עבור נפח קוקטייל הכולל של 100 μL.
הערה: למרות שאנו ממליצים titering נוגדנים, הכרכים הבאים עשויים לשמש כנקודת התחלה: CD64, 20 μL; CD95, 5 μL; CD30, 20 μL; CD5, 10 μL; CD20, 10 μL; CD15, 20 μL; CD40, 5 μL; ו CD45, 10 μL. - מעבירים את הבקבוקון מחנקן נוזלי לתוך דלי קרח המכיל קרח יבש כדי למנוע הפשרה. טרום חם 50 מ"ל של המדיום מופשר המכיל RPMI / 20% FCS / DNase (100 מיקרוגרם / מ"ל) בצינור חרוטי 50 מ"ל אמבטיה מים 37 מעלות צלזיוס.
- העברת 45 מ"ל של המדיום מופשר צינור חדש ולשמור אותו על 37 מעלות צלזיוס. להפשיר במהירות את התאים על ידי החזקת בקבוקון cryogenic ב 37 מעלות צלזיוס מיםH עד רק חלק קטן מאוד קפוא נשאר.
- יוצקים את תכולת הבקבוקון לתוך הצינור המכיל 45 מ"ל של המדיום הפשרה. שוטפים את בקבוקון cryogenic ריק 2 פעמים עם 1 מ"ל של המדיום מפשיר ולשלב את שטיפות.
- לדגור את התאים בטמפרטורת החדר למשך 15 דקות כדי לאפשר עיכול DNase תא equilibration מחדש. ספין את התאים ב XG 500 למשך 10 דקות ו לשאוב supernatant.
- Resuspend התאים μL כ 200 של המדיום מופשר שנערך בחזרה (5 מ"ל) ולתת לו לאזן לטמפרטורת החדר למשך 2-3 דקות. השחזור של 70% של תאים קיימא קיימא צפוי.
הערה: עבור טוהר גדול יותר של תאים HRS ממוינים שעשויים להיות rosetted על ידי תאי T המצורפים, קוקטייל של נוגדנים ללא תווית ניתן להוסיף בשלב זה (עיין בפרוטוקול אופציונלי). - הוסף 100 μL של קוקטייל נוגדן ו דגירה במשך 15 דקות בטמפרטורת החדר (RT), מוגן מפני האור. הוסף 3 מ"ל של המדיום מיון, לסובב את התאים ב 500 xgבמשך 10 דקות, וכן לשאוב supernatant.
- Resuspend התאים 1 מ"ל של המדיום מיון ולהעביר אותם ל 5 מ"ל צינור הזרימה העליון מסננת.
- שוטפים גם את צינור חרוטי 50 מ"ל מסננת התא עם 1 מ"ל נוסף של מיון בינוני ותאי המקום על הקרח.
3. (פרוטוקול אופציונלי) T Cell רוזטה חסימה
הערה: תאים HRS הם rosetted על ידי תאים T בסעיפים רקמה ההשעיה התא, ותאי אלה T עשוי לזיהום את החלק HRS מיון. אינטראקציות אלה מתווכות על ידי CD54 ו CD58 על התא HRS מחייב LFA-1 ו CD2 על תאי 4 , 11 . אינטראקציות אלה ניתן לחסום עם נוגדנים ללא תווית מולקולות אלה הידבקות.
- Aliquot 100,000 ל 500,000 תאים μL 100 של RPMI.
- דגירה ההשעיה התא עם נוגדנים ללא תווית CD2, CD54, CD58, ו LFA-1 (10 μL כל אחד) על קרח במשך 1 שעות. Tהוא ההשעיה התא יכול להיות מתויג עם נוגדנים פלורסנט.
4. HRS-, B-, בידוד T- התא באמצעות מיון תאים
הערה: למרות שהשתמשנו מכשיר מיוחד מחקר באמצעות 5 לייזרים (עיין גיליון חומרים), כל סדרן עם יכולת גילוי fluorochromes בשימוש בלוח הנוגדן צריך להיות מספיק. ביצוע השלבים הבאים דורש היכרות עם תוכנה 12 פונקציה ידע בסיסי של פעולות סדרן התא. עיין במדריך התוכנה המקוון לקבלת הוראות מפורטות.
- הגדרת Cytometer:
- הפעל את המחשב והתחבר. כוח על BSC (ו BSC שקעים), ולאחר מכן כוח על cytometer. המתן לפחות 90 s עבור המעבד הפנימי של cytometer כדי להתחיל, ולאחר מכן לפתוח את תוכנת בקרת הלייזר ולוודא כי כל לייזרים מופעלים. הפעל את התוכנה cytometer 13 ולהיכנס.
- בתוך התוכנה cytometer, לחץ על "Cytometer → הצגת תצורות." כאשר תיבת הדו שיח עבור תת תצורה התוכנית נפתחת, להדגיש את התצורה מותאמת אישית 130 מיקרומטר ולחץ על "הגדרת תצורה" ו "אישור". צא מתוכנית המשנה של התצורה.
- שים לב לתיבת הדו-שיח בתוכנת הציטומטר ולחץ על "השתמש בהגדרות CS & T". התקן זרבובית 130 מיקרומטר לתוך המכשיר ולהפוך את זרם על ידי לחיצה על כפתור "X האדום" בחלון הנחל. אפשר המכשיר להתחמם לפחות 30 דקות.
- הפעל בדיקת ביצועים על המכשיר בשיטה הרצויה (מודול תוכנה למעקב אחר ביצועים נכלל עם הסורר המתואר כאן, עיין במדריך התוכנה (עמ '117 - 122) ובסטנדרט הייחוס (עיין בחומרים עבור התקן המשמש התקנה זו).
- לחץ על "Cytometer → CST", ודא שהשדה "אפיון" מוגדר ל- "ChecK "בתפריט הנפתח, ולחץ על" הפעלה ". כאשר תתבקש על ידי התוכנה, לטעון צינורית של חלקיקים התייחסות על הדוגמה שלב ולחץ על" אישור ".
- לאחר סיום הריצה, לחץ על "סיום" וסגור את מודול התוכנה. לאחר תוכנת cytometer סיים להתחבר מחדש את cytometer, לחץ על "השתמש CS & T הגדרות" בתיבת הדו שיח שמופיע.
- קבעו את השהייה הנכונה של השחרור באמצעות תכונת השהייה האוטומטית של תוכנת הציטומטר (ראו עמ '154 - 161 של מדריך התוכנה).
- פתח את הניסוי "Drop Delay" בחלון "Browser" של תוכנת הציטומטר, התקן צינור של חלקיקי כיול על השלב המדגם, ולחץ על "טען" בחלון "רכישת לוח המחוונים". הפעל את התכונה ניטור אוטומטי זרם על ידי לחיצה על כפתור "מתוק נקודה" בחלון "זרם". השאר תכונה זו בכל עת עבור כל followinגרם צעדים.
- הפעל את מתח צלחת הסטייה על ידי לחיצה על כפתור "מתח" בחלון הנחל הצדדי ולאחר מכן הפעל את בדיקת המבחן על ידי לחיצה על הלחצן "Test Sort" הסמוך מיד ללחצן "מתח".
- התאם את כל הגדרות זרם הצד לאפס פרט לזרם הצד השמאלי. כוונן את הגדרת זרם הצד השמאלי כך ששני מקומות זרימה גלויים בחלון זרם הצד. לחץ על הלחצן "מסנן אופטי" וודא שכתם הזרם השמאלי ייפול בתוך התיבה השמאלית שמופיעה באזור השחור של חלון זרם הצד.
- כוונן את הגדרת הזרם השמאלי אם יש צורך. כבה את בדיקת המבחן על ידי לחיצה על הלחצן "בדוק מיון" שוב. בחלון "דפדפן", הרחב את האלמנט "Global Worksheets" של הניסוי "Drop Delay" על ידי לחיצה על "+".
- לחץ פעמיים על "מיין Layout_001" כדי לפתוח את פריסת מיון, לאמת על ידי בדיקה חזותיתיון כי האוכלוסייה המינית האוכלוסייה יש "P1" שהוקצו לו, ולחץ על "מיון" בחלון "מיון פריסה". בחלון "מיון פריסה", לחץ על "עיכוב אוטומטי" ולאחר מכן על "הפעלה".
- לאחר שהריצה תושלם, לחץ על "יציאה". התקן צינור של מים deionized סטרילי על השלב המדגם ולחץ על "טען" את "רכישת לוח המחוונים" חלון. הפעל את הצינור של מים לפחות 5 דקות כדי לנקות את חלקיקי הבדיקה שיורית מהמכשיר לפני שתמשיך.
- הפעל פקדים לפיצוי (באמצעות חרוזי פיצוי מגיליון החומרים או שווה ערך) באמצעות הגדרת פיצוי מובנית בתוכנת המזרן (ראה עמ '131 - 137 של מדריך התוכנה לפרטים נוספים).
- צור ניסוי חדש על ידי לחיצה על 'ניסוי חדש בניסוי'. בכרטיסייה "פרמטרים" של חלון "מצב מכשיר", מחק פרמטרים שאינם בשימוש, אם בכלל. לחץ על "ניסוי Compensatיון הגדרת פקדים פיצוי. "
- בחלון "דפדפן", הרחב את הדוגמה "Controls Controls" על-ידי לחיצה על הסימן "+". הפעל את צינורות בקרת הפיצויים מבלי להקליט את הנתונים, ואם יש צורך, כוונן את מתחי הגלאי (בכרטיסייה "פרמטרים" בחלון "מצב מכשיר"), כך שאוכלוסיית החרוזים המוכתמת באופן חיובי עבור כל פלואורוכרום היא בין ערוץ 10,000 ל -100,000, והם המבריקים בערוץ האיתור העיקרי שלהם.
- כתוב את המתח הנדרש עבור כל פרמטר עבור כל צינור. הדגש את הצינור "בקרה ללא שליטה" תחת הדגם "פיצויים שולטת" על ידי לחיצה על שמאל אחד. טען את צינור שליטה בלא כתם על הבמה המדגם ולחץ על "טען" בחלון "בקרת רכישה".
- הזן ידנית את המתחים שנקבעו על ידי הפעלת פקדים פיצויים בודדים לשדות מתח גלאי עבור כל הפרמטרים, וולאחר מכן לחץ על "הקלט" בחלון "בקרת רכישה". הפעל את כל פקדי הפיצויים הנותרים, הקלטת הנתונים מבלי לשנות את הגדרות הגלאי. התקן צינור של מים סטריליים, deionized במשך 5 דקות כדי לנקות את כל חומר שיורית מהמכשיר לפני שתמשיך.
- HRS תא gating:
- לרכוש ולהקליט לפחות 100,000 אירועים עבור gating הראשונית תוך התאמת קצב הזרימה לרכוש 3,000-4,000 אירועים / s (ראה שלב 4.1).
הערה: ייתכן שיהיה צורך להוסיף בינוני מיון נוסף לדלל את התאים אם ריכוז התא גבוה מדי. עצור את הרכישה. - שער תאים HRS באמצעות השלבים המתוארים באיור 1
הערה: ברוב המקרים CHL, בין 0.01% ל 0.1% של תאים יהיו תאים HRS.
- לרכוש ולהקליט לפחות 100,000 אירועים עבור gating הראשונית תוך התאמת קצב הזרימה לרכוש 3,000-4,000 אירועים / s (ראה שלב 4.1).
- B ו- T-cell gating:
- זיהוי שולט סומטי (B ותאי T) על ידי gating של CD20 ו CD5, respeCtively (gating לימפוציטים על ידי CD45 / SSH), ואחריו CD20 לעומת CD5 (ראה איור 1 ).
- איסוף אוסף זרמים לאוסף צינורות במעבר מראש צונן או דו כיוונית אוסף ארבעה. מילוי או צינורות זרימה או 15-mL צנטריפוגות צינורות בסגנון לפחות במחצית עם בינוני אוסף.
- הקצאת HRS ואוכלוסיות שליטה צינורות איסוף הנכון ההתקנה ההתקנה הבאה הוראות הספק. הפעל מחדש את רכישת התא ולהתחיל את המיון.
- לאסוף את כל התאים HRS עד 1 מיליון תאי B ו- T. לכוון את הזרמים אוסף צינורות איסוף במעמד מראש צונן ארבעה כיווני (או דו סטרי) מדף איסוף.
5. DNA החילוץ
- גלולה שנאספו תאים על ידי צנטריפוגה צינורות 1.5 מ"ל חרוטי ב XG 3000 במשך 10 דקות resuspend פעם עם 1 מ"ל של PBS לשטוף את התאים.
- גלולה שוב ב XG 3000 במשך 10 דקות ולהסיר את supernatant; היזהר מאוד לא להפריעאת גלולה זעירה.
- הוסף 150 μL של חיץ תמוגה (או נפח מתאים עבור ערכת בשימוש) לתאים שטף ומערבבים על ידי pipetting מעלה ומטה.
הערה: ניתן לאחסן lysate תא ב -70 ° C בשלב זה, במידת הצורך. - לבנות הרכבה עמודה על ידי הצבת עמודה מסנן בתוך הצינור ולהוסיף lysate מ צעד 5.5 לעמודה. ספין הרכבה ב XG 13,000 במשך 3 דקות.
- הסר את minicolumn מהרכבה ולזרוק את הנוזל בצינור אוסף. החלף את minicolumn בצינור איסוף.
- הוסף 650 μL של פתרון לשטוף עמודה לכל הרכבה. צנטריפוגה דקות 1 ב 13,000 x גרם. מחק את הנוזל מתוך צינור איסוף. חזור על שלב זה עבור סך של 4 שוטף.
- מחק את הנוזל מתוך צינור איסוף מחדש את הרכבה minicolumn. צנטריפוגה עבור 2 דקות ב XG 13,000 לייבש את המטריצה מחייב.
- מעבירים את minicolumn לצינור 1.5 מ"ל חדש ולהוסיף 25 μL של 10 מ"מ טריס- Cl, אשרהוא המועדף על צעד sonication הבאים, או Nuclease חינם מים מחומם ל 65 מעלות צלזיוס. לדגור על 2 דקות בטמפרטורת החדר צנטריפוגה הרכבה ב XG 13,000 במשך 1 דקות. חזור שוב פעם עם 25 μL עבור סכום כולל של 50 μL.
- מערבבים את elution DNA על ידי pipetting למעלה ולמטה לכמת באמצעות fluorimetry 14 .
6. בניית ספריה
- הכנה לפני תחילת:
- הגדרת sonicator ב מפלס המים 12, אינטנסיביות 5, מחזורים / פרץ 200, ו -7.
- בהתבסס על כמות DNA זמין שנקבע על ידי fluorimetry ועל העיצוב הניסויי, לקבוע את כמות ה- DNA לשימוש לבניית ספריה.
הערה: יש לזכור כי אם מעט מדי DNA משמש ואיכות הספרייה נפגעת, ייתכן שיש מעט יישומים עבור החומר שנותר מאחור למטרות אימות. עבור כניסות קלט תקני, ככל שקלט הקלט גבוה יותרהקיבולת של ספריית רצף שהתקבל. כמה הנחיות עבור פרוטוקול זה הן כדלקמן: 10 ng צריך להניב תוצאות טובות, 50 ng עשוי להיחשב מקסימלית גס. - להחליט על מתאם: הוספת יחס טוחנת מבוסס באופן רופף על הערכים בטבלה 1 .
- חישוב כמות המתאם לשימוש באופן הבא:
הערה: מספר שומות של קלט DNA, n
אני. N = 1.54e -12 12 * מסה קלט (ב ng) / (גודל קטע ממוצע)
Ii. כאשר גודל השבר הממוצע הוא 200 bp, זה מפשט ל:
N = 7.7e-15 * מסה קלט (ב ng)
מספר שומות המתאם לכלול, א
אני. N = r
נפח מלאי המתאם לשימוש בשלב קשירת המתאם, v
אני. V (ב μL ) = a / [ 10 -12 * * מתאםריכוז (ב מיקרומטר )]
הערה: אם ריכוז המתאם נמוך, כדי להבטיח את הכמות הנדרשת של מתאם כלול, פתרון מתאם ניתן להשתמש כדי לדלל את ריאגנטים תיקון סוף במקום מים.
לדוגמה: עבור 100 ng של קלט DNA עם גודל קטע ממוצע של 200 BP, עבור מתאם טוחנת הרצוי: להוסיף יחס של 15: 1 ואת ריכוז מלאי מתאם של 2 מיקרומטר, נפח המומלץ של מתאם להשתמש הוא 5.8 μL. - הקצה ברקודים באינדקס לדגימות.
הערה: ספריות שיתאגדו יחד לתגובת הכלאה או לנתיב בתא זרימה של רצף לא יכילו מדדים מיותרים.
- בניית הספריה - גז DNA:
- הוסף את מלוא כמות ה- DNA כדי לשמש צינור sonication. אם נפח המדגם המכיל את ה- DNA קלט לבד הוא פחות מ 50 μL, להוסיף EB חיץ עד נפח 50 μL הכולל לערבב.
- Sonicate עבור 30 s.
- הסר את הצינור ולבצע סיבוב מהיר בצנטריפוגה מיני רק מספיק כדי לאסוף כל תרסיס מהחלק העליון של הקירות של microtube.
- חזור על שלבים 6.2.2-6.2.3 עבור סכום כולל של שבעה מפגשי sonication 30-S עבור סך של 210 s של sonication.
הערה : אל תהסס להתנסות עם חלוקת 210 s לתוך פחות פגישות.
- בניית ספריה - תיקון סופי, זנב, קשירת מתאם:
הערה: הימנע לעשות כל בחירה גודל לפני צעד הגברה PCR הספרייה.- בצע את הוראות היצרן 19 עבור תיקון סוף ו- A-tailing לאחר sonication של ה- DNA המדגם.
- לאחר A-tailing, השתמש במספר המתאים של חפרפרות של מתאם (מחושב בשלב 6.1.3) בתגובה. שלב את המתאם, A- זנב DNA שברי, אנזים, חיץ ו דגירה לילה ב 20 מעלות צלזיוס במשך כ 16 שעות.
- ליבRary בנייה - הגברה ספריה:
- בצע ניקוי חרוז של התגובה קשירת מתאם. לאחר הוספת חרוזים למוצר ה- PCR, לחכות 5 דקות בטמפרטורת החדר. מניחים אותו נגד עמדה מגנטית להסיר את הנוזלים, לשטוף פעמיים μL 200 של אתנול 80%, יבש חרוזים מספיק זמן כדי להסיר את רוב הנוזל ללא ייבוש יתר, elute את ה- DNA של חרוזים ידי pipetting 25 μL של Nuclease ללא מים, כפי שהוצע, על חרוזים בעוד הצינור נשאר נגד מעמד מגנטי.
הערה: ייתכן שיהיה ניתן להתנסות עם שימור החרוזים ב polyethylene glycol
(PEG) עד לאחר הגברה במקום להשליך אותם, אבל זה לא נבדק. - כלול 0.6 μL של דילול 1: 1000 של צבע ירוק לכל μL 50 של תערובת הורים PCR. לחלופין, השתמש בזמן אמת PCR תואם צבע intercolating של בחירה בנפח המתאים עבור הציוד.
- תוכנית התגובה PCR ב 98 מעלות צלזיוס עבור45 s עבור denaturation הראשוני, ואחריו מחזור denaturation, חישול, וכן הרחבה ב 98 מעלות צלזיוס במשך 15 שניות, 60 מעלות צלזיוס למשך 30 שניות, ו 72 מעלות צלזיוס למשך 30 שניות, או לבחור את התוכנית המתאימה אם באמצעות חלופה אנזים פולימראז.
- הגדר את המכונה כדי לקחת נתונים הקרינה על 72 מעלות צלזיוס עבור כל מחזור. תוכנית הארכה הסופי ב 72 מעלות צלזיוס למשך דקה 1 ואחריו להחזיק ב 4 מעלות צלזיוס במשך כמות בלתי מוגבלת של זמן. PCR primers עבור הגברה של ספריית מתאם ligated באמצעות ריאגנטים בטבלה המשלימים הם: אוליגו 1, AATGATACGGCGACCACCGAGA ו אוליגו 2, CAAGCAGAAGACGGCATACGAG
- להגביר את הספרייה באמצעות התנאים PCR שתואר לעיל תוך התבוננות בעוצמת הקרינה ערכים בזמן אמת עם התוכנה qPCR, לעצור רק לפני סוף שלב הצמיחה המעריכית.
- לאחר הגברה, לעשות ניקוי חרוז סטנדרטי (ראה שלב 6.4.1) באמצעות 0.8x את עוצמת הקול של התגובה הגברה recovEred, בדרך כלל 0.8 x 50 = 40 μL של חרוזים. מוסיפים את החרוזים למוצר ה- PCR ומחכים 5 דקות בטמפרטורת החדר.
- מניחים אותו נגד עמדה מגנטית להסיר את הנוזלים, לשטוף פעמיים μL 200 של 80% אתנול, יבש חרוזים מספיק זמן כדי להסיר את רוב הנוזל ללא ייבוש יתר, elute ידי הוספת מים nuclease ללא חרוזים יבשים.
- לכמת את ה- DNA שנוצר באמצעות fluorimetry. לחזות את שברי הספרייה עבור גודל; עיין בסעיף על ציפיות נתונים QC לפרטים נוספים.
- בצע ניקוי חרוז של התגובה קשירת מתאם. לאחר הוספת חרוזים למוצר ה- PCR, לחכות 5 דקות בטמפרטורת החדר. מניחים אותו נגד עמדה מגנטית להסיר את הנוזלים, לשטוף פעמיים μL 200 של אתנול 80%, יבש חרוזים מספיק זמן כדי להסיר את רוב הנוזל ללא ייבוש יתר, elute את ה- DNA של חרוזים ידי pipetting 25 μL של Nuclease ללא מים, כפי שהוצע, על חרוזים בעוד הצינור נשאר נגד מעמד מגנטי.
7. Exome הכלאה
- שלב ארבע ספריות עם קודי מתאם שונים.
הערה: מסה על ידי fluorimetry וגודל על ידי ג 'ל משמשים לחישוב מולריות, ולאחר מכן ספריות משולבים כמויות equimolar עבור סכום כולל של 1,000 ng מסה של ספרייה מאוחסנת. מומלץ לשמור את כל זוגות הגידול הרגיל ביחד במקום להפריד אותם לבריכות נפרדות. - החל את פרוטוקול ללכוד exome Up class = "xref"> 15 ועושים 8 מחזורי PCR לאחר ניקוי הלכידה. אפשרויות אחרות של מטרות ללכוד עשוי להיות אפשרי.
8. רצף מרבב
- רצף לכידת הכלאה אחת המכילה ארבעה ספריות מרבב בנתיב אחד על פלטפורמת רצף המפנה בגיליון חומרים 16 .
הערה: תצורות אלטרנטיביות אפשריות עבור משתמשים מתקדמים המעוניינים להמשיך ולתכנן את היעד שלהם לקרוא עומק הכיסוי.
9. ניתוח (ניתן להחליף עם צינור חלופי (ים) אם רצוי)
- Snps ו indels קטן:
- מפת נתונים גולמיים לגנום הייחוס האנושי, UCSC hg19, באמצעות Burrows-Wheeler Aligner (BWA) 17 או אלגוריתם חלופי של בחירה. סינון או סימון קורא עם ציון איכות מיפוי מתחת 20 ו משכפלים PCR באמצעות Samtools 18 או פיקארדMaria 22> 19.
- זיהוי גרסאות נוקלאוטידים סומטיים indels קטן בדגימות HRS לעומת פקדים סומטיים T- התא באמצעות Strelka 20 או המתקשר משתנה של בחירה. החל snpEff 21 כדי להוסיף את הפלט Strelka. אם תרצה, לבדוק באופן שיטתי לוקוסים חלופה עבור artifacts באמצעות משולב Genome Viewer (IGV) 22 , 23 .
- העתק מספר שינויים:
- לחשב את יחס השתנה ליטר " ltr " עבור כל מרווח יעד exome " i " של ספריית intra ספריית נורמלי לקרוא בגידול נגד אלה של הנורמלי באופן הבא:
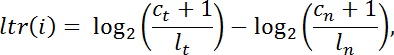
הערה: ג ' הוא מספר מיפוי הקריאה למרווח ללכוד נתון, l הוא גודל הספרייה הכולל, t מציין גידול, ו- n מציין נורמלי. - סנן את המרווחים עם כיסוי לא מספיק ( C + C n <100 reads) לניתוח נוסף. התנהגות פילוח inter- מרווח באמצעות DNAcopy v.1.0 24 מ Bioconductor ב ר.
הערה: שקול מקטעים שבהם הערך המוחלט של ה- ltr הממוצע מתחת ל -0.5 יהיה נייטרלי. המקטעים הנותרים יכולים להיקבע כמספר העתק, אם סימן ה - ltr הממוצע חיובי (במילים אחרות, יש יותר קריאות במדגם הגידול מאשר במדגם הרגיל לאחר הנורמליזציה,) או בהפסדים של מספר עותקים, אם השלט של ltr הממוצע הוא שלילי.
- לחשב את יחס השתנה ליטר " ltr " עבור כל מרווח יעד exome " i " של ספריית intra ספריית נורמלי לקרוא בגידול נגד אלה של הנורמלי באופן הבא:
Access restricted. Please log in or start a trial to view this content.
תוצאות
מגרש bioanalyzer צריך להילקח לאחר הגברה הספרייה ניקוי 0.8x חרוז. יש לראות "נורמלי כמו" התפלגות של גדלים שבר בטווח הרצוי ( איור 2 א ). סטיות מצורה זו, כגון "כתף" גלויה בעיקול, מציינות את נוכחותו של חפץ מולקולרי גבוה או נמוך. לדוגמה,
Access restricted. Please log in or start a trial to view this content.
Discussion
יישומים עתידיים או כיוונים לאחר מאסטרינג טכניקה זו
עבודה זו מאפשרת exome רצף מדגמים המכילים לפחות 10 ng של DNA. בהקשר הקליני, מגבלה זו אינה כוללת את רוב הדגימות של שאיבת המחט, בשל חומר לא מספיק, אך היא כוללת ביופסיות ליבה נאותות...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
הפיתוח של שיטה זו הפרויקט מומן על ידי המחלקה לפתולוגיה ו רפואה מעבדה של וייל קורנל המכללה הרפואית. אנו מכירים בתכנית ההכשרה המוסדית המשולבת בביולוגיה חישובית וברפואה למימון חלקי. ברצוננו להודות למדענים שחלקו עמם את זמנם ואת הידע שלהם, בייחוד את מריקה אפל; דן ברג'ס; Iwanka Kozarewa; צ'אד לוקלייר; וכולם מתוך מכללת וייל קורנל מכללת גנומיקס הליבה, כולל ג 'ני ג' אנג, Xiaobo (שון) ליאנג, דונג שו, ווי ג 'אנג, Huimin שאנג, טטיאנה בטסון, ו טאו ז' אנג.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Petri or Cell Culture Dish (sterile) | |||
| RPMI-1640 Media | Roswell Park Memorial Institute | ||
| Fetal Calf Serum (FCS), (heat inactivated) | |||
| Freezing Media (RPMI, 20% FCS, 10% dimethylsulfoxide (DMSO))-make fresh and keep sterile | |||
| RPMI with 2% FCS (make fresh or store for up to 1 month) | |||
| scalpel with fresh blade | |||
| 10 mL syringe (no needle) | |||
| Cryogenic vials | |||
| 50 mL conical centrifuge tubes, force | |||
| Centrifuge | capable of handling 50 mL conical centrifuge tubes and providing 400g | ||
| Hepes buffer(1 M, cell culture grade) | |||
| phosphate buffered saline (PBS) | |||
| Pluoronic-F68 | Thermo-Fisher | 24040-032 | |
| DNAase-I | Sigma-Aldrich, St. Louis, MO | D4527-10KU | store as 5 mg/mL in RPMI in -200 °C |
| Bovine Serum Albumin (BSA) | |||
| Sort Media (PBS + 2% BSA + 25 mM HEPES + Pluoronic –F68 (1x)) | |||
| CD64-FITC (22) | Beckman Coulter, Miami, FL | 20 μL suggested starting volume; Titering is suggested | |
| CD30-PE (BerH83) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD5-ECD (BL1a) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD40-PerCP-eFluor 710 (1C10) | Ebiosciences, San Diego, CA | 5 μL suggested starting volume; Titering is suggested | |
| CD20-PC7 (B9E9) | Beckman Coulter, Miami, FL | 10 μL suggested starting volume; Titering is suggested | |
| CD15-APC (HI98) | BD Biosciences, San Jose, CA | 20 μL suggested starting volume; Titering is suggested | |
| CD45 APC-H7 (2D1) | BD Biosciences, San Jose, CA | Can be substituted with 10 μL suggested volume of CD45-Krome Orange (J.33, Beckman Coulter); Titering is suggested | |
| CD95-Pacific Blue (DX2) | Life Technologies, Grand Island, NY | 5 μL suggested starting volume; Titering is suggested | |
| CD2 (5 μg; clone RPA-2.10) | Biolegend, San Diego, CA | For optional protocol; Titering is suggested | |
| CD54 (10 μg; clone 84H10) | Serotec, Oxford, United Kingdom | For optional protocol; Titering is suggested | |
| CD58 (10 μg; clone TS2/9) | eBioscience, San Diego, CA | For optional protocol; Titering is suggested | |
| LFA-1 (12 μg; clone MHM23) | Novus Biologicals, Littleton, CO | For optional protocol; Titering is suggested | |
| BD CS&T Beads | BD Biosciences, San Jose, CA | ||
| BD Accudrop Beads | BD Biosciences, San Jose, CA | ||
| BC Versa Comp antibody capture beads | Beckman Coulter, Miami, FL | Compensation Beads | |
| BD-FACS ARIA special research order instrument using 5 lasers | BD Biosciences, San Jose, CA | any BD-FACS aria with capabilities to detect the fluorochromes in the antibody panel should be sufficient | |
| Wizard | Promega | A2360 | |
| 10 mM Tris-Cl buffer | NA | ||
| Qubit dsDNA HS Assay kit | Life Technologies, Carlsbad, CA | ||
| S2 Sonicator | Covaris, Woburn, MA | Alternatives may be substituted | |
| microTUBE | Covaris, Woburn, MA | ||
| Low-Throughput Library Preparation Kit | Kapa Biosystems, Wilmington, MA | KK8221 | |
| Sybr Green | Sigma-Aldrich, St. Louis, MO | S9430 | |
| Agencourt AMPure XP Beads | Beckman Coulter, Miami, FL | ||
| Bioanalyzer | Agilent Technologies, Santa Clara, CA | ||
| SeqCap EZ Exome v.3.0 | Roche Nimblegen | 6465684001 | |
| HiSeq | Illumina | ||
| TruSeq-style Universal adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGAT*C*T | |
| TruSeq-style index adapter | Integrated DNA Technologies (IDT), Coralville, Iowa | HPLC purification; /5Phos/GATCGGAAGAGCACACGTCTGAACTCCAGTCACNNNNNNATCTCGTATGCCGTCTTCTGCTTG | |
| TruSeq-style PCR primer 1 | Integrated DNA Technologies (IDT), Coralville, Iowa | AATGATACGGCGACCACCGAGA | |
| TruSeq-style PCR primer 2 | Integrated DNA Technologies (IDT), Coralville, Iowa | CAAGCAGAAGACGGCATACGAG | |
| Nuclease Free Duplex Buffer | Integrated DNA Technologies (IDT), Coralville, Iowa | ||
| BD FACSDIVA software | BD Biosciences, San Jose, CA | ||
| BD Falcon Tubes | BD Biosciences, San Jose, CA | ||
| BD Flow Tubes | BD Biosciences, San Jose, CA |
References
- Abrams, J. National Cancer Institute's Precision Medicine Initiatives for the new National Clinical Trials Network. American Society of Clinical Oncology educational book / ASCO. American Society of Clinical Oncology. Meeting. , 71-76 (2014).
- Gagan, J., Van Allen, E. M. Next-generation sequencing to guide cancer therapy. Genome medicine. 7, 80(2015).
- Matsuki, E., Younes, A. Lymphomagenesis in Hodgkin lymphoma. Seminars in cancer biology. 34, 14-21 (2015).
- Fromm, J. R., Kussick, S. J., Wood, B. L. Identification and purification of classical Hodgkin cells from lymph nodes by flow cytometry and flow cytometric cell sorting. Am J Clin Pathol. 126 (5), 764-780 (2006).
- Fromm, J. R., Thomas, A., Wood, B. L. Flow cytometry can diagnose classical hodgkin lymphoma in lymph nodes with high sensitivity and specificity. Am J Clin Pathol. 131 (3), 322-332 (2009).
- Roshal, M., Wood, B. L., Fromm, J. R. Flow cytometric detection of the classical hodgkin lymphoma: clinical and research applications. Advances in hematology. 2011, 387034(2011).
- Reichel, J., et al. Flow sorting and exome sequencing reveal the oncogenome of primary Hodgkin and Reed-Sternberg cells. Blood. 125 (7), 1061-1072 (2015).
- Kozarewa, I. A Modified Method for Whole Exome Resequencing from Minimal Amounts of Starting DNA. PLoS ONE. 7 (3), e32617(2012).
- Brunicardi, F. C. Schwartz's Principles of Surgery. , 10th ed, McGraw-Hill Education / Medical. (2014).
- Kantor, A. B., Roederer, M. FACS analysis of leukocytes. Handbook of Experimental Immunology. 2, Blackwell Scientific. (1996).
- Sanders, M. E. Molecular pathways of adhesion in spontaneous rosetting of T-lymphocytes to the Hodgkin's cell line L428. Cancer Res. 48 (1), 37-40 (1988).
- Biosciences. FACSDiva Software v6.0. , Available from: http://www.bdbiosciences.com/in/instruments/software/facsdiva/resources/overview.jsp (2007).
- Biosciences. FACSAria II User's Guide. Part No. 644832, Revision A. , (2009).
- Life Technologies. Qubit 3.0 Fluorometer, Catalog Number Q33216. , Available from: https://www.thermofisher.com/order/catalog/product/Q33216 (2017).
- Roche Nimblegen. SeqCap EZ Library SR User's Guide ver. 4.1. , (2013).
- Illumina. HiSeq High-Throughput Sequencing System. , Available from: http://www.illumina.com/systems/hiseq_2500_1500.html (2016).
- Li, H., Durbin, R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics. 25 (14), 1754-1760 (2009).
- Li, H. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- The Broad Institute. Picard Tools. , Available from: http://broadinstitute.github.io/picard/ (2016).
- Saunders, C. T. Strelka: accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinformatics. 28 (14), 1811-1817 (2012).
- Cingolani, P., et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3. Fly (Austin). 6 (2), 80-92 (2012).
- Dobin, A. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in bioinformatics. 14 (2), 178-192 (2013).
- DNA copy number data analysis. v.R package version 1.34.0. , Available from: https://omictools.com/dnacopy-tool (2013).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved