A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
רצף מקביל בתפוקה גבוהה כדי כושר מדד לזיהום Leptospira interrogans Transposon ההכנסה מוטציות במהלך הזהב הסורי אוגר
In This Article
Summary
נתאר כאן טכניקה המשלבת מוטגנזה מכוונת transposon עם תפוקה גבוהה רצף כדי לזהות ולכמת transposon מוטציות leptospiral ברקמות לאחר אתגר של אוגרים. פרוטוקול זה יכול לשמש המוטנטים מסך עבור הישרדות והפצה בבעלי חיים, ניתן להחיל גם על מחקרים במבחנה .
Abstract
כתב יד זה, אנו מתארים transposon של רצף (Tn-Seq) טכניקה כדי לזהות ולכמת מוטציות Leptospira interrogans משתנה בכושר במהלך זיהום של אוגרים סוריים הזהב. Tn-Seq משלב מוטגנזה מכוונת transposon אקראיים עם הכוח של טכנולוגיה רצף תפוקה גבוהה. חיות מאותגרים עם בריכה של מוטציות transposon (בריכה קלט), ואחריו לקצירת של דם ורקמות כמה ימים מאוחר יותר כדי לזהות ולכמת את מספר מוטציות כל איבר (פלט בריכות). הבריכות פלט מושווים למאגר הקלט כדי להעריך את כושר ויוו של כל מוטציה. גישה זו מאפשרת הקרנת בריכה גדולה של מוטציות במספר מוגבל של בעלי חיים. עם שינויים מזעריים, פרוטוקול זה יכול להתבצע עם כל במודל חיה של לפטוספירוזיס, מאגר מודלים מארח כגון חולדות ומודלים זיהום אקוטי כמו אוגרים, כמו גם מחקרים במבחנה . Tn-Seq מספק כלי רב עוצמה למסך מוטנטים בעלי מומים כושר ב- vivo ו- in vitro לקשרי .
Introduction
זיהוי של גנים התקפה אלימה של חיידקים מסוימים, כגון Leptospira spp., קשה בגלל מספר מוגבל של גנטי הכלים הזמינים. גישה אחת נפוץ הוא יצירת אוסף של מוטציות על-ידי transposon אקראיים מוטגנזה מכוונת ואחריו הזיהוי של האתר הכניסה כל מוטציה ובדיקות התקפה אלימה של מוטציות transposon בודדים במודל חיה. גישה זו היא גוזלת זמן יקר, ודורש מספר גדול של בעלי חיים.
כאשר מוטגנזה מכוונת אקראי פותחה לראשונה על המחלה Leptospira interrogans, גנים המעורבים התקפה אלימה אותרו על-ידי בדיקת מוטציות בודדות המודל החייתי1. מוטציות נבחרו על סמך קריטריונים של תפקידיהם פוטנציאליים איתות או תנועתיות או שלהם החזוי הממברנה החיצונית או מיקום משטח. כמו ברוב חלקי leptospiral גנים קידוד חלבונים היפותטי של פונקציה לא מוכרות2, בחירת מוטציות בהתבסס על מגבלות קריטריונים אלה היכולת לגלות leptospiral הרומן גנים התקפה אלימה.
לאחרונה, בריכות של ל' interrogans transposon מוטציות הוקרנו על infectivity של אוגר ועכבר מודלים3. כל בעל חיים היה אתגר עם בריכה של מוטציות עד 10. Infectivity של מוטציה היה הבקיע חיובי אם זה זוהה על ידי ה-PCR של תרבויות המתקבל דם וכליות. בדיקת PCR היה מפרך כי זה נדרש לתגובה PCR נפרדים עבור כל מוטציה בבריכה. בגלל התדירות של כל מוטציה בתרבויות לא הייתה לכמת, הגישה היה מוטה לטובת זיהוי של מוטציות הקלוש ביותר.
אנו מתארים transposon של רצף (Tn-Seq) טכניקה, כאסטרטגיה למסך בצורה יעילה יותר עבור גנים התקפה אלימה. Tn-seq מורכב היצירה של ספריה של מוטציות על ידי מוטגנזה מכוונת transposon ואחריו רצף מקביל מסיבי4,5,6. בקצרה, מוטציות transposon איחדו, מחוסן חיות, התאוששה מאוחר יותר איברים שונים (פלט בריכות). ה-DNA של הבריכות פלט חילוץ, מתעכל עם אנזימי הגבלה או לכסנתם על ידי sonication. שני סיבובים של PCR פילוח של צמתים של האתרים ההכנסה transposon מבוצעות. שלב זה מאפשר התוספת של מתאמים הצורך עבור הרצף. המוצרים PCR וכתוצאה מכך הם נותחו על ידי רצף תפוקה גבוהה לזיהוי האתר הכניסה transposon של כל מוטציה של הבריכה יחד עם שפע היחסי שלהם, אשר הוא לעומת ההרכב הראשוני של הבריכה של מוטציה.
היתרון העיקרי של גישה זו היא היכולת למסך בו זמנית מספר רב של מוטציות עם מספר קטן של בעלי חיים. Tn-Seq אינו דורש ידע מוקדם של האתרים ההכנסה transposon אשר מגדיל את הסיכויים של גילוי חדש Leptospira-גנים ספציפיים המעורבים התקפה אלימה עם פחות זמן ויעילות רבה יותר. כי נטל leptospiral ברקמות הוא גבוה יחסית מכרסם מודלים זיהום רגישים קטלני (בדרך כלל 104 108 חיידקים/גרם של רקמת)7,8,9 באותה מידה כמו מאגר המארחים 10,11, רקמות ניתן לנתח ישירות ללא צורך תרבות, צמצום הטיות עקב הצמיחה במבחנה .
במחקרים Tn-Seq עם רוב פתוגנים חיידקיים שתוארו עד כה, התדר של מוטגנזה מכוונת insertional מותרים זיהום עם בריכות גדולות המכיל מוטציות קולקטיבי שיש הוספות המרווחות מקרוב transposon מרובות בתוך כל ג'ין4 ,12,13,14. Tn-Seq גם פותחה עבור חיידק אשר תדירות מוטגנזה מכוונת היא נמוכה יותר,6. עם Leptospira, ספרייה של מוטציות transposon יכול להיווצר על ידי החדרת את transposon על פלסמיד mobilizable על-ידי ההטיה כפי שתואר על ידי Slamti et al15. עם זאת, התדירות של מוטגנזה מכוונת transposon של ל' interrogans הוא נמוך. כאשר transposon Himar1 הוצג על פלסמיד conjugative, תדירות transconjugant דווחה רק 8.5 x 10-8 לכל נמען תא עם המתח לאי של ל' interrogans16 , סביר להניח שתהיה מסכן באופן דומה עם רוב זנים אחרים של ל' interrogans. פרוטוקול המתואר כאן הוא בחלקו מבוסס על שפותחה עבור Borrelia burgdorferi, שבו תדירות מוטגנזה מכוונת insertional transposon היא גם נמוכה6.
עבור שלנו פיילוט עם פרוטוקול ה-17, ערכנו מוטגנזה מכוונת transposon עם ל' interrogans serovar Manilae זן L495 בגלל ההצלחה של קבוצות אחרות לבודד מוטציות ההכנסה transposon בנימה זו יחד עם שלה נמוך LD50 (מנה קטלנית) התקפה אלימה1. אנו מוקרן מוטציות 42 על ידי Tn-Seq, זיהו כמה מועמדים מוטציה פגומה בהתקפה אלימה, כולל שני עם הוספות בגן אדנילאט cyclase המועמד. בדיקה אישית של המוטציות שני באוגרים אישר כי הם היו בנכונותם התקפה אלימה17.
Protocol
התראה: זנים פתוגניים של Leptospira spp. להיות מטופלת תחת נהלים הבלימה אבטחה ברמה 2 (BSL-2). חובה ללבוש ציוד מגן אישי המתאים (עיקרון השוויון הפוליטי). הקבינט Class II אבטחה יש להשתמש עבור כל המניפולציות של פתוגניים Leptospira spp.
1. הקמת Transposon ספריית מוטנטים15
-
העברת transposon לתוך spp Leptospira . על ידי ההטיה (איור 1)
- לחסן נפח של מעריכי-פאזי התרבות של spp. Leptospira פתוגניים המתאים 107 תאים לתוך 10 מ"ל של Ellinghausen-מקלו-ג'ונסון-האריס-18,(EMJH)-בינוני-19. דגירה ב 30 ° C עם סל"ד 150 לרעוד עד הצפיפות מגיע 2-8 x 108 תאים למ"ל.
הערה: הזמן ההכפלה של leptospires פתוגניים הוא 12 עד 24 שעות, בהתאם המתח. - לחסן µL 50 מתוך התורם Escherichia coli זן β216320 נושאת את פלסמיד (pCjTKS2) mobilizable transposon16 לתוך 5 מ"ל לוריא מרק (LB) בתוספת 0.3 מ מ 2, 6-diaminopimelicacid (DAP), µg 50/מיליליטר kanamycin (ק מ), 50 µg/mL spectinomycin (Spc) ומקום לילה בחממה 37 ° C-255 סל ד.
- לחסן µL 60 של e. coli תאים לתוך 3 מ"ל של EMJH בתוספת 0.3 מ מ של DAP (EMJH + DAP). דגירה ב 37 מעלות צלזיוס ב 255 rpm עצבנות עבור 3-4 h עד ≈600nmOD 0.3.
- להרכיב את יחידת סינון (איור 1), מניחים את הבסיס על 125 מ לנשק צד Erlenmeyer את הבקבוק, מקם מסנן תאית אצטט (גודל הנקבוביות 0.1 מ"מ; קוטר 25 מ מ) על בסיס, ו מהדק המשפך על גבי הבסיס. לחבר את יחידת סינון מערכת ואקום.
- הוסף 5 מ של Leptospira spp. תרבות, 0.5 mL התרבות החיידק לאבדון. אני יודע את הנוזל דרך המסנן.
- להעביר את המסנן עם פני השטח של חיידקים פונה כלפי מעלה אל EMJH + DAPplate. דגירה ב 30 מעלות צלזיוס למשך הלילה עם המסנן פונה כלפי מעלה.
- למקם את המסנן לתוך צינור 15 מ"ל המכיל 1 מ"ל של EMJH ו מערבולת 10 s כדי לשחרר את החיידקים לתוך כלי התקשורת. מורחים µL 200 של התליה על 5 EMJH platescontaining 50 µg/mL של ק מ באמצעות חרוזי זכוכית סטריליים 1 מ 10-15 או של מפזר חד פעמי סטרילי. לעטוף את הצלחות עם מצלמות-מיקרוסקופים, דגירה אותם הפוך ב 30 מעלות צלזיוס במשך 3-4 שבועות עד מושבות גלויים.
- העברת המושבות בנפרד לתוך 3 מ"ל של EMJH המכילה µg/mL 50 ק מ (EMJH + ק מ) ב 30° C תחת סל ד 150 עצבנות 7 עד 10 ימים עד התרבות מגיע צפיפות של ≈ 108/mL.
הערה: תרבויות שניתן לאחסן ב- 80 ° C או בחנקן נוזלי (עם 4% גליצרול).
- לחסן נפח של מעריכי-פאזי התרבות של spp. Leptospira פתוגניים המתאים 107 תאים לתוך 10 מ"ל של Ellinghausen-מקלו-ג'ונסון-האריס-18,(EMJH)-בינוני-19. דגירה ב 30 ° C עם סל"ד 150 לרעוד עד הצפיפות מגיע 2-8 x 108 תאים למ"ל.
-
זיהוי של אתר ההכנסה transposon על ידי ה-PCR מקוננים (איור 2)
- Lyse 50 µL של כל מוטציה transposon ב המבחנות על ידי דגירה ב 95 מעלות צלזיוס למשך 15 דקות.
הערה: ה-DNA אפשר לטהר במקום, באמצעות ערכת חילוץ הדנ א. - הכינו את התמהיל PCR עם Deg1 ו- Tnk1 תחל (טבלה 3) על פי טבלה 1. להעביר כל שפופרת PCR µL 23.7 של לערבב ולהוסיף 1.3 µL של תאים lysed. הפעל את התוכנית: 95 מעלות צלזיוס למשך 5 דקות; 40 מחזורי: 95 ° C 15 s, 40 ° C עבור 1 דקות, 72 מעלות צלזיוס למשך 2 דקות; 72 מעלות צלזיוס למשך 10 דקות.
- לעשות את המיקס PCR עם תג ו TnkN1 תחל (טבלה 3) על פי בטבלה 2. להעביר כל שפופרת PCR 24.2 µL של לערבב ולהוסיף 0.8 µL של ה-PCR #1 תגובה. הפעל את התוכנית: 95 מעלות צלזיוס למשך 5 דקות; מחזורים 35: 95 ° C 15 s, 55 ° C ל 30 s, 72 מעלות צלזיוס למשך 2 דקות; 72 מעלות צלזיוס למשך 10 דקות.
- להפעיל 3 µL של מוצרי ה-PCR על 1% agarose ג'ל עם מאגר 1 X טריס-אצטט-EDTA (טה) ב 10-15 V/ס מ (איור 2B).
- לטהר את מוצרי ה-PCR של דגימות חיוביות באמצעות ערכת טיהור PCR. Elute DNA עם נפח הנמוך ביותר המותר ע י הערכה על מנת למקסם את הריכוז של ה-DNA התאושש.
- שולח המוצרים PCR מטוהרים סנגר רצף באמצעות פריימר של TnkN1 (טבלה 3).
- לזהות את האתרים הכניסה על ידי השוואת את הרצף שנוצר עם רצף הגנום של המתח הורים על ידי ניתוח BLASTN (http://blast.ncbi.nlm.nih.gov/) או באמצעות מסד הנתונים (http://www.genoscope.cns.fr/agc/mage) SpiroScope21.
- אשר האתר ההכנסה של transposon על ידי ה-PCR באמצעות תחל ועוברים תהליך ריפוי כדי הרצפים מארח איגוף.
הערה: transposon מגדילה את הגודל של הרצף פראי-סוג ≈ 2 kb.
- Lyse 50 µL של כל מוטציה transposon ב המבחנות על ידי דגירה ב 95 מעלות צלזיוס למשך 15 דקות.
2. חיות ניסוי (איור 3)
-
תרבות של מוטציות Leptospira
- לגדול באופן אינדיבידואלי לכל transposon שנבחר מוטציה ב- 10 מ"ל של EMJH + ק ב 30 ° C-150 סל ד עצבנות על צפיפות של7-108 10 leptospires/mL.
- את leptospires בדילוגים של מיקרוסקופיית שדה אפל עם מונה Petroff-Hausser, או כפי שמתואר על ידי מילר23.
- לדלל כל תרבות ב EMJH כדי צפיפות זהה, למשל 106 תאים למ"ל.
- להרכיב את הבריכה קלט, מערבבים יחד את התרבויות מדולל בכמויות שוות.
הערה: כוללים פקדים בבריכה קלט על-ידי הוספת מוטנטים בעלי כושר הידוע מומים כגון loa2217,24 מוטציות עם כושר שהודעה כגון ligB17,25, בהתאמה. ניתן לאחסן בריכות של מוטציות עם 4% גליצרול ב-80 מעלות צלזיוס או חנקן נוזלי.
-
אתגר
הערה: בשיטות המתוארות כאן אושרו על ידי ותיקי לענייני גדול בלוס אנג'לס מוסדיים חיה על עצמך ועל שימוש הוועדה (פרוטוקול #09018-14).- מזריקים intraperitoneally 1 מ"ל של מאגר קלט כדי כל חיה מזרק אינסולין U-100 עם מחט 26G x ½".
הערה: בניסוי פיילוט, חיות 8 היו תגר עם 1 מ"ל של מאגר קלט, קרי, 106 חיידקים סה כ17. הזיהום הורשה להמשיך במשך 4 ימים לפני המתת חסד. - לאסוף 10 מ"ל של מאגר קלט, ספין במשך 20 דקות ב- 3,220 x g... הסר בזהירות את תגובת שיקוע מבלי להפריע בגדר. לאחסן בגדר תא ב- 80˚C עד לשימוש (שלב 3.1.3).
- לפקח על החיות מדי יום עד שהם מסתיימות כאשר הקצה שנקבע מראש. שוקל את האוגרים מדי יום ולחפש נקודת הקצה קריטריונים: אובדן תיאבון, הילוך או נושם קושי, השתטחות, פרווה הסתור, או אובדן > 10% מן המשקל המרבי השיגו.
- מזריקים intraperitoneally 1 מ"ל של מאגר קלט כדי כל חיה מזרק אינסולין U-100 עם מחט 26G x ½".
-
ניסויים במבחנה
- ביום של אתגר, לחסן 3 מבחנות של 25 מ של EMJH + ק מ 5 מ של מאגר קלט. לגדול תרבויות ב 30 ° C תחת סל ד 150 עצבנות.
- לספור את leptospires מדי יום באמצעות אחת מהשיטות של סעיף 2.1.2. הצפיפות מגיעה ≈ 1 x 108/mL, ספין לאורך כל השעיה במשך 20 דקות ב 3,220 x g.
- לאחסן את כדורי תא ב-80 מעלות צלזיוס עד השימוש.
-
איסוף ואחסון של רקמות
- המתת חסד החיות באמצעות אינהלציה איזופלוריין ואחריו ניקור חזה הדו-צדדיים26.
- מיד לאסוף 1 עד 2 מ של דם על ידי ניקוב לב עם מזרק 3 מ"ל ו מחט 25G x 5/8". להעביר את הדם לתוך שפופרת המכילה EDTA. מערבבים על-ידי היפוך, 5-6 פעמים.
- לאסוף את אחת הכליות לתבנית ל ½ של האונה החציוני הגחון של הכבד לתוך cryotubes.
- לאחסן לרקמות ב- 80 ° C עד השימוש.
3. הקמת ספריות גנומית עבור תפוקה גבוהה רצף (איור 4)
-
הפקת דנ א
- מיצוי DNA מהדם.
- העברה µL 100 של דם EDTA ברכבת התחתית צינור microcentrifuge.
- לטהר הדנ א באמצעות ערכת חילוץ הדנ א. בצע הוראות היצרן.
- מיצוי הדנ א של רקמות.
- באמצעות ואזמלי מנתחים ומספריים, הקוביות בין 50 ל-80 מ"ג של כל איבר לחתיכות קטנות (1 מ"מ x 1 מ"מ), להעביר אותם לתוך צינור יבשה סטרילית בורג-קאפ. למדוד את המשקל של רקמות עם מאזן דיוק.
- להוסיף 500 µL ל- PBS סטרילי לתוך הצינור.
- Homogenize דגימות באמצעות מפצל עבור 1 דקות ב- 5 תנועות לשניה.
- לחשב את עוצמת הקול המתאים 25 מ ג של רקמה באמצעות המשוואה הבאה:
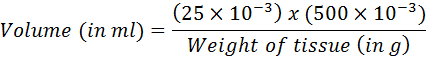
- להעביר את אמצעי האחסון מחושב לתוך ערכת חילוץ דנ א. להמשיך עם טיהור DNA ההוראות של היצרן.
- מיצוי DNA מתרבויות בריכה, במבחנה קלט.
- הפשרת כדורי חיידקי בטמפרטורת החדר למשך 5-10 דקות.
- להמשיך עם מיצוי DNA ההוראות המלווה את הקיט.
- מאגר DNA ב-80 מעלות צלזיוס עד השימוש.
- מיצוי DNA מהדם.
-
הטיית ה-DNA (איור 5)
- העברת µL 50 של DNA שחולץ על גבי צינור microcentrifuge 1.5 mL.
- מקם את הצינורות לתוך ארון התקשורת של הקרן sonicator כוס מלאה במים קרים (4 ° C).
- הפעל את sonicator למשך 3 דקות בעוצמה 80% עם 10 s על הדופק ו- 5 s מחוץ דופק.
התראה: ללבוש מצמדים האוזן או אטמי אוזניים להגן על השמיעה. - לרוץ µL 2.5 של הוטו דנ א על 2% agarose ג'ל כדי לאשר כי רוב ה-DNA הוא < 600 bp בגודלם.
-
תוספת של C-זנב
- למדוד ריכוז ה-DNA עם ספקטרופוטומטרים נפח קטן.
- חישוב נפח המתאימים 500 ננוגרם של ה-DNA באמצעות המשוואה הבאה:
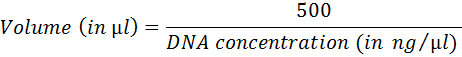
- להכין את התגובה עוקב (טבלה 4). דגירה h 1 ב 37 מעלות צלזיוס; הפוך ללא פעיל ב 75 מעלות צלזיוס למשך 20 דקות.
הערה: לקבלת דוגמאות המחייבים התאמת נפח גדול מ- 14.5 µL, להגדיל את נפח סופי של תגובה 40 µL ולמדרג הרכיבים הנותרים בהתאם. - לנקות את הדגימות עם ערכת טיהור PCR. Elute ה-DNA עם µL 12 • תנאי המאגר.
-
PCR מקוננים
- PCR #1
- הכנת תערובות PCR על פי טבלאות 3 ו -5. להעביר µL 22 של המיקס ספריית כל שפופרת PCR ולהוסיף 3 µL של ה-DNA מטוהרים. להמשיך באופן דומה עם המיקס שליטה.
הערה: פריימר TnkN317 היא ספציפית transposon והיא תחל olj3766 ספציפי זנב C. המיקס שליטה חסר מן המפתח TnkN3, אשר במיוחד מטרות של transposon. - הפעל את התוכנית הבאה: 95 מעלות צלזיוס למשך 2 דקות; מחזורים 24: 95 ° C ל 30 s, 60 ° C ל 30 s, 72 מעלות צלזיוס למשך 2 דקות; 72 מעלות צלזיוס למשך 2 דקות.
- הכנת תערובות PCR על פי טבלאות 3 ו -5. להעביר µL 22 של המיקס ספריית כל שפופרת PCR ולהוסיף 3 µL של ה-DNA מטוהרים. להמשיך באופן דומה עם המיקס שליטה.
- PCR #2.
- להכין את תערובות PCR השני על פי טבלאות 3 ו 6. µL 49 של המיקס ספריית להעביר כל שפופרת PCR ולהוסיף 1 µL של תגובה ספריית ה-PCR #1. להעביר µL 24.5 של המיקס שליטה כל שפופרת PCR ולהוסיף 0.5 µL של ה-PCR #1 שליטה תגובה.
הערה: פריימר pMargent2 ספציפית transposon, תחל IP6 מכילים 6-בסיס-זוג ברקוד רצפים ספציפיים מזוהה על-ידי פלטפורמת הדור הבא רצפי. - הפעל את התוכנית הבאה: 95 מעלות צלזיוס למשך 2 דקות; 18 מחזורים: 95 ° C ל 30 s, 60 ° C ל 30 s, 72 מעלות צלזיוס למשך 2 דקות; 72 מעלות צלזיוס למשך 2 דקות.
- להכין את תערובות PCR השני על פי טבלאות 3 ו 6. µL 49 של המיקס ספריית להעביר כל שפופרת PCR ולהוסיף 1 µL של תגובה ספריית ה-PCR #1. להעביר µL 24.5 של המיקס שליטה כל שפופרת PCR ולהוסיף 0.5 µL של ה-PCR #1 שליטה תגובה.
- הפעל 3 µL על 2% agarose ג'ל. הספרייה צריכה להראות כתם עם הרוב של האות בין 200 ל 600 bp (איור 6) ואין הגברה עבור התגובה שליטה.
- PCR #1
-
טיהור של מוצרי ה-PCR.
הערה: לנקות את ספריות גנומית עם ערכת טיהור PCR ההוראות של היצרן. Elute ה-DNA עם 30 µL • תנאי המאגר. - מודדים את ריכוז הדנ א באמצעות של fluorometer. קריאות בממוצע 2-3.
- לחשב את הנפח של כל ספריה שקול ל- 15 או 20 ng באמצעות המשוואה הבאה:
-
לערבב כל ספריות יחד על פי חישוב הקודם. לקבוע את ריכוז מולרי של ה-DNA עם המשוואה באתר האינטרנט הבא:
http://www.molbiol.edu.ru/eng/scripts/01_07.html
התראה: ריכוז ה-DNA ובדרישות אחסון תלויים פלטפורמת רצף.
4. תפוקה גבוהה רצף וניתוח נתונים
- רצף
- רצף ספריות כמו 64 bp יחיד-end קריאות באמצעות pMargent3 תחל את רצף מותאם אישית ומהצמיגים רצף מסחרי רגיל. פלטפורמת רצף יספק לכם FastQ קבצים עם כל רצף קריאות.
- לניתוח עם גלקסיה תוכנה
- הורד את הקובץ רצף הגנום
- בהבית של מסד הנתונים של SpiroScope (ראה שלב 1.2.7 בקישור), בחר את האורגניזם המשמש את הניסוי ולחץ על "טען לתוך הגנום הדפדפן".
- הכלים (בסמוך לחלקו העליון של דף הבית), בחר באפשרות "חיפוש/ייצוא > להוריד נתונים", בשורה "רצף (fasta)", לחץ על הגנום"כדי להוריד את הרצף.
- פתח את הקובץ באמצעות פנקס רשימות (PC) או TextEdit (Mac) ושנה את הכרומוזום (לדוגמה "המו"). לשמור על הפורמט fasta.
- בצע את השלבים אותם להוריד את רצף כרומוזום II. לשלב שני רצפים כרומוזום לתוך קובץ. txt יחיד על-ידי העתקה והדבקה.
הערה: רצף כרומוזום יכול גם להיות שהורדה מהאתר NCBI (https://www.ncbi.nlm.nih.gov) ושולבו קובץ. txt יחיד.
- העלאת קבצים לשרת גלקסי
הערה: גלקסיה הוא קוד פתוח, הפלטפורמה מבוססת אינטרנט לניהול נתונים ביואינפורמטיקה אינטנסיבית זרימות עבודה27,28,29,30 , ניתן לגשת https://usegalaxy.org/.- בתפריט כלים, בחר "לקבל נתונים > העלאת קובץ מהמחשב שלך". גרור להוריד את הקבצים .fastq שנוצר על-ידי הפלטפורמה רצף לתוך החלון ולאחר מכן לחץ על "התחל".
- בצע את השלבים באותו להעלות את הקובץ. txt Leptospira רצף הגנום.
- החתן קורא
- בחר "הגדרות: QC ומניפולציה > FASTQ התספורת" מתפריט כלים.
- ליד קובץ החתן, בחר את הספריות להעלות בשלב 4.2.2. בסוג ציוני איכות FASTQ קלט, בחר את מערכת רצף המתאים. לאפשרויות מתקדמות, תעזוב להסתיר מתקדם המשרד נבחר
- לחץ על "בצע".
- הסרת לכלוכים רצף
- בחר "הגדרות: QC ומניפולציה > להסיר לכלוכים רצף". ליד ספריה כדי לסנן, בחר את הקבצים מטופח שנוצר בשלב 4.2.3. לחץ על "בצע".
- להסיר את רצפי C-פלי
הערה: חזור על שלבים אלה פעם או פעמיים כדי להבטיח כל C-פלי יוסרו.- בחר "הגדרות: QC ומניפולציה > קליפ מתאם רצפים".
- בחר או הזן את הטקסט שלהלן:
ספריית לקליפ: בחר את הקבצים שנוצרו בשלב 4.2.4.
רצף מינימלי אורך: 15
מקור: להזין רצף מותאם אישית
הזן רצף מסיכה אישית: CCCCCCC
הזן ערך שאינו אפס כדי לשמור על רצפים מתאם ובסיסים x שבאות אחריה: 0
למחוק את רצפי עם בסיסים (N) לא ידוע: כן
אפשרויות פלט: פלט רצפים החתוכים והן שאינם נחתכים - לחץ על "בצע".
- הסר מתאם רצפים
- בחר "הגדרות: QC ומניפולציה > קליפ מתאם רצפים".
- בחר או הזן את הטקסט שלהלן:
- ספריית לקליפ: בחר קבצים שנוצרו בשלב 4.2.5.
- רצף מינימלי אורך: 15
- מקור: להזין רצף מותאם אישית
- הזן רצף מסיכה אישית: CGTATGCCGTCTTCTGCTTG
- הזן ערך שאינו אפס כדי לשמור על רצפים מתאם ובסיסים x שבאות אחריה: 0
- למחוק את רצפי עם בסיסים (N) לא ידוע: כן
- אפשרויות פלט: פלט רצפים החתוכים והן שאינם נחתכים
- לחץ על "בצע".
- קריאות מסנן בהתבסס על האיכות שלהם
- בחר "הגדרות: QC ומניפולציה > סנן לפי איכות".
הערה: כלי זה בוחר קריאות בהתבסס על איכות התוצאות. - בחר את הפריטים הבאים:
ספריית לסינון: בחר את הקבצים שנוצרו בשלב 4.2.6.
איכות ניתוק הערך: 20
אחוז בסיסים ברצף כי חייב להיות באיכות שווה ל/גבוה יותר מהערך ניתוק: 95 - לחץ על "בצע".
הערה: עם הגדרות אלו, קריאות קצר יותר מאשר 20 נוקלאוטידים או עם ציון האיכות של 20 או פחות 95% של המחזורים מתבטלים. להתאים הגדרות לכתחילה כדי הניסוי שלך.
- בחר "הגדרות: QC ומניפולציה > סנן לפי איכות".
- מפת קורא32
- בחר "הגדרות: מיפוי > Bowtie2"32.
- בחר את הפריטים הבאים את השדות בחלון הראשי:
הוא יחיד או מזווג הספרייה: יחיד-end
FASTQ קובץ: בחר את הספריה מסוננים לאיכות מהשלב 4.2.7.
לכתוב קריאות unaligned (בפורמט fastq) כדי להפריד בין קבצים: אין
לכתוב מיושר (בפורמט fastq) קריאות להפריד קבצים: אין
האם תבחר גנום הפניה מההיסטוריה שלך או להשתמש ליצירת אינדקס מובנה?: להשתמש גנום מההיסטוריה ולבנות אינדקס
בחר את הגנום הפניה: בחר לטעון את הקובץ genome.txt Leptospira בשלב 4.2.2.
סט קריאת קבוצות מידע?: לא הוגדר
בחירת ניתוח מצב: 1: ברירת המחדל של הגדרה בלבד
האם ברצונך להשתמש בהגדרות קבועות מראש?: לא, פשוט להשתמש ברירות מחדל
שמור את הנתונים הסטטיסטיים מיפוי bowtie2 ההיסטוריה: לא
עבודה משאב פרמטרים: להשתמש בפרמטרים משאב העבודה המהווה ברירת מחדל - לחץ על "לבצע" ליישור שאוחזרו כדי הגנום.
- להמיר קבצים
- בחר "הגדרות: SAMtools >. באם אל-סם להמיר באם סאם".
- בחר את הפריטים הבאים:
באם קובץ להמרת: בחר מיפוי ספריית מהשלב 4.2.8.
אפשרויות כותרת: כלול כותרת בפלט סאם (-h) - לחץ על "בצע".
- להמיר קבצים
- בחר "הגדרות: SAMtools > להמיר סאם מרווח".
- בחר את הפריטים הבאים:
בחר את הנתונים (dataset) כדי להמיר: בחרו בקובץ הספרייה ממופה סאם שנוצר בשלב 4.2.9.
להדפיס את כל?: כן - לחץ על "בצע".
- מיון קורא
- בחר "סינון ומיון > למיין נתונים לפי סדר עולה או סדר יורד".
- בחר את הפריטים הבאים:
מיון הנתונים (dataset): בחר את הקובץ מרווח שנוצר בשלב 4.2.10.
עמוד: 2
בטעם: סדר מספרי
הכל: סדר עולה - לחץ על "בצע".
- בחר קריאות התאמת כרומוזום אני
- בחר "סינון ומיון > בחר שורות התואמות את הביטוי".
- בחר את הפריטים הבאים:
בחר של קווים: בחר קובץ עם קריאות ממוינים שנוצר בשלב 4.2.11.
זה: התאמת
התבנית: הזן את השם של כרומוזום כמו החלטתי בשלב 4.2.1.3 (למשל, "אלוהים"). - לחץ על "בצע".
- בחר קריאות התאמת כרומוזום II
המשך בעקבות צעד 4.2.12. - קבוצה קורא על פי האתרים הוספות כרומוזום
- בחר "Join, להחסיר וקבוצת > לקבץ נתונים לפי עמודה ולבצע פעולה צבירה על עמודות אחרות".
- בחר את הפריטים הבאים:
בחר נתונים: בחר קובץ שנוצר משלב 4.2.12.
קבץ לפי עמודה: 2
התעלמות מרישיות תוך קיבוץ?: לא
להתעלם שורות המתחילות בתווים אלה: Ø
מבצע > + פעולת הוספת
סוג: ספירה
עמוד: 2
לעגל את התוצאה למספר השלם הקרוב: לא - לחץ על "בצע".
- קבוצה קורא על פי האתרים ההכנסה כרומוזום II
המשך בעקבות שלב 4.2.14. - מיון הכנסה אתרים על כרומוזום
- בחר "סינון ומיון > למיין נתונים לפי סדר עולה או סדר יורד".
- בחר את הפריטים הבאים:
סוג נתונים: בחר קובץ משלב 4.2.14.
עמוד: 1
בטעם: סדר מספרי
הכל: סדר עולה - לחץ על "בצע".
- למיין אתרים ההכנסה על כרומוזום II
המשך בעקבות שלב 4.2.16.
- הורד את הקובץ רצף הגנום
- ניתוח סטטיסטי
- להעביר את הנתונים גלקסי לתוך גיליון אלקטרוני קבצים על-ידי העתקה והדבקה של שתי העמודות מצעדי 4.2.16. 4.2.17. לתוך קובץ Excel.
הערה: העמודה הראשונה הוא הקואורדינטה נוקלאוטיד של אתר הכניסה transposon, העמודה השניה הוא מספר הקריאות בכל אתר ההכנסה. - לזהות את הגן מסתירים את transposon באמצעות הקואורדינטה נוקלאוטיד בטבלה.
הערה: לדוגמה, transposon להוסיפה אצל נוקלאוטיד #30718 של כרומוזום זה ממוקם הגן LIC10024 , אשר משתרע על פני נוקלאוטידים כרומוזום בתוך 29263-31539. - לחישוב יחסי תדרים (F) כל מוטציה בכל רקמה, בבריכה קלט בעקבות המשוואה להלן:
- חישוב יחסי הקלט/פלט (R) לכל מוטציה או רקמות באמצעות המשוואה הבאה:
- מבחן דרגה חתם Wilcoxon
- לנרמל את כל יחסי הקלט/פלט על-ידי הגדרת היחס החציוני עבור כל רקמות כל חיה ל- 1.0. יחס של 1.0 הוא נייטרלי, > 1.0 הוא חסרון, ו < 1.0 הוא יתרון33.
- להשוות בין יחסי קלט פלט ל- 1.0 (כושר נייטרלי) באמצעות הבדיקה דרגה Wilcoxon עם ערכים P < 0.05 נחשב משמעותי סטטיסטית.
- להעביר את הנתונים גלקסי לתוך גיליון אלקטרוני קבצים על-ידי העתקה והדבקה של שתי העמודות מצעדי 4.2.16. 4.2.17. לתוך קובץ Excel.
תוצאות
יצירה של ספריה של transposon מוטציות בל' interrogans על ידי ההטיה דורש יחידת סינון, כפי שמוצג באיור1. מצאנו את 100-200 transconjugants של כל הזדווגות.
האתר ההכנסה transposon מזוהה כל מוטציה על ידי קביעת רצף המוצר PCR שנוצר על ידי ה-PCR אקראי למחצה שמ...
Discussion
למרות תוצאות שלנו פיילוט עבור hamster תיגר intraperitoneally עם מוטציות ל' interrogans 42 מוצגים17, אנו מצפים כי בריכות גדולות יותר של מוטציות יכולים להיות מוקרן על ידי Tn-תת סעיף. בגלל התדירות של transconjugants הוא נמוך (100-200 transconjugants/הזדווגות), מספר matings נחוצים ליצור מספר מספיק של מוטציות לניסויים Tn...
Disclosures
המחברים אין לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי פרס הצטיינות לענייני חיילים משוחררים (עד D.A.H.), המכון הלאומי לבריאות להעניק R01 AI 034431 (D.A.H.).
Materials
| Name | Company | Catalog Number | Comments |
| Kanamycin sulfate from Streptomyces kanamyceticus | Sigma-Aldrich | K4000 | |
| 2,6-diaminopimelic acid | Sigma-Aldrich | D1377 | |
| Spectinomycin dihydrochloride pentahydrate | Sigma-Aldrich | S4014 | |
| Axio Lab A1 microscope with a darkfieldcondenser | Zeiss | 490950-001-000 | |
| DNeasy blood and tissue kit | Qiagen | 69504/69506 | |
| MinElute PCR Purification | Qiagen | 28004/28006 | |
| QIAquick PCR purification kit | Qiagen | 28104/28106 | |
| Model 505 Sonic Dismembrator | Fisher Scientific | FB-505 | |
| 2.5" Cup horn | Fisher Scientific | FB-4625 | |
| Bead Ruptor 24 | Omni International | 19-010 | Step 3.1.2.4 |
| Terminal deoxynucleotidyl transferase | Promega | M828C | |
| Master mix Phusion | Thermo Scientific | F531 | Preparation of genomic libraries, step 3.4. |
| DreamTaq Master Mix | Thermo Scientific | K9011/K9012 | Identification of the transposon insertion site, step 1.2. |
| dCTP | Thermo Scientific | R0151 | |
| ddCTP | Affymetrix/ USBProducts | 77112 | |
| T100 Thermal cycler | BioRad | 1861096 | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | step 3.6. |
| Qubit dsDNA HS assay kit | Invitrogen | Q32851/Q32854 | step 3.6. |
| Qubit assay tubes | life technologies | Q32856 | step 3.6. |
| PBS pH 7.2 (1X) | Gibco | 20012-027 20012-050 | |
| Disposable scalpel No10 | Feather | 2975#10 | |
| Plastic K2 EDTA 2 ml tubes | BD vacutainer | 367841 | |
| syringe U-100 with 26G x ½” needle | BD vacutainer | 329652 | IP challenge, step 2.2.1. |
| 3 mL Luer-Lok tip syringe | BD vacutainer | 309657 | Cardiac puncture, step 2.4.2. |
| 25G X 5/8” needle | BD vacutainer | 305901 | Cardiac puncture, step 2.4.2. |
| 25 mm fritted glass base with stopper | EMD Millipore | XX1002502 | Filtration unit system, step 1.1.7. |
| 25 mm aluminum spring clamp | EMD Millipore | XX1002503 | Filtration unit system, step 1.1.7. |
| 15 ml borosilcate glass funnel | EMD Millipore | XX1002514 | Filtration unit system, step 1.1.7. |
| 125 ml side-arm Erlenmeyer flask | EMD Millipore | XX1002505 | Filtration unit system, step 1.1.7. |
| Acetate-cellulose filter VVPP (pore size 0.1 mm; diameter 25 mm) | EMD Millipore | VVLP02500 |
References
- Murray, G. L., et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infect Immun. 77 (2), 810-816 (2009).
- Adler, B., Lo, M., Seemann, T., Murray, G. L. Pathogenesis of leptospirosis: the influence of genomics. Vet Microbiol. 153 (1-2), 73-81 (2011).
- Marcsisin, R. A., et al. Use of a high-throughput screen to identify Leptospira mutants unable to colonize the carrier host or cause disease in the acute model of infection. J Med Microbiol. 62, 1601-1608 (2013).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat Methods. 6 (10), 767-772 (2009).
- van Opijnen, T., Lazinski, D. W., Camilli, A. Genome-wide fitness and genetic interactions determined by Tn-seq, a high-throughput massively parallel sequencing method for microorganisms. Curr Protoc Microbiol. 36, 1E.3.1-1E.3.24 (2015).
- Troy, E. B., et al. Understanding barriers to Borrelia burgdorferi dissemination during infection using massively parallel sequencing. Infect Immun. 81 (7), 2347-2357 (2013).
- Wunder, E. A., et al. Real-time PCR reveals rapid dissemination of Leptospira interrogans after intraperitoneal and conjunctival inoculation of hamsters. Infect Immun. 84 (7), 2105-2115 (2016).
- Lourdault, K., Aviat, F., Picardeau, M. Use of quantitative real-time PCR for studying the dissemination of Leptospira interrogans in the guinea pig infection model of leptospirosis. J Med Microbiol. 58, 648-655 (2009).
- Coutinho, M. L., et al. Kinetics of Leptospira interrogans infection in hamsters after intradermal and subcutaneous challenge. PLoS Negl Trop Dis. 8 (11), e3307 (2014).
- Athanazio, D. A., et al. Rattus norvegicus as a model for persistent renal colonization by pathogenic Leptospira interrogans. Acta Trop. 105 (2), 176-180 (2008).
- Ratet, G., et al. Live imaging of bioluminescent Leptospira interrogans in mice reveals renal colonization as a stealth escape from the blood defenses and antibiotics. PLoS Negl Trop Dis. 8 (12), e3359 (2014).
- Gutierrez, M. G., Yoder-Himes, D. R., Warawa, J. M. Comprehensive identification of virulence factors required for respiratory melioidosis using Tn-seq mutagenesis. Front Cell Infect Microbiol. 5 (78), (2015).
- Gawronski, J. D., Wong, S. M., Giannoukos, G., Ward, D. V., Akerley, B. J. Tracking insertion mutants within libraries by deep sequencing and a genome-wide screen for Haemophilus genes required in the lung. Proc Natl Acad Sci U S A. 106 (38), 16422-16427 (2009).
- Gallagher, L. A., Shendure, J., Manoil, C. Genome-scale identification of resistance functions in Pseudomonas aeruginosa using Tn-seq. MBio. 2 (1), (2011).
- Slamti, L., Picardeau, M. Construction of a library of random mutants in the spirochete Leptospira biflexa using a mariner transposon. Methods Mol Biol. 859, (2012).
- Picardeau, M. Conjugative transfer between Escherichia coli and Leptospira spp. as a new genetic tool. Appl Environ Microbiol. 74 (1), 319-322 (2008).
- Lourdault, K., Matsunaga, J., Haake, D. A. High-throughput parallel sequencing to measure fitness of Leptospira interrogans transposon insertion mutants during acute infection. PLoS Negl Trop Dis. 10 (11), e0005117 (2016).
- Ellinghausen, H. C., McCullough, W. G. Nutrition of Leptospirapomona and growth of 13 other serotypes: Fractionation of oleic albumin complex and a medium of bovine albumin and polysorbate 80. Am J Vet Res. 26, 45-51 (1965).
- Johnson, R. C., Harris, V. G. Differentiation of pathogenic and saprophytic letospires. I. Growth at low temperatures. J Bacteriol. 94 (1), 27-31 (1967).
- Demarre, G., et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPa) conjugative machineries and their cognate Escherichia coli host strains. Res Microbiol. 156 (2), 245-255 (2005).
- Vallenet, D., et al. MicroScope: a platform for microbial genome annotation and comparative genomics. Database (Oxford). , bap021 (2009).
- Goodman, A. L., Wu, M., Gordon, J. I. Identifying microbial fitness determinants by insertion sequencing using genome-wide transposon mutant libraries. Nat. Protocols. 6 (12), 1969-1980 (2011).
- Miller, J. . Spirochetes in body fluids and tissues manual of investigative methods. , 22-23 (1971).
- Ristow, P., et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathog. 3 (7), (2007).
- Croda, J., et al. Targeted mutagenesis in pathogenic Leptospira species: disruption of the LigB gene does not affect virulence in animal models of leptospirosis. Infect Immun. 76 (12), 5826-5833 (2008).
- Haake, D. A. Hamster model of leptospirosis. Curr Protoc Microbiol. 12, 2 (2006).
- Giardine, B., et al. Galaxy: a platform for interactive large-scale genome analysis. Genome Res. 15 (10), 1451-1455 (2005).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 11 (8), R86 (2010).
- Blankenberg, D., et al. Galaxy: a web-based genome analysis tool for experimentalists. Curr Protoc Mol Biol. (Chapter 19), 11-21 (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Res. 8 (44), (2016).
- McCoy, K. M., Antonio, M. L., van Opijnen, T. MAGenTA; a Galaxy implemented tool for complete Tn-Seq analysis and data visualization. Bioinformatics. , 1367 (2017).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Kamp, H. D., Patimalla-Dipali, B., Lazinski, D. W., Wallace-Gadsden, F., Camilli, A. Gene fitness landscapes of Vibrio cholerae at important stages of its life cycle. PLoS Pathog. 9 (12), e1003800 (2013).
- Poggi, D., Oliveira de Giuseppe, P., Picardeau, M. Antibiotic resistance markers for genetic manipulations of Leptospira spp. Appl Environ Microbiol. 76 (14), 4882-4885 (2010).
- van Opijnen, T., Camilli, A. Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms. Nat Rev Microbiol. 11 (7), 435-442 (2013).
- Pappas, C. J., Picardeau, M. Control of gene expression in Leptospira spp. by Transcription activator-like effectors demonstrates a potential role for LigA and LigB in Leptospira interrogans virulence. Appl Environ Microbiol. 81 (22), 7888-7892 (2015).
- Koskiniemi, S., Sun, S., Berg, O. G., Andersson, D. I. Selection-driven gene loss in bacteria. PLoS Genet. 8 (6), e1002787 (2012).
- Troy, E. B., et al. Global Tn-seq analysis of carbohydrate utilization and vertebrate infectivity of Borrelia burgdorferi. Mol Microbiol. 101 (6), 1003-1023 (2016).
- Ramsey, M. E., et al. A high-throughput genetic screen identifies previously uncharacterized Borrelia burgdorferi genes important for resistance against reactive oxygen and nitrogen species. PLoS Negl Trop Dis. 13 (2), e1006225 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved