Method Article
דם מבוסס מבחן איתור ROS1 ואת RET פיוז'ן תעתיקים מלהסתובב חומצה ריבונוקלאית באמצעות תגובת שרשרת פולימראזית דיגיטלי
In This Article
Summary
זיהוי של מחזורי חומצה ריבונוקלאית (קרנה) מהדם נמצא צורך בתוכנית האבחון הקליני. כאן נתאר שיטות המאפיינות את קרנה של חולי סרטן ריאות שאינם קטנים התא באמצעות תגובת שרשרת של פולימראז דיגיטלי רגיש וספציפי. הבדיקות בדרישות עיצוב לאתר גרסאות פיוז'ן בתוך 72 שעות.
Abstract
פיתחנו שיטות הרומן בידוד ואפיון של נגזר הגידול במחזור חומצה ריבונוקלאית (קרנה) לביופסיה נוזלי מבוסס-דם. זיהוי חזקים קרנה התאוששה דם מייצג פתרון צורך קריטי בתוכנית האבחון הקליני. הבדיקה מתחילה עם האוסף של דם לתוך צינורות איסוף דם המכילים חומרים משמרים זה לייצב את קרנה. ללא תא, exosomal, ו- RNA טסיות-הקשורים מנותקת פלזמה במערכת זו הבדיקה. קרנה היא הפוכה עיבד ל- DNA משלימים (cDNA) מוגבר באמצעות תגובת שרשרת של פולימראז דיגיטלי (dPCR). דוגמאות מוערכים הן את סמן יעד, כמו גם גן הבקרה. בדיקת אימות כלל מגבלה של זיהוי, דיוק חוסן מחקרים עם דגימות אנליטית. השיטה פותחה בעקבות מחקרים אלו reproducibly לזהות מספר גרסאות פיוז'ן ROS1 (C-Ros proto-oncogene גרסאות 1; 8), RET (לסידור מחדש במהלך תרביות תאים proto-oncogene; 8 גרסאות). זרימת העבודה לדוגמה עיבוד מוטבה כך ניתן להפיק תוצאות הבדיקה באופן עקבי בתוך 72 שעות מיום קבלת מדגם.
Introduction
עד 25% של סרטן ריאות תאים שאינם קטנים (NSCLC) חולים אולי לא יש זמינות לבדיקה בעת ביצוע אבחון מספיק רקמות. אפילו במקרים בהם רקמה זמין, זה לא מספיק כמות או איכות לביצוע המומלצים בדיקות מולקולריות1,2. במקרים שבהם יש מספיק רקמה של ביופסיה עבור פרופיל מולקולרית, חולים שיש לחכות מספר שבועות או יותר לקבלת תוצאות, או להתחיל טיפול ללא תוצאות מולקולרית3,4. עם זאת, קריטי כי אבחון מולקולרי אינפורמטיבי יהיו זמינים בהתחשב כניסתו של אפשרויות טיפול ממוקד עבור חולים שאובחנו עם NSCLC. בדיקה של מחזורי ללא תא הדנ א (cfDNA) ביופסיה נוזלי הוא פתרון לאתגרים של רקמה מסורתית בדיקות4,5,6. אפשרויות הבדיקה הנוכחית מוטציות שבידיך NSCLC באמצעות cfDNA ו זרימת עבודה המבוסס על dPCR דומה ' לדור תוצאה מהירה, כוללים את קולטן גורם הגדילה באפידרמיס (EGFR) sensitizing מוטציות ΔE746-A750, L858R, EGFR ההתנגדות מוטציה T790M , גרסאות קראס proto-oncogene (קראס), B-Raf proto-oncogene (BRAF) משתנה V600E. אמנם לא כמו בהרחבה אומצו על-ידי השדה במחזור הגידול-derived RNA שליח (mRNA) מבודד ביופסיה נוזלי יכול גם לספק מידע קליני חשוב7,8,9. יש קודם לכן פיתחה ואנו דיווחו על שיטות זיהוי מרובבת קווצי עור Microtubule הקשורים חלבון כמו 4-אנפלסטית לימפומה קולטן טירוזין קינאז (EML4-ALK) פיוז'ן וריאנטים פלזמה דם10. במחקר זה, אנחנו האריכו שיטות אלה לכלול יעדים RNA מרובבת מסדר גבוה עבור ROS1 ורט, המכסה שמונה גרסאות פיוז'ן בתוך כל assay. המטרה הייתה לפתח טכניקה מהירה, רגיש, ספציפית, לשחזור איתור אלה גרסאות פיוז'ן של הפלזמה של חולים שאובחנו בעבר עם NSCLC.
תהליך הבדיקה מאותחלת במשרד של רופא בעזרת RNA ייצוב צינורות איסוף דם11. צינורות אלה מכילים משמרים כמו גם מעכבי RNase תא. הדגימות עדיפות שנשלחה ונישלח מרכזי המכללה של קאפ פתולוגים אמריקאי מוכר/קלינית מעבדה שיפור התיקונים (CLIA)-מעבדה מוסמכת (קליני מעבדתי) לעיבוד על-ידי כוח אדם מוכשר. ברגע שהתקבלו על-ידי מעבדה קלינית, כל שלב של עיבוד מתנהל תחת נהלי שאושרו (SOP). דם מלא הוא centrifuged לשחזר את הפלזמה, אשר משמש לאחר מכן כדי לבודד במחזור ה-RNA זה הוא או חופשי בדם או בתוך לבצע moieties, כגון exosomes וטסיות7,8,9. כדי לבודד RNA של תאים אלה, בחרנו את המערכת להתאוששות RNA המבוסס על השוואות של מספר שיטות החילוץ. הרנ א מבודד הוא מרוכז, הפוכה עיבד כדי cDNA. מספר אנזימים רוורס טרנסקריפטאז, גנים ספציפיים תחל הוערכו בזמן אופטימיזציה של שיטת סינתזה cDNA להגדיל את ROS1 ואת רט היעד התעתיק ההמרה10. . זה קריטי עבור שפע נמוך במחזור תעתיקים, כגון גרסאות פיוז'ן נגזר הגידול. בסופו של דבר, אנחנו אופטימיזציה ריכוזים פריימר ולאחר בדיקה dPCR כדי לאפשר זיהוי מרובבת של גרסאות פיוז'ן רט או ROS1, הגן בקרה, glucuronidase-β (GUSB). אנחנו ואז לשלב את התנאים הטובים ביותר מכל המחקרים אופטימיזציה פרוטוקול נעול הסופי לפני ביצוע האימות אנליטית המחקרים שתוארו בדו ח זה. פרוטוקול זה ותוצאות אלה מספקים את הבסיס עבור זרימת עבודה מהיר ורגיש איתור שגרתית גרסאות פיוז'ן נדיר במחזור.
Protocol
היצרנים ההנחיות עבור ריאגנטים המפורטות להלן, אלא אם כן שתואר אחרת. מבחני ה-PCR הם מוצרים זמינים מסחרית תוכננה לאתר fusions ROS1 ו רט.
1. עבודה עם ה-RNA כהכנה שעתוק במהופך (RT)-dPCR: מעבדה מומלצות
- ליצור סביבה נטולת RNase בעת עבודה עם ה-RNA.
- השתמש תרסיסים זמינים מסחרית שנועד להשבית RNases מזהמים.
- שימוש מוסמך RNase ללא ריאגנטים, טיפים, וצינורות. השתמש בעצות מכשול עבור ערכות-בדיקות ביוץ למניעת החדרת RNases או זיהום צולב של דגימות.
- תמיד ללבוש מעיל מעבדה כדי למנוע נפילה בין בגדים לתוך דגימת חלקיקים. לייעד את המעיל מעבדה ספציפי לשימוש עם עיבוד RNA.
- ללבוש כפפות כדי למנוע זיהום הדגימה מ RNases בעור. משתנים לעתים קרובות כפפות.
הערה: נניח מעבדה משטחים מזוהמים עם RNase מאז הם חשופים לסביבה. כפפות לפנות העור, השיער, ידיות, ידיות מקפיא, עטים/סמנים, וכו ' הם ההנחה תהיה כבר לא נטולת RNase. - Decontaminate פיפטורים, benchtops, צנטריפוגות ומשטחי עבודה אחרים עם ספריי איון RNase לכהן להשתמש.
- במידת האפשר, לשמור על ערכה של ציוד לשימוש עם ה-RNA בלבד.
- למזער שיבושים של זרימת האוויר באזורים מעבדה בעת עבודה עם דגימות RNA כדי למנוע חלקיקים מנפילה לתוך דוגמאות או מזהם את משטח העבודה.
- חנות טהור RNA ב-80 הלעפה תרוטרפמט.
- הימנע ההקפאה-thaws מרובים של דגימות ה-RNA, כמו זה יכול לגרום השפלה.
2. דור של חומר ה-RNA האנליטי עבור הפקדים חיובי
- עיצוב באמצעות DNA סינטטיים לאור רצפי mRNA גרסאות פיוז'ן של ריבית10.
- פיוז'ן נתון משתנה, בחר מרצף פיוז'ן mRNA הכוללת את אתר פיוז'ן בתוספת אורך מספיק איגוף מכל צד כדי לכסות את אמפליקון PCR.
- בחר רצפי בין 50-250 nt כדי לחקות את הגודל של מחזורי RNA שנלכדה באמצעות פלסמה מועשרת טסיות.
- הוספת רצף יזם T7 (5'-CAGAGATGCATAATACGACTCACTATAGGGAGA-3') 5' בסוף הרצף היעד.
- סדר רצפים סינתטיים כמו חומצה deoxyribonucleic נטושים כפול (DNA) קטעים.
- לשקם דנ א סינתטי במאגר טריס-EDTA (TE) כדי ריכוז סופי של 10 ng/µL.
- להמיר 60 דנ א סינתטי ng באמצעות במבחנה שעתוק RNA.
- לטהר תעתיקים RNA באמצעות ריאגנט מבוססות-פנול/guanidine12.
- כוללים DNase אני, נטולת RNase כדי להסיר תבנית שרידי דנ א.
- למדוד ריכוז מטוהרים במבחנה RNA באמצעות fluorometer זמינים מסחרית עם צבעי RNA ספציפיים ותקנים. ודא שהרנ א נמצא בטווח מקובל בסטנדרטים שבחרת. דילול עשוי להידרש.
- לאשר תמלול מוצלחת על ידי אלקטרופורזה בג'ל באמצעות ג'ל agarose 2% מעורבב עם הכתם ג'ל RNA ומגוון גבוה בסולם הרנ א כולל 50-250 nt גודל.
- עומס 500 ננוגרם של כל במבחנה -RNA על גבי ג'ל.
- הפעל את הג'ל של 5 V/ס מ.
- דמיינו להקות יחיד באמצעות תאורה, ולתעד את התוצאות.
- אשר תעתיק הצפוי גודל עבור כל אחד גרסאות פיוז'ן (מבוסס על עיצוב בשלב 2.1.2).
- לאשר זיהוי של כל במבחנה רנ א על ידי שימוש assay PCR הספציפי משתנה בהתאמה RT-dPCR (ראה שלבים 5-8 של פרוטוקול זה).
- אופציונלי: להכין תערובת equimolar של במבחנה RNA המכיל כל אחד על גרסאות פיוז'ן, הגן שליטה GUSB.
- אם מתבצע שלב 2.9: לאשר את זיהוי של כל אחד גרסאות פיוז'ן כלול את התערובת שליטה על-ידי dPCR באמצעות מבחני ה-PCR הספציפי משתנה (ראה שלבים 5-8 של פרוטוקול זה).
- לקבוע ריכוז קלט הרצוי עבור פקדים חיובי אנליטית על-ידי בדיקת ריכוזים ועד 0.25 2.5 fg10. לבחור ריכוז בהתבסס על פלט מספר העותק המבוקש.
- בעקבות אישור, להכין 10 µL aliquots לשימוש יחיד של RNA אנליטית לשימוש בקרה חיובית (שלב 4.4), חנות ב-80 הלעפה תרוטרפמט.
3. תורם דגימות
- איסוף דגימות דם כל אדם 10 מ ל 10 מ ל דם אוסף צינורות (BCT) המכיל כחומר משמר RNA נטול תאים.
הערה: כל התורמים האנושי יהיה מחקר שימוש ומאשר לא תורם ספציפי או פרטים מזהים יהיה להיות שנאספו בשימוש במהלך הבדיקה. - תהליך דגימות דם בתוך מסגרת הזמן שצוין על-ידי היצרן BCT.
- במאגר לפלסמה אנושית רגילה ניתן לרכוש ממקור מסחרי לשימוש בתוך הפקד חיובי אנליטית. להכין לשימוש יחיד, aliquots 1 מ"ל של במאגר לפלסמה אנושית רגילה, חנות ב-80 הלעפה תרוטרפמט לשימוש עם הפקד חיובי (שלב 4.4).
4. שחזור של מחזורי RNA של פלזמה
הערה: חשוב לעבוד במהירות במהלך הליך זה.
- צנטריפוגה צינורות דם מלא-200 g x עבור 20 דקות.
- לאסוף עד 4 מ"ל של פלזמה דם centrifuged אוסף שפופרת באמצעות פיפטה סרולוגית. יש להיזהר לא להפריע או תשאף השכבה המעיל באפי.
- לבודד במחזור RNA באמצעות ערכת זמינים מסחרית כי יכול ללכוד exosomes, טסיות דם, ללא תא RNA של פלזמה. לבודד RNA מהדגימה בקרה חיובית לצד כל אצווה.
-
להכין את הפקד חיובי כל אצווה של דגימות קליניות כדלקמן:
- הפשרת 1 מ"ל איחדו נורמלי לפלסמה אנושית aliquot (שלב 3.3).
- הפשרת 10 µL אנליטית RNA aliquot (שלב 2.12).
- להכין בקרה חיובית על-ידי הוספת 10 µL אנליטית RNA לתוך הדגימה לפלסמה אנושית רגילה, ברגע אתנול נוספה פלזמה lysate.
- Elute דגימות מים נטולי נוקלאז µL 100. פנה מיד עם RNA להתנקות וריכוז.
- דוגמאות עשויים להיות מאוחסנים על קרח רטוב, מכוסה, במשך שעה אחת.
- להתרכז RNA בשיטת עמודה מבוסס, elute ב µL 9 RNase ללא מים.
- מיד להמשיך בשלב 5, או לשמור דגימות על קרח רטוב במשך שעה אחת.
5. הפוך את שעתוק RNA ל cDNA
- המר מרוכז מדגם ה-RNA במחזור cDNA באמצעות ערכת התגובה שעתוק במהופך זמינים מסחרית, לרבות תחל אקראי (ראה טבלה 1 עבור רכיבים).
הערה: גנים ספציפיים תחל אופציונלי, ניתן לעצב עבור המבחן משתנים. תחל מעוצבים בהתבסס על המטרה רצף ה-RNA. השתמש פיוז'ן רצפים משתנה מ- 2.1 צעד.- כוללים לדוגמה שליטה אין רוורס טרנסקריפטאז ודגימת שליטה אין RNA (ראה טבלה 1).
- לבודד cDNA מתגובה שעתוק במהופך באמצעות עמודה ספין זמינים מסחרית של רכז הדנ א.
הערה: שלב זה מאפשר הסרה של אנזימים, תחל deoxynucleotide חינם triphosphates (dNTPs). - השתמש cDNA מיד ב- PCR תגובות או לאחסן ב-80 הלעפה תרוטרפמט.
6. PCR דיגיטלי
הערה: זו PCR היא ספציפית droplet PCR דיגיטלי (ראה טבלה של חומרים).
-
PCR מערבבים אמצעי זהירות.
- ללבוש כפפות מעילו ואת nitrile המעבדה חד פעמיות.
- שימוש PCR מערבבים ריאגנטים באזור הכנה ריאגנט ייעודי. לא מסתדר עם cDNA באזור הכנה ריאגנט בלבד.
- מכסים הגששים תוך כדי עבודה כדי להגן עליהם מפני אור. אור מופרז יכול צילום להלבין את הפלורסנט מחובר המכשיר.
- תחבורה תערובות, מכוסה, מוגן מפני אור, לתוך אזור קדם הגברה נפרדת לפני cDNA שיתווספו.
- להוסיף cDNA להיבדק כדי לערבב PCR בשכונה נקי PCR ממוקם באזור קדם הגברה.
- היכונו PCR mixes נפח התגובה האחרונה של 20 µL על פי בטבלה 2.
- להפיץ PCR mixes + cDNA ללוחות ה-PCR.
הערה: שימוש במיתאר צלחת כמו מומלץ לקחת מדריך. - מכסים את הצלחת באמצעות סילר לאיטום של צלחת נשלף.
- צנטריפוגה צלחות בקצרה לאסוף דגימות בתחתית הבארות.
- תערובת על צלחת שאכר על קביעת ערך נמוך מ-10 s.
- צנטריפוגה צלחות בקצרה כדי לאסוף דגימת בתחתית הבאר.
- הסר את לוחית סילר לאיטום. לבצע יצירת droplet עבור ה-PCR-cDNA לערבב עם גם מערכת דור droplet ידנית או אוטומטית.
- לדור droplet ידנית, העברה מיקס PCR µL 20 דוגמת בארות על הדיו הדור ' droplet '. הוסף µL 70 של שמן דור droplet. מכסים עם גומי אטם והעברת מחסנית לגנרטור droplet ידנית ליזום דור droplet. בעקבות יצירת droplet, להעביר טיפות צלחת PCR טריים באמצעות טיפים מומלצים על ידי היצרן. האחות, לחלק את טיפות לאט, מעל 5 עד 6 s כל אחד, בלי לגעת הפתיחה של קצה droplet מחסנית או צלחת.
- לדור droplet אוטומטיות, חותם את הצלחת עם כשסרט האטימה, העברת הגנרטור droplet. להבטיח כל העצות, מחסניות, צלחות במקום לפני תחילת יצירת droplet.
- בעקבות droplet בדור העברה של טיפות על צלחת PCR טריים, חותם עם סילר לאיטום צלחת עם רדיד אלומיניום, לוחות המחזור התרמי תוך שימוש בהגדרות בטבלה3.
- לאחר הצנטרפוגה תרמי בניהול הושלמה, לקרוא את הצלחת שימוש בקורא droplet. ליצור פריסה צלחת עבור תוכנת קורא שמזהה את המיקום של פקדים, דגימות, וכדומה, עומס לתוך תוכנת להתחיל לקרוא.
7. נתוני ניתוח, סקירה של דור של תוצאות
- לנתח את הצלחת לקרוא את התוצאות באמצעות תוכנה זמינים מסחרית.
- נווטו לתפריט Analyze כדי להציג מתווה דו מימדי משרעת (2D).
- להעריך את האיכות הכוללת של הנתונים על-ידי בדיקת הנתונים droplet.
- הערכת הנתונים עבור מספרים הכוללת אירוע מקובל שימוש בתפריט Events. אם ישנם אירועים פחות מ 10,000 לכל היטב, בקפידה להעריך את הנתונים עבור בעיות נוספות.
- בדוק נתונים עבור קרינה פלואורסצנטית סוטה amplitudes. הבדלים משמעותיים משרעת והבדלים ריכוז בין דגימות שכפל מצביעים על טיפול עניים או ערבוב של דגימות.
- ליצור הערות של droplet אשכולות עם דפוסים ספריי על ציר 45 מעלות, הרומז על מצבה העגום של טיפות באיכות ירודה או דגימות בעייתי.
- בדוק קודם בקרה חיובית, לא רוורס טרנסקריפטאז (לא RT) ונתוני בקרה RNA לא (NRC). בחר כל שליטה דוגמאות ולבחון אשכול איכות על ידי מגרש 2D. על סף תקין, הפרדה ברורה בין אשכולות droplet בלאו הכי.
- עבור כל משתנה assay, להגדיר את הסף מבוסס על שליטה בארות.
- ספי קבע על מגרשים 2D באמצעות הכלי הכוונת כדי להפריד בין האוכלוסייה droplet כפול שלילי מן האוכלוסייה ג'ין שליטה (המסומנת phosphoramidite 5'-hexachloro-fluorescein-CE בדיקה), ציר ה-y, אוכלוסיית הגן variant, אם להציג ( עם תוויות fluorescein amidite או 6-carboxyfluorescein בדיקה), ציר ה-x.
- סכום עותקים מכל קידוח שכפל דגימה בודדת.
- מבטאים את תוצאות הבדיקה כמו מספר העותקים variant זוהה.
הערה: כדי לקבוע את ערך הסף אנליטית שהתקשרת דגימה חיובית או שלילית, להפעיל רגיל מדגם תורם בריא קבע (לפחות 10 דוגמאות בודדות) בתהליך גיבוש ולהקים החיתוך מעל כל אות רקע לזיהוי בשביל המוטציה של ריבית. בנוסף, להקים את מספר העותקים ג'ין שליטה נדרש לקרוא תוצאה חיובית או שלילית. ניתוק ג'ין זה שליטה מתפקד בקרת איכות (QC) כדי להעריך את כמות ואיכות כל מדגם ה-RNA שמעובד פנימי.
8. אימות של תנאי ריאקציה RT-dPCR באמצעות שורות תאים (אופציונלי)
- כדי לוודא זיהוי של גרסאות פיוז'ן, השתמש שורות תאים זמינים מסחרית לבטא את פיוז'ן ROS1 או רט mRNA של עניין. פעל כדלקמן:
- Homogenize קפואה תאים בפתרון מבוסס-guanidinium פירוק ישירות ממצב למצב קפואים. מפשיר אפילו קצרה לפני המגון יכול לגרום RNA השפלה ואובדן.
- לבודד RNA תוך שימוש בעמודות ספין סיליקה-הממברנה מיועד RNA.
- למדוד את הריכוז של דגימות RNA באמצעות fluorometer עם ריאגנטים ספציפיים RNA ותקנים.
- לדלל RNA מבודד לתוך רקע של RNA פראי-סוג פלזמה או ממקור מסחרי אחר.
- לבצע צעדים מן הפוך שעתוק של רנ א cDNA, PCR דיגיטלי, ניתוח נתונים, סקירה, דור של תוצאות המופיעים בפרוטוקול זה כדי לאשר גילוי של הווריאציה הרצויה.
תוצאות
פרוטוקול זה מתאר מערכת הבדיקה שפותחה עבור הגילוי של RNA גרסאות פיוז'ן לשימוש בהמידה של מוטציות הנהג בתוך הפלזמה של חולי NSCLC (איור 1א'). פיוז'ן mRNA היו מוצרים מן הביטוי של rearrangements רט ו- ROS1 הנפוץ ביותר בקרב האוכלוסייה NSCLC מזוהה13,14,15,16,17. מבחני ה-PCR מרובבת תוכננו ואז לזהות על שמונה גרסאות התעתיק הנפוץ ביותר עבור כל מטרה NSCLC בתוך תגובה אחת. Translocations הנפוץ ביותר-מיקומה ROS1 ליצור שיוכי בחלקים 5' של CD74, SDC4, SLC34A2, EZR או TPM3 גנים (איור 1B). Translocations הנפוץ ביותר-מיקומה רט להוביל הסמיכות עם KIF5B, שעבורו assay מכסה שישה צמתים אקסון. שותפים רט נוספים מכוסים כוללים את אלה עם CCDC6 ו- TRIM33 (איור 1C). בסך הכל, מבחני מכסה כ- 88% של ROS1 ו- 99% של RET. שינויים עלולה להתרחש בתוך האוכלוסייה המטופלת NSCLC17.
ייחודה של הרכיבים assay הוערך קודם באמצעות שמונה בודדים במבחנה RNAs מכילים את רצף ה-mRNA לפי התרשימים פיוז'ן מכוסה על ידי ROS1 או רט מרובב מבחני. לכל מין RNA נבחנה נגד כל assay variant בודדים שכוללת את הגירסה מרובבת. היה לא אשיג של מבחני אלה, ובכך הפגנת 100% ירידה לפרטים אנליטית בתוך תוכנן מרובב מבחני (נתונים לא מוצג). כדי לקבוע את הגבול התחתון של זיהוי של פרוטוקול הבדיקה, הכולל RNA נגזר שורות תאים ביטוי variant פיוז'ן הכלולים וזמינותו היו מעורבבות רקע של RNA נורמלי בריכוזים 5%, 1%, 0.2% ו- 0.04%. מרובבת רט ו ROS1 variant PCR מבחני זוהה רק 0.2% פיוז'ן variant (איור 2א-ב). בנוסף, הכנה של 5% הנחה-יעד שורת התאים נגזר RNA (ביטוי של התעתיק פיוז'ן EML4-ALK) לא זוהה עם מרובבת ROS1, RET מבחני, בהמשך מדגימים ירידה לפרטים (איור 2א-ב).
דיוק בדיקה של תהליך RT-dPCR בוצע עבור שני ROS1 ומעובדים מיל' אנליטית בקרת החומר מורכב equimolar במבחנה RNAs היה בריכוזים שלוש (גבוהה, בינונית, נמוכה) דרך שעתוק במהופך, dPCR על שלוש הזדמנויות שונות בתוך באותו היום (אינטרה-יום), על שלושה ימים רצופים (inter-יום), ועם שני אופרטורים (inter-מפעיל). תוצאות בדיקות הדיוק הפגינו זיהוי מדויקת של התעתיק פיוז'ן מעניינים, כמו גם בקרת גנים, GUSB, אשר נכלל בתור מדד QC פנימי (איור 2C-D).
בנוסף בקרה פנימית של GUSB, שכל אצווה של דגימות קליניות הופעלה עם קבוצה של פקדים אצווה. פקד חיובי פותחה מתערובת של אנליטית חוץ גופית ב RNA מיוצגים כל אחד גרסאות פיוז'ן נבדק ב- RT-dPCR, כמו גם אנליטית במבחנה RNA עבור GUSB. RNA הזה נמזג לפלסמה אנושית רגילה lysate במהלך החילוץ-RNA, עובדה לצד הדגימות קליניים לאורך כל הפרוטוקול. הפקד רוורס טרנסקריפטאז (לא RT) לא היה פקד שלילי כדי לאשר את העדר של חומרים מזהמים בתהליך מיצוי העבודה RNA ומדגימים יחודיות של תחל עבור ה-RNA. הפקד RT לא נוצר באמצעות מאותו חומר כמו הפקד חיובית, אך היא אינה כוללת אנזימים בתוך התגובה סינתזה cDNA. אין RNA הפקד (NRC) הוא פקד שלילי כדי לוודא העדר מזהמות תעתיקים ברכיבי התגובה של שעתוק במהופך. פקד זה הוכנס שזרימת העבודה בשלב סינתזת cDNA, מים נוספה בהתגובה במקום תבנית ה-RNA. הפקדים לא RT ו- NRC חייב להיות שלילי בשני ערוצים, אם תוצאות מדויקות כדי להיגאל. טבלה 1 מציגה את רכיבי התגובה שעתוק במהופך עבור כל פקד. דוגמאות של החלקות 2D עבור כל אחד מהפקדים הללו מוצגים עבור ROS1 (איור 3 A-C), מבחני מולטיפלקס RET (איור 3 E-G). גרסאות פיוז'ן אותרו באמצעות בדיקה amidite (FAM) fluorescein, מיוצגים לאורך ציר ה-y, ואילו הגן בקרה, GUSB, זוהתה באמצעות בדיקה 5'-hexachloro-fluorescein-CE phosphoramidite (הקסדצימאלי) והוא בציר ה-x. פקדים אצווה אלה היו העריכו במשך 21 ימים כדי לקבוע assay חוסן. טיפות חיובי fusion ו GUSB בקרת גנים טיפות נצפו עבור ROS1 ורט ב כל הרצפים 21 להורג במהלך המחקר (איור 3ד', H). כל הפקדים שלילית (לא RT ו- NRC) הניבו תוצאות שליליות על-פני כל 21 הימים (נתונים לא מוצג).
היכולת לפתור בעיות היא מרכיב קריטי בכל פרוטוקול הבדיקה כדי להפעיל את ההגדרה קליני מעבדתי. כאן, אנו מספקים דוגמאות מהעולם האמיתי של תוצאות תת אופטימלית באמצעות פרוטוקול RT-dPCR. הראשון הוא מגרש 2D לדוגמה הממחיש את חשיבות אין רוורס טרנסקריפטאז הפקד (איור 4א). בדוגמה זו, טיפות מוטציה חיובית נכחו למרות שלא היתה ללא המרה cDNA בשל חוסר אנזים. התוצאה הייתה ככל הנראה בגלל תחל dPCR הגברה דנ א גנומי את המטרה. במקרה זה, העיצוב של וזמינותו פורש-אינטרון תמנע הגברה של הדנ א. לחלופין, אנזים DNase נטולת RNase יכול לשמש כדי לחסל את ה-DNA מזהמים, אך זה לא מומלץ לצורך זיהוי של מטרות נדיר, כי קצת השפלה RNA עלולה להתרחש במהלך הדגירה עם האנזים. העלילה 2D הבא דוגמה היה NRC עם טיפות חיובי בשני ערוצים (איור 4B). זה הצביעו על זיהום בשלב מסוים בכיוונון RT-dPCR. במקרה זה, ההמלצה היא למחוק את כל ריאגנטים שעשויות להיות מזוהמים משמש בדיקות, ביסודיות decontaminate כל הציוד של בדיקה חוזרת עם רכיבי התגובה טריים. השלישי דוגמה 2D העלילה שהוצגו כמו תרסיס של טיפות לאורך קו 45° (איור 4C). זו נגרמת לעתים קרובות על ידי הטיית התגבשו של טיפות. זהירות droplet טיפול לפני רכיבה על אופניים תרמי חיוני, כמו טיפות הם נוטים לגרום נזק. אנו ממליצים על השימוש של דור droplet אוטומטית, כאשר היא זמינה. אם העברת ידנית שנוצר על ידי טיפות, להיות בטוח לבחור את הטיפים נשא רחב מומלץ ולהעסיק זהיר בטכניקה pipetting. העברת droplet דורש איטי והשאיפה ומקבלים, בכל מקום לקחת יותר מ 5-6 שניות, וזה חיוני כי הפתח עצה פיפטה לא לגעת הדיו droplet או טוב. כאשר מחלק, שמור על קצה פיפטה ברמת נוזלי, אעלה את זה לאט. כמו טיפות הם ויתרו (תצוגת וידאו להדגמה). הדוגמה מגרש 2D הסופי מדגים חוסר הפרדה בין האוכלוסיות droplet חיוביים ושליליים (איור 4D). זה יכול להיות מספר סיבות. מעכבי PCR חזקה, כגון דטרגנטים בשימוש פירוק מאגרי עודף של הדנ א פגום מאוד, יכול לגרום לאובדן של הפרדה. במקרה זה, שקול להוסיף צעד לנקות בין cDNA סינתזה dPCR (כגון המתוארות בתוך בשלב 5 של פרוטוקול זה). בסופו של דבר, חוסר ההפרדה ניתן גם בשל תנאי הגברה תת אופטימלית, אופטימיזציה של השלב ה-PCR צריך להיות גם נחשב.
נתונים בתוך איור 5 מייצגים 984 בעולם האמיתי החולה לטעום סיבוב פעמים ומדגים את אופי זרימת עבודה זו בדיקה מהירה. תוצאות דווחו על הרופא המטפל מוקדם כמו בתוך 48 שעות (79% מהמקרים) קבלת דגימות ו ב 95% מהמקרים, בתוך 72 שעות. לסיכום, השימוש התייצב במחזור ה-RNA דם אוסף צינורות, RNA ממוטבת מיצוי הליכים מן הדם, ו- RT-dPCR לפעול על-פי פרוטוקול ממוטבת עם המתאים פנימי ופקדי אצווה, יכול לספק מערכת בדיקה מהירה זיהוי מדויק של פיוז'ן RNA משתנים רלוונטיים ב NSCLC.
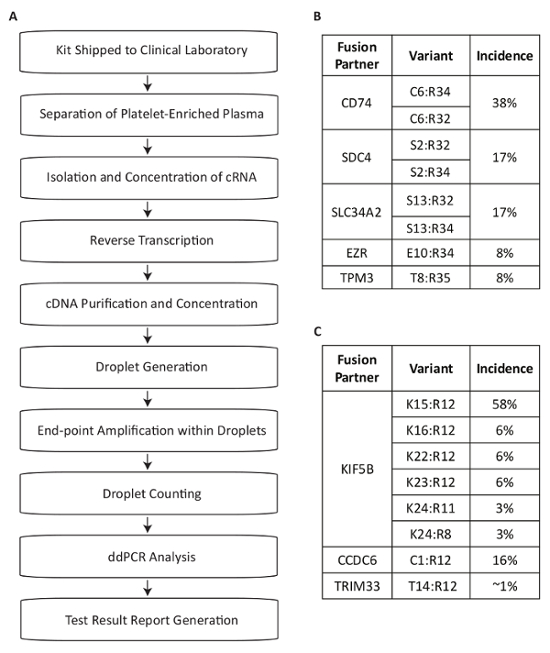
איור 1 : סקירה של דם דוגמת עיבוד השלבים לגילוי Variant היתוך באמצעות מבחני ספציפיים ביותר ונפוצים RET, גרסאות ROS1 ב NSCLC. (א) בדיקה של דגימות מתחילה כאשר דם משורטט, BCT נשלח בתוך ערכת איסוף הדגימה למעבדה קלינית. במחזור ה-RNA הוא התאושש ממקורות מרובים בתוך פלזמה מועשרת טסיות, הפוכה עיבד עם גנים ספציפיים לקרקע, מטוהרים לשימוש ב- dPCR. דוגמאות מעובדות באמצעות מערכת זמינים מסחרית, אשר מורכב של droplet דור (אמולסיה), הגברה וספירת droplet. ניתוח המידע באמצעות תוכנה זמינים מסחרית. תוצאות הבדיקה ואז מתועדים, דיווח על חזרה לרופא. מבקש בדיקה. התהליך מיועד לפעול בתוך פרק זמן של 72 שעות מעת קבלת דגימות למהדורה תוצאה. שמונה גרסאות עבור ROS1 (B) ו- (ג) RET מכוסים בתוך מבחני מרובבת. הותאם מהאתר Biodesix עם הרשאה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
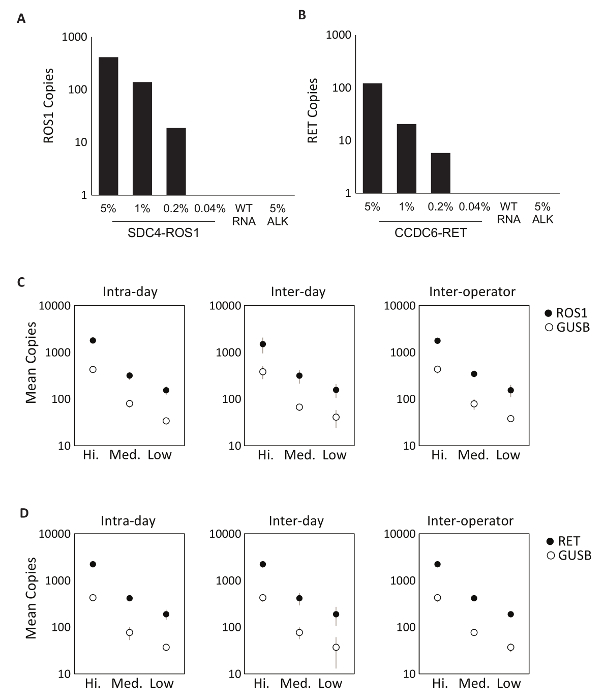
איור 2 : אימות אנליטית. שורות תאים לבטא (א) SDC4-ROS1 פיוז'ן ופיוז'ן CCDC6 (B)-RET היו מעורבבת עם רקע של RNA פראי-סוג האדם הכולל (WT RNA). עם כל משתנה פיוז'ן, המגבלה של זיהוי הוקמה באמצעות קריטריונים מוגדרים מראש עבור כל assay משתנה תדר משתנה של 0.2%. כל הדגימות מעל הסף הזה הכיל גם עותקים לפחות 21 של בקרת גנים. תקן EML4-ALK (ALK) 5% על רקע של RNA פראי-סוג נבחנה להדגים ירידה לפרטים assay, אשר אושר ע י תוצאה שלילית. סטנדרטים אנליטיים RNA מרובבת נמדדו ב גבוהה, בינונית, ריכוזים נמוכים עבור ROS1 (ג) ו- (ד) מיל' דיוק הוערך מעל שלושה מסלולי באותו יום (אינטרה-יום), פועל שלוש על שלוש יממות (inter-יום), ועם מפעיל עצמאי שני (inter-מפעיל). אמצעי העתקה מספר ואת סטיות תקן מוצגים. הותאם מהאתר Biodesix עם הרשאה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
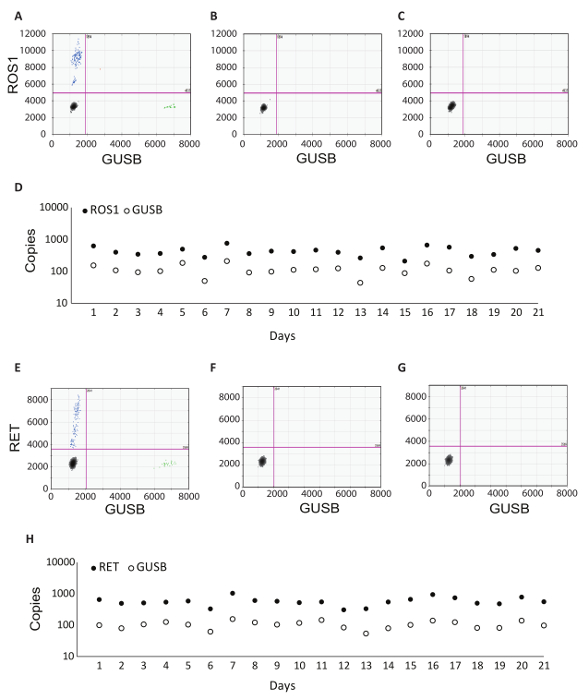
איור 3 : עיבוד שליטה דוגמאות ונתונים חוסן אצווה. 2D חלקת dPCR assay ROS1 מרובב תוצאות עבור שליטה חיובית (א), (B) אין רוורס טרנסקריפטאז ומערכת בקרת (C) אין שליטה תבנית הרנ א. פקדים (D) היו להפעיל על 21 ימים רצופים (למעט בסופי שבוע ובחגים). התכוונתי עותק מספר + /-סטיות תקן ROS1 חיובי הבקרה היו 439 + /-141. אין רוורס טרנסקריפטאז ופקדים תבנית לא היו גם להפעיל בכל יום, אלה היו כל שלילי (נתונים לא מוצג). 2D חלקת dPCR assay רט מרובב תוצאות עבור שליטה חיובית (E), (F) אין רוורס טרנסקריפטאז בקרה, (G) אין שליטה תבנית הרנ א. פקדים (H) היו להפעיל על 21 ימים רצופים (למעט בסופי שבוע ובחגים). התכוונתי עותקים + /-סטיות תקן רט חיובי הבקרה היו 586 + /-182. לא מוצגות אין רוורס טרנסקריפטאז ופקדים תבנית לא היו גם להפעיל בכל יום, היו שליליות. הותאם מהאתר Biodesix עם הרשאה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
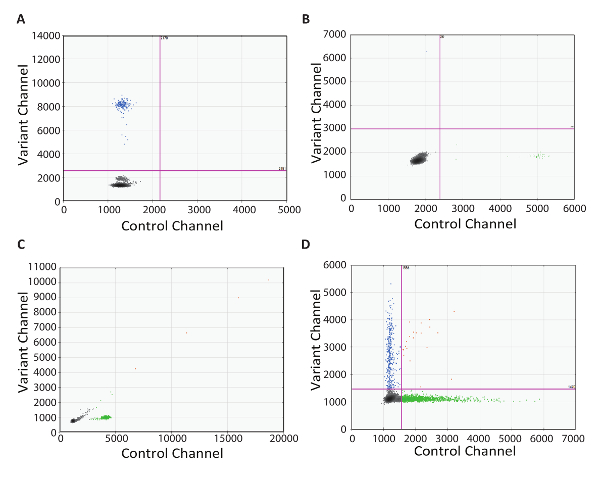
איור 4 : פתרון בעיות RT-dPCR. 2D מתווה וייצוג dPCR תת אופטימלית התוצאות המתקבלות כאשר יש זיהום () בתוך אין transcriptase הפוכה הפקד, זיהום (B) בתוך אין RNA הפקד, (ג) הטיה, התגבשו של טיפות, (D ) לקוי ממוטב PCR תנאים או עיכוב PCR. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
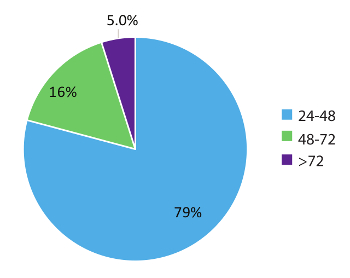
איור 5 : משך העבודה (TAT). תאת (בשעות) נערך עבור בדיקות מבקש של variant RNA (n = 984). נתונים לא כולל סופי שבוע, חגים וימי דגימות הוחזק > 24 שעות בשל מידע קליני לא שלם על טופסי בקשה בדיקת המעבדה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
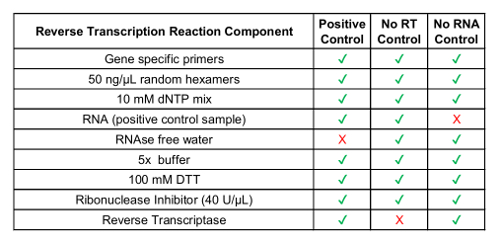
טבלה 1: הכנה של שעתוק במהופך ריאגנטים בקרות תהליכים.
| רכיב | נפח |
| 2 x dPCR supermix על הגששים (לא 2'-deoxyuridine 5'-טריפוספט) | 10 ΜL |
| 20 x היעד משתנה תחל/רגשים להגדיר (450 nmol/L תחל 250 בדיקה FAM nmol/L) | 1 ΜL |
| 20 x ערכת הבקרה היעד תחל/בדיקה (450 nmol/L תחל 250 בדיקה HEX nmol/L) | 1 ΜL |
| נוקלאז ללא מים | 1 ΜL |
| cDNA | 7 ΜL |
טבלה 2: הכנה של המיקס מאסטר dPCR.
| רכיבה על אופניים שלב | טמפרטורה | זמן | # מחזורים | כבש שער |
| הפעלת אנזים | 95 ° C | 10 דקות | 1 | ~ 2 oC/s |
| דנטורציה | 94 ° C | 30 s | 40 | |
| חישול/הרחבה | 55 ° C | 1 דקות | ||
| ביטול הפעלה של האנזים | 98 ° C | 10 דקות | 1 | |
| החזק (אופציונלי) | 4 ° C | אינסוף | 1 | ~ 1 oC/s... |
טבלה 3: תנאי הרכיבה תרמי.
Discussion
Rearrangements רט ו ROS1 ביחד יוצרים ~ 3% של מוטציות הנהג בתוך האוכלוסייה NSCLC18. למרות שזה נדיר, זיהוי שינויים גנטיים אלה היא חיונית. חולי NSCLC עם שינויים אלה עשויים להפיק תועלת הרפוי יישוב זה במיוחד לעכב את פעילות קינאז aberrant הנובעת onco-חלבון13. כמה טיפולים כאלה כבר המאושרים על ידי ה-FDA לשימוש ב NSCLC חיובית ROS1, בעוד אחרים הוכחו בתמימותה נגד רט ניסויים קליניים19.
טכנולוגיית ה-PCR דיגיטלית מספקת רגישות זה אידיאלי עבור יישומים ביופסיה נוזלי20. יש כבר אימוץ משמעותית של טכנולוגיה זו לשימוש עם מחזורי תא ללא ה-DNA של המדד של מוטציות הגידול בחולים עם NSCLC4,6,21,22,23 . בנוסף cfDNA, פיתחנו פרוטוקול המיועד למדידת חזקים גרסאות פיוז'ן הנפוצות ביותר בחולים עם NSCLC מלהסתובב הגידול RNA (איור 1א')10.
פרוטוקול הוקמה שלנו מאפשר לתחום האנליטי של זיהוי עד 0.2% (איור 2). בעוד RT-dPCR הוא ספציפי ורגיש במיוחד, מבחני מוגבלות הלוח של גרסאות פיוז'ן ידוע כי הם בחרו מרובב לצורך זיהוי ב- PCR וזמינותו. לפיכך, fusions תיכלל מבחני מרובבת יש לבחור בקפידה כדי להבטיח כיסוי הולם בתוך האוכלוסייה של חולים עם NSCLC. עיצבנו בהצלחה מבחני רט ו ROS1 בו זמנית לזהות שמונה תוצאות גרסאות פיוז'ן מן rearrangements של לוקוסים RET או ROS1, לכסות 99% 88% מן האוכלוסייה חיובי רט ו- ROS1, בהתאמה (איור 1בג )17.
המבחן האחרון זרימת העבודה כפי שמתואר במחקר זה כולל בקרת אצווה כדי להבטיח עקביות של תוצאות. פקדים אלה כוללות הן ברמה האנליטית חיוביים, כמו גם שני פקדים שלילי, המבטיחים ביחד אין זיהום או עיכוב PCR המתרחשים בתוך החבילה (איור 3). כדי להבטיח את היציבות של וזמינותו, בוצע מחקר באמצעות הפקדים אצווה במשך 21 יום (איור 3יח, ח). נתונים אלה מדגימים את העקביות של תהליך ה-RNA כפי שנקבעו במסגרת פרוטוקול זה.
שיטות מעבדה של הטיפול RNA הם מרכיבי המפתח של המבטיח תוצאות חזקות ומדויקות. שטח וציוד ייעודי לשימוש עם ה-RNA, ניקוי הציוד לאחר כל שימוש, בעזרת ריאגנטים RNase ללא חומרים מתכלים והחלת של ספריי איון RNase במרחב העבודה של כל לסייע בהפחתת RNases מזהמים. טיפול מצפוני של RNA דגימות על ידי טכנאים, כולל חלוק מעבדה ייעודית, תכופות שינויים הכפפות, עובדים במהירות על התהליך החילוץ RNA, והם לשמור דגימות על קרח בעל חשיבות עליונה לשמירה על שלמות הדגימה. ברגע RNA היה הפוך עיבד כדי cDNA, הדגימה היא בצורה יציבה יותר כי הוא נוטה פחות השפלה. בנוסף לשיטות העבודה התומכים RNA שלמות, רכיבי ה-PCR ודוגמאות צריך להישמר באזורים הפרדה כדי למנוע זיהום שעלול להוביל לתוצאות חיוביות כוזבות. ריאגנטים PCR מלאי והכנת mixes מאסטר PCR צריך להישמר נפרד מתבניות PCR, זהירות רבה יש לנקוט הפרדת תבנית מוגבר (post-PCR) של כל החומרים מראש מוגבר כולל ריאגנטים, רנ א cDNA דגימות. לבסוף, דור נכונה וטיפול mixes PCR emulsified לפני הגברה היא המרכזית לשמירה על תקינות droplet ותנאים אופטימליים dPCR. אמצעי זהירות כמו אלה הם קריטיים במהלך ביצוע של פרוטוקול זה כדי להשיג תוצאות עקבי ומדויק. כל הנתונים צריך להיבדק על ידי צוות מיומן לפני שחרורו של תוצאות כדי לוודא כי כל המדדים QC מולאו. במקרה של תוצאות שיוצרת (איור 4), האצווה חייב להיות נבדקו על ידי צוות טכני, מנהל המעבדה, עשוי לדרוש עיבוד מחדש.
RT-dPCR תוצאות יכול להיות מיוצר מוקדם ככל 24 שעות קבלת דגימות ו 95% של תוצאות המדגם בתוך המבחן לקבוע בשימוש במחקר זה (n = 984) דווחו לרופא ההזמנה תוך פחות מ- 72 שעות ממועד קבלת (איור 5). הפעם סיבוב מספקת לרופאים עם הרבה צריך מידע מולקולרית במסגרת זמן המאפשר חניכה של הטיפול המתאים. תוצאות אלו זמינים בדרך כלל מוקדם יותר מאלו שהושגו באמצעות ביופסיה רקמות קונבנציונלי. סמנים ביולוגיים נוספים NSCLC, סוגי סרטן אחרים ניתן לפתח באמצעות גישות מבוססות-RNA במחזור בדומה, ירוויחו באותו הזמן-כדי-תוצאות מהירות. לדוגמה, מדידה של תמליל mRNA מתוכנת מוות ליגנד 1 (PD-L1) באמצעות RT-dPCR יכול ליידע רופאים לגבי אפשרויות חיסוני. יש גם עניין הולך וגובר בכלי השירות של ביופסיה נוזלי dPCR בפיקוח על יעילות טיפולית. אינדיקציות קודמות של התחייה של הגידול באמצעות גנומית בדיקות עבור משתנים מסוימים עלולה לאפשר לרופאים להתאים משטרי הטיפול לפני המטופלים מפתחים סימפטומים על ידי תקן של אמצעי טיפול כגון הדמיה24. פרוטוקולים כמו האחד דיווחו במחקר זה הינם אידיאליים עבור ניטור שלהם הלא-invasiveness, רגישות, מהירה, סיבוב סביב הזמן וכתוצאה יחס עלות-תועלת. וזמינותו המתוארים בזאת מספק תוצאות תוך 72 שעות מעת קבלת דגימות, במחירים זיהוי חיובי-false מינימלי, אשר מקלה על החלטות לטיפול מהיר עוקף כמה מגבלות המנוסה המבוסס על רקמות ובדיקות4.
פרוטוקול ונתונים שלנו להפגין מערכת הבדיקה חזקים לזיהוי. גרסאות ה-RNA שפע נמוך, כמו גם את פוטנציאל מבוסס-דם מוטציה הבדיקה הקלינית. עבור אותם חולים אשר אין מנהל התקן שבידיך מוטציה מזוהה על ידי ביופסיה נוזלי יישוב מהיר גישות כמו זה, התוספת של הגנום מקיפה יותר, פרוטאום בדיקות רקמות והן דם עשויים לספק מידע קליני אפילו רחבה יותר כדי לתמוך תכנון טיפול.
Disclosures
H.M., אל. ג'יי, כ"ע ו G.A.P. עובדים של ולהחזיק מניות ב H.M. Biodesix, inc., אל. ג'יי, G.A.P. הם ממציאים על בקשה לרישום פטנט שהגיש Biodesix, המכסה אבחון מערכת איתור של מחזורי גרסאות גנטיים שאינם קטנים התא סרטן ריאות.
Acknowledgements
אנו מודים השותפים שלנו, סטפן ג'ונס, ניה Charrington, ד ר דיאנה Maar, ד ר קופר. סמנתה מן המרכז ביולוגיה דיגיטלית (ביו-ראד בע מ CA) בזכות העיצוב assay תמיכה; Nezar Rghei, Moemen ד ר עבדאללה (Norgen Biotek, קנדה) עצה קריטית בשעת מיטוב פרוטוקול החילוץ RNA; ובדוק שאנון קמפבל, סקוט תרסטון, ג'ף Fensterer, שאנון Martello ו Joellyn אנוס לעזרה עם דרישות וניטור מסחרי.
Materials
| Name | Company | Catalog Number | Comments |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1604071 |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1809353 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1604071 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1606077 |
| Phosphate Buffered Saline 1X, Sterile | Amresco | K812-500mL | 1446C189 |
| Phosphate Buffered Saline 1X, Sterile – 500 mL | Invitrogen | 10010023 | 1916C092 |
| RNase Zap (Life Tech) (250 mL) | Ambion | AM9780 | 353952 |
| Beta-Mercaptoethanol (BME) (250 mL) | CalbioChem | 6050 | W105B |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56054611 |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56238638 |
| Isopropyl Alcohol | VWR | 0918-4L | 2116C416 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 403648 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 288461 |
| DNase I | Thermo | K0441 | 371299 |
| QIAzol Lysis Reagent | Qiagen | 79306 | 54809699 |
| 20x TE buffer pH 8.0 | Alfa Aesar | J62388 | R13C548 |
| UltraPure Agarose | Invitrogen | 16500-100 | 552730 |
| 10x TBE buffer | Invitrogen | AM9863 | 353065 |
| Cell-Free RNA BCT | Streck | 218976 | 60110327 |
| Cell-Free RNA BCT | Streck | 218976 | 61900327 |
| Cell-Free RNA BCT | Streck | 218976 | 61480327 |
| Cell-Free RNA BCT | Streck | 218976 | 62320327 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 585849 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 588308 |
| Lysis Buffer | Norgen | 21205 | A5F61E |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 585848 |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 588309 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC186976 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188077 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188413 |
| Collection Tubes 500 pack | Zymo | C1001-500 | N/A |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 391657 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 392504 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 448001 |
| SuperScript IV Reverse Transcriptase | Life Technologies | 18090200 | 451702 |
| Qubit HS RNA Assay Kit (500) | Life Technologies | Q32854 | 1745264 |
| Qubit assay tubes (500) | Life Technologies | Q32856 | 13416Q311 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64031651 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64063941 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065740 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065741 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64079083 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64025320 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64052358 |
| gBlock KIF5B-RET K15:R12 | IDT | 151004172 | 4-Oct-16 |
| gBlock KIF5B-RET K16:R12 | IDT | 151004173 | 4-Oct-16 |
| gBlock KIF5B-RET K22:R12 | IDT | 151004174 | 4-Oct-16 |
| gBlock KIF5B-RET K23:R12 | IDT | 151004175 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R11 | IDT | 151004176 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R8 | IDT | 151004177 | 4-Oct-16 |
| gBlock CCDC6-RET C1:R12 | IDT | 151004178 | 4-Oct-16 |
| gBlock TRIM33-RET T14:R12 | IDT | 151004179 | 4-Oct-16 |
| RET exon 8 RT Gene Specific Primer | IDT | 150554385 | 28-Sep-16 |
| 5’-CTCCACTCACACCTG-3’ | IDT | 150554385 | 28-Sep-16 |
| RET exon 11 RT Gene Specific Primer | IDT | 150554384 | 28-Sep-16 |
| 5’-GCAAACTTGTGGTAGCAG-3’ | IDT | 150554384 | 28-Sep-16 |
| RET exon 12 RT Gene Specific Primer | IDT | 150554383 | 28-Sep-16 |
| 5’-CTGCCTTTCAGATGGAAG-3’ | IDT | 150554383 | 28-Sep-16 |
| gBlock CD74-ROS1 C6:R34 | IDT | 152324366 | 15-Nov-16 |
| gBlock CD74-ROS1 C6:R32 | IDT | 152324367 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R32 | IDT | 152324368 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R34 | IDT | 152324369 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R32 | IDT | 152324370 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R34 | IDT | 152324371 | 15-Nov-16 |
| gBlock EZR-ROS1 E10:R34 | IDT | 152324372 | 15-Nov-16 |
| gBlock TPM3-ROS1 T8:R35 | IDT | 152324373 | 15-Nov-16 |
| ROS1 exon 34 RT Gene Specific Primer | IDT | 152704983 | 21-Nov-16 |
| 5’-CCTTCCTTGGCACTTT-3’ | IDT | 152704983 | 21-Nov-16 |
| ROS1 exon 35 RT Gene Specific Primer | IDT | 152704985 | 21-Nov-16 |
| 5’-CTCTTGGGTTGGAAGAGTATG-3’ | IDT | 152704985 | 21-Nov-16 |
| ALK Gene Specific Primer | IDT | 140035422 | 26-Aug-16 |
| 5’-CAGTAGTTGGGGTTGTAGTCG-3’ | IDT | 140035422 | 26-Aug-16 |
| EML4-ALK Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| SLC34A2-ROS1 Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| CCDC6-RET Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| Human Brain Total RNA | Ambion | AM7962 | 1703548 |
| PrimePCR ddPCR Expert Design Assay: K15:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K16:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K22:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K23:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R11 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R8 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C1:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: T14:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: E10:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: T8:R35 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| (version 2) | Bio-Rad | 12003909 | 213939881 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 13-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 20170112v3.2 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851151 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 207383915 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 195995635 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851152 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 213949301 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 20160914 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 211383227 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 1065C220 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052953 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052358 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 1065C320 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64052952 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64064127 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000065883 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084276 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000079928 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084395 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084634 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20160627 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161107 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161206 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161216 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20170125 |
| Pipet Tips for Automated Droplet Generator | Bio-Rad | 1864120 | PR125340 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206894 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206893 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 1409850 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 100402 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 145851 |
| Microseal 'B' seals | Bio-Rad | MSB1001 | BR00428490 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64039089 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049253 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049255 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64081870 |
| DNA Lo Bind Tube 0.5 mL | Eppendorf | 22431005 | E1629620 |
| DNA Lo Bind Tube 1.5 mL | Eppendorf | 22431021 | F16698K |
| DNA Lo Bind Tube 2 mL | Eppendorf | 22431048 | E160610I |
| 50 mL Conicals, Polypropylene (25) | Thermo | 339652 | G5ZF5W8118 |
| TempAssure PCR 8-Strips, Optical Caps, Natural, polypropylene (120) | USA Scientific | 1402-4700 | 16202 |
| For Rainin LTS Pipettors 0.5-20 µL tips | Pipette.com | LF-20 | 40155-642C4-642C |
| For Rainin LTS Pipettors 5-200 µL tips | Pipette.com | LF-250 | 40154-642C4-642B |
| Tips LTS 200 ul Filter 960/10 RT-L200F (10 boxes) | Rainin | 17002927 | 1635 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F1175551-1108 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F118054L-1720 |
| Pipet Tips, 100 ul TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1180-1740 | 0014961Q-2501 |
| Pipet Tips, 200 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1180-8710 | E116684P-1540 |
| Pipet Tips, 1000 ul XL TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1182-1730 | F118815P |
| 5 mL Standard Racked Gilson-fit Reference Tips | Scientific Specialties | 4411-00 | 14312 |
| Combitips advanced, 0.1 mL Biopur | Eppendorf | 003 008 9618 | F165414H |
| Combitips advanced, 0.2 mL Biopur | Eppendorf | 0030 089.626 | F166689J |
| Combitips advanced, 5 mL Biopur | Eppendorf | 0030.089 669 | F166054J |
| Combitips advanced, 50 mL Biopur | Eppendorf | 003.008.9693 | F166055I |
| Reagent Reservoir | VWR | 89094-680 | 141500 |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165029I |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165028G |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Green | Eppendorf | 951020346 | F166183K |
| Equipment Type | Equipment ID | ||
| Analytical Balance | EQP0125 | ||
| Cryogenic Freezer 1, -80oC | EQP0095 | ||
| Refrigerator 6.1 cu ft GP06W1AREF | EQP0139 | ||
| -20oC Freezer | EQP0140 | ||
| Beckman Coulter Microfuge 22R | EQP0025 | ||
| Beckman Coulter Microfuge 22R | EQP0124 | ||
| Thermo Scientific Hereaus Megafuge 8 | EQP0104 | ||
| Mini Centrifuge | EQP0131 | ||
| Mini Centrifuge | EQP0136 | ||
| Mini Centrifuge | EQP0134 | ||
| Mini Centrifuge | EQP0235 | ||
| Mini Centrifuge | EQP0216 | ||
| Thermo Scientific HeraTherm Incubator | EQP0105 | ||
| Pipette 0.1 - 2.5 μL | EQP0182 | ||
| Pipette 0.1 - 2.5 μL | EQP0072 | ||
| Pipette 0.1 - 2.5 μL | EQP0070 | ||
| Pipette 0.5-10 μL | EQP0218 | ||
| Pipette 0.5-10 μL | EQP0075 | ||
| Pipette 0.5-10 μL | EQP0169 | ||
| Pipette 0.5-10 μL | EQP0074 | ||
| Pipette 0.5-10 μL | EQP0147 | ||
| Pipette 2 - 20 μL | EQP0128 | ||
| Pipette 2 - 20 μL | EQP0160 | ||
| Pipette 2 - 20 μL | EQP0018 | ||
| Pipette 2 - 20 μL | EQP0146 | ||
| Pipette 10 - 100 μL | EQP0079 | ||
| Pipette 10 - 100 μL | EQP0181 | ||
| Pipette 10 - 100 μL | EQP0085 | ||
| Pipette 10 - 100 μL | EQP0077 | ||
| Pipette 20 - 200 μL | EQP0088 | ||
| Pipette 20 - 200 μL | EQP0087 | ||
| Pipette 20 - 200 μL | EQP0231 | ||
| Pipette 100 - 1000 μL | EQP0050 | ||
| Pipette 100 - 1000 μL | EQP0158 | ||
| Pipette 100 - 1000 μL | EQP0217 | ||
| Pipette 100 - 1000 μL | EQP0082 | ||
| Pipette 100 - 1000 μL | EQP0183 | ||
| Pipette 100 - 1000 μL | EQP0083 | ||
| Pipette 5 mL | EQP0153 | ||
| Timer | S/N 140623950 | ||
| Hamilton SafeAire VAV Fume Hood | EQP0206 | ||
| Biosafety Cabinet | EQP0205 | ||
| Biosafety Cabinet | EQP0204 | ||
| Qubit 3.0 | EQP0102 | ||
| Benchmark Digital Heat Block | EQP0108 | ||
| Benchmark Digital Heat Block | EQP0231 | ||
| Polaroid Z2300 Instant Print Digital Gel Camera with WiFi and 16GB SDHC memory card | EQP0111 | ||
| Electrophoresis Power Unit | EQP0113 | ||
| Electrophoresis Small Gel Box | EQP0116 | ||
| Maestro Transilluminator | EQP0118 | ||
| Microwave | EQP0215 | ||
| Multichannel 8-well Pipette 2 - 20 μL | EQP0207 | ||
| Multichannel 8-well Pipette 10 - 100 μL | EQP0090 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0094 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0161 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0162 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0163 | ||
| Vortex Genie 2 | EQP0052 | ||
| Vortex Genie 2 | EQP0007 | ||
| Vortex Genie 2 | EQP0132 | ||
| Vortex Genie 2 | EQP0137 | ||
| Vortex Genie 2 | EQP0135 | ||
| Air Clean PCR Workstation | EQP0203 | ||
| Air Clean PCR Workstation | EQP0096 | ||
| Air Clean PCR Workstation | EQP0148 | ||
| Air Clean PCR Workstation | EQP0097 | ||
| QX200 Droplet Generator | EQP0202 | ||
| QX200 Droplet Generator | EQP0121 | ||
| Automated Droplet Generator | EQP0179 | ||
| PX1 PCR Plate Sealer | EQP0123 | ||
| PX1 PCR Plate Sealer | EQP0186 | ||
| C1000 Touch Cycler w/96W FS RM | EQP0120 | ||
| S1000 Cycler w/96W FS RM | EQP0174 | ||
| S1000 Cycler w/96W FS RM | EQP0173 | ||
| T100 Thermal Cycler | EQP0180 | ||
| T100 Thermal Cycler | EQP0175 | ||
| QX200 Droplet Reader | EQP0194 | ||
| QX200 Droplet Reader | EQP0122 |
References
- Ignatiadis, M., Lee, M., Jeffrey, S. S. Circulating Tumor Cells and Circulating Tumor DNA: Challenges and Opportunities on the Path to Clinical Utility. Clin Cancer Res. 21 (21), 4786-4800 (2015).
- Alix-Panabieres, C., Pantel, K. Clinical Applications of Circulating Tumor Cells and Circulating Tumor DNA as Liquid Biopsy. Cancer Discov. , (2016).
- Paxton, A. Is Molecular AP testing in sync with guidelines. CAP Today. , (2014).
- Sacher, A. G., et al. Prospective Validation of Rapid Plasma Genotyping for the Detection of EGFR and KRAS Mutations in Advanced Lung Cancer. JAMA Oncol. , (2016).
- Sozzi, G., et al. Quantification of free circulating DNA as a diagnostic marker in lung cancer. J Clin Oncol. 21 (21), 3902-3908 (2003).
- Oxnard, G. R., et al. Noninvasive detection of response and resistance in EGFR-mutant lung cancer using quantitative next-generation genotyping of cell-free plasma DNA. Clin Cancer Res. 20 (6), 1698-1705 (2014).
- Best, M. G., et al. RNA-Seq of Tumor-Educated Platelets Enables Blood-Based Pan-Cancer, Multiclass, and Molecular Pathway Cancer Diagnostics. Cancer Cell. 28 (5), 666-676 (2015).
- Rodriguez, M., et al. Different exosome cargo from plasma/bronchoalveolar lavage in non-small-cell lung cancer. Genes Chromosomes Cancer. 53 (9), 713-724 (2014).
- Kalluri, R. The biology and function of exosomes in cancer. J Clin Invest. 126 (4), 1208-1215 (2016).
- Mellert, H., et al. Development and Clinical Utility of a Blood-Based Test Service for the Rapid Identification of Actionable Mutations in Non-Small Cell Lung Carcinoma. J Mol Diagn. 19 (3), 404-416 (2017).
- Qin, J., Williams, T. L., Fernando, M. R. A novel blood collection device stabilizes cell-free RNA in blood during sample shipping and storage. BMC Res Notes. 6, 380 (2013).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. Biotechniques. 15 (3), 532-537 (1993).
- Kohno, T., et al. Beyond ALK-RET, ROS1 and other oncogene fusions in lung cancer. Transl Lung Cancer Res. 4 (2), 156-164 (2015).
- Takeuchi, K., et al. ROS1 and ALK fusions in lung cancer. Nat Med. 18 (3), 378-381 (2012).
- Rimkunas, V. M., et al. Analysis of receptor tyrosine kinase ROS1-positive tumors in non-small cell lung cancer: identification of a FIG-ROS1 fusion. Clin Cancer Res. 18 (16), 4449-4457 (2012).
- Tsuta, K., et al. RET-rearranged non-small-cell lung carcinoma: a clinicopathological and molecular analysis. Br J Cancer. 110 (6), 1571-1578 (2014).
- Forbes, S. A., et al. COSMIC: somatic cancer genetics at high-resolution. Nucleic Acids Res. 45 (1), 777-783 (2017).
- Salgia, R. Diagnostic challenges in non-small-cell lung cancer: an integrated medicine approach. Future Oncol. 11 (3), 489-500 (2015).
- Cagle, P. T., Raparia, K., Portier, B. P. Emerging Biomarkers in Personalized Therapy of Lung Cancer. Adv Exp Med Biol. 890, 25-36 (2016).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci U S A. 96 (16), 9236-9241 (1999).
- Oxnard, G. R., et al. Association Between Plasma Genotyping and Outcomes of Treatment With Osimertinib (AZD9291) in Advanced Non-Small-Cell Lung Cancer. J Clin Oncol. 34 (28), 3375-3382 (2016).
- Reckamp, K. L., et al. A Highly Sensitive and Quantitative Test Platform for Detection of NSCLC EGFR Mutations in Urine and Plasma. J Thorac Oncol. 11 (10), 1690-1700 (2016).
- Yanagita, M., et al. A prospective evaluation of circulating tumor cells and cell-free DNA in EGFR mutant non-small cell lung cancer patients treated with erlotinib on a phase II trial. Clin Cancer Res. , (2016).
- Abbosh, C., et al. Phylogenetic ctDNA analysis depicts early-stage lung cancer evolution. Nature. 545 (7655), 446-451 (2017).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-534 (1993).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved