A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בחירת קבוצות משנה מרובים סמן עם הופעות איבחונים יעיל באופן דומה
In This Article
Summary
קיימים אלגוריתמים לייצר פתרון אחד עבור dataset זיהוי ' סמן '. פרוטוקול זה מדגים את קיום פתרונות מרובים לתוצאה דומה ומציג תוכנה ידידותית למשתמש כדי לסייע לחוקרים ביו לחקור datasets שלהם בשביל האתגר המוצע. מדעני מחשב עשויים גם כן לספק תכונה זו בשלהם סמן זיהוי אלגוריתמים.
Abstract
סמן זיהוי היא אחת מהשאלות ביו יותר חשוב לחוקרים תפוקה גבוהה 'טכנולוגיות"ולהפיק כמעט כל האלגוריתמים זיהוי סמן קיימת תת-קבוצה סמן אחד עם מדידה מיטביים עבור dataset נתון . עם זאת, מחקר שנערך לאחרונה הפגינו את קיום קבוצות משנה מרובים סמן עם הופעות סיווג לתוצאה דומה או אפילו זהה. פרוטוקול זה מציג מתודולוגיה פשוטה לגילוי קבוצות משנה סמן עם הופעות איבחונים, טוב יותר על-ידי המשתמש ניתוק. הפרוטוקול מורכב נתוני הכנה, טעינה, סיכום מידע תוכנית בסיסית, פרמטר הכוונון, סמן ההקרנה, תוצאה ויזואליזציה, פרשנות, סמן גנטי ביאורים, exportation תוצאה והדמיה - איכות הפרסום. סמן המוצע הקרנת אסטרטגיה הוא אינטואיטיבי ומדגים כללי לפיתוח אלגוריתמים גילוי סמן. ממשק משתמש גרפי ידידותי למשתמש (GUI) פותחה באמצעות שפת התיכנות פיתון, ומאפשר ביו לחוקרים יש גישה ישירה אל התוצאות שלהם. ניתן להוריד את קוד המקור ואת המדריך של kSolutionVis http://www.healthinformaticslab.org/supp/resources.php.
Introduction
חקר איבחונים, נפוץ ביותר, כריית בעיות באזור ביו, מאתגר נתונים משמש לבניית מודל סיווג מאומן על שתי קבוצות של דגימות עם המדויקות ביותר אפליה כוח1, 2 , 3 , 4 , 5 , 6 , 7. עם זאת, הנתונים הגדול שנוצר בתחום הביו-רפואי יש את הגלום "גדול p קטנה n" הפרדיגמה, עם מספר תכונות בדרך כלל הרבה יותר גדול מאשר מספר דוגמאות6,8,9. לכן, חוקרים ביו יש להפחית את הממד תכונה לפני ניצול של אלגוריתמים כדי להימנע overfitting בעיה8,9. אבחון סמנים ביולוגיים מוגדרים קבוצת משנה של תכונות שזוהו המפריד בין המטופלים של מחלה נתונה מ בקרה בריאים דגימות10,11. חולים הם בדרך כלל מוגדר כ הדגימות החיוביות, הפקדים בריא מוגדרים דגימות שליליות12.
מחקרים שנעשו לאחרונה הראו כי קיים פתרון אחד או יותר עם הופעות סיווג זהים או לתוצאה דומה עבור ביו הנתונים (dataset)5. כמעט כל תכונה את האלגוריתמים הבחירה הם אלגוריתמים דטרמיניסטי, לייצר פתרון אחד בלבד ערכת הנתונים זהה. אלגוריתמים גנטיים בו-זמנית עשוי ליצור פתרונות מרובים עם הופעות דומות, אבל הם עדיין מנסים לבחור פתרון אחד עם הפונקציה כושר הכי טוב בתור הפלט עבור הנתונים (dataset) נתון13,14.
התכונה בחירה אלגוריתמים ניתן לקבץ בערך כמו מסננים או עטיפות12. אלגוריתם הסינון בוחר העליון -k התכונות מדורגת על ידי שיוכם בודדים משמעותית עם תוויות מחלקה בינארי מבוסס על ההנחה כי התכונות אינם תלויים אחד בשני15,16,17 . אף על פי הנחה זו לא מחזיקה נכון עבור כמעט כל datasets בעולם האמיתי, הכלל מסנן האוריסטי מבצע טוב במקרים רבים, למשל, האלגוריתם (מינימום יתירות והרלוונטיות המרבי) mRMR, Wilcoxon בדיקה בהתאם תכונת הסינון (WRank) אלגוריתם, ואת העלילה ROC (מאפיין ההפעלה מקלט) המבוסס על אלגוריתם הסינון (ROCRank). mRMR, הוא אלגוריתם יעיל מסנן כי זה קרוב הבעיה שערוך קומבינטורית עם סדרה של בעיות הרבה יותר קטן, השוואת מרבי-תלות תכונה בחירת אלגוריתם, שכל אחד מהם כרוך רק שני משתנים, ו לכן משתמש pairwise הסתברויות משותף אשר הם עמידים יותר18,19. עם זאת, mRMR עשוי לזלזל התועלת של תכונות מסוימות כמו זה לא למדוד את האינטראקציות בין תכונות אשר יכול להגביר את הרלוונטיות, ובכך מחטיא כמה שילובים תכונה חסרי תועלת בנפרד, אבל הם שימושיים רק כאשר משולב. האלגוריתם WRank חישוב ציון פרמטרית של איך שמסווגת תכונה בין שתי מחלקות של דגימות, והוא ידוע בזכות החוסן שלה ליניאריים20,21. יתר על כן, האלגוריתם ROCRank מעריך כמה משמעותית האזור תחת ROC העקומה (AUC) של תכונה מסוימת היא איבחונים ובדוקים ביצועים22,23.
מצד שני, עטיפת מוערך של המסווג מוגדרים מראש הביצועים של תת-ערכה תכונה נתונה, iteratively שנוצר על ידי כלל היריסטי, ויוצר את ערכת המשנה של תכונה עם מדידת הביצועים הטובה ביותר24. עטיפת בדרך כלל outperforms מסנן הביצועים סיווג אך פועל לאט יותר25. לדוגמה, האלגוריתם27 26,היער אקראי התפלגות (RRF) משתמש כלל חמדן, המעריכה את התכונות על קבוצת משנה של הנתונים הדרכה בכל צומת יער אקראי, ציונים החשיבות של מי תכונה מוערכים על ידי מדד גיני . הבחירה של תכונה חדשה נענש אם שלה רווח מידע לא לשפר את התכונות שבחרת. בנוסף, הניתוח חיזוי עבור מיקרו-מערכים (פאם)28,אלגוריתם29 , גם באלגוריתם מעטפת, מחשבת של centroid עבור כל התוויות מחלקה ובוחרת ואז תכונות כדי לכווץ את centroids ג'ין כלפי הכולל הכיתה centroid. . פאם היא חזקה עבור תכונות המרוחקים.
פתרונות מרובים עם ביצועים סיווג העליון עשוי להיות נחוץ עבור כל ערכת נתונים נתונה. ראשית, המטרה אופטימיזציה של אלגוריתם דטרמיניסטי זו מוגדרת על ידי הנוסחה המתמטית, למשל, שיעור שגיאה מינימום30, אשר אינה בהכרח אידיאלי עבור דגימות ביולוגיות. שנית, dataset ייתכן מרובות, פתרונות שונים באופן משמעותי, עם הופעות דומות יעיל או אפילו זהים. כמעט כל תכונה קיימת בחירה אלגוריתמים יבחר אחד הפתרונות האלה באופן אקראי כמו פלט31.
מחקר זה תציג פרוטוקול אנליטית אינפורמטיקה ליצירת פתרונות הבחירה תכונה מרובים עם הופעות דומות עבור כל נתון איבחונים הנתונים (dataset). בהתחשב בכך כי החוקרים ביותר ביו אינם מכירים טכניקות informatic או בקידוד ממוחשב, ממשק משתמש גרפי ידידותי למשתמש (GUI) פותחה כדי להקל על הבדיקה המהירה של איבחונים ביו datasets. פרוטוקול אנליטית מורכבת טעינת נתונים המסכם, פרמטר הכוונון, צינור ביצוע ואת התוצאה פרשנויות. בלחיצה פשוטה, החוקר הוא מסוגל לייצר את סמן קבוצות משנה וחלקות ויזואליזציה באיכות פרסום. הפרוטוקול נבדקו באמצעות את transcriptomes של שני איבחונים datasets חריפה לימפוציטית לוקמיה (כל), דהיינו, ALL1 ו- ALL212. Datasets ALL1 ו ALL2 היו להוריד רחבה המכון הגנום ניתוח במרכז הנתונים, לרשותכם http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi. ALL1 מכיל דגימות 128 עם תכונות 12,625. הדוגמאות הללו, 95 נמצאים תאי B כל 33 הם T-cell כל. ALL2 כוללת 100 דגימות עם תכונות 12,625 גם כן. הדוגמאות הללו, ישנם חולים 65 שסבלו relapse ומטופלים 35 זה לא. ALL1 היה dataset איבחונים קל, עם דיוק המינימלי ארבעת עטיפות להיות 96.7% ו- 6 של אלגוריתמי הבחירה של תכונה 8 להשגת 100%12ומסננים ארבע. בעוד ALL2 היה dataset קשה יותר, עם תכונת 8 לעיל את הבחירה האלגוריתמים להשגת לא יותר 83.7% דיוק12. למידת הדיוק הזה הושג עם תכונות 56 זוהה על ידי האלגוריתם מעטפת, בחירת תכונה מבוססת קורלציה (CFS).
Protocol
הערה: פרוטוקול הבאים מתאר את פרטי ההליך האנליטי אינפורמטיקה וקודים מדומה של המודולים העיקריים. מערכת ניתוח אוטומטיות פותחה באמצעות פיתון גרסה 3.6.0 ו פנדה מודולים של פייתון, abc, numpy, scipy, sklearn, sys, PyQt5, sys, mRMR, מתמטיקה, matplotlib. החומרים אשר השתמשו במחקר זה מפורטים בטבלה של חומרים.
1. מכינים את מטריצת נתונים ושיעור תוויות
- להכין קובץ מטריקס הנתונים כקובץ מטריקס או פסיק-טאבים, כמופיע ב איור 1A.
הערה: כל שורה יש את כל הערכים של תכונה, הפריט הראשון הוא שם התכונה. תכונה הוא מזהה probeset עבור ערכת הנתונים מבוסס-microarray transcriptome או מזהה ערך אחר כמו משקע ציסטאין עם הערך מתילציה שלו ב- methylomic dataset. כל עמודה נותן את ערכי תכונה של מדגם נתון, עם הפריט הראשון הוא השם לדוגמה. שורה מופרדת לעמודות באמצעות טאב (איור 1B) או פסיק (איור 1C). קובץ מופרד באמצעות טאב מטריקס מזוהה על-ידי .tsv סיומת קובץ, קובץ מופרד מטריקס כולל את סיומת. csv. קובץ זה עשוי להיווצר על-ידי שמירת מטריצה כמו גם את תבנית .tsv או ה-. csv של תוכנות כגון Microsoft Excel. המטריקס נתונים עשוי להיווצר גם על-ידי המחשב קידוד. - הכנת קובץ תווית ה-class כקובץ או פסיק-טאבים מטריקס (איור 1D), דומה לקובץ הנתונים מטריקס.
הערה: העמודה הראשונה נותן השמות לדוגמה, התווית מחלקה של כל מדגם ניתנת בעמודה שכותרתו הכיתה. תאימות מירבית נחשב בתהליך כתיבת קוד, כך ניתן להוסיף עמודות נוספות. ניתן לעצב קובץ המחלקה תווית קובץ .tsv או ה-. csv. השמות בעמודה מחלקה עשויים להיות בכל תנאי, אולי יש יותר משני שיעורים של דגימות. המשתמש יכול לבחור שניים המחלקות עבור הניתוח הבא.
2. לטעון את מטריצת נתונים ושיעור תוויות
- לטעון את מטריצת נתונים ותוויות שיעור לתוך התוכנה. לחץ על לחצן מטריצת נתונים עומס כדי לבחור את הקובץ מטריצת נתונים מוגדרים על-ידי המשתמש. לחץ על לחצן תוויות מחלקה עומס כדי לבחור קובץ תווית המחלקה המתאימה.
הערה: לאחר שני הקבצים נטענים, kSolutionVis ינהל מסך שגרתית של ההתאמה בין שני הקבצים. - מסכמים את כל המאפיינים, דגימות מקובץ הנתונים מטריקס. להעריך את הגודל של קובץ הנתונים מטריקס.
- מסכמים את דגימות ושיעורי מהקובץ תווית הכיתה. להעריך את הגודל של קובץ תווית המחלקה.
- בדוק כל דגימה מתוך מטריצת נתונים כולל תווית הכיתה. מסכמים את מספר הדגימות עם תוויות הכיתה.
3. יסכם ויציג את הסטטיסטיקה הבסיסית של הנתונים (dataset)
- לחץ על לחצן סיכום, ללא כל מילת המפתח שצוינה קלט, התוכנה תציג 20 תכונות אינדקס ושמות התכונות המתאימות.
הערה: משתמשים עליך לציין את שם התכונה שהם רוצים למצוא לראות שלה-סטטיסטיקה בסיסית והפצה ערך התואם בין כל דוגמאות קלט. - מספקים של מילת המפתח, למשל "1000_at", בתיבת הטקסט של תכונה למצוא תכונה מסוימת יש לסכם. לחץ על לחצן סיכום כדי לקבל את הסטטיסטיקה הבסיסית עבור תכונה זו נתונה.
הערה: מילת המפתח עשויים להופיע בכל מקום שמות התכונות היעד, להקל על תהליך חיפוש עבור המשתמשים. - לחץ על לחצן סיכום למצוא תכונה יותר מפעם אחת עם מילת המפתח נתון ולאחר מכן ציין את מזהה ייחודי כדי להמשיך לשלב לעיל של סיכום תכונה מסוימת אחת.
4. לקבוע את התוויות הכיתה ומספר תכונות המדורגים עליונים
- בחר את השמות של חיובי ("(33) P") ושיעורי שלילי ("(95) N") בתיבות רשימה נפתחת כיתה חיוביות וגם שליליות מחלקה, כפי שמוצג באיור 2 (באמצע).
הערה: מומלץ לבחור שסיווג בינארי מאוזן dataset, דהיינו, ההפרש בין המספרים של דגימות חיוביות ושליליות היא מזערית. מספר דוגמאות ניתנת גם בתוך סוגריים לאחר השם בכל תווית מחלקה של שתי תיבות רשימה נפתחת. - בחרו 10 ככל שמספר תכונות המדורגים עליונים (פרמטר pTopX) בתיבה הנפתחת Top_X (?) עבור מסך מקיף של המשנה-התכונה.
הערה: התוכנה באופן אוטומטי מדרג את כל התכונות על ידי P-ערך מחושב על ידי מבחן t של כל תכונה משווה את המחלקות חיוביים ושליליים. תכונה עם קטנים P-ערך יש כוח יותר להפלות בין המעמדות שתי דוגמאות. המודול הקרנה מקיפה הוא שהמפתחות אינטנסיבית. הפרמטר pTopX הוא 10 כברירת מחדל. משתמשים יכולים לשנות פרמטר זה בטווח של 10 עד 50, עד שהם מוצאים סיפוק כוללים קבוצות משנה עם הופעות סיווג טוב.
5. לכוון את מערכת פרמטרים עבור הופעות שונות
- בחר את מדידת ביצועים (pMeasurement) דיוק (Acc) בתיבה הנפתחת Acc/bAcc (?) עבור המסווג הנבחר מכונת למידה קיצונית (אלם). אפשרות נוספת של פרמטר זה היא מדידת דיוק מאוזנת (bAcc).
הערה: תן TP, FN, TN, ולהיות FP המספרים של אמת תוצאות חיוביות, שליליות כוזבות, התשלילים נכון מוטעות, בהתאמה. המדידה Acc מוגדר (TP+TN)/(TP+FN+TN+FP), אשר פועלת באופן הטוב ביותר ב- dataset מאוזנת6. אבל מסווג אופטימיזציה עבור Acc נוטה להקצות כל הדגימות למחלקה שלילי אם מספר דוגמאות שליליות הוא הרבה יותר גדול מזה של אלה חיובית. BAcc מוגדר (Sn + Sp) / 2, איפה Sn = TP/(TP+FN) ו Sp = TN/(TN+FP) התעריפים כראוי החזוי עבור חיובי ושלילי דגימות, בהתאמה. לכן, bAcc מווסת את ההופעות חיזוי על שתי הכיתות, עלול להוביל הופעה חיזוי מאוזנת על פני שתי מחלקות לא מאוזן. Acc הוא הבחירה המהווה ברירת מחדל של pMeasurement. התוכנה משתמשת המסווג אלם כברירת מחדל כדי לחשב ההופעות סיווג. המשתמש לבחור גם מסווג מכונת וקטורים תומכים (מכונת וקטורים תומכים), KNN (k השכן הקרוב), עץ ההחלטות או נאיבית Bayes. - בחר את ערך הסף 0.70 (פרמטר pCutoff) עבור המידה ביצועים שצוין בתיבת הקלט pCutoff:.
הערה: Acc , bAcc בטווח שבין 0 ל-1, והן המשתמש יכול לציין pCutoff ערך של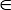 [0, 1] כמו החיתוך כדי להציג את הפתרונות מתאימים. התוכנה מבצעת מקיפה תכונה-משנה ההקרנה, המתאימה pCutoff יגרום 3D להדמיה יותר אינטואיטיבי ומפורש. ערך ברירת המחדל עבור pCutoff הוא 0.70.
[0, 1] כמו החיתוך כדי להציג את הפתרונות מתאימים. התוכנה מבצעת מקיפה תכונה-משנה ההקרנה, המתאימה pCutoff יגרום 3D להדמיה יותר אינטואיטיבי ומפורש. ערך ברירת המחדל עבור pCutoff הוא 0.70.
6. הפעל את הצינור ולייצר את התוצאות מטמיעים אינטראקטיבי
- לחץ על לחצן Analyze כדי להפעיל את הצינור ולהפיק החלקות ויזואליזציה, כפי שמוצג באיור 2 (למטה).
הערה: הטבלה הימנית נותן כל תכונת ערכות המשנה שלהם pMeasurement שמחשבת את אסטרטגיית האימות קרוס 10-fold המסווג אלם, כפי שתואר לעיל5. שני מגרשים פיזור 3D וחלקות שתי שורות נוצרות עבור ההליך ההקרנה תכונה-ערכה עם קביעות הפרמטר הנוכחי. - בחרו ' 0.70 ערך ברירת המחדל pMeasurement החיתוך (פרמטר piCutoff, קלט בתיבת הערך), ו-10 כברירת המחדל של מספר קבוצות משנה התכונה הטובה ביותר (פרמטר piFSNum).
הערה: הצינור תבוצע באמצעות פרמטרים pTopX, pMeasurement, pCutoff. התכונה שזוהו קבוצות משנה יכול להיות עוד יותר הוקרן באמצעות חיתוך של piCutoff, עם זאת piCutoff לא יכול להיות קטן יותר pCutoff. לכן, piCutoff מאותחל כמו pCutoff , ניתן לאבחן רק תכונה קבוצות משנה עם ≥ מדידת ביצועים piCutoff . ערך ברירת המחדל של piCutoff הוא pCutoff. לפעמים kSolutionVis יזהה פתרונות רבים, רק את הטוב ביותר piFSNum (ברירת מחדל: 10) ניתן לאבחן את התכונה קבוצות משנה. אם מספר קבוצות משנה תכונה שזיהה את התוכנה הוא קטן יותר piFSNum, ניתן לאבחן כל תכונה קבוצות משנה. - לאסוף ולתרגם את התכונות שזוהה על-ידי התוכנה, כפי שמוצג באיור3.
הערה: הטבלה בתיבה השמאלית מציגה תת-קבוצות תכונה שזוהו ומדידות ביצועים שלהם. השמות של שלוש העמודות הראשונות הם "F1", "F2" ו- "F3". שלוש תכונות בקבוצת משנה כל תכונה ניתנת לפי סדר הדירוג שלהם בשורה אחת (F1 < F2 < F3). העמודה האחרונה נותן את מדידת ביצועים (Acc או bAcc) של כל תכונה משנה, את שם העמודה (Acc או bAcc) הוא הערך של pMeasurement.
7. לפרש את פיזור 3D המגרשים-דמיינו ולפרש תת-קבוצות תכונה עם הופעות איבחונים לתוצאה דומה באמצעות פיזור 3D חלקות
- לחץ על לחצן נתח ליצירת העלילה פיזור תלת-ממד של תת-קבוצות תכונה העליון 10 עם מופעי סיווג הטוב ביותר (Acc או bAcc) זוהה על ידי התוכנה, כפי שמוצג באיור 3 (תיבת האמצעי). למיין את שלוש תכונות בקבוצת משנה תכונה לפי סדר עולה של שורותיהם ולהשתמש בסולם הדרגות של שלוש תכונות כמו F1/F2/F3 הצירים, קרי, F1 < F2 < F3.
הערה: הצבע של נקודה מייצגת את ביצועי איבחונים המשנה תכונה המתאימה. Dataset ייתכן קבוצות משנה מרובים תכונה עם מדידות ביצועים יעילים באופן דומה. לכן, עלילה אינטראקטיבית ומפושטת פיזור נחוץ. - שנה את הערך ל 0.70 בתיבת הקלט pCutoff: , ולחץ על הלחצן נתח ליצירת העלילה פיזור תלת-ממד של תת-קבוצות תכונה עם הביצועים מדידה ≥ piCutoff, כפי שניתן לראות באיור 3 (תיבת נכון). לחץ על לחצן כוונון תלת-ממד כדי לפתוח חלון חדש לכוון ידנית את זוויות צפייה של העלילה פיזור תלת-ממד.
הערה: כל תת-קבוצה תכונה מיוצגת על ידי נקודה באותו אופן כמו לעיל. העלילה פיזור תלת-ממד נוצר ב זווית ברירת המחדל. כדי להקל את לוויזואליזציה תלת-ממדית, כוונון, חלון נפרד נפתח על-ידי לחיצה על לחצן 3D כוונון. - לחץ על הלחצן הקטן כדי להפחית את היתירות של ערכות המשנה תכונה שזוהו.
הערה: אם משתמשים רוצים בחר השלישייה תכונה ולצמצם את היתירות של ערכות המשנה תכונה נוספת, התוכנה גם מספקת פונקציה זו באמצעות האלגוריתם הנבחר של תכונה mRMR. לאחר לחיצה על הכפתור הקטן , kSolutionVis להסיר תכונות אלה מיותר בתוך השלישייה תכונה והפק את הטבלה ופיזור שתי החלקות הנ ל. התכונות שהוסר של השלישייה תכונה יוחלפו על-ידי מילת המפתח בטבלה. הערכים ללא ציר F1/F2/F3 מסומן כמו הערך של piFSNum (הטווח של הערך נורמלי של F1/F2/F3 הוא [1, top_x]). לכן, הנקודות הכוללים ערך אף עשויים להופיע מתווה "חריג חשוד טעות" נקודות ב- 3D. החלקות תלת-ממדיים באופן ידני tunable ניתן למצוא ב "כוונון ידני של החלקות נקודה תלת-ממד" בחומר משלים.
8. למצוא גנים ביאורים ואגודות שלהם עם מחלות אנושיות
הערה: השלבים 8-10 ימחיש כיצד להוסיף ביאורים גן מרמת רצף ה-DNA וחלבון. ראשית, סמל הגן של כל מזהה סמן מ שלעיל יאוחזרו ממסד הנתונים של דוד32ולאחר מכן שני שרתי אינטרנט נציג ישמש כדי לנתח את הסמל הזה ג'ין מן הרמות של ה-DNA וחלבון, בהתאמה. השרת GeneCard מספק לביאור תפקודית מקיפה של סמל הגן נתון, ומספק חוקי התורשה מקוון במסד אדם (OMIM) את curation המקיף ביותר של המחלה-גן עמותות. שרת ה UniProtKB הוא אחד של המאגר המקיף ביותר של חלבון, וחוזה שרת מערכת חיזוי מבוססות קבוצה (GPS) איתות זירחון של רשימה גדולה מאוד של kinases.
- העתק ו הדבק קישור האינטרנט של מסד הנתונים, דוד דפדפן אינטרנט לפתוח את דף האינטרנט של מסד נתונים זה. לחץ על הקישור ג'ין מזהה ההמרה ראה איור 4A ולהזין את התכונה מזהים 38319_at/38147_at/33238_at של הראשון סמן ערכת המשנה של הנתונים (dataset) ALL1 (איור 4B). לחץ על הקישור לרשימת גנים ' ולחץ על שלח רשימה , כפי שמוצג באיור 4B. אחזר את הביאורים עניין ולחץ על הצג רשימת גנים (איור 4C). לקבל את רשימת סמלים ג'ין (איור 4D).
הערה: הסמלים ג'ין לאחזר כאן ישמש עבור עוד יותר פונקציונלי ביאורים בשלבים הבאים. - העתק ו הדבק קישור האינטרנט של מסד הנתונים, ג'ין כרטיסי דפדפן אינטרנט לפתוח את דף האינטרנט של מסד נתונים זה. חפש את השם של הגן CD3D בתיבת הקלט של שאילתת מסד נתונים ולמצוא את הביאורים של הגן הזה מן הגן כרטיסי33,34, כפי שמוצג בטבלה 1 ו- 5A איור.
הערה: כרטיסי ג'ין הוא גנטי מקיף ראשוניים, מתן נומנקלטורה, גנומיקה, פרוטאומיקס, לוקליזציה subcellular, ואת המסלולים המעורבים מודולים פונקציונליים אחרים. הוא גם מספק קישורים חיצוניים למסדי נתונים שונים אחרים ביו כמו PDB/PDB_REDO35, ג'ין Entrez36, OMIM37UniProtKB38. אם שם התכונה אינה סמל גן רגיל, להשתמש במסד הנתונים ENSEMBL כדי להמיר אותו39. CD3D הוא השם של הגן T-Cell קולטן T3 דלתא שרשרת. - העתק ו הדבק קישור האינטרנט של מסד הנתונים OMIM דפדפן אינטרנט לפתוח את דף האינטרנט של מסד נתונים זה. חפש את השם של הגן CD3D ולמצוא את הביאורים של הגן הזה ממסד הנתונים OMIM37, כפי שמוצג בטבלה 1 ו- 5B איור.
הערה: OMIM משמש כעת באחד ממקורות מוסמכים ומקיף ביותר של הגן האנושי התקשרויות עם מחלות הניתנות להעברה בירושה. OMIM היה ביוזמת ד ר ויקטור א McKusick לקטלוג של מוטציות גנטיות הקשורות למחלות40. OMIM עכשיו מכסה מעל 15,000 גנים אנושיים, הפנוטיפים מעל 8,500, בדצמבר 1סנט 2017.
9. הוספת ביאורים החלבונים מקודד ואת השינויים Post-Translational
- העתק ו הדבק קישור האינטרנט של מסד הנתונים UniProtKB דפדפן אינטרנט לפתוח את דף האינטרנט של מסד נתונים זה. חפש את השם של הגן CD3D בתיבת הקלט שאילתה של UniProtKB ולמצוא את הביאורים של הגן הזה של מסד הנתונים38, כפי שמוצג בטבלה 1 ו- 5C איור.
הערה: UniProtKB אוספת מקור עשיר של הביאורים של חלבונים, כולל המינוח ומידע פונקציונלי. מסד נתונים זה מספק גם קישורים חיצוניים למסדי נתונים אחרים בשימוש נרחב, כולל PDB/PDB_REDO35, OMIM37ו- Pfam41. - העתק ו הדבק קישור האינטרנט של שרת האינטרנט GPS דפדפן אינטרנט לפתוח את דף האינטרנט של שרת אינטרנט זה. אחזר את רצף החלבון מקודדת על ידי הגן סמן CD3D מתוך מסד הנתונים UniProtKB38 ולחזות שאריות post-translational השינוי (PTM) של החלבון בעזרת כלי מקוון GPS, כפי שמוצג בטבלה 1 ואיור 5D.
הערה: מערכת ביולוגית הוא דינמי ומסובך, ולאסוף מסדי הנתונים הקיימים רק מידע. לכן, כלים מקוונים חיזוי הביו-רפואית, כמו גם תוכניות מנותק עשוי לספק ראיות שימושיות כהשלמה מנגנון ממוצע. GPS כבר מפותחת ומשופרות עבור מעל 12 שנה7,42 , עשוי לשמש כדי לחזות שאריות של חלבון PTM בפפטיד נתון רצף43,44. כלים זמינים גם עבור נושאי מחקר שונים, לרבות התחזית של מיקום subcellular45 , שעתוק מקדם האיגוד מוטיבים 46 בין היתר של חלבון.
10. להוסיף ביאורים אינטראקציות חלבון-חלבון ומודולים פונקציונלי מועשר שלהם
- העתק ו הדבק קישור האינטרנט של שרת האינטרנט מחרוזת דפדפן אינטרנט לפתוח את דף האינטרנט של שרת אינטרנט זה. חפש את הרשימה על הגנים CD3D של P53, ולמצוא את תכונותיהם מתוזמר משימוש במסד הנתונים מחרוזת47. ההליך יכול להתבצע באמצעות שרת אינטרנט אחר, דוד32.
הערה: מלבד הביאורים הנ של גנים יחידניים, ישנם כלים אינפורמטיקה בקנה מידה גדול רבים זמינים לחקור את המאפיינים של קבוצת גנים. מחקר שנערך לאחרונה הראה כי הגנים סמן בנפרד רע אולי מהווים הרבה-משופרת ג'ין קבע5. לכן, זה שווה את העלות החישובית למסך של סמנים ביולוגיים מורכבים יותר. מסד הנתונים מחרוזת אוכל לדמיין את החיבורים אינטראקציה ידוע או החזוי, השרת דוד עשוי לזהות את המודולים פונקציונלי עם פנוטיפ משמעותית-עמותות הגנים המאוחזרים47,32. שונים כלי ניתוח אינפורמטיקה בקנה מידה גדול אחרים זמינים גם הם.
11. לייצא קבוצות משנה סמן שנוצר, החלקות ויזואליזציה
- ייצוא תת-קבוצות סמן שזוהו כקובץ טקסט .tsv או ה-. csv עבור ניתוח נוסף. לחץ על לחצן ייצוא השולחן מתחת לשולחן של כל סמן שזוהו ערכות המשנה ובחרו באיזה פורמט טקסט לשמור כקובץ.
- לייצא החלקות ויזואליזציה קובץ תמונה. לחץ על לחצן שמור תחת כל חלקה ובחרו באיזה פורמט התמונה לשמור כקובץ.
הערה: התוכנה תומכת של פיקסל. בתבנית png של .svg תבנית וקטורית. תמונות פיקסל טובים להצגה על מסך המחשב, ואילו תמונות וקטור ניתן להמיר בכל רזולוציה הנדרש למטרות פרסום כתב העת.
תוצאות
המטרה של זרימת עבודה זו (איור 6) היא לזהות קבוצות משנה מרובים סמן בעזרת יעילות דומה עבור dataset איבחונים. התהליך כולו מודגם על ידי שני datasets דוגמה ALL1, ALL2 מופק של זיהוי התפרסמו לאחרונה סמן ללמוד12,48. משתמש יכול להתקין kSolutionVis על פי ה...
Discussion
מחקר זה מציג פרוטוקול זיהוי ואפיון של easy-to-בצע פתרון רב סמן עבור dataset איבחונים מוגדרים על-ידי המשתמש. התוכנה שמה דגש על ידידותיות למשתמש ממשקים גמיש ייבוא/ייצוא בתבניות קובץ שונות, ומאפשר חוקרת ביו לחקור את הנתונים (dataset) בקלות באמצעות GUI של התוכנה. מחקר זה מדגיש גם את הצורך של יצירת פתרון אחד...
Disclosures
יש לנו שאין ניגודי אינטרסים הקשורים בדו ח זה.
Acknowledgements
עבודה זו נתמכה על-ידי התוכנית האסטרטגית של מחקר עדיפות של האקדמיה הסינית למדעים (XDB13040400) ואת המענק הפעלה מאוניברסיטת ג'ילין. הסוקרים אנונימיים ומשתמשים בדיקות ביו היו להערכה על הערותיהם בונה על שיפור את השימושיות ואת הפונקציונליות של kSolutionVis.
Materials
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
References
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved