Method Article
שילוב ניתוח של דנ א ב מיצוי Virion גולמי עם הניתוח של RNA של עלים נגועים לגלות הגנום וירוס חדש
In This Article
Summary
כאן אנו מציגים גישה חדשה לזיהוי וירוסים עם כפול-גדיל DNA הגנום. אנו משתמשים בשיטות הרגילות כדי לחלץ DNA ו- RNA עלים נגועים ולבצע רצף הדור הבא. כלים Bioinformatic להרכיב רצפים לתוך contigs, לזהות contigs המייצג את הגנום של וירוס והקצה הגנום לקבוצות בטקסונומיה.
Abstract
גישה metagenome זו משמשת לזיהוי וירוסים עם הגנום DNA מעגלי וגיליון הציונים שלהם. צמח לעיתים קרובות וירוסים DNA מתרחשים titers נמוכה של המארח שלהם או לא יכול להיות מחוסן באופן מכני לארח עוד קשים להפיץ כדי להשיג עם כייל נוגדנים גדולה יותר של חומר זיהומיות. עלים נגועים הם הקרקע במאגר קלה עם pH אופטימלי והרכב יוניים מומלצת לטיהור רטרווירוסים ברוב של פארא bacilliform. אוריאה משמש כדי לשבור את גופות הכללה השמנה virions כדי להמיס את מרכיבי התא. צנטריפוגה דיפרנציאלית מספק יותר הפרדה בין virions של הצמח מזהמים. אז טיפול proteinase K מסיר את capsids. לאחר מכן ה-DNA הנגיפי הוא מרוכז, המשמש עבור הדור הבא רצפי (הגדרות). הגדרות הנתונים משמשים כדי להרכיב contigs אשר מוגשים ל NCBI-BLASTn לזיהוי תת-ערכה של וירוס רצפים ב- dataset שנוצר. צינור מקבילים, RNA הוא מבודד עלים נגועים בשיטה סטנדרטית מבוססי עמודה RNA החילוץ. ואז ריבוזום דלדול מבוצעת להעשיר של תת-קבוצות של תעתיקים mRNA ווירוסים. רצפים שהורכב נגזר RNA רצף (RNA-seq) הוגשו NCBI-BLASTn לזיהוי תת-ערכה של רצפי וירוס זה הנתונים (dataset). במחקר שלנו, זיהינו שני הגנום קשורים badnavirus באורך מלא ב- datasets שני. שיטה זו היא העדיף גישה נפוצה אחרת אשר מחלץ את האוכלוסייה צבירה של רצפי RNA קטנים כדי לשקם רצף גנומית של וירוס צמח. וירוס משחזרת צינור זה metagenomic האחרון הקשורים רצף זה תעתיק רטרו אלמנטים מוכנסים לתוך הגנום הצמח. זה הוא מצמידים מבחני ביוכימית או מולקולרית להבחין עוד יותר את פעיל מדבקים. הגישה שתועדו במחקר זה, משחזרת רצפים נציג של שכפול וירוסים צפויה להצביע על זיהום בנגיף פעיל.
Introduction
מחלות צמחים המתעוררים כונן החוקרים לפתח כלים חדשים כדי לזהות את agent(s) סיבתי הנכון. דוחות ראשונית של מחלות וירוס חדש או חוזרות מבוססים על סימפטומים המתרחשים בדרך כלל כמו פסיפס של מומים של עלים, וריד ניקוי, גמדות, כמישה, נגעים, נמק, או תסמינים אחרים. התקן עבור דיווח וירוס חדש כמו סוכן סיבתי של מחלה היא להפריד אותו פתוגנים מזהמים אחרים, להפיץ את זה בפונדקאי מתאים, לשחזר את המחלה על ידי מזריקים לתוך צמחים בריאים של המין המארח המקורי. המגבלה בגישה זו היא כי סוגים רבים של וירוסים תלויים חרק או וקטורים אחרים לשידור פונדקאי מתאים או בחזרה המין המארח המקורי. במקרה זה, חפש את הווקטור המתאים יכול להיות ממושך, ייתכנו קשיים להקים מעבדה מושבות של וקטור ולאחר בהמשך המאמצים הדרושים כדי לתכנן עבור שידור ניסיוני פרוטוקול. אם לא ניתן להשיג את התנאים במחקרי מעבדה מוצלחת שידור, ואז העבודה נופל קצר התקן לדיווח מחלה וירוס חדש. וירוסים המתרחשים מארחיהם טבעי-titers נמוך מאוד, חוקרים עליך לזהות מארחים חלופי עבור הפצת כדי לשמור על מלאי זיהומיות מספיקות לביצוע המחקר. עבור מינים וירוס להדביק רק כמה צמחים זה יכול להיות גם מכשול לגידול תרבויות מניות1.
בשנים האחרונות מדענים הם להעסקת לעתים קרובות יותר תפוקה גבוהה הגדרות וגישות metagenomic לחשוף את רצפי וירוס הקיימים בסביבה, אשר קיים קשור מחלה ידועה, אך ניתן להקצות מינים בטקסונומיה ועוד סוגים 2 , 3 , 4. כזו גישות גילוי של אפיון חומרים גנטיים בסביבה ברורים לספק דרך לתאר את וירוס המגוון בטבע או נוכחותם מערכת אקולוגית מסוימת אבל לא בהכרח אישור מסגרת עבור הגדרת סוכני סיבתי עבור מחלה נראית לעין.
הסוג Badnavirus שייך למשפחת Caulimoviridae של pararetroviruses. וירוסים אלה הם bacilliform במצב עם גדיל כפול מעגלית DNA הגנום של-7 עד 9 kb. Pararetroviruses כל לשכפל דרך ביניים RNA. Pararetroviruses קיימים episomes וישכפלו עצמאית של הצמח בהכפלת ה-DNA5,6. מחקרים בתחום של אוכלוסיות וירוס עולה כי אוכלוסיות וירוס אלה מורכבים מבחינה גנטית. בנוסף, המידע שבידי במגוון של הצמח הגנום רצפי התפוקה גבוהה חשפו דוגמאות רבות badnavirus הגנום שברי המוכנסים על-ידי שילוב לגיטימי אירועים צמח הגנום. רצפי אנדוגני badnavirus אלה אינם קשורים בהכרח זיהום7,8,9,10,11. כתוצאה מכך, השימוש של הגדרות לזיהוי badnaviruses חדש כסוכן סיבתי של המחלה הינו מסובך המגוון subpopulation של הגנום episomal, כמו גם המופע של רצפי אנדוגני12,13.
אמנם לא אחת צינור אופטימלית עבור הגילוי של הגנום pararetrovirus הרומן, ישנן שתי גישות נפוצות כדי לזהות וירוסים אלה כסוכני סיבתי למחלה. שיטה אחת היא להעשיר עבור רצפי RNA קטן של עלים נגועים, ואז להרכיב את רצפי אלה צריך להשרות וירוס genome(s)14,15,16,17. גישה אחרת היא הגברה מעגל מתגלגל (RCA) כדי להגביר את הגנום של וירוס ה-DNA מעגלי18. ההצלחה של RCA תלוי גיל העלה כייל את וירוס בתוך הרקמה שנבחרו. המוצרים RCA נתון עיכול הגבלה, משובטים לתוך פלסמידים עבור ישיר רצף19,20,21.
Canna לנמר צהוב וירוס (CaYMV) badnavirus, מתואר הגורם etiological של מחלת לנמר צהוב קאנה, למרות רק שבר bp 565 של הגנום היה בעבר מבודד מן צפורנים נגוע22. מחקר עכשווי מזוהה CaYMV ב Alpinia purpurata (פריחה ג'ינג'ר; CaYMV-Ap)23. מטרתו של מחקר זה היה להתאושש רצפי הגנום badnavirus מלאה canna נגוע חבצלות. אנחנו מתארים עבור טיהור וירוס מן הצמח מזהמים ולאחר מכן לבודד את ה-DNA הנגיפי של הכנת פרוטוקול והכן ספריית DNA לשימוש המיתרים. גישה זו מבטלת את הצורך עבור שלבי ביניים הגברה מולקולרית. אנחנו גם לבודד mRNA מצמחים נגועים עבור ה-RNA-תת סעיף המיתרים, אשר כוללת RNA-seq בוצע באמצעות כל הכנה חומצת גרעין. Contigs התאספו נמצאו להתייחס טקסון Badnavirus ב שני datasets באמצעות המרכז הלאומי עבור ביוטכנולוגיה ו מידע (NCBI) יישור המקומי בסיסי בכלי החיפוש עבור חומצות גרעין (BLASTn). זיהינו הגנום של badnavirus שני מינים24.
Protocol
1. כללי וירוס טיהור על ידי צנטריפוגה דיפרנציאלי בשיטה סטנדרטית על ידי קובי. et al. 25
- ראשית, לחתוך 80-100 גר' עלים של צמחים, טוחנים בבלנדר וארינג ב 4 ° C באמצעות 200 מ ל טחינת מאגר (0.5 M NaH2PO4, 0.5 M נה2HPO4 (pH 7.2). ו- 0.5% (w/v) נה2אז3). ללבוש מעיל מעבדה וכפפות עבור כל שלבי של הליך זה.
- לאחר מכן, העבר את homogenate (300 מ ל) גביע 1.0 L. להוסיף homogenate בתוך ברדס כימי 18 גרם אוריאה ו- 25 מיליליטר 10% nonionic דטרגנט (t-Oct-C6H4-(ו2CH2) או9).
הערה: בשלב זה מומלץ ללבוש בטיחות משקפי מגן ומסכה נשימה פשוטה להגנה אישית. - מערבבים עם פגים בקצרה בשכונה ולכסות את. הספל עם מסכל. ואז להעביר את הספל מכוסה בנייר כסף חדר קר ומערבבים בעזרת פגים בן לילה ב 4 º C.
- להעביר את homogenate כדי צנטריפוגה הרוטור בקבוקים (מכולות 250 מ ל), צנטריפוגה, רוטור זווית קבועה ב 4,000 x g 10 דקות ב 4 º C. בשכונה fume כימי, לשחזר את תגובת שיקוע וסינון באמצעות 4 שכבות של גזה.
- לחלק את homogenate בין צינורות פוליפרופילן צנטריפוגה 38.5 mL צנטריפוגה עבור 2.5 h ב g 40,000 x-4 מעלות צלזיוס. בדרך כלל, לבדוק נוכחות של גלולה ירוק בחלק התחתון של הצינור, גלולה לבנה לאורכו של הצינור. יוצקים את תגובת שיקוע ולשמר את שני כדורי; במקום דוגמאות על קרח.
הערה: בגדר ירוק מכיל מהכלורופלסט עמילן, organelles אחרים. - עובד בברדס כימי, השתמש שוטר גומי כדי להפריד את כדורי. Resuspend בגדר לבן בבקבוק כל רוטור ב 1 מ"ל של ddH2O במהלך 1-2 h תוך שמירה שהמתלים לילה ב 4 ° C כדי לאפשר את החומרים לפזר את מלא פתרון. Centrifuge התליה בגובה 6000 x g ו- 4 מעלות צלזיוס למשך 10 דקות לפנות את הפסולת הנותרת.
- Centrifuge התליה מרוכזת בשעה 136,000 g x עבור 2 h-4 ° C עד הצניפה virions. Resuspend כדורי ב 1 מ"ל של מאגר (50 מ מ טריס-HCl, pH 7.5, 5 מ מ MgCl2).
הערה: שלב אופציונלי זה לטפל virions עם DNAse אני (10 µg/mL) למשך 10 דקות ב 37 ° C כדי להסיר DNA שאינם encapsidated, קרי, מזהם כלורופלסט, דנ א מיטוכונדריאלי. לאחר מכן, בטל את DNAse אני על-ידי הוספת EDTA 1 מ מ. - לשבש את virions עם 40 µL של proteinase µg/µL 2 K ב 37 מעלות צלזיוס למשך 15 דקות.
- העבודה בתוך ברדס כימי להתאושש virion DNA על ידי מיצוי אורגני. ללבוש מגן הפנים, כפפות, מעיל מעבדה במהלך החילוץ להגנה מפני השפעות בריאותיות חריפה פוטנציאליים. להוסיף 1 כמות אלכוהול פנול-כלורופורם-isoamyl (49:50:1) הדגימה, לנער בעבודת יד 20 ס' צנטריפוגה בטמפרטורת החדר במשך 5 דקות ב- 16,000 x ג להסיר את פאזה מימית העליון ואת העברת צינור חדש. חזור על החילוץ הזה פעמיים או יותר. השלך שלב אורגני על-ידי הצבתו בבקבוק פסולת זכוכית עבור סילוק נכון כימי מוסדיים26.
- לרכז את הדנ א באמצעות אתנול משקעים. השתמש 0.3 M הריכוז הסופי של סודיום אצטט (pH 5.2) ואמצעי אחסון 2.5 של 95% אתנול. הצב דגימות ב-20 ° C 30-60 דקות, צנטריפוגה-13,000 g x עבור 10-20 דקות הצניפה דנ א26.
- עובד על ספסל מעבדה, resuspend בגדר דנ א ב 1 מ"ל של 0.1 מ מ טה מאגר (pH 8.0). במיון ההשעיה דרך הג'ל המסחרי סינון העמודה (בדרך כלל משמש עבור תגובת שרשרת פולימראזית (PCR) לנקות) לחסל את מלחי והחומרים משקל מולקולרי נמוך עלול לעכב את המיתרים.
- לנתח את הדגימות ידי 1% agarose בג'ל באמצעות אתידיום ברומיד מכתים כדי להציג את האיכות של ההכנות. להעריך את האיכות של הדנ א באמצעות ספקטרופוטומטרים nanodrop.
הערה: יחס של ספיגת מדגם 260 λ ו λ 280 בין 1.85 ו- 2.0 מציין בדרך כלל כי ההכנה הוא "נקי" של זיהומים ויש באיכות הרצויה ביותר. - לנתח את האיכות של ה-DNA (השתמש pg 5 עד 10 ng) באמצעות שבב המבוסס על כלי נימי אלקטרופורזה.
הערה: פלט באיכות מראה נקי פסגות, המייצג את שברי DNA בחלוקה לפי גודל לאורך ציר x. שיא הגובה מצביע על שפע של השבר. פסגות משוננות מציינים שברי מפורק חלקית או מזהמים כימיים. עקומות עגול מייצגים כתם של DNA המציין באיכות ירודה
2. הספרייה באמצעות DNA והכנה מבוסס-אמולסיה הגברה המשובטים (emPCR הגברה)
הערה: הספרייה בדרך כלל מוכן על ידי מתקן הגדרות אשר מבוצעות מונחה לקוח.
- להטות את הפתרון של DNA (> 200 ng) באמצעות לתת לה משאף אשר ממירה את ה-DNA קטעים. מאתרים ומפסיקים את המתאמים מסחרי לפי ההוראות של המדריך27.
- לבצע את emPCR הגברה של דגימת דנ א על פי הוראות היצרן28,29,30. חזור על השלב שטיפת שלוש פעמים, לשטוף אחרי זה, גלולה החרוזים ב- minicentrifuge עבור ביטול ס' 10 את תגובת שיקוע לשטוף אחרי זה.
הערה: ההליך מתחיל עם הכנת חרוזים לכידה על ידי שטיפה במאגר שטיפת מסחרי מסופק עם הקיט. emPCR משמש בדרך כלל עבור תבנית הגברה עבור הגדרות. - חום denature את ה-DNA או RNA ב 95 מעלות צלזיוס במשך 2 דקות ולאחר מכן 4 ° C עד מוכן לשימוש. שימוש 200 - מיליון מולקולות של DNA/RNA ל 5 מיליון לכידת חרוזים באמצעי אחסון הסופי של µL 30. להכין מדגם מעושה לצד דגימת DNA/RNA ולבצע את הפעולות הבאות עם הדגימה חומצת גרעין, כמו גם את הדגימה מעושה.
- לבצע תחליב על-ידי vortexing ברכבת התחתית של תחליב שמן 10 s במהירות המרבית, ואז שופכים את כל התוכן (4 מ ל) לתוך פלסטיק זע צינור התואמת מהמגן פלטפורמה. במקום הצינור מלהיב על פלטפורמת לערבב האמולסיה ב-2000 סל ד במשך 5 דקות.
- לוותר על 100 aliquots µL של אמולסיה קאפ חשפנות-8 צינורות או לתוך צלחת 96-ובכן. קאפ הצינורות או לאטום את הצלחת ולבצע emPCR באמצעות תוכנית מומלצת של היצרן28.
הערה: לאחר השלמת ה-PCR, בדוק הבארות כדי לראות אם האמולסיה ללא פגע ולאחר מכן המשך. למחוק את הבאר כולו אם האמולסיה שבור. - שייפרס ולעבוד בשכונה כימי כדי לאסוף את החרוזים DNA מוגבר (ADB). ואקום תשאף האמולסיה מן הבארות ולאסוף את החרוזים בשפופרת 50 מ. לשטוף את הבארות פעמיים עם 100 µL של אלכוהול איזופרופיל, תשאף על רינס 50 מל אותו לשפופרת הזו.
- מערבולת שנאספו אמולסיות, resuspend של ADB עם אלכוהול איזופרופיל לאמצעי הסופי של 35 מ ל. גלולה ADB-g 930 x עבור 5 דק. הסר את תגובת שיקוע והוסף 10 מ"ל של שיפור מאגר. מערבולת ADB, לשטוף לאחר מכן על-ידי הוספת אלכוהול איזופרופיל לאמצעי הסופי 40 מ"ל, צנטריפוגה להשליך תגובת שיקוע לאחר כל שטוף, חזור על השלב לשטוף פעמיים.
- לבצע שטיפה הסופי באמצעות אתנול במקום אלכוהול איזופרופיל. להוסיף שיפור מאגר נפח סופי מ"ל 35, מערבולת של גלולה החרוזים-930 g x עבור 5 דק. להסיר את תגובת שיקוע אבל להשאיר 2 מ של שיפור מאגר.
- להעביר את המתלים צינור microcentrifuge, בקצרה צנטריפוגה כדי גלולה של ADB. לאחר השמטת את תגובת שיקוע, לשטוף בגדר ADB פעמיים עם 1 מ"ל של מאגר וחלוקתו. צנטריפוגה ולמחוק את תגובת שיקוע לשטוף אחרי זה.
- כדי להכין העשרת חרוז של ספריית ה-DNA, להוסיף 1 מ"ל של 1 N NaOH החרוזים. מערבולת ADB ואז דגירה למשך 2 דקות בטמפרטורת החדר. צנטריפוגה וזורקים את תגובת שיקוע. חזור על שלב זה לשטוף פעם אחת.
- להוסיף 1 מ"ל מאגר מחזק, ואז מערבולת ADB, דגירה למשך 2 דקות בטמפרטורת החדר. בקצרה צנטריפוגה ולמחוק את תגובת שיקוע. חזור על שלב זה שוב באמצעות µL 100 מאגר מחזק.
- כדי anneal תחל רצף הדי, להוסיף µL 15 של פריימר Seq A ו- µL 15 של Seq פריימר B המסופק בערכה. בקצרה לערבב על ידי vortexing ומניחים את הצינור microcentrifuge בתוך גוש חום-65 מעלות צלזיוס במשך 5 דק העברה קרח למשך 2 דקות.
- לשטוף שלוש פעמים עם 1.0 מ"ל של חישול מאגר. מערבולת עבור 5 s ו להשליך תגובת שיקוע בכל פעם.
- לפני רצף, למדוד את מספר חרוזים באמצעות מונה חרוז מסחרי. צריך להיות לפחות 500,000 חרוזים מועשר.
הערה: הדלפק חרוז הוא מכשיר מיוחד שמודד את החרוזים צינור microcentrifuge שסופקו.
3. גנרל mRNA בידוד, dsDNA סינתזה החל נגוע קאנה יוצא כי מבחן מאת RT-PCR על CaYMV באמצעות דיווח על אבחון תחל
- לובשים מעיל מעבדה, כפפות גומי להגנה אישית כל השלבים הבאים. עובד על ספסל מעבדה, לאסוף דגימות 12 מן העלים, טיבלו את הדגימות חנקן נוזלי. השתמש טחנה חרוז המגון. להשתמש ערכת מסחרי המספק שיטה המבוססת על עמודה סטנדרטית למפעל הכולל בידוד ה-RNA. הוספת המאגר פירוק guanidine-isothiocyanate שסופקו על-ידי ערכת דגימת קרקע ו- shake 20 s.
- הוספת אתנול ומערבבים באופן יסודי, על-פי הוראות ערכת. להוסיף homogenate כל עמודה ספין המאגד RNA הקרום. רוחצים שלוש פעמים את elute את הרנ א לתוך שפופרת התאוששות24.
- לכמת את הרנ א באמצעות ספקטרופוטומטרים כדי למדוד את היחס בין ספיגת-260 λ ותקינות λ 280. בדוק הרנ א באמצעות 1% agarose בג'ל מוכתם אתידיום ברומיד.
הערה: יחס ספיגת בין 1.85 ו- 2.0 מציינת כי ההכנה הוא באיכות הרצויה ביותר. מתייחסים RNA עם DNase אני (10 µg/mL) למשך 10 דקות ב 37 º C. להשתמש בעמודת ספין מסחרי דמעות RNA RNase ללא מים31. דוגמאות בריכה RNA לפני שתמשיך. - להשתמש ערכת להסרת rRNA כדי להסיר צמח ribosomal RNA. Aliquot beads מגנטי microcentrifuge tube, לשטוף פעמיים עם RNase ללא מים. מערבולת הצינור aliquot כדי resuspend, מניחים את הצינורית על דוכן מגנטי ולחכות נוזלי לנקות. למחוק את תגובת שיקוע והחלף הפתרון resuspension חרוז מגנטי. מערבולת resuspend ולהוסיף 1 µL של RNase מעכב.
הערה: ערכות כאלה להשתמש oligo-dT קשורה beads מגנטי אשר hybridize ל mRNA. השיטה משתמשת בטכנולוגיית ההפרדה חרוז מגנט רגיל לשחזר תעתיקים24. - Ng לשלב 500 כדי µg 1.25 של RNA, RNase ללא מים, מאגרי התגובה שסופקו על-ידי ערכת. מניחים את התערובת למשך 10 דקות ב 50 º C. מסירים מהאש ומוסיפים beads מגנטי שטף במים חינם RNAse. מערבולת בקצרה ולהגדיר בטמפרטורת החדר במשך 5 דקות.
- מניחים על דוכן מגנטי ולחכות הנוזל לנקות. להעביר את תגובת שיקוע צינור microcentrifuge טריים. הגדר על קרח.
- להשתמש בשיטה המבוססת על פתרון לכידת העשרה של exosomes ל- 200 ng של RNA להכין ספריית cDNA.
הערה: ספריית cDNA גדיל כפול בדרך כלל מוכן על ידי מתקן הגדרות אשר מבוצעות מונחה לקוח. - קטע את הרנ א באמצעות פתרון פיצול RNA מסחרי (0.136 g ZnCl2 ו- 100 מ מ טריס-HCl pH 7.0). להוסיף 2 µL של פתרון µL 18 של RNA (200 ng סה כ). ספין צינורות בקצרה microcentrifuge, מקום הדגימות ב 70 ° C s 30, ואת העברת לקרח. לעצור את התגובה באמצעות 2 µL של 0.5 M EDTA pH 8.0 ו 28 µL של 10 מ מ טריס-HCl pH 7.5.
- לאגד RNA beads מגנטי על ידי ערבוב בטמפרטורת החדר במשך 10 דקות שימוש concentrator מגנטי כדי לאסוף את החרוזים ולמחוק את תגובת שיקוע. לשטוף את החרוזים שלוש פעמים עם 200 µL של 70% אתנול. ביטול כל לשטוף, ואז האוויר יבש החרוזים pelleted בטמפרטורת החדר במשך 3 דקות Resuspend ב- 19 µL של 10 מ מ טריס-HCl pH 7.5.
- Anneal תחל אקראית ל RNA מפוצלים על ידי חימום עד 70 מעלות צלזיוס למשך 10 דקות ולאחר מכן מקם את הצינורית על הקרח 2 דק. הכן הראשון סטרנד, השני cDNA סטרנד באמצעות ערכת סינתזה cDNA מסחרי רגיל.
- לטהר את cDNA כפול-strand באמצעות רכז חרוז מגנטי. לשטוף עם 800 µL של 70% אתנול שלוש פעמים. ביטול כל לשטוף, אוויר יבש כדורי בטמפרטורת החדר במשך 3 דקות Resuspend ב 16 µL של 10 מ מ טריס-HCl pH 7.5. השתמש במסוע חרוז המגנטי להפריד את החרוזים cDNA גדילי כפול, אשר כעת בפתרון. הסר את cDNA מאת pipetting לתוך צינור חדש של ה-PCR 200 µL.
- לבצע קטע סיום תיקון באמצעות אנזימים ותערובת של deoxyribonucleotides שסופקו על-ידי ערכת הכנה ספריית מסחרי. ערכת מסחרי מספק מתאמי מדולל מראש כדי להוסיף על שני קצוות cDNA כפול-strand באמצעות ליגאז מסחרי ב 25 מעלות צלזיוס למשך 10 דקות.
4. הגדרות של DNA ספריית המוכן dsDNA ספריית מקריסטלים של mRNA והכנה וירוס גולמי
- השתמש מכשיר pyrosequencing תפוקה גבוהה רגיל ופעל פרוטוקולים מומלץ כל היצרנים לייצר המפרט ישירה של רצפי DNA. שימוש מסחרי רצף ריאגנטים, כולל fluorescently תוויות נוקלאוטידים.
הערה: עבור פרטים עיין ההוראות של היצרן שסופקו עם המכשיר. - לבצע רצף שלאחר ניתוח באמצעות תוכנה מכלול הגנום אשר באופן אוטומטי מרכיב קריאות לייצר את הסט הראשון של contigs באורך ממוצע של < 700 bp. להשתמש בתוכנה FastQC באתר האינטרנט של iPlant/CyVerse אשר מבצעת בקרת איכות בדיקות נתונים גולמיים רצף32. בחר רצפי עם ציונים Phred ≥ 30 להמשיך לבנות רצפים ארוכים יותר מ קטן רצף קריאות24 באמצעות מיפוי ותוכנות אמפליקון.
הערה: עבור פרטים עיין הוראות היצרן. - שלח אלה contigs התאספו כדי ניתוח NCBI-BLASTn באמצעות מודול ברירת המחדל MEGABLAST, כמו גם Viridplantae (TaxID: 33090) ווירוסים (TaxID: 10239) כפי organismal המגבילה את שמות33. לאסוף subpopulation של contigs שמראים דמיון גבוה ל דיווח על הגנום Badnavirus לדוח.
- ודא כי פיגומים הצטרף המייצגים אחד או יותר המועמד וירוס באורך מלא הגנום, לייצר כראוי במסגרת רצפים שיש באותו הארגון כמו הגנום badnavirus רגיל. כדי לעשות זאת, קלט את הגנום של וירוס באורך מלא המועמד לתוך פלסמיד תוכנת ציור. ואז לאשר נוקלאוטידים קודם 15 מורכב tRNAפגש (TGGTATCAGAGCGAG) אשר מאוד כולו בין badnaviruses. אתר את הסימן פוליאדנילציה פוטנציאליים, לקראת סיום 3' של הגנום. הוספת ביאורים הגנום המלא כדי לזהות הנוכחות של שני ORFs קטנים, אחד ORF גדול קידוד של polyprotein. מכן להשתמש בפורטל ExPASy לתרגם את הכלי כדי לזהות את badnavirus ORF1, ORF2 ו- ORF3 תרגום מוצרים34.
הערה: תוכנה מדעית זו היא בחינם, להפיק DNA מעגלי, לזהות קריאה פתוחה כל מסגרות, ומספק פלט מיידית כדי לוודא כי הרצף מייצג את הגנום DNA מעגלי באורך מלא. - השתמש בקוד פתוח כלי השוואה רצף מרובים, שריר, CLUSTALW, כדי להשוות בין הגנום וירוס המתקבל DNA ו- RNA ניתוחים35,36.
- חפש את מסד הנתונים נוקלאוטיד NCBI כדי להשיג את רצפי הגנום המלא של 30 badnavirus מינים ולייצא אותם כמסמך בתבנית .fasta. להעלות רצפים תוכנה אבולוציוני גנטי אנליזה של רצפי יחד עם רצפי הגנום וירוס מתקבל על ידי הגדרות. צור מספר רצף היישורים ועצים מקסימום הסבירות באמצעות שריר37.
5. איכות הערכה של דה נובו רצף על ידי ה-PCR הגברה של הגנום של וירוס מצמחים נגועים
- קלט את רצפי הגנום שזוהה badnavirus באורך מלא (בפורמט .fasta) לתוך הכלי Primer3 מקוון חינם כדי להפיק PCR תחל38. לזהות ערכות פריימר יהיה לייצר מוצרים רציף של 1000-1500 bp לכל אורכו של genome(s) וירוס. שלח את רצפי למתקן שירות לסנתז ולספק תחל ה-PCR.
הערה: הפלט מזהה פריימר מקובל זוגות עם טמפרטורות ההיתוך נפוץ ומקובל ומקומות פריימר מדויק לאורך הרצף הציג. - עובד על ספסל מעבדה, לובשת חלוק מעבדה וכפפות, לבודד µg 5 של ה-DNA מן העלים הנגועים בנגיף שליטה בריאים באמצעות שיטה אוטומטית המערבת תאית פאראמגנטיים סטנדרטי חלקיקים כדי לבודד דנ א של הצמח גשמי39 . להקפיא עלים חומר (20-40 מ ג) בחנקן נוזלי בשפופרת microcentrifuge ומשתפשף באמצעות מפעל חרוז. לשלב את הדגימה עם פירוק המאגר בצינור microcentrifuge ולהוסיף RNase A כל דגימה. מערבולת המדגם עבור 10-20 s ו בקצרה לסובב את הדגימה כדי להסיר חלקיקים מוצקים.
הערה: תאית פאראמגנטיים חלקיקים יש קיבולת גבוהה של איגוד ה-DNA, לבודד תפוקה גבוהה של דנ א טהור. השיטות עמודה סיליקה מסחרי רגיל עבור בידוד של דנ א לא ביעילות לחלץ DNA מתוך מגוון רחב של מיני צמחים. כתוצאה מכך, עשרות שיטות קיימות הן שינויים של הליכים אלה כדי לשפר את היעילות של מיני צמחים בודדים. השיטה של חלקיקים תאית פאראמגנטיים אוטומטיות נבחר בגלל זה מניב יותר ואיכותי יותר DNA מינים עשבוניים תוכן יותר מ 2540. - השתמש ריאגנט מסחרי מחסניות לבידוד DNA פאראמגנטיים אוטומטית. להוסיף 300 µL של מים בחינם נוקלאז כל ריאגנט מסחרי להעביר ועם מחסנית צמח lysate את המחסנית זהה. הכנס את מיכל הדיו המדף מחסנית, מקום פומפה בבאר קרוב אל הצינור • תנאי ולמקם • תנאי מאגר לתוך הצינור • תנאי. לטעון מחסניות לתוך המכונה בידוד חומצת גרעין אוטומטית ולהפעיל את הצמח DNA בידוד פרוטוקול41,42.
- לבצע את ה-PCR להפיק סדרת מוצרי ה-PCR חופפים. השתמש 5 מיקרומטר של כל פריימר ואחורה עם 35 מחזורי PCR הגברה. השתמש אופניים התנאים הבאים: דנטורציה ב 95 ° C עבור 60 s, חישול ב 50 מעלות צלזיוס במשך 45 s, והסיומת ב-72 מעלות למשך 1-2 דקות עם סיומת הסופי ב-72 מעלות במשך 7-10 דקות שימוש בעמודה סינון ג'ל ארוזים מראש כדי למנוע מלחי והחומרים משקל מולקולרי נמוך כמו בשלב 1.231.
- לחשב יחס טוחנת 3:1 של מוצר ה-PCR וקטוריים כדי לקבוע את כמות המוצר PCR מאתרים ומפסיקים 50 ng של pGEM ליניארית פלסמיד43. השתמש בפקד הכנס ה-DNA כדי לקבוע אם ligations לעבוד בצורה יעילה. לבצע את מצדו תוך שימוש T4 DNA ליגאז (3 U µL) ב 4 º C. ואז שינוי צורה מסחרית מוכנה JM109 המוסמכת Escherichia coli תאים. שימוש לשלוט pg 100 של נימולים פלסמיד DNA כפקד חיובי לשינוי יעיל. צלחת 100 µL של תאים טרנספורמציה על גבי לוחות LB-אגר עם הבחירה אנטיביוטי ו כחול/לבן לשחזר פלסמידים מחוברים26. דגירה לוחות עבור 16-24 h ב- 37 מעלות צלזיוס.
הערה: וקטור pGEM יש גן lacZ שמקודד β-galactosidase. חיידקים טרנספורמציה גדל על צלחת המכיל אמפיצילין µg/mL 100, 0.5 מ מ IPTG, 80 µg/mL 5-bromo-4-chloro-3-indoyl-β-D-galactopyranosidase (X-גל) יהפוך כחול בגלל β-galactosidase פעילות. פלסמיד pGEM זו לליניארית באופן משבש את הגן lacZ . המושבות המכילות את הכיסויים מוצר ה-PCR לשבש את הגן lacZ, לא לעכל X-גל. מושבות אלו הם לבנים. לפיכך ניתן להבחין מושבות עם מוסף מאלה בלי מוסף לפי הצבע של המושבה (לבן מול הכחול)26. - לבודד את ה-DNA של שלוש מושבות באמצעות ערכת של בידוד פלסמיד מבוססי עמודה סטנדרטית39. רצף שלושה פלסמידים של שינוי מוצר. להשוות כל רצף ה-DNA הגנום של וירוס דה נובו התאספו המיוצר על ידי המיתרים. השתמש CLUSTALW כדי ליישר את רצפי ולהבטיח כי הם מסודרים כראוי.
תוצאות
שיטת טיהור זה וירוס ששונה מסופקים של העשרה של וירוס DNAs שימושית לצורך זיהוי שני מינים וירוס הגדרות, ביואינפורמטיקה. לאחר homogenate היה centrifuged-g 40,000 x עבור 2.5 h, היה גלולה ירוק בחלק התחתון של הצינור, גלולה לבנה לאורכו. בגדר ירוק היה resuspended לתוך שפופרת אחת microcentrifuge, בגדר לבן היה resuspended לתוך שני צינורות microcentrifuge. PCR בוצע באמצעות תקן ה-PCR CaYMV תחל אבחון, מוצרים אותרו בגדר לבן solubilized, לא בגדר ירוק (איור 1א'). מדגם של הכנת גס נבדק על ידי במיקרוסקופ אלקטרונים הילוכים, הבחנו חלקיקים bacilliform מדידה 124-133 ננומטר אורך (איור 1B). זה במסגרת אורך מודאלי החזוי badnaviruses רוב. דנ א היה מופק כדורי לבן וירוק, resuspended בנפרד. איור 1C, שנעמיס 5 µL של ה-DNA שחולצו מן הירוק, הדגימה גלולה לבנה (1.6 µg של ה-DNA של השבר ירוק) ו- 3.1 µg של ה-DNA של השבר הלבן כדי 0.8% agarose ג'ל אלקטרופורזה וניתח את ה-DNA בעקבות אתידיום ברומיד מכתים. השבר ירוק הכיל משקל מולקולרי נמוך דנ א ואילו השבר הלבן הפיק שתי להקות של משקל מולקולרי גבוה יותר דנ א, כמו גם משקל מולקולרי נמוך דנ א (איור 1C). הג'ל המוצגים באיור 1C היה לרוץ במשך 40 דקות ב- 100 וולט ומציעה השמצות בנתיב 3 כי המתח ג'ל צריך להיגרע לייצר להקות יותר ברור. נתונים אלו מראים כי בגדר לבן היה מועשר עבור virions. הריכוז (0.6 µg/mL) ה-DNA שחולצו מן המדגם לבן היה נמוך, אבל נאותה המיתרים, אשר דורשת מינימום של 10 ng של ה-DNA כדי להמשיך. DNAs מפוצלים שימשו כדי להתכונן ספריה המיתרים.
במקביל, RNA שהופק מצמחים נגועים קאנה (איור 1D) עבור תפוקה גבוהה RNA-תת סעיף זרימת עבודה סטנדרטי בוצע עבור ספריית הכנה, המיתרים, יצירת contigs וזיהוי של רצפי גנום ויראלי (איור 1E). תוצאות הפלט באמצעות DNA ו- RNA כמו חומרי מוצא הושוו.
השגנו 188,626 קריאות ה-DNA raw על-ידי הגדרות באמצעות DNA מבודד הכנת וירוס גולמי. קריאות כונסו לתוך 13,269 contigs, BLASTn נעשה שימוש כדי לחפש את ערכת הנתונים NCBI של רצפי (באמצעות Viridplantae TaxID: 33090, וירוס TaxID: 10239 בשם המגביל האורגניזמים) (איור 1E). התוצאות NCBI-BLASTn גילה כי 93% contigs דה נובו התאספו היו הסלולר רצפים, 22% היו ידועות, 0.3% היו וירוס contigs (איור 2א). רוב contigs, מסווגות רצפים הסלולר אותרו גם מיטוכונדריאלי או כלורופלסט הדנ א. בתוך ערכת הנתונים של וירוס contigs, 32% של contigs וירוס היו קשורים לחברי Caulimoviridae (שלא היו רצפים Badnavirus) ו- 58% מאלה שהיו קשורים Badnavirus. של contigs וירוס, 29% היו מאוד דומות (e < 1 x 10-30) עם ג ' ין V17 ORF3 ולבודד CaYMV (EF189148.1), קנה סוכר bacilliform וירוס לבודד Batavia D, הגנום המלא (FJ439817.1), ושל בננה פס CA וירוס להשלים את הגנום ( KJ013511). בתוך אוכלוסייה זו, היו contigs רב כשהופיעה שני באורך מלא הגנום.
תפוקה גבוהה RNA-seq המיוצר 153,488 רצף בודדים נקי קריאות עם ממוצע לקרוא אורך < 500 bp. Contig הרכבה מופחת זה כדי 8,243 contigs. אלה הוגשו NCBI-BLASTn (באמצעות Viridplantae TaxID: 33090, וירוס TaxID: 10239 בשם המגביל האורגניזמים), ואת התוצרים יכניסו 76% contigs קטגוריה של רצפים תאי צמח, 23% היו ידועות. 0.1% היו כידידותיים וירוס contigs ( איור 2 B). בחינה מדוקדקת יותר של האוכלוסייה באוכלוסייה 0.1% של וירוס contigs נקבע כי 68% מאלו שהוזמנו Caulimoviridae (איור 2B). 3 contigs גדולות בתוך אוכלוסייה זו זוהו עם דמיון גבוה (e < 1 X 10-30) CaYMV לג'ין ולבודד V17 ORF3 (EF189148.1), קנה סוכר bacilliform וירוס לבודד Batavia D, הגנום המלא (FJ439817.1) ופס בננה וירוס CA הגנום המלא (KJ013511). בחינת contigs של שלוש, הצטרפנו ידנית שתיים כאלה כדי לייצר גנום הוירוס באורך מלא.
השווינו את contigs אורך הגנום של וירוס המיוצר על ידי רצף DNA ו- RNA בדומה לפיגום הדדית כדי לאשר הנוכחות של שני הגנום של וירוס באורך מלא. גנום הוירוס באורך מלא אחד של 6,966 bp באופן לא סופי בשם וירוס לנמר צהוב הקשורים Canna 1 (CaYMAV-1) (איור 3א). הגנום השני היה 7,385 bp ווריאציה של CaYMV הדבקה Alpinia purpurata (CaYMV-Ap01) (איור 3א).
לבסוף, תחל PCR אשר נועדו שיבוט ~ 1,000 שבר bp של וירוס, שימשו באופן שונה לזהות שני הגנום בקרב אוכלוסיה של צמחים canna 227 המייצגים תשע זנים מסחריים. במקרים רבים נדבקו צמחים בודדים עם שני וירוסים. אנו מספקים דוגמא RT-PCR לזיהוי של CaYMAV-1 ו- CaYMV-Ap01 את הצמחים 12. שלושת אלה היו חיוביות רק עבור CaYMV-Ap01, 9 היו חיוביים שני וירוסים (איור 3ב).

איור 1 : וירוס חומצת גרעין ההכנות של הגדרות זרימת העבודה. (א) Agarose (1.0%) ג'ל אלקטרופורזה של 565 bp PCR שברי הגנום CaYMV. שני מוצרים PCR התגלו דגימות שהוכנו בגדר לבן (מסלולים 1, 2) אך לא הדגימה צניפה ירוק (ליין 3). בקרה חיובית (+) מייצג את מוצר ה-PCR מוגבר מצמח נגוע דנ א זה הופרדה באמצעות שיטה אוטומטית מעורבים חלקיקים תאית פאראמגנטיים סטנדרטי. ליין L מכיל בסולם הדנ א המשמש כסטנדרט למדידת בגודל של להקות DNA ליניארי של נתיבים מדגם. (B) דוגמה חלקיקי וירוס להצגה על-ידי במיקרוסקופ אלקטרונים הילוכים ב בגדר לבן לשחזר אותו באמצעות fractionation גסה של canna נגוע עלים. (ג) Agarose (0.8%) ג'ל אלקטרופורזה של דנ א התאוששה הירוק (ליין 1) ולבן (ליין 2). גללים זה חיובי שנבדקו על ידי ה-PCR בלוח א הנקודות אדום וצהוב ליד ליין 2 לזהות שתי להקות DNA משקל מולקולרי גבוה המתרחשים השבר הלבן. (ד) Agarose (1%) ג'ל אלקטרופורזה של RNA הכולל התאושש על ידי טיהור RNA מבוססי עמודה. ליין L מכיל בסולם הדנ א המשמש כסטנדרט למדידת הגודל של להקות ליניארית ב נתיבים מדגם. ליין 1-6 מכיל RNA מבודד canna נגוע עלים אשר היו איחדו מדגם יחיד עבור דלדול ribo ו- RNA-תת סעיף (E) צינור סכמטי של חומצת גרעין ומשום, ספריית הכנה, רצף, contig הרכבה גנום הוירוס גילוי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
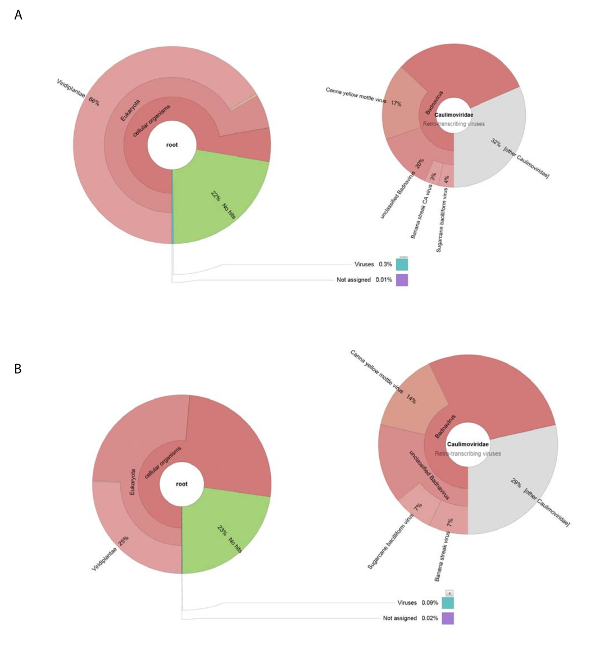
איור 2 : תרשימים כתר להמחיש את קטגוריות בטקסונומיה contigs. (א) התרשים על המופעים השמאלי שפע והפצה טקסונומי של contigs התכנסו from הכנת וירוס גולמי. התרשים הנכון מתארת את הפרופורציות של וירוס contigs הקשורים עם משפחת Caulimoviridae , Badnavirus סוג של שלושה מינים קרובים. (B) הלוח בצד השמאל מציג השפע של contigs נגזר RNA-seq בהתבסס על התפלגות בטקסונומיה שלהם. מימין נמצאת הגרף המתאר את השפע של contigs של contigs וירוס הקשורים עם משפחת Caulimoviridae , Badnavirus סוג של שלושה מינים קרובים בתוך האוכלוסייה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
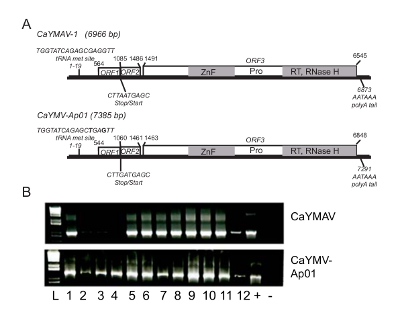
איור 3 . אפיון של הגנום CaYMAV-1 ו- CaYMV-Ap01. (א) Diagrammatic ייצוג של Canna צהוב לנמר לשייך וירוס 1 (CaYMAV) ו Canna לנמר צהוב וירוס דומה הגנום מבודד מן Alpinia purpurata (CaYMV-Ap01). נוקלאוטיד עמדות 1-10 מזוהה בתור ההתחלה של הגנום ומכיל tRNAפגש anticodon אתר אופייני של רוב הגנום badnavirus. העמדות להפסיק ולהתחיל לתרגום של מסגרת קריאה פתוחה (ORF) 1 ו- 2 סמוכים. חלבונים אלה יש נודע פונקציות. ORF3 הוא polyprotein המכיל אבץ אצבע (ZnF), פרוטאז (Pro), רוורס טרנסקריפטאז (RT) ותחומים RNAse H. 3' poly(A) אות רצף כולו עבור שניהם הגנום של וירוס. (B) RT-PCR ניתוח בוצע בעזרת RNA מבודד מן העלים וירוס נגוע, תחל המאתרות CaYMAV ו- CaYMV-Ap01. באוכלוסייה זהה של 12 תחנות, שלושה היו נגועים CaYMV-Ap01 בלבד, ואילו שאר נדבקו בנגיף הן CaYMAV והן CaYMV-Ap01. (+) מצביע על בקרה חיובית (-) מציין שליטה שלילי. נתון זה לשכפל/השתנו מ. Wijayasekara et al. 24 בעלי הרשאה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
בשנים האחרונות מגוון שיטות יש כבר מועסקים ללמוד מגוון ביולוגי צמחים וירוס סביבות טבעיות אשר כוללים מעשירה חלקיקי כמו וירוס (הוי) או וירוס מסוים RNA או DNA2,3,44, 45,46 . שיטות אלה, מלוות המיתרים וניתוח bioinformatic. מטרתו של מחקר זה היתה למצוא את סוכן סיבתי של מחלה נפוצה במפעל מעובדים. המחלה דווח כדי להיות התוצאה של הווירוס בעל חלקיקים שאינם העטופים bacilliform, ועל אשר כבר רק רסיס bp 565 משובטים47. מידע זה היה מספיק עבור חוקרים קודמים להקצות באופן היפותטי הווירוס הסוג Badnavirus בתוך המשפחה Caulimoviridae. בעוד דוחות קודמים שיערו כי מחלת לנמר canna canna חבצלות היה התוצאה של badnavirus יחיד, באמצעות גישה metagenomics המתוארים במחקר זה, קבענו כי המחלה נגרמה badnavirus סופית שני מינים24. לכן, הכוח של שימוש בגישה metagenome לגלות את סוכן סיבתי של המחלה הוא כי אנחנו מסוגל לזהות מצבים שבהן עשוי להיות גורם אחד או יותר.
הגישה שלנו שילוב נתונים רצפי DNA ו- RNA היא יסודית, גם מדגים כי התוצאות באמצעות שתי גישות הניבו תוצאות עקביות, אישר את נוכחותם של שני וירוסים הקשורים. אנו המועסקים הליך שונה עבור בידוד של caulimoviruses, המיוצר מדגם זה היה מועשר עבור חומצות גרעין וירוס הקשורים ועל זה היו מוגנים בתוך capsid וירוס. מעבדת שירות היה מתכווץ כדי לבצע את רצפי DNA. המושג חיוני עבור רצף דה נובו הוא שאת ה-DNA פולימראז משלבת את פלורסנט שכותרתו נוקלאוטידים לתוך בשרך תבנית ה-DNA במהלך מחזורי רציף של סינתזת ה-DNA. Contigs התאספו שהופעלו על-ידי הגדרות הוגשו לתוך זרימת עבודה bioinformatic בהפקת contigs כמה שמצבן contigs וירוס. אישור נוסף של וירוס שני הגנום10,24,48,49,50 הושג באמצעות ניתוח bioinformatic של RNA-seq והנתונים שהתקבלו מדולדל ribo ההכנות RNA. אחד התוצאה מעניינת היה ללמוד כי באוכלוסיות של רצפי לשחזר אותו באמצעות רצפי DNA ו- RNA מסופקים התפלגויות דומות של חומצות גרעין נגיפי וויראליים. עבור רצפי DNA ו- RNA, < 0.5% של רצפי היו ממוצא וירוס. בתוך האוכלוסייה של וירוס רצפים 78-82% היה שייך למשפחת Caulimoviridae. על ידי השוואת את contigs וירוס שהורכב מ רצפי DNA ו- RNA, אנחנו אישר כי הגנום התאספו שני התרחשה ב שני נתונים (datasets).
חשש של באמצעות רצפי DNA היחידה לזיהוי הגנום וירוס חדש הוא הגנום badnavirus הוא ה-DNA מעגלי פתוח. אנו לשער כי רצפים חופפים שיבושים הגנום יכול להציג מכשולים להרכבה הגנום של contigs. בדיקה ראשונית של התוצאות רצפי DNA חשף שתי הגנום וירוס דומה. שיערנו כי הגנום אלה מיוצגים גם מגוון גנטי של זן לא נחקרה, או מייצגים שני מינים משותפת מדביק אותו צמח24. לפיכך, ניתוח bioinformatic הקולקטיבי של נתונים (datasets) מתקבל על ידי הגדרות DNA ו RNA רצף, איפשרה האישור של הנוכחות של שני באורך מלא הגנום.
יש עוד דו ח אשר פיתח שיטה חלופית עבור חילוץ וי ו חומצות גרעין של צמח homogenates ללימודים metagenomic, המבוססים על שגרות להתאושש DNA כרובית פסיפס וירוס (CaMV; caulimovirus)3. גישה זו מזוהה הרומן RNA וחלבונים וירוס DNA בצמחים שאינם מעובדים. השלבים נגזר מן ההליך בידוד caulimovirus השתמשו במחקר זה לגלות את סוכן סיבתי של מחלה של צמחים מתורבתים הם בניגוד השלבים נגזר לחילוץ וי מ צמחים נגועים באופן טבעי24. ההצלחה של שתי השיטות ששונה מרמז כי ההליך במסגרת caulimovirus בידוד עשוי להיות נקודת התחלה יקר ללימודים metagenomic של הצמח וירוסים באופן כללי.
Disclosures
המחברים אין לחשוף.
Acknowledgements
המחקר מומן על-ידי אוקלהומה מרכז לקידום המדע, הטכנולוגיה להחיל מחקר התוכנית שלב II AR 132-053-2; ומחקר, אוקלהומה משרד החקלאות המומחיות יבולים גרנט תוכנית. אנו מודים דוקטור HongJin פאנג, ביואינפורמטיקה אוסו הליבה מתקן אשר נתמכה על ידי מענקים NSF (EOS-0132534) ו- NIH (2P20RR016478-04, 1P20RR16478-02, 5P20RR15564-03).
Materials
| Name | Company | Catalog Number | Comments |
| NaH2PO4 | Sigma-Aldrich St. Louis MO | S5976 | Grinding buffer for virus purification |
| Na2HPO4 | Sigma-Aldrich | S0751 | Grinding buffer for virus purification |
| Na2SO3 | Thermo-Fisher Waltham, MA | 28790 | Grinding buffer for virus purification |
| urea | Thermo-Fisher | PB169-212 | Homogenate extraction |
| Triton X-100 | Sigma-Aldrich | X-100 | Homogenate extraction |
| Cheesecloth | VWR Radnor, PA | 21910-107 | Filter homogenate |
| Tris | Thermo-Fisher | BP152-5 | Pellet resuspension& DNA resuspension buffers |
| MgCl2 | Spectrum, Gardena, CA | M1035 | Pellet resuspension buffer |
| EDTA | Spectrum | E1045 | Stops enzyme reactions |
| Proteinase K | Thermo-Fisher | 25530 | DNA resuspension buffer |
| phenol:chloroform:isoamylalcohol | Sigma-Aldrich | P2069 | Dissolve virion proteins |
| DNAse I | Promega | M6101 | Degrade cellular DNA from extracts |
| 95% ethanol | Sigma-Aldrich | 6B-100 | Virus DNA precipitation |
| Laboratory blender | VWR | 58984-030 | Grind leaf samples |
| Floor model ultracentrifuge &Ti70 rotor | Beckman Coulter, Irving TX | A94471 | Separation of cellular extracts |
| Floor model centrifuge and JA-14 rotor | Beckman Coulter | 369001 | Separation of cellular extracts |
| Magnetic stir plate | VWR | 75876-022 | Mixing urea into samples overnight |
| Rubber policeman | VWR | 470104-462 | Dissolve virus pellet |
| 2100 bioanalyzer Instrument | Agilent Genomics, Santa Clare, CA | G2939BA | Sensitive detection of DNA and RNA quality and quantity |
| 2100 Bioanalyzer RNA-Picochip | 5067-1513 | Microfluidics chip used to move, stain and measure RNA quality in a 2100 Bioanalyzer | |
| 2100 Bioanalyzer DNA-High Sensitive chip | 5067-4626 | Microfluidics chip used to move, stain and measure DNA quality in a 2100 Bioanalyzer | |
| Nanodrop spectrophotometer | Thermo-Fisher | ND-2000 | Analysis of DNA/RNA quality at intermediate steps of procedures |
| Plant total RNA isolation kit | Sigma-Aldrich | STRN50-1KT | Isolate RNA for RNA-seq |
| RNase-free water | VWR | 10128-514 | Resuspension of DNA and RNA for NGS |
| RNA concentrator spin column | Zymo Research, Irvine, CA | R1013 | Prepare RNA for RNA-seq |
| rRNA removal kit | Illumina, San Diego, CA | MRZPL116 | Prepare RNA for RNA-seq |
| DynaMag-2 Magnet | ThermoFisher | 12321D | Prepare RNA for RNA-seq |
| RNA enrichment system | Roche | 7277300001 | Prepare RNA for RNA-seq |
| Agarose | Thermo-Fisher | 16500100 | Gel analysis of DNA/RNA quality at intermediate steps of procedures |
| Ethidium bromide | Thermo-Fisher | 15585011 | Agarose gel staining |
| pGEM-T +JM109 competent cells | Promega, Madison, WI | A3610 | Clone genome fragments |
| pFU Taq polymerase | Promega | M7741 | PCR amplify virus genome |
| dNTPs | Promega | U1511 | PCR amplify virus genome |
| PCR oligonucleotides | IDT, Coralvill, IA | Custom order | PCR amplify virus genome |
| Miniprep DNA purification kit | Promega | A1330 | Plasmid DNA purification prior to sequencing |
| PCR clean-up kit | Promega | A9281 | Prepare PCR products for cloning |
| pDRAW32 software | ACAClone | Computer analysis of circular DNA and motifs | |
| MEGA6.0 software | MEGA | Molecular evolutionary genetics analysis | |
| Primer 3.0 | Simgene.com | ||
| Quant-iT™ RiboGreen™ RNA Assay Kit | Thermo-Fisher | R11490 | Fluorometric determination of RNA quantity |
| GS Junior™ pyrosequencing System | Roche | 5526337001 | Sequencing platform |
| GS Junior Titanium EmPCR Kit (Lib-A) | Roche | 5996520001 | Reagents for emulsion PCR |
| GS Jr EmPCR Bead Recovery Reagents | Roche | 5996490001 | Reagents for emulsion PCR |
| GS Junior EmPCR Reagents (Lib-A) | Roche | 5996538001 | Reagents for emulsion PCR |
| GS Jr EmPCR Oil & Breaking Kit | Roche | 5996511001 | Reagents for emulsion PCR |
| GS Jr Titanium Sequenicing kit* | Roche | 5996554001 | Includes sequencing reagents, enzymes, buffers, and packing beads |
| GS Jr. Titanium Picotiter Plate Kit | Roche | 5996619001 | Sequencing plate with associated reagents and gaskets |
| IKA Turrax mixer | 3646000 | Special mixer used with Turrax Tubes | |
| IKA Turrax Tube (specialized mixer) | 20003213 | Specialized mixing tubes with internal rotor for creating emulsions | |
| GS Nebulizers Kit | Roche | 5160570001 | Nucleic acid size fractionator for use during library preparations |
| GS Junior emPCR Bead Counter | Roche | 05 996 635 001 | Library bead counter |
| GS Junior Bead Deposition Device | Roche | 05 996 473 001 | Holder for Picotiter plate during centrifugation |
| Counterweight & Adaptor for the Bead Deposition Devices | Roche | 05 889 103 001 | Used to balance deposition device with picotiter plate centrifugation |
| GS Junior Software | Roche | 05 996 643 001 | Software suite for controlling the instrument, collecting and analyzing data |
| GS Junior Sequencer Control v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Run Processor v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS De Novo Assembler v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Reference Mapper v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Amplicon Variant Analyzer v. 3.0 | Roche | (Included in item 05 996 643 001 above) |
References
- Dijkstra, J., Jager, C. P. Practical Plant Virology : Protocols and Exercises. , Springer-Verlag. Berlin Heidelberg. 1 edn (1998).
- Roossinck, M. J. Plant virus metagenomics: biodiversity and ecology. Annu Rev Genet. 46, 359-369 (2012).
- Melcher, U., et al. Evidence for novel viruses by analysis of nucleic acids in virus-like particle fractions from Ambrosia psilostachya. J Virol Methods. 152 (1-2), 49-55 (2008).
- Stobbe, A. H., Schneider, W. L., Hoyt, P. R., Melcher, U. Screening metagenomic data for viruses using the e-probe diagnostic nucleic Acid assay. Phytopathology. 104 (10), 1125-1129 (2014).
- Borah, B. K., et al. Bacilliform DNA-containing plant viruses in the tropics: commonalities within a genetically diverse group. Mol Plant Pathol. 14 (8), 759-771 (2013).
- Bousalem, M., Douzery, E. J., Seal, S. E. Taxonomy, molecular phylogeny and evolution of plant reverse transcribing viruses (family Caulimoviridae) inferred from full-length genome and reverse transcriptase sequences. Arch Virol. 153 (6), 1085-1102 (2008).
- Geering, A. D., et al. Banana contains a diverse array of endogenous badnaviruses. J Gen Virol. 86, Pt 2 511-520 (2005).
- Kunii, M., et al. Reconstruction of putative DNA virus from endogenous rice tungro bacilliform virus-like sequences in the rice genome: implications for integration and evolution. BMC Genomics. 5, 80(2004).
- Laney, A. G., Hassan, M., Tzanetakis, I. E. An integrated badnavirus is prevalent in Figure germplasm. Phytopathology. 102 (12), 1182-1189 (2012).
- Gambley, C. F., Geering, A. D., Steele, V., Thomas, J. E. Identification of viral and non-viral reverse transcribing elements in pineapple (Ananas comosus), including members of two new badnavirus species. Arch Virol. 153 (8), 1599-1604 (2008).
- Gayral, P., et al. A single Banana streak virus integration event in the banana genome as the origin of infectious endogenous pararetrovirus. J Virol. 82 (13), 6697-6710 (2008).
- Lyttle, D. J., Orlovich, D. A., Guy, P. L. Detection and analysis of endogenous badnaviruses in the New Zealand flora. AoB Plants. 2011, 008(2011).
- Le Provost, G., Iskra-Caruana, M. L., Acina, I., Teycheney, P. Y. Improved detection of episomal Banana streak viruses by multiplex immunocapture PCR. J Virol Methods. 137 (1), 7-13 (2006).
- Singh, K., Talla, A., Qiu, W. Small RNA profiling of virus-infected grapevines: evidences for virus infection-associated and variety-specific miRNAs. Funct Integr Genomics. 12 (4), 659-669 (2012).
- Alfson, K. J., Beadles, M. W., Griffiths, A. A new approach to determining whole viral genomic sequences including termini using a single deep sequencing run. J Virol Methods. 208, 1-5 (2014).
- Kreuze, J. F., et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs: a generic method for diagnosis, discovery and sequencing of viruses. Virology. 388 (1), 1-7 (2009).
- Zheng, Y., et al. VirusDetect: An automated pipeline for efficient virus discovery using deep sequencing of small RNAs. Virology. 500, 130-138 (2017).
- James, A. P., Geijskes, R. J., Dale, J. L., Harding, R. M. Molecular characterisation of six badnavirus species associated with leaf streak disease of banana in East Africa. Annals of Applied Biology. 158 (3), 346-353 (2011).
- Baranwal, V. K., Sharma, S. K., Khurana, D., Verma, R. Sequence analysis of shorter than genome length episomal Banana streak OL virus like sequences isolated from banana in India. Virus Genes. 48 (1), 120-127 (2014).
- Sukal, A., Kidanemariam, D., Dale, J., James, A., Harding, R. Characterization of badnaviruses infecting Dioscorea spp. in the Pacific reveals two putative novel species and the first report of dioscorea bacilliform RT virus 2. Virus Res. 238, 29-34 (2017).
- BÖmer, M., Turaki, A. A., Silva, G., Kumar, P. L., Seal, S. E. A sequence-independent strategy for amplification and characterisation of episomal badnavirus sequences reveals three previously uncharacterised yam badnaviruses. Viruses. 8 (7), (2016).
- Momol, M. T., Lockhart, B. E. L., Dankers, H., Adkins, S. Canna yellow mottle virus detected in Canna in Florida. Plant Health Progress. , August 2-4 (2004).
- Zhang, J., et al. Characterization of Canna yellow mottle virus in a new host, Alpinia purpurata, in Hawaii. Phytopathology. 107 (6), 791-799 (2017).
- Wijayasekara, D., et al. Molecular characterization of two badnavirus genomes associated with Canna yellow mottle disease. Virus Res. 243, 19-24 (2018).
- Covey, S. N., Noad, R. J., al-Kaff, N. S., Turner, D. S. Caulimovirus isolation and DNA extraction. Methods Mol Biol. 81, 53-63 (1998).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Molecular cloning: A laboratory manual. 2nd edn. , Cold Spring Harbor Press. (1989).
- Radford, A. D., et al. Application of next-generation sequencing technologies in virology. J Gen Virol. 93, Pt 9 1853-1868 (2012).
- Kanagal-Shamanna, R. Emulsion PCR: Techniques and Applications. Methods Mol Biol. 1392, 33-42 (2016).
- Getts, D. R., et al. Targeted blockade in lethal West Nile virus encephalitis indicates a crucial role for very late antigen (VLA)-4-dependent recruitment of nitric oxide-producing macrophages. J Neuroinflammation. 9, 246(2012).
- van Dijk, E. L., Jaszczyszyn, Y., Thermes, C. Library preparation methods for next-generation sequencing: tone down the bias. Exp Cell Res. 322 (1), 12-20 (2014).
- Gel filtration principles and methods. GE Healthcare. , (2010).
- Goff, S., et al. The iPlant Collaborative: Cyberinfrastructure for Plant Biology. Frontiers in Plant Science. 2, (2011).
- Lin, Z., et al. Next-generation sequencing and bioinformatic approaches to detect and analyze influenza virus in ferrets. J Infect Dev Ctries. 8 (4), 498-509 (2014).
- Artimo, P., et al. ExPASy: SIB bioinformatics resource portal. Nucleic Acids Res. 40, Web Server issue 597-603 (2012).
- Edgar, R. C. MUSCLE: a multiple sequence alignment method with reduced time and space complexity. BMC Bioinformatics. 5, 113(2004).
- Hung, J. H., Weng, Z. Sequence Alignment and Homology Search with BLAST and ClustalW. Cold Spring Harb Protoc. 2016 (11), (2016).
- Sohpal, V. K., Dey, A., Singh, A. MEGA biocentric software for sequence and phylogenetic analysis: a review. Int J Bioinform Res Appl. 6 (3), 230-240 (2010).
- Untergasser, A., et al. Primer3--new capabilities and interfaces. Nucleic Acids Res. 40 (15), 115(2012).
- Dhaliwa, A. DNA extraction and purification. Mater Methods. 3, 191(2013).
- Moeller, J. R., Moehn, N. R., Waller, D. M., Givnish, T. J. Paramagnetic cellulose DNA isolation improves DNA yield and quality among diverse plant taxa. Appl. Plant Sci. 2 (10), (2014).
- Moeller, J. R., et al. Paramagnetic cellulose DNA isolation improves DNA yield and quality among diverse plant taxa. Appl. Plant Sci. 2 (10), (2014).
- Grooms, K. Review: Improved DNA Yield and Quality from Diverse Plant Taxa. , (2015).
- Nishimori, A., et al. In vitro and in vivo antivirus activity of an anti-programmed death-ligand 1 (PD-L1) rat-bovine chimeric antibody against bovine leukemia virus infection. PLoS One. 12 (4), 0174916(2017).
- Rojas, M. R., Gilbertson, R. L. Plant Virus Evolution. Roossinck, M. J. 1, Springer-Verlag. 27-51 (2008).
- Roossinck, M. J. The big unknown: plant virus biodiversity. Curr Opin Virol. 1 (1), 63-67 (2011).
- Roossinck, M. J., Martin, D. P., Roumagnac, P. Plant Virus Metagenomics: Advances in Virus Discovery. Phytopathology. 105 (6), 716-727 (2015).
- Momol, M. T., Lockhart, B. E. L., Dankers, H., Adkins, S. Plant Health Progress. , Online (2004).
- Eni, A., Hughes, J. D., Asiedu, R., Rey, M. Sequence diversity among badnavirus isolates infecting yam (Dioscorea spp.). Archives of Virology. 153 (12), Ghana, Togo, Benin and Nigeria. 2263-2272 (2008).
- Harper, G., et al. The diversity of Banana streak virus isolates in Uganda. Arch Virol. 150 (12), 2407-2420 (2005).
- Muller, E., Sackey, S. Molecular variability analysis of five new complete cacao swollen shoot virus genomic sequences. Arch Virol. 150 (1), 53-66 (2005).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved