Method Article
דיסקציה של משפר תפקוד באמצעות הפרעות משפר מבוססי CRISPR מולטיפלקס בשורות תא
In This Article
Summary
פרוטוקול זה מתאר את השלבים הדרושים כדי לתכנן ולבצע מיקוד מרובבת של משפרי עם החלבון פיוז'ן deactivating SID4X-dCas9-קראב, המכונה גם משפר הפרעה (משפר-i). פרוטוקול זה מאפשר זיהוי משפרי המווסתים ביטוי גנים ואסטמה ומקילה על ניתוח היחסים בין משפרי ויסות גנים מטרה משותפת.
Abstract
משפרי מרובים לעיתים קרובות לווסת גן מסוים, אך עבור רוב הגנים, עדיין לא ברור איזה משפרי נחוצים עבור ביטוי גנים, ולשלב כמה אלה משפרי לייצר היענות תעתיק. כמו מיליוני משפרי זוהו, תפוקה גבוהה כלים נדרשים כדי לקבוע משפר תפקוד בקנה מידה הגנום כולו. שיטות לימוד משפר תפקוד כוללים בדיקה גנטית מחיקות בעזרת נוקלאז שולטים Cas9, אך קשה ללמוד את השפעות קומבינטורית משפרי מרובים באמצעות טכניקה זו, כמו מספר שורות רצופות תא המשובטים חייב להיות שנוצר. כאן, אנו מציגים שיפור-i, CRISPR מבוססת על התערבות שיטה המאפשרת לחקירה פונקציונלי של משפרי מרובים בו זמנית ב שלהם לוקוסים אנדוגני. משפר-אני עושה שימוש בשני תחומים דכאניים דבוקה נוקלאז לקויה Cas9, סיד, קראב, כדי להשיג משפר הביטול באמצעות היסטון deacetylation-יישוב לוקוסים. פרוטוקול זה מנצל תקנים ארעית של מדריך RNAs כדי לאפשר איון ארעית של אזורים יישוב, והוא יעיל במיוחד חוסמת inducible תעתיק תגובות לגירויים בהגדרות תרביות רקמה. משפר-i הוא מאוד ספציפי הן מיקוד גנומית שלה והן את השפעותיו על ביטוי גנים גלובלית. התוצאות המתקבלות מפרוטוקול זה לעזור להבין אם שיפור תורמת ביטוי גנים, היקף התרומה של ההשפעה התרומה על ידי אחרים משפרי הסמוך.
Introduction
בקנה מידה גדול רצף פרויקטים כגון קדד1, מפת הדרכים Epigenomics2שהפאנטום3 זיהו מיליוני משפרי בשם בתוך הגנום האנושי על פני מאות סוגי תאים. ההערכה היא כי כל יזם משייכת ממוצע של משפרי 4.9, משפר כל קשר בממוצע 2.4 גנים3, רומז ביטוי גנים הוא לעתים קרובות התוצאה של השילוב של מספר האינטראקציות רגולציה מבוזרת. אתגר משמעותי הנותרים הוא להגדיר לא רק כמה בודדים משפרי לתרום ביטוי גנים, אבל איך שהם משתלבים להשפיע על הביטוי. גישות גנטיות משמשות כדי לזהות קשרים בין משפרי אורגניזמים דגם דרוזופילה4 עכברים5. עם זאת, ניסויים אלה הם ועתירת בתפוקה נמוכה לחקר משפרי מרובים-גנים מרובים.
גישה אחת ללמוד משפר תפקוד בקנה מידה גדול כולל מבחני בנפט במקביל הכתב. מבחני אלה מאפשרים ההקרנה סימולטני של אלפי רצפי DNA על יכולתם לנהוג את הביטוי של גנים כתב6. בזמן מבחני אלה הראו כי רצף ה-DNA לבדו יכול להיות מספיק כדי להעביר מידע גנטי תקנה7, הם באים עם ההליכים של המתבצעת מחוץ להקשר כרומטין מקורי ועם מקדם heterologous. בנוסף, בגודל של רצף ה-DNA עוברים בדיקה ב בנפט במקביל הכתב מבחני הוא בדרך כלל פחות מ-200 basepairs, אשר עשויים לכלול הרלוונטיים רצף שמסביב. חשוב, כמו מבחני כתב למדוד רק את הפעילות של רצף אחד בכל פעם, הם לא לוקחים בחשבון את קשרי הגומלין המורכבים שיכול להתקיים בין מוצרי טיפוח טבעיים. לכן, בעוד בנפט במקביל הכתב מבחני יכול להיות אינפורמטיבי אודות הפעילות מהותי של רצף ה-DNA, הם לא בהכרח להודיע לנו על הפונקציה של רצף ה-DNA בהקשר של הגנום.
לאחרונה CRISPR/Cas9 פיתח כלים8 יש הקלה המחקר של הכונה כפי שהם מאפשרים המחיקה של משפרי-מיקומה אנדוגני. עם זאת, מחיקת משפרי מרובים בו-זמנית עלולה להוביל אי יציבות גנומית, וזה זמן רב כדי ליצור שיפור רצופים מחיקות בשורה תא בודד. בנוסף, נוצר רצף גנומית חדש באתר של מחיקה בעקבות תיקון, רצף זה עשוי לקבל פונקציית רגולציה. גירסה חלופית Cas9 פותחה במיוחד עבור להתכוונן ביטוי גנים, בהסתמך על fusions של הפעלת9,10 או הדחקת תחומים12 11,לטופס נוקלאז לקויה של Cas9 (dCas9). חלבונים אלה פיוז'ן הינם אידיאליים עבור הלומדים לוקוסים מרובים בו זמנית כמו שהם לא פיזית לשנות את רצף ה-DNA, במקום לווסת אפיגנטיקה על מנת לחקור את אזור רגולטוריות. פיוז'ן דכאניים הנפוצה ביותר הוא קראב, אשר מגייס המתחם מדכא. שבולם שיתוף KAP1, קידום בתצהיר של דיכוי-הקשורים היסטון H3 ליזין 9 trimethylation (H3K9me3)13. dCas9-קראב, הידוע גם CRISPR הפרעה14, שימש כדי היעד ומסך משפרי בודדים על תרומתם לחיזוק ביטוי גנים15,16; עם זאת, זה לא הותאם מבחינת פילוח אזורים מרובים בו-זמנית. גרסה אחת של הפרעה CRISPR מולטיפלקס עבור מוצרי טיפוח טבעיים, פסיפס-seq17, משתמשת תא בודד RNA-seq כמו הבדיקה, אבל טכנולוגיה זו הוא יקר ומתאים רק לצורך המחקר של גנים ביטוי מאוד בשל הרגישות הנמוכה של תא בודד רנ א-תת סעיף.
אנחנו ביקשו לפתח שיטה המבוססת על הפרעות CRISPR ניקוד משפר קומבינטורית פונקציה בתוך ההקשר של תגובה תעתיק אסטרוגן. כמחצית גנים אסטרוגן מגיב מכילים 2 או יותר משפרי מחויב על ידי אלפא קולטן אסטרוגן (ER) בקרבת מקום18, רומז כי משפרי מרובים עשויים להיות השתתפות התגובה אסטרוגן, הבנת שהלוגיקה של הרגולציה ידרוש פילוח משפרי מרובים בו-זמנית. כפי מחקרים ראשוניים באמצעות התערבות CRISPR ב היזמים שהציע כי לא כל היזמים הם מגיבים באותה מידה דיכוי בתיווך קראב19, אנחנו הסיק כי התוספת של תחום דכאניים ברורים כדי dCas9 עשוי להקל על שחרור משרות של משפרי מגוונות. בחרנו את Sin3a אינטראקציה תחום של Mad1 (SID)20 כמו זה מוביל הגיוס של היסטון deacetylases21, אשר להסיר קבוצות אצטיל-שינויים היסטוניים הקשורים עם פעילות גנים ברמת השעתוק. חשוב, התחום סיד היה יעיל להפחתת ביטוי גנים כאשר דבוקה dCas922 וסיפורי23, Sin3a הוכח להיות גורם משותף דכאניים חזק מגוון משפר רצף בהקשרים24. אנחנו נעשה שימוש SID4x-dCas9-קראב (משפר-i) כדי להתמקד 10 משפרי שונה על-ידי המיון, לזהות אתרי קישור ER (ERBS) שהם נחוצים על התגובה תעתיק אסטרוגן בגיל 4 גנים18. אנחנו גם לפלח את השילובים של משפרי לזהות את האתרים לשתף פעולה בייצור של התגובה תעתיק אסטרוגן. מצאנו כי עד 50 אתרי העלול ניתן לפלח בו זמנית עם שינויים בביטוי הגנים לזיהוי. באמצעות שבב-seq ו RNA-seq, הפגנו כי Enhancer-i הוא טכניקה ספציפי מאוד ללמוד משפרי מרובים בו-זמנית.
ב פרוטוקול זה, אנו מתארים את השלבים הכרוכים בביצוע Enhancer-i, טכניקה גמישה המאפשרת לימוד משפרי מרובים בו-זמנית באווירה תרביות רקמה תפקודית. משפר-i הוא מאוד מתואם עם מחיקת גנטי אבל מספק לנטרול ארעי התלויים היסטון deacetylases (HDACs). מספקים את המדריך RNAs באמצעות תקנים ארעי ולא יציב אינטגרציה באמצעות וקטורים ויראלי, פרוטוקול זה מונע התצהיר והפצת פוטנציאלי של H3K9me3. עיצוב מדריך RNA פרטים זה פרוטוקול שכפול באמצעות הרכבה גיבסון, תרביות תאים של מדריך RNAs באמצעות lipofection, הניתוח של ביטוי גנים וכתוצאה מכך משתנה על-ידי qPCR. נשלב גם את השיטות להערכת יחודיות של שיפור-i מיקוד ברמה של הגנום של transcriptome. בעוד טכניקה זו פותחה כדי ללמוד הכונה על ידי מיון בחבלי משפרי שורות תאים סרטניים אנושיים, זה החלים על ניתוח כל שיפור יונקים.
Protocol
1. דור של תא קווים Stably לבטא SID4X-dCas9-קראב
הערה: התנאים תרביות תאים וריכוזי סמים המובאת כאן מוטבו עבור תאים אישיקאווה, קו התא סרטן רירית הרחם, גדל בתקשורת RPMI 1640 בתוספת 10% FBS ו 1% פניצילין/סטרפטומיצין (RPMI מלאה). שורות תאים אחרים עשויים לדרוש תנאים שונים תרביות תאים וריכוזי סמים. משתמשים יכולים לבצע גם תקנים ארעי ניסויים בתאי פראי-סוג, במקום לייצר קו תא יציבה, עם פלסמיד לבטא SID4X-dCas9-קראב יחד עם מדריך RNA לבטא פלסמידים; עם זאת, תוצאות transfections ארעי ייתכן שתתקשה להתרבות כמו SID4X-dCas9-קראב רמות עשויה להשתנות לפי תקנים.
- צלחת אישיקאווה תאים בבארות לפחות 2 של צלחת 6-ובכן-זרימה 30-50% (כ 300,000 אישיקאווה תאים) ב- 3 מ"ל של RPMI מלאה.
- האחות המדיה מן התאים. לשטוף את התאים פעם אחת עם 1 x PBS (pH 7.4). האחות של PBS ולהוסיף טריפסין (4 מ ל צלחת 10 ס מ או 5 מ עבור בקבוקון T-75).
- דגירה התאים ~ 5 דקות ב 37 מעלות צלזיוס, בדיקת כל 2 דקות עבור תאים מנותקת ורעדתי בעדינות את הכלי.
- ברגע תאים יש תלושים, פיפטה trypsinized תאים לכל אורך כמה שעות, פיפטה בעדינות במורד הצד של כלי השיט לשחרר תאים המצורפת.
- להעביר את התאים צינור חרוטי 15 מ"ל, ספין התאים למטה במשך 5 דקות ב 250 x g.
- האחות של טריפסין, resuspend את התאים ב- 5-10 מ"ל של מדיה. השתמש פיפטה של P1000 מביצועם תא גושים במידת הצורך.
- לספור את התאים ולקבוע את אמצעי האחסון הדרושים כדי צלחת ~ 300,000 תאים לכל באר הנפח הכולל של 3 מ. להוסיף את התאים 2 בארות נפרדים של צלחת 6-. טוב. למלא כל אחד טוב עד 3 מ"ל עם RPMI מלאה.
- לנער בעדינות את הצלחת כל 5 דקות הראשון באינטרקום לאחר ציפוי כדי להבטיח כי התאים מופצים בצורה שווה על הצלחת. השתמש במיקרוסקופ כדי להבטיח כי התאים התפזרו מהאמצע של הבאר.
- בתוך 24 שעות של ציפוי, לבצע את transfections הבאות באמצעות ריאגנט של תרביות תאים המתאימים עבור שורת תאים של עניין. עבור תאים אישיקאווה, השתמש בהליך כמתואר להלן.
הערה: פרוטוקול זה מבוסס על ההנחה השימוש ריאגנטים cationic ליפוזום מבוססות תקנים. אלקטרופורציה מספק שיטה חלופית עבור סוגי תאים אשר רגישים מאוד ריאגנטים אלה או זה להפגין יעילות נמוכה תרביות תאים עם lipofection. חייב להיות מוטבת תנאים תקנים עבור שורת תאים עניין לפני שתנסה ניסויים Enhancer-i.- ב- mL 1.7 גלאים שפופרת, למהול 2.5 μg של SID4X-dCas9-קראב פלסמיד ו-800 ng של פלסמיד ביטוי חלבון פלואורסצנטי לתוך המדיה ללא סרום כך שאמצעי האחסון האחרון בצינור 155 µL והוא הריכוז הסופי של פלסמיד µg 0.020/µL.
- צינור אחר, µg 3.3 שתדללו של פלסמיד שאינו מכיל קלטת התנגדות neomycin, כגון pCMV-GFP, בתקשורת ללא סרום כך שאמצעי האחסון האחרון בצינור 155 µL הריכוז הסופי של פלסמיד הוא µg 0.020/µL.
- מערבולת כל צינור בקצרה, ספין למטה באמצעות של microfuge.
- להוסיף µL 9.9 מהתרכובת תרביות תאים (טבלה של חומרים) כל שפופרת. מערבבים על-ידי vortexing בקצרה על מהירות נמוכה. ספין הצינורות למטה עם microfuge.
- דגירה הצינורות בטמפרטורת החדר במשך לפחות 5 דקות, אך לא יותר מ 20 דקות.
- ב אבטחה הקבינט, להוסיף 150 µL של המיקס דנ א מוכן: ריאגנט dropwise טוב על הצלחת 6-. טוב. חזור על הצינור השני של DNA: ריאגנט מוכן לערבב. לערבב את הצלחות על ידי מתערבל בעדינות את הצלחת ולחזור החממה.
- יום 2 פוסט תרביות תאים, לשנות את המדיה, להשלים עם G418 כדי ריכוז סופי של 600 ng/μL. ריכוז זה ייתכן שתצטרך להיות ממוטבים עבור סוג התא.
- שנה מלאה RPMI מדיה, להשלים עם G418 בכל יום אחר במשך 2-4 שבועות עד התאים בקרה transfected מתים ולהיות הבארות המכיל SID4X-dCas9-קראב confluent. הסכום המדויק הנדרש בתאים לשחזר תלויות זמן הכפלה של התאים.
- כאשר התאים הופכים confluent, המעבר אל קיבול T-25 או T-75, השלימו RPMI עם מינון נמוך של G418 (300 ng/μL עבור תאים אישיקאווה). במהלך המסע, להפוך aliquots 2 ~ 100,000 תאים כל (בערך 1/10בתאנון של צלחת 6-ובכן) לתוך 2 צינורות ריפרבאן מ"ל 1.7 נפרד לבידוד RNA ו- DNA, בהתאמה. לסובב את הצינורות האלה למטה (5 דקות, 250 גרם x), להסיר טריפסין על-ידי pipetting, להקפיא את הצינורות ב-20 ° C לשימוש עתידי.
- לבודד דנ א גנומי באמצעות ערכות זמינים מסחרית ולבצע PCR בעזרת "pAC95_PCR" או "SID4X_PCR" תחל (טבלה 1) כדי לוודא המצאות חלבון כימרי בתוך הקו הסלולרי. השתמש דנ א גנומי שחולצו מן הקו הורים כפקד שלילית, SID4x-dCas9-קראב פלסמיד DNA כפקד חיובי. השתמש תערובת בסיס אמינות גבוהה פולימראז עם 50-100 ננוגרם של הדנ א, רכיבה על אופניים התנאים הבאים: 98 ° C ל 30 s, 25 מחזורי של (98 ° C עבור 10 s, 58 ° C ל 30 s, 72 מעלות צלזיוס למשך 2 דקות), 72 מעלות צלזיוס למשך 5 דקות , תחזיק על 4 מעלות צלזיוס.
- כדי לוודא את הביטוי של החלבון פיוז'ן ברמת ה-RNA, לבצע qPCR עם ה-RNA שחולצו מן הקו הסלולרי באמצעות ערכות זמינים מסחרית. השתמש תחל את "dCas9_qPCR" (טבלה 1), פרוטוקול qPCR בשלב אחד מספק בשלב 6.3 של פרוטוקול זה.
- כדי לוודא הביטוי ברמת חלבון של היתוך גרעיני, לבצע מערבון ביחסינו lysates מתוך שורת תאים. השתמש נגד דגל או אנטי-HA נוגדנים כדי לזהות את החלבון פיוז'ן.
2. מדריך RNA עיצוב
הערה: פרוטוקול זה מיועד לשימוש עם הרנ א מדריך U6 שיבוט וקטורית שנוצרה על ידי המעבדה הכנסייה וזמין על Addgene (Addgene 41824). כדי ליצור גרסה של זה וקטורית שמכילה התנגדות puromycin המותרת באותה אסטרטגיה שיבוט כמו 41824, עברנו מרובים באתר שיבוט זה לתוך הווקטור pGL3-U6-sgRNA-PGK-puromycin (Addgene 51133). Addgene 41824 או את הגרסה שלנו עם puromycin (Addgene 106404) תואמים האסטרטגיה שיבוט המפורטות להלן.
- להשיג basepairs 600-900 של רצף ה-DNA לכל אזור הרגולציה של עניין. השתמש אתרי קישור פקטור שעתוק ו/או נגישות כרומטין להנחיה היכן להגדיר את האזור של הריבית (איור 2 א).
הערה: בזמן הדוגמה באיור 2 א כולל משפרי ויוצאת, זה גם אפשרי למקד את הרגולציה אלמנטים ממוקם בתוך אינטרונים. - מקם כל רצפי שהושג בקובץ טקסט יחיד באמצעות התבנית FASTA.
- לזהות אחד לפחות אזור בקרה שלילית זה לא צפוי להשתנות במהלך ניסיוני תנאים, כגון אדם המקדם את גן כי אינו מתבטא בשורה תא של ריבית. להשיג את רצף ה-DNA עבור אזור זה ולהוסיפו לקובץ טקסט בתבנית FASTA.
הערה: אנו משתמשים מדריך RNAs מיקוד יזם IL1RN 25 כפקד שלילי עבור כל האזורים שאנחנו היעד. המשתמשים יכולים גם לבחור רצף intergenic ליד האזור עניין שאינו מכיל אתרי קישור פקטור שעתוק כפקד שלילי. עם זאת, אם הם מהווים מטרה לוקוסים מרובים בו זמנית, אזור שליטה שלילי יחיד מפשט עיצוב ניסיוני, פרשנות של תוצאות. אם משפר יישוב intronic, ייתכן מועיל למטרה אזור intronic-מיקומה אותו שאינו מכיל רכיב תקינה בשם בתור פקד שליליים נוספים, כמו הפיוז'ן dCas9 עלולים להפריע שעתוק. - לזהות אזורים בקרה חיובית, כגון היזמים כי הם המטרות בשם של האזורים הרגולציה של עניין או היזמים של גנים אשר הם מאוד עיבד בשורת תאים של ריבית. להשיג את רצף ה-DNA עבור אזורים אלה והוסף אותו בקובץ טקסט בתבנית FASTA.
- השתמש בתוכנית כגון e-פריך26 (http://www.e-crisp.org/E-CRISP/) רצפי DNA שנוצר כדי למצוא את המדריך RNAs עם נמוך את המטרות (אידיאלי 0-3). מדריך RNAs מורכב 20 נוקלאוטידים במעלה הזרם של protospacer הסמוך מוטיב (פאם), אשר לוקח את הטופס "הגולה" עבור dCas9 מ ס הפישחה ינפל תומימת.
- באתר e-פריך, בחר את האורגניזם עניין שימוש בתפריט הנפתח. מכלול הגנום מופיע בצד ימין של השם מינים.
- בחר בלחצן האפשרות קלט הוא FASTA רצף . העתק את רצפי FASTA מלמעלה, והדבק אותם בתיבת הדו-שיח. ודא כותרת FASTA כלולות עבור כל רצף.
הערה: עד 50 רצפים יכול להיות שאילתה בעת ובעונה אחת. - בחר בלחצן האפשרות בינונית את עיצוב יחיד בתפריט הנפתח.
- לחץ על הלחצן התחל חיפוש sgRNA. כרטיסיה חדשה של הדפדפן יפתח, יוצגו תוצאות. הורד את רצפי המועמד על-ידי לחיצה על לחצן הורד דוח טבלאי של Excel מעוצב על כל רצפי השאילתה יחד.
- פתח את דוח טבלאי באמצעות Excel, או בתוכנית לעריכת טקסט.
- השתמש את הדפדפן הגנום UCSC בלאט המועמד gRNA באורך מלא רצפים (23 basepairs) כדי הגנום.
- בדפדפן, נווט אל אתר דפדפן הגנום UCSC (http://genome.ucsc.edu). תחת המקטע הכלים שלנו, לאתר את המילה BLAT ולחץ על זה. הכלי חיפוש בלאט יפתח.
- השתמש בתפריטים הנפתחים ממוקם מתחת לטקסט בלאט חיפוש הגנום כדי לבחור את מכלול אורגניזם ו גנום של עניין.
- העתק את רצפי RNA מדריך מהדוח טבלאיים שנוצרו על ידי e-פריך, והדבק אותם בתיבת הדו-שיח. ודא כי כל רצף יש כותרת עליונה FASTA ייחודי, ואז לחץ על Submit בתחתית תיבת הדו-שיח.
הערה: עד 25 רצפים יכולים להיבדק בעת ובעונה אחת. בדף תוצאות החיפוש בלאט , יופיע במערכים של כל מדריך רצף ה-RNA, עם כל שורה מייצגת ישור. באופן אידיאלי, צריך להיות יישור אחד עבור כל מדריך RNA, המציין את הייחודיות של המדריך RNA. - למנוע את קווי יישור שמיישרים במספר מיקומים הגנום במידת האפשר.
- לבחון מדריך RNA לוקליזציה והפצה בתוך האזור בעל עניין, לחץ על הקישור הדפדפן תחת המקטע פעולות לאחד RNAs מדריך המאוחזרים. הדפדפן הגנום יופיע ואת ימורכז על המדריך שנבחר RNA. השתמש בלחצנים התרחקות בחלק העליון של הדף כדי להמחיש את ההפצה של אחרים מדריך RNAs המזוהה על-ידי e-פריך בתוך האזור של ריבית.
- בחר 4 רצוי שאינם חופפים מדריך RNA רצפים המופצים ברחבי האזור בעל עניין (איור 2B). אם האזור של ריבית עולה 600 bp, שקול להוסיף 1-2 מדריכים נוספים. הימנע מדריך RNAs עם homopolymeric נמתח, תוכן קיצוני GC, כמו תכונות אלה יכולים לעכב את הרנ א מדריך שכפול התהליך ולהפחית RNA מדריך מיקוד יעילות.
- ברגע המדריכים נבחרו, ליצור קובץ המכיל את המדריך המלא רצף ה-RNA (23 נוקלאוטידים) עבור כל מדריך הרצוי ולאחר מכן הסר את נוקלאוטיד 5', כמו גם את פאם (הגולה) מקצה 3'. שלב זה מאפשר הזמנת oligo.
- להוסיף את הרצף הבא לסוף 5' של הרצף oligonucleotide: GTGGAAAGGACGAAACACCG.
- להוסיף את הרצף הבא לסוף 3' של הרצף oligonucleotide: GTTTTAGAGCTAGAAATAGC.
הערה: הרצף הסופי צריך להיות נוקלאוטידים 59 זמן תראה ככה: GTGGAAAGGACGAAACACCG-יעד (19 nt)-GTTTTAGAGCTAGAAATAGC. - להבטיח שלכל רכיב רגולטורי כדי להיות ממוקד עם משפר-i יש לפחות 4 oligonucleotides ייחודי המיועד לכך. להזמין את רצפי אלה יחד עם תחל "U6_internal" המופיעים בטבלה 1.
3. מדריך RNA שיבוט
הערה: מדריך RNA שכפול באמצעות הרכבה גיבסון הוכיחה להיות יעילים ביותר בידיים שלנו, מניב מאות מושבות לכל צלחת, עם נוכח השליטה היחידה וקטור מעטים אם בכלל מושבות. יעילות כזה חיוני לשמירה על מורכבות במהלך שכפול במאגר. יתרון נוסף של גיבסון הרכבה שיבוט הוא למשתמשים אין מה לדאוג הנוכחות של אנזים ההגבלה לחתוך האתר במדריך RNA הם מנסים להכניס הווקטור שיבוט U6. למרות זאת, פרוטוקול זה ניתן להתאים עבור מסורתי אנזים הגבלה המבוסס על שיבוט במידת הצורך.
- לשקם מדריך RNA oligos-ריכוז סופי של 100 μM במים הנדסה גנטית (RNase-חופשית, נטולת DNase). צריך להיות לפחות 4 מדריך נפרד RNA oligos עבור כל אזור בעל עניין.
- עבור כל אזור הרגולציה של עניין, ליצור מאגר של כל oligos המתאים לאזור של ריבית. בשפופרת גלאים, לשלב 5 μL של כל oligo RNA מדריך משוקם נפרדים עבור כל אזור. לערבב את הבריכה היטב על ידי vortexing, ולאחר מכן להסיר את 1 μL, לדלל את 1:200 aliquot במים הנדסה גנטית.
הערה: אם רצונך בכך, בשלוליות פילוח אזורי רגולטוריות בודדים ניתן עוד יותר לשלב כדי ליצור בריכה מורכבים מיקוד אזורים מרובים. עד 50 אזורים רגולטוריות עשויות להיות ממוקדות בו-זמנית בבריכה יחיד (איור 3C). - לבצע PCR קצר עם תחל U6 לצרף הומולוגיה אזורים oligos לפני ההרכבה גיבסון. בסיסים כ-40 יתווספו כל oligo, מניב מוצר bp ~ 100 המכיל הומולוגיה מספיקות כדי הווקטור U6 בשני הקצוות.
- מדריך כל בריכה RNA, להגדיר μL 20 PCR עם תערובת בסיס אמינות גבוהה פולימראז, הרכיבים הבאים: 1 μL של הבריכה oligo מדולל מהשלב 3.2, μL 1 של פריימר לפנים U6 (10 μM), μL 1 של U6 הפוך פריימר (10 μM) , מים μL עד 20.
- דגירה ב הצנטרפוגה תרמי עם התנאים הבאים: 98 ° C ל 30 s, 10 מחזורים של (98 ° C עבור 10 s, 55 ° C ל 30 s, 72 מעלות צלזיוס למשך 2 דקות), 72 מעלות צלזיוס במשך 5 דקות, והחזק ב 4 º C.
- הפעל μL 5 התגובה על 1-2% agarose ג'ל עם סולם נמוך משקל מולקולרי. המוצר הסופי צריך להיות ~ 100 basepairs (איור 2C).
- לנקות את התגובה הרחבה עם ערכת טיהור DNA מבוססי עמודה, elute, μL 20 • תנאי מאגר המסופק בערכה.
הערה: כמו המוצר הוא קצר, הימנע משימוש מבוסס-חרוז לנקות קופצים, אשר נועדו לשלול שברים קטנים במרחק של פחות מ 100 bp. - לכמת את ה-DNA מטוהרים באמצעות fluorometer או ספקטרופוטומטרים (מצופה התשואה הוא 10-20 ng/μL). מדריך RNA מוסיף שניתן לאחסן ב-20 ° C, או יכול לשמש מיד בהרכבה גיבסון עם וקטור U6 ליניארית.
- כדי להכין את המטופל U6 וקטור שיבוט להרכבה גיבסון, להגדיר את תקציר אנזים הגבלה. אם תגובות רבות של הרכבה גיבסון לביצוע, להגדיר מעכל מרובים כדי להבטיח תשואה מספקת של וקטור לחתוך.
- השתמש 20 יחידות של האנזים AflII μg 1 של פלסמיד בתגובה 20 μL עם המאגר אנזים הגבלה המתאים. דגירה ב 37 מעלות צלזיוס במשך 1-2 h.
- לנקות את התקציר עם חרוזים או ערכת מבוססי עמודה עבור טיהור DNA, elute ב μL 20 • תנאי המאגר. לכמת את ה-DNA מטוהרים באמצעות fluorometer או ספקטרופוטומטרים. דוגמאות שיכול להיות קפוא ב-20 ° C לשימוש מאוחר יותר.
- לבצע הרכבה גיבסון על וקטור מוכן והכנס.
- להגדיר את גיבסון הרכבה תגובות על קרח. שימוש 50 ng של וקטור ו- 7 ננוגרם של הקדמי בתגובה 20 μL. לדלל 1:10 מוסיף במים הנדסה גנטית כדי להקל על pipetting. להגדיר את וקטור היחידה גיבסון הרכבה ריאקציה, באמצעות 50 ng של וקטור, מחליף את תותב עם מים.
- דגירה גיבסון מכלול התגובות למשך 15 דקות ב 50 מעלות צלזיוס, ואחריו אחיזה ב 4 º C.
- להעביר את המוצרים מורכבים לקרח. לדלל את המוצרים מורכבים 1:4 במים הנדסה גנטית על קרח. לדוגמה, הוסף 5 μL של גיבסון הרכבה מוצר 15 μL של מים הנדסה גנטית.
- להפוך את המוצרים מכלול גיבסון מדולל.
- הפשרת תאים המוסמכת יעילות גבוהה על קרח ולהפוך aliquots 25 μL עבור כל שינוי. אם בריכה מורכבים מיקוד באתרים מרובים רצוי, להפשיר מספיק תאים לתוך צינורות שונים כדי לבצע העתקות עצמאיות מרובות באותה הבריכה gRNA מורכבים.
- עבור כל מוצר מדולל, להוסיף 1 μL של זה דילול mL 1.7 גלאים שפופרת המכילה 25 μL של תאים המוסמכת. מערבבים על ידי מצליף בקצרה את הצינור. דגירה הצינורות על קרח למשך 30 דקות.
- חום לזעזע את התאים עבור 30 s-42 ° C, ואז להעביר מיד קרח למשך 2 דקות.
- הוספת μL 300 SOC מדיה (2% טריפטון, 0.5% תמצית שמרים, 10 מ מ NaCl, 2.5 מ מ. אשלגן כלורי, 10 מ מ MgCl2, 10 מ מ MgSO4ו- 20 מ מ גלוקוז) ולתת את התאים לשחזר עבור h 1 ב 37 ° C עם טלטול (300 סל ד). במהלך תקופה זו, חמים צלחות אגר עם אמפיצילין/carbenicillin עד 37 מעלות צלזיוס בתוך אינקובטור. השתמש לוח אחד עבור כל שינוי.
- צלחת μL 50 תאים ולמקם את הצלחות חממה 37 ° C בלילה. הבריכות מיקוד אתרים בודדים, במקום ישירות ב- 3-5 מ ל מרק ליברות (טבלה של חומרים) המכיל אמפיצילין/carbenicillin (1 מ"ג/מ"ל), דגירה בין לילה ברעידות-250 סל"ד ב 37 ° C עבור minipreps.
- לקצור את התאים, לבודד דנ א.
- עבור ספריות RNA גדול מדריך מיקוד באתרים מרובים, להשתמש במגרדת צלחת של איסוף כל המושבות שהלוח בודדים לתוך אחד maxiprep (150 מ ל נוזל תרבות). זה ניתן להקל על ידי שפיכת ~ 5 מ של ליברות עם האנטיביוטיקה המתאימה לתוך צינור בז 50 מ ל ולשרוט את המושבות לתוך הצינור. עבור ספריות מיקוד אתרים בודדים, לגרד את הצלחות לתוך miniprep (3-5 מ ל נוזל תרבות).
- דגירה תרבויות אלה עם האנטיביוטיקה המתאימה עבור h 3-5 ב 37 מעלות צלזיוס ברעידות-250 סל"ד.
- לבצע הפקת דנ א באמצעות ערכה שתוצאתו נטולת אנדוטוקסין preps.
- לכמת הדנ א באמצעות fluorometer או ספקטרופוטומטרים. פלסמידים יכולים להיות בשימוש מיד תרביות תאים או המאוחסנים ב-20 ° C לשימוש עתידי.
- לאשר הנוכחות של מדריך רצף ה-RNA בתוך הווקטור U6 בריכות קטנות מיקוד אתרי יחיד, להשתמש סנגר רצף-miniprep מוכן עם פריימר "U6_PCR_R" מפורטות בטבלה 1. בשל איגום של מדריך RNAs, הרצף היעד 19 basepair gRNA תניב בסיסים מעורבת, אבל המקדם U6 ואת מדריך RNA לגרדום המקיפים את הרצף הזה צריך להיות שלם.
4. תקנים של שיפור-i
הערה: עבור המצור מוצלחת מתגובה אסטרוגן באמצעות שיפור-i בתאים אישיקאווה, זה הכרחי כדי לשלול את התאים של אסטרוגן במשך 5-7 ימים לפני תרביות תאים על ידי שמירה על אותם פנול אדום חינם RPMI עם 10% פחם-הפשיטו FBS ו- 1% פניצילין/סטרפטומיצין. התאים צריכים להיות מחונן בתקשורת זו במהלך ואחרי תרביות תאים אם מנסה לחסום מענה אסטרוגן אנו ממליצים על השימוש של טריפסין חינם פנול אדום עבור המעבר של תאים מלאה פנול אדום חינם RPMI.
- יום לפני תרביות תאים, צלחת התאים (פראי-סוג או לבטא stably SID4X-dCas9-קראב) לתוך צלחת 24-ובכן-30-50% confluency (~ 60,000 תאים לכל טוב עבור תאים אישיקאווה). צלחת מספיק תאים כך transfections ניתן לבצע שכפול, והם כוללים בארות כדי להיות transfected עם שליטה מדריך RNAs. ודא כי התאים הם מופץ באופן שווה על פני הבאר ע י ניעור בעדינות את הצלחת לאחר יופיצ תא כמו שלב 1.1.7.
הערה: פרוטוקול זה מבוסס על ההנחה השימוש ריאגנטים cationic ליפוזום מבוססות תקנים. אלקטרופורציה מספק שיטה חלופית עבור סוגי תאים הרגישים מאוד ריאגנטים אלה. חייב להיות מוטבת תנאים תקנים עבור שורת תאים עניין לפני שתנסה ניסויים Enhancer-i. - למחרת, להכין את transfections לפי הוראות של תרביות תאים הכימית של בחירה. עבור תאים אישיקאווה, שימוש 550 ng של פלסמיד הכולל עבור כל טוב של צלחת 24-. טוב. לדלל את פלסמידים כדי ריכוז סופי של μg/μL 0.020 בתקשורת ללא סרום (1.1 μg של ה-DNA ב- 52 μL של הנפח הכולל למעיינות 2 transfecting). השתמש μL 3 של ריאגנט תרביות תאים עבור כל μg 1 של ה-DNA, מערבולת דגירה כפי שמתואר בשלב 1.2. להוסיף 25 μL של התערובת הסופית מכל קידוח.
הערה: כדי שילובים היעד של אתרים, השתמש באותו המשקל של פלסמיד עבור כל אתר בודדים ולאחר מכן למלא את המשקל שאריות עם פלסמיד שליטה (RNA מדריך ריק שיבוט וקטוריים או מדריך RNAs פילוח אזור בקרה שלילית כגון IL1RN יזם). לשימוש transfections ארעי, יחס של 3:2 Cas9 פיוז'ן: מדריך RNA פלסמיד. ניתן להוסיף פלסמידים המכיל כתבים פלורסנט לפקח על תרביות תאים יעילות. - ב 36 h פוסט תרביות תאים, לשנות את התקשורת באמצעות פנול אדום חינם RPMI עם 10% הפשיטו פחם FBS ו 1% פניצילין/סטרפטומיצין (עבור תאים אישיקאווה) והענק puromycin (הריכוז הסופי: 1 μg/mL) ו neomycin (ריכוז סופי: 300 ng/mL). אם תאים רגישים ריאגנט תרביות תאים, התקשורת ניתן לשינוי מוקדם יותר, אך יש להוסיף אנטיביוטיקה לא מוקדם יותר תקנים פוסט 24 שעות ביממה.
הערה: המתן לפחות 24 שעות לאחר הוספת אנטיביוטיקה לפני קציר תאים. שינויים בביטוי עקב שיפור-i יכול להתגלות מוקדם ככל 48 שעות פוסט תרביות תאים, עד 5 ימי פוסט תרביות תאים. אם עובדים עם תאים אישיקאווה נמנעו של אסטרוגן, ביצוע של 8-h 10 nM 17β-אסטרדיול (E2) אינדוקציה יום אחרי טיפול אנטיביוטי, ואז לקצור תאי מיד.
5. תא המסיק והפקת RNA
- להכין מאגר פירוק עם 1% β-mercaptoethanol (BME). ודא שיש מספיק תערובת פירוק-BME (300 μL עבור כל טוב שנקטפו).
- האחות התקשורת באמצעות ליניקת אבק.
- לשטוף את התאים פעם אחת עם אמצעי אחסון שווה ל- 1 x PBS (500 μL), לשאוב כדי להסיר כמה שיותר PBS ככל האפשר.
- להוסיף 300 μL של פירוק-BME פתרון מכל קידוח באמצעות פיפטה רב-ערוצי. Pipette הפתרון פירוק למעלה, למטה 8 - 10 פעמים, והעברת באר לוחית או 1.7 mL גלאים צינורות על קרח. רנ א ניתן באופן מיידי, או שיכול להיות קפוא lysates ב-80 מעלות צלזיוס לעיבוד עתידי.
- כדי לחלץ RNA lysates, להשתמש ערכת זמינים מסחרית הכוללת טיפול DNase. Elute באמצעי האחסון המומלץ הקטן ביותר של מים הנדסה גנטית (RNase-חופשית, נטולת DNase) או • תנאי מאגר ולכמת RNA. עבור מספר קטן של דוגמאות, להשתמש fluorometer או ספקטרופוטומטרים. עבור מספרים גדולים של דגימות, השתמש בדיקה פלורסנט שמזהה RNA ולמדוד על קורא צלחת. שיכול להיות קפוא דגימות ב-80 מעלות צלזיוס לפני או אחרי כמת.
6. שינויים בביטוי גנים באמצעות צעד אחד qPCR ו- RNA-seq לכימות
- להשיג qPCR תחל עבור הגנים של עניין, ג'ין משק אחד לפחות מתבטא ליד רמת הגנים יישוב, לא משנה על פני תנאי הניסוי. באופן אידיאלי, אלה תחל תתפרס על צומת אקסון-אקסון להימנע הגברה של הדנ א.
- מבחן אלה תחל ב- RNA המתקבל הקו תא עניין. שימוש להמיס עקומת ניתוח כדי לאמת את הייצור של מוצר אחד. אם מוצר אחד אינו מיוצר, לבדוק פריימר נוספים זוגות.
- עבור כל שיפור-i ובקרה מדריך RNA מטופלים מדגם, לזהות כמה גנים צריך להיות לבדיקה בדוגמת זו. זו קבוצה של גנים צריך לכלול את הגנים משק בית, כגון CTCF או GAPDH.
- להגדיר תגובות qPCR.
- לדלל כל דוגמאות לריכוז אותו במים, כך 50 ng של RNA הכולל הוא pipetted בקלות, ויש מספיק מדולל RNA עבור כל תגובה. לדוגמה, לדלל RNA ל ~16.6 ng/μL ולהשתמש μL 3 של RNA בתגובה לכל. לשמור RNA על הקרח תוך הגדרת mixes מאסטר.
- הכנת תערובות מאסטר נפרד לכל הגן ניתן למדוד באמצעות ערכות qPCR זמינים מסחרית של צעד אחד. השתמש אמצעי התגובה 20 μL μL 1 של כל פריימר (10 μM מניות פתרון). להגדיר תגובות אלו על קרח.
- צלחת התגובה המתאימה הצנטרפוגה תרמי, להוסיף דוגמאות RNA ואחריו mixes מאסטר. חותם עם סילר לאיטום צלחת ומערבבים בעדינות על-ידי vortexing או pipetting. Centrifuge בקצרה את הצלחת (140 x גר' 60 s) על מנת להבטיח את הנוזלים נמצאת בתחתית הבארות.
- דגירה את הצלחת ב הצנטרפוגה תרמי כדלקמן (או כמו ערכת מורה): 48 º C למשך 30 דקות, 95 מעלות צלזיוס למשך 10 דקות, 40 מחזורי של (95 ° C 15 s, 60 מעלות צלזיוס במשך 1 דקה).
- להשיג ערכי Ct עבור כל ג'ין נמדד בתוך כל דגימה. השתמש בשיטת Ct השוואתי כדי לזהות שינויים בביטוי הגנים.
- להחסיר ה-Ct של הגן משק מן ה-Ct של כל גן מעניין עבור כל דגימה כדי להפיק ערכים Ct מנורמל.
- הדגימות שליטה מטופלים, סעו ממוצע של הערכים Ct מנורמלת עבור כל הגן. יומן בסיס 2-סולם מתקפל דיכוי אז יכול להיות מחושב על-ידי חיסור המדגם Enhancer-i יחס מנורמל Ct עבור כל הגנים של אותו ערך של המדגם שליטה טיפל עבור הגן המתאים.
- כדי לקבוע כללי שינויים בביטוי הגנים לאחר הטיפול משפר-i, להכין את הדגימות רצפי RNA באמצעות ערכת זמינים מסחרית תואם טכנולוגיית רצף של המשתמש. שימוש ~ 500 ננוגרם של RNA להפעלת החומר ולהכין ספריות ושכפול ביולוגית לפחות 2.
7. אימות של פילוח גנומית ספציפיים על-ידי-SID4X-dCas9-קראב באמצעות שבב-seq
הערה: החלבון פיוז'ן SID4X-dCas9-קראב מכילה תג epitope דגל והן תג epitope HA, אך התוצאות הטובות ביותר עבור שבב-seq התקבלו עם נוגדנים נגד דגל. במידת הצורך, המשתמש יכול לבצע ניסויים שבב-seq נוספים עבור גורמי שעתוק שעשוי להיות מושפע Enhancer-i, או עבור H3K27ac, סימן של פעילות וריח מופחת לפי Enhancer-i. עם זאת, כל ניסוי שבב-seq דורש 10 x 106 תאים, כך בהתאם.
- Transfect תאים עם בריכות Enhancer-i.
- צלחת 10 x 106 תאים בצלחת תרביות רקמה 15 ס מ. כל מנה מייצג 1 ניסוי שבב-seq עבור הפקטור 1 עניין.
- למחרת, transfect את התאים באמצעות μg 20 של DNA הכולל לכל תבשיל. עבור transfections בשורות תא stably לבטא SID4X-dCas9-קראב, הדנ א צריך להיות בריכה פלסמיד של מדריך RNAs מיקוד כל אתרי עניין וכאופציה פלסמיד ביטוי חלבון פלואורסצנטי. לשימוש transfections ארעי, יחס של 3:2 dCas9 פיוז'ן חלבון: מדריך RNA בריכה. עבור שבב-seq של שר או שינויים היסטון אחרות, לבצע בקרה נוספים אחד לפחות-מדריך RNA תרביות תאים על תבשיל אחר.
- פנקו את הכלים עם puromycin (1 μg/mL), neomycin (300 ng/mL)-24-48 שעות פוסט תקנים. להמתין לפחות 24 שעות לפני הקטיף כרומטין.
- כרומטין הקציר של מנות.
הערה: כדי לחקור את ההשפעות של שיפור-i ב- ER איגוד גנומית בתאים אישיקאווה, לבצע טיפול nM E2 1 h 10 מנות transfected עם שליטה RNAs הוראות Enhancer-i לפני הקציר. כדי לבחון את השפעות Enhancer-i-H3K27ac, ביצוע של nM 8 h 10 E2 אינדוקציה על מנות transfected עם שליטה RNAs הוראות Enhancer-i לפני הקציר.- ΜL 500 של 37% פורמלדהיד חלות על כל מנה (סופי בריכוז 1%). מערבולת הלוחות בקצרה. תן את הצלחות לשבת בטמפרטורת החדר במשך 10 דקות.
- להוסיף 1 מ"ל של 2.5 מטר גליצין (ריכוז סופי של 125 מ מ). מערבולת הלוחות בקצרה.
- יוצקים את התקשורת עם פורמלין, גליצין. הוסף אמצעי שווה (~ 20 מ ל) ל- PBS קר 1 x.
- יוצקים את טוב, מגניב. האחות עם העצמות ואקום להסיר כמה שיותר PBS ככל האפשר. מניחים את הכלים על קרח.
- להוסיף 3-5 מ ל 1 קר x מאגר פירוק PBS או פצאל (5 מ מ צינורות pH 8.0, 85 מ מ אשלגן כלורי, 0.5% NP-40) עם 1 x מעכב פרוטאז (נוסף רק לפני השימוש) כל צלחת. לגרד את המנה עם במגרדת צלחת ולהעביר את הפתרון צינור חרוטי 15 מ"ל על קרח.
- גלולה כרומטין מאת ספינינג למטה צינורות ומפרידה למשך 5 דקות ב 4 ° C-g 1000 x. להשליך את תגובת שיקוע, לאחסן את כדורי ב- 80 ° C לשימוש עתידי, או להמשיך עם פרוטוקול seq שבב של בחירה באמצעות נוגדן אנטי-דגל או נוגדנים לעבר השני גורמי שעתוק או היסטון שינויים מעניינים (H3K27ac, H3K9me3).
תוצאות
איור 1 מראה סכימטי של זרימת העבודה המתוארת בפרוטוקול. כדי לקבוע התרומות של ER מכורך משפרי ליד הגן מוסדר אסטרוגן MMP17, הכולל 3 אתרי קישור בקרבת מקום כפי שהוגדר על ידי שבב-seq (איור 2 א), מדריך RNAs תוכננו עבור כל אזור. עיצוב מדריך RNAs, חלון bp 600-900 של רצף סביב כל חדר המיון אתר קישור עניין היה נבחר ולשים לתוך תוכנית לעיצוב של RNA מדריך. כתוצאה מדריך RNA וחלבונים בעלי חזה מהמטרה אתרים היו המיושר הגנום האנושי באמצעות בלאט 0-2. ארבעה שאינם חופפים מדריך RNAs שהשתרעו האזור המוגדר על-ידי צ'יפ-seq, רגישות יתר DNaseI נבחרה לצורך מיקוד (איור 2B). רצף נוספים (טבלה 1) נוספה בכל קצה כדי להקל על שיבוט במורד הזרם, וכתוצאה מכך שברי נוקלאוטיד 59 שהוזמנו. עם ההגעה, מדריך RNAs היו מדולל, איחדו על ידי האתר, של ה-PCR קצר בוצעה להוספת אזורים הומולוגיה לפני ההרכבה גיבסון. איור 2C מראה המוצר RNA מדריך הצפוי לאחר PCR קצר באמצעות תחל "U6_internal" (טבלה 1), אשר יוסיף basepairs 20 של רצף בכל צד של basepair 59 מדריך קטע RNA, וכתוצאה מכך רצף ~ 100 basepair. בעקבות הרכבה גיבסון, בשלוליות מדריך RNA הפכו חיידקים, פלסמיד minipreps היו מוכנים ביום המחרת. איור דו-ממדי מראה תוצאות ניסוי לנתיחה משפר, איפה משפרי מרובות הסמוכים MMP17 מיועדים לבד, בשילוב באמצעות שיפור-i. אתרים ממוקד על ידי שיפור-i מסומנים עם משושה שחור. מדריך RNA פלסמידים מיקוד האתרים המצוינים היו transfected לתוך אסטרוגן-מקופח אישיקאווה תא קו stably לבטא SID4X-dCas9-קראב. יומיים לאחר מכן, התקשורת השתנתה, puromycin נוספה כדי להעשיר transfected בתאים. למחרת, התאים נבצרו בעקבות טיפול אסטרדיול 8 h 10 ננומטר. RNA היה מבודד, של צעד אחד qPCR בוצעה. בדוגמה זו, אתרים 1 ו-2 הם הכרחי לתגובה אסטרוגניים מלאה של MMP17, בעוד אתר 3 לא תורם בתנאים אלה (איור דו-ממדי, מסלולים ii-iv). כאשר רק אתרים 2 או 3 פעילים (השישי, השביעי), התגובה אסטרוגן דומה כאשר אין אתרי הם פעילים (השמיני), רומז כי אתרים אלו לא יכולה לתרום באופן עצמאי. אתר 1 יכול לתרום איזה ביטוי בפני עצמו (v), אך הפעילות הגדול נתפסת כאשר אתרים 1 ו- 2 פעילים (iv).
לתמרן משפרי 10 ליד 4 גנים שונים בו זמנית (איור 3 א), מתחם בריכות של מדריך RNAs נוצרו המכיל מדריכים משפר 42 ומדריכים יזם 16. מדריך RNA oligos היו איחדו לפני המדריך הראשוני RNA סיומת ה-PCR (שלב 3.3), וכתוצאה מכך PCR היו מטוהרים ומוצרים בשילוב עם ריק puromycin U6 וקטור שיבוט באמצעות הרכבה גיבסון. לאחר ההרכבה גיבסון, המרות עצמאית מרובים לבצע, מצופה. הלוחות היו שיפשף לתוך ליברות, מותר לגדל החוצה עבור 2-4 שעות לפני maxiprep. איור 3B מציג הפחתות נציג בביטוי הגנים לפי qPCR כאשר בשלוליות RNA מדריך היו transfected לתוך אסטרוגן-מקופח אישיקאווה תא קו stably לבטא SID4X-dCas9-קראב שטופלו כפי שתואר לעיל (איור דו-ממדי). הפחתות ממשפר-i דומים לאלו שהושגו על ידי מיקוד האמרגן של הגן יעד בשם. איור 3C מציג ההשפעות של דילול של מדריך RNAs על הפחתת התגובה אסטרוגן באמצעות שיפור-i. 1:50 דילול של בריכת מדריך RNA מיקוד משפר את יד G0S2 עדיין מניבה הפחתה משמעותית של ביטוי גנים, רומז כי Enhancer-אני יכול לשמש למטרה עד 50 אתרים בו זמנית. עם זאת, שחרור משרות יכול להיות מדולל, המציין כי מאות אתרים לא ניתן לפלח בו זמנית אלא אם כן שיטות זיהוי רגיש יותר מועסקים.

איור 1. פרוטוקול סכמטי של דיסקציה משפר מולטיפלקס באמצעות שיפור-אני מדריך RNAs (אדום וכחול) תוכננו באמצעות e-פריך ו שנבחר באמצעות דפדפן הגנום UCSC. מדריך ארבע RNAs נבחרים זה להקיף את האזורים של הריבית (שעתוק מקדם אתרי קישור כפי שהוגדרו על ידי שבב-seq). מדריך RNA oligonucleotides זה יש כבר איחדו על-ידי האזור של הריבית (אדום וכחול) עוברים על ה-PCR להוספת אזורים הומולוגיה (כתום) לפני גיבסון הרכבה ושינוי. בריכות פלסמיד וכתוצאה מכך הם transfected ויה lipofection לתוך שורות תאים stably לבטא SID4X-dCas9-קראב או פראי-סוג התאים בשיתוף עם SID4X-dCas9-קראב פלסמיד. מדריך RNA פלסמיד בריכות יכול להיות transfected בנפרד למטרה אתר אחד בכל פעם, או בשילוב היעד באתרים מרובים בו-זמנית. תאים transfected מטופלים באנטיביוטיקה כדי להעשיר עבור תאים המכיל מדריך RNAs. ב ~ 72 h פוסט תרביות תאים, התאים נקצרים. חומצות גרעין ניתן לחלץ qPCR, RNA-seq או שבב-תת סעיף אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
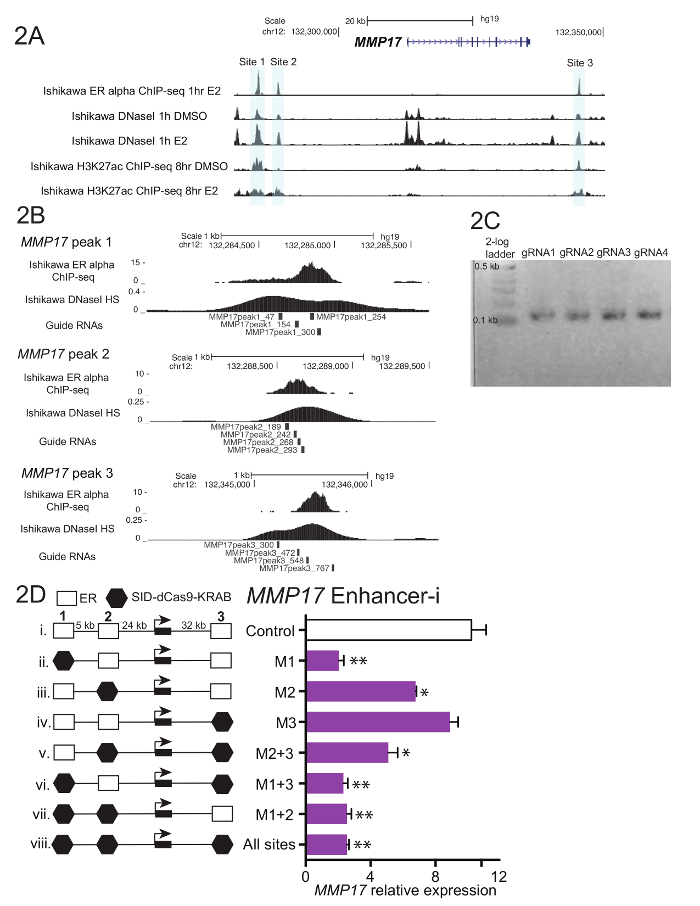
באיור 2. מדריך ניתוח ועיצוב משפר RNA עבור MMP17. (א) הגנום דפדפן צילום מסך של חיזוק אלפא מכורך ER (אפור) כדי להיות ממוקד ליד MMP17. איור זה שונה מ- Carleton, et al. 18. (B) מדריך RNA עיצובים עבור 3 איגוד אתרי18. האתר מחייב מיון כפי שהוגדרו על ידי שבב-seq היא המטרה, המשבצת RNAs מדריך 4 ברחבי האזור. האות רגישות DNaseI, אשר משתרע על פני אתר איגוד, יכול לשמש גם כדי להגדיר יעד רצף עבור מדריך עיצוב ה-RNA. שבב-seq והן DNaseI HS הנתונים התקבלו מתאי אישיקאווה שטופלו אסטרדיול nM 10 עבור ח' 1 (ג) מדריך נציג RNA רצפים המוכנים להרכבה גיבסון, שעברו של PCR קצר כדי להוסיף הומולוגיה אזורים. (ד) ביטוי היחסי של MMP17 נמדד באמצעות qPCR בעקבות המיקוד של אזורים ספציפיים עם משפר-i טיפול אסטרדיול nM 8-h10. הביטוי הוא יחסי CTCF ורמת ביטוי של MMP17 בתאים אינו מטופל באמצעות אסטרדיול. מדריך שליטה RNAs היעד האמרגן של IL1RN. כל קווי השגיאה מייצגים SEM, כוכביות זוגי לציין p < 0.01 ורווק הכוכביות מציינות p < 0.05 ב מבחן t מזווג. איור זה שונה מ- Carleton, ואח. 18. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
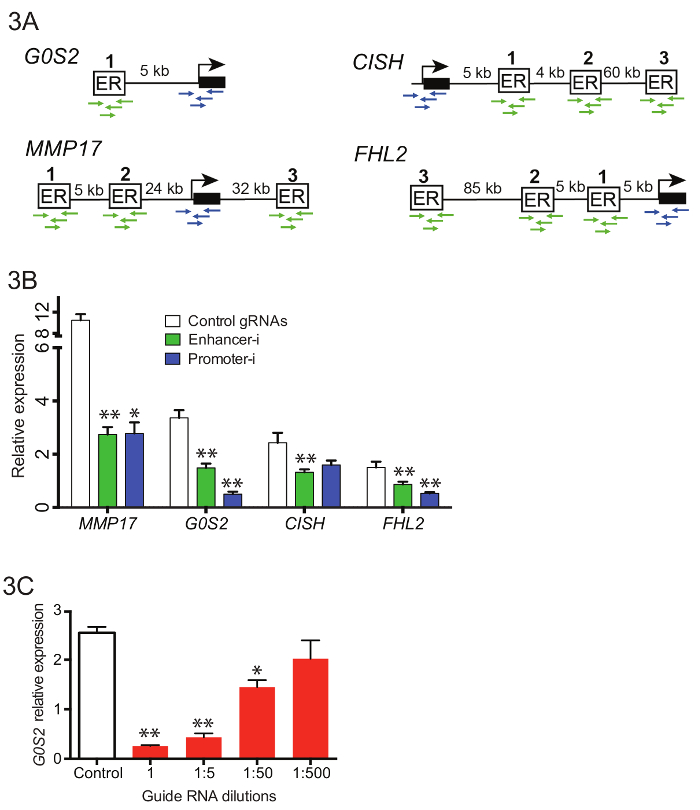
איור 3. פילוח משפרי מרובים בקרבת גנים שונים בו זמנית עם איחדו Enhancer-אני תיאור סכמטי של איגוד אתרים, היזמים כדי להיות ממוקד משפר במאגר-i (א) . (B) ההשפעות על ביטוי כפי שהיא נמדדת qPCR לאחר E2 טיפול בתאי אישיקאווה transfected עם בריכה פלסמיד Enhancer-i (ירוק), יזם-i פלסמיד הבריכה (כחול) או פקד gRNAs (לבן)18. הפחתה משמעותית על כל הגנים הוא ציין עם משפר-i. איור זה שונה מ- Carleton, ואח. 18. (ג) ההשפעות על רמות ביטוי G0S2 לאחר הטיפול E2 בתאי אישיקאווה transfected עם כמויות שונות של מדריך RNAs מיקוד G0S2. ניתן לראות ירידה משמעותית אפילו עם כמויות קטנות של מדריך RNA (1:50 דילול), מציע את זה עד 50 אתרים ייתכן ממוקד בו זמנית. כל קווי השגיאה מייצגים SEM, כוכביות זוגי לציין p < 0.01 ורווק הכוכביות מציינות p < 0.05 ב מבחן t מזווג. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
| שם | רצף |
| U6_internal_F | TTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCG |
| U6_internal_R | GACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC |
| U6_PCR_F | CCAATTCAGTCGACTGGATCCGGTA |
| U6_PCR_R | AAAAAAAGCACCGACTCGGTGCCA |
| gRNA_qPCR_F | GCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG |
| gRNA_qPCR_R | AAAAAGCACCGACTCGGTGCC |
| dCas9_qPCR_F | GTGACCGAGGGAATGAGAAA |
| dCas9_qPCR_R | AGCTGCTTCACGGTCACTTT |
| pAC95_PCR_F | AGAAGAGAAAGGTGGAGGCC |
| pAC95_PCR_R | CGTCACCGCATGTTAGAAGG |
| SID4X_PCR_F | CAATAGAAACTGGGCTTGTCG |
| SID4X_PCR_R | TCGTGCTTCTTATCCTCTTCC |
טבלה 1. תחל משמש מדריך RNA סיומת, רצף, qPCR, וזיהוי של החלבון פיוז'ן.
Discussion
פרוטוקול זה מתאר שיטה פשוטה וגמישה ניקוד פונקציית מעצם מיקומה גנומית אנדוגני מבלי פיזית לשנות את רצף ה-DNA. בזמן דומה ברעיון שפורסמו בעבר פרוטוקולי להפרעה CRISPR באמצעות dCas9-קראב27, משפר-i שונה פרוטוקולים אלה ב 3 דרכים הראשי. ראשית, משפר-אני מנצל המחשבים שמעצבת SIN3A של MAD120 כדי להשיג שיפור הביטול. משפר הביטול יכול להינצל באמצעות מעכבי HDAC, רומז המנגנון העיקרי של הביטול הוא HDAC תלויים. שלא כמו הפרעות CRISPR עם dCas9-קראב, משפר-i לא תוביל בתצהיר של H3K9me3. זה סביר בשל העובדה כי Enhancer-i נסמכת על מבוא ארעית של מדריך RNAs, עם התאים להיות נקצרו 3 ימים לפרסם תקנים. בהפרעות CRISPR, נצפית עלייה H3K9me3 ב 7 ימים-פוסט-התמרה חושית-12. לבסוף, פרוטוקול Enhancer-i מספק אסטרטגיה היעד באתרים מרובים בו-זמנית ולעקוב אחר היעילות של מיקוד. פסיפס-seq17, dCas9-קראב משמש למטרה משפרי מרובים בו זמנית, אבל טכניקה זו מסתמכת על רצפי RNA בתא יחיד כדי לזהות שינויים בביטוי, גנים רבים (כגון אסטרוגן מגיב גנים) להתגלות עקב נמוכה הרגישות של תא בודד RNA-תת סעיף. משפר-i מספק שיטה אמינה ללמוד משפרי בנפרד, בשילוב עבור כל הגן.
השלב הקריטי ביותר של שיפור-i הוא תרביות תאים, אשר צריך להיות ממוטבים עבור שורת תאים של עניין. פרוטוקול זה מסתמך על טיפול puromycin להעשיר עבור תאים transfected, אבל זה אפשרי המדריך משותפת transfecting RNAs עם החלבון הניאון ומיון עבור פלורסנט תאים באמצעות cytometry זרימה עשויה להוכיח שיטת העשרה יותר באיזה תא סוגי. אנו ממליצים ממעקב את רמת הביטוי של מדריך RNAs ואת SID4x-dCas9-קראב qPCR לפתור בעיות, לאשר תרביות תאים. אם מדריך RNA רמות נמוכות (מחזור הסף > 30), משתמשים עשויים גם לשקול חלופי מדריך RNA אסטרטגיות הייצור כגון במבחנה שעתוק28. זה גם אפשרי כי למרות רמות gRNA גבוהות, מדריך RNA פילוח של החלבון SID4x-dCas9-קראב הוא לא יעיל, ובמקרה בחירה שונה מדריך רצפי RNA עשוי להיות נחוץ. על ידי ביצוע שבב-seq על חלבון פיוז'ן עם כרומטין מתאי Enhancer-i מטופלים, ניתן לנטר את היעילות של מיקוד. אם אות גבוה של SID4x-dCas9-קראב-האזור של עניין, אין שינויים בביטוי בגן שלה יעד בשם מזוהים, ואז האזור סביר שאינה תורמת הביטוי של הגן הזה תחת התנאים למד.
מגבלה אחת פוטנציאלי של Enhancer-i היא כי את המטרה אפקטים עלולים להצטבר אם אתרים רבים מדי מיועדים בו זמנית. בכל זאת, CRISPR אסטרטגיות התערבות של נוקאאוט יש פחות את היעד תופעות מאשר RNAi29, במיוחד כאשר קו תא polyclonal לבטא dCas9-קראב משמש. בזמן שראינו את המטרה איגוד גנומית של SID4X-dCas9-קראב כשמתכוונים 10 אתרים בו זמנית, לא זיהינו שינויים ביטוי הגן בעקבות אירועים אלה מחייב. כמו כמה משפרי ניתן לפנות היזמים מרובים ו/או מוצרי טיפוח טבעיים אחרים, זה אפשרי כי גנים רבים עשויים להשתנות ביטוי לפי פילוח של תוספת יחיד, שלא ברור אם זה סוג של הכונה היא נפוצה. כדי לאשר השינויים ביטוי נצפו הן בשל המיקוד תוספת ספציפית, לא רחוק ממסלול הנחיתה-אפקטים, יוכלו המשתמשים לבצע שיפור-i עם שתי קבוצות נפרדות של מדריך שאינם חופפים RNAs פילוח באותו האזור. בנוסף, המחיקה גנטי של האזור באמצעות Cas9 נוקלאז-מוכשר נוסף יכול לאשר את השפעותיו על ביטוי גנים.
כפונקציות Enhancer-i דרך deacetylation היסטון, זה אפשרי כי יכולותיו לנטרול מוגבלות למוצרי טיפוח טבעיים בעלי רמות ניכר של היסטון acetylation. ישנם מגוון fusions דכאניים חלופיים שעשויים להיות יעילה יותר מיקוד משפרי ספציפיים. ה-DNA fusions methyltransferase כדי dCas9 יכול לשמש כדי להפחית ביטוי גנים כאשר ממוקד משפרי דיסטלי30, אבל הדיכוי הזה הוא לעתים קרובות לא חולף. פיוז'ן דכאניות אחר משתמש התחום חבר של GATA1 (FOG1), אשר מוביל אל היסטון H3 ליזין 27 trimethylation, represses ביטוי גנים ברמות דומה dCas9-קראב במגוון של היזמים וקווי תא31. מעניין, הוספת יותר עותקים של FOG1 dCas9 לצמצם את הפוטנציאל דכאניים אצל היזמים, רומז כי עותק יחיד של התחום סיד עשוי לספק לנטרול משפר עוד יותר העותקים 4 בשימוש כיום במשפר-i. זה אפשרי כי יש לוקוסים עשויה להועיל מיקוד כפול על ידי שילובים שונים של fusions dCas9 לעיל. לדוגמה, דיכוי ארוך טווח יציב יכולה להיות מושגת על ידי התמרה חושית סימולטני של dCas9-DNMT3a ו- dCas9-קראב32. רוב fusions דכאניים אלה רק סומנו כמטרות כדי לוקוס בודד בכל פעם, והוא נותר לא ברור שזה יעיל ביותר מניפולציה משפרי מרובים בו-זמנית.
משפר-i, בעוד שיטה מתאימה ללמוד שילובים של מרחיבי קומץ של גנים, עדיין קצת מוגבל בתפוקה אם המשתמש מבקש ללמוד בשם מרחיבי מאות גנים. יישומים עתידיים של טכניקה זו תשלב טכנולוגיות מבוססות-הדמיה לכמת גנים מרובים בדגימות מרובות בו-זמנית. חשוב לציין, טכנולוגיות אלה תואמים זיהוי ישיר של מולקולות RNA מן lysate, ומבטל את הצורך לבידוד RNA גוזלת זמן. עיבודים אלה יקל על חקירתו של קבוצות גדולות של מוצרי טיפוח טבעיים.
Disclosures
המחברים אין לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי NIH/NHGRI R00 HG006922 HG008974 R01 NIH/NHGRI אל האם, ואת מכון הסרטן הצייד. J.B.C. נתמכה על ידי תוכנית אימונים NIH ב גנטיקה של T32GM007464.
Materials
| Name | Company | Catalog Number | Comments |
| ZR 96-well Quick-RNA Kit | Zymo Research | R1053 | |
| Power SYBR Green RNA-to-CT 1-Step | Applied Biosystems | 4389986 | |
| AflII restriction enzyme | NEB | R0520S | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531L | |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | |
| FuGENE HD | Promega | E2312 | |
| DNA Clean & Concentrator Kit | Zymo Research | D4013 | |
| Buffer RLT Plus | Qiagen | 1053393 | |
| b-estradiol | Sigma-Aldrich | E2758 | |
| Human: Ishikawa cells | ECACC | 99040201 | |
| H3K27ac rabbit polyclonal | Active Motif | 39133 | |
| H3K9me3 rabbit polyclonal | Abcam | ab8898 | |
| FLAG mouse monoclonal | Sigma-Aldrich | F1804 | |
| ER alpha rabbit polyclonal | Santa Cruz | sc-544 | |
| pGL3-U6-PGK-Puro plasmid | Addgene | 51133 | Shen et al., 2014 |
| gRNA_cloningVector plasmid | Addgene | 41824 | Mali et al., 2013 |
| AflII U6 puromycin plasmid | Addgene | 106404 | Carleton et al., 2017 |
| SID4X-dCas9-KRAB plasmid | Addgene | 106399 | Carleton et al., 2017 |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-10ML | |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977-023 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985070 | |
| KAPA Stranded mRNA-Seq Kit, with KAPA mRNA Capture Beads | Kapa Biosytems | KK8420 | |
| Pierce Protease and Phosphatase Inhibitor Mini Tablets | ThermoFisher Scientific | A32959 | |
| Formaldehyde solution | Sigma-Aldrich | 252549-25ML | |
| Geneticin Selective Antibiotic (G418 Sulfate) (50 mg/mL) | ThermoFisher Scientific | 10131035 | |
| LB Broth | ThermoFisher Scientific | 10855001 | |
| Quick-DNA Miniprep Kit | Zymo Research | D3020 | |
| Quick-Load Purple 2-Log DNA Ladder | NEB | N0050S |
References
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Roadmap Epigenomics, C., et al. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Perry, M. W., Boettiger, A. N., Bothma, J. P., Levine, M. Shadow enhancers foster robustness of Drosophila gastrulation. Curr Biol. 20 (17), 1562-1567 (2010).
- Lam, D. D., et al. Partially redundant enhancers cooperatively maintain Mammalian pomc expression above a critical functional threshold. PLoS Genet. 11 (2), e1004935 (2015).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Savic, D., et al. Promoter-distal RNA polymerase II binding discriminates active from inactive CCAAT/ enhancer-binding protein beta binding sites. Genome Res. 25 (12), 1791-1800 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23 (10), 1163-1171 (2013).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol. , (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods. 12 (5), 401-403 (2015).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods. 12 (12), 1143-1149 (2015).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6 (3), e1000869 (2010).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Fulco, C. P., et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science. 354 (6313), 769-773 (2016).
- Joo, J. Y., Schaukowitch, K., Farbiak, L., Kilaru, G., Kim, T. K. Stimulus-specific combinatorial functionality of neuronal c-fos enhancers. Nat Neurosci. 19 (1), 75-83 (2016).
- Xie, S., Duan, J., Li, B., Zhou, P., Hon, G. C. Multiplexed Engineering and Analysis of Combinatorial Enhancer Activity in Single Cells. Mol Cell. 66 (2), 285-299 (2017).
- Carleton, J. B., Berrett, K. C., Gertz, J. Multiplex Enhancer Interference Reveals Collaborative Control of Gene Regulation by Estrogen Receptor alpha-Bound Enhancers. Cell Syst. 5 (4), 333-344 (2017).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Ayer, D. E., Laherty, C. D., Lawrence, Q. A., Armstrong, A. P., Eisenman, R. N. Mad proteins contain a dominant transcription repression domain. Mol Cell Biol. 16 (10), 5772-5781 (1996).
- Alland, L., et al. Role for N-CoR and histone deacetylase in Sin3-mediated transcriptional repression. Nature. 387 (6628), 49-55 (1997).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Rennoll, S. A., Scott, S. A., Yochum, G. S. Targeted repression of AXIN2 and MYC gene expression using designer TALEs. Biochem Biophys Res Commun. 446 (4), 1120-1125 (2014).
- Stampfel, G., et al. Transcriptional regulators form diverse groups with context-dependent regulatory functions. Nature. 528 (7580), 147-151 (2015).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods. 10 (10), 973-976 (2013).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nat Methods. 11 (2), 122-123 (2014).
- Parsi, K. M., Hennessy, E., Kearns, N., Maehr, R. Using an Inducible CRISPR-dCas9-KRAB Effector System to Dissect Transcriptional Regulation in Human Embryonic Stem Cells. Methods Mol Biol. , 221-233 (2017).
- Romanienko, P. J., et al. A Vector with a Single Promoter for In Vitro Transcription and Mammalian Cell Expression of CRISPR gRNAs. PLoS One. 11 (2), e0148362 (2016).
- Smith, I., et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map. PLoS Biol. 15 (11), e2003213 (2017).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Res. 45 (17), 9901-9916 (2017).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved