A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
עיצוב, סינתזה של ארון אקורדיון דנ א Reconfigurable
* These authors contributed equally
In This Article
Summary
אנו מתארים פרוטוקול מפורט עבור עיצוב, הדמיה, רטוב-מעבדת ניסויים וניתוח עבור מתלה reconfigurable של אקורדיון הדנ א של 6 על ידי 6 רשתות.
Abstract
במערכות מבוססות-ננו-מבנה ה-DNA מכני או ננו משינס הדנ א, אשר מייצרים תנועה הננומטרי מורכבות ב- 2D and 3D ננומטר לרזולוציה ångström, הראה פוטנציאל גדול בתחומים שונים של ננו-טכנולוגיה כגון הכורים מולקולרית, משלוח סמים, ומערכות nanoplasmonic. Reconfigurable DNA אקורדיון המדף, אשר באופן קולקטיבי שבאפשרותך לתפעל רשת הננומטרי 2D או 3D של אלמנטים, בשלבים מרובים בתגובה הקלט דנ א, מתואר. פלטפורמת יש פוטנציאל להגדיל את מספר רכיבים ננו משינס DNA ניתן לשלוט של מספר רכיבים כדי מידה רשת בשלבים רבים של שינוי תצורה.
ב פרוטוקול זה, אנו מתארים את התהליך ניסיוני של reconfigurable ה-DNA אקורדיון ארון התקשורת של 6 על ידי 6 רשתות שינוי. הפרוטוקול כולל עיצוב כלל וסימולציות הליך של המבנים ושל ניסוי רטוב-lab סינתזה, שינוי תצורה. בנוסף, ניתוח המבנה באמצעות TEM (במיקרוסקופ אלקטרונים הילוכים) וקשת/קשתית (העברת אנרגיה של קרינה פלואורסצנטית תהודה) נכלל בפרוטוקול. שיטות הדמיה ועיצוב חדשניים מכוסה פרוטוקול זה יסייע לחוקרים להשתמש המדף אקורדיון DNA עבור יישומים נוספים.
Introduction
מערכות מכאניות המבוסס על ה-DNA nanostructures או DNA ננו משינס1,2,3,4,5 הם ייחודיים משום שהם מייצרים תנועה הננומטרי מורכבות ב- 2D and 3D ננומטר ל רזולוציה ångström, על-פי3,2,גירויים למערכות ביולוגיות שונות6. על ידי הצמדת חומרי פונקציונלי על מבנים אלה ושליטה על עמדותיהם, מבנים אלה ניתן ליישם בתחומים שונים. לדוגמה, ה-DNA ננו משינס הוצעו עבור הכור מולקולרית7, משלוח סמים8ו-9,מערכות nanoplasmonic10.
בעבר, הצגנו reconfigurable DNA אקורדיון המדף, אשר יכול לתפעל רשת הננומטרי 2D או 3D של אלמנטים11 (איור 1 א'). שלא כמו אחרים ננו משינס DNA רק לשלוט כמה אלמנטים, פלטפורמת יכולים באופן קולקטיבי לתמרן אלמנטים 2D או 3D מעת לעת ערוכים לשלבים שונים. אנו צופים כי רשת תגובה כימית וביולוגית לתכנות או מערכת מחשוב מולקולרי יכול להיבנות מהמערכת שלנו, על ידי הגדלת מספר הרכיבים לשליטה. המתלה אקורדיון הדנ א הוא מבנה, שבו הרשת של קורות DNA מרובים מחובר המפרקים המורכב חד גדילי הדנ א (איור 1B). המתלה אקורדיון שנוצר על ידי הקורות דנ א למנוחת מאת המנעולים הדנ א, אשר hybridize לחלק דביק של קורות, לשנות את הזווית בין הקורות על פי אורך החלק גישור של המנעולים (מצב סגור). בנוסף, שינוי תצורה צעד מרובת מומחש הוספת מנעולים חדשים לאחר היווצרות המדינה בחינם על ידי ניתוק DNA מנעולים עד12,הזחה סטרנד מבוססי מקום אחיזה לרגל13.
ב פרוטוקול זה, אנו מתארים את התהליך כולו של סינתזה ועיצוב של ארון התקשורת אקורדיון דנ א reconfigurable. הפרוטוקול כולל עיצוב, הדמיה, רטוב-מעבדת ניסויים וניתוח לסינתזה של המדף אקורדיון הדנ א של 6 על ידי 6 רשתות שינוי, שינוי של תצורה אלה. המבנה מכוסה הפרוטוקול הוא הדגם הבסיסי של ה מחקר קודמות11 והוא 65 ננומטר על ידי 65 ננומטר בגודלם, בהיקף של 14 קורות. מבחינת עיצוב, הדמיה, העיצוב מבנית של המדף אקורדיון שונה קונבנציונאלי DNA אוריגמי14,15 (קרי, בצפיפות). לכן, כלל עיצוב, הדמיה מולקולרית שונו שיטות מסורתיות. כדי להדגים, אנו מראים את טכניקת עיצוב באמצעות הגישה ששונה caDNAno14 והסימולציה של המדף אקורדיון באמצעות16,oxDNA17 עם תסריטים נוספים. לבסוף, מתוארים שני הפרוטוקולים של TEM, סריג לצורך ניתוח של מבנים מתלה אקורדיון שתצורתו נקבעה.
Protocol
1. עיצוב של 6 על ידי ארון תקשורת אקורדיון דנ א 6 עם caDNAno14
- להוריד ולהתקין תוכנה caDNAno 2.014 לעצב ארון אקורדיון דנ א (caDNAno 2.5 זמינה גם ב- https://github.com/cadnano/cadnano2.5). פתח caDNAno14 ' ולחץ על ' כלי מרובע כדי להוסיף תפקיד חדש עם סריג מרובע '.
- מספר כל קרן של המדף אקורדיון ולצייר בלוח השמאלי סריג של caDNAno14 (איור 2).
- לחץ על הכלי עיפרון וצייר בכל הקורה בלוח העריכה נכון caDNAno14. הפסקה קורות בכל 32 bp, אשר עבור המפרקים בין קורות סמוכות. מקום סיכות ג'יי ג'יי אברהמס באותה העמדה המפרקים. להשתמש בכלי הוספת הכלי עיפרון caDNAno14 לתת את המפרקים יש נוספת ג'יי ג'יי אברהמס חד גדילי.
- לחץ על הכלי עיפרון וחבר המפרקים. לכל קרן יש שבעה בתי עסק.
- צור ג'יי ג'יי אברהמס לגרדום כדי למזג פיגומים לתוך הלולאה יחיד על-ידי שימוש בפיגומים שדווחה בעבר ניתוב אלגוריתם11. אל תתנו את התחום מחייב מינימום בין הגדילים לגרדום והידוק להיות פחות מ 8 bp (איור 3).
- למקם פיגומים שלא נעשה בהם שימוש באסיפה על הקודקודים ממוקם משני צדי המדף אקורדיון, כפי שמוצג באיור3.
- לחץ על לשבור את הכלי. לשבור את הגדילים איפה סיכות גדילי מעגלית או יותר מ 60 bp.
- לעצב את נעילת התאים.
- לחץ על לשבור את הכלי. הפסקה 8 bp של אזור ה-DNA סיכות כדי להפוך חלק דביק ולמחוק 8 bp של אזור ה-DNA סיכות. ישנם 18 חלקים דביק (איור 1) בארון 6 על ידי 6 אקורדיון.
- מקום רצפים לאחור משלים החלקים דביק בשני קצותיו של מנעול קווצות ולחבר אותם על ידי אזור הגישור, אשר מורכב של פולי T קווצות לאורך הרצוי (איור 1B).
- עבור תצורת, להוסיף 8 bp של רצפי מקום אחיזה לרגל בקצה של ה-DNA מנעולים סטרנד העקירה. הרצף מקום אחיזה לרגל בשימוש הוא בטבלה מס ' 2.
- להכין את הגדילים poly A אשר נמצאים הפוך משלימים לאזור גישור.
- גדילי עיצוב שמבטלות משלים המנעולים DNA לניסוי. שינוי תצורה.
- לחץ על הכלי רצף ולחץ על DNA לגרדום. לבחור לגרדום כמו M13mp18 רגיל. לחץ על כלי ייצוא , שמירת רצף בתבנית csv (טבלה 1).
2. לדמות את המבנה עם oxDNA
- הורד והתקן את16,oxDNA17. קוד המקור האחרון זמין על https://sourceforge.net/projects/oxdna/files/.
- להפוך קבצי תצורה החל מקובץ14 caDNAno שימוש ב- script פייתון 'cadnano_interface.py', אשר מסופק באריזה17 16,oxDNA. השימוש הוא כדלקמן: 'פייתון cadnano_interface.py cadnano_file.json ר'. טופולוגיה הקובץ של קובץ התצורה נוצרים עכשיו.
הערה: הקובץ טופולוגיה כולל כמה קווצות, נוקלאוטידים נמצאים המבנה ואת מידע לגבי עמוד שדרה-עמוד השדרה הקשרים בין נוקלאוטידים. קובץ התצורה כוללת מידע כללי כגון timestep, אנרגיה, גודל התיבה. התמצאות מידע כגון מיקום וקטורי בסיס עמוד השדרה וקטור, וקטור נורמלי, מהירות, מהירות זוויתית של נוקלאוטידים הוא גם כלולים (איור 4). - לשנות את המידע בקובץ התצורה וטופולוגיה מ caDNAno14 כדי לגרום להם משקפים את המידע המבני אמיתי של ארון התקשורת אקורדיון. קורות כל מסודרים במקביל כאשר הם דמיינו את הטופולוגיה וקובצי תצורה מ caDNAno14 . עם זאת, המדף אקורדיון הוא מבנה סריג אז המרחק בין נוקלאוטידים בונדד רחוק עבור הסימולציה (איור 5).
- לסובב כל קרן ולעבור מבנה סריג הרצוי. 9 טורים משמאל בקובץ התצורה הם וקטור המיקום, עמוד שדרה-בסיס וקטור, וקטור נורמלי (איור 4). כדי לסובב את קרן, לסובב כל עמדה, וקטורים. בסיס עמוד השדרה, ואני רגילה באמצעות טרנספורמציה המסתובבת. לאחר מכן להזיז קרן על-ידי שינוי וקטור המיקום כדי לאתר אותו, כמוצג באיור5.
- להירגע למבנה באמצעות קובץ ה-script מסופק באריזה oxDNA (עיין בדוגמה $oxDNA/דוגמאות/RELAX_INITIAL_CONFIGURATION למידע נוסף).
- הפעל את הסימולציה דינמיקה מולקולרית עבור 10 מיליון שלבים באמצעות קובץ התצורה רגועה. השימוש הוא כדלקמן: '. / oxDNA < input >' שמירת נתוני כל 5000 או 10000 צעדים.
- ויזואליזציה
הערה: המבנים היו visualized באמצעות כמו.- הורד והתקן את הגירסה העדכנית ביותר של כמו (https://sourceforge.net/projects/cogli1/).
- להפעיל את כמו עם קבצי תצורה וטופולוגיה סימולציה oxDNA. השימוש הוא כדלקמן: '. / cogli1 -t < טופולוגיה קובץ >< קובץ תצורה >'.
- להסתיר את תיבת על-ידי הקשה על b.
3. סינתזה של המבנה
הערה: שיטת סינתזה מותאמת מן הקודם פרוטוקול15,18.
- לרכוש במקום DNA מעוצב מ ספק oligonucleotide.
- התאם את הריכוז של אלה סיכות דנ א כדי 100 μM בעזרת נוקלאז ללא מים.
- בריכה כל צירוף ד. נ המהווה את מבנה 'שחררו את המדינה' לתוך שפופרת אחת ולהתאים את ריכוז μM 2 על גדיל אחד.
- מאגר ה-DNA נעל קווצות לפי אורך ומספר אתרי נעל לתוך צינורות ולהתאים את ריכוז μM 2 על גדיל אחד. אתרי נעל 18, 9 ו- 4 משמשים. הוסף פולי A גדילי אשר משלימים לאזור גישור-ריכוז זהה.
- גדילי בריכה שמבטלות משלימים ל- DNA לנעול חוטים האורך לתוך צינורות ולהתאים את ריכוז μM 2 על גדיל אחד.
- להכין תמיסת MgCl-2 , 300 ננומטר על ידי ערבוב 70 μL של מים נטולי נוקלאז ו 30 μL של הפתרון2 1 μM MgCl. הכן 5 x טריס EDTA פתרון על ידי ערבוב μL 95 נטול נוקלאז מים ו 5 μL של הפתרון טריס EDTA 100 x.
- להוסיף 2 μL של הידוק ה-DNA, μL 1.1 בפתרון2 MgCl, 2 μL של פתרון טריס EDTA, 7.6 μL של מים נטולי נוקלאז ו- 7.3 μL של ה-DNA לגרדום אשר הריכוז הוא 110 nM כדי להפוך μL 20 מניות מעורבת. להגדיר את הריכוז הסופי של לגרדום דנ א ל-40 ננומטר, להדק את הדנ א 200 ננומטר, MgCl2 16 מ"מ ו- EDTA טריס לפתרון 0.5 x.
- במהירות חום הפתרון מלאי מעורבות של הצנטרפוגה תרמי עד 80 ° C וקריר עד 60 ° צלזיוס בקצב של 4 דקות לכל ° C וקריר מ- 60 ° C עד 4 ° C בקצב של 40 דקות לכל מעלות צלזיוס.
4. טיהור של המבנה
הערה: דוגמאות של כל המבנים היו טהור לפני ניתוח. בסעיף זה, נתאר הפרוטוקול של פג-טיהור, אשר מותאם בין ספרות הקודם19. המדגם יכול גם להיטהר על ידי אלקטרופורזה בג'ל כפי שמתואר בספרות הקודם15,18.
- להכין 5 מ' של NaCl, טריס x 100-EDTA.
- הכן משקעים-מאגר על ידי ערבוב μL 150 של 8000 פג, 500 μL של 100 x טריס EDTA, μL 101 של 5 M NaCl 249 μL של נוקלאז ללא מים.
- הכן היעד-מאגר על ידי ערבוב μL 5.5 בפתרון nM MgCl2 300 מ סעיף 3.3, μL 10 בפתרון טריס EDTA x 5 של סעיף 3.3 ו μL 84.5 נוקלאז ללא מים.
- מיקס 20 μL של המבנה מסונתז בסעיף 3, μL 20 משקעים-מאגר של סעיף 4.2. ואז לסובב המניה מעורב ב g x 16000-4 מעלות צלזיוס. הסר את תגובת שיקוע, להמיס צניפה במאגר-המטרה של סעיף 4.3.
5. שינוי תצורה של ארון התקשורת אקורדיון ממצב' חינם' כדי 'נעול המדינה'
- לסנתז את המבנה ללא DNA מנעולים לניסוי תצורה.
- להכין את נעילת התאים של סעיף 3.
- להוסיף 2 μL של DNA נעל קווצות לאורך הרצוי לתוך 20 μL של המבנה מסונתז. הריכוז של גדילי נעל ה-DNA הוא גבוה פי חמישה מאשר המבנה.
- דגירה המדגם על 0, 10, 25, 50 או 100 דקות כדי לראות כמה מהר שינוי תצורה מתרחשת.
- הדגירה 100 דקות, דגירה המדגם ב 50 מעלות צלזיוס למשך 30 דקות, מגניב לאט עד 25 ° C בקצב של 0.33 ° C/דקה.
- הדגירה 50 דקות, דגירה המדגם ב 50 מעלות צלזיוס במשך 15 דקות, מגניב לאט עד 25 ° C בקצב של 0.66 מעלות/דקות.
- הדגירה 25 דקות, דגירה המדגם ב 50 מעלות צלזיוס במשך 7.5 דקות, מגניב לאט עד 25 ° C בקצב של 1.32 ° C/דקה.
- הדגירה 10 דקות, דגירה המדגם ב 50 מעלות צלזיוס במשך 3 דקות, מגניב לאט עד 25 ° C בקצב של 3.3 ° C/דקה.
- עבור הדגירה 0 דקות, לאחסן לדוגמה 4 ° C מימין לאחר הוספת מנעולי התאים.
- מיד לאחר השלב attaching, במהירות לקרר את הדגימה עד 4 ° C כדי למנוע דנטורציה בלתי רצויים.
6. שינוי תצורה של ארון התקשורת אקורדיון מ 'נעול במצב' למצב' חינם'
- לסנתז את המבנה עם מנעולים הדנ א לאורך הרצוי עבור הניסוי תצורה.
- להכין את הגדילים משלימים הפוכה של סעיף 3.
- להוסיף 2 μL של קווצות הם להפוך משלים המנעול גדילי לאורך הרצוי לתוך 20 μL של המבנה מסונתז. הריכוז של גדילי נעל ה-DNA הוא גבוה פי חמישה מאשר המבנה.
- דגירה המדגם על 0 12, 60, 120, 240 דקות לראות באיזו מהירות שינוי תצורה מתרחשת.
- ה-12, 60, 120, דגירה 240 דקות, במהירות לחמם את הדגימה עד 40 ° צלזיוס, לאט להתקרר עד 20 מעלות צלזיוס למשך הזמן המתאים לכל. מיד לאחר השלב התולש, במהירות לקרר את הדגימה עד 4 ° C כדי למנוע דנטורציה בלתי רצויים.
- עבור הדגירה 0 דקות, לאחסן לדוגמה 4 ° C מימין לאחר הפוכה גדילים משלימים נוספים.
7. TEM הדמיה
הערה: פרוטוקול הדמיה TEM הותאמה ספרות קודמת18,20.
- הכן 1.25 מ' NaOH פתרון על ידי ערבוב 87.5 μL של מים נטולי נוקלאז μL 12.5 בפתרון NaOH 10 מ'.
- להוסיף 1 μL של 1.25 מ' NaOH פתרון μL 50 של הפתרון formate uranyl 2%.
- מערבולת הפתרון במשך 3 דקות, צנטריפוגה-מהירות מקסימלית במשך 3 דקות. להפקיד μL 3 המדגם מטוהרת על הרשת TEM משוחררים-זוהר במשך 3 דקות, במהירות לשטוף עם נייר סינון.
- להפקיד 7 μL של uranyl מוכן formate פתרון למשך 30 שניות, במהירות לשטוף עם נייר סינון.
- למדוד את האורך והזווית של המבנה אקורדיון עם תמונה מאת TEM.
8. סריג ניתוח
- השתמש אטו 550 וצבע אטו 647N, אשר המרחק פורסטר הוא 6.5 ננומטר. החלף גדילי fluorescently שכותרתה הידוק 58 ו- 117 הידוק בטבלה 1. ואז לסנתז את המבנה עם גדילים fluorescently עם תוויות ברורות על ידי השיטה המתוארת בסעיף 3.
- למדוד את הריכוז של המדגם מטוהרים.
- לנרמל את הדגימה כדי 10 ננומטר, טען 50 μL כדי microplates 384 טוב.
- לרגש את הדגימה עם צבעי התורם ואת מקבל ב 550 ננומטר ולמדוד ספקטרום קרינה פלואורסצנטית מ- 570 nm 800 nm עם fluorometer.
- למדוד את ספקטרום קרינה פלואורסצנטית המדגם התורם בלבד באותה דרך.
- לרגש את צבעי המדגם-650 nm ואת ספקטרום פלורסצנטיות מדידה של 670 nm 800 ננומטר. . זה כדי למדוד את ריכוז מקבל...
- להשיג את סטיות תקן באמצעות חזרה על אותו ניסוי עם שלוש דוגמאות, מסונתז ומותאמים מטוהרים בנפרד.
- לחשב את היעילות סריג עם יחס שיטה כפי שתואר על ידי המשוואה להלן21.
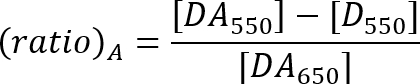
DA550: מקבל שיא עוצמת קרינה פלואורסצנטית המדגם ועם התורם מקבל ב 550 ננומטר עירור.
D550: עוצמת קרינה פלואורסצנטית פליטה מקבל טווח המדגם התורם בלבד-550 ננומטר עירור.
DA650: מקבל שיא עוצמת קרינה פלואורסצנטית המדגם ועם התורם מקבל ב 650 nm עירור.
תוצאות
המתלה אקורדיון של הדנ א 6 תוכנן על ידי 6 מדומה של ה16,oxDNA17 , התוצאות מוצגות באיור6. מן התוצאה סימולציה, הוא אישר כי המבנה המיועד נוצר ללא עיוותים של המבנה.
הדימויים TEM איור 7 הם תמ?...
Discussion
פרוטוקול זה מציג את כל התהליך של עיצוב, הדמיה, סינתזה, וניתוח של בסיסי 2D DNA אקורדיון ארון התקשורת. עיצוב ששונה והכללים סימולציה תוארו כי החוק עיצוב שונה מזו של אוריגמי DNA רגיל, בכך המדף אקורדיון DNA יש נוקלאוטידים נוספים ג'יי ג'יי אברהמס גמישות14,15. מכאן, אנו מצפי?...
Disclosures
המחברים אין לחשוף
Acknowledgements
מחקר זה מומן בחלקו על-ידי התוכנית מרכז פיתוח מחקר הכללית דרך נבחרת מחקר קרן של Korea(NRF) ממומן על ידי משרד המדע, ICT (MSIT) (2015K1A4A3047345) Nano· פיתוח חומרים טכנולוגיה תוכנית דרך לאומי מחקר קרן של קוריאה (ב- NRF) ממומן על ידי משרד המדע ICT (MSIT) (2012M3A7A9671610). המכון למחקר הנדסי-אונ' סאול מסופקים מכוני מחקר לעבודה זו. מחברים להכיר תודתי לכיוון יון טה-יאנג (מדעי הביולוגיה, אונ' סאול) בנוגע ספקטרוסקופיה זריחה על הניתוח סריג.
Materials
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
References
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -. G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127 (2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102 (2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101 (2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272 (2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486 (2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved