A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הדמית מבוססת תמונה אוטומטית של מלכודות נויטרופילים באמצעות NETQUANT
In This Article
Summary
כאן, אנו מציגים פרוטוקול להפקת נויטרופילים מלכודות (רשתות) ו-NETQUANT הפעלה, תוכנה אוטומטית לחלוטין לכמת של רשתות בתמונות immunofluorescence.
Abstract
מלכודות נויטרופילים מסחטות (רשתות) הם מבנים מיקרוביאלית כמו האינטרנט המורכב של DNA ו גרגר עושה נגזר חלבונים מיקרוביאלית. מיקרוסקופ immunofluorescence ומבוססי תמונה שיטות הקוונפיקציה להישאר כלים חשובים כדי ככמת היווצרות נויטרופילים המלכודת המבנה. עם זאת, קיימות מגבלות מפתח לשיטות המבוססות על מודלואונציה הזמינות כעת לכימות רשתות. שיטות ידניות של התמונה מבוסס הקוונפיקציה נטו הם לעתים קרובות סובייקטיבי, נוטה שגיאה מייגע עבור משתמשים, במיוחד שאינם מנוסים משתמשים. כמו כן, כיום אפשרויות תוכנה זמינות עבור כימות הן למחצה או אוטומטי או דורשים הכשרה לפני המבצע. כאן, אנו מדגימים את היישום של שיטת כימות החיסונית אוטומטית המבוססת על הטיפול כדי להעריך את היווצרות נטו בשם NETQUANT. התוכנה קלה לשימוש ויש לה ממשק משתמש גרפי ידידותי למשתמש (GUI). הוא מחשיב פרמטרים רלוונטיים ביולוגית כגון גידול בשטח פני השטח DNA: היחס החלבון סמן נטו, ואת דפורמציה גרעינית כדי להגדיר היווצרות נטו. יתר על כן, כלי זה בנוי כיישום זמין באופן חופשי, ומאפשר לכמת זיהוי תא בודד וניתוח.
Introduction
נויטרופילים הם מתווכים חיוניים של תגובות ההגנה המולדת מארח נגד מגוון רחב של פתוגנים מיקרוביאלית1. הם מבצע פונקציות מיקרוביאלית שלהם על ידי שחרור גרגירים שלהם המכיל מגוון רחב של חלבונים מיקרוביאלית2, לייצר מינים חמצן תגובתי (ROS) ו רדמה1, ודרך phagocyציטוזה3. בנוסף, בריקמן ואח '. 4 תיאור נויטרופילים מסחטות מלכודות (רשתות) כמו מנגנון הרומן שבו המלכודת נויטרופילים לחסל פתוגנים הפולשים. מאז התגלית שלהם לפני קצת יותר מעשור, הרשתותהיו מעורבים במגוון רחב של זיהומיות5,6 ולא זיהומיות7 morbidities. היווצרות נטו הוא תהליך פעיל ותוצאות שחול של דנ א כרומטין מצופה עם granule-נגזר מיקרוביאלית חלבונים8. חלק מהשינויים המרכזיים במבנה הסלולר והגרעין הקשורים למערך הרשת כוללים את אובדן המבנה הגרעיני, הגיוס של חלבונים מציטופלסמה לגרעין וגידול בקוטר הגרעיני והסלולר8,9.
רשתות כמו האינטרנט, אשר עשויים להופיע כמבנים לפזר מעט יותר גדול מתא או כמו מבנים מספר פעמים גדול יותר מאשר נויטרופילים בודד נחשבים אינדיקטורים שלnetosis 5,10. באמצעות מיקרוסקופ ניאון, רשתות ניתן לזהות על ידי בודק דנ א עם בדיקה פלורסנט כגון 4 ', 6-diamidino-2-פניינילידול (DAPI) ועל ידי immunofluorescence כתמים נגד חלבונים נטו מאוגד כגון נויטרופילים elastase. הכמת של אזורים חופפים של כתמים עבור החלבונים ו-DNA נטו מאוגד קובע את השטח הכולל תחת רשתות בתמונה11.
מספר אפשרויות ניתוח תמונה זמינות כדי לבצע כימות קרינה פלואורסצנטית המבוססת על רשתות11,12. אך אפשרויות תוכנה אלה מציגות מגבלות שאינן ידידותיות למשתמש ו/או אוטומטיות לחלוטין. במאמר זה, אנו מדגימים את הפעולה של NETQUANT13, יישום זמין באופן חופשי שיכול לבצע משוחדת לחלוטין אוטומטית immunofluorescence מיקרוסקופ תמונה מבוססת נטו המבוסס. האפליקציה יש ממשק גרפי ידידותי למשתמש (GUI) והוא יכול לבצע ניתוח תא בודד. התוכנה לכמת את NETosis בתמונה על ידי זיהוי שינויים מורפולוגיים באזור של DNA-NET-מאוגד סמן, כרומטין הקשורות דפורמציה של הגרעין ולהגדיל את ה-DNA: היחס הנקי חלבון. ביחד, הקריטריונים של NET definition מרובים מאפשרים לכמת את הנטו המחמירים על פני מספר ערכות נתונים בצורה לא משוחדת.
Protocol
ועדת האתיקה של אוניברסיטת לונד אישרה את אוסף דם הורידים ממתנדבים בריאים בהתאם להכרזת הלסינקי (2013/728). כל המתנדבים סיפקו את הסכמת הידע שלהם בכתב.
1. בידוד דם היקפיים נויטרופילים באמצעות צפיפות-הדרגתי צנטריפוגה
- לאסוף דם הורידים האנושיים בצינורות המכילים הפארין ולאפשר לצינורות להגיע לטמפרטורת החדר.
הערה: לפחות 16 מ ל של דם מתורם בריא נדרש להניב כדורית גדולה מספיק. - מערבבים את הדם בנפח אחד של 2% תוספי ב מלוחים (0.9% הנאל) ולאפשר משקע בטמפרטורת החדר עבור 30 דקות ב שפופרת סטרילי מ50 mL של צנטריפוגה.
- מנושף את supernatant לתוך שפופרת סטרילי 50 mL וצנטריפוגה ב 200 x g עבור 10 דקות ב 4 ° c.
- משלב זה ואילך נמשיך בבידוד ב -4 ° צ' או בקרח.
- להשעות את הגלולה ב 5 מ ל של קרח קר מלוחים ושכבה על גבי 5 מ ל של בידוד לוקיציט מעבר הצבע (9.1% נתרן diatrizoate עם 5.7% dextran, w/v) בשפופרת סטרילי 15 mL סטרילית.
- צנטריפוגה עבור 30 דקות ב 400 x g ב 4 ° c.
- . ולהשליך את הסופרנטאנט
- Lyse כדוריות הדם האדומות על ידי השעיית הגלולה ב 3 מ ל של מים קרים קרח עבור 30 s. מייד להוסיף 1 מ ל 3.6% הנאגל ולאחר מכן למלא עם 10 מ ל של תמיסת מלח קר הקרח.
- צנטריפוגה את ההשעיה התא עבור 10 דקות ב 350 x g.
- להסיר את הסופרנטאנט, לאסוף את הגלולה התא ולהשעות אותו מחדש 1 מ ל של תמיסת מלח. הגדר הצידה 10 μL בצינור מיקרוצנטריפוגה להערכת מספר התאים והכדאיות באמצעות טריטופה כחול בחדר בורקר.
- הוסף 10 μL של השעיית התא ל 90 μL של 0.4% הפתרון הכחול. קח 10 μL של השעיית התא בחדר בורקר. לספור את התאים 4 ריבועים המאוגדים על ידי 3 שורות בכל פינה של החדר. תאים המופיעים כחול כהה לקליטת הצבע הם לא קיימא, להוציא אותם ממספר התא הכולל.
- בטא את מספר הטלפון כתאים/mL כפי שהוגדר על-ידי המשוואה שלהלן.
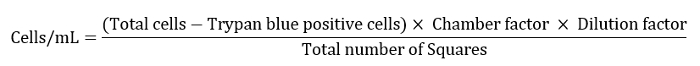
הערה: להלן מקדם החדר היה 10,000, מקדם דילול היה 10 ומספר הריבועים הכולל היה 4. - דלל את השעיית התאים הנותרים ל-10 מ ל לשלב הכביסה הסופי.
- צנטריפוגה עבור 5 דקות ב 200 x g.
- להשעות את נויטרופילים ב-RPMI-1640 עם 2 mg/mL חום-inactivated פעלה האדם סרום אלבומין (HSA) בריכוז של 5 x 105 תאים/mL.
2. הכנת כיסוי וגירוי של נויטרופילים
- מקום אחד coverslip (10 מ"מ, #1) בכל באר של 12-היטב צלחת מעיל שמיכות על ידי הוספת 200 μL של 0.01% פולי-L-ליזין פתרון ולהשאיר אותו ב 37 ° c בלילה.
- שטוף כיסוי עם 300 μL פוספט מאגר תמיסת מלח (PBS) פעם ולהשאיר יבש.
- הוסף 400 μL של 5 x 105 נויטרופילים/mL לכל טוב ומארג בטמפרטורת החדר עבור 15 דקות.
- להעביר את הצלחת המכילה נויטרופילים לאינקובטור ב 37 ° צ' עם 5% CO2 עבור 15 דקות.
- . הסר את הסופרנטאנט הוסף 400 μL של prewarmed RPMI-1640 בינוני עם 2 מ"ג/mL HSA לפקדים. הוסף 300 μL טרום מחומם RPMI עם 20 ננומטר בפורובול 12-myristate 13-אצטט (PMA) עבור גירוי.
- לעורר נויטרופילים עבור 150 דקות ב 37 ° צ' עם 5% CO2.
3. ויזואליזציה של רשתות
- הסר את supernatant ולשטוף את הדגימות 2x עם 200 μL של PBS.
- תקן דגימות על-ידי הוספת 200 μL של 4% פאראפורמלדהיד (בתחתית) ב-PBS במשך 20 דקות ב 37 ° c.
שימו לב: הכדורגלן רעיל ויש לטפל בו בזהירות. - שטוף דגימות 3x עם 200 μL של PBS.
- החדירות הדגימות על ידי הוספת 50 μL של 0.5% טריטון X-100 עבור 30 s.
- שטוף את דגימות 3x עם 200 μL של PBS.
- חסום את הדגימות באמצעות סרום עז בגובה 5% ב-PBS בשעה 37 ° c.
- הוסף 300 μL של הארנב הראשי אנטי האדם נויטרופילים elastase בפתרון חסימת בדילול של 1:500 עבור 90 דקות ב 37 ° c.
- שטוף את דגימות 3x עם 300 μL של PBS.
- הוסף 300 μL של עז משנית אנטי ארנבת נוגדן פלורסנט בדילול של 1:1000 עבור 90 דקות ב 37 ° c.
- לשטוף את שמיכות 3x עם 300 μL של PBS.
- הסר את שמיכות מן הבארות ולטעון את coverslips עם 10 μL הרכבה בינונית המכילה DAPI. אחסן לילה בטמפרטורת החדר בחשכה כדי לייבש את הדגימות.
הערה: הצביעת של ה-DNA עם DAPI הוא בהחלט צעד קריטי בשיטה. המשתמשים יכולים גם לפתור בעיות על ידי הוספת פתרון DAPI האקסוסוגני בטווח ריכוז סופי של 0.1 \ u 20120.5 μg/mL עבור 2 \ u20123 min, ואחריו 3 שטיפת שלבים עם 300 μL PBS. - לרכוש תמונות עם מיקרוסקופ לשדה רחב מאוד באמצעות המטרה 20X.
4. ניתוח וקוונפיקציה של רשתות באמצעות NETQUANT
הערה: ניתן להוריד את NETQUANT על ידי לחיצה על קובץ ההתקנה שנמצא על הארכיון של Zenodo Github או באתר המעבדה נורדנחש (https://nordlab.med.lu.se/?page_id=34).
- ייבוא ערכות נתונים לניתוח, שמות ערוצים והמרת תמונות
- פתח את לשונית ההתקנה ב-NETQUANT.
- בחרו בתיקיית המקור לניתוח בלחיצה על האפשרות ' קבל נתיב ' בתפריט המקור ובחרו בתיקייה המכילה את רצפי התמונות לניתוח.
- לחצו על האפשרות ' קבל נתיב ' בתפריט היעד ובחרו בתיקייה לשמירת הנתונים שאחרי ניתוח התמונה.
- שם הערוצים כך "ערוץ ה-DNA" מתאים עם כתמים DNA (g, DNA או dapi) ו-"NET-channel" מתארת את מכתים החלבון המאוגד (למשל, net, נויטרופילים elastase) בתמונות. עבור התפקוד החלק של התוכנה, (מומלץ) שם התיקיה המכילה את קבצי תמונת הבקרה "שליטה".
הערה: ערוץ NET מתייחס לסמן החלבון המאוגד לרשת בלבד. - האכילו את המטא-נתונים של התמונה בתוכנה על-ידי לחיצה על לחצן ' טען נתוני תמונה ' בתפריט המשנה של מידע התמונה .
- בחר את סדר הערוץ הנכון הכלול בתמונות בתפריט המשנה של הזמנת הערוץ . אפשרות זו נכללה במצב של כשל כדי למנוע אי התאמות בשוגג.
- לרכוש מאפייני תמונה ראשית מהנתונים הגולמיים ולהמיר את התמונות על-ידי לחיצה על לחצן הכן נתונים. התמונות שהומרו מופיעות בתפריט המשנה סוג לדוגמה . לחץ על תפריט סוג לדוגמה להצגה ובחירה של כל ערכות הנתונים הנרכשים לניתוח.
- בחרו תמונה מתפריט המשנה ' סוג דגימה ' ולחצו על הלחצן ' הצג נתוני תמונה ' כדי להציג את התמונות שפוצלו לתוך הערוץ ה-DNA ו-NET בהתאמה.
- פילוח של תאים בערוץ ה-DNA ובערוץ NET
- בחר את שיטת הפילוח על-ידי לחיצה על תפריט המשנה של השיטה הן בערוץ ה-DNA והן בערוץ NET.
הערה: שיטת ברירת המחדל של פילוח מוגדרת כאדפטיבית והיא ההגדרה המומלצת. אפשרויות אחרות זמינות גם כולל כללי, קצה ו-Chan-Vese. אפשרות לפרשת-החוצה כלולה גם כדי להבדיל בין תאים או רשתות ממוקמות באופן הדוק. - הזן את הכרטיסייה פילוח כדי להתאים את תאי הבקרה תחילה בשני הערוצים על-ידי לחיצה על האפשרות דגימות פקד קטע .
- בחרו PMA מתפריט המשנה לדוגמה ולחצו על אפשרות האצווה (מומלץ) להתחלת פילוח של כל התמונות הכלולות בערכת הנתונים. בחר את התמונות בתפריט סוג לדוגמה ולחץ על כפתור נתוני התמונה כדי להמחיש ולאמת את מסיכות התמונה הבינאריות (מסיכת DNA ו-NET mask) שנוצרו לאחר פילוח.
- בחר את שיטת הפילוח על-ידי לחיצה על תפריט המשנה של השיטה הן בערוץ ה-DNA והן בערוץ NET.
- ניתוח תא בודד של מאפיינים הניתנים לזיהוי
- היכנס ללשונית הניתוח ונתח את דגימות הבקרה על-ידי לחיצה על לחצן קביעת הסף .
- שנה את סוג המדגם ל-PMA ולחץ על הלחצן ' קבל מאפייני תא ' כדי להשלים את הניתוח של דגימות מוסטימולציה.
- בחרו תמונה מתפריט המשנה ' סוג דגימה ' ולחצו על הלחצן ' הצג נתוני תמונה ' כדי להציג את כיסוי השכבה ואת מספר התאים והתאים היוצרים את NET בתמונה.
- השוואה בין מאפייני תא לזיהוי תאים היוצרים את הרשת
- בחרו דגימה מתפריט המשנה ' סוג דגימה ' ולחצו על הלחצן ' נתח רשתות ' כדי להשלים את הניתוח. תמונות בודדות ניתן לבחור מסוג לדוגמה תת תפריט לניתוח או את כל האצווה של תמונות ניתן לנתח על ידי בחירת אפשרות אצווה (מומלץ).
- התאם את קריטריוני NET באופן ידני כדי להניב תוצאות אופטימליות עבור דוגמה נתונה. השוו רשתות מזוהות עם התמונות המקוריות כדי להעריך את איכות הזיהוי.
הערה: לאחר מכן ניתן להשתמש בקריטריוני NET בכל התמונות בערכת הנתונים. שינויים בקריטריוני האינטרנט יוחלו בו בכל דגימות הבקרה. פעולה זו מגבילה את האפשרות של כל הבדלים פוטנציאליים שעלולים לנבוע עקב התאמה מרבית של פרמטרי NET. ההגדרות בקריטריוני NET יכולות להיות מותאמות בהתאם לדרישות המשתמש. היחסים בין שיעורי גילוי שווא ו-NETQUANT כבר נחקרו בעבר13. טווחים טיפוסיים לגידול באזור הם 2 \ u20124, מעגליות להיות 0.7 \ u 20120.9 ו-DNA/היחס נטו להיות 0.6 – 2.0. - בדוק את סיכום הנתונים בתפריט המשנה של נתוני התא שבו מוצגים מספר התמונות, ספירת תאים לכל תמונה ואחוז של רשתות לכל תמונה.
הערה: האחוז הכולל של הרשתות בערכת הנתונים כולה מוצג על-ידי "מד הרשת". ספירת התמונות הכוללת, ספירת התאים, אחוז הרשתות במדגם (רשתות%) והרשתות% במדגם הפקדים מוצגות בטבלת סטטיסטיקת התקציר מתחת למד הרשת. אנו ממליצים שנתוני הבקרה ידווחו לצד הנתונים שהתקבלו מדגימות מוסטימולציה.
- תפוקות תוצאות
- הזן את כרטיסיית הפלט כדי לבחור ולהציג פלטי תוצאות.
- לחקור ולהשוות את פלטי נתונים שונים שנוצרו ניתוח של השליטה PMA על ידי בחירת הטופס של הפלט ולחיצה על לחצן תוצאות פלט .
הערה: כל הנתונים שהופקו לאחר ניתוח עבור הפקדים והאיסופים נשמרים בתיקיית הניתוח כפי שנבחרה בתפריט המשנה של היעד. נתונים נשמרים בתבניות. csv או. pdf. - הפעל את קובץ השיטה כדי לקבל את הגירסה של התוכנה והקריטריונים הנטו המשמשים לניתוח (להיכלל בסעיף שיטות למטרות פרסום).
- לחץ על טבלת נתוני התוצאות כדי להמחיש את נקודות הנתונים הבודדות במדגם נתון.
- דמיינו את התפלגות השטח נטו ו-DNA: יחס נטו בדגימות. הקו האדום מציין את ערך הסף בגרפים.
- קבע את אזור NET לעומת צורת ה-DNA על ידי לחיצה על קובץ ההפצה Bivariate .
- טעינת הניתוח הקודם והאצווה כל השלבים
- טען הגדרות ניתוח שהצליחו בעבר ב-NETQUANT באמצעות לחצן ' טען ניתוח קודם '.
- השתמש בלחצן האצווה כל השלבים הכלולים בתפריט ההתקנה כדי להפעיל את שלבים 5 \ u201212 (איור 1, איור 2, איור 3, איור 4, איור 5) ישירות לקבלת הפלט הסופי.
תוצאות
5 x 105 נויטרופילים/mL הושמו על כיסוי ממוקם בצלחת 12-באר ומגורה עם 20 ננומטר או או שמאל לא מגורה עבור 150 דקות. הדגימות היו לאחר מכן מוכתם באמצעות הארנב הראשי אנטי האדם נוגדנים נויטרופילים elastase, עז משנית נגד הארנב fluorophore מצופף נוגדנים DAPI-צבע מתויג פלואורוסקופים כי כתמי DNA (לר...
Discussion
היווצרות נטו הוא התוספת העדכנית יחסית של נויטרופילים מגוונת הארמנטריום4 והיה גל מורגש של עניין ללמוד את המשמעות של רשתות במגוון רחב של תחומי מחקר5,7,14,15. הקניית תמונות בעזרת מיקרוסקופ Immunofluorescence והשימוש העו?...
Disclosures
TM ו-PN יש פטנט בהמתנה הקשורים לאלגוריתמים המשמשים NETQUANT.
Acknowledgements
העבודה ממומנת על ידי קרן Crafoord (TM ו-PN), מענק שוודי מחקר המענק (PN, TM), המועצה למחקר שוודי (PN) ו Groschinsky קרן (TM, PN).
Materials
| Name | Company | Catalog Number | Comments |
| BD Vacutainer Heparinised plastic tubes | BD Biosciences | 367885 | |
| Lymphoprep | Axis-Shield | 114547 | |
| RPMI-1640 with L-Glutamine | Gibco | 11835-030 | |
| 50mL conical flasks | Sarstedt | 62.547.004 | |
| 15mL conical flasks | Sarstedt | 62.554.002 | |
| 12-well Tissue culture plates | Falcon | 10626491 | |
| #1 Coverslips 10mm | Menzel Glaser | CS10100 | |
| Glass slides | Menzel Glaser | 631-0098 | |
| Primary anti-human elastase | DAKO | DAKO rabbit 1373, contract immunization | |
| Secondary fluorophore conjugated goat anti-rabbit | Life technologies | A-11072, A-11070 | |
| PROLONG-Gold Antifade reagent with DAPI | Life technologies | P36930 | Mounting medium |
| Goat serum | Sigma-Aldrich | G9023 | |
| Phorbol 12-myristate 13-acetate (PMA) | Sigma-Aldrich | 79346 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Nikon Ti-E Epifluorescence microscope | Nikon | ||
| CCD camera | Andor Zyla | ||
| Plan Apochromat 20x, 40x objectives | Nikon | ||
| Windows 10 | Microsoft | Operating system | |
| macOS Sierra 10.12 | Apple | Operating system | |
| MATLAB | Mathworks |
References
- Nauseef, W. M., Borregaard, N. Neutrophils at work. Nature Immunology. 15 (7), 602-611 (2014).
- Borregaard, N., Cowland, J. B. Granules of the human neutrophilic polymorphonuclear leukocyte. Blood. 89 (10), 3503-3521 (1997).
- Nordenfelt, P., Tapper, H. Phagosome dynamics during phagocytosis by neutrophils. Journal of Leukocyte Biology. 90 (2), 271-284 (2011).
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- Sorensen, O. E., Borregaard, N. Neutrophil extracellular traps - the dark side of neutrophils. Journal of Clinical Investigation. 126 (5), 1612-1620 (2016).
- Yipp, B. G., Kubes, P. NETosis: how vital is it?. Blood. 122 (16), 2784-2794 (2013).
- Jorch, S. K., Kubes, P. An emerging role for neutrophil extracellular traps in noninfectious disease. Nature Medicine. 23 (3), 279-287 (2017).
- Fuchs, T. A., et al. Novel cell death program leads to neutrophil extracellular traps. J Cell Biol. 176 (2), 231-241 (2007).
- Mohanty, T., et al. A novel mechanism for NETosis provides antimicrobial defense at the oral mucosa. Blood. 126 (18), 2128-2137 (2015).
- Hakkim, A., et al. Activation of the Raf-MEK-ERK pathway is required for neutrophil extracellular trap formation. Nature Chemical Biology. 7 (2), 75-77 (2011).
- Brinkmann, V., Goosmann, C., Kuhn, L. I., Zychlinsky, A. Automatic quantification of in vitro NET formation. Frontiers in Immunology. 3, 413 (2012).
- Coelho, L. P., et al. Automatic determination of NET (neutrophil extracellular traps) coverage in fluorescent microscopy images. Bioinformatics. 31 (14), 2364-2370 (2015).
- Mohanty, T., Sorensen, O. E., Nordenfelt, P. NETQUANT: Automated Quantification of Neutrophil Extracellular Traps. Frontiers in Immunology. 8, 1999 (2017).
- Kaplan, M. J., Radic, M. Neutrophil extracellular traps: double-edged swords of innate immunity. Journal of Immunology. 189 (6), 2689-2695 (2012).
- Cedervall, J., Olsson, A. K. Immunity Gone Astray - NETs in Cancer. Trends in Cancer. 2 (11), 633-634 (2016).
- Ginley, B. G., et al. Computational detection and quantification of human and mouse neutrophil extracellular traps in flow cytometry and confocal microscopy. Scientific Reports. 7 (1), 17755 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved