Method Article
באמצעות מקור פתוח MALDI תוף-MS IDBac הניתוח של חלבון מיקרוביאלית ונתונים מיוחדים מטבוליט
In This Article
Summary
IDBac הוא מקור פתוח המסה ספקטרומוטוריקה מבוססי צינור ביואינפורמטיקה המשלבת נתונים מחלבונים שלמים, ספקטרום מטבוליזם מיוחדים, שנאסף על החומר הנייד מגורד ממושבות חיידקי. צינור מאפשר לחוקרים לארגן במהירות מאות אלפי מושבות חיידקי לתוך קבוצות מטקמיים ועוד להבדיל אותם מבוסס על ייצור מטבוליזם המקצועית.
Abstract
על מנת להמחיש את היחסים בין בקטריאלי תולדות והפקה מטבוליזם המקצועית של מושבות חיידקי גדל על מזינים אגר, פיתחנו idbac-בעלות נמוכה ותפוקה גבוהה באמצעות מטריצה לייזר בסיוע בלייזר/יינון ביומוטמטריה (MALDI-תוף MS)-הצינור הגדול של הטיסה. תוכנת IDBac מיועד שאינם מומחים, הוא זמין באופן חופשי, ומסוגל לנתח כמה אלפי מושבות חיידקי. כאן, אנו מציגים הליכים להכנת מושבות חיידקי לניתוח MS MALDI-תוף, הפעלת כלי MS, עיבוד נתונים והדמיה ב-IDBac. בפרט, אנו מורים למשתמשים כיצד לאשכול חיידקים לתוך העצים בהתבסס על טביעות אצבעות חלבון MS ו אינטראקטיבי ליצור רשתות האגודה מטבולייט (מאנס) מתוך נתונים מיוחדים מטבוליזם.
Introduction
מחסום מרכזי לחוקרים הלומדים פונקציה חיידקית היא היכולת להעריך במהירות ובו בו את הזהות הטקקיות של מיקרואורגניזם ויכולתו לייצר מטבוליטים מיוחדים. זה מנע התקדמות משמעותית בהבנת הקשר בין הקימוט החיידקי וייצור מטבוליזם מיוחדים ברוב החיידקים מבודדים מהסביבה. למרות שיטות מבוססות MS המשתמשות בטביעות אצבע של חלבונים לקבוצה וזיהוי חיידקים מתוארים היטב1,2,3,4, מחקרים אלה בדרך כלל בוצעו על קבוצות קטנות של בודד, באופן ספציפי למין. חשוב מכך, מידע על ייצור מטבוליזם מיוחדים, הנהג העיקרי של תפקוד חיידקים בסביבה, נשאר משולב במחקרים אלה. סילבה ואח '5 לאחרונה סיפקה היסטוריה מקיפה המפרט את השימוש התחתון של MALDI-תוף MS לנתח מטבוליטים מיוחדים ומחסור של תוכנה כדי להקל על צווארי בקבוק הנוכחי בביואינפורמטיקה. על מנת לטפל בחסרונות אלה, יצרנו IDBac, צינור ביואינפורמטיקה המשלב הן ליניארי ו הרפלקסים של מצבי MALDI-תוף MS6. זה מאפשר למשתמשים להמחיש במהירות ולהבדיל חיידקי בודד מבוסס על החלבון הן מטבוליזם וטביעות אצבעות MS מיוחדים, בהתאמה.
IDBac הוא חסכוני, תפוקה גבוהה, ומיועד המשתמש שכבה. היא זמינה באופן חופשי (chasemc.github.io/IDBac), ומחייבת גישה לספקטרומטר מסה של MALDI-תוף (במצב של הרפלקסים הדרושים לניתוח מטבוליזם). הכנה לדוגמא מסתמכת על השיטה הפשוטה "המורחבת ישירה" שיטה7,8 ונתונים נאספים עם ברציפות ליניארי ו-הרפלקסים של רכישות במקום יחיד maldi-יעד. עם IDBac, אפשר לנתח את הקימוט הפוטוטיבית וייצור מטבוליזם מיוחדות של מאות מושבות בפחות מארבע שעות, כולל הכנה לדוגמא, רכישת נתונים, ויזואליזציה של נתונים. זה מציג זמן משמעותי ויתרון עלות על שיטות מסורתיות של זיהוי חיידקים (כגון רצף גנים), וניתוח פלט מטבולית (כרומטוגרפיה נוזלית המסה ספקטרומטריה [LCMS] ושיטות כרומוגרפיות דומות).
באמצעות נתונים שהושגו בניתוח מצב ליניארי, IDBac משתמש באשכולות הירארכיים כדי לייצג את המצב החדש של ספקטרום החלבונים. כיוון שהספקטרום מייצג בעיקר חלבונים ריבוזומניים, הם מספקים ייצוג של הגיוון הפילוגנטי הקיים במדגם. בנוסף, IDBac משלבת רפלקסים במצב נתונים כדי להציג טביעות אצבעות מיוחדות לייט מטבוליזם כמו רשתות האגודה מטבוליט (מאנס). מאמינים הם רשתות דו-צדדי המאפשרים הדמיה קלה של ייצור המטמיטבוליט משותף ייחודי בין בודד בקטריאלי. הפלטפורמה IDBac מאפשר לחוקרים לנתח הן חלבונים ונתוני מטבוליזם מיוחדים בטנדם, אבל גם בנפרד, אם רק אחד מסוג הנתונים הוא רכשה. חשוב מכך, IDBac מעבד נתונים גולמיים מ Bruker ו ש'יאמן מכשירים, כמו גם txt, tab, csv, mzXML, ו Mzxml. פעולה זו מבטלת את הצורך בהמרה ידנית ובעיצוב של ערכות נתונים, ומפחיתה באופן משמעותי את הסיכון של שגיאת משתמש או משנה של נתוני MS.
Protocol
1. הכנת מטריצת MALDI
- להכין 10 מ"ג/mL MALDI-כיתה, ו/או recrystallized-cyano-4-hydroxycinnamic חומצה (CHCA) ב-MS-כיתה ממיסים: 50% acetonitrile (ACN), 47.5% מים (H2O), 2.5% trifluoroacetic חומצה (tfa). דוגמה: 100 μL פתרון = 50 μL ACN + 47.5 μL H2O + 2.5 ΜL tfa + 1 מ"ג chca
- הכינו לפחות 1 μL של פתרון מטריצה לנקודה לוחית MALDI ומערבולת או sonicate עד בפתרון (כ 5 דקות sonication או לא מוצקים גלויים).
התראה: TFA היא חומצה חזקה שיש לטפל בה בתוך מעטפת כימית בזמן שהוא חובש ציוד הגנה אישי נאות, שכן הוא יכול לפגוע בעור, בעיניים ובאיירווייז במגע או באינהלציה.
הערה: chca הוא דייב ורגיש בהיר ויש לאחסן בבקבוקונים ענבר ב desiccator. יש הרבה אפשרויות מטריצה MALDI זמין. CHCA הוא הנפוץ ביותר עבור פרופיל חלבון של חיידקים, אבל עובד גם עבור ניתוח מטבוליזם המקצועית. הבחירה של מטריקס תלויה בצורכי משתמש/ניסוי בודדים.
- הכינו לפחות 1 μL של פתרון מטריצה לנקודה לוחית MALDI ומערבולת או sonicate עד בפתרון (כ 5 דקות sonication או לא מוצקים גלויים).
2. הכנת לוחיות היעד של MALDI
הערה: ראו שאואר et al.7, לפרטים נוספים.
- לשטוף את הצלחת MALDI עם מתנול (כיתה או גבוהה) וניגוב יבש עם מגבונים נייר רכים. אין להשתמש במברשות משפשפים בעת ניקוי לוחיות היעד, משום שפעולה זו עלולה לפגוע לצמיתות במשטח של לוחית היעד.
- הקצאת חלבון ומטבוליט מיוחדים מטבוליזם כתמים. ארגן את כתמי הכיול באופן שווה לאורך האוכלוסיה לדוגמה, כדי להתחשב בחריגות מMALDI-פלייט ובשעת סחף מכשירים לאורך זמן. הקצה מספר מתאים של נקודות מדיה/מטריצה ריקות עבור המחקר; כתמים אלה יכילו רק מדיה ומטריצה, או רק מטריצה.
- באמצעות קיסם סטרילי, להעביר חלק קטן של מושבה חיידקית למקום המתאים על צלחת MALDI. לפזר את המושבה החיידקית. באופן שווה במקום המקום אמור להיראות שטוח ככל האפשר.
הערה: יהיה קל יותר לשטח מושבות חיידקית שאינן מוזהות/אמורפיות יותר. עבור מושבות נוקשות/מוצקות יותר, הימנע מעזיבת אשכולות גלויים של מסת התאים במקום MALDI (איור 1).
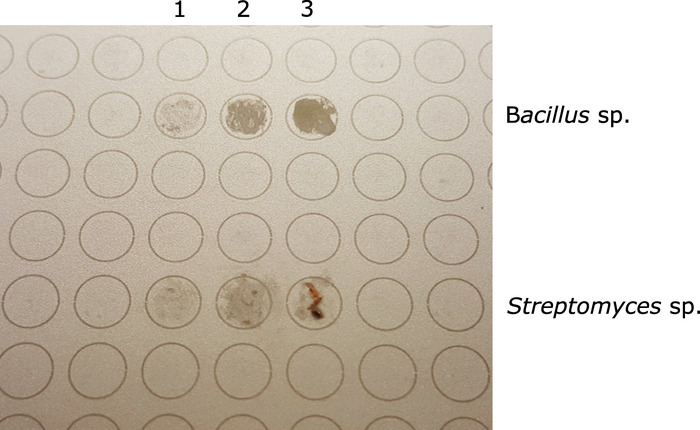
איור 1: מmaldi לוחית היעד מראה שני מבודד שונים לפני הוספת חומצה פורמית ו maldi מטריקס (העליון 3 מקומות- מקרתגו sp.; התחתון 3 מקומות- Streptomyces sp.). עבור שניהם, עמודה 3 מייצגת מדגם עודף; עמודה 2 מייצגת את כמות המדגם המתאימה; עמודה 1 מייצגת מדגם לא מספיק עבור ניתוח maldi. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- הכינו בקרת מטריצה/מדיה באמצעות קיסם שיניים סטרילי להעברת כמות מינימלית של אגר/מדיה אל הספוט (ים) המתאימים בלוח MALDI.
- שכבת-על 1 μl של 70% מהספקטרומטר המסה של חומצה פורמית על כל נקודה לדוגמה, כולל כתמי בקרת מטריקס. אפשר חומצה לאוויר יבש לחלוטין בתוך מייבש כימי (כ 5 דקות).
התראה: חומצה פורמית היא כימיה קאוסטית, ויש לטפל בה בתוך מנדפים כימיים. זה יכול לפגוע בנתיבי. הנשימה אם נשאף - הוסף 1 μL של פתרון המטריצה המוכנה MALDI לכל נקודה לדוגמה, כמו גם לנקודות בקרת מטריצה/מדיה. אפשר פתרון מטריצה לאוויר יבש לחלוטין (כ 5 דקות).
הערה: ניתן לאחסן את הצלחת בdesiccator, בחשיכה, עד לניתוח ספקטרומטר מסה של MALDI-תוף. זמני האחסון המותרים עשויים להשתנות בהתאם ליציבות המדגם. - הוסף 0.5 – 1.0 μL מכייל את כתמי הכיול שהוקצו ואחריו פתרון מטריצה 1 μL MALDI. פיפטה את הפתרון שנוצר למעלה ולמטה כדי לערבב. אפשר לכל המקומות להתייבש באוויר לפני המבוא לספקטרומטר המסה של MALDI-תוף.
הערה: חלבון ומטבוליזם מיוחדים כאשר יש להוסיף בתוך 30 דקות של ניתוח MALDI, כמו שניהם רגישים השפלה.
3. רכישת נתונים
הערה: הפרמטרים הכלליים לרכישת נתונים מפורטים בטבלה 1.
| פרמטר | חלבון | מטבוליט מיוחדים |
| התחלה המונית (Da) | 1920 | 60 |
| קצה המונים (Da) | 21000 | 2700 |
| הטיה המונית (Da) | 1900 | 50 |
| יריות | 500 | 1000 |
| תדירות (Hz) | 2000 | 2000 |
| גודל לייזר | גדול | בינוני |
| MaxStdDev (ppm) | 300 | 30 |
. שולחן 1
- בעקבות פרוטוקולים ספציפיים למכשיר בשימוש, להגדיר את שני החלבון ואת המטכיול לייט מיוחדים.
- בדוק כמה מקומות יעד נפרדים כדי לקבוע את עוצמת הלייזר האופטימלי ולהשיג גלאי להשתמש בעת רכישת ספקטרום (זה יהיה שונה היום ובאמצעות מכשיר).
הערה: איור 2A ואיור 3a הצג ספקטרום אופטימלי, בעוד איור 2a ואיור 3a הם דוגמאות של ספקטרום באיכות נמוכה.
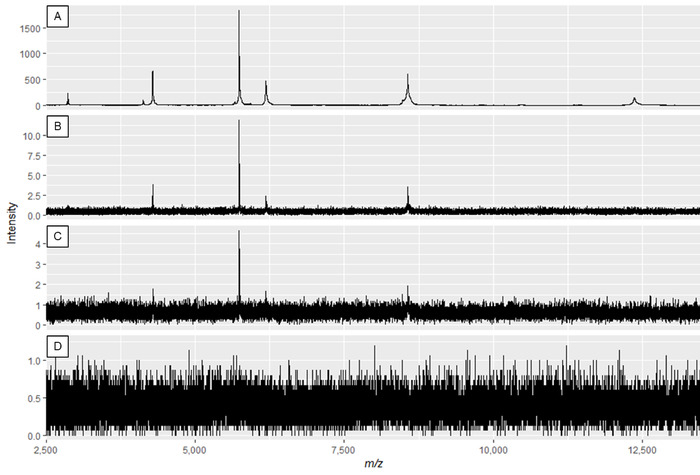
איור 2: דוגמה ספקטרום החלבון מציג את ההשפעה של שינוי כוח לייזר ולהשיג גלאי. האיכות של ספקטרה היא הטובה ביותר בלוח A, ופוחתת עד מספיק איכות ספקטרום בחלוניות C ו- D. בעוד הספקטרום בלוח B עשוי לגרום לפסגות שמיש, פאנל A מציג נתונים אופטימליים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
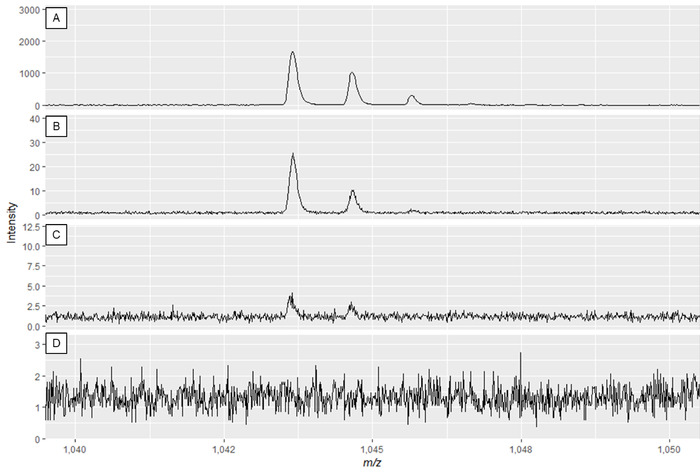
איור 3: דוגמה מיוחדים ספקטרום מטבוליזם להציג את האפקט של שינוי כוח לייזר ולצבור גלאי. האיכות של ספקטרה היא הטובה ביותר בלוח A ופוחתת עד מחסור באיכות ספקטרום בחלוניות C ו- D. בעוד הספקטרום בלוח B עשוי לגרום לפסגות שמיש, פאנל A מציג נתונים אופטימליים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- לרכוש ספקטרה, שמירת ספקטרום החלבון לתוך תיקיה אחת המקצועית ספקטרום מטבוליזם לתוך תיקיה שנייה, נפרדת.
4. ניקוי לוחית היעד של MALDI (מותאם משאואר ואח '7)
- הסר את לוחית היעד MALDI ממחזיק שלה ולשטוף עם אצטון.
- לשטוף עם סבון נוזלי לא שוחקים כדי להסיר מעקב חלבונים ושומנים, ומגבונים נייר רך/מברשת שיניים רכה.
- לשטוף עם מים דה מייהים עבור כ 2 דקות כדי להסיר לחלוטין סבון.
- הSonicate את לוחית היעד במים (כיתה) במשך 5 דקות.
- שטפו את לוחית היעד במים (כיתה או גבוהה יותר).
- שטפו את לוחית היעד עם מתנול (כיתה).
5. התקנת תוכנת IDBac
- הורד את תוכנת IDBac.
הערה: גיבויים קבועים זמינים גם להורדה (עיין בטבלת החומרים). - לחץ פעמיים על ההורדה "Install_IDBac. exe" כדי ליזום את תוכנית ההתקנה ובצע את ההוראות המופיעות על המסך.
6. החל מהנתונים הגולמיים
הערה: הסברים מפורטים והוראות של כל שלב עיבוד נתונים מוטבעים בתוך IDBac, אולם הניתוחים העיקריים וכניסות אינטראקטיביות מתוארים להלן.
- לחץ פעמיים על קיצור הדרך השולחני IDBac להשיק IDBac. IDBac ייפתח בכרטיסיה מבוא כברירת מחדל.
- השתמש בלחצן ' בדוק עדכונים ' כדי לוודא שהגירסה העדכנית ביותר של idbac נמצאת בשימוש (מחייבת גישה לאינטרנט). אם קיימת גירסה חדשה יותר, IDBac להוריד ולהתקין באופן אוטומטי את העדכון, לאחר IDBac יבקש להפעיל מחדש.
- לחץ על הכרטיסיה החל מהנתונים הגולמיים ובחר מהתפריט את סוג הנתונים שישמשו עם idbac; להמשיך בעקבות ההוראות ב-app.
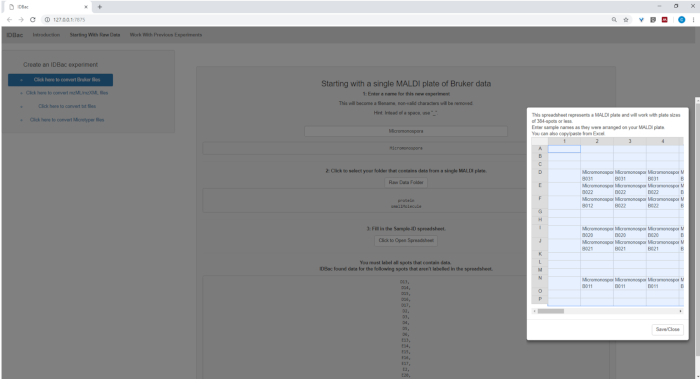
- בעת הגדרת ההמרה והעיבוד של קבצי נתונים, הזן שם תיאורי לניסוי שממנו תתבקש (ראה איור 4). ניסויים מאוחר יותר יוצגו בסדר אלפביתי, ולכן אסטרטגיה מועילה היא להתחיל שמות הניסוי עם התכונה קבוצה (למשל, "באקלוס-trials_experiment-1"; "באקלוס-trials_experiment-2").
איור 4: IDBac המרת נתונים וצעד טרום עיבוד. הפונקציה IDBac ממירה ספקטרום גולמי לתבנית mzML הפתוחה ומאחסנת mzML, רשימות שיא ומידע לדוגמה במסד נתונים עבור כל ניסוי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
7. עבודה עם ניסויים קודמים
- לאחר המרת קבצים ועיבוד אותם עם IDBac, או בכל עת שהוא רוצה לנתח מחדש ניסוי, לנווט לעבודה עם הניסויים הקודמים דף ולבחור ניסוי לעבוד עם (איור 5).
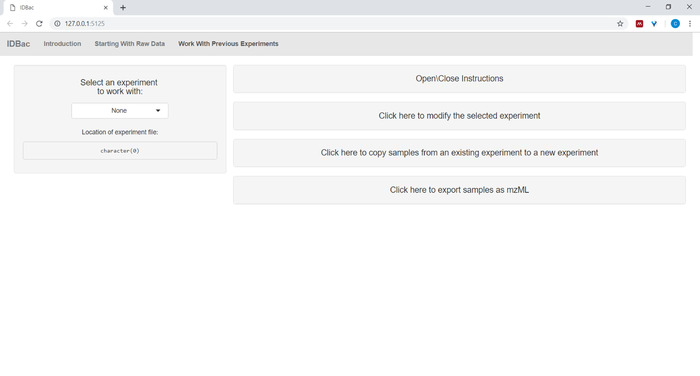
איור 5: עמוד "עבודה עם ניסויים קודמים". השתמש בעמוד ' עבודה עם ניסויים קודמים ' של IDBac כדי לבחור ניסוי לניתוח או לשינוי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- אופציונלי הוסף מידע על דגימות באמצעות התפריט לחץ כאן כדי לשנות את הניסוי שנבחר. מידע כניסה לגיליון האלקטרוני המאוכלס באופן אוטומטי ולחץ על שמור (איור 6). אפשרות זו מאפשרת למשתמש לקודד בצבע נתונים במהלך ניתוח.
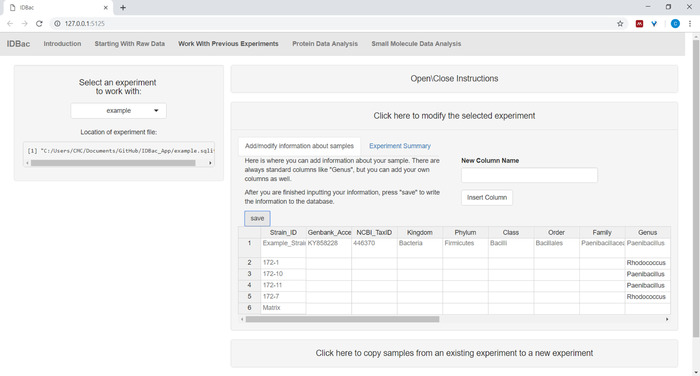
איור 6: מידע לדוגמה לקלט. במסגרת העמוד "עבודה עם ניסויים קודמים" משתמשים יכולים להזין מידע על דגימות כגון זהות מטקבית, מיקום גבייה, תנאי בידוד ועוד. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- אופציונלי העבר הכל, או קבוצת משנה של דגימות לניסוי חדש או אחר על-ידי לחיצה על דגימות העברה מניסויים קודמים לניסויים חדשים/אחרים ובעקבות ההוראות המפורטות (איור 7).
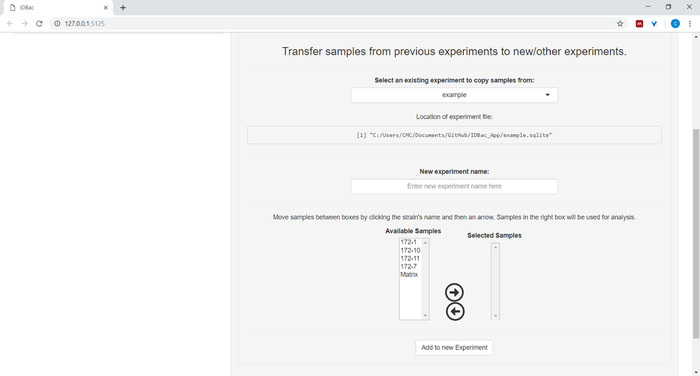
איור 7: העברת נתונים. הדף "עבודה עם ניסויים קודמים" מכיל את האפשרות להעביר נתונים בין ניסויים קיימים לניסויים חדשים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- כאשר אתה מוכן להתחיל בניתוח, ודא שהתהתנסות לעבודה עם נבחרה. בחר באפשרות ניתוח נתונים בחלבון או בניתוח נתונים של מולקולה קטנה.
8. הגדרת ניתוח נתוני חלבונים ויצירת מגרשים לשיקוף
- אם תנתח נתוני חלבונים, נווט תחילה לדף ' ניתוח נתוני חלבון '. בחר הגדרות איסוף בשיא והערכת ספקטרום חלבונים של דגימות באמצעות מגרשים השיקוף המוצג (איור 8).
הערה: בחלקות המראות, פסגה אדומה מרמזת על נוכחות השיא רק בספקטרום העליון, ואילו פסגות כחולות מייצגות את אלה המתרחשות בשני הספקטרום.
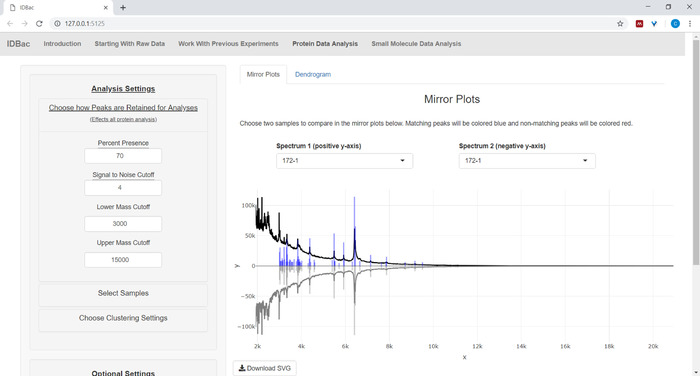
איור 8: לבחור כיצד הפסגות נשמרות לצורך ניתוח. לאחר בחירת ניסוי לניתוח, ביקור בעמוד "ניתוח נתוני חלבונים" ולאחר מכן פתיחת התפריט "בחר כיצד פסגות נשמרות לניתוח" מאפשרת למשתמשים לבחור הגדרות כגון יחס אות לרעש לצורך שמירה על פסגות. מחלקת השיקוף המוצגת (או הגודגרמה) תעדכן באופן אוטומטי כדי לשקף את ההגדרות שנבחרו. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- כוונן את אחוז השכפל בו יש להציג את הפסגה כדי שהיא תיכלל לניתוח (לדוגמה, אם הסף מוגדר ל-70% והשיא מתרחש בלפחות 7 מתוך 10 משכפל, הוא ייכלל).
- השימוש בחלקות המראה כהנחיה חזותית, מתאים את האות לניתוק הרעש שנשמר ברוב הפסגות "המקוריות" והרעש הנמוך ביותר, ומציין כי יותר משכפל וערך "אחוז שיא" גבוה יותר יאפשר בחירה של אות נמוך יותר לניתוק הרעש.
- ציין את האותיות הקטנות והתחתונות של m/z , ומכתיב את טווח ערכי המסה בתוך כל קשת שישמשו בניתוח נוסף על-ידי idbac.
9. דוגמאות לאשכולות שימוש בנתוני חלבון
- בעמוד ' ניתוח נתוני חלבון ', בחרו בכרטיסייה ' שמנת עץ '. הדבר מאפשר לקבץ דגימות לתוך העצים בהתאם לאמצעי המרחק שנבחרו על-ידי המשתמש ולאלגוריתמים של קיבוץ באשכולות.
- לחץ על בחר דוגמאות בתפריט ובצע את ההוראות כדי לבחור דוגמאות כדי לכלול בניתוח. רק דגימות המכילות ספקטרום חלבונים יוצגו בתיבת הדוגמאות הזמינות (איור 9).
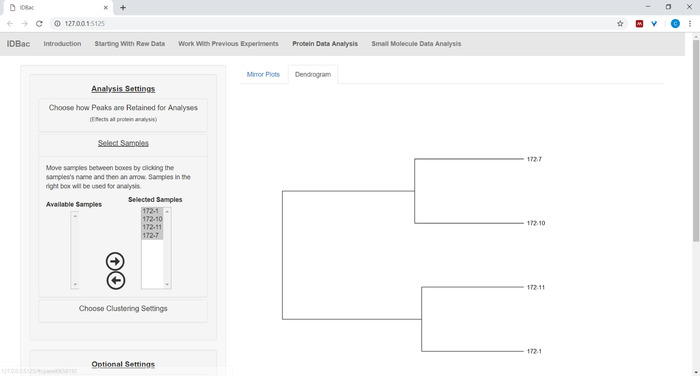
איור 9: דוגמאות מתוך הניסוי שנבחר לכלול בתוך העצים המוצגים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- השתמש בערכי ברירת המחדל או, תחת בחר הגדרות קיבוץ באשכולות, בחר את המרחק הרצוי ואת אלגוריתמי האשכולות שיוחלו על הדור של הגודגרמה.
- בחר באפשרות נוכחות/היעדרות כקלט. לחילופין, אם בטוחים בשיא השיאים של הדגימות (למשל, לאחר ביצוע מחקר להערכת שינויים בעוצמת שיא), בחרו עוצמות כקלט.
הערה: בזמן הפרסום, IDBac מספק גמישות בהגדרות עבור קיבוץ באשכולות, תוך הסתמכות על משתמשים כדי לבחור את השילובים המתאימים. אם לא היתה מוכרת לאפשרויות אלה, היא מוצעת לזוג מרחק A: "קוסינוס" ו-"ממוצע (UPGMA)" באשכולות; או B: מרחק "אוקלידי" ו-"וורד D2" באשכולות. - כדי להציג ערכי אתחול על הגודגרמה, הזן מספר בין 2 ל-1000 תחת Bootstraps.
- בעת דיווח על תוצאות, העתק את הטקסט בתוך הפיסקה הצעות לדיווח על ניתוח חלבון . פעולה זו מספקת את ההגדרות המוגדרות על-ידי המשתמש שיצרו את הגודגרמה הספציפית.
10. התאמה אישית של החלבונים בחלבון
- כדי להתחיל להתאים אישית את הגיגרם, פתח את התפריט התאמת העצים (איור 10).
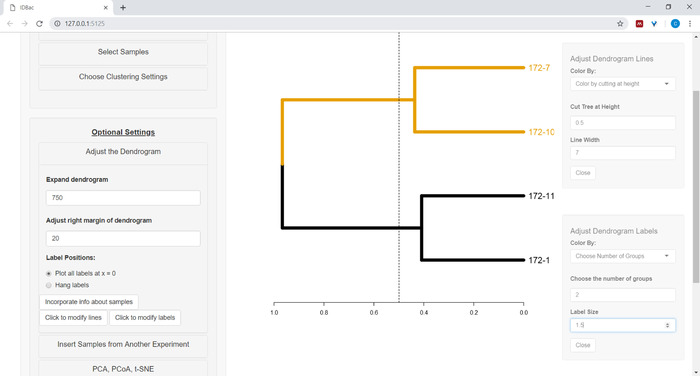
איור 10: כוונן את הגודגרמה. IDBac מספק מספר אפשרויות לשינוי אופן המראה של הגודגרמה, אלה ניתן למצוא בתוך התפריט "התאם את הטבעות". זה כולל ענפים צביעה ותוויות על ידי k-אמצעים, או על ידי "חיתוך" העצים על גבי גובה המשתמש מסופק. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- כדי לצבוע את הקווים ו/או התוויות של הגודגרם בחרו בלחצן המתאים: לחץ כדי לשנות שורות או לחץ כדי לשנות תוויות ובחר את האפשרויות הרצויות.
- כדי להתוות מידע מהגיליון האלקטרוני שליד הגודגרמה (ראה שלב 7.2), בחר את הלחצן לשלב מידע אודות דגימות. פעולה זו תפתח לוח שבו קטגוריה (עמודה בגיליון האלקטרוני) תאכלס את עצמה בהתבסס על הערכים שהוזנו (איור 11).
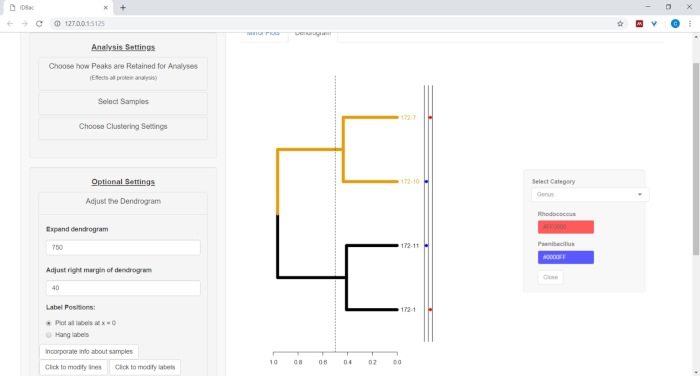
איור 11: שילוב מידע על דגימות. בתוך התפריט "כוונן את הגודגרמה" היא האפשרות "שילוב מידע על דגימות". בחירה באפשרות זו תאפשר התוויית מידע אודות דגימות שליד הגודגרמה. מידע לדוגמה הוא קלט בעמוד "עבודה עם ניסויים קודמים". אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
11. הוספת דגימות מניסוי נפרד לתוך העצים
- כדי להוסיף דגימות מניסוי אחר, בחר את לחצן התפריט הוסף דוגמאות מניסוי אחר. בצע את ההוראות בלוח החדש שנפתח (איור 12).
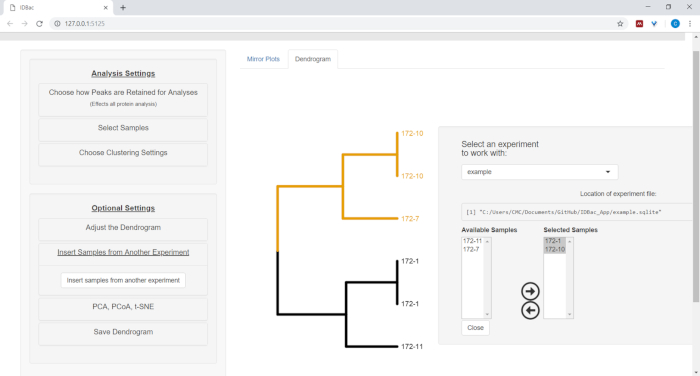
איור 12: הוספת דוגמאות מתפריט ' ניסוי אחר '. לפעמים מועיל להשוות דגימות מניסוי אחר. השתמש בתפריט "הוסף דוגמאות מניסוי אחר" כדי לבחור דוגמאות שיכללו בתוך השימוש הנוכחי בעצים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
12. ניתוח נתוני מטבוליט מיוחדים ורשתות התאגדות מטבוליט (מאמינים)
- המשך אל דף רשת השיוך של מטבוליט (מולקולה קטנה) . דף זה מאפשר הדמיה של נתונים באמצעות ניתוח רכיבים עקרוניים (pca) ומאמינים, המשתמשים ברשתות דו-צדדי כדי להציג את הקורלציה של ערכי m/z של מולקולה קטנה עם דגימות.
- אם נוצר על ידי חלבון (סעיף 9), הוא יוצג גם בעמוד זה. לחץ-וגרור על העצים כדי להדגיש דוגמאות לבחירה של עניין לניתוח. אם אין דוגמיות מודגשות או לא נוצר החלבון, אדם של תת-קבוצה אקראית או כל הדגימות יופיעו, בכבוד (איור 13).
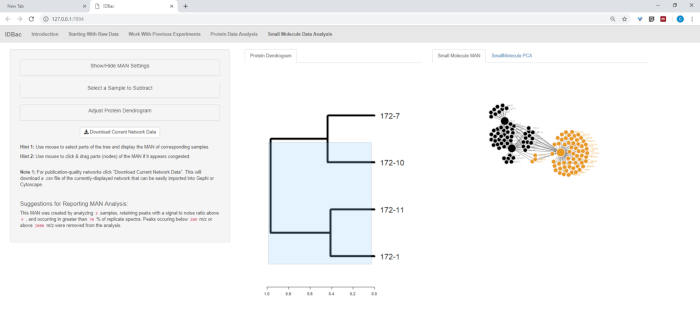
איור 13: דף ניתוח נתונים של מולקולה קטנה. אם נוצרה מעטפת חלבונים, היא תוצג בעמוד "ניתוח נתונים של מולקולה קטנה". דף זה יציג גם את רשתות השיוך של מטבוליט (מאמינים) וניתוח רכיבים עקרוניים (PCA) עבור נתוני מולקולה קטנה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- כדי להפחית מטריצה/מדיה ריקה ב-MAN, פתח את התפריט בחר דוגמה לחסר ובחר בדוגמה המתאימה לשימוש כריק.
- פתח את התפריט הצג/הסתר הגדרות אדם כדי לבחור את הערכים הרצויים עבור אחוז של נוכחות השיא בשכפל, אות לרעש, ו העליון והתחתון cutoffs המסה, כפי שנעשה עבור ספקטרום החלבון בסעיף 9. השתמש בעלילות השיקוף הקטנות כדי להנחות את הבחירה של הגדרות אלה.
- בחר באפשרות "הורד נתוני רשת נוכחיים" כדי לשמור את הנתונים של האדם המוצג כעת. ניתן להשתמש בנתונים אלה בתוכנת ניתוח רשת שאינה IDBac.
- כדי לדווח על תוצאות, העתק את הטקסט בתוך הפיסקה ' הצעות לדיווח על ניתוח אדם '. פעולה זו מספקת את ההגדרות המוגדרות על-ידי המשתמש להפקת האדם הנוצר.
13. שיתוף נתונים
- הניסוי כל IDBac נשמר כמסד נתונים בודד SQLite. הוא מכיל את המרה של ספקטרום raw של mzML, שזוהו פסגות, ואת כל מידע קלט משתמש על דגימות. לכן, כדי לשתף ניסוי IDBac פשוט לשתף את הקובץ SQlite בעל אותו שם כמו הניסוי (מיקום הקובץ מוצג בעבודה עם הניסויים הקודמים הדף).
תוצאות
ניתחנו שישה זנים של מיקרוונונוספורסיה ושני זנים של באקלוס subtilis, אשר היו מאופיינים בעבר6, באמצעות נתונים זמינים בדוי: 10.5281/zenodo. 2574096. להלן ההנחיות בכרטיסיה ' התחלה עם נתוני Raw ', בחרנו באפשרות לחץ כאן כדי להמיר קבצי bruker ובעקבות ההנחיות שסופקו על-ידי idbac עבור כל ערכת נתונים (איור 14).
לאחר ההמרה אוטומטי מראש/השיא השלבים הסתיימו, התקדמנו ליצור ניסוי IDBac משולב חדש על ידי העברת דגימות משני ניסויים לתוך ניסוי אחד המכיל את מקרתגו וגם דגימות מיקרומטר (איור 15). הניתוח שהתקבל מעורב בהשוואת ספקטרום החלבון באמצעות חלקות מראה, כתמונה באיור 16, שהיה שימושי להערכת איכות הספקטרום והתאמת הגדרות איסוף השיא. איור 17 מציג צילום מסך של תוצאות באשכולות החלבון עם הגדרות ברירת המחדל שנבחרו. הגודגרמה הייתה צבועה על ידי התאמת הסף לעלילה (מופיע כקו מנוקד). של הערה היא ההפרדה הבהירה בין genera, עם שתי הצ מבודדת ו B. subtilis מבודד אשכולות בנפרד.
איור 18, איור 19, ואיור 20 הדגש את היכולת ליצור מאמינים של אזורים שנבחרו על ידי המשתמש על-ידי לחיצה וגרירה על-פני עץ החלבון. עם זאת היינו מסוגלים במהירות ליצור מאמינים כדי להשוות רק את הזנים B. subtilis (איור 18), רק M. הצ זנים (איור19), ואת כל הזנים בו זמנית (איור 20). התפקיד העיקרי של רשתות אלה היא לספק לחוקרים סקירה רחבה של מידת החפיפה המקצועית מטבוליזם בין חיידקים. עם נתונים אלה בהישג יד, החוקרים יש כעת את היכולת לקבל החלטות מושכלות מכמות קטנה בלבד של חומר מגורד מתוך מושבה חיידקית.
איור 14: העיבוד של ספקטרה. ספקטרום ברוקר autoFlex שהורד הומרו ועובדו באמצעות IDBac. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
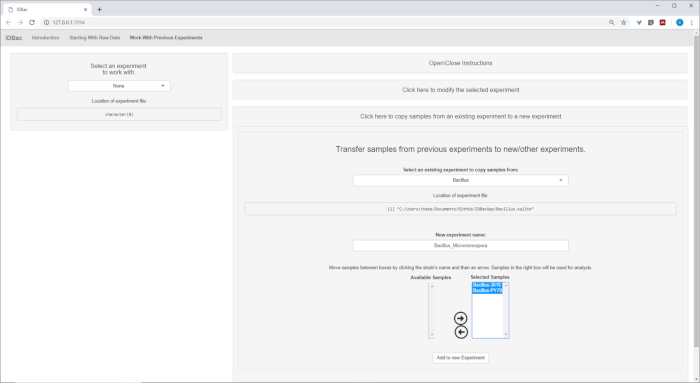
איור 15: ניסוי IDBac משולב. מכיוון שהמיקרוונוספורט ובאקלוס ספקטרום נאספו בלוחות מטרה שונים, שני הניסויים המשולבים לאחר מכן בניסוי יחיד-"Bacillus_Micromonsopora". זה נעשה בתוך הכרטיסייה "עבודה עם ניסויים קודמים", בעקבות ההנחיות בתוך התפריט "דגימות העברה מניסויים קודמים לניסויים חדשים/אחרים". אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
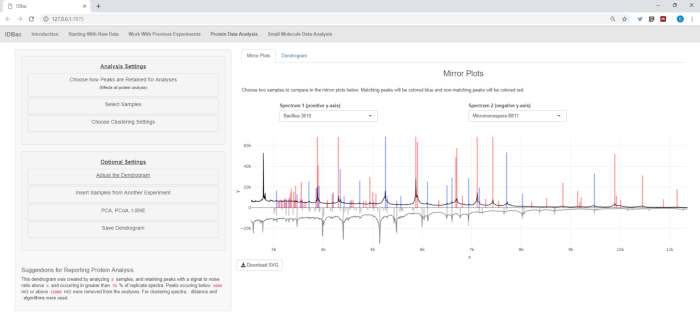
איור 16: השוואה. מיקרואוספורא ובאקלוס ספקטרום הושוו באמצעות מחלקות המראה בתוך דף "ניתוח נתוני חלבון". בסופו של דבר, הגדרות השיא המוגדר כברירת מחדל נבחרו. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
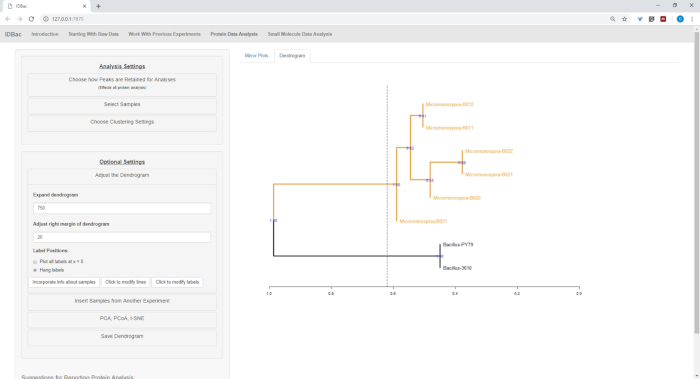
איור 17: קיבוץ באשכולות הירארכי. קיבוץ באשכולות הירארכי, שימוש בהגדרות ברירת המחדל, מקובצים באופן תקין באמצעות מיקרולוס ומיקרומטר. הגוגיגרמה הייתה צבועה על ידי "חיתוך" הגודגרמה בגובה שרירותי (מוצג כקו מקווקו) ו-100 האתחול השתמשו כדי להראות ביטחון בהסתעפות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
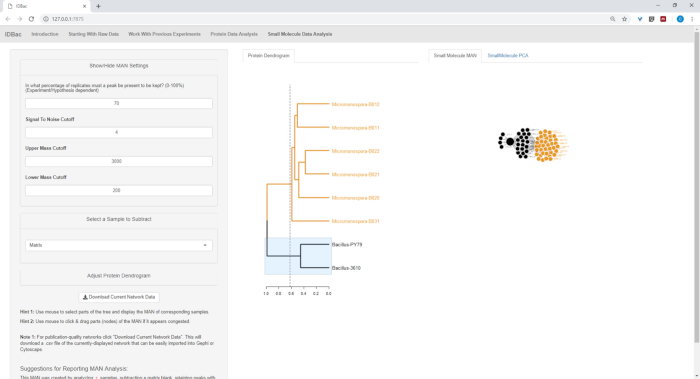
איור 18: האדם נוצר על ידי בחירת הזנים של מקרתגו sp. מן החלבונים החלבון הראו הפקה דיפרנציאלית של מטבוליטים מיוחדים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
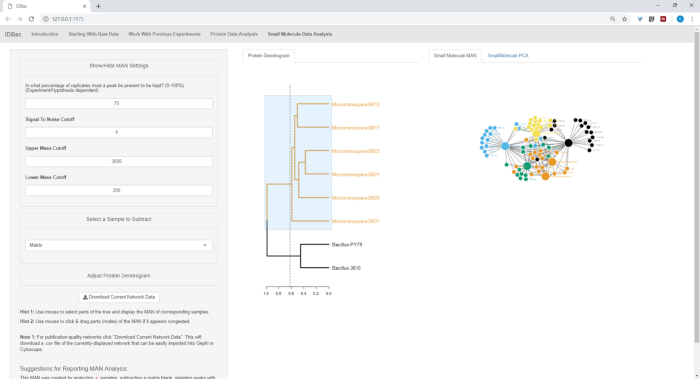
איור 19: האדם נוצר על ידי בחירת ששת מיקרומטר sp. זנים מן החלבונים החלבון הראו הייצור הדיפרנציאלי של מטבוליטים מיוחדים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
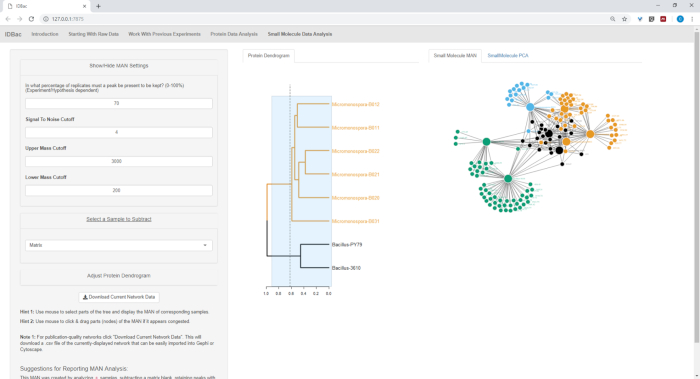
איור 20: האיש של באקלוס sp. ו מיקרוונוספורמטר sp. זנים המציגים ייצור דיפרנציאלי של מטבוליטים מיוחדים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
פרוטוקול IDBac פרטים חלבון חיידקי והתמחה מטבוליזם נתונים רכישה וניתוח של עד 384 חיידקי בודד 4 h על ידי חוקר אחד. עם idbac אין צורך לחלץ DNA מבודד בקטריאלי או ליצור תמציות מטבוליט מיוחדות מתוך התסיסה נוזלי ולנתח אותם באמצעות שיטות כרומטוגרפי. במקום זאת, חלבונים ונתונים מטבוליזם מיוחדים נאספים על ידי הפצת חומר פשוט ממושבות חיידקי ישירות על צלחת היעד MALDI. זה מפחית במידה ניכרת את הזמן ואת העלות הקשורים טכניקות חלופיות כגון רצף הגנים של 16 rRNA ו LCMS9.
חשוב להוסיף כתמים של מטריצה ריקה וכיול ללוח MALDI, ואנו ממליצים להשתמש במספר מתאים של שכפול כדי להבטיח שימוש בלתי מוצפן ובטחון סטטיסטי. מספרי השכפל יהיו תלויי-ניסוי. לדוגמה, אם משתמש מתכוון להבדיל בין אלפי מושבות מאוסף של לוחות גיוון סביבתי, ייתכן שיהיה צורך בפחות משכפל (המעבדה שלנו אוספת שלושה משכפל טכני לכל מושבה). לחילופין, אם משתמש מבקש ליצור מסד נתונים מותאם אישית של זנים מתוך מטקבקטריאלי ספציפיים כדי לקבוע במהירות סיווגים תת מיני של מבודד לא ידוע, אז משכפל יותר מתאים (המעבדה שלנו אוספת שמונה משכפל ביולוגי לכל מתח).
IDBac הוא כלי להבדיל במהירות מאוד הקשורים בקטריאלי בהתבסס על מידע מטקמיים וייצור מטבוליזם המקצועית. זה יכול להשלים או לשמש כמבשר לשיטות אורתוגונאליות כגון ניתוחים גנטיים מעמיק, מחקרים הכרוכים בייצור מטבוליזם ותפקוד, או אפיון של מבנה מטבוליט מיוחדים על ידי ספקטרוסקופיית תהודה מגנטית גרעינית ו/או LC-MS/MS.
ייצור מטבוליזם המקצועית (IDBac מאנס) הוא פגיע מאוד לתנאי גידול חיידקי, במיוחד באמצעות מדיה שונים, אשר היא מגבלה פוטנציאלית של השיטה. עם זאת תכונות אלה ניתן לנצל על ידי המשתמש, כמו IDBac יכול בקלות ליצור מאמינים מראה את ההבדלים בייצור מטבוליזם המקצועית תחת מגוון רחב של תנאי גדילה. חשוב לציין כי בעוד טביעות אצבעות מיוחדים מטבוליזם עשוי להשתנות בתנאי הצמיחה, הצגנו בעבר כי טביעות אצבע של חלבונים נשארים יציבים יחסית על פני משתנים אלה (ראה קלארק ואח '6). כאשר מתעסקים עם לוחות מגוון סביבתי, אנו ממליצים לטהר מבודד חיידקי לפני ניתוח כדי להפחית את התרומות האפשריות מפני שכנים בקטריות שכנות.
לבסוף, חוסר במאגר המידע הציבורי הניתן לחיפוש של טביעות אצבעות של חלבון MS הוא קיצור מרכזי המגיע השימוש בשיטה זו כדי לסווג חיידקים סביבתיים לא ידועים. יצרנו idbac עם זה בראש, וכללה המרה אוטומטית של נתונים לתוך פורמט מקובל הקהילה קוד פתוח (mzml)10,11,12 ועיצב את התוכנה כדי לאפשר חיפוש, שיתוף, ויצירה של מסדי נתונים מותאמים אישית. אנחנו בתהליך של יצירת מסד נתונים ציבורי גדול (> 10000 מאופיין במלואו), אשר יאפשר סיווג של כמה בודד לרמת המינים, כולל קישורים למספרי ההצטרפות של GenBank כאשר הם זמינים.
IDBac הוא מקור פתוח והקוד זמין עבור כל אחד כדי להתאים אישית את הצרכים שלהם ניתוח נתונים והדמיה. אנו ממליצים למשתמשים להתייעץ עם גוף ספרות נרחב (זאואר ואח '7, סילבה et al.5) כדי לסייע בתמיכה ובעיצוב המטרות הנסיוניות שלהם. אנו מארחים פורום לדיון ב: https://groups.google.com/forum/#!forum/idbac ואמצעים כדי לדווח על בעיות עם התוכנה ב: https://github.com/chasemc/IDBacApp/issues.
Disclosures
. למחברים אין מה לגלות
Acknowledgements
עבודה זו היתה נתמכת על ידי המכון הלאומי של מדעי הרפואה הכללית גרנט R01 GM125943, נשיונל ג'יאוגרפיק CP-044R-17; מלגת קרן המחקר האיסלנדי 152336-051; ואוניברסיטת אילינוי. בקרנות האתחול של שיקגו כמו כן, אנו מודים לתורמים הבאים: ד ר אמנדה בולמן לקבלת סיוע בפרמטרים לרכישת חלבונים באמצעות מלנדי-תוף MS; ד ר טרי מור וד ר מריה ג'יין עבור התקן אלפא-cyano-4-hydroxycinnamic חומצה מטריקס (CHCA).
Materials
| Name | Company | Catalog Number | Comments |
| Acetonitrile | Fisher | 60-002-65 | LC-MS Ultra CHROMASOLV |
| Autoflex Speed LEF MALDI-TOF instrument | Bruker Daltonics | ||
| Bruker Daltonics Bacterial test standard | Fisher | NC0884024 | Bruker Daltonics 8604530 |
| Bruker Peptide Calibration standard | Fisher | NC9846988 | Bruker Daltonics 8206195 |
| Formic Acid | Fisher Chemical | A117-50 | 99.5+%, Optima LC/MS Grade |
| MALDI-TOF target Plate | Bruker Daltonics | ||
| Methanol | Fisher Chemical | A456-500 | Optima LC/MS Grade |
| Toothpicks | any is ok | ||
| Trifluoroacetic acid | Fisher | AC293810010 | 99.5%, for biochemistry, ACROS Organics |
| Water | VWR | 7732-18-5 | LC-MS |
| α-Cyano-4-hydroxycinnamic acid | Sigma | 28166-41-8 | (C2020-25G) ≥98% (TLC), powder |
References
- Sandrin, T. R., Goldstein, J. E., Schumaker, S. MALDI TOF MS profiling of bacteria at the strain level: A review. Mass Spectrometry Reviews. 32 (3), 188-217 (2013).
- Cain, T. C., Lubman, D. M., Weber, W. J., Vertes, A. Differentiation of bacteria using protein profiles from matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 8 (12), 1026-1030 (1994).
- Holland, R. D., Wilkes, J. G., et al. Rapid identification of intact whole bacteria based on spectral patterns using matrix-assisted laser desorption/ionization with time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 10 (10), 1227-1232 (1996).
- Rahi, P., Prakash, O., Shouche, Y. S. Matrix-assisted laser desorption/ionization time-of-flight mass-spectrometry (MALDI-TOF MS) based microbial identifications: challenges and scopes for microbial ecologists. Frontiers in Microbiology. 7, 1359 (2016).
- Silva, R., Lopes, N. P., Silva, D. B. Application of MALDI mass spectrometry in natural products analysis. Planta Medica. 82, 671-689 (2016).
- Clark, C. M., Costa, M. S., Sanchez, L. M., Murphy, B. T. Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4981-4986 (2018).
- Freiwald, A., Sauer, S. Phylogenetic classification and identification of bacteria by mass spectrometry. Nature Protocols. 4 (5), 732-742 (2009).
- Schulthess, B., Bloemberg, G. V., Zbinden, R., Böttger, E. C., Hombach, M. Evaluation of the Bruker MALDI Biotyper for identification of Gram-positive rods: development of a diagnostic algorithm for the clinical laboratory. Journal of Clinical Microbiology. 52 (4), 1089-1097 (2014).
- Schumann, P., Maier, T. MALDI-TOF mass spectrometry applied to classification and identification of bacteria. Methods in Microbiology. 41, 275-306 (2014).
- Chambers, M. C., Maclean, B., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534 (2008).
- Martens, L., Chambers, M., et al. mzML-a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved