A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
וקטור lentiviral פלטפורמה למסירה יעיל של כלי עריכת Epigenome לתוך האדם המושרה מודלים המחלה נגזר תאי גזע Pluripotent
In This Article
Summary
לפלח epigenome DNA עריכה מייצג גישה טיפולית רב-עוצמה. פרוטוקול זה מתאר את הייצור, טיהור, הריכוז של ה-all-in-one וקטורים lentiviral מסתירים את transgene CRISPR-dCas9-DNMT3A עבור יישומים לעריכת epigenome בתא גזע pluripotent המושרה אנושי (hiPSC)-נגזר נוירונים.
Abstract
השימוש של תאים hiPSC, נגזר מייצג בגישה יקר ללמוד מחלות ניווניות אנושי. כאן, אנו מתארים את פרוטוקול ממוטב עבור הבידול של hiPSCs נגזר מטופל עם triplication של אלפא-synuclein ג'ין (SNCA) מיקומה לתוך מחלת פרקינסון (PD)-אוכלוסיות עצביים וכולינרגיים הרלוונטיים. לצבירת ראיות הראה כי רמות גבוהות של SNCA הם סיבתי לפיתוח של הכרה PD. קליניות צריכים לייסד רומן גישות טיפוליות עבור PD, במיוחד אלה מיקוד ברגולציה של ביטוי SNCA , אנחנו לאחרונה פיתחה מערכת CRISPR/dCas9-מתילציה-מבוסס DNA epigenetically לווסת שעתוק SNCA על ידי העשרת רמות מתילציה האזור רגולטוריות אינטרון 1 SNCA . כדי לספק את המערכת, המורכב מת (שהפעלתם בוטלה) הגירסה של Cas9 (dCas9) התמזגו עם המחשבים קטליטי של 3A אנזים methyltransferase ה-DNA (DNMT3A), וקטור lentiviral משמש. מערכת זו חלה על תאים המכילים triplication של לוקוס SNCA ומקטין את SNCA-mRNA ואת רמות החלבון בכ 30% באמצעות מתילציה DNA יישוב של אינטרון SNCA 1. Downregulation מכויל של רמות SNCA מציל הקשורים למחלות פנוטיפים הסלולר. בפרוטוקול הנוכחי, אנו שואפים לתאר את הליך שלב אחר שלב עבור המבדילים hiPSCs לתוך ובתאים עצבית (NPCs) ואת הממסד אימות של מבחני pyrosequencing על ההערכה של הפרופיל מתילציה SNCA אינטרון 1. חלוקה לרמות ביתר פירוט את המערכת lentivirus-CRISPR/dCas9 שימוש בניסויים אלה, פרוטוקול זה מתאר כיצד לייצר ולטהר, להתרכז וקטורים lentiviral כדי לסמן להתאמתם עבור epigenome ואת הגנום-יישומים לעריכת באמצעות hiPSCs ו- NPCs. הפרוטוקול ניתנת להתאמה בקלות והוא יכול לשמש כדי לייצר כייל נוגדנים גבוה lentiviruses ליישומים במבחנה ויוו.
Introduction
פלטפורמות מרובות לעריכת epigenome פותחו לאחרונה להתמקד על כל רצפי DNA אזורים ששולטים ג'ין ביטוי1,2. הכלים לעריכת epigenome שנוצר מתוכננים (i) להסדיר שעתוק, (ii) לשנות שינוי היסטון posttranslational, (iii) לשנות מתילציה DNA, (iv) לווסת את רכיב אינטראקציות. הגישה כדי לעגן את מכפילי תמלול/כרומטין Cas9 (מת) שהפעלתם בוטלה (dCas9) מופעל מבעבר מפותח לעריכת epigenome פלטפורמות, כגון אבץ אצבע חלבונים (ZFPs) ו effectors כמו מפעיל שעתוק (אגדות), מחסה תחום אפקטור תעתיק חזק (אד) התמזגו לתחום ה-DNA מחייב מעוצב (DBD)3. התוצאות של פנוטיפ הרצויה כגון הפעלה או דיכוי מוגדרת על-ידי המולקולה אפקטור מעוגנת את לוקוסים אנדוגני (איור 1). כדי ליצור activators תעתיק לתכנות, dCas9/gRNA מודולים מקושרים VP164,5,6 (איור 1 א'), תחום הפעלה ויראלי מגייס Pol II והמנגנון שעתוק כללי. השינוי של מערכת זו כללה VP64, tetramer של תחומים VP16, מתן אפילו יותר חזקה הפעלת קצב5,6. המערכת כבר מועסקים בהצלחה להפעיל קידוד ואזורים noncoding על ידי מיקוד היזמים ואלמנטים רגולטוריות. חשוב, אף-על-פי מולקולות VP64 ישירות אל תשנה את הכרומטין באזור היעד, היא מגייסת מכפילי כרומטין אשר מאגד תוצאות בעדות של הסימנים פעיל (euchromatin), כולל acetylation H3/H4, H3-K4 di / tri-מתילציה5,6. בנוסף VP64, יחידת משנה p65 של המתחם NF-κB האנושי יש כבר קשור את מודול dCas9/gRNA7. מעניין, קשירה של אלה effectors לאזורים במעלה הזרם של שעתוק התחלה אתרים (אזעקת דייט) ובתוך היזמים תוצאות אינדוקציה גנים חזקים. למרות זאת, effectors VP64 ו p65 שיכולים להפעיל גם את ההשפעות activatory תוך כדי להיות מקושר לאזורים ממוקם במורד הזרם של אזעקת דייט, משפרי דיסטלי7,8. כדי מביאות לתגובה תעתיק עמיד יותר, מספר dCas9-VP64 או dCas9-p65 fusions צריך להתגייס על יעד בודד לוקוס9,10. ככזה, ההתפתחות האחרונה של מפעילים הדור הבא, אשר לגייס תחומים אפקטור מרובים על-ידי מורכבת dCas9 יחיד-gRNA, כגון SunTag, הביא חזק הפעלה יכולת השוואה בין dCas9-VP64 פיוז'ן עמיתיהם11 , 12. תעתיק הפעלה משופרת התקבל באמצעות היתוך גרעיני של VP64, p65 Rta (VPR), transactivation לתחום של גמא-herpesviruses, קצה קרבוקסילי של dCas913 (איור 1 א'). מערכות CRISPR/dCas9 דומות התפתחו במטרה הדיכוי (איור 1B).
ג'ין אנדוגני דיכוי יכולה להיות מושגת עם fusions מדכא. שבולם שעברו הנדסה לאחור באמצעות מגוון רחב של מנגנונים (איור 1B). הוכח כי מערכות CRISPR/dCas9, לקשר את מדכא. שבולם DBD (אפילו בלי אפקטור תחום/s), ביעילות תסתמי כבר ביטוי גנים בעוד קשור יזם או נגד הזרם/במורד הזרם-TSS אזורים3,6 ,14. ההשפעות על שעתוק נגרמת על ידי הפרעה הסטריים של איגוד פקטור שעתוק ועיבוד RNA פולימראז. עם זאת, גישות מקיף יותר נדרשים, כמו ג'ין דיכוי מאת לבד הסטריים מכשלה הוא לעתים קרובות לא מספיק בשביל להשתיק חזקים. ההתפתחות האחרונה של הדור הבא של משתיקי קול בהתבסס על מערכות CRISPR/dCas9 נושא מדכא. שבולם תעתיק תחומים (TRDs), מכפילי היסטון (H3-K9 di-/ tri-מתילציה, H3-K27 di-/ tri-מתילציה; H3-K36 di-/ tri-מתילציה, H3/H4 deacetylation), ואת מתילציה DNA (CpG) הוביל את בניית כלים epigenetic ומאפשר עמידים יותר להשתיק אפקטים4,5,15,16, 17,18,19,20. זה הוכח, כי הגיוס של אלה epigenetic מכפילי ה-DNA עשוי להוביל להיווצרות עוד כרומטין סגור, והם מרוכז, אשר בדרך כלל להפיק יותר חזק שתיקה התוצאה21,22. התחום שתיקה הנפוץ ביותר בשימוש עם DBDs הוא4,Krüppel-הקשורים תיבת (קראב)5. הגיוס של הגורם הוכח כדי להתכתב עם כרומטין השינויים; ובכל זאת, המנגנון של שינויים אלה הם עדיין להיות מבואר16,17,18. לאחרונה, הוכח כי הלוקליזציה של קראב ל- DNA יכול לקדם את מכלול של methyltransferase היסטון את SETDB1, את מתחמי NuRD deacetylation (HDAC) היסטון, מציעה את האפשרות כי אינטראקציות אלה לתווך את היווצרות עיבוי כרומטין, תעתיק שתיקה3,13. כגישה חלופית, אפקטור תחומים יכול להיות דבוקה DBDs ליצירת חלבון שתיקה epigenetic מותאם אישית. מערכת זו מזרז ישירות דכאניים סימוני הדנ א או שינויים היסטון.
לאחרונה, השימוש במערכות CRISPR/dCas9 סינתטי קשור האנזים DNMT3A יש כבר לשנות את ייעודו עבור הביטול תעתיק. DNMT3A מזרז מתילציה DNA זה מפעילה דיכוי גנים ברמת השעתוק במהלך היווצרות של הטרוכרומטין על היזמים ג'ין אנדוגני, אחרים אזורים רגולטוריות (איור 1B)18,20. מקדונלד ואח18 ו- Vojta et al.20 היו המחברים הראשונים לדווח על כך שניתן להשתמש מתילציה DNA epigenome-ג'ין להחרשת או דיכוי, הממחיש המערכת פיוז'ן המועבר פלסמיד dCas9-DNMT3A יכול לשפר מנע בעוצמה ציטוזין מתילציה סביב ה TSS18,20. מקדונלד ועמיתים הפגינו התעסוקה של האסטרטגיה עלולה לגרום לירידה משמעותית (כ-40%) בגן מדכאי, רמות ה-mRNA CDKN2A 18. באופן דומה, פילוח האזור מקדם unmethylated של גנים באך או IL6ST מראה מתילציה CpG מוגבר זה היה בקורלציה עם הפחתה כפולה של ביטוי גנים20. המעבדה שלנו יש לאחרונה לשנות את ייעודו השימוש מתילציה DNA attenuating התוצאה פתולוגית של SNCA ביטוי (איור 2)23. האסטרטגיה מבוססת על שיפור סלקטיבי מתילציה DNA בתוך אזור אינטרון 1 SNCA , כפי שדווח בעבר להיות hypomethylated PD, דמנציה עם לוי גופים (DLB) המוח24,25, 26. Hypomethylation זה נקשר ביטוי SNCA , ובכך מציעים ליעד אטרקטיבי עבור התערבות טיפולית24,27,28. אנחנו לאחרונה הראו רמה נמוכה של מתילציה DNA אינטרון SNCA 1, אזור hiPSC, נגזר וכולינרגיים NPCs המתקבל מטופל PD עם triplication SNCA 23. היתרון של מודל ניסיוני זה היא כי NPCs ניתן robustly מופצות בתרבות או נוסף הבדיל לתוך נוירונים בוגרים, הפעלת של סינון יעיל לזהות גורמים גנטיים המתווכות פנוטיפים הסלולר, כולל חמצוני מתח, אפופטוזיס29. יתר על כן, מערכת מודל זה מאפשר למדענים לסכם את האירועים התפתחותית זו לפני תחילת סימפטום בחולים. בנוסף, נגזר hiPSC NPCs מייצגים כלי נהדר כדי לבדוק את המסלולים תאית ומולקולרית המשויך ביטוי גנים. חשוב, NPCs נגזר hiPSC, בשילוב עם טכנולוגיית CRISPR/Cas9-epigenome המדינה-of-the-art יכול מאוד להקל על הפיתוח של "הדור הבא סמים" למחלות ניווניות רבות.
כדי להפחית את רמות פתולוגי של ביטוי SNCA, שפיתחנו לאחרונה מערכת מבוססת lentivirus נושא של חלבון כימרי dCas9-DNMT3A ו- gRNA כדי במיוחד היעד מתילציה CpG בתוך אינטרון SNCA 1 (איור 2 א)23. פרוטוקול זה יתאר וקטור lentiviral (LV) עיצוב וייצור בפירוט. הזינו אותו מייצגים אמצעי יעיל להעברת רכיבים CRISPR/dCas9 מכמה סיבות, כלומר מוסיף (i) את יכולת לשאת DNA מגושם, (ii) יעילות גבוהה של מגוון רחב של תאים, כולל תאים החלוקה והן nondividing30 transducing , וכן (iii) את היכולת שלהם זירוז תגובות ציטוטוקסיות ו immunogenic מינימלי. לאחרונה, אנחנו שהוחל מערכת LV נוירונים דופאמין hiPSC, נגזר מן המטופל עם triplication של מיקומה SNCA והפגינו את הפוטנציאל הטיפולי של אותו למסירה של עריכת epigenome מתילציה כלים23 ( איור 2B). ואכן, מערכת LV-gRNA/dCas9-DNMT3A גורמת עלייה משמעותית מתילציה DNA-האזור אינטרון 1 SNCA . גידול זה מתכתב עם הירידה ברמות של mRNA, חלבון SNCA 23. יתר על כן, SNCA downregulation מציל הקשורות PD פנוטיפים ב- SNCA triplication/hiPSC-derived וכולינרגיים נוירונים (למשל, מיטוכונדריאלי ROS ייצור ותא הכדאיות)23. חשוב לציין, הפגנו כי ההפחתה בביטוי SNCA על ידי מערכת LV-gRNA-dCas9-DMNT3A הוא מסוגל היפוך על הפנוטיפים האופייניים של נוירונים דופאמין hiPSC, נגזר מן המטופל PD שביצע את SNCA triplication, כגון מיטוכונדריאלי ROS ייצור ותא הכדאיות23. המטרה של פרוטוקול זה הוא 1) כדי לחלק לרמות הפרוטוקול של הפקה וריכוז של פלטפורמת LV ממוטב ליצירה צייץ בשיא ההכנות נגיפי ו 2) כדי לתאר את הבידול של hiPSCs לתוך NPCs בדוגמת להפוך וכולינרגיים בוגרת נוירונים31,32 , אפיון רמות מתילציה של האזור יישוב בתוך אינטרון SNCA 1.
פלטפורמות lentiviral יש יתרון גדול על פלטפורמת וקטור הפופולרי ביותר, כלומר adeno-הקשורים וקטורים (AAVs), אשר הוא היכולת של לשעבר להכיל מוסיף גנטי גדול33,34. AAVs ניתן ליצור תשואות גבוהות באופן משמעותי, אבל בעלי קיבולת אריזה נמוך (< 4.8 kb), להתפשר על השימוש בהם לצורך אספקת מערכות CRISPR/Cas9 ה-all-in-one. לפיכך, נראה כי אותו יהיה הפלטפורמה-של-הבחירה ביישומי מעורב אספקה של כלים CRISPR/dCas9. לכן, פרוטוקול שמפורטות כאן יהיה כלי רב ערך עבור חוקרים שרוצים להעביר ביעילות רכיבים לעריכת epigenome תאים ואיברים. פי הפרוטוקול, מתאר את האסטרטגיה כדי להגדיל את יכולות הייצור וביטוי של הווקטורים באמצעות שינוי ב- cis הרכיבים בתוך30,35קלטת ביטוי וקטור. האסטרטגיה מבוססת על הרומן מערכת שפותחה למד במעבדה שלנו ואני מדגיש את היכולת שלה לייצר חלקיקים נגיפיים בטווח של 1010 יחידות ויראלי (וו) /mL30,35.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. מערכת העיצוב והייצור וירוס
- פלסמיד עיצוב ובנייה
הערה: הבנייה של וקטור LV-gRNA-dCas9-DNMT3A ה-all-in-one מתבצע באמצעות הפקה- ואת קלטת ביטוי ממוטב ביטוי שפורסמו על-ידי Ortinski et al.30. בקלטת וקטור נושא שידור חוזר של האתר זיהוי של גורם שעתוק Sp1 ומחיקה של המדינה-of-the-art בתוך לא מתורגם (U3') אזור של טרמינל 3' באורך חוזר (משמאל לימין) (איור 2 א)30,36. עמוד השדרה וקטור נמצאו יעילים אספקת ולבטא30,CRISPR/Cas935.- להשיג את הגירסה deactivate (המלח) של SpCas9 (dCas9) באמצעות מערכות מוטגנזה דגן (נתונים לא מוצג). החלף את השיבוט מחסה מוטציות D10A ו- H840A בתחומים קטליטי HNH ו- RuvC של האנזים, בהתאמה, עם Cas9 פעיל pBK30129 על ידי החלפת בין שברי AgeI-BamHI (איור 3).
- שואבים את התחום קטליטי DNMT3A pdCas9-DNMT3A-eGFP (ראה טבלה של חומרים) על ידי הגברה של DNMT3A חלק BamHI-429/R 5 '- GAGCGGATCCCCCTCCCG - 3' BamHI-429/L 5' - CTCTCCACTGCCGGATCCGG - 3' (איור 3). כדי להגביר את האזור המכיל DNMT3A, השתמש התנאים הבאים: (1) 95 ° C 60 s, (2) 95 ° C עבור 10 s, (3) 60 ° C עבור 20 s, (4) 68 ° C עבור 60 ס' חזור על תנאי 2 עד 4 30 x. עבור הסיומת הסופי, להשתמש 68 מעלות צלזיוס למשך 3 דקות והחזק 4 ° C.
- לשכפל השבר DNMT3A, מתעכל על ידי אנזים הגבלה BamHI, לתוך האתר BamHI של וקטור שונה pBK301 נושא dCas9. ודא השיבוט על ידי סנגר ישירה רצף. שימו לב כי פלסמיד הביא בנמלים dCas9-DNMT3A-p2a-puromycin transgene. פלסמיד מבטא gRNA לגרדום מ- U6 אנושי המקדם (איור 3).
- החלף את הגן כתב puromycin חלבון פלואורסצנטי ירוק (GFP) כדי ליצור dCas9-DNMT3A-p2a-GFP. לעכל dCas9-DNMT3A-p2a-פורו פלסמיד עם FseI. לטהר את המקטע וקטור בעזרת שיטת טיהור ג'ל. הכן את תותב באמצעות עיכול pBK201a (pLenti-GFP) עם FseI. לשכפל את מקטע FseI לתוך וקטור. PBK539 פלסמיד הביא בנמלים transgene dCas9-DNMT3A-p2a-GFP (איור 3).
- Culturing HEK-293T תאים, ציפוי תאים עבור תרביות תאים
הערה: כליה אנושית עובריים 293T (HEK-293T) תאים מתורבתים ב גבוהה מלאה-גלוקוז Dulbecco המתואמת של בינוני הנשר (DMEM; 10% עגל שור סרום, אנטיביוטיקה-antimycotic 1 x, 1 x נתרן פירובט, 1 x לא חיוניות חומצת אמינו, 2 מ מגלוטמין) ב 37 ° C עם 5% CO 2. עבור הפארמצבטית הפרוטוקול, מומלץ לבחון את הנסיוב עגל כאשר מחליפים הרבה/אצווה שונים. עד 15 6 ס מ לוחות הדרושים לייצור lentiviral.- השתמש התאים נמוך-המעבר להפעלת תרבות חדשה (נמוך יותר מאשר מעבר 20). לאחר התאים להגיע למפגש 90%-95%, וארוקן את המדיה ואת בעדינות לשטוף אותו עם עקר 1 x באגירה פוספט תמיסת מלח (PBS).
- להוסיף 2 מ של טריפסין-EDTA (0.05%) דגירה זה ב 37 מעלות צלזיוס למשך 3-5 דקות. כדי להשבית הכימית דיסוציאציה, להוסיף 8 מ של DMEM גבוהה-גלוקוז מלאה, ו x x-15 פיפטה 10 עם פיפטה סרולוגית 10 מ ליצירת תא בודד השעיה של 4 x 106 תאים למ"ל.
- עבור transfections, מעיל צלחות 15 ס מ עם ג'לטין 0.2%. הוסף 22.5 מ של גלוקוז גבוה בינוני, זרע התאים על-ידי הוספת 2.5 מ של התא השעיה (סה כ ~ 1 x 107 תאים/צלחת). דגירה הלוחות ב 37 ° C עם 5% CO2 עד 70%-80% הנהרות.
- תרביות תאים תאים HEK-293T
- להכין 2 x פתרון באגירה ולבס BBS CaCl 1 מ'2, על פי Vijayraghavan, קנטור35. לסנן את הפתרונות על ידי הכנסתם מסנן מיקרומטר 0.22 ואחסן אותם ב 4 º C. המיקס תרביות תאים חייב להיות ברור לפני תוספת שלה על התאים. אם השילוב הופך מעוננים במהלך הדגירה, להכין טרי x BBS 2 (ה-pH = 6.95).
- כדי להכין את תערובת פלסמיד, להשתמש של פלסמידים ארבע כמפורט (התמהיל הבא הוא מספיק עבור לוח 15 ס מ אחד): 37.5 µg של וקטור CRISPR/dCas9-העברה (pBK492 [DNMT3A-פורו-לא-gRNA] או pBK539 [DNMT3A-GFP-לא-gRNA]); 25 µg של pBK240 (psPAX2); µg 12.5 של pMD2.G; µg 6.25 של pRSV-rev (איור 4A). חישוב נפח פלסמידים מבוסס על ריכוז ולהוסיף הכמויות הנדרש לתוך צינור חרוטי 15 מ"ל. להוסיף µL 312.5 של 1 מ' CaCl2 להביא את עוצמת הקול הסופי 1.25 mL, בעזרת סטרילי dd-H2O. בעדינות להוסיף mL 1.25 של פתרון 2 x BBS בעת vortexing המיקס. תקופת דגירה של 30 דקות בטמפרטורת החדר. התאים הם מוכנים תרביות תאים, ברגע שהם יהיו confluent 70%-80%.
- האחות התקשורת ולהחליף אותו עם 22.5 מ ל שזה עתה הוכנו DMEM גבוהה-גלוקוז ללא סרום. להוסיף 2.5 מ של תערובת תרביות תאים dropwise כל צלחת 15 ס מ. מערבולת הלוחות, דגירה ב 37 ° C עם 5% CO2 על 2-3 h.
- לאחר 3 שעות, להוסיף 2.5 מ ל (10%) של נסיוב לכל צלחת דגירה בין לילה ב 37 ° C עם 5% CO2.
- ביום 1 לאחר תרביות תאים, לבחון את התאים כדי לוודא שלא תהיה שום או מוות של תאים מינימלית, כי התאים יצרו תרבות confluent (100%).
- לשנות את התקשורת על-ידי הוספת 25 מ ל שזה עתה הוכנו סרום DMEM ו-10% גלוקוז גבוהה כל צלחת.
- דגירה ב 37 ° C עם 5% CO2 במשך 48 שעות.
- הקציר של הוירוס
- לאסוף את תגובת שיקוע של כל התאים transfected, בריכה להם צינורות חרוט 50 מ. צנטריפוגה ב 400-450 x g עבור 10 דקות לסנן את תגובת שיקוע דרך יחידת מסנן ואקום 0.45 מיקרומטר. לאחר סינון, ניתן לשמור את תגובת שיקוע ב 4 ° C לאחסון לטווח קצר (עד 4 ימים). לאחסון לטווח ארוך, להכין aliquots ואחסן אותם ב- 80 ° c
הערה: ההכנות נגיפי nonconcentrated צפויים להיות ~ 2 x 107 3 x 107 וו/mL (עיין בסעיף 1.5 לקביעת כייל נוגדנים). מומלץ להכין aliquots לשימוש יחיד, מאז מספר מחזורים ההקפאה-הפשרה תגרום לאובדן 10% – 20% ב- titers תפקודית.
- לאסוף את תגובת שיקוע של כל התאים transfected, בריכה להם צינורות חרוט 50 מ. צנטריפוגה ב 400-450 x g עבור 10 דקות לסנן את תגובת שיקוע דרך יחידת מסנן ואקום 0.45 מיקרומטר. לאחר סינון, ניתן לשמור את תגובת שיקוע ב 4 ° C לאחסון לטווח קצר (עד 4 ימים). לאחסון לטווח ארוך, להכין aliquots ואחסן אותם ב- 80 ° c
- ריכוז של נגיפים
הערה: לטיהור, שיטת כפול-סוכרוז שני שלבים מעורבים צעד מעבר צבע סוכרוז, צעד כרית סוכרוז הוא ביצע (איור 4B).- ליצירת מעבר צבע סוכרוז, להכין את צינורות ultracentrifugation חרוט לפי הסדר הבא: 0.5 מ של 70% סוכרוז ב- PBS 1 x, 0.5 מ של 60% סוכרוז DMEM, 1 מ"ל של 30% סוכרוז ב DMEM ו 2 מ של 20% סוכרוז ב- 1 x PBS.
- . בזהירות, מוסיפים את תגובת שיקוע, שנאספו לפי סעיף 1.4, מעבר הצבע. מאחר לנפח הכללי שנאסף ארבעה לוחות 15 ס מ 100 מ ל, להשתמש 6 צינורות ultracentrifugation לעבד את תגובת שיקוע ויראלי.
- באותה מידה להפיץ את תגובת שיקוע ויראלי בקרב כל שפופרת ultracentrifugation. כדי למנוע שבירה צינור במהלך צנטריפוגה, מלא הצינורות ultracentrifugation כדי לפחות שלוש וארבע של קיבולת הנפח הכולל שלהם. לאזן את הצינורות עם 1 x PBS. Centrifuge את הדגימות ב x 70,000 g עבור 2 h ב-17 מעלות צלסיוס
הערה: כדי לשמור על השכבה סוכרוז במהלך השלבים האצה והאטה, לאפשר את ultracentrifuge לאט להאיץ ולהאט הרוטור מ-0 ל-200 x g ומ-200 0 x g במשך 3 דקות הראשון והאחרון של הספין, בהתאמה. - בעדינות לאסוף 30% – 60% סוכרוז שברים לתוך צינורות נקיים (איור 4B). להוסיף 1 x למעלה (קר) PBS 100 מ של הנפח הכולל. מערבבים על-ידי pipetting מספר פעמים.
- . בזהירות, stratify ההכנה ויראלי על כרית סוכרוז על-ידי הוספת מ 4 ל 20% סוכרוז (ב- 1 x PBS) ברכבת התחתית. המשך על-ידי pipetting ~ 20 – 25 מ של הפתרון ויראלי למחזור בכל. למלא את הצינורות 1 x PBS אם אמצעי האחסון של התוכן שלהם הוא פחות מ 3-4 למחזור. בזהירות לאזן בין הצינורות. צנטריפוגה ב 70,000 x g עבור 2 h ב-17 מעלות צלסיוס . רוקן את תגובת שיקוע, היפוך הצינורות על מגבות נייר כדי לאפשר את הנותרים נוזלי כדי לנקז.
- להסיר את כל הנוזל על ידי בזהירות כ רפה בעברית את הנוזל הנותר. שימו לב כי, בשלב זה, פלטות המכילה הוירוס בקושי נראה קטן. נקודות שקופות. להוסיף µL 70 ל- 1 x PBS צינור הראשון כדי resuspend בגדר. ביסודיות pipette ההשעיה, להעביר אותו הצינור הבא עד כל החבילות הן resuspended.
- לשטוף את הצינורות עם µL 50 נוספים של 1-PBS ומיקס כמו בעבר. שימו לב כי, בשלב זה, עוצמת הקול של התליה האחרון ~ 120 µL, מופיע מעט חלבי. כדי לקבל השעיה ברורה, להמשיך עם צנטריפוגה s 60 ב x 10,000 ג'י. להעביר את תגובת שיקוע צינור, להפוך 5 µL aliquots, ולאחסן אותם ב- 80 ° c
הערה: ההכנות וקטור lentiviral רגישים מחזוריות החוזרות ונשנות הקפאה והפשרה. בנוסף, הוא הציע כי השלבים הנותרים הם לעשות תרביות רקמה בלימה או ב המיועד אזורים מוסמך במונחים של להיות ברמה נאותה של תקני אבטחה (איור 4B).
- כימות של titers ויראלי
הערה: ההערכה של נגיפי titers מבוצע בשיטת p24 מקושרים-אנזים immunosorbent assay (אליסה) (p24מחסום פה אליסה), על-פי התוכנית חיסון לאיידס הלאומית המכונים לבריאות (NIH) פרוטוקול עבור HIV-1 p24 ללכוד הנוגדנים עם שינויים קלים.- השתמש µL 200 של 0.05%-Tween-20 x 1 קר ב- PBS (PBS-T) כדי לשטוף את הבארות של צלחת 96-ובכן 3 x.
- כדי להרגיע את הצלחת, השתמש µL 100 של נוגדן monoclonal anti-p24 מדולל ב- 1:1,500 ב- 1 x PBS. דגירה את הצלחת בן לילה ב 4 º C.
- להכין ריאגנט חסימה (1% שור אלבומין [BSA] ב- 1 x PBS) ולהוסיף 200 µL בכל טוב כדי למנוע מחייב לא ספציפית. השתמש µL 200 של PBS-T כדי לשטוף את x 3 טוב לפחות שעה בטמפרטורת החדר.
- להמשיך עם הכנת המדגם. בעת עבודה עם ההכנות וקטור מרוכז, לדלל וקטור ב- 1: 100, באמצעות µL 1 מדגם µL 89 של dd-H20, 10 µL של טריטון X-100 (ריכוז סופי של 10%). על ההכנות nonconcentrated, לדלל את הדגימות-1:10.
- לקבל HIV-1 סטנדרטים באמצעות כפולה לדילול טורי (הריכוז ההתחלתי הוא 5 ng/mL).
- לדלל את הדגימות מרוכז (להכין בשלב 1.6.4) ב- RPMI 1640 בתוספת 0.2% BSA Tween 20 ו- 1% כדי להשיג 1:10, 000, 1:50, 000, 1:250,000 דילולים. באופן דומה, לדלל את הדגימות nonconcentrated (להכין בשלב 1.6.4) ב- RPMI 1640 בתוספת 0.2% Tween 20 ו- 1% BSA להקים שבערך, 1:2, 500, ו דילולים 1:12,500.
- הוסף את הדגימות ואת תקני בצלחת ב- triplicates. דגירה בין לילה ב 4 º C.
- למחרת, לשטוף את הבארות 6 x.
- להוסיף 100 µL של הארנב polyclonal נוגדן anti-p24, מדולל ב- 1:1,000 ב RPMI 1640, 10% סרום שור עוברית (FBS), BSA 0.25% ו- 2% העכבר הרגיל סרום (NMS), דגירה ב 37 מעלות צלזיוס במשך 4 שעות.
- לשטוף את הבארות 6 x. להוסיף עז ארנב נגד חזרת peroxidase IgG מדולל ב- 1:10,000 ב RPMI 1640 בתוספת 5% עז נורמלית בסרום, 2% NMS, BSA 0.25% ו- 0.01% Tween 20. דגירה ב 37 ° C עבור 1 h.
- לשטוף את הבארות 6 x. הוסף שוייץ peroxidase המצע, דגירה בטמפרטורת החדר למשך 15 דקות.
- כדי להפסיק את התגובה, להוסיף 100 µL של 1 N HCl. בקורא microplate, למדוד את ספיגת ב 450 ננומטר.
- מדידה של הכתב פלורסנט בעוצמה
- השתמש ההשעיה ויראלי להשגת 10-fold לדילול טורי (מתוך 10-1 כדי 10-5) ב 1 x PBS.
- צלחת 5 x 10 תאים5 HEK-293T כל טוב של צלחת 6-. טוב. החל µL 10 של כל דילול ויראלי לתאים, דגירה ב 37 ° C עם 5% CO2 במשך 48 שעות.
- להמשיך תא לפעיל על-ידי קרינה פלואורסצנטית מיון (FACS) ניתוח כדלקמן: ניתוק תאים על-ידי הוספת µL 200 של 0.05% טריפסין-EDTA פתרון. דגירה התאים ב 37 מעלות צלזיוס למשך 5 דקות, resuspend אותם ב 2 מ של DMEM בינוני (עם סרום). לאסוף הדגימות לתוך צינור חרוטי 15 מ"ל צנטריפוגה-x 400 g ב- 4 מעלות צלזיוס. Resuspend בגדר ב µL 500 ל- PBS קר 1 x.
- לתקן את התאים על-ידי הוספת µL 500 של 4% paraformaldehyde (PFA), תקופת דגירה של 10 דקות בטמפרטורת החדר.
- צנטריפוגה ב x 400 g ב 4 ° C, resuspend בגדר ב 1 מ"ל של PBS 1 x. לנתח את הביטוי GFP באמצעות מכשיר FACS, כפי שמתואר Ortinski et al.30. כדי לקבוע כייל פונקציונלי של וירוס, השתמש בנוסחה הבאה.

כאן,
Tg = מספר תאים GFP-חיוביות;
Tn = סה כ מספר תאים;
N = מספר כולל של תאים transduced;
V = אמצעי המשמש עבור התמרה חושית (ב microliters).
- ספירת תאים GFP-חיובית
הערה: לקבוע את הריבוי של זיהום (MOI) כי הוא מועסק עבור התמרה חושית. מבחן של טווח רחב של MOIs (מ- MOI = 1 ל- MOI = 10).- הזרע 3 x 105 לתאים5 HEK-293T עונה 4 פרק 10 לכל כל טוב של צלחת 6-. טוב.
- כאשר התאים להגיע > 80% הנהרות, מגלי אותם עם וקטור למשרד הפנים של עניין.
- דגירה ב 37 ° C עם 5% CO2, לנטר את האות ה-GFP בתאים 1-7 ימים.
- לספור את מספר תאי ה-GFP-חיוביות. מעסיקים מיקרוסקופ פלואורסצנטי (תוכנית 4 x אובייקטיבי, 0.1 נ. א, 40 x הגדלה) באמצעות מסנן GFP (עירור אורך גל = 470 ננומטר, אורך גל פליטה = 525 ננומטר). השתמש untransduced תאים כדי להגדיר שליטה אוכלוסיית תאים GFP-שלילי.
- מעסיקים את הנוסחה הבאה כדי לקבוע את כייל פונקציונלי של הנגיף.
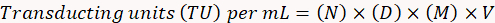
כאן,
N = מספר תאים GFP-חיוביות;
D = גורם לדילול;
M = פקטור ההגדלה;
V = נפח של וירוס המשמש עבור התמרה חושית.
הערה: לחשב התוצאות בעקבות דוגמה זו: עבור תאים GFP-חיובית 10 (N) נספרים לדילול (ד) 10-4 (1:10, 000) בהגדלה 20 x (ז) במדגם 10 µL (V), יהיה טו למיליליטר (10 x 104) x (20) x (10) x (100) = 2 x 108 וו/mL.
2. הבידול של העצבי דופאמין ובתאים
- Culturing hiPSCs
הערה: hiPSCs מחולה עם triplication של מיקומה SNCA, ND34391, התקבלו מן המכון הלאומי של הפרעות נוירולוגיות ו קו (NINDS) קטלוג (ראה טבלה של חומרים).- תרבות hiPSCs בתנאים מזין תלויית תרבות ESC-iPSC נטולת מזין בינוני (ראה טבלה של חומרים) על גבי הממברנה המלא hESC מטריקס בסיסי (BMM)-מצופה לוחות (ראה טבלה של חומרים). לשטוף את המושבות confluent עם 1 מ"ל של DMEM/F12, להוסיף 1 מיליליטר דיסוציאציה ריאגנט (ראה טבלה של חומרים), דגירה למשך 3 דקות בטמפרטורת החדר.
- האחות הכימית דיסוציאציה ולהוסיף 1 מ"ל של תרבות ESC-iPSC נטולת מזין בינוני.
- לגרד את הצלחת השימוש של מרים תא, resuspend את המושבות ב mL 11 נטולת מזין ESC-iPSC תרבות בינוני על ידי pipetting x-5 4 x באמצעות פיפטות בורוסיליקט.
- לוחית רישוי 2 מ"ל של המושבה השעיה על גבי צלחות מצופה BMM ולמקם את הצלחת ב 37 ° C עם 5% CO2. לבצע שינוי בינונית מדי יום, לפצל את התאים כל 5-7 ימים.
- בידול לתוך העצבי דופאמין ובתאים
הערה: הבידול של hiPSCs לתוך וכולינרגיים עצבית ובתאים (MD NPCs) מתבצעת באמצעות פרוטוקול זמינים מסחרית אינדוקציה עצבי בינוני לפי הוראות היצרנים, עם שינויים קלים (32 31, ראה טבלה של חומרים). היום הראשון של הבידול נחשב יום 0. HiPSCs באיכות גבוהה נדרשים עבור בידול עצבית יעילה. אינדוקציה של MD NPCs הוא performedusing גוף embryoid (EB)-מבוסס פרוטוקול.- לפני שמתחילים את הבידול של hiPSCs, להכין צלחת תרבות microwell (ראה טבלה של חומרים) על פי הוראות היצרן שלה.
- לאחר הכנת לוח תרבות microwell, להוסיף 1 מ"ל של אינדוקציה עצבי בינוני (נים; ראה טבלה של חומרים) בתוספת 10 מיקרומטר Y-27632.
- להפריש את הצלחת עד מוכן לשימוש.
- לשטוף את hiPSCs עם DMEM/F12, להוסיף 1 מ"ל של פתרון ניתוק התא (ראה טבלה של חומרים), דגירה למשך 5 דקות ב 37 ° C עם 5% CO2.
- Resuspend תאים בודדים ב- DMEM/F12, centrifuge אותם ב 300 x גרם במשך 5 דקות.
- וארוקן את תגובת שיקוע ובזהירות resuspend התאים נים + 10 מיקרומטר Y-27632 בריכוז הסופי של 3 x 106 תאים/מ ל....
- להוסיף 1 מ"ל של התליה תא בודד לבאר בודדת של צלחת תרבות microwell, centrifuge את הצלחת ב x 100 g למשך 3 דקות.
- בוחנים את הצלחת מתחת למיקרוסקופ כדי להבטיח ריפודי כתופיים התאים בין microwell, דגירה התאים ב 37 ° C עם 5% CO2.
- ימים 1-4, לבצע שינוי יומי בינוני חלקית.
- באמצעות micropipette של 1 מ"ל, להסיר 1.5 מ של המדיום וזורקים. . לאט, להוסיף 1.5 מ של נים טריים ללא Y-27632.
- חזור על שלב 2.2.10 עד יום ד'.
- יום 5, מעיל אחד טוב של צלחת 6-ובכן עם BMM.
- מניחים מסננת הפיך מיקרומטר 37 (ראה טבלה של חומרים) על גבי צינור חרוטי 50 מ ל (פסולת). הצבע על החץ של מסננת הפיך כלפי מעלה.
- הסר את המדיום לצלחת תרבות microwell מבלי להפריע את EBs בנוי.
- להוסיף 1 מ"ל של DMEM/F12, מיד לאסוף את EBs עם פיפטה בורוסיליקט ולסנן אותם דרך מסננת.
- חזור על שלב 2.2.15 עד EBs כל יוסרו לצלחת תרבות microwell.
- היפוך של מסננת מעל צינור חרוטי 50 מ"ל ולהוסיף 2 מ של נים כדי לאסוף את כל EBs.
- לוחית רישוי 2 מ"ל של ההשעיה EB לבאר בודדת של צלחת מצופים BMM באמצעות פיפטה בורוסיליקט. דגירה של EBs ב 37 ° C עם 5% CO2.
- יום 6, להכין 2 מ"ל של נים + 200 ng/mL ששש (ראה טבלה של חומרים) ולבצע שינוי בינונית מדי יום.
- יום 8, לבחון את האחוז של אינדוקציה עצביים.
- לספור EBs מחובר ו, באופן ספציפי, לקבוע את מספר כל EB בודדים הממולא עם רוזטות עצבית. לכמת את אינדוקציה רוזט עצביים באמצעות הנוסחה הבאה.
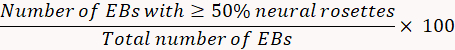
הערה: אם תנאי הגיוס עצבית < 75%, הבחירה רוזט עצבית עשוי להיות לא יעיל. - יום 12, להכין 250 מ של מדיום N2B27 119 מ של neurobasal בינוני, 119 מ של DMEM/F12 בינוני, 2.5 מ ל GlutaMAX, 2.5 מ ל NEAA, 2.5 מ של תוספת N2, 5 מ ל B27. ללא ויטמין A, μL 250 של גנטמיצין (50 מ"ג/מ"ל) , ו- 19. ΜL 66 של BSA (7 מ"ג/מ"ל).
- כדי להכין 50 מ של מדיום N2B27 מלאה, להוסיף 3 μM CHIR99021, 2 μM SB431542, bFGF 20 ng/mL, 20 ng/mL EGF ו 200 ng/mL ששש.
הערה: חשוב להכין את השלמת בינונית לפני השימוש. - האחות בינוני מן הבארות המכיל רוזטות עצבית, לשטוף עם 1 מ"ל של DMEM/F12.
- מודעה 1 מ"ל של ריאגנט הבחירה רוזט עצבית (ראה טבלה של חומרים) דגירה ב 37 ° C עם 5% CO2 לשעה.
- הסר בחירה הכימית, באמצעות pipettor של 1 מ"ל, מכוון ישירות האשכולות רוזט.
- להוסיף את המתלים צינור חרוטי 15 מ"ל וחזור על שלבים 2.2.25 ו- 2.2.26 עד רוב האשכולות רוזט עצבית שנאסף.
הערה: כדי למנוע זיהום, עם סוגי תאים nonneuronal, לא overselect. - Centrifuge התליה רוזט ב x 350 גרם במשך 5 דק. לשאוב את תגובת שיקוע, resuspend רוזטות עצבית N2B27 + 200 ng/mL ששש. להוסיף את המתלים רוזט עצבית לבאר, מצופים BMM, דגירה את הצלחת ב 37 ° C עם 5% CO2.
- ימים 13-17, לבצע שינוי בינונית מדי יום באמצעות בינוני N2B27 הושלמה. המעבר את התאים כאשר התרבויות confluent 80% – 90%.
- כדי לפצל את התאים, להכין צלחת BMM מצופים.
- לשטוף את התאים עם 1 מ"ל של DMEM/F12, תשאף המדיום ולהוסיף 1 מיליליטר דיסוציאציה ריאגנט (ראה טבלה של חומרים).
- תקופת דגירה של 5 דקות ב 37 מעלות צלזיוס, להוסיף 1 מ"ל של DMEM/F12, לסלק תאים המחוברים על ידי pipetting למעלה ולמטה. לאסוף את המתלים NPC צינור חרוטי 15 מ"ל. צנטריפוגה ב x 300 גרם במשך 5 דקות.
- וארוקן את תגובת שיקוע, resuspend תאי 1 מ"ל של N2B27 מלאה + 200 ng/mL ששש.
- לספור את התאים ואת צלחת אותם על צפיפות של 1.25 x5 10 תאים/cm2, דגירה התאים ב 37 ° C עם 5% CO2.
- לשנות את המדיום בכל יום אחר, באמצעות N2B27 מלאה + 200 ng/mL ששש.
הערה: -קטע זה, NPCs נחשבים מעבר (P) 0. ניתן למשוך ששש של המדיום N2B27-P2. - המעבר את התאים ברגע שיגיעו למפגש 80% – 90%.
- בשלב זה, לאשר כי התאים להביע סמנים Nestin ו- FoxA2 באמצעות immunocytochemistry ו- qPCR. פרוטוקול זה מוביל הדור של 85% תאי זוגית-חיובית עבור הסמנים Nestin ו- FoxA2.
- עבור passaging תאים, חזור על שלבים 2.2.31–2.2.36.
- להקפיא את התאים, החל מהמעבר P2. עבור הקפאת תאים, חזור על השלבים 2.2.31–2.2.36, resuspend בגדר תא-עונה 2 פרק 106 4 x 106 תאים למ"ל באמצעות קר קדמון עצבית קפוא בינוני (ראה טבלה של חומרים).
- להעביר 1 מ"ל של התליה תא לתוך כל cryovial, להקפיא את התאים באמצעות מערכת סטנדרטית קירור להאט את קצב-מבוקר. לאחסון לטווח ארוך, שמור את התאים חנקן נוזלי.
- מפשיר MD NPCs
- להכין צלחת מצופים BMM וחמים N2B27 מלאה. להוסיף 10 מ של DMEM חמים/F12 צינור חרוטי 15 מ"ל. המקום cryovial בתוך גוש חום 37 º C למשך 2 דקות.
- להעביר את התאים cryovial הצינור המכיל DMEM/F12. צנטריפוגה ב x 300 גרם במשך 5 דקות.
- וארוקן את תגובת שיקוע, resuspend תאי 2 מ של N2B27, ולהוסיף התליה תא טוב אחד של הלוח מצופה BMM. דגירה התאים ב 37 ° C עם 5% CO2.
- להכין צלחת מצופים BMM וחמים N2B27 מלאה. להוסיף 10 מ של DMEM חמים/F12 צינור חרוטי 15 מ"ל. המקום cryovial בתוך גוש חום 37 º C למשך 2 דקות.
3. התמרה חושית של MD NPCs וניתוח של מתילציה שינויים
-
התמרה חושית של MD NPCs
- מגלי MD NPCs ב-70% למפגש עם וקטורים LV-gRNA/dCas9-DNMT3A-MOI = 2. להחליף את posttransduction h 16 בינונית N2B27.
- הוסף N2B27 5 puromycin µg/mL, 48 שעות לאחר התמרה חושית. התרבות התאים למשך 3 שבועות לתוך N2B27 פלוס puromycin כדי להשיג את הקווים MD NPC יציב. התאים מוכנים עבור יישומי הזרם (ה-DNA, RNA, חלבון ניתוח, אפיון פנוטיפי23, קפוא, passaging).
-
אפיון לפרופיל מתילציה של אינטרון SNCA 1
- לחלץ את הדנ א של כל תא stably transduced קו באמצעות מיצוי הדנ א של קיט (ראה טבלה של חומרים).
- שימוש 800 ng של ה-DNA כדי לבצע המרה ביסולפאט, שימוש זמינים מסחרית קיט (ראה טבלה של חומרים). לאחר ההמרה ביסולפאט, elute ה-DNA ביסולפאט המרה כדי 20 ng/µL.
-
PCR לניתוח pyrosequencing
- הכנת המיקס מאסטר PCR נוקלאז ללא צינור. עבור כל תגובה, להשתמש µL 0.4 של פריימר הפוכה (10 מיקרומטר), µL 0.4 של פריימר לפנים (10 מיקרומטר), µL 1.6 של MgCl2 (25 מ מ), 2 µL של 10 x CoralLoad להתרכז, 10 µL של מיקס מאסטר x PCR 2, µL 4 5 x Q-פתרון, µL 1 של ה-DNA , ו- 0.6 µL של נוקלאז ללא מים.
- להעביר את הצלחת התגובה thermocycler ולבצע PCR תוך שימוש בתנאים הבאים: 95 מעלות צלזיוס למשך 15 דקות, 50 מחזורים של 94 ° C ל 30 s, 56 ° C ל 30 s, ו- 72 מעלות צלזיוס ל 30 s, בצעד האחרון מינימום 10 הרחבה-72 מעלות צלזיוס. תחל המשמש את pyrosequencing של SNCA אינטרון 1 מוצגים בטבלה 1, איור 7 או משלים איור 1.
- לאחר הגברה, דמיינו את amplicons באמצעות µL 2 של מוצר ה-PCR עם אתידיום ברומיד מכתים, הבאים agarose בג'ל.
- מבחני Pyrosequencing מאומתות באמצעות תערובות של unmethylated (U), מפוגל DNAs המרה ביסולפאט (ז) ב- היחס הבא, דהיינו 100U:0M, 75U:25M, 50U:50M, 25U:75M ו 0U:100M (ראה טבלה של חומרים).
- התנהגות pyrosequencing בעזרת ריאגנטים pyrosequencing (ראה טבלה של חומרים), לחשב את הערכים מתילציה עבור כל אתר CpG באמצעות תוכנת pyrosequencing. לקבלת פרוטוקול מפורט pyrosequencing, עיין Bassil et al.37.
Access restricted. Please log in or start a trial to view this content.
תוצאות
אימות של titers הייצור של הווקטורים LV-dCas9-DNMT3A-GFP/פורו בהשוואה לכנף GFP תמים
אנו לבצע p24מחסום פה אליסה כדי להשוות בין titers הפיזי של LV-dCas9-DNMT3A-GFP/פורו עם עמיתיהם GFP/פורו תמימה. התוצאות נציג, המוצגים באיור 5A, מדגימים כי ה?...
Access restricted. Please log in or start a trial to view this content.
Discussion
הזינו אותו החלו להסתמן כמו הרכב של epigenome עריכה, במיוחד בהקשר של מחלות גנטיות, בעיקר בגלל היכולת שלהם (i) להכיל מטענים דנ א גדולים ו- (ii) מגלי ביעילות מגוון רחב של חלוקת תאים nondividing. היעילות אריזה גדולה של אותו מועיל במיוחד עבור יישומים הכוללים אריזות של מערכות CRISPR/dCas9 אשר גדולות מדי. מנקודת מבט ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
אוניברסיטת דיוק הגיש בקשה לרישום פטנט זמני הקשורים במחקר זה.
Acknowledgements
עבודה זו הייתה מומן בחלקו על ידי בפרס הפיתוח Neurotechnology קאהן (כדי האו. סי) ואת נבחרת מוסדות של בריאות/לאומי המכון של הפרעות נוירולוגיות שבץ (NIH/NINDS) (NS085011 R01 כדי האו. סי).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| 0.22 μM filter unit, 1 L | Corning | 430513 | |
| 0.45-μm filter unit, 500 mL | Corning | 430773 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 50 mL conical centrifuge tubes | Corning | 430291 | |
| 6-well plates | Corning | 3516 | |
| Aggrewell 800 | StemCell Technologies | 34811 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| BD FACS | Becton Dickinson | 338960 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| Eppendorf Cell Imaging Slides | Eppendorf | 30742060 | |
| High-binding 96-well plates | Corning | 3366 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Reversible Strainer | StemCell Technologies | 27215 | |
| SW32Ti rotor | Beckman Coulter | 369650 | |
| VWR® Disposable Serological Pipets, Glass, Nonpyrogenic | VWR | 93000-694 | |
| VWR® Vacuum Filtration Systems | VWR | 89220-694 | |
| xMark™ Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| Accutase | StemCell Technologies | 7920 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| Anti-FOXA2 Antibody | Abcam | Ab60721 | |
| Anti-Nestin Antibody | Abcam | Ab18102 | |
| Antibiotic-antimycotic solution, 100x | Sigma Aldrich | A5955-100ML | |
| B-27 Supplement (50x), minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| BES | Sigma Aldrich | B9879 - BES | |
| Bovine Albumin Fraction V (7.5% solution) | Thermo Fisher Scientific | 15260037 | |
| CHIR99021 | StemCell Technologies | 72052 | |
| Corning Matrigel hESC-Qualified Matrix | Corning | 08-774-552 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| DMEM-F12 | Lonza | 12-719 | |
| DMEM, high glucose media | Gibco | 11965 | |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | |
| EpiTect PCR Control DNA Set | Qiagen | 596945 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Gentamicin | Thermo Fisher Scientific | 15750078 | |
| Gentle Cell Dissociation Reagent | stemCell Technologies | 7174 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Human Recombinant bFGF | StemCell Technologies | 78003 | |
| Human Recombinant EGF | StemCell Technologies | 78006 | |
| Human Recombinant Shh (C24II) | StemCell Technologies | 78065 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific | 11140050 | |
| mTeSR1 | StemCell Technologies | 85850 | |
| N-2 Supplement (100x) | Thermo Fisher Scientific | 17502001 | |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| PyroMark PCR Kit | Qiagen | 978703 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| SB431542 | StemCell Technologies | 72232 | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| STEMdiff Neural Induction Medium | StemCell Technologies | 5835 | |
| STEMdiff Neural Progenitor Freezing Medium | StemCell Technologies | 5838 | |
| TaqMan Assay FOXA2 | Thermo Fisher Scientific | Hs00232764 | |
| TaqMan Assay GAPDH | Thermo Fisher Scientific | Hs99999905 | |
| TaqMan Assay Nestin | Thermo Fisher Scientific | Hs04187831 | |
| TaqMan Assay OCT4 | Thermo Fisher Scientific | Hs04260367 | |
| TaqMan Assay PPIA | Thermo Fisher Scientific | Hs99999904 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| Y27632 | StemCell Technologies | 72302 | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Goat serum, Sterile, 10 mL | Sigma | G9023 | Working concentration 1:1000 |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Normal mouse serum, Sterile, 500 mL | Equitech-Bio | SM30-0500 | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| pMD2.G | Addgene | 12253 | |
| pRSV-Rev | Addgene | 52961 | |
| psPAX2 | Addgene | 12259 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsmBI | New England Biolabs | R0580S | |
| BsrGI | New England Biolabs | R0575S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
References
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gaj, T., Gersbach, C. A., Barbas, C. F. 3rd ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Thakore, P. I., Black, J. B., Hilton, I. B., Gersbach, C. A. Editing the epigenome: technologies for programmable transcription and epigenetic modulation. Nature Methods. 13 (2), 127-137 (2016).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Horlbeck, M. A., et al. Nucleosomes impede Cas9 access to DNA in vivo and in vitro. eLife. 5, (2016).
- Chavez, A., et al. Comparison of Cas9 activators in multiple species. Nature Methods. 13 (7), 563-567 (2016).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21 (3), 440-446 (2018).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Holtzman, L., Gersbach, C. A. Editing the Epigenome: Reshaping the Genomic Landscape. Annual Review of Genomics and Human Genetics. , (2018).
- Perez-Pinera, P., et al. Synergistic and tunable human gene activation by combinations of synthetic transcription factors. Nature Methods. 10 (3), 239-242 (2013).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nature Methods. 12 (12), 1143-1149 (2015).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- McDonald, J. I., et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biology Open. 5 (6), 866-874 (2016).
- Huang, Y. H., et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biology. 18 (1), 176(2017).
- Vojta, A., et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research. 44 (12), 5615-5628 (2016).
- Razin, A., Kantor, B. DNA methylation in epigenetic control of gene expression. Progress in Molecular and Subcellular Biology. 38, 151-167 (2005).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18786-18791 (2009).
- Kantor, B., et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Molecular Therapy. , (2018).
- Jowaed, A., Schmitt, I., Kaut, O., Wullner, U. Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. Journal of Neuroscience. 30 (18), 6355-6359 (2010).
- Wang, Y., et al. A DNA methyltransferase inhibitor, 5-aza-2'-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson's disease-related genes in dopaminergic neurons. CNS Neuroscience & Therapeutics. 19 (3), 183-190 (2013).
- Matsumoto, L., et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson's disease. PLOS One. 5 (11), e15522(2010).
- Desplats, P., et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel mechanism for epigenetic alterations in Lewy body diseases. Journal of Biological Chemistry. 286 (11), 9031-9037 (2011).
- Ai, S. X., et al. Hypomethylation of SNCA in blood of patients with sporadic Parkinson's disease. Journal of the Neurological Sciences. 337 (1-2), 123-128 (2014).
- Tagliafierro, L., Chiba-Falek, O. Up-regulation of SNCA gene expression: implications to synucleinopathies. Neurogenetics. 17 (3), 145-157 (2016).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Molecular Therapy - Methods & Clinical Development. 5, 153-164 (2017).
- Tagliafierro, L., et al. Genetic analysis of alpha-synuclein 3' untranslated region and its corresponding microRNAs in relation to Parkinson's disease compared to dementia with Lewy bodies. Alzheimer’s & Dementia. 13 (11), 1237-1250 (2017).
- Tagliafierro, L., Zamora, M. E., Chiba-Falek, O. Multiplication of the SNCA locus exacerbates neuronal nuclear aging. Human Molecular Genetics. , (2018).
- Kantor, B., McCown, T., Leone, P., Gray, S. J. Clinical applications involving CNS gene transfer. Advances in Genetics. 87, 71-124 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Advances in Genetics. 87, 125-197 (2014).
- Vijayraghavan, S., Kantor, B. A Protocol for the Production of Integrase-deficient Lentiviral Vectors for CRISPR/Cas9-mediated Gene Knockout in Dividing Cells. Journal of Visualized Experiments. (130), e56915(2017).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Molecular Therapy. 16 (12), 1968-1976 (2008).
- Bassil, C. F., Huang, Z., Murphy, S. K. Bisulfite pyrosequencing. Methods in Molecular Biology. 1049, 95-107 (2013).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Research. 43 (13), 6450-6458 (2015).
- Nishimasu, H., et al. Crystal Structure of Staphylococcus aureus Cas9. Cell. 162 (5), 1113-1126 (2015).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. Journal of Virology. 68 (4), 2632-2648 (1994).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. Journal of Virology. 71 (8), 6113-6127 (1997).
- Goffin, V., et al. Transcription factor binding sites in the pol gene intragenic regulatory region of HIV-1 are important for virus infectivity. Nucleic Acids Research. 33 (13), 4285-4310 (2005).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. Journal of Biological Chemistry. 280 (22), 21545-21552 (2005).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Molecular Therapy. 19 (3), 547-556 (2011).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved