A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הערכה של סרטן המעי הגס סיכון ושכיחות על ידי שרפרף DNA זיהוי שלמות
In This Article
Erratum Notice
Summary
ערכת האבחון המוצגת חליל-DNA היא שיטת חיסכון בזמן וידידותי למשתמש כדי לקבוע את ההסתברות האמינה של נוכחות של נגעים סרטן המעי הגס.
Abstract
בימינו, שרפרף DNA יכול להיות מבודד ומנותח על ידי מספר שיטות. שרידים ארוכים של דנ א בצואה ניתן להבחין על ידי הספק qPCR, המספק סבירות אמינה לנוכחות של נגעים טרום הנאופלסטית או נאופלסטית המעי הגס. שיטה זו, המכונה DNA ארוך הזריחה (FL-DNA), הוא הליך מהיר, פולשני שהוא שיפור על מערכת המניעה העיקרית. שיטה זו מבוססת על הערכה של שלמות ה-DNA צואה על ידי הגברה כמותית של יעדים ספציפיים של דנ א גנומית. בפרט, הערכה של שברי DNA יותר מ 200 bp מאפשר זיהוי של חולים עם נגעים המעי הגס עם ספציפיות מאוד. עם זאת, מערכת זו וכל בדיקות ה-DNA שרפרף הזמין כרגע להציג כמה בעיות כלליות שצריך להיות ממוען (למשל, את התדר שבו יש בדיקות צריך להתבצע מספר אופטימלי של שרפרף שנאסף בכל נקודת זמן עבור כל אדם). עם זאת, היתרון העיקרי של FL-DNA היא האפשרות להשתמש בו בשיתוף עם מבחן המשמש כיום בתוכנית ההקרנה CRC, המכונה כימיקלים מבוססי חיסוני בדיקת דם סמוי (iFOBT). אכן, שתי בדיקות ניתן לבצע על אותו מדגם, הפחתת עלויות והשגת חיזוי טוב יותר של נוכחות בסופו של דבר של נגעים המעי הגס.
Introduction
סרטן המעי הגס (CRC) נובע תהליך רב שלב שבו האפיתל בריא לאט מתפתחת לתוך אדנומות או פוליפים, אשר התקדמות קרצינומות ממאירים לאורך זמן1,2. למרות שיעור השכיחות הגבוה של CRC, מגמה כלפי מטה באחוז מקרי המוות נצפתה בעשור האחרון3. אכן, כלים אבחון מוקדם שאומצה תוכניות הקרנה הובילו גילוי מוקדם והסרה של אדנומות טרום הנאופלסטית או פוליפים4. עם זאת, בשל המגבלות הטכניות השונות, אף אחת מהשיטות הללו אינה אופטימלית. ואכן, כדי לשפר את הרגישות והספציפיות, הוצעו בדיקות דנ א שרפרף רבות בלבד או בשילוב עם בדיקות אבחון5שגרתיות 5,6.
בדרך כלל, רירית בריאה שופך את זרם הצואה האפוציטים, בעוד רירית החולה מפילינג הקולונציטים בלתי אפוטוטיות. שברים של 200 bp או יותר באורך לאפיין את ה-DNA לא אפוטוטית. ה-DNA הזה נקרא DNA ארוך (L-DNA) והפך utilizable ביוקרה לאבחנה מוקדמת CRC. ה-dna יכול להיות מבודד מדגימת צואה וכימות על ידי qpcr באמצעות בדיקת מחוץ למערכת האבחון של מבחנה-dna בערכה7,8,9,10,11,12.
הבדיקה מורכבת משני שאומר לאיתור שברי FL-DNA החל 138 bp כדי 339 bp. כל שיטת הזמן מאפשרת הגברה של FL-DNA (משפחה) כמו גם ספייק-DNA (HEX). כדי להבטיח הגברה אופטימלית של כל השברים, הבדיקה חולקה לשתי שמות (בשם "A" ו-"B"). השיטת A מזהה שני אזורים של אקסון 14 של גן הנגמ ש (NM_001127511) וחלק של אקסון 7 של הגן TP53 (NM_001276760). שיטת B מזהה קטע של אקסון 14 של הנגמ ש (NM_001127511) ושני אזורים של אקסון 5 ו-8 של הגן TP53 (NM_001276760). ההסכמה אינה מבדילה בין האזורים שזוהו. ה-DNA של הדקר מתאים ל-DNA הoncorהלופון הסלמון מאפשר אימות כי ההליך נעשה כראוי ובודק את הנוכחות האפשרית של מעכבי, אשר עשוי להניב תוצאות שליליות שווא. הריכוז חליל-DNA מוערך על ידי קוונפיקציה מוחלטת באמצעות שיטת העקומה הסטנדרטית והוא מתבטא כ ng/התגובה.
השיטה FL-dna היא לא פולשנית וזולה שרפרף DNA מבחן, כי, בשילוב עם מבוסס מבחינה כימית חיסוני בדיקת דם סמוי (ifobt), משמש כיום תוכניות הקרנת crc ומאפשר תחזיות טוב יותר של CRC ו/או בסיכון גבוה הנגעים אדנומה12.
Protocol
המטופלים גויסו בבית החולים הקונטולוו רומאנולו לולו סטודיו e לה דה טוורי (IRST) של מלנדולה (FC, איטליה) בין 2013 ל 2015. חולים נרשמו היו לתוך פרוטוקול IRSTB002, אושרה על ידי ועדת האתיקה של IRST-IRCCS AVR (25/10/2012, ver .1). כל השיטות בוצעו בהתאם להנחיות ולתקנות רלוונטיות. הסכמה מושכלת בכתב הושגה מכל המטופלים.
1. חילוץ דנ א מצואה
- השתמשו בערכה להכנת דגימות צואה (ראו טבלת חומרים). בחר וטפל בחומר הצואה על-ידי ביצוע החילוץ בהתאם להוראות היצרן. להגביר את הדנ א הטהור ישירות או לאחסן ב-20 ° c לניתוח הבא.
2. הכנת בקרה חיובית, סטנדרטים, ספייק-DNA, ודגימות קליניות
- הכנת תקנים ודגימות
- כדי להכין את השליטה החיובית, סטנדרטים, ספייק-ב-DNA, ואת כל הדגימות הקליניות, צנטריפוגה מראה של שליטה חיובית, תקנים, ו-DNA-ב, לאחר מכן להשעות מחדש כל מגיב על ידי הוספת כמות נכונה של מים שסופקו (ראה להלן). ואז, מערבולת בזהירות את השליטה החיובית, סטנדרטי, ו-DNA-in-in, ולאחר מכן צנטריפוגה עבור 10 s. כדי להשיג השעיה מלאה של ריאגנטים יבש, לאחסן את ריאגנטים נוזלי בטמפרטורת החדר (RT) עבור 30 דקות לפני השימוש.
- השליטה החיובית היא דנ א אנושי בפורמט יבש. השהה מחדש כל נורית עם 750 μL של מים.
- ה-DNA של הדקר הוא סלמון (Oncorהלופון keta) dna, אשר משמש כפקד פנימי אקסוסוגני כדי לאמת את הנוכחות האפשרית של מעכבי בדגימות DNA שחולצו מצואה. השהה מחדש כל נורית עם 100 μL של מים.
- כדי להכין את עקומת התקן, ליצור ארבעה 1:5 מדלל החל מהפתרון במניה. הנקודות הסטנדרטיות חייב להיות 10 ng/התגובה, 2 ng/התגובה, 0.4 ng/התגובה, ו 0.08 ng/התגובה.
- כדי להכין את השליטה החיובית, סטנדרטים, ספייק-ב-DNA, ואת כל הדגימות הקליניות, צנטריפוגה מראה של שליטה חיובית, תקנים, ו-DNA-ב, לאחר מכן להשעות מחדש כל מגיב על ידי הוספת כמות נכונה של מים שסופקו (ראה להלן). ואז, מערבולת בזהירות את השליטה החיובית, סטנדרטי, ו-DNA-in-in, ולאחר מכן צנטריפוגה עבור 10 s. כדי להשיג השעיה מלאה של ריאגנטים יבש, לאחסן את ריאגנטים נוזלי בטמפרטורת החדר (RT) עבור 30 דקות לפני השימוש.
- הכנת ה-DNA של הדקר הראשונה
- הכן את בקרת ה-DNA של הדקר ישירות לפני השימוש.
- הכן את 1 x ספייק בקרת דנ א על ידי ערבוב 5 μL של FL-DNa ספייק עם 20 μL של מים סטריליים. מספר של 1 x ספייק דגימות בקרת DNA יהיה מוכן על פי מספר הדגימות שיש לנתח, בתוספת שליטה חיובית.
- הכנת דגימות
- מערבבים 75 μL של דגימות (דגימות קליניות או בקרה חיובית) עם 25 μL של 1 x ספייק-ב-DNA, מניב נפח כולל של 100 μL.
3. הגברה ונחישות של הערך FL-DNA באמצעות qPCR Easy PGX
הערה: תערובות הגברה מלאות המכילות את התחל והבדיקות הספציפיות המכוונות ל-dna האנושי והשליטה הפנימית מסופקים בתבנית ליטליבית ב -8 רצועות היטב לתמהיל fl-dna mix a ו-fl-dna mix B. תקנים, שליטה חיובית ושלילית, והדגימות חייבות להיות מוגברה עם תערובות ליטו דגימות קליניות רק צריך להיות מוגבר רק בשכפול עם שני מתערבב ליקומי.
- עיין בטבלת החומרים של הכלי ותוכנת ההפעלה של qpcr.
- פתח את תוכנת ההפעלה והגדר את הלוחית והפרופיל התרמי:
- הגדר את הצלחת כפי שמוצג בטבלה 1.
- הגדר את סוג הבאר עבור כל שמונת המיקומים בעמודה 1 כרגיל.
- הגדר את סוג הבאר עבור הבארות A2 ו-B2 כ- Ntc.
- הגדר את סוג הבאר עבור C2 ו-D2 (הפקדים החיוביים) כבלתי ידועים.
- הגדר את סוג הבאר עבור כל המיקומים האחרים כבלתי ידועים.
- בחר את כל המיקומים 96, ולהוסיף את משפחה צבע הקסדצימאלי. לחץ על לוח סנכרון.
- הגדר את הפרופיל התרמי לפי טבלה 2.
- הגדר את הצלחת כפי שמוצג בטבלה 1.
- צנטריפוגה את המספר הדרוש של רצועות עבור 10 s כדי להביא את התוכן לתחתית הצינור.
- להסיר בעדינות את חותמות מן הרצועות, תוך כדי לשים לב לשמור את התוכן, ולהוסיף את הרצועות המתאימות: שליטה שלילית: 20 μL של מים; לדוגמה: 20 μL של דנ א; עקומת סטנדרטי: 20 μL של תקן 1, 2, 3 או 4; שליטה חיובית: 20 μL של שליטה חיובית.
- סגור בקפידה את כל הרצועות באמצעות 8 רצועה כמוסות אופטיות שטוחות ומערבולת לכמה שניות.
- צנטריפוגה את הרצועות עבור 10 ולטעון אותם לתוך המכשיר. . אז תתחיל לרוץ
- פתח את תוכנת ההפעלה והגדר את הלוחית והפרופיל התרמי:
4. ניתוח נתונים
הערה: ניתן לבצע ניתוח נתונים באופן אוטומטי או ידני בהתאם לתוכנה (ראה טבלת חומרים).
- בסוף ההפעלה, בחר עמודות A, C, E, G עבור "משפחה: FL-DNA-A" ו "HEX: IC", ועמודות B, D, F, H עבור "המשפחה: FL-DNA-B" ו-"hex: IC".
- הגדר את הפרטים הבאים עבור כמות ההתחלתי של כמויות: 10 ng/התגובה עבור בארות A1 ו-B1, 2 ng/תגובה עבור C1 ו-D1, 0.4 ng/התגובה עבור E1 ו F1, ו 0.08 ng/התגובה עבור G1 ו-H1.
- הגדר ערכי הזריחה של הסף ל 150 משפחה (FL-dna A ו-FL-dna B) ו-HEX (IC) ערוצים.
- בתיבה תוצאות טבלה, לחץ על אפשרויות עמודה | בחר הכל | אישור כדי לקבל את התוצאות בשני הערוצים עם cq המתאים שלהם (∆ R) ו ∆ r הערכים האחרונים.
הערה: ערכים אלה מסופקים על-ידי תוכנת הכלי של מכשיר ה-PCR בזמן אמת. ΔR האחרון מתייחס לערך הזריחה המנורמל למחזור ההגברה האחרון. - בתיבה תוצאת טבלה, לחץ לחיצה ימנית על הטבלה כדי לפתוח את התפריט תלוי ההקשר ולחץ על שלח ל-Excel כדי לייצא את הנתונים הגולמיים.
- בדוק את ערכי התקנים כדי לאמת את ההתאמה של העקומה הסטנדרטית.
- עבור כל שילוב FL-DNA, לבדוק את R2 ["r ² (∆ R)" עמודה] ויעילות ["יעילות (%)" עמודה] ערכים. אם הם נמצאים בטווח מקובל, ניתן להמשיך בניתוח בהתאם להוראות היצרן (שולחן 3).
- אם תוצאות ערוץ ה-משפחה אינן נמצאות בטווח הצפוי, השמט נקודה אחת של העקומה הרגילה והפעל מחדש את ההפעלה.
- קבע את ערכי הפקדים השליליים והחיוביים עם הנוסחה הבאה, בהתחשב בערכים "No Cq" כאפס:
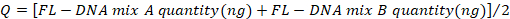
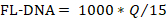
- השווה בין הערכים המתקבלים לאלה המדווחים בטבלה 4.
- אם שולטת התגובה הם בטווח של ערכים צפויים, להמשיך עם ניתוח של דגימות.
הערה: ודא שערכי Cq שהתקבלו מופקים מתגובת הגברה אמיתית (עקומת פלואורסצנטית מסיבית) ולא מחפץ (עקומת פלואורסצנטית לינארית). - כדי לנתח את ההתאמה של המדגם עבור כל שילוב FL-DNA, להשוות את ערכי Cq של ערוץ HEX. אם הערך הוא ≥ 16, המשך בניתוח הדגימות. אם הערך הוא < 16 או שאין Cq, סביר להניח שהסיבה לכך היא שגיאה מחוץ לספייק-DNA. לכן, לא ניתן לנתח את הדגימות.
- חשב את הממוצע של ערכי Cq בערוץ "HEX" של הפקד החיובי באמצעות הנוסחה הבאה:

- חשב את הממוצע של ערכי Cq בערוץ "HEX" של המדגם הכפול באמצעות הנוסחה הבאה:
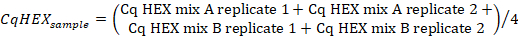
- חשב את הערכים ∆ CqHEX בהתאם לנוסחה הבאה:
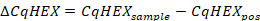
- השווה את ערכי ∆ CqHEX של הדגימות עם אלה שדווחו בטבלה 5.
- עבור כל תערובת (Mix A ו-Mix B), השווה את ערכי ה-Cq של ערוץ המשפחה עם אלה המדווחים בטבלה 6.
- כדי לקבוע את ערך FL-DNA של כל דוגמה מתאימה, השתמש בנוסחה הבאה, בהתחשב בערכי "No Cq" כאפס:
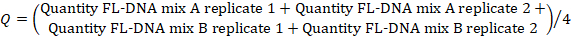
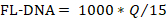
הערה: הסיכון לסרטן המעי הגס ושכיחות היא פונקציה של הערכות iFOBT ו-FL-DNA על פי התוצאות nomogram Fagan שהתקבלו על ידי Rengucci ואח '12 (שולחן 7).
תוצאות
זרימת העבודה של פרוטוקול זה מוצגת באיור 1. זרימת העבודה מספקת שני צעדי בקרה ופעולות שונות בהתאם לתוצאות שלב אלה. ראשית, אם מדגם מציג פקדים לא מתאימים, יש לחזור על הגברה. שנית, אם הגברה מעכבות, יש לעבד את המדגם מההתחלה או לסווג כערך לא יקר.
איור 2 מציג את עקומות ...
Discussion
מחקרים קודמים הוכיחו כי שלמות DNA ניתוח של שרפרפים שחולצו על ידי גישות ידניות וחצי אוטומטי יכול לייצג כלי אלטרנטיבי לגילוי מוקדם של נגעים המעי הגס7,8,9,10,11,12. מולקולרית, בדיקות ההקרנה ?...
Disclosures
מאורה מנאגי הוא עובד במשרה מלאה of Diatech פרמקוגנטיקה srl.
Acknowledgements
למחברים אין ותודות.
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL polypropylene twist-lock tubes (DNase-, RNase-, DNA-, PCR inhibitor-free) | Consumables required for DNA extraction and Real Time PCR | ||
| Absolute Ethanol (quality of analytical degree) | Reagent required for DNA extraction | ||
| Benchtop centrifuge | Maximum speed of 20000 x g. Instrument required for DNA extraction | ||
| EasyPGX analysis software version 2.0.0 | Diatech Pharmacogenetics | RT800-SW | Analysis software |
| EasyPGX centrifuge/vortex 8-well strips | Diatech Pharmacogenetics | RT803 | Instrument recommended for the Real Time PCR assay |
| EasyPGX qPCR instrument 96 | Diatech Pharmacogenetics | RT800-96 | Instrument recommended for the Real Time PCR assay |
| EasyPGX ready FL-DNA | Diatech Pharmacogenetics | RT029 | Kit required for the Real Time PCR assay |
| Micropipettes (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Powder-free disposable gloves | Consumables required for DNA extraction and Real Time PCR | ||
| QIAamp Fast DNA Stool | Qiagen | 51604 | Kit recommended for the DNA extraction and purification from stool |
| Sterile filter tips DNase-, RNase-free (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Thermal block e.g. EasyPGX dry block | Diatech Pharmacogenetics | RT801 | Instrument required for DNA extraction |
| Vortex e.g. EasyPGX centrifuge/vortex 1.5 ml | Diatech Pharmacogenetics | RT802 | Instrument required for DNA extraction |
References
- Fearon, E. R. Molecular Genetics of Colorectal Cancer. Annual Review of Pathology. 6, 479-507 (2011).
- Sears, C. L., Garrett, W. S. Microbes, Microbiota, and Colon Cancer. Cell Host and Microbe. 15, 317-328 (2014).
- Levin, B., et al. Screening and Surveillance for the Early Detection of Colorectal Cancer and Adenomatous Polyps, 2008: A Joint Guideline From the American Cancer Society, the US Multi-Society Task Force on Colorectal Cancer, and the American College of Radiology. Gastroenterology. 134, 1570-1595 (2008).
- Bosch, L. J., et al. Molecular tests for colorectal cancer screening. Clinical Colorectal Cancer. 10, 8-23 (2011).
- Ahlquist, D. A. Molecular detection of colorectal neoplasia. Gastroenterology. 138, 2127-2139 (2010).
- Calistri, D., et al. Fecal multiple molecular tests to detect colorectal cancer in stool. Clinical Gastroenterology and Hepatology. 1, 377-383 (2003).
- Calistri, D., et al. Detection of colorectal cancer by a quantitative fluorescence determination of DNA amplification in stool. Neoplasia. 6, 536-540 (2004).
- Calistri, D., et al. Quantitative fluorescence determination of long-fragment DNA in stool as a marker for the early detection of colorectal cancer. Cellular Oncology. 31, 11-17 (2009).
- Calistri, D., et al. Fecal DNA for noninvasive diagnosis of colorectal cancer in immunochemical fecal occult blood test-positive individuals. Cancer Epidemiology Biomarkers and Prevention. 19, 2647-2654 (2010).
- De Maio, G., et al. Circulating and stool nucleic acid analysis for colorectal cancer diagnosis. World Journal of Gastroenterology. 20, 957-967 (2014).
- Rengucci, C., et al. Improved stool DNA integrity method for early colorectal cancer diagnosis. Cancer Epidemiology Biomarkers and Prevention. 23, 2553-2560 (2014).
Erratum
Formal Correction: Erratum: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection
Posted by JoVE Editors on 9/28/2020. Citeable Link.
An erratum was issued for: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection. An affiliation was updated.
The first affiliation was updated from:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST)
to:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) IRCCS, Meldola, Italy
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved