A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
צינור ביואינפורמטיקה לנתח באופן מדויק ויעיל את המייקרואורפה בצמחים
* These authors contributed equally
In This Article
Summary
קו ביואינפורמטיקה, כלומר miRDeep-P2 (miRDP2 בקיצור), עם קריטריונים מעודכנים של הצמח Mirdeep ואלגוריתם מקוצר, יכול לנתח באופן מדויק וביעילות מיקרוסקריפט בצמחים, במיוחד עבור מינים עם genomes מורכבים וגדולים.
Abstract
MicroRNAs (miRNAs) הם 20-עד 24-נוקלאוטיד (nt) RNAs קטנים (sRNAs) הקיימים באופן מקיף בצמחים ובעלי חיים המשחק תפקידים חזקים בוויסות ביטוי הגנים ברמה שלאחר ההמרה. רצפי הספריות sRNA לפי רצף הדור הבא (NGS) שיטות המועסקים באופן נרחב כדי לזהות ולנתח miRNA הטרנססקריפט בעשור האחרון, והתוצאה היא עלייה מהירה של גילוי miRNA. עם זאת, שני אתגרים עיקריים נובעים הביאור miRNA הצמח בשל הגדלת עומק של ספריות sRNA רצף, כמו גם את הגודל והמורכבות של הצמח genomes. ראשון, סוגים רבים אחרים של sRNAs, בפרט, RNAs מתערב קצר (siRNAs) מספריות Srnas, הם מסומן בטעות כמו miRNAs על ידי כלים חישוביים רבים. שנית, זה הופך להיות תהליך רב למדי זמן לניתוח miRNA הטרנססקריפט במינים צמחיים עם genomes גדולים ומורכבים. כדי להתגבר על האתגרים הללו, שודרגו לאחרונה miRDeep-P (כלי פופולרי עבור הפעלת ניתוח Mirdeep) כדי miRDeep-P2 (miRDP2 בקיצור) על-ידי שימוש באסטרטגיית סינון חדשה, בגרירת האלגוריתם הבקיע ושילוב הצמח החדש שעודכן Mirdeep קריטריוני ביאור. בדקנו miRDP2 נגד אוכלוסיות sRNA בחמישה צמחים מייצגים עם הגדלת מורכבות גנומית, כולל Arabidopsis, אורז, עגבניה, תירס וחיטה. התוצאות מעידות על כך שmiRDP2 עיבד משימות אלה ביעילות גבוהה מאוד. בנוסף, miRDP2 ביצעו כלים אחרים חיזוי לגבי רגישות ודיוק. יחד, התוצאות שלנו להפגין miRDP2 ככלי מהיר ומדויק עבור ניתוח הצמח miRNA transcript, ולכן כלי שימושי לסייע לקהילה ביאורים טובים יותר Mirna בצמחים.
Introduction
אחת התגליות המרגשות ביותר בשני העשורים האחרונים בביולוגיה היא התפקיד המוחלק של מינים sRNA בוויסות פונקציות מגוונות של הגנום1. במיוחד, mirnas מהווה מחלקה חשובה של 20-ל 24-nt srnas ב eukaryotes, ובעיקר לתפקד ברמה post-transcript כמו הרגולטורים גנים בולטים במהלך שלבי פיתוח מחזור חיים, כמו גם בתגובות גירוי וסטרס2,3. ב צמחים, mirnas נובעים התעתיקים העיקריים בשם פרי-mirnas, אשר משותילים בדרך כלל על-ידי RNA פולימראז II כיחידות תמלול בודדות4,5. מעובד על ידי מכונות סלולר שימור אבולוציונית (drosha rnase III ב בעלי חיים, dicer כמו צמחים), פרי-mirnas הם מרוטרים לתוך מיידית precursors מוקדם, pre-mirnas, אשר מכילים רצפים להרכיב גזע פנים מולקולרי מבנים6,7. Pre-mirnas מעובדים מכן לתוך intermediates כפול תקוע, כלומר דופלקסים mirnas, המורכב סטרנד פונקציונלי, בוגרת mirnas, ואת שותף פחות פונקציונלי, mirnas *2,8. לאחר שנטען לתוך מתחם ההשתקה המושרה RNA (risc), mirnas בוגרת יכול לזהות מטרות mrna שלהם מבוסס על משלימה רצף, וכתוצאה מכך פונקציה תקינה שלילית2,8. mirnas יכול לערער את התעתיקים היעד שלהם או למנוע תרגום יעד, אבל הדרך הקודמת נשלטת בצמחים8,9.
מאז התגלית של מירנה הראשונה באתר המלון 10,11, מחקרים רבים כבר מחויבת לזיהוי mirna וניתוח פונקציונלי שלה, במיוחד לאחר הזמינות של שיטת ngs. היישום רחב של שיטת NGS קידם במידה רבה את הניצול של כלים חישוביים שתוכננו ללכוד את התכונה הייחודית של miRNAs, כגון מבנה לולאה גזע של טרום סמנים הצטברות שלהם מועדפים של רצף קורא על Mirnas בוגרת Mirnas *. כתוצאה מכך, החוקרים השיגו הצלחה יוצאת דופן בזיהוי miRNAs במינים שונים. בהתבסס על מודל הסתברות שתוארה בעבר12, פיתחנו Mirdeep-P13, שהיה כלי החישוב הראשון לגילוי mirdeep הצמח מתוך נתונים ngs. mirdeep-P הייתה מיועדת במיוחד לכבוש את האתגרים של פענוח צמח mirdeep שמציעות יותר משתנה באורך מקודparalogous גדול משפחות13,14,15. לאחר השקתו, תוכנית זו כבר הורידה אלפי פעמים והשתמשו כדי להוסיף ביאורים miRNA הטרנססקריפט ביותר מ 40 מינים צמח16. מונעת על ידי כלים מבוססי NGS כמו miRDeep-P, יש כבר עלייה דרמטית במספר של Mirdeep רשום במאגר Mirdeep הציבור Mirdeep17, שם מעל 38,000 מוצרים mirdeep מתארחים כעת (שחרור 22.1) בהשוואה רק ~ 500 פריטים mirdeep (שחרור 2.0) ב 200818.
עם זאת, שני אתגרים חדשים עלו מן הצמח miRNA ביאור. ראשון, היחס הגבוה של שווא-חיוביים השפיעו בכבדות על איכות של צמח mirna ביאורים16,19 מהסיבות הבאות: 1) מבול של rnas קצר הפרעה (sirnas) מ-ngs srna ספריות היו מסומן בטעות כמו mirna בשל חוסר קריטריונים מחמירים mirna ביאור; 2) עבור מינים ללא מידע מירנה פריורי, שווא-תוצאות החזוי בהתבסס על נתוני NGS קשה לחסל. באמצעות miRBase כדוגמה, טיילור ואח '20 נמצא שליש של הצמח מירנה ערכים במאגר הציבורי21 (שחרור 21) לא היה משכנע הראיות התומכות אפילו שלושה-ארבעה של משפחות צמח mirbase היו בספק. שנית, זה הופך להיות תהליך רב למדי זמן לחיזוי mirnas צמח עם גנום גדול ומורכב16. כדי להתגבר על האתגרים הללו, התעדכנו miRDeep-P על-ידי הוספת אסטרטגיית סינון חדשה, משיכת יתר של אלגוריתם הניקוד ושילוב קריטריונים חדשים עבור צמח Mirdeep ביאור, ושוחרר גירסה חדשה miRDP2. בנוסף, בדקנו miRDP2 באמצעות NGS sRNA נתונים עם הגדלת בהדרגה הגנום גדלים: Arabidopsis, אורז, עגבניה, תירס וחיטה. לעומת אחרים חמישה כלים בשימוש נרחב הגירסה הישנה שלה, miRDP2 נותחו אלה נתונים sRNA וניתח miRNA ההמרה מהר יותר עם דיוק ורגישות משופרת.
תוכן חבילת miRDP2
חבילת miRDP2 מורכב משש סקריפטים מתועדים Perl כי יש להפעיל ברציפות על ידי סקריפט bash מוכן. מתוך ששת התסריטים, שלושה (convert_bowtie_to_blast. pl, filter_alignments. plו- excise_candidate. pl) עוברים בירושה מ-mirdeep-P. הסקריפטים האחרים משתנים מהגירסה המקורית. פונקציות של ששת הסקריפטים מתוארות בהמשך:
preprocess_reads. pl מסננת קורא, כולל קריאות שארוך מדי או קצר מדי (< 19 nt או > 25 nt), וקורא בקורלציה עם רצפי Rfam ncRNA, כמו גם קורא עם RPM (קורא לכל מיליון) פחות מ 5. קובץ ה-script מאחזר קריאות בקורלציה לרצפים מבוגרים ידועים של miRNA. קובצי הקלט הם קריאות מקוריות בתבנית FASTA/FASTQ ו-bowtie2 פלט של קריאות מיפוי לרצפי miRNA ו-ncRNA.
הנוסחה לחישוב סל ד היא כדלקמן:
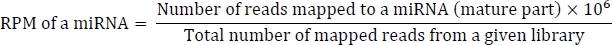
convert_bowtie_to_blast. pl משנה את תבנית עניבת הקשת לתוך תבנית מנותח הפיצוץ. תבנית שנותחה הפיצוץ היא תבנית מותאמת אישית מופרדת טבלאית הנגזרת מתבנית תקן של NCBI.
filter_alignments. pl מסננת את המערכים של ברצף מעמיק קורא הגנום. הוא מסנן מערכים חלקיים, כמו גם קריאות מיושרות מרובות (סף תדירות שצוין על-ידי המשתמש). הקלט הבסיסי הוא קובץ בתבנית שנותחה הפיצוץ.
excise_candidate. pl גוזר רצפי מספרים פוטנציאליים מרצף התייחסות באמצעות היישור מקריא כהנחיות. הקלט הבסיסי הוא קובץ בתבנית שנותחה הפיצוץ וקובץ FASTA. הפלט הוא כל רצפי מקודמן פוטנציאליים בפורמט FASTA.
mod-miRDP.pl זקוק לשני קבצי קלט, קובץ חתימה מבנה קובץ, אשר שונה מן האלגוריתם mirdeep-P הליבה על ידי שינוי מערכת הניקוד עם פרמטרים מסוימים הצמח. קבצי הקלט הם קובץ מבנה המבנה הקודמי וקורא קובץ חתימת הפצה.
mod-rm_redundant_meet_plant. pl זקוק לשלושה קבצי קלט: chromosome_length, סמנים מוקדמים וoriginal_prediction שנוצרו על-ידי mod-miRDP.pl. הוא מייצר שני קבצי פלט, קובץ חזוי שאינם יתירים קובץ החזוי מסוננים על ידי החדש העדכון קריטריונים miRNA הצמח. פרטים על תבנית קובץ הפלט מתוארים בסעיף 1.4.
Protocol
1. התקנה ובדיקה
- הורד את יחסי התלות הדרושים: Bowtie222 ו-RNAfold23. מומלץ לעשות חבילות שעברו קומפילציה.
- הורד Bowtie2, כלי מיפוי קריאה, מהאתר הביתי שלה (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml).
- הורד RNAfold, כלי של חבילת וינה המשמש לחיזוי המבנה המשני של RNA, מ http://www.tbi.univie.ac.at/~ivo/RNA/.
- לפני התקנת miRDP2, ודא ששני יחסי התלות האלה מותקנים כראוי והתאם אישית את קובץ סביבת הbash (לדוגמה,. ba,) כדי להגדיר נתיב נכון עבור שתי יחסי תלות אלה.
הערה: כלי מיפוי אחרים כגון Bowtie24 מתאימים גם לmiRDP2; או Bowtie או Bowtie2 ניתן להשתמש לאחר גירסה 1.1.3.
- כדי להוריד את חבילת miRDP2, עבור אל https://sourceforge.net/projects/mirdp2/files/latest_version/ ולהביא את הקבצים אוסך.
- לפני התקנת miRDP2, ודא Perl בנתיב. כדי להתקין את miRDP2, חלץ את כל התוכן של קובץ ה-אוסך שהורדת לתיקייה אחת (שורות פקודה כמו ב-1.4.2) ולאחר מכן הגדר את נתיב התיקיה לתוך הנתיב.
הערה: צומת מחשב או מיחשוב עם לפחות 8 GB זיכרון RAM ו 100 GB מומלץ להפעיל miRDP2. - בדוק את צינור MiRDP2.
- כדי לבדוק אם miRDP2 הותקן כראוי, השתמש בנתוני הבדיקה ובפלט הצפוי שנמצא ב- https://sourceforge.net/projects/mirdp2/files/TestData/. נתוני הבדיקה מכילים קובץ ברצף GSM מעוצב אחד וקובץ הגנום האחד.
- העבר את כל הקבצים שהורדו לספריית העבודה הנוכחית:
mv miRDP2-v*. tar. Gz מידע. tar. gz ncRNA_rfam, tar. gz < user_selected_folder >
< user_selected_folder cd > - חלץ את קבצי ה-אוסך הדחוסים:
טאר – xvzf miRDP2-v*. tar. gz
tar -xvzf testdata. tar. gz
טאר – xvzf ncRNA_rfam. טאר. gz - בניית אינדקס הגנום Arabidopsis:
bowtie2-build -f/testdata/TAIR10_genome. פא./מTAIR10_genome - בנה את אינדקס ההפניות ncRNA:
bowtie2-בנה -אפ/ncRNA_rfam פא/1.1.3/script/index/rfam_index - הפעל את צינור miRDP2:
bash ./1.1.3/mirdp2-v1.1.3_pipeline. bash – g./testdata/TAIR10_genome. פא-i./testdata/tair 10_genome – f./Testdata/gsm2094927.fa – o.
הערה: פקודות לינוקס המשמשות הן בגופנים מודגשים ונטויים, עם אפשרויות שורת פקודה בכתב נטוי. * מציין את הגירסה של miRDP2 (הגירסה הנוכחית היא 1.1.3). הפקודה bowtie2-build אמורה להימשך כ -10 דקות, וצינור miRDP2 צריך להסתיים תוך מספר דקות
- בדוק פלטי בדיקה.
- שים לב שתיקיה בשם ' GSM2094927-15-0-10 ' נוצרת באופן אוטומטי ב- < user_selected_folder >, המכילה את כל קבצי הביניים והתוצאות.
- בדוק שקובץ הפלט המופרד בטאבים GSM2094927-15-0-10_filter_P_prediction, הפלט הסופי של miRNAs החזוי, מכיל עמודות המציינות מזהה כרומוזום, כיוון סטרנד, נציג קורא מזהה, מזהה קודמן, מיקום Mirnas בוגרת, מקודמן מיקום, רצף מבוגר, ורצף מקודמן. שים לב לקובץ המיטה הנוסף שנגזר מקובץ זה כדי להקל על הניתוח.
- בדוק את הקובץ "progress_log", המספק מידע אודות שלבים מוגמרים והקבצים "script_log" ו-"script_err", המכילים פלט ואזהרות של תוכניות.
הערה: כרגע, יש לנו נבדק miRDP2 על שתי פלטפורמות לינוקס, כולל CentOS שחרור 6.5 על שרת אשכול, ו Cygwin 2.6.0 ב-PC Windows מערכת, ו miRDP2 צריך לעבוד על מערכות דומות שתומכות Perl.
2. זיהוי מירנס הרומן
- לפני הפעלת הצינור, ודא שהקלט מעובד מראש בתבנית נאותה.
הערה: הגירסה החדשה 1.1.3 של miRDP2 יכול לקבל קבצים בפורמט FASTQ המקורי כקלט, למרות תהליך קריאות העיצוב מבוצע כמו בגירסאות קודמות.- ראשית, להסיר את המתאמים מתוך 5 ' ו-3 ' מסתיים ברצף מעמיק קריאות (אם קיים).
- שנית, לנתח את מבנה הרצף העמוק קורא בפורמט FASTA.
- שלישית, הסר יתירות כגון קריאה עם רצף זהה מיוצגים באמצעות ערך FASTA יחיד וייחודי.
- לבסוף, ודא שכל מזהי FASTA הם ייחודיים. כל מזהה רצף חייב להסתיים ב-' _x ' ובמספר שלם, המציין את מספר ההעתקה של הרצף המדויק שאוחזר בערכות הנתונים ברצף העמוק. דרך אחת להבטיח מזהה FASTA ייחודי היא לכלול מספר פועל במזהה. לעיון, עיין בקובץ GSM2094927. fa בנתוני הבדיקה (https://sourceforge.net/projects/mirdp2/files/TestData/).
- עיין בפרטים הבאים לקבלת דוגמאות של קריאות מעוצבות בצורה נכונה:
> read0_x29909
הובלה מרבית
> read1_x36974
הגנה מפני הקובץ
> read2_x32635
מיכאל שלמה
- בניית מדדי התייחסות.
- עבור התייחסות הגנום, כדי לחסוך בזמן, להוריד קבצי אינדקס Bowtie2 מהאתר iGenomes (https://support.illumina.com/sequencing/sequencing_software/igenome.html) אם רצפי הגנום של מינים של עניין היו באינדקס. אחרת, המשתמשים משתמשים באינדקס לרצפים ושומרים את קובץ האינדקס למשך זמן מה עד שהפרוייקט מסתיים מאחר שייתכן שיהיה צורך ליצור אינדקס מחדש של רצף הגנום. פרטים על אופן האינדקס התייחסות הגנום נכללים במדריך bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml).
- אינדקס נוסף שאינו של miRNA ncRNA צריך גם כדי לסנן רצפים רועשים מקטעי RNA אחרים שאינם קידוד. הקובץ הוא אוסף של רצפי ncRNA הראשי מ Rfam, כולל rRNA, tRNA, snRNA, ו שנחנה. כדי לבנות אינדקס זה, נא עיין בחלק 1.4, כלומר יש להציב את האינדקס בצורה נכונה, דהיינו < miRDP2_version >/script/מrfam_index.
- . הפעל את miRDP2
- כדי להשתמש ב-miRDP2 לזיהוי מירנאס חדשים מתוך נתונים ברצף עמוק, הפעל את ה-script של bash בחבילה כדי להפעיל את צינור הניתוח (דוגמה ניתן למצוא בשלב 1.4):
< path_to_miRDP2_folder >/miRDP2-v. * _pipeline. bash – g < genome_file >-אני < path_to_index/index_prefix >-f < seq_file >-או < output_folder
כאשר * מציין את הגירסה של סקריפט bash הצינור. קיימים שלושה פרמטרים הניתנים לשינוי: 1) מספר המיקומים השונים שניתן היה למפות ל-, 2) את מספר האי-התאמה של הפעלת bowtie2 ו-3) את הסף של סל ד (קריאות למיליון). שינוי אלה באמצעות האפשרויות – L,-M ו-R, בהתאמה. הסבר מפורט הוא בסעיף 3.1.
- כדי להשתמש ב-miRDP2 לזיהוי מירנאס חדשים מתוך נתונים ברצף עמוק, הפעל את ה-script של bash בחבילה כדי להפעיל את צינור הניתוח (דוגמה ניתן למצוא בשלב 1.4):
- בדוק את miRDP2 תפוקות.
- שים לב שתיקיית הפלט נוצרת באופן אוטומטי תחת < output_folder >, ונקראת ' < seq_file_name >-15-0-10 '; שלושת המספרים האחרונים מציינים את הערכים (ברירת המחדל במקרה זה) עבור פרמטרים 1, 2 ו-3, בהתאמה. הקובץ < seq_file_name > _filter_P_prediction מכיל מידע של miRNAs האחרונה החזוי מספק את קריטריוני הביאור שעודכנו לאחרונה של צמח Mirnas. פרטים על תבנית קובץ הפלט מתוארים בחלק 1.4.
3. שינויים וזהירות באמצעות miRDP2
- פרמטרים שניתן לשנות
- השתמש באפשרות '-L ' כדי להגדיר את המגבלה של מספר המיקומים שניתן למפות לקריאה (פרמטר 1). ייתכן שמיפוי הקריאה לאתרים רבים מדי משויך לרצפים חוזרים, וסביר להניח שהוא מוכן ל-miRNAs. הגדרת ברירת המחדל היא 15. עבור מינים ספציפיים, אם יש משפחות miRNA עם חברים רבים, הפרמטר הראשון עשוי להיות מוגבר באופן ידני כדי להסתגל לנוף הגנום.
- השתמש באפשרות '-M ' כדי להגדיר את האי-התאמות המותר עבור עניבת הקשת (פרמטר 2). הגדרת ברירת המחדל היא 0.
- השתמש באפשרות '-R ' כדי להגדיר את הסף לקריאה העשויה להיות מתאימה ל-miRNAs בוגרת (פרמטר 3). כדי להקטין את צריכת הזמן ואת התוצאות החיוביות, המסנן קורא באמצעות RPM. רק קריאות שחורגות מסף סל ד מסוים עשויות לייצג רצפים בוגרים של miRNAs ולא רעשי רקע, והוא יישמר לצורך ניתוח נוסף. הגדרת ברירת המחדל היא 10 סל ד.
- שים לב ששינוי פרמטרים אלה עלול להשפיע על צריכת הביצועים והזמן. באופן כללי, גידול של פרמטר 1 ו-2 וירידה בפרמטר 3 יפיק תוצאה מחמירה פחות וזמן רב יותר ולהיפך.
- יתירות ומירנה *
- שים לב כי miRNAs פלט מ miRDP2 עשוי להיות שונה miRNAs הידוע. מצאנו כי זה בעיקר בשל אחת משתי סיבות: טרוגניות של miRNAs בוגרת או השפע היחסי של Mirnas ו Mirnas *. מצאנו כי זה לא משפיע על מבחר האורך האופטימלי של טרום סמנים ואת הפרופיל של הגנים הידועים miRNA.
תוצאות
צינור הביאור mirna, miRDP2, תיאר בזאת מוחל על 10 הציבור srna-seq ספריות מ 5 מינים צמח עם אורך הגנום גדל בהדרגה, כולל arabidopsis thaliana, oryza סאטיבה (אורז), solanum lycopersicum (עגבניה), zea מייז (תירס) ו triticum חיטת (באיור 1א באופן כללי, לכל מין, 2 ספריות sRNA מייצגות מרקמות שונות (...
Discussion
עם הופעתו של ngs, מספר גדול של מירנה מקום זוהו מתוך כמות גוברת של רצף נתונים של srna במינים שונים29,30. במסד הנתונים הקהילתי מרוכז miRBase21, הפריטים mirbase הופקד גדל כמעט 100 פעמים בעשור האחרון. עם זאת, בהשוואה mirnas בעלי חיים, צמח mirnas יש תכונות ייחודיות רבות העו...
Disclosures
. למחברים אין מה לגלות
Acknowledgements
עבודה זו נתמכת על ידי בייג האקדמיה לחקלאות יערנות מדעי (KJCX201917, KJCX20180425, ו KJCX20180204) ל-XY ולאומי המדע הטבעי הקרן של סין (31621001) ל-LL.
Materials
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
References
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved