A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מיפוי גנטי של הבדלי התרמוסבילות בין מינים של שמרים באמצעות הגנום ההופכי לניתוח המייזוליסיטי
In This Article
Summary
השפעה הדדית באמצעות רצף (RH-seq) היא שיטה חדשה ורבת עוצמה למפות את הבסיס הגנטי של הבדל תכונה בין מינים. בריכות המייזוליטיס נוצרות על ידי הטרנספורזיס והכושר שלהם מתבצע באמצעות הצמיחה התחרותית באמצעות רצף ברמה גבוהה. ניתוח הנתונים הנובעים מנקודות גנים שבבסיס התכונה.
Abstract
מטרה מרכזית של גנטיקה מודרנית היא להבין כיצד ומדוע אורגניזמים בטבע שונים ב פנוטיפים. עד היום, השדה התקדם בעיקר על כוחה של שיטות מיפוי הצמדה ושיוך, אשר מעקב אחר הקשר בין משתנים רצף ה-DNA ו לפנוטיפ באמצעות רקומביננטי צאצאים מתוך מצות בין אנשים של מין. גישות אלה, אם כי חזקות, אינן מתאימות באופן מתאים להבדלים בין מינים מבודדים מחדש. כאן אנו מתארים שיטה חדשה לניתוח הגנום-רחב של וריאציה תכונה טבעית שניתן להחיל בקלות על מינים לא תואמים. האסטרטגיה שלנו, RH-seq, היא יישום הגנום הרחב של מבחן ההופכי הופכי. אנו לרתום אותו כדי לזהות את הגנים האחראים על הצמיחה המרשימה בטמפרטורה גבוהה של שמרים סכביסים cerevisiae ס ביחס paradoxus המינים של אחותה . RH-seq מנצל טרנספסון מוטאנסיס כדי ליצור בריכה של הדדיות הדדית, אשר מסומנים לאחר מכן באמצעות תחרות בטמפרטורות גבוהות באמצעות רצף התפוקה הגבוהה. העבודה שלנו RH-seq כפי שהונחו כאן מספק קפדנית, דרך משוחדת כדי לנתח העתיקה, תכונות מורכבות ב שמרים לבשלה, עם האזהרה כי ברצף אינטנסיבי משאבים מרחוק נדרש כדי להבטיח כיסוי גנומית עבור מיפוי גנטי. כאשר העלויות ברצף ירידה, גישה זו מחזיקה הבטחה גדולה לשימוש עתידי על פני eukaryotes.
Introduction
מאז השחר של השדה, זה היה מטרה מעולה בגנטיקה כדי להבין את הבסיס המכונה של וריאציה על אנשים פראיים. כאשר אנו ממפים את הבית בבסיס תכונה של עניין, הגנים מתהווה יכול להיות שימוש מיידי כמטרות אבחון ותרופות, והוא יכול לשפוך אור על עקרונות האבולוציה. תקן התעשייה לקראת המטרה הזאת הוא לבחון את הקשר בין הגנוטיפ לבין הפנוטיפ באמצעות הצמדה או אסוציאציה1. רב עוצמה כמו גישות אלה, יש להם מגבלה אחת מפתח – הם סומכים על פאנלים גדולים של צאצאי רקומביננטי מן הצלבים בין אנשים הפרעות. הם אינם בשימוש בחקר המינים שאינם יכולים להזדווג ליצור צאצאים מלכתחילה. ככזה, לשדה יש יכולת מועטה לניתוח משוחד של הבדלים בין מינים מבודדים מחדש של2.
בעבודה זו אנו מדווחים על התחתון הטכני של שיטה חדשה, RH-seq3, עבור סקרים הגנום בקנה מידה של הבסיס הגנטי של וריאציה תכונה בין מינים. גישה זו היא גרסה מקבילה בנפט של הבחינה הדדית מבחן4,5, אשר היה לראשונה הגה כדרך להעריך את ההשפעות פנוטימית של הבדלים allelic בין שני רקעים שונים גנטית ב לוקוס מסוים (איור 1a). בערכה זו, שני האנשים מפוצלים הם הראשונים ליצור היברידית, מחצית הגנום אשר מגיע מכל אחד ההורים בהתאמה. ברקע זה, זנים מרובים מופקים, כל אחד המכיל עותק קטוע או נמחק של כל ההורה של כל הורה של לוקוס. זנים אלה הם hemizygous מאז הם נשארים דיפלואידי בכל מקום בגנום למעט במיקום של עניין, שם הם נחשבים haploid, והם מכונים הדדית מאז כל אחד חסר רק אלל של ההורה אחד, עם אלל הנותרים נגזר ה הורה אחר. על-ידי השוואת הפנוטיפים של זנים אלה he, הדדיים הדדית, אפשר להסיק אם משתנים רצף ה-DNA על מקום מניפולציות לתרום את התכונה של עניין, מאז המשתנים לוקוס הם ההבדל הגנטי היחיד בין הדדית זנים המיזוליציטים. בדרך זו, ניתן לקשר בין הבדלים גנטיים בין מינים לבין הבדל פנוטיוני ביניהם בכיוונון ניסיוני מבוקר. עד כה היישומים של בדיקה זו היו במסגרת מועמד-גן-כלומר, מקרים בהם ההשערה כבר בהישג יד כי וריאציה טבעית על לוקוס מועמד עשוי להשפיע על תכונה.
במה שלהלן, אנו מניחים את הפרוטוקול למסך הופכי בקנה מידה של הגנום, באמצעות שמרים כמערכת מודל. השיטה שלנו יוצרת השלמה גנומית של מוטציות heמיזרגוט, על ידי יצירת מוטאנטים של F1 כלאיים החיים בין המינים והוא מרכיב אותם לטרנספסון. אנחנו מאגר heמיזוליטיס, למדוד פנוטיפים שלהם לפי רצף מבוסס, ולבדוק הבדלים בתדר בין שיבוטים של הבריכה הנושאת שני ההורים ' אללים של גן נתון. התוצאה היא קטלוג של הבית שבו משתנים בין המינים משפיעים על תכונת הריבית. אנו מיישמים את זרימת העבודה RH-seq כדי להבהיר את הבסיס הגנטי של הבדלים התרמותרמיים בין שני מינים שמרים הנצה, סכביסים cerevisiae ס ו -S. paradoxus, אשר התפצל ~ 5,000,000 לפני שנה6.
Protocol
1. הכנת הפלאבאק המכיל את הפלמיד לטרנספורמציה
- פס החוצה מושבות יחיד את זן E. coli מחסה פלpJR487 באמצע ליברות + carbenicillin אגר צלחת. מודטה ללילה אחד ב 37 ° c או עד מושבות יחיד להופיע.
הערה: תיאור האופן בו ניתן למצוא את הpJR487 בעבודה הקודמת3. - מחסן 1 L של LB + carbenicillin ב 100 μg/mL עם מושבה אחת של E. coli המכיל pJR487 בקבוקון זכוכית 2 L. לגדול לילה ב 37 ° צ' עם טלטול ב 200 סל ד עד רווי (OD600 ≥ 1.0).
- לטהר את הדנ א הפלמטרי מהתרבות באמצעות ערכת בקנה מידה גדול הכנה הפלסטיות כפי שהורו בפרוטוקול שפורסם של היצרן (ראה טבלת חומרים לפרטים). מאגר ה-DNA לאחר הדגירה של 10 דקות עם 5 מ ל של המאגר מחומם ל 37 ° c.
- למדוד את הכמות והאיכות של ה-DNA פלמיד עם ספקטרוסקופיה (ראה טבלת חומרים לפרטים).
- חזור על שלבים 1.2 – 1.4 עד סכום כולל של לפחות 11 mg פלאמיד DNA ב A260: יחס280 של לפחות 1.8 מבודדים. זה עשוי לקחת כמה preps, בהתאם ליעילות.
- מערבבים את כל הפלבמיד שיטת ' יחד לתוך צינור אחד ולהביא את הנפח הכולל של עד 20 מ ל עם מאגר להתחמק או מים. מדדו שוב את הכמות והאיכות הסופיים באמצעות ספקטרוסקופיה. הריכוז של פלבאמצע צריך להיות לפחות 538 ng/μL ב זה 20 מ"ל נפח האחרון. אם הריכוז הוא גבוה יותר מ 538 ng/μL, לדלל את הפלסמיד עם מאגר להתחמק או מים כדי 538 ng/μL. פלמיד יכול להיות מאוחסן ב -4 ° c עד כמה שבועות עד השימוש.
2. יצירת בריכה של הגנום ההופכי שאינו ממוקד, כולו
- הכנת תאי שמרים היברידיים לטרנספורמציה
- פס out JR507 מ-80 מלאי ° c מקפיא המקפיא למושבות יחיד על צלחת YPD אגר. מודטה ב -26 ° c עבור יומיים או עד שמושבות מופיעות.
הערה: JR507 הוא זן היברידית שנעשו דרך תא בודד ההזדווגות של נבגים של הפלואיד של ס. cerevisiae ס DBVPG1373 ו S. paradoxus Z1 (באמצעות מיקרוסקופ הניתוח tetrad)3. - האיחישוב 100 mL של YPD נוזלי בתוך בקבוקון זכוכית 250 mL עם מושבה אחת של JR507 ולנער ב 28 ° c, 200 rpm עבור 24 שעות, או עד השלב הנייח הוא הגיע.
- למחרת, למדוד את הצפיפות האופטית ב 600 ננומטר (OD600) של התרבות הלילה. ליצור תרבות חדשה על ידי לדלל חלק התרבות הלילה עם YPD נוזלי טרי לתוך בקבוקון חדש 1 L זכוכית כדי OD600 של 0.2 וכמות של 500 mL.
הערה: חישוב לדוגמה של דילול גב אם לתרבות הלילה יש OD600 של 5.0, כאשר C הוא צפיפות אופטית ו-V הוא אמצעי אחסון: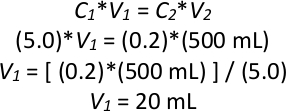
כך, 20 מ ל של תרבות לילה רווי יתווספו 480 mL של נוזל YPD לעשות סך של 500 mL של התרבות ב OD600 של 0.2. - חזור על השלב 2.1.3 שלוש פעמים נוספות כדי לעשות סך של 4 500 mL ב-OD600 של 0.2 בארבע 1 L זכוכית מבחנות, באמצעות תרבות לילה זהה לכל ארבע תרבויות חדשות. מודטה את כולם ב 28 ° צ' עבור 6 שעות (2-3 דורות) רועד ב 200 סל ד.
- לשלב שתי תרבויות 500 mL ליצירת תרבות 1 L. לשלב את שאר התרבויות 2 500 mL ליצירת תרבות נוספת 1 L. בשלב זה, ישנן שתי תרבויות L. כל אחת מהתרבויות הללו תהיה כפופה לטרנספורמציה עם pJR487 בשלבים הבאים.
- פס out JR507 מ-80 מלאי ° c מקפיא המקפיא למושבות יחיד על צלחת YPD אגר. מודטה ב -26 ° c עבור יומיים או עד שמושבות מופיעות.
- טרנספורמציה של pJR487 לתוך תאים שמרים היברידית
- פצל כל אחת מן התרבויות 1 L לתוך 70 mL הali, ב 20 מנורות חרוט פלסטיק עבור סך של 40 צינורות. להניח בצד 20 שפופרות ולבצע את השלבים הבאים על 20 שפופרות בכל פעם.
- צנטריפוגה כל אחד עשרים צינורות עבור 3 דקות ב 1,000 x g כדי גלולה תאים שמרים. . מחק את הסופרנטאנט
- השהה מחדש כל גלולה עם 25 מ"ל של H סטרילי2O על ידי vortexing. צנטריפוגה עבור 3 דקות ב 1,000 x g. . מחק את הסופרנטאנט
- השהה מחדש כל גלולה עם 5 מ ל של 1 x TE, 0.1 M LiOAc מאגר על ידי vortexing. צנטריפוגה עבור 3 דקות ב 1,000 x g. . מחק את הסופרנטאנט
- חזור על שלב ה2.2.4. בעוד התאים הם תפרידו, להכין לפחות 120 mL של פתרון של 39.52% פוליאתילן גליקול, 0.12 M LiOAc ו 1.2 x טריס-EDTA מאגר (12 mM טריס-HCl ו 1.2 mM EDTA). . חנות על קרח
- כדי להכין את ה-DNA פלמיד לשינוי, תחילה להרתיח 4 מ ל של דנ א זרע סלמון ב 100 ° c עבור 5 דקות ומיד מגניב אותו על הקרח עבור 5 דקות. לאחר מכן, לערבב 20 מ ל של pJR487 (שהתקבלו בסעיף 1) בריכוז של 538 ng/μL עם 4 מ ל של הזרע הסלמון מקורר דנ א עבור נפח כולל של 24 מ ל. . שמור על הקרח עד השימוש
- הוסף 600 μL של דנ א פלמיד מעורבב עם דנ א זרע סלמון על גבי כל גלולה תא. אל-השהה עדיין.
- הוסף 3 מ ל של הפתרון יתד-LiOAc-TE עשה בשלב 2.2.5 לכל גלולה. מחדש את הגלולה על ידי ליטוף למעלה ולמטה והורטקנג.
- מודקון כל צינור במשך 10 דקות בטמפרטורת החדר.
- התחשמלות מחממת כל צינור במשך 26 דקות באמבט מים הממוקם עד 39 ° c.
הערה: כל כמה דקות, להפוך כל צינור כדי למנוע את התאים להתיישב בתחתית הצינור. - צנטריפוגה כל צינור עבור 3 דקות ב 1,000 x g. למחוק את הסופרנטאנט ולהשעות מחדש כל גלולה ב 10 מ ל של YPD על ידי vortexing. לשלב את כל עשרים צינורות לתוך מבחנה זכוכית חדשה. הנפח הכולל של תאים צריך להיות ~ 200 mL.
- העברת 66.6 mL של תאים לבקבוקון חדש 1 L זכוכית ולהביא נפח של 500 mL עם נוזל YPD. חזור על שתי פעמים נוספות כדי להשתמש בשלם 200 mL של תאים שעברו טרנספורמציה. למדוד את OD600 של כל חדש 500 mL תרבות (מצפה od600 של ~ 0.35-4).
- לנער את כל שלושת מבחנות ב 28 ° צ' עבור 2 שעות להתאושש (< 1 דור) ב 200 סל ד.
- הוסף 0.5 mL של 300 mg/mL G418 לכל אחד שלושת מבחנות, לריכוז הסופי של 300 μg/mL G418 ולהחזיר לרעוד על 28 ° צ', 200 סל ד.
הערה: לפני שלב זה, התאים ההיברידית השתנה כבר מתאושש שינוי. עם תוספת של G418, הנוכחות של הפלבין pJR487 מסומנת. כל התאים שלא לקחו את הפלמיד במהלך השינוי יתחילו למות. - חזור על שלבים 2.2.2 – 2.2.14 עם שאר 20 הצינורות החרוטים של תאים. בשלב זה צריך להיות שש 1 מבחנות זכוכית, כל אחד עם 500 mL של תאים עם G418 נוסף.
- דגירה כל ששת מבחנות של תאים 28 ° צ', רועד ב 200 סל ד, במשך כ 2 ימים או עד OD600 של ~ 2.3 הוא הגיע בכל בקבוקון. לשלב את כל ששת מבחנות יחד כדי ליצור תרבות אחת.
הערה: למרות שכל התאים בתרבות זו לא ישמשו בצעדים במורד הזרם, המטרה של שימוש בכרכים גדולים כאלה הייתה ליצור אירועי שינוי ייחודיים רבים ככל האפשר ולנרמל כל מיני בעיות לאורך שינוי יחיד על-ידי האגירה של כולם יחד. - השימוש בתרבות שנוצר ב2.2.16 לאיחסן שני חדש 1 מבחנות עם 500 mL של YPD + G418 (300 μg/mL) ל-OD600 של 0.2. יהיו שאריות תרבות. שאפשר להיפטר מהם
- שניהם 1 L מבחנות ב 28 ° c לילה, עם טלטול ב 200 סל ד, עד שכל אחד מהם מגיע OD600 של ~ 2.2 (~ 3.5 דורות). לשלב את שתי התרבויות לתוך תרבות אחת ולמדוד את OD600 של התרבות המשולבת שוב.
הערה: בשלב זה, התרבות צריכה להיות כמעט לגמרי מורכבת מתאים מחסה פלpJR487 באמצע. בחלק מאוכלוסיית התאים, הטרנספבאק האחד יהיה משורבב מהפלסטלינה לתוך הגנום על ידי הטרנספציה המבוטאת מהפלסמיד. עם זאת, המשך הביטוי המתמשך יכול להוביל לטרנספוזיציה במהלך מבחר, שהיה מטשטש את הקשר בין הגנוטיפ לבין הפנוטיפים. מטרת השלבים הבאים היא לבצע בחירה נגדית כנגד נוכחותם של הפלביניים, כדי להבטיח שאין יותר ביטוי של הטרנספזדאז. הבריכה כתוצאה מכך הוא שילוב של תאים עם או בלי הטרנספסון משולב לתוך הגנום, אבל רק תאים המכילים את הטרנספסון מזוהים במהלך שלבי המיפוי הבאים. הזמן בטרנספורמציה שבמהלכו מתבטא הטרנספלסה, לפני שהקידוד הפלשני אובד, עשוי לשלוט בסיכוי ששיבוט נתון לאחר שהמוטמוזיס מקיים יותר מאשר החדרת משנה אחת. התדירות של אלה, אשר מתבטא כמוטציות "משניות" בניתוח של כל גן אחד בכל פעם, יכול להיות מוערך על ידי הגדרת מספר מוגדר של מושבות לאחר מוטזיס, ולאחר מכן שילוב ה-DNA שלהם ורצף-המאשר את מספר ההכנסה העצמאית עמדות בבריכה - צנטריפוגה 25 מ ל של תרבות זו עבור 3 דקות ב 1,000 x g. חשב את המספר של סך OD600 יחידות של תאים הנמצאים במרחק של 25 מ ל (ראה חישוב להלן). להיפטר supernatant ולהשעות מחדש מספיק H2O כדי ליצור השעיית תא של 1.85 OD600/ml על ידי vortexing.
הערה: חישוב לדוגמה עבור השעיה של תאים במים אם OD600 של תרבות משולבת היה 2.2: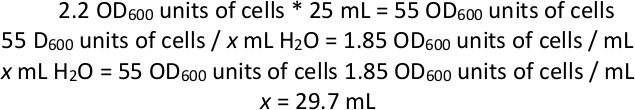
כך, לאחר ספינינג 25 מ ל של תרבות התא ומחיקת supernatant, להוסיף מספיק H2O לתאים כדי להביא את הנפח הכולל של תאים ומים עד ~ 29.7 ml (מאז הגלולה התא יהיה גם אמצעי אחסון, להוסיף פחות מ 29.7 ML של H2O). - באמצעות חרוזי זכוכית, צלחת 1 מ ל של תאים מושעה במים על כל 12 ריבוע גדול להשלים אגר לוחיות סינתטי עם 5-אני. מודקת כל צלחת ב 28 ° c עבור 1-2 ימים או עד צורות הדשא על הצלחת.
- באמצעות מסחטת סטרילי קטנה, לגרד את התאים של כל אחד 6 צלחות לתוך צינור עם 35 mL של מים סטריליים. חזור עם 6 הצלחות האחרות עבור מספר כולל של שתי שפופרות של תאים ומים. שלב את כל השתלים בתא בשפופרת אחת. למדוד את OD600 של השעיה זו, באמצעות מים כריק. להביא את OD600/ml ריכוז של תאים כדי 44.4 OD600 יחידות/ml עם מים. בניסיון שלנו, הטרנספוזיציה יעילות (החלק של התאים KAN + כי הם URA-) הוא בממוצע 50%.
- לקבוע את מספר-80 מניות ° צ' מקפיא של תאים לאחסן. ניתן להשתמש בכל סדרת מחלקים בעתיד עבור ניסוי אחד.
הערה: בהתחשב בזמן הצורך ביצירת המאגר, יש לאחסן מספר מבחנות במקרה של שימוש לרעה בשוגג או לביצוע ניסויים בשכפול. 20-30 מניות הם מספר סביר. - כל מלאי המקפיא יכיל 40 OD600 יחידות של תאים 1 מ ל של 10% dmso. הוסף 900 μL של תאים ל 100 μL של DMSO. חזור על המספר הכולל של מניות ההקפאה שנוצרו. אחסן כל אחד ב-80 ° c לשימוש עתידי.
3. מבחר ההימיזכים הדדיים בפורמט במאגר
- הפשרת מ-80 ° c מקפיא מקום אחד של המזרח השני במאגר מקטע 2 בטמפרטורת החדר.
הערה: אל תתנו את השיר לשבת זמן רב בטמפרטורת החדר ברגע שהוא מגיע, השתמש בו מיד. - השתמש במלואו 1 mL כדי לאיחסן 150 mL של נוזל YPD בבקבוק זכוכית 250 mL. למדוד את ה-OD600 של תרבות זו, ולאחר מכן הדגירה ב 28 ° c, רועד ב 200 rpm, עבור ~ 7 שעות, או עד ה2-3 תרבות עברה טווח אוכלוסייה האוכלוסיה. בשלב זה, התרבות היא מוכנה לשמש לחיסונים תרבויות שעברו בחירה.
הערה: חישוב לדוגמה: אם ה-OD600 של הבקבוקון המקורי מודד 0.25, מודדת את התרבות עד שהוא מגיע ל-OD600 של לפחות 1.0. אם נקודות דגימה כלשהן רצוי ב "זמן-אפס" (T-0), כדרך לחקור את אוכלוסיית heמיזיואת האוכלוסייה לפני הבחירה, כדורי תא ניתן לקחת עכשיו על ידי תפרידו 5-10 mL של תרבות לכל גלולה ב 1,000 x g עבור 3 דקות, להשליך את הסופרנטאנט ואת קופא ב-80 ° c. - השתמש בבריכת ההימיחיטה הגדולה כדי לשחזר תרבויות לבחירה בשיטת שכפול מתאימה, בטמפרטורה גבוהה (39 ° c) ובטמפרטורת מתירני (28 ° c). לכל הפחות, להגדיר שלוש תרבויות ביולוגיות לשכפל בחירה בכל טמפרטורה, עבור סך של שישה תרבויות הבחירה.
- צור כל תרבות בחירה עם 500 mL הכולל בקבוקון זכוכית 2 L עם YPD נוזלי והאיחסן ל-OD600 של 0.02. לנער כל תרבות בחירה ב 100 סל"ד ב 28 ° צ' או 39 ° צ' עד 6-7 טווח האוכלוסייה אירעו (מתאים OD600 של ~ 1.28-2.56). נסה להתאים באופן הדוק ככל האפשר את הסופי OD600 של כל התרבויות הבחירה.
הערה: תרבויות הבחירה ב -28 ° c יגדלו מהר יותר מאשר תרביות בחירה ב-39 ° c. כתוצאה מכך, תרביות בחירה ב-39 ° צ' יבלו תקופה ארוכה יותר בחממה. המשך עם השלבים הבאים עם כל בקבוקון כפי שהוא הופך להיות מוכן, ללא קשר למספר הכולל של שעות בילו בחממה. בניסיון שלנו, תרבויות ב -28 ° צ' או 39 ° צ' לקחו ~ 12 או ~ 18 שעות, בהתאמה, כדי להגיע OD של ~ 2.0. בחירות ארוכות יכול להיות יתרון של הגברה של אפקטי כושר קטן, אבל גם לאפשר דה נובו מוטציות רקע להתעורר, אשר להציג רעש לתוך התפלגות הסופי של מוטציות על פני טרנספסון מוטנטים בכל גן אחד/allele. ככזה חשוב להגביל את זמן הבחירה בניסוי מסוג RH-seq.
- צור כל תרבות בחירה עם 500 mL הכולל בקבוקון זכוכית 2 L עם YPD נוזלי והאיחסן ל-OD600 של 0.02. לנער כל תרבות בחירה ב 100 סל"ד ב 28 ° צ' או 39 ° צ' עד 6-7 טווח האוכלוסייה אירעו (מתאים OD600 של ~ 1.28-2.56). נסה להתאים באופן הדוק ככל האפשר את הסופי OD600 של כל התרבויות הבחירה.
- הקציר כדורי תא מכל תרבות בחירה. לחשב את אמצעי האחסון הנדרש כדי לקבל 7 OD600 יחידות של תאים וצנטריפוגה ב 1,000 x g עבור 3 דקות לפחות ארבעה כדורי כרך זה מכל תרבות הבחירה כשכפל טכני עבור הכנה לספרייה ורצף (ראה סעיפים 4 ו-5 , להלן). השמט את הסופרנטנט והחנות בשעה-80 ° c.
הערה: לדוגמה, אם לבקבוקון בחירה יש מנת מ600 אחרונה של 2.0: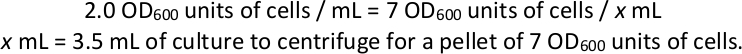
4. Tn-מבנה הספריה seq ורצף הארה כדי לקבוע את השפע של טרנספסון מוטציה המיזוליטס
- להפשיר על קרח כל כדור כל תא מסעיף 3 כי עומד להיות רצף.
- בודד דנ א גנומית סה (gDNA) מכל גלולה תא באמצעות ערכת טיהור שמרים gDNA לאחר הוראות היצרן. להשעות מחדש את ה-DNA ב 50 μL של מאגר הימנעות מחומם ל 65 ° c.
- לכמת את כמות gDNA מכל גלולה באמצעות פלואורימטר. הכמות הכוללת המינימלית של gDNA נדרש עבור כל גלולה תא ליצירת רצף הדור הבא (NGS) הספריה עבור Tn-seq באמצעות ההליך הבא הוא 1 μg.
הערה: פחות מ-1 μg של gDNA ניתן להשתמש כדי ליצור ספרייה, אבל הכמות הסופית והאיכות של הספרייה תסבול. - עקוב אחר פרוטוקול מבוסס ליצירת ספריות Tn-seq7. שים לב למידע הרלוונטי הבא הייחודי לפרוטוקול זה:
- לאחר הטיית gDNA, תיקון סוף ולאחר מתאם, להגביר את gDNA המכיל את הטרנספסון דרך ה-PCR. עבור ה-PCR, השתמש באפשרות התחל הבאה והפוכה, שהן ספציפיות עבור מתאמי הטרנספסון ו-NGS, בהתאמה:
קדימה (N – נוקלאוטיד אקראי)
5 ' ATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACG
CTCTTCCGATCTNNNNNNAGCAATATTTCAAGAATGCATGCGTCAAT 3 '
הפוך (מתיחה של Ns מייצגת מדד ייחודי 6-bp המשמש עבור ריבוב. ראה להלן מידע נוסף על אינדקסים)
5 ' CAAGCAGAAGACGGCATACGAGATNNNNNNGTGACTGGAGTTCAG
מיכל האור 3 - השתמש בשלבי הניקוי הנכללים עם חרוזים בגודל-סלקטיבי כדי למזער את הפרופורציה של קטעים משוכפלות בספריה הסופית, שיהיו קצרים מכדי לכלול רצף גנומי הניתן למיפוי.
הערה: לאחר שעקבו אחר הדרישות המינימליות לשכפול עד היום לתרבויות הבחירה, יהיו 24 דגימות gDNA בודדות לרצף. בהינתן העלות הנוכחית לרצף, לא סביר כי כל מדגם יפעל בעצמו. כדי לשלב דגימות על אותו נתיב, ליצור מספר התחל הפוך, כל אחד עם אינדקס ייחודי 6 בסיס זוג. ניתן לשלב דגימות באמצעות אינדקסים שונים לאותו נתיב רצף ולאחר מכן להפריד בין בחינה חישובית אופן פרופורציונלי.
- לאחר הטיית gDNA, תיקון סוף ולאחר מתאם, להגביר את gDNA המכיל את הטרנספסון דרך ה-PCR. עבור ה-PCR, השתמש באפשרות התחל הבאה והפוכה, שהן ספציפיות עבור מתאמי הטרנספסון ו-NGS, בהתאמה:
- רצף בודד 150 bp קורא מתוך כל ספריה באמצעות טכנולוגיות NGS על פני שמונה נתיבים.
הערה: הכמות הנדרשת ברצף הפעולות הנדרשות תלויה במידה רבה באיכות הספריות המוכנות בשלב הקודם (כלומר, שיעור ה-dna בספריה המכיל את ה-DNA של טרנספסון, המייצג דנ א המגיע מההדמיגוטים הדדיים). ישנם שני גורמים עיקריים התורמים לכך. ראשית, כיוון שתאים ללא טרנספורסון משולבים אינם מסומנים כנגד במהלך יצירת הבריכה, כל תרבות תהיה תערובת של תאים עם ובלי הטרנספסון. שנית, אפילו בתוך הגנום של טרנספסון המכיל הדדיות הדדית, רוב הגנום הוא לא טרנספסון המכיל רצף, ו gDNA זה יהיה בלתי נמנעת להיות חלק ההכנה בספרייה. המטרה של הגברה סופית של ה-PCR של טרנספסון המכיל דנ א היא להגדיל את היחס של הטרנספסון המכיל דנ א לשני מקורות אלה של רקע gDNA. ככל שהגברה זו יעילה יותר, החלק הגבוה יותר של הקריאות יהיה מסוגל לשמש בניתוח במורד הזרם. האיכות הנמוכה ביותר של הספריות, יהיה צורך לבצע את הרצף יותר, כיוון שחלק הולך וגובר של קריאות לא יכיל את ה-DNA של הטרנספסון ולא יהיה שימושי. בהינתן האילוצים הנ ל, שמונה מסלולים של רצף היו מסוגלים לעקוב אחר הליך המייזוליט הדדית במידה סבירה. יותר רצף מאפשר. ניתוח עמוק יותר
5. מיפוי מיקומים של הוספות טרנספסון ואנליזה של RH-seq
הערה: ניתוח הנתונים הבא הושג עם סקריפטים מותאמים אישית פיתון (נמצא באינטרנט ב https://github.com/weiss19/rh-seq), אבל יכול להיות משופץ באמצעות שפות scripting אחרות. להלן, השלבים העיקריים בתהליך מתוארים באופן מיתאר. בצע את הפעולות הבאות בכל אחד משכפול קובץ הקריאה, אלא אם צוין לשלב אותם.
- חיתוך רצפים של מתאם מחוץ לקריאה ולהפרדת כל הקריאות של שכפול בהתאם לאינדקס.
- מצא את הקריאות המכילות את. הטרנספסון-גנום כדי לבצע זאת, חפש בתוך כל אחד מהם 20 הבסיס זוגות של הטרנספסון, המקוקאכטציטקציה. בטל את כל הקריאות שאינן מכילה רצף זה.
הערה: מניסיוננו, שיעור הקריאות מיפוי עד סוף הטרנספסון הוא 83-95%. - חתוך את הנותרים, המכיל את הטרנספסון לקריאה כדי להכיל רק את הרצף במורד הזרם של הקצה של 3 הטרנספסון. על ידי מיפוי רצף זה לגנום שמרים, לקבוע את ההקשר גנומית של החדרת הטרנספסון עבור כל לקרוא (שלב 5.4 להלן).
- השתמש בכלי מיפוי שווה ערך כדי למפות את הרצף במורד הזרם של הטרנספסון ל- s. cerevisiae ס DBVPG1373 x s. paradoxus Z1 הגנום היברידי (שם סקריפט: map_and_pool_BLAT. py).
- התעלם מקריאות שבהן יש פחות מ-50 זוגות בסיסים של רצף שמיש במורד של 3 מסוף הטרנספסון. רצפים קצרים קשה למפות באופן ייחודי.
- בשעת שימוש בבלט, השתמשו בפרמטרים הבאים: זהות = 95, גודל אריח = 12.
- ליצור גנום היברידית בסיסית להשתמש עבור מיפוי על ידי שרשור הגירסאות העדכניות של התייחסות גנום של s. cerevisiae ס S288c ו-s. paradoxus CBS432.
הערה: קובץ ביאור בסיסי המתאר את גבולות גנומית של גנים בודדים ברחבי הגנום היברידית ניתן למצוא במאגר Github המפורטים לעיל (שם הקובץ: YS2 + CBS432 + plasmid_clean). השתמש רק בקריאה איזו מפה למיקום יחיד בגנום היברידי (כלומר, הם ייחודיים ל-S. cerevisiae ס או S. paradoxus). תדירות אחידה של אירועים הכנסה ברחבי הגנום צפוי; הפצת עמדות ההכנסה ברחבי הגנום מדווחת במקום אחר3.
- לספור את המספר הכולל של קריאות מיפוי לכל מיקום ייחודי הכנסת הטרנספסון, אשר אנו מסיק שכל מקורם בתאים של שיבוט מוטציה בודד הכנסה אחת. הסכום של כל הערכים האלה מספריה בודדת מכונה המספר הכולל של קריאות ממופות עבור אותה ספריה.
- במקרים בהם יש מיפוי הוספות מרובים בתוך 3 זוגות בסיסים אחד של השני, לשלב את כולם לנקודת הכנסה אחת, הקצאת כל הקריאות למיקום היחיד עם ספירת הקריאה הגבוהה ביותר. ערך זה, ninsert, מייצג את השפע של שיבוט הכניסה בתוך הגלולה התא שממנו gdna היה רצף. בשלב זה, יהיו רשימות של n הוסף, כל השפע של החדרת הטרנספסון ייחודי ממופה, רשימה אחת עבור כל כדורית תא ברצף.
הערה: הטרנספסון מוסיף ברצפי TTAA בגנום, ברצף של 4 זוגות בסיסים. כך, אנו מסיק כי מיפוי הוספות בתוך 3 זוגות בסיס אחד של השני חייב להיות מקורו באותו אתר TTAA. - מאחר שיהיה מספר שונה במקצת של הקריאות הכוללות המגיעים מכל ספריה ברצף, יש לנרמל את ערכי ninsert בכל הקבצים אם יש להשוותו. בצע זאת על-ידי הטבתה של המספר הכולל של קריאות ממופות מכל ספריה בודדת, nגלולה, ולקחת את הממוצע של כל nגלולה בכל הספריות, < nגלולה>. הכפל כל n הוסף בנתונים של ספריה בודדת על-ידי היחס בין < nגלולה>/n כדי לחשב הוספה, השפע המנורמל של שיבוט ההכנסה הנתונה של טרנספסון.
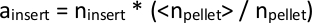
לחילופין, ניתן להעריך את גודל הספרייה באמצעות כלים זמינים כגון DESeq28 (שם קובץ Script: total_reads_and_normalize. py). - בטאת הקבוצה של כל הוספות שמופו בכל הספריות. עבור הוספות שנמצאו בספריות מסוימות אך לא באחרים , הגדר הוספה = 1 עבור חישובים במורד הזרם.
- סנן את הקריאות כדי למצוא הוספות שנופלות בתוך גנים בהתאם לקובץ הביאור (שם Script: remove_NC_and_plasmid_inserts. py).
- עבור כל הכנסה ייחודית, לחשב את השפע הממוצע על פני משכפל טכני של כל בחירה (כל תרבות ב 28 ° צ'או 39 ° c), < הכנס >טכני (שם Script: combine_tech_reps_V2. py).
- עבור כל הכנסה ייחודית, לחשב את השפע הממוצע על פני משכפל ביולוגי של כל טמפרטורה, <הוספה>סך, על ידי לקיחת הממוצע של כל < הכנס >טכני בכל טמפרטורה. במקביל, לחשב את מקדם הווריאציה עבור כל הוספה, CVinsert, סה כ על פני ה< הכנס >טכני (שם Script: combine_bio_reps. py).
הערה: בשלב זה, עבור כל טמפרטורה, 28 ° צ' ו-39 ° c, יש רשימה של הוספות שינויים ייחודיים, השפע הממוצע שלהם ומקדם הווריאציה בין משכפל ביולוגי לכל. נתונים אלה עבור הניסוי שלנו מדווחים במקום אחר3. - סנן את רשימת כל הוספות עבור אלה שיש להם, ב 28 ° צ' או 39 ° c, <הוספה>סך > 1.1, וCV להוסיף,סה"כ ≤ 1.5 (שם קובץ: filter_inserts. py).
- עבור כל הכנסה ייחודית, חשב את יומן הרישום2(< הוספה >סכום, 28 ° צ' /< מ>כולל הכולל, 39 ° c). ערך זה מייצג את "הרגישות התרמותרמית" של שיבוט מוטציה של הכנסה מסוימת (שם קובץ Script: fitness_ratios. py).
- מיין את כל הוספות ייחודי על ידי הגן על ידי אלל (ש. cerevisiae ס או s. paradoxus), ולטבול את מספר הוספות בכל אלל. לסנן גנים כך שרק גנים שיש להם לפחות 5 הוספות בכל אלל מנותח (שם סקריפט: organize_and_filter_genes. py).
הערה: הוספות ייחודיות מרובות בכל אלל מאפשרות מידה מדויקת יותר של הרגישות התרמוטתית הדדית של ההימיזיוט. הפחתת מספר הוספות הנדרש לכל אלל אפשרי אך להתפשר על הדיוק של מידה זו ולהגדיל את העומס בדיקות מרובות על ידי מתן הגנים יותר להיבדק. בנוסף, סינון גנים עם מעט מדי הוספות לכל אלל יסייע להפחית את ההשפעה על תוצאות הבדיקה של שיבוט heמיזיייוט בודדים מחסה מוטציה באתר משני המביאה פנוטיפים שונים מאוד. - עבור כל הגנים שנותרו בערכת הנתונים לאחר הסינון לעיל, להשוות את thermotolerances (היומן2 יחסי) של כל הוספות ב -s. cerevisiae ס אלל לאלה ב s. paradoxus אלל באמצעות מבחן מאן-ויטני U. לחילופין, ניתן ליישם מודל רגרסיה, המותאם מ-DESeq28 (שם Script: mann_whitney_u. py).
- תקן ערכי- pעבור בדיקות מרובות באמצעות שיטת בנימיני-הוכברג.
- גנים עם ערכי pמשמעותיים (אומרים, ≤ 0.01) הם מועמדים גנים חשובים הבדלים התרמוסטטים בין שני המינים.
תוצאות
מיזלנו את ס. cerevisiae ס ו- s paradoxus ליצור היברידית סטרילית, אשר אנו נתונים לטרנספסון מוטגנזה. כל שיבוט מוטגני היה he, היברידית דיפלואידי שבו אחד אלל של גן אחד מופרת (איור 1a, איור 2). היינו מתמודדים נגד השני באמצעות גידול ב39 ° c ובניסוי נפרד כפקד, ב -28 ...
Discussion
היתרונות של RH-seq על פני שיטות סטטיסטיות-גנטיות קודמות הן מספר קיפול. בניגוד לניתוח ההצמדה וההתאגדות, הטיפול ב-RH-seq מעניק רזולוציית מיפוי של גנים בודדים; ככזה, סביר להניח שהיא תהיה בעלת תועלת משמעותית גם במחקרים של וריאציה של תכונה על פני אנשים ממין נתון, כמו גם הבדלים בין-ספציפיים. כמו כן, נסי...
Disclosures
. למחברים אין מה לגלות
Acknowledgements
. אנחנו מודים לג רואופ, ר. האקלי, אני. גריגוריה, א. ארקין וג סקלקר על תרומתם למחקר המקורי, פ. אלזבאן, א. פלאם, ג. גגלמן, ג'יי הונג, י. קים, מ. מOltrogge, ו-ל. לסיוע טכני, ד. סאבאג ' לנדיבות עם מיקרוסקופ משאבים, ו-B. Blackman, ס. קוראדטי, א. פלמהולץ, נ' גואצ'י, ד. כורטלנד, ס. נלסון ו-א. ססיקומאר לדיונים; כמו כן, אנו מודים לג דובר (המחלקה לביוהנדסה, אוניברסיטת ברקלי) עבור הפלאבאק. עבודה זו נתמכת על ידי R01 GM120430-A1 ועל ידי הקהילה רצף הפרויקט 1460 RBB בארה ב. המחלקה לאנרגיה משותפת מכון הגנום, משרד DOE של מתקן משתמש המדע. העבודה שנערכה על ידי האחרונה נתמכת על ידי משרד המדע של משרד האנרגיה של ארצות הברית תחת חוזה לא. דה-AC02-05CH11231.
Materials
| Name | Company | Catalog Number | Comments |
| 1-2 plasmid Gigaprep kits | Zymo Research | D4204 | The number of kits required depends on how efficient your preps are in each kit. This kit comes with 5 individual plasmid prep columns. Run 1 L of saturated E. coli culture through each prep column, as using more than 1 L per column can cause clogging of the prep filter, leading to low yield and poor quality DNA. |
| 10X Tris-EDTA (TE) buffer (100 mM Tris-HCl and 10 mM EDTA) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 1M LiOAc | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 300 mg/mL Geneticin (G418) | Gibco | 11811023 | |
| 52% polyethylene glycol (PEG) 3350 | Sigma | 1546547 | Dissolve in water and filter sterilize through a 0.22 μm filter before use. 1X trafo mix: 228 uL 52% PEG, 36 uL 1M LiOAc, 36 uL 10X TE buffer |
| Autoclaved LB liquid broth | BD Difco | 244620 | Make LB liquid broth using your powder from any brand, and milliQ water. Autoclave it before use. |
| Carbenicillin stock in water (100 mg/mL) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| Complete synthetic agar plates (24.1cm x 24.1cm) with 5-fluoroorotic acid (5-FOA) [0.2% drop-out amino acid mix without uracil or yeast nitrogen base (YNB), 0.005% uracil , 2% D-glucose, 0.67% YNB without amino acids, 0.075% 5-FOA] | 5-FOA: Zymo Research, Drop-out mix: US Biological, Uracil: Sigma, D-glucose: Sigm), YNB: Difco | 5-FOA: F9001-5, Drop-out mix: D9535, Uracil: U0750, D-glucose: G8270, YNB: DF0919 | |
| DMSO | Any | N/A | |
| E. coli strain carrying pJR487 (CEN-/ARS+ piggyBac-containing plasmid) | N/A | N/A | Request from Brem lab. |
| Hybrid yeast strain JR507 (S. cerevisiae DBVPG1373 x S. paradoxus Z1, URA-/URA-) | N/A | N/A | Request from Brem lab. |
| Illumina Hiseq 2500 | used for SE-150 reads | ||
| Large shaking incubators with variable temperature settings | Any | N/A | |
| LB + carbenicillin agar plates (100 μg/mL) | Agar: BD Difco | Agar: 214010 | Make LB agar plates as normal and add carbenicillin to 100 μg/mL before drying. |
| Nanodrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit Fluorimeter | Thermo Scientific | Q33240 | |
| Salmon sperm DNA | Invitrogen | 15632011 | |
| Water bath at 39°C | Any | N/A | |
| Yeast fungal gDNA prep kit | Zymo Research | D6005 | |
| Yeast peptone dextrose (YPD) liquid media | BD Difco | Peptone: 211677, Yeast Extract: 212750 | Add filter-sterilized D-glucose to 2% after autoclaving. |
| YPD + G418 agar plates (300 μg/mL) | Agar: BD Difco | Agar: 214010 | Make YPD agar plates as normal and add G418 to 300 μg/mL before drying. |
| YPD agar plates | Agar: BD Difco | Agar: 214010 |
References
- Flint, J., Mott, R. Finding the molecular basis of quantitative traits: successes and pitfalls. Nature Reviews Genetics. 2, 437-445 (2001).
- Allen Orr, H. The genetics of species differences. Trends in Ecology and Evolution. 16, 343-350 (2001).
- Weiss, C. V., et al. Genetic dissection of interspecific differences in yeast thermotolerance. Nature Genetics. 50, 1501-1504 (2018).
- Stern, D. L. Identification of loci that cause phenotypic variation in diverse species with the reciprocal hemizygosity test. Trends in Genetics. 30, 547-554 (2014).
- Steinmetz, L. M., et al. Dissecting the architecture of a quantitative trait locus in yeast. Nature. 416, 326-330 (2002).
- Scannell, D. R., et al. The Awesome Power of Yeast Evolutionary Genetics: New Genome Sequences and Strain Resources for the Saccharomyces sensu stricto Genus. G3 (Bethesda). 1, 11-25 (2011).
- Wetmore, K. M., et al. Rapid quantification of mutant fitness in diverse bacteria by sequencing randomly bar-coded transposons. MBio. 6, e00306-e00315 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15, 550 (2014).
- Wilkening, S., et al. An evaluation of high-throughput approaches to QTL mapping in Saccharomyces cerevisiae. Genetics. 196, 853-865 (2014).
- Kim, H. S., Huh, J., Riles, L., Reyes, A., Fay, J. C. A noncomplementation screen for quantitative trait alleles in saccharomyces cerevisiae. G3 (Bethesda). 2, 753-760 (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved