Method Article
הדור הבא של RNA/DNA ויראלי יישומי הכלאה באתרם במחקר נגיף הכשל החיסוני האנושי/וירוס הכשל החיסוני של סימיאן
In This Article
Summary
כאן אנו מציגים בדיקת הכלאה במקום של הדור הבא לזיהוי רצפי RNA או DNA נגיפיים ספציפיים ברקמות משובצות פרפין קבועות פורמלין (FFPE). גישה זו מאפשרת הדמיה של עותקים נמוכים של RNA ו-DNA בפחות מ-24 שעות עם רגישות וסגוליות גבוהות מאוד.
Abstract
הכלאה באתרה היא טכניקה רבת עוצמה לזיהוי רצפי RNA או DNA ספציפיים בתוך תאים בודדים בקטעי רקמה, ומספקת תובנות חשובות לגבי תהליכים פיזיולוגיים ופתוגנזה של מחלות. הכלאה באתרה (ISH) משמשת במשך שנים רבות להערכת מיקומם של תאים נגועים בנגיפים, אך לאחרונה פותחה גישת ISH מהדור הבא עם אסטרטגיית תכנון בדיקה ייחודית המאפשרת הגברת אותות ודיכוי רקע בו זמנית כדי להשיג הדמיה של מולקולה בודדת תוך שמירה על מורפולוגיה של רקמות. הדור הבא של ISH מבוסס על גישה כמו PCR מסועף, אך מבוצע באתרו והוא קל, רגיש וניתן לשחזור יותר משיטות ISH קלאסיות או גישות PCR באתרן בזיהוי שגרתי של RNA או DNA ברקמות משובצות פרפין קבועות בפורמלין (FFPE). בשנים האחרונות המעבדה שלנו מיישמת פלטפורמת ISH זו לאיתור תאים חיוביים של כשל חיסוני אנושי (HIV) וכשל חיסוני סימיאני (SIV), RNA נגיפי (vRNA) ו/או DNA נגיפי (vDNA) בתוך מספר רב של רקמות FFPE. עם כתב היד הטכני המפורט הזה, ברצוננו לחלוק את הידע והעצות שלנו עם כל האנשים המעוניינים להשתמש בדור הבא של ISH במחקר שלהם.
Introduction
ISH היא הגישה הניסיונית המשמשת למיקוד והדמיה של DNA, RNA או גדילי חומצות גרעין משלימים (כלומר, בדיקות) לרצפי DNA או RNA ספציפיים בתוך תא או קטע של רקמה. ISH מאפשר לוקליזציה והדמיה ספציפית של רצפי גרעין ספציפיים ברקמות, חשוב להבנת רמת הביטוי, הארגון, ההפצה והאינטראקציות בין המטרה לסביבה התאית שלה, שהיא מידע רב ערך שלא ניתן להשיג באמצעות טכניקות פופולריות אחרות, כגון qPCR. עד לאחרונה, ISH בוצע בדרך כלל עם DNA משלים מסומן או RNA משלים (ריבופרוב). בדיקות אלה היו מצומדות ישירות עם בסיסים מסומנים ברדיו, פלואורסצנט או אנטיגן (למשל, 35S, FITC ו-digoxigenin) ולאחר מכן אותרו וכימתו ברקמה באמצעות אוטורדיוגרפיה, מיקרוסקופיה פלואורסצנטית או גישות זיהוי אימונוהיסטוכימיה, בהתאמה. בעוד שטכנולוגיות אלה באתרן ממשיכות להיות גישות בעלות ערך, יש מקום רב לשיפור כדי לפתח גישות פחות עתירות עבודה, פשוטות יותר, מהירות יותר, רגישות וספציפיות.
גישת ISH מסחרית חלופית מהדור הבא (למשל, בדיקת RNAscope), שתוארה לראשונה ב-2012, לזיהוי RNA שליח מארח (mRNA) מבוססת על PCR מסועף. זיהוי ה-mRNA מבוצע בתאי FFPE וברקמות, עם רגישות המתקרבת להדמיה של מולקולת RNA בודדת בתאים בודדים1. הספציפיות של גישה זו מושגת בתנאי הייחודי ששני בדיקות מטרה כפולות Z נקשרות ברציפות לרצפי ה-RNA (או ה-DNA) המשלימים שלהן בהתאמה עבור מגבר אות לקשור1 ברצף. זה מאפשר התחלה של מפל הגברת אותות באמצעות שלבי הכלאה עוקבים הדומים ל-DNA מסועף (bDNA)1,2. בנוסף, גישה זו מהירה וקלה להפליא, עם תוצאות המתקבלות תוך יום אחד בלבד (<8 שעות), יתרון משמעותי בהשוואה לעד 4 שבועות בטכניקות חלופיות, כולל Radio-ISH 1,2. הדור הבא של ISH פתח נקודות מבט והזדמנויות חדשות לחקר HIV/SIV. המכשולים העיקריים לריפוי HIV הם מאגרי התאים והרקמות הנוצרים בשלבים המוקדמים של המחלה 3,4. המטרה הכוללת של טכניקה זו היא לזהות, למקם ובסופו של דבר להבין את תאי הרקמה העיקריים הפועלים כמאגר ויראלי ומתמידים בתוך מארח נגוע. זה בתורו יעזור בפיתוח אסטרטגיות ריפוי יעילות נגד HIV.
בכתב יד זה, אנו מסבירים בפירוט את פרוטוקול ה-RNA/DNA multiplex ISH הדופלקס שלנו מהדור הבא (למשל, RNAscope/DNAscope) ומסבירים כיצד שינינו את פרוטוקול ה-RNA ISH הקיים כדי לייעל את ה-ISH של הדור הבא לדגימות ולמטרות הספציפיות שלנו. פרוטוקול זה מאפשר ויזואליזציה, לוקליזציה וכימות של RNA נגיפי HIV/SIV ו-DNA ויראלי בתוך מקטעי רקמה של 5 מיקרומטר. הדמיה סימולטנית של vRNA ו-vDNA מתבצעת על ידי שילוב של שתי ערכות בדיקה מותאמות אישית: חוש אחד, המכוון לגדיל המקודד vDNA (C1 SIVmac239 Gag-Pol-Sense בדיקה [416141-C1]), ואנטי-חוש אחד, המכוון לתעתיקי vRNA (בדיקה C2 SIVmac239 Vif-Env-Nef-Tar-Anti-Sense [416131-C2]) המכסה אזורים שונים בגנום הנגיפי (טבלה 1), תוך שימוש בשני ערוצי הדמיה שונים, C1 ו-C2. בפרוטוקול זה, ערוצים C1 ו-C2 מאפשרים לנו לדמיין אותות בצבעים שונים (כלומר, AP באדום ו-HRP בחום) ולזהות את הבדיקות בגישות שונות. למעט עיבוד וחיתוך קיבוע הרקמות, בדיקה זו אורכת יומיים. מוצג כאן פרוטוקול ההכלאה הדופלקס vRNA ו-vDNA באתרו שניתן לבצע על כדורי תאים או קטעי רקמה.
Protocol
1. הכנת חתכים ושקופיות
- חתוך את גושי הפרפין והשתמש במיקרוטום כדי לחתוך חלקים של 5 +/- 1 מיקרומטר. הרכיבו את החלקים או כדורי התא על מגלשות מיקרוסקופ טעונות באמבט מים ללא RNase של 40-45 מעלות צלזיוס. מגלשות יבשות באוויר למשך הלילה ב-37 מעלות צלזיוס או RT.
הערה: ניתן לאחסן את השקופיות עד 3 חודשים בטמפרטורת החדר (RT) ו-6 חודשים ב-4 מעלות צלזיוס. - Deparaffinize שקופיות FFPE.
- אופים שקופיות בתנור יבש למשך שעה בחום של 60 מעלות צלזיוס.
- במכסה אדים, מלאו שני כלי צביעה בשקופיות עם ~200 מ"ל קסילן טרי, ושני כלי צביעה נוספים עם ~200 מ"ל אתנול טרי 100%. מכסים את המכולות במכסים.
- מניחים את השקופיות ברשת וטובלים בכלי הראשון המכיל קסילן. דגירה למשך 5-10 דקות ב-RT עם תסיסה.
- מניחים את השקופיות לתוך הכלי השני המכיל קסילן ודוגרים במשך 5-10 דקות ב-RT עם תסיסה.
- מניחים מיד את השקופיות בכלי המכיל 100% אתנול. דגרו את המגלשות למשך 5-10 דקות ב-RT עם תסיסה.
- מניחים מיד את השקופיות בצלחת השנייה המכילה 100% אתנול ודוגרים למשך 5-10 דקות ב-RT עם תסיסה.
- הסר את המתלה מהאתנול, הקש בעדינות על צד המתלה כדי להסיר עודפי אתנול, ושטוף במים נטולי RNase למשך 5-15 דקות.
2. הכנת תנור
- מפעילים את תנור ההכלאה ומכוונים את הטמפרטורה ל-40 מעלות צלזיוס.
- הניחו מטלית או מגבת נייר סופגת יציבה במגש והרטיבו לחלוטין במים מזוקקים כפולים כדי לאפשר בקרת לחות.
- הכניסו את המגש המכוסה לתנור וסגרו את דלת התנור. מחממים את המגש למשך 30 דקות לפחות בחום של 40 מעלות צלזיוס לפני השימוש. שמור את המגש בתנור כאשר אינו בשימוש.
3. אחזור אפיטופים המושרה על ידי חום
- הכן מאגר אחזור יעד הכלאה של 0.5x ציטראט ISH (10 ננומול/ליטר, pH = 6, ראה טבלת חומרים). מביאים לרתיחה בכוס על צלחת החימום.
- בצע אחזור אפיטופ המושרה על ידי חום על ידי הנחת השקופיות למאגר אחזור מטרה רותח למשך 30 דקות.
- הסר את השקופיות ממאגר אחזור היעד ושטוף מיד במים מזוקקים כפולים. יש לייבש ב-100% אתנול למשך 5 דקות לפני הייבוש באוויר.
- לאחר שהמגלשות התייבשו באוויר, החל עט מחסום הידרופובי כדי להקיף את קטע הרקמה במגלשה. הקפד לאפשר למחסום ההידרופובי להתייבש לחלוטין באוויר.
4. טיפול מקדים בפרוטאז
- הניחו את השקופיות המיובשות על מתלה שקופיות נעילה, ולאחר מכן הכינו את הריאגנטים לטיפול מקדים בפרוטאז (תמיסת עיכול פרוטאז, 2.5 מיקרוגרם/מ"ל) על ידי דילול עם PBS סטרילי, קר, ביחס של 1:5. מערבבים היטב.
הערה: שלושה ריאגנטים שונים של פרוטאז בריכוזים שונים מסופקים בערכה הזמינה מסחרית. פרוטאז III (סטנדרטי), פרוטאז IV (חזק) ופרוטאז פלוס (קל). בדוק באופן אמפירי את זמן העיכול והדילול של הפרוטאז לפני היישום במחקר מכיוון שהתנאים האופטימליים ישתנו בהתאם לסוג הרקמה, הקיבוע והעובי (ראה דיון). - יש להוציא את תמיסת הפרוטאז המדוללת על שקופיות כדי לכסות לחלוטין את חלקי הרקמה. דגרו מיד את השקופיות למשך 20 דקות בחום של 40 מעלות צלזיוס בתנור (הוכן בשלב 1.4), וודא שהמגלשות אטומות במגש ההכלאה הלח. אל תתנו לחלקי הרקמות להתייבש למשך שארית הפרוטוקול.
- יש לשטוף מיד פי 3 על ידי טבילת מתלה הנעילה לתוך מגש כביסה מלא במים מזוקקים כפולים.
- בצע את חסימת הפרוקסידאז האנדוגנית על ידי הטלת תמיסת הפרוקסידאז על כל חלק רקמה כדי לכסות אותו לחלוטין. דגרו את המגלשות למשך 10 דקות ב-RT. לאחר שתסיים, שטפו את החלקים פי 3 במים מזוקקים כפולים.
5. הכלאת בדיקה והגברת אותות
הערה: כדי למנוע אידוי, ודא שהמגש מבוקר הלחות אטום כהלכה כדי שהרקמות לא יתייבשו במהלך שלבי הדגירה. החזירו את תא הלחות לתנור במהלך שלב הכביסה כדי להבטיח שהוא יישאר בטמפרטורה של 40 מעלות צלזיוס.
- מערבבים את הגשושית C2 והגשושית C1 ביחס של 1:50 על ידי פיפטינג של נפח אחד של בדיקה C2 ל-50 נפחים של בדיקה C1 לתוך צינור כפי שהוצע על ידי היצרן. הפוך את הצינור מספר פעמים. מחממים את תערובת בדיקת המטרה בתנור 40 מעלות צלזיוס למשך ~10 דקות כדי להמיס כל משקעים לפני השימוש.
הערה: ניתן לאחסן את בדיקות המטרה המעורבות בטמפרטורה של 4 מעלות צלזיוס למשך עד 6 חודשים. - הסר את המגלשות מהמים. שטפו והקישו או החליקו את השקופיות כדי להסיר עודפי מים מחלקי הטישו. הוצא מיד את הגשושית על השקופיות, וודא שכל קטע רקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את תערובת הבדיקה בתא הלחות למשך הלילה בטמפרטורה של 40 מעלות צלזיוס.
- למחרת, הוציאו את השקופיות מהתנור והניחו אותן במגש הכביסה המכיל מאגר כביסה פי 0.5 למשך 5 דקות ב-RT. חזור על שלב הכביסה פעם נוספת.
- הסר את השקופיות ממאגר הכביסה. שטפו והקישו או החליקו את השקופיות כדי להסיר עודפי מאגר כביסה מחלקי הטישו.
- הוצא מסחרית מגיב AMP 1 מוכן לשימוש (2 ננומול/ליטר) במאגר הכלאה B (20% פורממיד, 5x SSC, 0.3% ליתיום-דודציל סולפט, 10% דקסטרן סולפט, ריאגנטים חוסמים) על השקופיות, מה שמבטיח כיסוי מלא של קטע הרקמה ללא בועות אוויר. דגירה למשך 30 דקות בטמפרטורה של 40 מעלות צלזיוס בתא הלחות. חזור על שלבים 1.6.3-1.6.4, ובצע את הכביסה במשך 2 דקות כל אחד.
- מחלק זמין מסחרית AMP 2. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 15 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלבים 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחד.
- מחלק זמין מסחרית AMP 3. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 30 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלבים 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחד.
- מחלק זמין מסחרית AMP 4. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 15 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלבים 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחד.
- מחלק זמין מסחרית AMP 5. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 30 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלב 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחת.
- מחלק זמין מסחרית AMP 6. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 15 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלב 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחת.
- לפני הזיהוי, שטפו את השקופיות פעם אחת ב-1x TBS-Tween 20 (0.05% v/v). הסר את השקופיות ממאגר הכביסה, שטוף והקש או החלק את השקופיות כדי להסיר עודפי מאגר כביסה מחלקי הרקמות. מניחים מיד במגש הכביסה המלא במאגר 1x TBS-Tween.
6. זיהוי אות יעד של ערוץ 1 (C1)
הערה: זה מבוצע באמצעות פוספטאז אלקליין אדום וכרומוגן אדום מהיר Amplification 6 מערכות זיהוי 2-plex (ראה טבלת חומרים) המכילות תוויות פוספטאז אלקליין, וזיהוי כרומוגני. הוא משתמש באדום מהיר כמצע ליצירת אות אדום.
- הכן פתרון עבודה אדום מהיר (FR) באמצעות דילול של 1:60 מ-Fast RED-B ל-Fast RED-A. מערבבים היטב. כדי להפחית משקעים ולקבל אות נקי יותר, סנן את תמיסת הכרומוג'ן דרך קרום MCE של 0.45 מיקרומטר באמצעות מזרק.
הערה: השתמש בתמיסת Fast RED-B תוך 5 דקות. אין לחשוף לאור שמש ישיר או לאור UV. - הסר את השקופיות מ-TBS-Tween, שטוף והקש או החלק את השקופיות כדי להסיר עודף מאגר מחלקי הרקמה.
- יש להוציא תמיסת FR מעורבת ומסוננת על כל חלק של טישו, ולוודא שכל חלק מכוסה לחלוטין. דגירה ב- RT למשך 6-8 דקות. התבוננו במיקרוסקופ.
- שוטפים את השקופיות במאגר כביסה פי 0.5. הסר את השקופיות ממאגר הכביסה, שטוף והקש או החלק את השקופיות כדי להסיר עודפי מאגר כביסה מחלקי הרקמות.
- מחלק זמין מסחרית AMP 7. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 10 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלב 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחת.
- מחלק זמין מסחרית AMP 8. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 15 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלב 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחת.
- מחלק זמין מסחרית AMP 9. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 30 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלב 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחת.
- מחלק זמין מסחרית AMP 10. ודא שחלק הרקמה מכוסה לחלוטין ללא בועות אוויר. דגרו את המגלשות בתא הלחות למשך 30 דקות בטמפרטורה של 40 מעלות צלזיוס. חזור על שלב 5.3-5.4, ובצע את הכביסה למשך 2 דקות כל אחת.
7. זיהוי אותות יעד של ערוץ 2 (C2)
הערה: פעולה זו מתבצעת באמצעות ערכות כרומוג'ן Brown HRP ו-DAB הזמינות מסחרית (ראה טבלת חומרים). ההגברה 10 מזיהוי 2-plex מכילה תוויות פרוקסידאז חזרת, וזיהוי כרומוגני מתבצע באמצעות DAB ליצירת אות חום.
- לזיהוי אופטימלי של אות ה-DAB, השתמש בערכות זמינות מסחרית ופעל לפי הוראות היצרן (ראה טבלת חומרים). התבוננו במיקרוסקופ.
זהירות: DAB רעיל. פעל לפי אמצעי הזהירות המתאימים והנחיות הבטיחות בעת טיפול וסילוק של כימיקל זה.
8. מכתים והרכבה
- מכתים את השקופיות עם המטוקסילין.
- צבעו את השקופיות למשך 30 שניות עם 50% המטוקסילין מסונן טרי תוך כדי ערבוב השקופיות. השקופיות יופיעו בסגול. יש לשטוף מיד במים זורמים תוך כדי ערבוב המגלשות למעלה ולמטה עד שהמים צלולים. חלקי הרקמה יישארו סגולים.
- כדי להשיג ניגודיות טובה יותר, הניחו שקופיות מוכתמות במים מזוקקים רוויים בליתיום קרבונט למשך דקה. יש לשטוף היטב במים זורמים לפחות פי 3 תוך כדי ערבוב המגלשות. השתמש במים מזוקקים כפולים לשטיפה האחרונה.
- מכיוון ש-FR רגיש לממיסים אורגניים, שקופיות מוכתמות ב-FR יצטרכו להיות מכוסות במדיום הרכבה על בסיס מים ולייבש למשך הלילה ב-RT.
- הרכיב את השקופיות.
- ודא שחלקי הרקמות המכוסים במדיום הרכבה על בסיס מים יבשים.
- טבלו שקופיות בקסילן לפני החלקת הכיסוי באמצעות מגיב הרכבה. הקפד למנוע או להסיר בועות אוויר בין הכיסוי לחלק הטישו והניח להתייבש במשך 16 שעות ב-RT.
9. פרוטוקול ניתוח תמונה כמותי ל-RNAscope באמצעות CellProfiler5
- בקצרה, ודא שהתוכנה תפריד כתמי המטוקסילין ו-FR לתמונות נפרדות. זהה ומדוד את האובייקטים המעניינים: גרעינים, ויריונים, תאים חיוביים וצביעה חיובית מצטברת של FR. אחסן מדידות בקובץ CSV ושמור את התמונה שנותחה.
- בחר באפשרות "Unmix colors", המפרידה בין כתמים, ומפצלת את אזור העניין המקורי (ROI) לתמונות המטוקסילין ו-FR נפרדות.
- אם המוסידרין, קעקוע או תכונות דומות מפריעים לניתוח, הוסף שלב "Unmix" שני עם המטוקסילין, FR ו-DAB. השתמש בתמונת ה-FR השנייה כדי למצוא את פיקסלי ה-FR החזקים ביותר. התמונה השנייה הזו תשמש כמסכה לצביעה אמיתית של FR.
- לחלופין, החלק את התמונות המוכתמות לפני סף שלהן. זו החלטה אמפירית ולא תמיד נחוצה. סף את התמונות המוכתמות הבודדות באמצעות "IdentifyPrimaryObjects" כדי לבחור פיקסלים חיוביים.
- זהה את שלושת סוגי האובייקטים השונים (ויריון, אגרגטים של ויריון, תא יצרני).
- ודא שהגרעינים הם אובייקטים בקוטר 4-100 פיקסלים (פיקסלים) המוכתמים בהמטוקסילין. יש לפרק ולמלא חורים לאחר הסף. אובייקטים "IntenseFastRed" הם בקוטר 4-100 פיקסלים וכוללים ויריונים, תאים חיוביים וצביעה חיובית מצטברת, כמו שנראה על תאים דנדריטיים זקיקים (FDC) בזקיקי תאי B (BCF). תמונה זו משמשת לסינון תוצאות חיוביות שגויות (למשל, המוסידרין).
- ודא שהחיובים הקטנים של FR הם אובייקטים בקוטר 2-12 פיקסלים. מדידה זו כוללת ויריונים ותאי vDNA+. השלך חפצים מחוץ לטווח זה. יש לפרק ולמלא חורים לאחר הסף.
- ודא שחיובי FR גדולים: אובייקטים בקוטר 9-100 פיקסלים. מדידה זו כוללת תאים חיוביים של vRNA+ וצביעה חיובית מצטברת. ירידה לאחר הסף. הגודל של אובייקטים חיוביים FR קטנים וגדולים יכול לחפוף. הם יופרדו בשלב מאוחר יותר.
הערה: התוכנה (למשל, Cellprofiler) משתמשת בפיקסלים עבור גדלי אובייקטים, ואלה המשמשים כאן נגזרים משקופיות שנסרקו ב-0.2510 מיקרומטר/פיקסל (40x).
- הגדר וחלץ תוצאות.
- לאחר זיהוי אובייקטים כלשהם, הגדר וחלץ תוצאות עבור מספר הוויריונים, תאים נגועים יצרניים וגרעינים.
- זהה ויריונים על ידי מיסוך FastRedSmallPositives עם האובייקט IntenseFastRed מוגדר להסרת תוצאות חיוביות שגויות (למשל, המוסידרין).
- לאחר מכן, זהה את התאים החיוביים וצביעה חיובית FR מצטברת. הסר תוצאות חיוביות שגויות וויריונים מ- FastRedLargePositives על ידי מיסוך עם IntenseFastRed ועם ערכת אובייקטי הוויריון.
- חלץ תאים חיוביים מה-FastRedLargePositives המזוקק על ידי מיסוך שוב עם גרעינים. פצל אובייקטים שכבר אינם נוגעים זה בזה, וסנן את התוצאות לפי אזור האובייקט, תוך הסרת אובייקטים קטנים (≤6 פיקסלים). זה מסיר את הכתמים שנוצרו על ידי מיסוך חפיפת הגרעינים. התוצאה היא התאים החיוביים.
- לבסוף, הגדר חיובי FR מצטבר. שלב זה משתמש ב- IdentifyTertiaryObjects, לאיתור אובייקטים הכלולים באובייקט אב גדול יותר. במקרה זה, קבוצת האובייקטים המעודנת FastRedLargePositives היא האב ותאים חיוביים מופחתים.
- ספרו את מספר הוויריונים והתאים החיוביים.
- מדוד את האזור החיובי המצטבר והמר אותו מפיקסלים למ"מ2. לחלופין, רשום את השטח התפוס על ידי ויריונים ותאים חיוביים במ"מ2 אם הניתוח דורש שימוש בתא סטנדרטי ובגדלי ויריונים במקום ספירה ישירה.
- שכב על האובייקטים החיוביים בתמונה המקורית ושמור את התוצאה.
הערה: נתוני ה-ISH מדווחים על ידי מספר הוויריונים לכל 106 גרעינים (תאים) ומספר תאי ה-vRNA+ הנגועים באופן פרודוקטיבי לכל 106 גרעינים (תאים) להבנה טובה יותר וכדי להקל על ההשוואה עם נתוני qPCR, אך ניתן לדווח על תוצאות גם לפי שטח הרקמה במ"מ2.
תוצאות
בכתב יד קודם 2,6,7,8,9,10,11, דיווחנו כי ניתן לשלב את פלטפורמות ה-ISH של הדור הבא המזהות vRNA או vDNA, באמצעות בדיקות חישה (vDNA) המכוונות לחלק ה-5' gag-pol של הגנום SIV/HIV ובדיקות אנטי-סנס (vRNA) המכוונות לגנים במחצית ה-3' של הגנום (vif, vpx, vpr, tat, env ו-nef) כמו גם אלמנט ה-TAR בגנום 5' (טבלה 1). גישה זו מבדילה בין תאים פעילים מבחינה שעתוק (vRNA+, vDNA+) לבין תאים נגועים לא פעילים מבחינה שעתוק (כביכול סמויים) או תאים המכילים פרו-וירוסים לא כשירים מבחינה שעתוק (vRNA-, vDNA+) באותו קטע רקמה2 (איור 1).
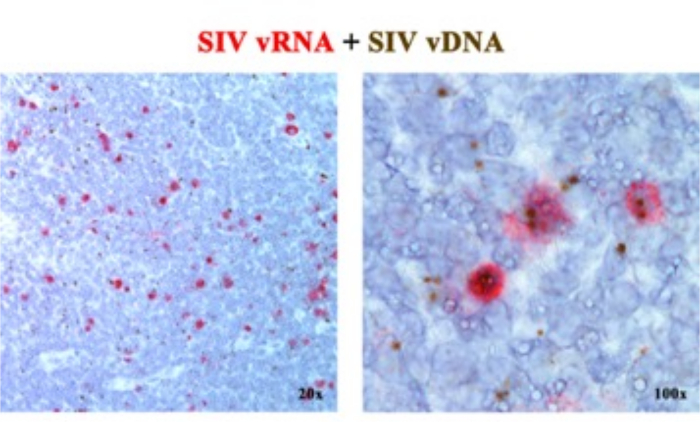
איור 1: זיהוי RNA ויראלי ו-vDNA באותו קטע רקמה. שילוב של בדיקת היברידיזציה של RNA (אדום) והכלאת DNA (חום) בבלוטת לימפה RM נגועה ב-SIV באופן חריף, המדגים את היכולת לזהות vRNA ו-vDNA באותו קטע רקמה ומספק גישה רבת עוצמה לזיהוי תאי vDNA+ vRNA שקטים מבחינה שעתוק באתרם. נתון זה שונה מ-Deleage C. et al.2. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| ערכות בדיקה של Plex RNA/DNAscope יחיד | |||
| שם | קטלוג ACD # | מספר ZZ | תיאור |
| SIVmac239 (אנטי-סנס) | 312811 | 83 | מיקוד בדיקה אנטי-חושי בתוך 1251-9420bp של D01065.1 (gag, pol, vif, vpx, vpr, tat, env ו-nef) |
| SIVmac239 (חוש) | 314071 | 83 | בדיקת חישה המכוונת לגדיל הפוך בתוך 1251-9420bp של D01065.1 (gag, pol, vif, vpx, vpr, tat, env ו-nef) |
| V-HIV1-Clade A (אנטי-סנס) | 416101 | 80 | בדיקה אנטי-חושית מכוונת בתוך 879-7629bp של קונצנזוס HIV-1 Clade A (gag, pol, vif, vpr, tat, rev, vpu, env ו-nef) |
| V-HIV1-Clade A (סנס) | 426341 | 80 | בדיקה חושית המכוונת לגדיל הפוך בתוך 879-7629bp של HIV-1 Clade A קונצנזוס (gag, pol, vif, vpr, tat, rev, vpu, env ו-nef) |
| V-HIV1-Clade B (אנטי-סנס) | 416111 | 78 | בדיקה אנטי-חושית מכוונת בתוך 854-8291bp של AF324493.2, HIV-1 Clade B NL4-3 (gag, pol, vif, vpr, tat, rev, vpu, env ו-nef) |
| V-HIV1-Clade B (סנס) | 425531 | 78 | בדיקה חושית המכוונת לגדיל הפוך בתוך 854-8291bp של AF324493.2, HIV-1 Clade B NL4-3 (gag, pol, vif, vpr, tat, rev, vpu, env ו-nef) |
| V-HIV1-Clade D (אנטי-סנס) | 416121 | 76 | בדיקה אנטי-חושית מכוונת בתוך 894-7697bp של קונצנזוס HIV-1 Clade D (gag, pol, vif, vpr, tat, rev, vpu, env, nef) |
| V-HIV1-Clade D (סנס) | 426351 | 76 | בדיקה חושית המכוונת לגדיל הפוך בתוך 894-7697bp של קונצנזוס HIV-1 Clade D (gag, pol, vif, vpr, tat, rev, vpu, env, nef) |
| ערכות בדיקה של Multi-Plex RNA/DNAscope | |||
| שם | קטלוג ACD # | מספר ZZ | תיאור |
| V-SIVmac239-gag-pol-Sense-C1 | ברקוד 416141-C1 | 40 | בדיקת חישה המכוונת לגדיל הפוך בתוך 1251-4093bp של D01065.1 (gag ו-pol) |
| V-SIVmac239-vif-env-nef-tar-C2 (אנטי-סנס) | ברקוד 416131-ג2 | 47 | מיקוד בדיקה אנטי-חושי בתוך 5381-10257bp של D01065.1 (vif, vpx, vpr, tat, env, nef ואלמנט TAR) |
| V-HIV1-Clade_B-gag-pol-sense-C1 | ברקוד 444051-ג1 | 40 | בדיקה חושית המכוונת לגדיל הפוך בתוך 854-3940bp של AF324493.2, HIV-1 Clade B NL4-3 (gag ו-pol) |
| V-HIV1-Clade_B-vif-vpr-tat-rev-vpu-env-nef-tar-C2 (אנטי-סנס) | ברקוד 444061-ג2 | 40 | בדיקה אנטי-חושית מכוונת בתוך 5042-9673bp של AF324493.2,, HIV-1 Clade B NL4-3 (vif, vpr, tat, env, nef ואלמנט TAR) |
| V-HIV1-Clade_C-gag-pol-sense-C1 | ברקוד 444021-ג1 | 48 | בדיקת חישה המכוונת לגדיל הפוך בתוך 888-5032bp של רצף קונצנזוס HIV-1 Clade C (gag ו-pol) |
| V-HIV1-Clade_C-vif-vpr- rev-vpu-env-nef-tar-C2 (אנטי-סנס) | ברקוד 444041-ג2 | 49 | בדיקה אנטי-חושית מכוונת בתוך 5078-9698bp של רצף קונצנזוס HIV-1 Clade C (vif, vpr, tat, env, nef ואלמנט TAR) |
| V-HIV1-Clade_AE-gag-pol-sense-C1 | ברקוד 444011-ג1 | 55 | בדיקה חושית המכוונת לגדיל הפוך בתוך 890-4812bp של AF259954.1, HIV-1 Clade AE (gag ו-pol) |
| V-HIV1-Clade_AE-vif-vpr-tat-rev-vpu-env-nef-tar-C2 (אנטי-סנס) | ברקוד 444031-ג2 | 57 | בדיקה אנטי-חושית מכוונת בתוך 5052-9694bp של AF259954.1, HIV-1 Clade AE (vif, vpr, tat, env, nef ואלמנט TAR) |
טבלה 1: רשימת בדיקות להתמקדות ב-vRNA ו-vDNA של HIV-1 ו-SIV.
Discussion
הכלאה באתרה היא בדיקה קפדנית הדורשת קפדנות וידע בסיסי בכימיה של חומצות גרעין, ביולוגיה של התא והיסטולוגיה כדי להיות מסוגל להתאים כל שלב קריטי למיקום מטרה בסביבה שמורה היטב. בדיון זה ברצוננו להדגיש את השלבים הקריטיים שבהם פתרון בעיות הוא חיוני להשגת תוצאות מדויקות וניתנות לפרשנות.
הקיבוע והעיבוד של הרקמות הם קריטיים ויש לטפל בהם מראש כדי לוודא שהבדיקה יכולה להניב את התוצאות הטובות ביותר. קיבוע PFA ניטרלי (4% מוכן טרי) הוא אופטימלי לבדיקת הדופלקס. עם זאת, ניתן לבצע את הבדיקה גם על רקמות קפואות (OCT) עם תנאי הקיבוע המתאימים לאחר ההקפאה.
טיפול מקדים בקטעי הרקמה הוא שלב מכריע. ישנם שני שלבי טיפול מקדים בבדיקה זו: הראשון הוא אחזור אפיטופ המושרה על ידי חום (HIER). שלב זה חשוב להיפוך קישורי גשר מתילן ושיקום מבני חלבון, הדרוש ברקמות קבועות. היעילות של טיפול זה תלויה בזמן, בטמפרטורה, בסוג מאגר האחזור וב-pH. הטיפול המקדים השני הוא אחזור אפיטופ המושרה על ידי פרוטאז (PIER). שלב זה מבקע פפטידים, חושף את האנטיגן או הנוקלאוטידים, ומשתמש באנזימים כולל פרוטאינאז K, טריפסין ופפסין. זהו צעד רגיש ביותר שעלול לפגוע הן במורפולוגיה של הרקמות והן ביעד העניין. ריכוז האנזים, כמו גם זמן וטמפרטורת הדגירה הם קריטיים בתהליך זה. עיכול יתר מוביל לתיחום גרעינים לקוי ולקושי בשלבי כמות. זה קריטי למצוא איזון בין גישה אופטימלית למטרת ה-RNA/DNA לבין תנאי טיפול מקדימים שאינם פוגעים ברקמה או ביעד המבוקש. לכל סוג רקמה יש רמת רגישות שונה לכל אחד מהטיפולים המקדימים הללו ויש לבדוק אמפירית כל פרמטר (ריכוז אנזים, זמן, טמפרטורה).
המחמירה של מאגר הכביסה מבוססת על שלושה פרמטרים עיקריים: טמפרטורה, ריכוז מלחים וחומר ניקוי וזמן. מאגר הכביסה הוא מאגר נתרן ציטראט מלוח (SSC), וריכוז המלח בתוך המאגר שולט בחומרה במהלך שלבי הכביסה. בפרוטוקול שלהם, ACD ממליץ להשתמש במאגר הכביסה בריכוז סופי של 0.1x SSC, 0.03% ליתיום דודציל סולפט. תוך כדי עבודה על אופטימיזציה של DNAscope ומולטיפלקס, קבענו ששימוש במאגר הכביסה בריכוז סופי של 0.05x SSC נתן לנו תוצאות טובות יותר להמחשת אות ה-DNA ועזר במידה ניכרת להפחית הכלאה לא ספציפית מחוץ למטרה הנובעת מהדגירה הלילית של בדיקת החישה.
יש לחשוב על הבחירה בגישת הזיהוי, כרוגן (אדום או חום) לעומת פלואורסצנטי על סמך סוג הרקמה והמטרה לפני תחילת הבדיקה. הגישה הכרומוגנית האדומה תיתן ניגודיות נחמדה, מכיוון שאדום אינו נמצא באופן טבעי ברקמות. כרומוג'ן חום ייתן תוצאות דומות לכרומוגן אדום. עם זאת, חשוב לזכור שלחלק מתוצרי פירוק הדם הקיימים ברקמה יש צבע דומה, ויהיה קשה להפריד דיו קעקוע מהאות החום בזמן הכימות. גישת זיהוי פלואורסצנטי תאפשר הבחנה ברורה בין סמנים תאיים שונים והריבוב יציע בדיקה מושלמת לפנוטיפ של התאים המכילים vRNA ו/או vDNA.
יש צורך בבקרות מרובות כדי להבטיח את הספציפיות של הבדיקות ואת איכות הבדיקה. כל בדיקה חדשה שתוכננה חייבת להיבדק על רקמות בקרה או כדורי תאים חיוביים ושליליים ידועים. לעתים קרובות אנו מייצרים פלסמידים המכילים את הרצף הממוקד שלנו ומבצעים טרנספקציה לקווי תאים כדי ליצור בקרות חיוביות. לכל ריצה אנו מוסיפים רקמה שלילית ידועה (HIV או SIV שלילית), בקרה ללא בדיקה המכילה רק את מדלל הבדיקה, ובקרה מטופלת RNase כדי להבטיח את האיכות והספציפיות של הבדיקה.
הכימות הוא שלב חשוב ביותר ויש לבצע אותו באמצעות הכלים והאלגוריתם המתאימים המבוססים על השאלה שנשאלה. בכתב יד זה הצגנו תוכנה לניתוח תמונות (למשל, Cellprofiler), שבחרנו לאחר הערכה של מספר אפשרויות. הערכנו שתוכנה זו היא התוכנה הטובה ביותר לצרכים שלנו, אך ישנן תוכנות רבות לניתוח תמונות שניתן להשתמש בהן.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
פרויקט זה מומן במלואו במימון פדרלי מהמכון הלאומי לסרטן, המכונים הלאומיים לבריאות, תחת חוזה מס. HHSN261200800001E ועל ידי המרכז הלאומי לחקר פרימטים של אורגון P51OD011092 מענק NIH (JDE). התוכן של פרסום זה אינו משקף בהכרח את ההשקפות או המדיניות של מחלקת הבריאות ושירותי האנוש, ואזכור של שמות מסחריים, מוצרים מסחריים או ארגונים אינו מרמז על תמיכה של ממשלת ארה"ב. הדופלקס פותח בעזרת Advanced Cell Diagnostics.
Materials
| Name | Company | Catalog Number | Comments |
| ACD HybEZII Hybridization system (110V) with ACD EZ-Batch Slides system | ACD | 321710 | Hybridization oven |
| CAT Hematoxylin | Biocare medical | CATHE-GAL | colorstain |
| Clear-Mount | ELECTRON MICROSCOPY SCIENCES | 17985-15 | mounting reagent for red chromogen |
| Immpact DAB Peroxidase Kit | Vector | SK-4105 | Used to reveal HRP - DAB (Brown) to replace the DAB coming in the ACD kit |
| lithium carbonate | Fisher chemical | L119-500 | bluing solution |
| paraformaldehyde | ELECTRON MICROSCOPY SCIENCES | 15714-S | for tissue fixation (4%) |
| PBS | life technology | 14190-136 | |
| Permount Mounting Medium | ThermoFisher Scientific | SP15-100 | mounting regaent for brown chromogen |
| Prolong Gold | ThermoFisher Scientific | P36930 | mounting regaent for fluorescence |
| ribonucleases A | ThermoFisher Scientific | 12091039 | for RNAse treatment in DNAscope protocol |
| ribonucleases T1 | Roche | R1003 | for RNAse treatment in DNAscope protocol |
| RNAscope 2.5, 2-plex detection reagent | ACD | 322430 | Brown and red kit chromogen detection |
| RNAscope Target Retrieval Reagents | ACD | 322000 | retrieval buffer |
| SuperFrost Plus Glass Slides | ThermoFisher Scientific | 12-550-17 | |
| TBS | BOSTON BIOPRODUCTS | BM-301-4L | for washes |
| TSA Plus Fluorescence palette kit (Cy3, Cy5, TMR, Fluorescein) | Perkin elmer | NEL760001KT | HRP Fluorescence detection |
| Tween 20 | SIGMA | P1379-1L | for washes |
| XYLENE 20LT | ThermoFisher Scientific | AC422680200 |
References
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnosis. 14 (1), 22-29 (2012).
- Deleage, C., et al. Defining HIV and SIV Reservoirs in Lymphoid Tissues. Pathogens and Immunity. 1 (1), 68-106 (2016).
- Sengupta, S., Siliciano, R. F. Targeting the Latent Reservoir for HIV-1. Immunity. 48 (5), 872-895 (2018).
- Churchill, M. J., Deeks, S. G., Margolis, D. M., Siliciano, R. F., Swanstrom, R. HIV reservoirs: what, where and how to target them. Nature Reviews Microbiology. 14 (1), 55-60 (2016).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), e2005970(2018).
- Deleage, C., Chan, C. N., Busman-Sahay, K., Estes, J. D. Next-generation in situ hybridization approaches to define and quantify HIV and SIV reservoirs in tissue microenvironments. Retrovirology. 15 (1), 4(2018).
- Deleage, C., Turkbey, B., Estes, J. D. Imaging lymphoid tissues in nonhuman primates to understand SIV pathogenesis and persistence. Current Opinion in Virology. 19, 77-84 (2016).
- Estes, J. D., et al. Defining total-body AIDS-virus burden with implications for curative strategies. Nature Medicine. 23 (11), 1271-1276 (2017).
- Mavigner, M., et al. Simian Immunodeficiency Virus Persistence in Cellular and Anatomic Reservoirs in Antiretroviral Therapy-Suppressed Infant Rhesus Macaques. Journal of Virology. 92 (18), (2018).
- Peterson, C. W., et al. Differential impact of transplantation on peripheral and tissue-associated viral reservoirs: Implications for HIV gene therapy. PLoS Pathogen. 14 (4), e1006956(2018).
- Deleage, C., et al. Impact of early cART in the gut during acute HIV infection. Journal of Clinical Investigation Insight. 1 (10), e87065(2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved