A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
קביעת יעילות מעכבי כימית נגד הצמיחה טוקסופלזמה Gondii באמצעות שיטת לוציפראז מבוססי צמיחה
In This Article
Summary
מוצג כאן הוא פרוטוקול להעריך את היעילות עכבות של תרכובות כימיות נגד הצמיחה מחוץ למוח טוקסופלזמה gondii באמצעות שיטת לוציפראז מבוססי צמיחה. הטכניקה משמשת כדי לאשר את העיכוב ספציפיות על ידי מחיקה גנטית של הגן היעד המתאים. עיכוב של LHVS נגד TgCPL פרוטאז מוערך כדוגמה.
Abstract
טוקסופלזמה gondii הוא מעין פתוגן שמשפיע רבות על אוכלוסיית האדם. האנטיביוטיקה הנוכחית המשמשת לטיפול בטוקסופלזמוזיס קלינית מוגבלת. בנוסף, הם מפגינים תופעות לוואי שליליות בקבוצות מסוימות של אנשים. לכן, גילוי של therapeutics הרומן עבור טוקסופלזמוזיס קליני הוא הכרחי. השלב הראשון של פיתוח אנטיביוטי הרומן הוא לזהות תרכובות כימיות מראה יעילות גבוהה בעיכוב של גידול טפיל באמצעות אסטרטגיית הקרנת תפוקה גבוהה. כפתוגן מחייב, טוקסופלזמה יכול רק לשכפל בתוך תאים מארחים, אשר אוסרת על שימוש במידות ספיגה אופטית כאינדיקטור מהיר לצמיחה. המוצג כאן הוא פרוטוקול מפורט עבור שיטת הצמיחה מבוססי לוציפראז. כדוגמה, שיטה זו משמשת כדי לחשב את זמן ההכפלה של מסוג פראי טוקסופלזמה טפילים ולמדוד את היעילות של מוראואינאוריאה-leucyl-הומוסקסיל-ויניל סולפון פניאיל (lhvs, מתחם המיקוד של ציטוקינים ציסטאין) לגבי עיכוב של הצמיחה האנטיתאיים הטפיל. מתואר גם, הוא מבוסס CRISPR-Cas9 מבוססי הגן הפרוטוקול בטוקסופלזמה באמצעות 50 bp הומולוגי אזורים עבור שילוב מחדש תלויי הומולוגיה (HDR). על ידי כימות efficacies העכבות של LHVS ב פראי-סוג ו Tgcpl (טוקסופלזמה צנתור psin L-כמו פרוטאז)-טפילים לקוי, זה מראה כי lhvs מעכב את הצמיחה פראי סוג טפיל ביעילות רבה יותר מאשר Δcpl צמיחה, הרומז כי TGCPL הוא מטרה כי lhvs נקשר ב טוקסופלזמה. רגישות גבוהה ותפעול קל של שיטת הגדילה הזאת לוציפראז מבוססי להפוך אותו מתאים ניטור טוקסופלזמה התפשטות והערכת יעילות הסמים באופן תפוקה גבוהה.
Introduction
טוקסופלזמה gondii הוא טפיל מצליח מאוד מאוד מחייב, אשר מדביק כ שליש מהאוכלוסייה האנושית. שיעור התמסורת הגבוה ברובו נובע מנתיבי שידור מגוונים, כולל צריכת בשר מבושל, חשיפה למאגרים ממיונקים ושידור מולד במהלך הלידה. T. gondii בעיקר גורם לזיהומים אופורטוניסטים שיכולים להוביל לתחלואה חמורה ותמותה באנשים בסכנה1,2,3,4,5,6. האנטיביוטיקה המשמשת כיום לטיפול בטוקסופלזמוזיס חריפה יעילה במיוחד לטיפול בזיהומים מולדים וסמויים ולגרום לתגובות חמורות אצל אנשים מסוימים3,7,8. לכן, יש צורך דחוף לזהות therapeutics הרומן קיים. הבנת ההבדלים בתהליכים תת-תאיים בתוך טוקסופלזמה והמחשב המארח שלה יסייע לזהות יעדי סמים פוטנציאליים. לכן, יעיל ונוח טכניקות מניפולציה הגנום נדרשים ללמוד את התפקידים של גנים בודדים בתוך טוקסופלזמה. בנוסף, הטוקסופלזמה שייך ל-מערכה apicomplexa, הכולל מספר פתוגנים אנושיים משמעותיים, כגון פלסטלינה והצפנה . מכאן, טוקסופלזמה יכול לשמש כאורגניזם מודל לעזור ללמוד ביולוגיה בסיסית של טפילים apicomplexan אחרים.
כדי לזהות אנטיביוטיקה הרומן נגד פתוגנים חיידקים, הקרנת תפוקה גבוהה של ספרייה של תרכובות כימיות מבוצעת בתחילה כדי לקבוע את יעילותה בדיכוי של גידול חיידקים. עד כה, מספר הצמיחה מבוססי מיקרופלייט מבוסס כבר פותחו למדידת צמיחה תאיים של T. gondii (כלומר, רדיואקטיבי 3H-uracil התאגדות המבוססת על כימות9, מבוססי אליסה מבוסס על זיהוי טפיל באמצעות T. gondii-נוגדנים ספציפיים10,11, כתבת חלבון מדידה באמצעות β-גלטוסידאז או yfp-הביע טוקסופלזמה זנים12,13, ו שפותחה לאחרונה תוכן גבוה הדמיה ה14
לכל האסטרטגיות הבודדות יש יתרונות ייחודיים; עם זאת, מגבלות מסוימות גם מגבילות את יישומותיהם. לדוגמה, מכיוון ש- טוקסופלזמה יכול לשכפל רק בתוך תאי בעלי חיים מסוימים, התאמה אוטומטית ומחייבת לא ספציפית של נוגדנים אנטי-T. gondii כדי לארח תאים גורמים להפרעות במידות המבוססות על קרינה פלואורסצנטית. יתר על כן, השימוש באיזוטופים רדיואקטיביים דורש תאימות בטיחות מיוחדת ובעיות בטיחות פוטנציאליות. חלק מהאספניות הללו מתאימות יותר להערכת הצמיחה בנקודת זמן יחידה ולא בניטור רציף של הצמיחה.
מוצג כאן הוא פרוטוקול לוציפראז מבוסס על כימות הצמיחה טוקסופלזמה תאיים. במחקר הקודם, הננו גן לוציפראז היה משוכפל תחת טוקסופלזמה טובולין יזם, ואת זה ביטוי לוציפראז לבנות היה מנוכר לתוך פראי סוג (RHΔku80Δhxg המתח) טפילים כדי ליצור RHΔKu80Δhxg::nluc זן (המכונה RHΔku80::nluc להלן)15. זן זה שימש את זן הורים לקביעת צמיחה תאיים ומחיקה גנטית במחקר זה. שימוש RHΔku80::nluc זן, הגידול הטפיל בפיברותקיעות העורלה האנושית (hffs) היה מנוטר מעל 96 h תקופה לאחר הדלקת כדי לחשב את זמן ההכפלה של טפיל.
בנוסף, את היעילות עיכוב של LHVS נגד הצמיחה טפיל יכול להיקבע על ידי התוויית טוקסופלזמה שיעורי גדילה נגד ריכוזי lhvs סדרתי כדי לזהות את הערך IC50 . הספרות הקודמות דיווחה כי tgcpl הוא מטרה מרכזית של lhvs בטפילים, כי הטיפול עם lhvs מקטין את ההתפתחות של זיהומים טוקסופלזמה כרוניים חריפה16,17,18,19. בנוסף, RHΔku80:: nluc שימש זן הורים לשינוי הגנום כדי ליצור מאמץ לקוי של tgcpl(RHΔku80Δcpl::nluc), ואת העיכוב של lhvs נמדד נגד מוטציה זו. על ידי התבוננות upshift של IC50 ערכים עבור LHVS ב tgcplלקויה טפילים לעומת זן WT, זה היה מאומת כי tgcpl הוא ממוקד על ידי lhvs ב vivo.
בפרוטוקול זה, RHΔku80::nluc משמש זן הורים, אשר חסר יעיל בלתי הומוולוגי מסלול להצטרף בסוף (nhej), ובכך להקל כפול מוצלב הומולוגיה התלויים שילוב מחדש (HDR)20,21. בנוסף, האזורים 50 bp הומולוגיים מקיפים בשני קצותיו של קלטת התנגדות לסמים על ידי ה-PCR. המוצר PCR משמש תבנית תיקון כדי להסיר את מקום הגן כולו באמצעות HDR באמצעות CRISPR-Cas9-מבוססי הגנום כלים לעריכת. ניתן לשלב בקלות את אזורי ההומוולוגי הקצרים האלה לתוך התחל, ולספק אסטרטגיה נוחה לייצור תבנית התיקון. פרוטוקול זה ניתן לשנות כדי לבצע מחיקה גנטית אוניברסלית תיוג גנים אנדוגני.
למשל, בפרסום האחרון שלנו, שלושה גנים הפרוטאז, Tgcpl, tgcpb (טוקסופלזמה צנתור psin B-כמו פרוטאז), ו TgSUB1 (טוקסופלזמה sub sin כמו פרוטאז 1), היו מבחינה גנטית בתוך tgcpl (טוקסופלזמה כלורוקין-התנגדות טרנספורטר)-טפילים לקוי בשיטה זו15. בנוסף, tgamn (הבנה n [tgamn, TGGT1_221310]) היה מתויג באופן שורש15. מעבדת Lourido דיווחו גם באמצעות אזורים הומולוגיים קצרים בטווח של 40-43 bp למבוא של מוטציה באתר מכוון גנים והתיוג הגן אנדודוגני בגנום טוקסופלזמה באמצעות שיטה דומה22. אלה שינויים הגנום מוצלחת מראים כי אזור bp הומולוגי 40-50 מספיק עבור DNA יעיל שילוב מחדש במתח TgKU80-לקוי, אשר מפשט מאוד את מניפולציה הגנום ב טוקסופלזמה gondii.
Protocol
טוקסופלזמה gondii מסווג בקבוצת סיכון 2 ויש לטפל ברמת בטיחות ביולוגית 2 (bsl-2). הפרוטוקול נבדק ואושר על ידי ועדת הביובטיחות המוסדית באוניברסיטת קלמסון.
1. לוציפראז מבוסס טוקסופלזמה הצמיחה המבוססת
- זרעי העורלה האנושית (HFFs) 1 שבוע לפני הטפיל חיסון כדי להבטיח כי תאים מארחים הם באופן מלא שוטפת. בצע שיטת מדמה בלוח שקוף כדי להבטיח שטפילים יישארו תאיים לאורך תקופת ההערכה.
הערה: הבקשה מתבצעת ב-96 מיקרולוחיות היטב. על פי צרכים ניסיוניים, זה יכול להיות מוקטן עד 384 או 1536 מיקרוצלחות היטב. - להעביר טוקסופלזמה טפילים לתוך hffs שוטפת 2 ימים לפני השימוש על ידי העברת ~ 0.3-0.4 מ ל של טפילים מלאים לגמרי לתוך בקבוקון T25. מודטה תאים מארחים נגועים ב 37 ° צ' עם 5% CO2 עבור 2 ימים.
- מזרק 5 מ ל של טפילים טריים באמצעות מחט בטיחות 21 G 5x כדי לשחרר טפילים תאיים, ואז לעבור דרך מסנן 3 יקרומטר כדי להסיר פסולת תא מארח. לשטוף טפילים שיורית מתוך הבקבוקון באמצעות 7 מ ל של פנול אדום חינם D10 בינוני, ואז לעבור את המסנן שוב.
- הטפילים צנטריפוגה ב 1000 x g עבור 10 דקות בטמפרטורת החדר (RT). יוצקים את supernatant ולהשעות מחדש את הגלולה ב 10 מ ל של פנול אדום חינם D10 מדיה.
- . כדי לקבוע את הריכוז
- לדלל טפילים 1 x 104 טפילים/mL עבור זן פראי (WT) מאמץ. לגידול זנים טפיל לקויה, להגדיל את הריכוז בהתאם להתבונן עלייה משמעותית אותות לוציפראז.
- מדידת מדיה בזהירות מ 96 מיקרופלטות היטב מקדם הזריעה עם hffs ו חסן 150 μl של טפיל מחדש לתוך בארות בפורמט של שלוש עמודות חמש שורות, אשר מייצגת שלושה משכפל טכני חמש זמן.
- מודקת את המיקרופלייט ב 37 ° צ' ו 5% CO2 עבור 4 h.
- מנושף מדיה בזהירות מן הבארות כדי להסיר טפילים שאינם פלשו, ולאחר מכן למלא את הבארות עם מדיה לחינם RT-האדום מתוך שורה (פרט לשורה הראשונה).
- מערבבים כמויות שוות של PBS ו 2x מאגר שיטת ללוציפראז ולדלל את המצע ללוציפראז כדי 12.5 μm.
- הוסף 100 μL של המצע מדלל לוציפראז לתוך כל טוב של השורה העליונה. מודאת המיקרופלטות ב-RT עבור 10 דקות כדי לאפשר את התאים כדי באופן מלא lyse.
- למדוד את הפעילות הלוציפראז באמצעות קורא מיקרופלייט. הגדרות קורא הצלחות מפורטות בטבלה 1. כל קריאה מייצגת את המספר הראשוני. של טפילים במשך 4 שעות לאחר ההדבקה
- חזור על שלבים 1.9-1.12 עבור כל שורה כל 24 שעות במשך 4 ימים מבלי לשנות את המדיום. קריאות אלה משקפות את המספר הכולל של טפילים משוכפלים ב 24 h, 48 h, 72 h, ו 96 h לאחר ההדבקה.
- לחשב את הקריאות הממוצעת בכל זמן ולחלק אותם על ידי הקריאות הממוצעת ב 4 h כדי לקבוע את שינויי הקיפול בגידול טפיל לאורך זמן.
- התווה את הנתונים באמצעות תוכנת גרפים. שולחן קריאה הצמיחה הנציג ומגרשים של RHΔku80::nluc טפילים מוצגים באיור 1א, ב.
- כדי לחשב את זמן ההכפלה, התווה את ערכי log2 של שינויי קיפול בנקודות הזמן הבודדות על-פני תקופת הדגירה. השתמש בפונקציית רגרסיה ליניארית כדי לחשב מדרון, המייצג את זמן ההכפלה של כל זן (איור 1א, ג).
2. הערכה של יעילות מתחם כימי עכבות נגד טוקסופלזמה צמיחה
הערה: כאן, הערכה של עיכוב של LHVS ב טוקסופלזמה צמיחה מוצגת כדוגמה. שמונה ריכוזים שונים של LHVS נבדקים, ושלושה משכפל טכני מבוצעים עבור כל אחד שלוש משכפל ביולוגי עבור שני RHΔku80::NLUC ו RHΔKu80Δcpl::nluc זנים.
- לפני הידבקות הטפיל, הזרע HFFs כדי 96 מיקרופלטות היטב בפורמט של שלוש שורות ותשע עמודות עבור שכפול ביולוגי אחד לכל משטח לכל זן. תאים מארחים יורשו לגדול לפחות 7 ימים לפני השימוש.
- לעבור RHΔku80:: NLUC ו RHΔKu80Δcpl::nluc טפילים עבור 2 ימים לפני השימוש. בצע את שלבים 1.2-1.6 עבור טיהור טפיל וכימות. השעיה מחדש של טפילים ב-פנול מדיה ללא אדום ב 1 x 104 טפילים/mL.
- מתיף מדיה מלוחית המכילה את הטיפקס confluent ומחסן כל טוב עם 150 μL של טפיל מחדש. מודקת את המיקרופלייט ב 37 ° צ' ו 5% CO2 עבור 4 h.
- הכינו LHVS בשמונה ריכוזים שונים ב 12 מאגר היטב על ידי דילול סדרתי. באופן כללי, הריכוזים מופחתו באמצעות שלוש מקפלים בצורת דילול טורית.
הערה: הריכוז הנמוך ביותר מופחת על ידי 6,561 מתקפל ביחס לריכוז הגבוה ביותר. השינוי הקיפול של הדילול ניתן להתאמה בהתאם למאפיינים שונים של תרכובות בודדות. - ב 4 h לאחר ההדבקה, מתוך מדיה להסיר טפילים שאינם פלשו ולמלא כל היטב מתוך עמודות 2-9 עם 150 μL של מדיה בתוספת LHVS בריכוזים שונים. השאר את העמודה הראשונה מלאה במדיום רגיל כדי לשמש כפקד שאינו מטופל.
- מודקת את המיקרופלייט ב 37 ° צ' ו 5% CO2 עבור 96 h נוספים.
- בצע את שלבים 1.9-1.11 ולמדוד פעילות הלוציפראז של בארות בודדות.
- ממוצע הפעילויות הלוציפראז של שלושה משכפל טכני מבארות של כל הריכוז LHVS של כל אדם.
- לחלק את הפעילות לוציפראז הממוצע עבור כל הריכוז LHVS על ידי הפעילות לוציפראז הממוצע נגזר טפילים שאינם מטופלים כדי לחשב את פעילות הלוציפראז מנורמלת כאחוז.
- התווה את הפעילות הלוציפרתית המנורמלת נגד ריכוזי LHVS הבודדים באמצעות תוכנת גרפים (איור 2). עיכוב פירימתמין נגד הצמיחה טפיל נמדד גם כפקד. פירומתמין הוא אנטיביוטיקה קלינית המשמשת לטיפול טוקסופלזמוזיס חריפה על ידי עיכוב מטבוליזם חומצה פולית ב טוקסופלזמה.
- חשב את ערכי ה-IC50 עבור תרכובות בודדות באמצעות השיטה המוטבעת בתוכנה הגראפית, תגובה מנורמלת לעומת [מעכב], תחת תוכנית הרגרסיה "מינון-תגובה". ה-IC50 מחושב באמצעות הנוסחה הבאה:
Y = 100/(1 + X/IC50)
כאשר: Y מייצג את הפעילות לוציפראז מנורמל של תאים נגועים תחת ריכוזים שונים של מעכב, ו-X מייצג ריכוזים בודדים של מעכב.
3. CRISPR-Cas9 מבוסס גנים מחיקה ב טוקסופלזמה טפילים
- דור של בניית פלסמאמצע ביטוי מדריך RNA (sgRNA) ו Cas9 למחיקת גן של עניין
- עבור אל www.ToxoDB.org ולאחזר את רצף קידוד הגנים כולו, כולל introns ו exons, יחד עם 1.5 kb 5 '-utrs ו 3 '-utrs (אזורים בלתי מתורגמים).
הערה: כאן, Tgcpl (TGGT1_321530) מיועדת כדוגמה מייצגת. - העתק את רצף הבקרה של tgcpl לתוכנת ניתוח הרצף (עיין בטבלה של חומרים עבור השם והגירסה) וסמן את האזורים 5 '-ו-3 '-utr.
- בחר את סמל הכלים בשורת התפריטים העליונה ולאחר מכן בחר בשכפול | מצא את אתרי CRISPR.
- בחר 3 ' (Cas9) ' עבור מיקום האתר פם ובחר את התיקייה המכילה את רצף הגנום טוקסופלזמה בסעיף הניקוד ספציפיות. השאר את שאר ההגדרות כברירות מחדל.
- בחר sgRNA עם שני הקריטריונים הבאים: 1) מראה ציון ספציפיות גבוה, בדרך כלל > 98%, ו 2) חסר G לאחר NGG, מוטיב הסמוך ביותר (פאם) רצף. SgRNA שנבחר ממוקם בדרך כלל באתרים קרוב ההתחלה ולהפסיק המאתרים של הגן של עניין.
- העתק את הרצף של sgRNA שנבחר והדבק אותו בתבנית התחל הבאה.

החלק באדום מייצג את TgCPL רצף ה-tgsgrna שנבחר. זה יכול להיות מוחלף sgRNAs שונים עבור גנים שונים של עניין.
הערה: אם sgRNA שנבחר אינו מתחיל ב-G, הוסף G בתחילת sgRNA כדי לסייע בשיפור הביטוי שלו. - לבצע תגובת PCR כדי לשנות את הקיים מראש ביטוי הפלסטיות sgrna (איור 3a) כי מטרות טוקסופלזמה אורציל פוספוריםבטברואז (tguprt) גן23 באמצעות ה-PCR הקדמה עם ההגדרות המסופקות בטבלה 2.
- הפעל את מוצר ה-PCR על ג'ל שנוצר כדי לאשר הגברה מוצלחת. מוצר ה-PCR של 10 kb צפוי להיות מוגבר (איור 3ב).
- חלץ את מוצר ה-PCR באמצעות ערכת הפקת ג'ל לבדיקת דנ א ומעגליות באמצעות ערכת מוטזיס מכוונת-אתר. התייחס לטבלה 3 עבור המתכון. דגירה התגובה עבור 10-20 דקות ב RT.
- הפוך את המוצר PCR מעגלי לתוך E. coli ולבחור 10 שיבוטים לאימות נוסף של שילוב sgRNA מעוצב.
- לגדל שני שיבוטים ולחלץ פלמידים. חתוך את הפלטידים המטוהרים. עם באמהי ואכרוב פלמידים מועמדים תניב שתי להקות ב 2.4 kb ו 7.2 kb (איור 3ג).
- שלח את הפלמידים עבור רצפי הרצף באמצעות M13 הפוך התחל כדי לאשר החלפה מוצלחת של Tguprt sgrna עם sgrna תוכנן (איור 3ד).
- עבור אל www.ToxoDB.org ולאחזר את רצף קידוד הגנים כולו, כולל introns ו exons, יחד עם 1.5 kb 5 '-utrs ו 3 '-utrs (אזורים בלתי מתורגמים).
- יצירת תבנית תיקון למחיקה גנטית באמצעות מנגנון HDR
- לדברי המיקוד אתרים של sgRNA שנבחרו, לאתר 50 bp של 5 '-UTRs או 3 '-UTRs של הגן היעד עבור שילוב מחדש תלויי הומולוגיה (HDR, ראה סעיף דיונים). בחירת האזורים עוקבת אחר הקריטריונים המפורטים להלן, בהתאם למיקום היעדים sgRNA.
- אם באתר המחשוף על ידי Cas9 ממוקם במעלה הזרם מן ההתחלה קודון, בחר את הבאים: 50 bp רצף ה-dna במעלה מאתר המחשוף כמו אזור hdr השמאלי, ו-50 רצף dna bp במורד מן העצירה קודון כמו אזור hdr הימני.
- אם באתר המחשוף על ידי Cas9 הוא בין ההתחלה ולהפסיק הקשר, לבחור את הבאים: 50 bp רצף DNA במעלה מן ההתחלה codons כמו האזור HDR השמאלי, ו-50 רצף DNA bp במורד מן לעצור codons כמו אזור HDR הימני.
- אם באתר המחשוף על ידי Cas9 ממוקם במורד הזרם מן קודון לעצור, בחר את הבאים: 50 bp רצף dna במעלה מן ההתחלה קודון כמו האזור hdr השמאלי, ו-50 רצף dna bp במורד האתר המחשוף כמו באזור hdr הימני.
הערה: האתר ממוקם בין ההתחלה והפסקת הצליל של האינטרנט . לפיכך, התחל הבא נועד להגברה של תבנית התיקון באמצעות pMDC64 כתבנית, אשר מקודד את הקלטת ההתנגדות פירימתמין. הרצפים שחורים לספח pMDC64 באמצע עבור הגברה PCR. האזורים המסומנים באדום הם Tgcplרצפים ספציפיים לשילוב מחדש של הומוולוגי.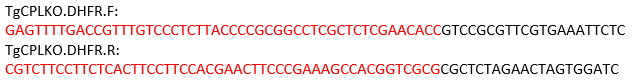
- לבצע PCR באמצעות ה-pcr בתנאים המתוארים בטבלה 4.
- הפעל את המוצר ה-PCR על ג'ל agarose (איור 3E), ואחריו חילוץ ג'ל והליכים כלפי תקן חומצות גרעין.
הערה: אם הלהקה הצפויה לא יכולה להיות מוגברת בהצלחה, מטב את תנאי ה-PCR ו/או החלפת זוגות.
- לדברי המיקוד אתרים של sgRNA שנבחרו, לאתר 50 bp של 5 '-UTRs או 3 '-UTRs של הגן היעד עבור שילוב מחדש תלויי הומולוגיה (HDR, ראה סעיף דיונים). בחירת האזורים עוקבת אחר הקריטריונים המפורטים להלן, בהתאם למיקום היעדים sgRNA.
- טוקסופלזמה
- לעבור RHΔku80::nluc טפילים עבור 2 ימים בבקבוקון T25 המכיל hffs confluent. בקבוקון T25 של טפילים מלאים. מספיק בשביל שניים עד שלוש העברה
- מזרק ומסננים לטהר טפילים כפי שמתואר בשלב 1.2. השעיה מחדש של טפילים במאגר cytomix ו ספין למטה ב 1,000 x g עבור 10 דקות ב-RT.
- לשטוף טפילים הפלטים עם 10 מ ל של מאגר cytomix ולסובב את הטפילים ב 1,000 x g עבור 10 דקות ב-RT.
- שופכים בזהירות את supernatant ולהשעות את הטפילים באותו מאגר בריכוז של 1 x 108 טפילים/mL.
- מערבבים 2 μg של תקן ה-DNA של תבנית תיקון עם 20 μg של sgRNA/Cas9 ביטוי פלמידים (יחס המוני = 1:5, שווה ערך ל 1:3 היחס הטוחנת). אם תפוקת הגברה של תבנית התיקון נמוכה, הפחת את הקלט של שתי חתיכות ה-DNA בהתאם. מינימום של 0.5 μg של תבנית תיקון ניתן להשתמש.
- מערבבים 400 μL של טפיל מחדש, דנ א ו-5 μL של 200 mM ATP/500 מ"מ גלוטתיון מופחת (GSH) בשפופרת צנטריפוגה 1.5 mL. הביאו את אמצעי האחסון הכולל ל-500 μL עם מאגר cytomix, במידת הצורך.
- להעביר את התערובת של טפילים DNA כדי קובט אלקטרופורציה (4 מ"מ רוחב הפער) ולבצע אלקטרופורציה (2 kV מתח, 50 Ω התנגדות) באמצעות מנגנון אלקטרופורציה.
- העברת טפילים חשמליים לבקבוקון T25 המכיל הטיפקס במדיום D10 טרי. החלת אנטיביוטיקה הולמת לבחירת סמים לאחר 24 שעות.
- לשמור על לחץ בררני לסמים עד שצמיחת הטפילים הטרנסגניים יציבה.
- לטהר את ה-DNA גנומית מאוכלוסיית הנוקאאוט ולבדוק שילוב של הקלטת ההתנגדות פירימתמין לתוך tgcpl לוקוס ידי ה-PCR. לאחר האימות, המשך לסעיף 3.4. אם לא, בצעו סבב נוסף של. מעבר טפיל ובחירת סמים חוסר יכולת לזהות את השילוב הנכון של הקלטת התנגדות לסמים מרמזת בדרך כלל כי הגן היעד חיוני או שאין גישה לוקוס הגן.
- שיבוט של טפילים הנוקאאוט
- זרעי 2 96 היטב microplates עם תאים HFF ו-דגירה ב 37 ° צ' ו 5% CO2 במשך 1 שבוע לפני טפילים שיבוט.
- מעבר ~ 0.3-0.4 מ ל מאוכלוסיית הטפילים הטרנסגניים בבקבוקון T25 המכיל את הטיפקס ומגדלים אותם במשך יומיים. שקול להעביר טפילים נוספים. אם המוטציה מראה פגמים בגדילה
הערה: כדי להשיג את התשואה הטובה ביותר ואת הכדאיות, התאים המארחים הם נגועים בכבדות על ידי טפילים, ורוב טפילים נשמרים בשלב תאיים. - מזרק הנגועים בתאים מארחים ומסננים לטהר טפילים טריים כפי שהוזכר בשלב 1.3. להשעות את הטפילים ב D10 בינוני ולסובב אותם למטה ב 1,000 x g עבור 10 דקות ב-RT.
- להשעות את הטפילים הפלטים ב -10 מ ל של D10 בינונית.
- לספור טפילים באמצעות הומוציטוטומטר כדי לקבוע את הריכוז הטפיל.
- לבצע דילול שני צעדים כדי להביא את הריכוז 10 טפילים/mL ב D10 בינוני בתוספת עם אנטיביוטיקה המתאים. בדרך כלל, הטפיל הראשונית מחדש מדולל על ידי 1,000-קיפול, ואחריו דילול שני ל 10 טפילים/mL.
- מכילים מדיה מ96 היטב מיקרופלטות המכילות hffs ו-חסן 150 μl של טפילים מדולל לתוך כל באר.
- הצלחות דגירה ב 37 ° צ' עם 5% CO2 עבור 7 ימים ללא הפרעה כדי לאפשר היווצרות פלאק. תקופת הדגירה יכולה להיות ארוכה יותר אם טפילים טרנסגניים מציגים פגמים בגדילה.
- המסך את הצלחות באמצעות מיקרוסקופ שלב-ניגודיות וסמן רק את הבארות המכילות לוח יחיד.
- לבצע את ה-PCR המושבה כדי לזהות שיבוטים נכונים.
- השימוש עצות הפיפטה כדי לגרד את החלק התחתון של כל באר להרים נגועים HFF monolayers.
- Pipet 75 μL של התא מחדש מכל אחד מסומן היטב לתוך 1.5 מיקרוצנטריפוגה שפופרות.
- צינורות צנטריפוגה עבור 10 דקות במהירות מקסימלית ב RT. בזהירות ומתה את supernatant ולהשעות את הגלולה ב 10.25 μL של מאגר הליזה המכיל מאגר דילול ותוסף שחרור ה-DNA המסופק בערכה (טבלת חומרים).
- דגירה את הדגימות עבור 4 דקות ב RT, ואז 2 דקות ב 98 ° c. לאחר מכן, ניתן להשתמש בדגימות ל-PCR או לאחסנם ב-20 ° c עד לשימוש. שלושה סטים של תגובות ה-PCR משמשים לבדיקת השילוב של הקלטת התנגדות לסמים ואובדן גן העניין (איור 4א). פנה לטבלה 5 להגדרת התגובה של ה-PCR וללוח 6 עבור הגדרות התרמוטרטרנר.
- לזהות את המשובטים הנכונים ולהעביר ארבעה שיבוטים לתוך T25 צלוחיות המכילות HFFs confluent.
- לאחר שיבוטים בודדים lyse תאים מארחים, טיהור DNA גנומית עבור אימות PCR נוסף.
- אם נוגדן מזהה את חלבון הריבית זמין, בצע את ההליך החיסוני הסטנדרטי כדי לוודא אובדן של חלבון היעד ב טוקסופלזמה outs הנכון. תמונות מייצגות להקרנה מוטציה המחיקה Tgcplמופיעים באיור 4ב, ג.
תוצאות
איור 1 מייצג דוגמה לעקומת גדילה עבור RHΔKu80::nluc זן והחישוב הנגזר עבור זמן ההכפלה שלו. באופן כללי, המנה מבוצעת בשלוש משכפל טכני עבור כל אחד משלושת המשכפלת הביולוגית לחשבון עבור וריאציות של קריאות פעילות לוציפראז. על מנת לחשב את שינוי קיפול מנורמל של ...
Discussion
+ + פרוטוקול זה מתאר פרוטוקול לוציפראז מבוסס להערכת צמיחה טוקסופלזמה תאיים ולהעריך את היעילות עיכוב של תרכובות כימיות נגד גידול טפיל. בהשוואה אסטרטגיות קיימות זמין למדידת צמיחה תאיים טוקסופלזמה , שיטה זו מציגה רגישות גבוהה וספציפיות. בעת ניטור הצמיחה טפיל, שינון מבוים ברור 96 היט?...
Disclosures
. למחברים אין מה לגלות
Acknowledgements
המחברים רוצים להודות לד. סיבלי וקארותרס לשיתוף pSAG1-Cas9-sgRNA-TgUPRT פלאמיד ואנטי-TgCPL ו-TgActin נוגדנים. עבודה זו נתמכת על ידי קרן ההפעלה קלמסון (כדי Z.D.), אבירים הטמפלרים עין קרן רפואת עיניים הקריירה-Starter מחקר מענק (כדי Z.D.), מענק הטייס של מענק NIH COBRE P20GM109094 (כדי Z.D.), ו-NIH R01AI143707 (כדי Z.D.). לתורמים לא היה כל תפקיד בתכנון לימוד, איסוף נתונים וניתוח, החלטה לפרסם או הכנת כתב היד.
Materials
| Name | Company | Catalog Number | Comments |
| Agarose gel extraction kit | New England BioLabs | T1020L | |
| BamHI | New England BioLabs | R0316S | |
| Biotek Synergy H1 Hybrid Multi-Mode Microplate Reader | BioTek Instuments | ||
| BTX Gemini Twin Waveform Electroporation System | Harvard Apparatus | ||
| Chemically competent E. coli cells | New England BioLabs | C29871 | |
| CloneAmp HiFi PCR premix | Takara Bio | 639298 | |
| Coelenterazine h | Prolume | 301-10 hCTZ | |
| EcoRV | New England BioLabs | R3195S | |
| Phire Tissue Direct PCR Master Mix | Thermo Scientific | F170L | |
| Plasmid miniprep kit | Zymo Research | D4054 | |
| Q5 Site-Directed Mutagenesis kit | New England BioLabs | E0554S | |
| Software | |||
| Geneious software for sgRNA design (version: R11) | |||
| GraphPad Prism software (8th version) | |||
| SnapGene for molecular cloning (version: 4.2.11) |
References
- Blader, I. J., Coleman, B. I., Chen, C. T., Gubbels, M. J. Lytic Cycle of Toxoplasma gondii: 15 Years Later. Annual Review of Microbiology. 69 (1), 1-23 (2014).
- Jones, J. L., Kruszon-Moran, D., Rivera, H., Price, C., Wilkins, P. P. Toxoplasma gondii Seroprevalence in the United States 2009-2010 and Comparison with the Past Two Decades. The American Journal of Tropical Medicine and Hygiene. 90 (6), (2014).
- Kieffer, F., Wallon, M. Congenital toxoplasmosis. Handbook of Clinical Neurology. 112, 1099-1101 (2013).
- Hoffmann, S., Batz, M. B., Morris, G. J. Annual cost of illness and quality-adjusted life year losses in the United States due to 14 foodborne pathogens. Journal of Food Protection. 75 (7), 1292-1302 (2012).
- Dubey, J. Toxoplasmosis. Journal of the American Veterinary Medical Association. 205 (11), 1593-1598 (1994).
- Lindsay, D., Dubey, J. Toxoplasma gondii: the changing paradigm of congenital toxoplasmosis. Parasitology. 138 (14), 1-3 (2011).
- Deng, Y., Wu, T., Zhai, S., Li, C. Recent progress on anti-Toxoplasma drugs discovery: Design, synthesis and screening. European Journal of Medicinal Chemistry. 183, 111711 (2019).
- Butler, N. J., Furtado, J. M., Winthrop, K. L., Smith, J. R. Ocular toxoplasmosis II: clinical features, pathology and management. Clinical & Experimental Ophthalmology. 41 (1), 95-108 (2013).
- Pfefferko, E., Pfefferko, L. C. Specific Labeling of Intracellular Toxoplasma gondii with Uracil. Journal of Eukaryotic Microbiology. 24 (3), 449-453 (1977).
- Merli, A., Canessa, A., Melioli, G. Enzyme immunoassay for evaluation of Toxoplasma gondii growth in tissue culture. Journal of Clinical Microbiology. 21 (1), 88-91 (1985).
- Derouin, F., Chastang, C. Enzyme immunoassay to assess effect of antimicrobial agents on Toxoplasma gondii in tissue culture. Antimicrobial Agents and Chemotherapy. 32 (3), 303-307 (1988).
- McFadden, D., Seeber, F., Boothroyd, J. Use of Toxoplasma gondii expressing beta-galactosidase for colorimetric assessment of drug activity in vitro. Antimicrobial Agents and Chemotherapy. 41 (9), 1849-1853 (1997).
- Gubbels, M. J., Li, C., Striepen, B. High-Throughput Growth Assay for Toxoplasma gondii Using Yellow Fluorescent Protein. Antimicrobial Agents and Chemotherapy. 47 (1), 309-316 (2003).
- Touquet, B., et al. High-content imaging assay to evaluate Toxoplasma gondii infection and proliferation: A multiparametric assay to screen new compounds. PLoS ONE. 13 (8), e0201678 (2018).
- Thornton, L. B., et al. An ortholog of Plasmodium falciparum chloroquine resistance transporter (PfCRT) plays a key role in maintaining the integrity of the endolysosomal system in Toxoplasma gondii to facilitate host invasion. PLOS Pathogens. 15 (6), e1007775 (2019).
- Larson, E. T., et al. Toxoplasma gondii cathepsin L is the primary target of the invasion-inhibitory compound morpholinurea-leucyl-homophenyl-vinyl sulfone phenyl. The Journal of Biological Chemistry. 284 (39), 26839-26850 (2009).
- Dou, Z., McGovern, O. L., Cristina, M., Carruthers, V. B. Toxoplasma gondii Ingests and Digests Host Cytosolic Proteins. mBio. 5 (4), e01188-14 (2014).
- Cristina, M., et al. Toxoplasma depends on lysosomal consumption of autophagosomes for persistent infection. Nature Microbiology. 2, 17096 (2017).
- Parussini, F., Coppens, I., Shah, P. P., Diamond, S. L., Carruthers, V. B. Cathepsin L occupies a vacuolar compartment and is a protein maturase within the endo/exocytic system of Toxoplasma gondii. Molecular Microbiology. 76 (6), 1340-1357 (2010).
- Huynh, M. H., Carruthers, V. B. Tagging of endogenous genes in a Toxoplasma gondii strain lacking Ku80. Eukaryotic cell. 8 (4), 530-539 (2009).
- Fox, B. A., Ristuccia, J. G., Gigley, J. P., Bzik, D. J. Efficient gene replacements in Toxoplasma gondii strains deficient for nonhomologous end joining. Eukaryotic Cell. 8 (4), 520-529 (2009).
- Sidik, S. M., Hackett, C. G., Tran, F., Westwood, N. J., Lourido, S. Efficient Genome Engineering of Toxoplasma gondii Using CRISPR/Cas9. PLoS ONE. 9 (6), e100450 (2014).
- Shen, B., Brown, K. M., Lee, T. D., Sibley, D. L. Efficient Gene Disruption in Diverse Strains of Toxoplasma gondii Using CRISPR/CAS9. mBio. 5 (3), e01114-14 (2014).
- Radke, J. R., et al. Defining the cell cycle for the tachyzoite stage of Toxoplasma gondii. Molecular and Biochemical Parasitology. 115 (2), 165-175 (2001).
- Ran, A. F., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B., Valen, E. CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Research. 44 (W1), W272-W276 (2016).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 2812 (2014).
- Peng, D., Tarleton, R. EuPaGDT: a web tool tailored to design CRISPR guide RNAs for eukaryotic pathogens. Microbial Genomics. 1 (4), e000033 (2015).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature Biotechnology. 32 (12), 1262-1267 (2014).
- Sidik, S. M., et al. A Genome-wide CRISPR Screen in Toxoplasma Identifies Essential Apicomplexan Genes. Cell. 166 (6), 1423-1435 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved